Prácticas integradas de Ecología molecular microbiana
|
|
|
- Elisa Castro Mendoza
- hace 8 años
- Vistas:
Transcripción
1 Prácticas integradas de Ecología molecular microbiana Indice. Página 1. Introducción 1 2. Cronograma 1 3. Esquema de trabajo 2 4. Reactivos utilizados 3 5. Preparación de las Muestras 3 6. Hibridación in situ con sondas fluorescentes (FISH) 5 7. Extracción del ADN 7 8. Amplificación por PCR 8 9. Electroforesis en gel con gradiente desnaturalizante (DGGE) Amplificación del gen del ARNr 16S para la construcción de genotecas 16
2 1. Introducción Las técnicas de microbiología tradicionales requieren la obtención de cultivos puros (lo que no siempre es posible) y múltiples ensayos de laboratorio, por lo que son poco adecuadas para estudiar la biodiversidad de un ecosistema. Durante la década de los 90 se desarrollaron una serie de técnicas de biología molecular que han supuesto una revolución en el campo de la Ecología microbiana. Dichas técnicas, basadas en el 16S rrna, permiten estudiar la biodiversidad microbiana de un ecosistema sin necesidad de proceder al aislamiento de las especies que lo integran. En estas prácticas vamos a emplear tres de las principales técnicas aplicables a estudios de ecología molecular microbiana: la electroforesis en gel con gradiente desnaturalizante (DGGE/TGGE), la hibridación in situ con sondas de DNA marcadas con un cromóforo fluorescente (FISH), y la construcción y rastreo de genotecas. Se trabajará con muestras naturales (suelo) y artificiales: flóculos de la depuradora de fangos activos de la UAM (aerobia) y biomasa (lodo granular) un reactor UASB (anaerobio). 2. Cronograma Este grupo de prácticas se estructura en cinco sesiones intensivas de laboratorio (4-6 horas de duración), más una sesión teórica previa y una sesión de análisis de resultados posterior. 1ª Sesión. Fijación de las muestras para FISH (lo harán los monitores por la mañana) Preparación de las muestras Extracción y limpieza del DNA Análisis del DNA en gel de agarosa. PCR gen 16S rrna utilizando cebadores universales para los dominios Bacteria (para genoteca) y Archaea (para PCR anidada). Dejar overnight. Lavado de las muestras para FISH. 2ª Sesión PCR utilizando cebadores universales para los dominios Bacteria y Archaea (para DGGE, fragmento del gen 16S rrna). Si es posible, hacer PCR directa y anillada. Preparar y correr gel de agarosa para analizar productos PCR de la primera sesión. Preparar geles de agarosa para analizar la PCR para DGGE. FISH: observar mediante contraste de fases o tras tinción con DAPI para ver dilución adecuada para conteo. 1
3 Clonación: ligación para hacer una genoteca. Dejar overnignt. 3ª Sesión Preparar las placas LB Clonación: transformación y plaqueo en placas LB-Amp Preparar gel DGGE Correr gel de agarosa para analizar la PCR para DGGE FISH: fijar las muestras en portas. Recoger y guardar placas genoteca 4ª Sesión FISH: hibridación y lavado Picar colonias genoteca en placa maestra. Cálculo eficiencia clonación. Correr DGGE (o.n.) 5º Sesión Revelar y ver DGGE Observación revertientes Análisis microscópico (cuantificación) de las hibridaciones del FISH. 3. Esquema de trabajo. MUESTRA Extracción de ADN Hibridación in situ PCR DGGE Genoteca 2
4 4. Reactivos utilizados. 29-HindIII ADN 4,6-diamino-2-fenol-dihidrocloruro (DAPI) Acrilamida / bisacrilamida (40%) (37,5:1) Acido clorhídrico Acido bórico Agarosa Bromuro de etidio Citifluor AF-1 Cloruro sódico dntps Dodecilsulfato sódico (SDS) EcoRI Etanol Formaldehído Formamida Sau3AI Taq-polimerasa Tritiplex III (EDTA) Tris-Trizma Base Sondas FISH: ver tabla. Cebadores 341f-GC, 622fGC, 907r, 1492r, 27f, 948f, 1492r Kit Fast DNA kit for soil BIO101 Kit Microcon-Centrifugal filter devices Kit pgemt-easy vector Ampicilina Células competentes Agar X-gal CBM MERCK BIORAD MERCK GIBCO-BRL SIGMA SIGMA CITIFLUOR Ltd. MERCK PHARMACIA MERCK BIOLABS MERCK MERCK PHARMACIA BIOLABS PROMEGA /BIOTOOLS MERCK SIGMA BIOTOOLS/ TAPER QUIAGEN MILLIPORE PROMEGA BRITEPEN CBM PRONADISA DUCHEFOR 5. Preparación de las muestras. Las muestras serán preparadas según sus características físicas y su procedencia. Puesto que las muestras para hacer FISH deben fijarse en el momento de su recogida, los procedimientos descritos a continuación se realizarán con dos alícuotas independientes: una fijada en formaldehído (para hacer FISH, protocolo 6) y otra sin fijar (para extraer DNA, protocolo 7). Muestra de suelo o lodo de depuradora. Para extracción de DNA: 1. Pesar aproximadamente 0,5 2 g, resuspender en 5 ml de NaCl 0,9% 2. Disgregar por sonicación (potencia media 0.50 w, frecuencia constante, durante 3 minutos). Agitar la muestra en el vortex durante 10 minutos. 3. Dar un pulso de centrífuga para eliminar el material inerte grueso y quedarse con el sobrenadante. 3
5 4. Centrifugar éste a 5000 rpm durante 10 minutos y tomar el pellet (desechar el sobrenadante). 5. Resuspender el pellet en 1000 l de PBS 1X 1. Guardar a 4 C. Para FISH: Las muestras se entregarán fijadas. Lo más habitual es hacerlo con formaldehído (el concentrado está al 37%) a una concentración final del 4% (v/v) en PBS. El tiempo de fijación suele variar entre 2 y 16 horas a 4 C. 1. Centrifugar la suspensión en formaldehído durante 5 minutos a r.p.m. Resuspender el pellet en 1000 l de PBS. 2. Repetir el lavado con PBS. Centrifugar 7 minutos a r.p.m. 3. Resuspender el pellet en PBS o NaCl y continuar con los pasos 2-4 del protocolo anterior. 4. Resuspender el pellet resultante en 1000 l en Etanol: PBS (1:1). Conservar a -20 C. Lodo granular. Para la extracción de DNA. Tomar 2-5 gránulos, resuspenderlos en 1 ml de PBS 1X y disgregar por sonicación (potencia media 0.50 w, frecuencia constante, durante 30 segundos) o con un potter. Guardar a 4 C. Para FISH 1. Decantar el formaldehído y lavar abundantemente con PBS. 2. Repetir el lavado con PBS para eliminar el formaldehído externo a los gránulos. 3. Pesar aproximadamente 0.05 g (5-10 gránulos), resuspender en 1 ml de PBS y disgregar por sonicación (potencia media 0.50 w, frecuencia constante, durante 30 segundos). Puede requerirse repetir el proceso para romper y disgregar bien los gránulos. Alternativamente, los gránulos pueden disgregarse con un potter y un vortex. 4. Centrifugar a baja velocidad (3.000 rpm, 1 minuto) para eliminar los restos de gránulos. 5. Descartar el pellet y centrifugar el sobrenadante durante 5-7 minutos a rpm. 6. Resuspender el pellet en 1 ml de PBS. 7. Centrifugar 5-7 min a rpm. 8. Resuspender el pellet resultante en 1 ml de la mezcla PBS:etanol (1:1). Conservar a -20 ºC hasta su utilización. PBS (buffer fosfato sódico): 130 mm NaCl 10 mm Na 2 HPO 4 / NaH 2 PO 4 Ajustar el ph a 7.2 Esterilizar en autoclave. 4
6 6. Hibridación in situ con sondas fluorescentes (FISH). El FISH permite identificar in situ la presencia de microorganismos al nivel taxonómico que se requiera. Las sondas son oligonucleótidos de DNA (16-20 nucleótidos) marcadas en su extremo 5 con un cromóforo fluorescente cuya secuencia es complementaria de una región del 16S rrna. Debido a su pequeño tamaño, son capaces de penetrar en las células fijadas, hibridando (apareamiento DNA-RNA) in situ con los ribosomas de las células completas, generando una señal fluorescente que permite su identificación, localización y posterior cuantificación. La especificidad de la sonda utilizada, que es función de la conservación de la secuencia de rrna seleccionada, nos permite detectar/identificar al nivel taxonómico que deseemos: desde dominio hasta especie. El proceso de hibridación in situ se divide en cuatro etapas: i) toma de muestra y fijación en el momento de la recogida para preservar la integridad celular y, especialmente, de sus ribosomas; ii) hibridación con una sonda específica marcada con un fluorocromo; iii) tinción de contraste con un marcador universal (DAPI, que se une al DNA de cualquier célula) u otra sonda más general marcada con otro fluorocromo de distinto color; iv) visualización mediante un microscopio de fluorescencia. La cuantificación puede hacerse mediante contaje directo en el microscopio (epifluorescencia o láser confocal), análisis de imágenes (tratamiento de las imágenes tomadas con cámara CCD o microscopio láser confocal) o citometría de flujo. La relación entre las células hibridadas con la sonda específica de grupo y las teñidas con DAPI o hibridadas con la sonda general nos dará el porcentaje de nuestras bacterias en la muestra con respecto al número total de bacterias presentes o con respecto al grupo general cuya sonda hayamos utilizado (v.g. bacterias o arqueas). Es la única técnica cuantitativa, aunque el proceso puede ser tedioso y subjetivo (microscopio de epifluoroscencia) o muy complejo (microscopio láser confocal). Permite estudiar la estructura de agregados microbianos (biopelículas, flóculos, gránulos), si bien lo ideal para ello sería disponer de un microscopio confocal. Su principal limitación es la disponibilidad de sondas para el taxón o grupo bacteriano que queremos localizar. Si no es así, es preciso diseñar la sonda y poner a punto sus condiciones de hibridación, lo que requiere tiempo y experiencia. 5
7 Tabla 1. Sondas para FISH y sus características Sonda Secuencia (5-3 ) Formamida (%) / Especificidad NaCl (mm) EUB338 GCTGCCTCCCGTAGGAGT 35 / 80 Bacteria ARC915 GTGCTCCCCCGCCAATTCCT 20 / 225 Archaea ALF968 GGTAAGGTTCTGCGCGTT 20 / 225 Alpha-proteobacteria BET42a GCCTTCCCACTTCGTTT 35 / 80 Beta-proteobacteria HGC69A TATAGTTACCACCGCGT 25 / 159 Gram-positivos con alto G+C (Actinobacteria) MX825 (**) TCGCACCGTGGCCGACACCT 20 / 225 Methanosaeta AGC (*): solo en el lodo granular. 6.1 Fijación. Las muestras se fijan con formaldehído o para-formaldehído a una concentración final del 4%. El tiempo más habitual es de 4 horas a 4 o C, si bien puede variar entre 2-20 dependiendo de la muestra. El procedimiento de fijación y lavado del formaldehído es el descrito en al apartado 5. Las muestras en PBS:etanol pueden conservarse a -20 o C durante meses. 6.2 Hibridación y lavado Paso 1: estimación de la dilución adecuada. Preparar cuatro diluciones (1/5, 1/10, 1/100, 1/1000 en etanol:pbs 1:1) -de la muestra preparada en 5- y añadir 5 l de cada suspensión a un pocillo del portaobjetos multipocillos ( 0,5 cm. FEDELCO. ER-308). Secar durante aprox. 15 minutos en estufa (30-46 o C) y teñir con DAPI (1 mg/ml), durante 3 minutos. El exceso de DAPI se lava primero con H 2 O Milli-Q y luego con Etanol 70%. Dejar secar y añadir una gota de CITIFLUOR en cada pocillo, poner el cubre y contar las bacterias teñidas de azul al microscopio de fluorescencia con el filtro para DAPI. Para realizar la hibridación debemos seleccionar la dilución en la cual contemos aproximadamente bacterias por campo. Paso 2: hibridación. Añadir 5 l de la dilución seleccionada en un pocillo del portaobjetos excavado multipocillos. Secar. Deshidratar la muestra introduciéndola en disoluciones de etanol de concentración creciente (50%, 80%, 100%), 3 minutos en cada concentración. Dejar secar a temperatura ambiente. Añadir 8 l del tampón de hibridación (tablas 2, 3) y 1 l de cada sonda de FISH a utilizar (tabla 1). Poner el porta en una cámara de hibridación y dejar hibridando durante 2 horas (entre 1.5 y varias horas) a 46 o C. Paso 3: lavado. Tras la hibridación debe eliminar el tampón de hibridación (cuidado con los restos. La formamida es tóxica!) mediante un lavado con alta fuerza iónica. Para esto, llenar la cámara de hibridación con 50 ml de tampón de lavado (tablas 2 y 3) y mantenerlo durante 15 minutos ( exactos!) a 48 o C (tener el baño pre-calentado). Pasado este tiempo eliminar el tampón de lavado, quitar los restos con agua y secar (aire, estufa). Teñir con DAPI (1 mg/ml), 6
8 durante 3 minutos y eliminar el exceso de DAPI lavando primero con H 2 O Milli-Q y luego con Etanol 70%. Tabla 2: Composición química del tampón de hibridación y del tampón de lavado. (Los valores de X,Y,Z se indican en la tabla 3). Tampón de Hibridación Concentración Cantidad NaCl 5 M 180 l Tris HCl 1 M ( ph = 8) 20 l Formamida 100 X SDS 10 % 1 l Agua Milli-Q PURA Hasta 1000 l Tampón de lavado Concentración Cantidad NaCl 5 M Y Tris HCl 1 M ( ph = 8) 1000 l EDTA 0.5 M ( ph = 9) Z SDS 10 % 50 l Agua Milli-Q PURA Hasta 50 ml Tabla 3: Valores de X, Y, Z. Estos dependen del porcentaje de formamida utilizada para cada sonda (V hibridación = 1 ml; V lavado = 50 ml). Formamida (%) X( l)= Formamida Y ( l) = NaCl (5M) Z ( l) = EDTA (0.5M) Paso 4: observación y conteo. Las muestras se observan al microscopio con los filtros específicos para cara fluorocromo. Para proteger la fluorescencia añadir una gota de citifluor. 7- Extracción del ADN de los microorganismos presentes en la muestras utilizando el FastDNA Kit for soil BIO Tomar la muestra preparada en el paso 5 y mezclar con 120 μl de MT Buffer. Añadir la mezcla en los tubos (Lysing Matriz E tube). 7
9 2- Romper las bacterias en el equipo Fastprep FP120, con los siguientes parámetros: Cuatro ciclos de ruptura a 5,5 potencia y 40 segundos. Enfriar 5 minutos en hielo entre el segundo y tercer ciclo. 3- Centrifugar el tubo a rpm, durante 1 minuto y transferir el sobrenadante a un eppendorf de 1,5 ml. 4- Añadir al sobrenadante 250 μl de PPS y mezclar invirtiendo 12 veces. 5- Centrifugar a rpm durante 5 minutos. 6- Transferir el sobrenadante a un eppendorf de 5 ml y añadirle 1 ml de Binding Matriz Suspensión. 7- Agitar la mezcla lentamente en un rotor vertical durante 20 minutos a 4 o C. 8- Filtrar en un SPIN filter, añadiendo al filtro volúmenes no superiores a 600 μl de la mezcla, centrifugar a rpm durante 1 minuto y desechar el sobrenadante. Repetir las veces que sean necesarias hasta filtrar toda la mezcla. 9- A la resina retenida en el filtro añadirle 500 μl de SEWS-M y centrifugar a rpm durante 1 minuto. 10- Centrifugar el SPIN filter a rpm durante 3 minutos, para secar la muestra. 11- Descartar el tubo eppendorf, meter el filtro en un nuevo tubo y dejarlo abierto 5 minutos para que se seque. 12- Añadirle 100 μl de DES y resuspender la resina (homogenizar 2 minutos). 13- Centrifugar a rpm durante 3 minutos. El líquido filtrado contiene el ADN. Conservarlo a 4 o C. 8- Amplificación por PCR de un fragmento del gen del ARNr 16S para DGGE. La reacción en cadena de la polimerasa (PCR) es un método rápido y versátil de amplificación in vitro de secuencias específicas de ADN presentes en una mezcla compleja de secuencias (ADN total). Para poder llevar a cabo una amplificación selectiva es necesario conocer las secuencias flanqueantes a la diana. Con ellas se construyen dos oligonucleótidos, de entre 15 y 30 bases, que actúan como cebadores (primer) en la reacción. Estos cebadores están diseñados para que se pueda iniciar la reacción de síntesis de ADN en presencia de una ADN polimerasa termoestable adecuada (Vg. Taq, TthI,..) y de los precursores del ADN (los cuatros desoxinucleótidos trifosfato: datp, dctp, dgtp y dttp). Cada cebador inicia la síntesis de una hebra de ADN complementaria a una de las hebras del segmento de la diana con lo que las dos hebras de nueva síntesis serán complementarias entre sí. Las hebras de ADN de nueva síntesis servirán de moldes para las reacciones de síntesis en los ciclos posteriores. En estas prácticas la PCR se utilizará para amplificar fragmentos de DNA que serán separados 8
10 mediante DGGE (apartado 9) y para la formación y rastreo de librerías genéticas (apartados 10 y 11). Condiciones de la PCR para posterior DGGE (volumen de 100 μl) Tampón 10X (sin MgCl 2 ) 10 μl. Solución de MgCL 2 (25 mm).. 12 μl 1 dntps (50 mm). 1 μl Cebadores (50 μm) μl (de cada uno: Forward y Reverse). ADN. Depende de las características de la muestra y su Concentración (1-10 μl. Puede ser necesaria su dilución previa) Taq ADN Polimerasa. 0,5 H 2 O MilliQ..Hasta 100 μl 1 : la concentración final de Mg 2+ es de 3 mm. Si el tampón 10X incluye Mg 2+ debe tenerse en cuenta. 2 : 50 pmoles/ μl Para amplificar por PCR fragmentos del gen del ARNr 16S a partir de una muestra compleja, y que posteriormente serán resueltos mediante DGGE se utilizarán los siguientes pares de cebadores (tabla 4): 341F-GC - 907R (T = 52 o C) para bacterias y 622F-GC R (T = 42 o C) para arqueas. Programa: 94 o C 94 o C 10 min. 1 min. T 72 o C 72 o C 2 min. 10 min. 1 min. 4 o C 35 Ciclos Tabla 4. Cebadores utilizados para la amplificación por PCR del gen ARNr 16S de Bacterias y Arqueas. Cebador Diana Secuencia (de 5 a 3 ) Especificidad Referencia 907R CCGTCAATTCMTTTGAGTTT Dominio Bacteria Brosius, F-GC* CCTACGGGAGGCAGCAG Dominio Bacteria Brosius, F-GC* TGAAATCYYRTAATCCC Dominio Arquea Chan, R TACGGYTACCTTGTTACGACTT Universal Lane, 1991 Nota: GC*: secuencia de 40 nucleótidos rica en GC, unida al extremo 5 del cebador. La secuencia GC es: 5 - CGC CCG CCG CGC CCC GCG CCC GTC CCG CCC CCG CCC-3. (M = C:A, Y = C:T, R = A:G) 9
11 9. Electroforesis en gel con gradiente desnaturalizante (DGGE). La DGGE/TGGE se basa en la diferente movilidad electroforética de fragmentos de DNA parcialmente desnaturalizados con igual longitud pero diferente secuencia, lo que da lugar a un patrón de bandas reflejo de la diversidad genética presente en la comunidad microbiana. El número de bandas corresponde con el número de miembros predominantes, y su secuenciación y posterior análisis filogenético dan una buena perspectiva sobre la composición de dicha comunidad. A partir de la muestra que se quiera estudiar se lleva a cabo la extracción de DNA y RNA. Mediante PCR se procede a una amplificación parcial (entre pb) del gen del 16S rrna utilizando cebadores específicos. Las distintas moléculas de DNA correspondientes a diferentes microorganismos o unidades taxonómicas operacionales (OTUs) se separan mediante electroforesis desnaturalizante. Para ello se utilizan geles de poliacrilamida con un gradiente creciente de urea-formamida (DGGE) o de temperatura (TGGE). Cada molécula de DNA se desplaza hacia el polo positivo y deja de moverse cuando la concentración de ureaformamida/temperatura provoca su desnaturalización (separación de las dos moléculas del DNA de doble hebra según su temperatura de fusión -Tm). Cada banda obtenida en el gel corresponde a un microorganismo presente en la muestra original, por lo que el simple patrón de bandas puede ser suficiente para determinados estudios. Las bandas pueden ser recortadas y el DNA secuenciado. Comparando las secuencias resultantes con una base de datos de 16S DNA de bacterias y arqueas, podremos saber con que organismos tienen mayor similitud los microorganismos presentes en nuestra muestra. La DGGE/TGGE y otras técnicas de huella genética son técnicas rápidas y sencillas si se limitan a la obtención de un patrón de bandas, aunque puede aumentarse su complejidad y la información obtenida si se incluye la secuenciación y análisis filogenético de las mismas. Generan una información taxonómica y filogenética relativamente precisa (menos que la clonación, pero más que el FISH) sobre los microorganismos predominantes en el sistema, y es útil para seguir la evolución espacial y temporal de las poblaciones de microorganismos y su respuesta a cambios físico-químicos y nutricionales. G P 9.1- Preparación del gel de DGGE. 1- Para hacer el gel se utilizan 2 cristales: (G)- cristal grande (20cm x 20cm) y (P)- cristal pequeño (20cm x 18cm). Además se utilizan dos separadores (1 mm de espesor). 1 10
12 G P 2 2- Se colocan los separadores encima del cristal grande (G). Se puede poner un poco de vaselina en la superficie de los separadores para que el cierre lateral sea más hermético. 3- Posteriormente se coloca el cristal pequeño encima Se utilizan dos pinzas (A, B) para ajustar los cristales. A B 4 5- Primero se pone una pinza y luego se coloca la otra. 5 11
13 6 6- Ajustar bien las pinzas ( cuidado!: no apretar excesivamente. Tanto las pinzas como los vidrios pueden romperse) y verificar que los cristales están colocados correctamente. Poner el sistema formador de gel sobre una base, que tiene en la parte inferior una lámina de plástico que sella herméticamente todo el sistema. 7- El sistema formador de gel se ajusta a la base mediante dos llaves laterales Formación del gel: Montar un sistema como se indica en la figura 8. Se prepararán dos soluciones de urea-formamida (20% y 70%) a partir de los stock (0% y 80%). H L 8 Composición del Stock 0%: 37,5 ml de Acrilamida/Bisacrilamida 40%. 5 ml de TAE 50X llevar hasta 250 ml con agua MilliQ. 12
14 Composición del Stock 80%: 37,5 ml de Acrilamida/Bisacrilamida 40%. 5 ml de TAE 50X 84 g de Urea 80 ml formamida desionizada. llevar hasta 250 ml con agua MilliQ. Preparación de las soluciones de urea-formamida Solución al 20%: Se mezclan 2,5 ml del stock al 80% y 7,5 ml del stock al 0%. Homogenizar levemente y añadir 6,9 μl de TEMED y 53 μl de APS (1%). Solución al 70%: Se mezclan 8,75 ml del stock al 80% y 1,25 ml del stock al 0%. Homogenizar levemente y añadir 6,9 μl de TEMED y 53 μl de APS (1%). TEMED : N,N,N,N -tetrametilentilendiamina. APS: Peroxidisulfato de amonio. Las soluciones 20% y 70% se añaden en los depósitos del formador de gradientes. La solución del 20% en el compartimiento (L) y la solución del 80 en el compartimiento (H). Cerciorarse que la comunicación entre ambos compartimientos este cerrada. Al compartimiento (H) se le introduce una mosca magnética y todo el sistema se coloca encima de un agitador magnético para garantizar que la mezcla se homogenicé de forma continua. El agitador magnético se pone en marcha en cuanto se añaden las dos soluciones. Posteriormente, de forma simultánea se pone en marcha la bomba peristáltica y se abre la comunicación entre los compartimientos (H) y (L). Observaremos como en el compartimiento (H) se van mezclando poco a poco ambas soluciones y la bomba peristáltica bombea esta mezcla al sistema formador del gel. Cuando prácticamente no queda solución en los compartimientos, se inclina el formador del gradiente para bombear todo. Posteriormente se mezclan 10 ml de la solución stock del 0% con 6,9 μl de Temed y 53 μl de APS (1%) y se homogeniza. De esta mezcla se adiciona 9 ml al sistema formador del gel, por la parte superior con la ayuda de una pipeta pasteur. Luego se introduce suavemente el peine formador de los pocillos y añade más mezcla hasta completar el gel. Dejar polimerizar, a 4 o C, durante un mínimo de 4 horas y un máximo de 24 h. 13
15 Se preparan 7,5 L de TAE 1X a partir del stock de TAE 50X ( mezclar 242 g de Tris base, 57,1 ml de ácido acético glacial, 100 ml de 0,5 M EDTA, disolver hasta 1 L con agua destilada y esterilizar 20 minutos a 1 atm) 9- Después de gelificado se saca el peine y se lavan los pocillos con el buffer TAE 1X. Se coloca el sistema formador de gel sobre la pieza de sujeción Introducir el conjunto en la carcasa que contiene 7,5 L del buffer TAE 1X Las muestras (producto de PCR) que se van a cargar en el gel se concentran al vacío hasta reducir su volumen a unos 15 μl. Añadir a cada muestra 10 μl tampón de carga y cargarlas en los pocillos con mucho cuidado y evitando que pase muestra de un pocillo a otro. El gel se corre con las siguientes condiciones electroforéticas: Potencial eléctrico: 200 v Tiempo: 4 horas.temperatura: 60 o C 12- Después de correr el gel de DGGE, se apaga la fuente, el sistema de agitación y calentamiento y se saca el conjunto de la carcasa
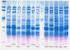
16 13- El sistema formado de gel se separa de la pieza de sujeción Se quitan las 2 pinzas con mucho cuidado y con la ayuda de los separadores se quita solo el cristal pequeño. Quedando el gel pegado al cristal grande. Luego el gel (junto con el cristal grande) se tiñe durante 20 minutos en una solución de Bromuro de Etidio (alternativa: GelRed) y, posteriormente, se lava con agua durante 10 minutos Foto de un gel de DGGE (gradiente 20-70), después de teñido. Cada carril corresponde a una muestra y, en teoría, cada banda a un microorganismo con diferente secuencia. Estas bandas se cortan, se reamplifican con los mismos primer, se purifican y se secuencian. 15
17 10. Amplificación del gen del ARNr 16S para la construcción de genotecas. A partir de los ADNs extraídos en el punto 7, vamos a volver a amplificar el ADN correspondiente al gen que codifica el ARN 16S. El producto de la PCR se utilizará para la construcción de genotecas en plásmidos. La amplificación se realizará utilizando los siguientes cebadores: Cebador Secuencia Especificidad 27f AGAGTTTGATCMTGGCTCAG Bacteria 25f CYGGTTGATCCTGCCRG Archaea 1492r TACGGYTACCTTGTTACGACTT Universal Y: C/T; R: A/G Las condiciones de la reacción de PCR son las siguientes: 2,5 L de tampón de reacción (10x), 5pmol de cada cebador, 3,75mM MgCl 2, 0,2mM dntps, 5 g de Albúmina de suero bovino (BSA) y 0.5-1u de ADN polimerasa, en un volumen final de 25 L. El programa de PCR consiste en 5min de desnaturalización a 94 C seguido por 25 ciclos de 1min a 94 C (desnaturalización), 1 min a 55ºC (anillamiento), 2 min a 72 C (elongación) y un ciclo final de 10 min a 72 C. Los productos de PCR se clonarán en el plásmido pgemt easy vector de Promega (Madison, E.E.U.U.). Para ello, se realizará la siguiente reacción de ligación: 2X Rapid ligation buffer 5 l P-Gem vector 1 l Producto de PCR 3 l T4 DNA Ligasa 1 l Los tubos se incubarán como mínimo 1 hora a temperatura ambiente (alternativa: 4 C toda la noche, lo que aumenta la eficiencia). Los productos de las ligaciones se transformarán en células competentes de E. coli DH5, según el siguiente protocolo: 1. Dejar un tubo de células competentes (si fuera necesario hacer alícuotas de 50 l) en hielo hasta que se descongele (unos 5 min). Mezclar suavemente. 2. Centrifugar las ligaciones con un breve pulso. Añadir 2 l de cada reacción de ligación a un tubo esteril de 1,5 ml. Colocar en hielo 3. Incubar 30 min en hielo 16
18 4. Incubar la mezcla durante 1 30 a 42 o C (shock térmico). No agitar. Inmediatamente poner los tubos en hielo otros 2 min. 5. Añadir 500 l de medio SOC o LB a temperatura ambiente a las reacciones de transformación. 6. Incubar 1 / 1.5 h a 37 o C, con agitación. 7. Plaquear 100 l de la transformación en una placa de LB/ampicilina/X-Gal 8. Centrifugar el resto de la transformación, eliminar la mayoría del líquido y plaquear otra placa de LB/Ampicilina/X-Gal (Amp: 100 ng/ l ; X-Gal: 100 ng/ l) 9. Incubar las placas a 37 o C durante la noche. 17
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
3. COMPONENTES. Tampón de electroforesis concentrado 2 x 50 ml 10 X (2 envases 500ml)
 PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS
 Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL
 LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
DETECCIÓN DE C. jejuni EN HAMBURGUESA DE POLLO POR PCR TRADICIONAL PRT-712.04-082
 Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz porosa
 INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
DETERMINACIÓN DEL FACTOR Rh por PCR
 Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
ELECTROFORESIS BASICA
 Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA. Grado en Medicina
 DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES
 PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
ELECTROFORESIS AVANZADA
 Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
3/22/2010. Iván Ferrer Rodríguez, PhD Catedrático. En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR.
 Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I
 UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I DRAS. MARÍA LUISA ORTIZ Y ANA PEDREGOSA 1 PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA
UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I DRAS. MARÍA LUISA ORTIZ Y ANA PEDREGOSA 1 PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA
DANAGENE RNA PURIFICATION KIT
 DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz
 IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDS-PAGE. Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE)
 ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDSPAGE Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE) Método rápido, reproducible y de bajo costo Utilizado para cuantificar, comparar
ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDSPAGE Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE) Método rápido, reproducible y de bajo costo Utilizado para cuantificar, comparar
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR
 PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
Reacción en cadena de la polimerasa (PCR)
 Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
DANAGENE SALIVA KIT. Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones
 DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
CUESTIONES. 1. Por qué la PCR convencional no es cuantitativa?
 2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
TÉCNICAS GENÓMICAS. 2) Cuantificación del número de virus presentes en muestras de sangre o suero: carga vírica
 TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
Procesado de muestras en el laboratorio de la clínica (I)
 12.prevención de la salud Procesado de muestras en el laboratorio de la clínica (I) A lo largo de esta primera parte veremos como realizar un manejo correcto de las muestras de sangre, orina y líquidos
12.prevención de la salud Procesado de muestras en el laboratorio de la clínica (I) A lo largo de esta primera parte veremos como realizar un manejo correcto de las muestras de sangre, orina y líquidos
Código: IDK-010 Ver: 1. Proteína G C825T. Sistema para la detección de la mutación C825T en el gene de la proteína G.
 C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
Protocolos de secuenciación de ADN
 1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
Instrucciones técnicas Aparato de extracción y filtración en caliente para sistemas de microondas
 Instrucciones técnicas Aparato de extracción y filtración en caliente para sistemas de microondas Las condiciones experimentales de un experimento con microondas dependen de los datos técnicos del aparto
Instrucciones técnicas Aparato de extracción y filtración en caliente para sistemas de microondas Las condiciones experimentales de un experimento con microondas dependen de los datos técnicos del aparto
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Laboratorio Biología Molecular: EPSH. Universidad de Zaragoza
 GEL DE POLIACRILAMIDA A) Preparación del soporte del gel: Para hacer el gel se utilizan dos láminas de vidrio (cristal en "U" y cristal recto) unidas con cinta adhesiva. Estas láminas son diferentes, se
GEL DE POLIACRILAMIDA A) Preparación del soporte del gel: Para hacer el gel se utilizan dos láminas de vidrio (cristal en "U" y cristal recto) unidas con cinta adhesiva. Estas láminas son diferentes, se
5. MATERIALES Y MÉTODOS
 5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO
 Actividad Experimental SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO Investigación previa 1.- Investigar las medidas de seguridad que hay que mantener al manipular KOH y H SO, incluyendo que acciones
Actividad Experimental SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO Investigación previa 1.- Investigar las medidas de seguridad que hay que mantener al manipular KOH y H SO, incluyendo que acciones
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS.
 NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
Práctico: Genética bacteriana 2007.
 Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
Prácticas integradas de Ecología molecular microbiana
 Prácticas integradas de Ecología molecular microbiana Indice Página 1. Introducción 1 2. Cronograma 1 3. Esquema de trabajo 2 4. Reactivos utilizados 3 5. Preparación de las muestras 3 6. Extracción del
Prácticas integradas de Ecología molecular microbiana Indice Página 1. Introducción 1 2. Cronograma 1 3. Esquema de trabajo 2 4. Reactivos utilizados 3 5. Preparación de las muestras 3 6. Extracción del
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico
 17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
LABORATORIO: MICROBIOLOGÍA BIOQUÍMICA Y HEMATOLOGÍA.
 LABORATORIO: MICROBIOLOGÍA BIOQUÍMICA Y HEMATOLOGÍA. CS Illes Columbretes Página 1 En el laboratorio de microbiología se realizan investigaciones microbiológicas (diagnóstico bacteriológico, micológico,
LABORATORIO: MICROBIOLOGÍA BIOQUÍMICA Y HEMATOLOGÍA. CS Illes Columbretes Página 1 En el laboratorio de microbiología se realizan investigaciones microbiológicas (diagnóstico bacteriológico, micológico,
Normalización de soluciones de NaOH 0,1N y HCl 0,1N.
 Laboratorio N 1: Normalización de soluciones de NaOH 0,1N y HCl 0,1N. Objetivos: - Determinar la normalidad exacta de una solución de hidróxido de sodio aproximadamente 0,1 N, utilizando biftalato de potasio
Laboratorio N 1: Normalización de soluciones de NaOH 0,1N y HCl 0,1N. Objetivos: - Determinar la normalidad exacta de una solución de hidróxido de sodio aproximadamente 0,1 N, utilizando biftalato de potasio
TRABAJO PRÁCTICO N 2: TÉCNICAS DE ESTERILIZACIÓN Y CULTIVO DE MICROORGANISMOS Objetivos:
 TRABAJO PRÁCTICO N 2: TÉCNICAS DE ESTERILIZACIÓN Y CULTIVO DE MICROORGANISMOS Objetivos: -Conocer las metodologías actuales de control y eliminación de microorganismos. -Obtener dominio de los métodos
TRABAJO PRÁCTICO N 2: TÉCNICAS DE ESTERILIZACIÓN Y CULTIVO DE MICROORGANISMOS Objetivos: -Conocer las metodologías actuales de control y eliminación de microorganismos. -Obtener dominio de los métodos
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O. Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua
 C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
2.3 SISTEMAS HOMOGÉNEOS.
 2.3 SISTEMAS HOMOGÉNEOS. 2.3.1 DISOLUCIONES. Vemos que muchos cuerpos y sistemas materiales son heterogéneos y podemos observar que están formados por varias sustancias. En otros no podemos ver que haya
2.3 SISTEMAS HOMOGÉNEOS. 2.3.1 DISOLUCIONES. Vemos que muchos cuerpos y sistemas materiales son heterogéneos y podemos observar que están formados por varias sustancias. En otros no podemos ver que haya
39. Aislamiento y purificación del DNA de un plásmido recombinante
 39. Aislamiento y purificación del DNA de un plásmido recombinante Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento de Bioquímica
39. Aislamiento y purificación del DNA de un plásmido recombinante Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento de Bioquímica
MANUAL DE PRÁCTICAS DE LABORATORIO
 MANUAL DE PRÁCTICAS DE LABORATORIO CARRERA Ingeniería en Biotecnología ASIGNATURA: Microbiología Gral. FICHA TECNICA Fecha: Nombre del catedrático: 13-SEPT-2012 FICHA TÉCNICA MICROBIOLOGÍA GENERAL Jesús
MANUAL DE PRÁCTICAS DE LABORATORIO CARRERA Ingeniería en Biotecnología ASIGNATURA: Microbiología Gral. FICHA TECNICA Fecha: Nombre del catedrático: 13-SEPT-2012 FICHA TÉCNICA MICROBIOLOGÍA GENERAL Jesús
Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a
 Introducción Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a los investigadores verificar la expresión de una proteína, determinar la cantidad relativa
Introducción Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a los investigadores verificar la expresión de una proteína, determinar la cantidad relativa
Practica la genética Ficha didáctica del profesorado Bachillerato
 Ficha didáctica del profesorado Bachillerato www.eurekamuseoa.es Extracción de ADN Cuál es la función de cada uno de los ingredientes utilizados para realizar la disolución tampón para la visualización
Ficha didáctica del profesorado Bachillerato www.eurekamuseoa.es Extracción de ADN Cuál es la función de cada uno de los ingredientes utilizados para realizar la disolución tampón para la visualización
PCR gen 18S ARNr humano
 PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA"
 P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
COPROPACK PLUS. Fijación y concentración de parásitos fecales. Noviembre 2012
 COPROPACK PLUS Noviembre 2012 A 7 Km 1081 P.O. Box 20 - E43870 AMPOSTA / SPAIN1 Tel. ++ 34 (977) 70 14 39 Fax ++ 34 (977) 70 30 40 1 DETECCIÓN DE PARÁSITOS FECALES La metódica habitual para la separación
COPROPACK PLUS Noviembre 2012 A 7 Km 1081 P.O. Box 20 - E43870 AMPOSTA / SPAIN1 Tel. ++ 34 (977) 70 14 39 Fax ++ 34 (977) 70 30 40 1 DETECCIÓN DE PARÁSITOS FECALES La metódica habitual para la separación
AUTOR: JORGE CONTRERAS PINEDA 1. Titulo:
 Página 1 de 1 1. Titulo: Electroforesis de DNA 2. Objetivo Conocer los principios básicos de la electroforesis horizontal en geles de agarosa y aplicarlo para la separación de DNA humano, plasmídico, recombinante
Página 1 de 1 1. Titulo: Electroforesis de DNA 2. Objetivo Conocer los principios básicos de la electroforesis horizontal en geles de agarosa y aplicarlo para la separación de DNA humano, plasmídico, recombinante
SECUENCIACIÓN SANGER DE ADN: GUÍA DE SOLUCIONES TÉCNICAS
 SECUENCIACIÓN SANGER DE ADN: GUÍA DE SOLUCIONES TÉCNICAS Secugen recomienda que para la visualización de las secuencias se usen programas que permitan ver el dato crudo, ya que a partir de ese dato se
SECUENCIACIÓN SANGER DE ADN: GUÍA DE SOLUCIONES TÉCNICAS Secugen recomienda que para la visualización de las secuencias se usen programas que permitan ver el dato crudo, ya que a partir de ese dato se
PCR. Técnica inventada por Kary Mullis :1987. Premio Nobel 1993. Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN
 PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
Curso de biología molecular CIMAT 2010
 Curso de biología molecular CIMAT 2010 INTRODUCCIÓN Este curso-taller tiene como propósito mostrar de manera teórica y práctica algunos principios básicos de biología molecular e ingeniería genética. Conocerás
Curso de biología molecular CIMAT 2010 INTRODUCCIÓN Este curso-taller tiene como propósito mostrar de manera teórica y práctica algunos principios básicos de biología molecular e ingeniería genética. Conocerás
3. ESTANDARIZACIÓN DE DISOLUCIONES VALO- RANTES.
 3. ESTANDARZACÓN DE DSOLUCONES ALO- RANTES. 3.1 NTRODUCCÓN Si la disolución valorante no se ha preparado a partir de un patrón primario, su concentración no será exactamente conocida, y por lo tanto, habrá
3. ESTANDARZACÓN DE DSOLUCONES ALO- RANTES. 3.1 NTRODUCCÓN Si la disolución valorante no se ha preparado a partir de un patrón primario, su concentración no será exactamente conocida, y por lo tanto, habrá
ECOLOGÍA MICROBIANA Y COMPORTAMIENTO BACTERIANO COMUNITARIO
 5 TRABAJO PRÁCTICO ECOLOGÍA MICROBIANA Y COMPORTAMIENTO BACTERIANO COMUNITARIO OBJETIVOS: 1 Observar y comprender la diversidad bacteriana que coexiste en un mismo nicho en un dado ecosistema y las interrelaciones
5 TRABAJO PRÁCTICO ECOLOGÍA MICROBIANA Y COMPORTAMIENTO BACTERIANO COMUNITARIO OBJETIVOS: 1 Observar y comprender la diversidad bacteriana que coexiste en un mismo nicho en un dado ecosistema y las interrelaciones
1 MATERIALES Y MÉTODOS
 1 MATERIALES Y MÉTODOS El presente protocolo experimental contempla la amplificación del DNA de las bacterias y virus causantes de las ETS Neisseria gonorrhoeae, Chlamydia trachomatis y VPH mediante PCR
1 MATERIALES Y MÉTODOS El presente protocolo experimental contempla la amplificación del DNA de las bacterias y virus causantes de las ETS Neisseria gonorrhoeae, Chlamydia trachomatis y VPH mediante PCR
V MATERIALES Y MÉTODOS
 V MATERIALES Y MÉTODOS 5.1 Aislamiento de bacterias e identificación 5.1.1 Cepas tomadas del cepario de la Universidad de las Américas-Puebla En el cepario del Departamento de Química y Biología hay algunas
V MATERIALES Y MÉTODOS 5.1 Aislamiento de bacterias e identificación 5.1.1 Cepas tomadas del cepario de la Universidad de las Américas-Puebla En el cepario del Departamento de Química y Biología hay algunas
I. 15microlitros de agua esteril, perforar el pozo y diluir. II. Se tomaron 2 microlitros para la primera transformación.
 23 de agosto 2007 Se comenzó la elaboración de la bitácora Medio LB Broth, Billar (Luria-Bertani) 25gr/litro Se preparó 500 mililitros agregando 12.5 gramos Se diluyó y esterilizó por autoclave Medio LB
23 de agosto 2007 Se comenzó la elaboración de la bitácora Medio LB Broth, Billar (Luria-Bertani) 25gr/litro Se preparó 500 mililitros agregando 12.5 gramos Se diluyó y esterilizó por autoclave Medio LB
Centro de Capacitación en Informática
 Fórmulas y Funciones Las fórmulas constituyen el núcleo de cualquier hoja de cálculo, y por tanto de Excel. Mediante fórmulas, se llevan a cabo todos los cálculos que se necesitan en una hoja de cálculo.
Fórmulas y Funciones Las fórmulas constituyen el núcleo de cualquier hoja de cálculo, y por tanto de Excel. Mediante fórmulas, se llevan a cabo todos los cálculos que se necesitan en una hoja de cálculo.
Técnicas de ADN recombinante: la manipulación genética
 Técnicas de ADN recombinante: la manipulación genética A partir de los años 70 se desarrollaron las herramientas de la biología molecular o la ingeniería genética o lo que se ha llamado técnicas del ADN
Técnicas de ADN recombinante: la manipulación genética A partir de los años 70 se desarrollaron las herramientas de la biología molecular o la ingeniería genética o lo que se ha llamado técnicas del ADN
PIP 4º ESO IES SÉNECA TRABAJO EXPERIMENTAL EN FÍSICA Y QUÍMICA
 MEZCLAS Las mezclas son agrupaciones de dos o más sustancias puras en proporciones variables. Si presentan un aspecto uniforme son homogéneas y también se denominan disoluciones, como la de azúcar en agua.
MEZCLAS Las mezclas son agrupaciones de dos o más sustancias puras en proporciones variables. Si presentan un aspecto uniforme son homogéneas y también se denominan disoluciones, como la de azúcar en agua.
2) PRÁCTICAS DE BIOLOGÍA (2º de Bachillerato) IDENTIFICACIÓN DE CROMOSOMAS HUMANOS Y REALIZACIÓN DE UN IDEOGRAMA DE UN CARIOTIPO
 2) PRÁCTICAS DE BIOLOGÍA (2º de Bachillerato) IDENTIFICACIÓN DE CROMOSOMAS HUMANOS Y REALIZACIÓN DE UN IDEOGRAMA DE UN CARIOTIPO OBJETIVO El objetivo de esta práctica es aprender a reconocer los cromosomas
2) PRÁCTICAS DE BIOLOGÍA (2º de Bachillerato) IDENTIFICACIÓN DE CROMOSOMAS HUMANOS Y REALIZACIÓN DE UN IDEOGRAMA DE UN CARIOTIPO OBJETIVO El objetivo de esta práctica es aprender a reconocer los cromosomas
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa
 38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
23. Hidrólisis ácida y enzimática del glucógeno
 23. Hidrólisis ácida y enzimática del glucógeno Emilia Martínez Galisteo, Carmen Alicia Padilla Peña, Concepción García Alfonso, José Antonio Bárcena Ruiz, Jesús Diez Dapena. Departamento de Bioquímica
23. Hidrólisis ácida y enzimática del glucógeno Emilia Martínez Galisteo, Carmen Alicia Padilla Peña, Concepción García Alfonso, José Antonio Bárcena Ruiz, Jesús Diez Dapena. Departamento de Bioquímica
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR)
 METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS
 GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS Leidy Diana Ardila Leal Docente. INTRODUCCIÓN En esta práctica se va a realizar la extracción
GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS Leidy Diana Ardila Leal Docente. INTRODUCCIÓN En esta práctica se va a realizar la extracción
CÁMARA THOMA Y NEUBAUER IMPROVED PARA EL RECUENTO DE LEVADURAS (TIRAJE)
 1/5 CÁMARA THOMA Y NEUBAUER IMPROVED PARA EL RECUENTO DE LEVADURAS (TIRAJE) En este documento se pretende explicar de forma clara y sencilla el proceso de recuento y viabilidad de las levaduras para el
1/5 CÁMARA THOMA Y NEUBAUER IMPROVED PARA EL RECUENTO DE LEVADURAS (TIRAJE) En este documento se pretende explicar de forma clara y sencilla el proceso de recuento y viabilidad de las levaduras para el
ORGANIZACIÓN DEL LABORATORIO DE CULTIVO DE TEJIDOS
 ORGANIZACIÓN DEL LABORATORIO DE CULTIVO DE TEJIDOS Las actividades relacionadas con cultivo in vitro de tejidos deben realizarse en ambientes asépticos, con iluminación y temperatura controladas, para
ORGANIZACIÓN DEL LABORATORIO DE CULTIVO DE TEJIDOS Las actividades relacionadas con cultivo in vitro de tejidos deben realizarse en ambientes asépticos, con iluminación y temperatura controladas, para
Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez
 Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez 1 Objetivos Entender la técnica: Reacción de Polimerasa en Cadena (PCR por sus siglas en inglés)
Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez 1 Objetivos Entender la técnica: Reacción de Polimerasa en Cadena (PCR por sus siglas en inglés)
BIOTECNOLOGÍA. 1.- Ingeniería Genética, ADN recombinante
 BIOTECNOLOGÍA 1.- Ingeniería Genética, ADN recombinante Técnica del ADN recombinante: es la más usada en ingeniería genética, esta basada en el uso de las enzimas de restricción o endonucleasas de restricción
BIOTECNOLOGÍA 1.- Ingeniería Genética, ADN recombinante Técnica del ADN recombinante: es la más usada en ingeniería genética, esta basada en el uso de las enzimas de restricción o endonucleasas de restricción
VALORACIÓN ÁCIDO-BASE. Conocer y aplicar el método volumétrico para realizar una titulación ácido-base
 EXPERIMENTO 3 VALORACIÓN ÁCIDO-BASE Objetivo general Conocer y aplicar el método volumétrico para realizar una titulación ácido-base Objetivos específicos 1.- Determinar el punto de equivalencia de una
EXPERIMENTO 3 VALORACIÓN ÁCIDO-BASE Objetivo general Conocer y aplicar el método volumétrico para realizar una titulación ácido-base Objetivos específicos 1.- Determinar el punto de equivalencia de una
MMP. MÉTODOS DE MUESTREO Y PRUEBA DE MATERIALES
 LIBRO: PARTE: TÍTULO: CAPÍTULO: MMP. MÉTODOS DE MUESTREO Y PRUEBA DE MATERIALES 5. MATERIALES PARA SEÑALAMIENTO Y DISPOSITIVOS DE SEGURIDAD 01. Pinturas para Señalamiento 003. Contenido de Pigmento en
LIBRO: PARTE: TÍTULO: CAPÍTULO: MMP. MÉTODOS DE MUESTREO Y PRUEBA DE MATERIALES 5. MATERIALES PARA SEÑALAMIENTO Y DISPOSITIVOS DE SEGURIDAD 01. Pinturas para Señalamiento 003. Contenido de Pigmento en
PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO
 PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO I. OBJETIVO Determinar la presencia de bacterias Mesófilas Aerobias en una muestra de agua potable por la técnica de
PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO I. OBJETIVO Determinar la presencia de bacterias Mesófilas Aerobias en una muestra de agua potable por la técnica de
TRABAJO PRACTICO ESTERILIZACION
 TRABAJO PRACTICO ESTERILIZACION Introducción La esterilización es un proceso de suma importancia para la industria de las fermentaciones. Para comenzar la explicación de este tema es conveniente dejar
TRABAJO PRACTICO ESTERILIZACION Introducción La esterilización es un proceso de suma importancia para la industria de las fermentaciones. Para comenzar la explicación de este tema es conveniente dejar
LABORATORIO BIO 9000 PROCEDIMIENTO PE-F-27 PARA LA DETERMINACION DE CLORO COMBINADO EN AGUAS
 LABORATORIO BIO 9000 PROCEDIMIENTO PARA LA Rev. nº: 02 Fecha de revisión: 08/08/06 BIO 9000 Distribución de documentos Copia Controlada Nº Asignada a: Fecha: / / REV. FECHA HOJA/S CAUSA DEL CAMBIO 01 17/08/04
LABORATORIO BIO 9000 PROCEDIMIENTO PARA LA Rev. nº: 02 Fecha de revisión: 08/08/06 BIO 9000 Distribución de documentos Copia Controlada Nº Asignada a: Fecha: / / REV. FECHA HOJA/S CAUSA DEL CAMBIO 01 17/08/04
EL MICROSCOPIO SECCION ESPAÑOLA CIENCIAS INTEGRADAS ESCUELA EUROPEA DE LUXEMBURGO 3º SECUNDARIA
 EL MICROSCOPIO Introducción En el estudio de las ciencias naturales ocupa una parte particularmente importante el, ya que permite observaciones que están fuera del alcance de la visibilidad directa del
EL MICROSCOPIO Introducción En el estudio de las ciencias naturales ocupa una parte particularmente importante el, ya que permite observaciones que están fuera del alcance de la visibilidad directa del
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
OBTENCIÓN DE CULTIVOS PUROS
 Introducción Objetivo Fundamento Trabajo Práctico Nº 4 OBTENCIÓN DE CULTIVOS PUROS Procedimiento Resultados Instrucciones para el uso del Sistema Anaeróbico Gaspak Bibliografía INTRODUCCIÓN En los ambientes
Introducción Objetivo Fundamento Trabajo Práctico Nº 4 OBTENCIÓN DE CULTIVOS PUROS Procedimiento Resultados Instrucciones para el uso del Sistema Anaeróbico Gaspak Bibliografía INTRODUCCIÓN En los ambientes
EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS
 EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS Objetivos: Mª Jesús González García Mª Amparo Mora Alcácer COLEGIO AVE Mª DE PENYA-ROJA Aprender a trabajar en el laboratorio y apreciar el orden,
EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS Objetivos: Mª Jesús González García Mª Amparo Mora Alcácer COLEGIO AVE Mª DE PENYA-ROJA Aprender a trabajar en el laboratorio y apreciar el orden,
Operación de Microsoft Excel. Guía del Usuario Página 79. Centro de Capacitación en Informática
 Manejo básico de base de datos Unas de las capacidades de Excel es la de trabajar con listas o tablas de información: nombres, direcciones, teléfonos, etc. Excel puede trabajar con tablas de información
Manejo básico de base de datos Unas de las capacidades de Excel es la de trabajar con listas o tablas de información: nombres, direcciones, teléfonos, etc. Excel puede trabajar con tablas de información
La separación de mezclas de las cuales existen dos tipos como son las homogéneas y heterogéneas
 Introducción En el tema operaciones fundamentales de laboratorio se dan una serie e pasos muy importantes para el desarrollo del programa de laboratorio por ejemplo podemos citar varios procedimientos
Introducción En el tema operaciones fundamentales de laboratorio se dan una serie e pasos muy importantes para el desarrollo del programa de laboratorio por ejemplo podemos citar varios procedimientos
PRÁCTICA Nº 5 REACCIONES DE POLIMERIZACIÓN. Objetivos
 PRÁCTICA Nº 5 REACCINES DE PLIMERIZACIÓN bjetivos - Realizar un ejemplo práctico de reacción de polimerización por condensación: Preparación de un poliéster. Preparación de una poliamida. - Resaltar la
PRÁCTICA Nº 5 REACCINES DE PLIMERIZACIÓN bjetivos - Realizar un ejemplo práctico de reacción de polimerización por condensación: Preparación de un poliéster. Preparación de una poliamida. - Resaltar la
PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN DE LAS COMPETENCIAS PROFESIONALES CUESTIONARIO DE AUTOEVALUACIÓN PARA LAS TRABAJADORAS Y TRABAJADORES
 MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
Dispositivo de Permeación Integrado en el GC
 Dispositivo de Permeación Integrado en el GC Diseño Integrado en el GC Nivel de Calibración desde PPB a PPM No se necesitan Cilindros o Reguladores Opción doble Horno Rentable, Seguro, Limpio, Flexible,
Dispositivo de Permeación Integrado en el GC Diseño Integrado en el GC Nivel de Calibración desde PPB a PPM No se necesitan Cilindros o Reguladores Opción doble Horno Rentable, Seguro, Limpio, Flexible,
MATERIALES Y MÉTODOS. Cepas Utilizadas
 MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
Prácticas de Laboratorio MICROSCOPIO ÓPTICO
 Prácticas de Laboratorio EL MICROSCOPIO ÓPTICO Objeto de la práctica El objeto de la práctica es el uso del microscopio y las observaciones que se pueden hacer con el mismo. Materiales utilizados El microscopio.
Prácticas de Laboratorio EL MICROSCOPIO ÓPTICO Objeto de la práctica El objeto de la práctica es el uso del microscopio y las observaciones que se pueden hacer con el mismo. Materiales utilizados El microscopio.
NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN
 NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN 1. PROCEDIMIENTOS BÁSICOS: En este apartado pretendemos detallar algunos factores muy importantes a la hora de realizar
NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN 1. PROCEDIMIENTOS BÁSICOS: En este apartado pretendemos detallar algunos factores muy importantes a la hora de realizar
DESTILACIÓN. DETERMINACIÓN DEL GRADO ALCOHÓLICO DEL VINO
 1. INTRODUCCION La destilación es un proceso que consiste en calentar un líquido hasta que sus componentes más volátiles pasan a la fase de vapor y, a continuación, enfriar el vapor para recuperar dichos
1. INTRODUCCION La destilación es un proceso que consiste en calentar un líquido hasta que sus componentes más volátiles pasan a la fase de vapor y, a continuación, enfriar el vapor para recuperar dichos
La PCR. La Reacción en Cadena de la Polimerasa es la herramienta más utilizada
 La PCR La Reacción en Cadena de la Polimerasa es la herramienta más utilizada PCR- Ventajas y Usos Amplificación ilimitada de secuencias de interés. Rápida, Sencilla y Eficaz. Técnica altamente sensible
La PCR La Reacción en Cadena de la Polimerasa es la herramienta más utilizada PCR- Ventajas y Usos Amplificación ilimitada de secuencias de interés. Rápida, Sencilla y Eficaz. Técnica altamente sensible
MÓDULO: GESTIÓN DE RESIDUOS TEMA: DESMINERALIZACIÓN
 MÓDULO: GESTIÓN DE RESIDUOS TEMA: DESMINERALIZACIÓN DOCUMENTACIÓN ELABORADA POR: NIEVES CIFUENTES MASTER EN INGENIERIÁ MEDIOAMBIENTAL Y GESTIÓN DEL AGUA ÍNDICE 1. INTRODUCCIÓN 2. INTERCAMBIO IÓNICO 3.
MÓDULO: GESTIÓN DE RESIDUOS TEMA: DESMINERALIZACIÓN DOCUMENTACIÓN ELABORADA POR: NIEVES CIFUENTES MASTER EN INGENIERIÁ MEDIOAMBIENTAL Y GESTIÓN DEL AGUA ÍNDICE 1. INTRODUCCIÓN 2. INTERCAMBIO IÓNICO 3.
Grupo Nodrissa. Marta Bacardit Abellán EXTRACCIÓN, ALMACENAMIENTO Y CONSERVACIÓN DE LA LECHE MATERNA
 Grupo Nodrissa. Marta Bacardit Abellán EXTRACCIÓN, ALMACENAMIENTO Y CONSERVACIÓN DE LA LECHE MATERNA 1. Extracción de la leche materna Motivos de extracción: Incorporación al trabajo. Para estimular la
Grupo Nodrissa. Marta Bacardit Abellán EXTRACCIÓN, ALMACENAMIENTO Y CONSERVACIÓN DE LA LECHE MATERNA 1. Extracción de la leche materna Motivos de extracción: Incorporación al trabajo. Para estimular la
Actividad: Qué es la anomalía del agua?
 Nivel: 1º Medio Subsector: Ciencias químicas Unidad temática: El agua Actividad: Seguramente ya has escuchado sobre la anomalía del agua. Sabes en qué consiste y qué es algo anómalo? Se dice que algo es
Nivel: 1º Medio Subsector: Ciencias químicas Unidad temática: El agua Actividad: Seguramente ya has escuchado sobre la anomalía del agua. Sabes en qué consiste y qué es algo anómalo? Se dice que algo es
IES Menéndez Tolosa 3º ESO (Física y Química)
 IES Menéndez Tolosa 3º ESO (Física y Química) 1 De las siguientes mezclas, cuál no es heterogénea? a) azúcar y serrín. b) agua y aceite. c) agua y vino d) arena y grava. La c) es una mezcla homogénea.
IES Menéndez Tolosa 3º ESO (Física y Química) 1 De las siguientes mezclas, cuál no es heterogénea? a) azúcar y serrín. b) agua y aceite. c) agua y vino d) arena y grava. La c) es una mezcla homogénea.
INGENIERÍA GENÉTICA 5 GAATTC 3 3 CTTAAG 5
 INGENIERÍA GENÉTICA 1. Fundamentos básicos de la ingeniería genética 2. Desnaturalización e hibridación del ADN 3. Reacción en cadena de la polimerasa (PCR) 4. Nuevas disciplinas surgidas de la ingeniería
INGENIERÍA GENÉTICA 1. Fundamentos básicos de la ingeniería genética 2. Desnaturalización e hibridación del ADN 3. Reacción en cadena de la polimerasa (PCR) 4. Nuevas disciplinas surgidas de la ingeniería
TEMA 3: EN QUÉ CONSISTE?
 Módulo 7 Sesión 3 5/16 TEMA 3: EN QUÉ CONSISTE? La metodología seguida para aplicar correctamente la técnica de RGT se basa en cuatro fases (Figura 1). En la primera de ellas, se seleccionan los elementos
Módulo 7 Sesión 3 5/16 TEMA 3: EN QUÉ CONSISTE? La metodología seguida para aplicar correctamente la técnica de RGT se basa en cuatro fases (Figura 1). En la primera de ellas, se seleccionan los elementos
FACULTAD REGIONAL ROSARIO
 FACULTAD REGIONAL ROSARIO CATEDRA BIOTECNOLOGIA ROFESOR: Ing. Eduardo Santambrosio JTP: Ing. Marta Ortega AUX 1ª : Ing. Pablo Garibaldi PCR (Polymerase chain reaction) Reacción en cadena de la polimerasa
FACULTAD REGIONAL ROSARIO CATEDRA BIOTECNOLOGIA ROFESOR: Ing. Eduardo Santambrosio JTP: Ing. Marta Ortega AUX 1ª : Ing. Pablo Garibaldi PCR (Polymerase chain reaction) Reacción en cadena de la polimerasa
Introducción al diseño híbrido con ZW3D
 Introducción al diseño híbrido con ZW3D Con este tutorial podrá aprender el diseño 3D con un programa CAD 3D híbrido de modelado de sólidos y superficies combinadas. El objetivo es dibujar un grifo en
Introducción al diseño híbrido con ZW3D Con este tutorial podrá aprender el diseño 3D con un programa CAD 3D híbrido de modelado de sólidos y superficies combinadas. El objetivo es dibujar un grifo en
