2. MATERIAL Y MÉTODOS
|
|
|
- Carlos Toledo Robles
- hace 8 años
- Vistas:
Transcripción
1 39 2. MATERIAL Y MÉTODOS
2
3 Material y métodos CEPAS BACTERIANAS Y PLÁSMIDOS Las cepas bacterianas utilizadas en este trabajo se detallan en las tablas y 2.1.2, en su mayoría pertenecientes a Salmonella enterica serovar Typhimurium: Tabla Cepas bacterianas de Salmonella enterica serovar Typhimurium utilizadas en este trabajo. Cepas Genotipo relevante Referencia/Origen LT2 cepa salvaje Lilleengen, 1948 TT1704 his-9533 Torreblanca y Casadesús, 1996 SV3081 pslt - (isogénica de LT2) Torreblanca et al., 1999 YFER TT1704 yfer::mudj Prenafeta, 1999 YFER2 TT1704 yfer - Este trabajo YFERYGDP TT1704 yfer::mudj ygdp::tn10dcam Prenafeta, 1999 YGDP TT1704 ygdp::tn10dcam Polo, 2000 C52 rpos::cam Kowarz et al., 1994 TTRPOS TT1704 rpos::cam Este trabajo YFEH TT1704 yfeh - Este trabajo cepa salvaje ATCC SV4280 lrp - J. Casadesús Tabla Cepas de Escherichia coli utilizadas en este trabajo. Cepas Genotipo relevante Referencia HB101 supe44, supf58, hsds3(r - B m - B ), reca13, ara- Sambroock et al., , proa2, lacy1, galk2, rpsl20, xyl-5, mtl-1 BL21(DE3) plyse; hsds, gal, lcits857, ind1, Sam7, Nin5, Studier y Moffatt, 1986 lacuv5-t7gene1
4 Material y métodos 42 A continuación se describen las características de los plásmidos (tabla 2.1.3) y bacteriófagos (tabla 2.1.4) utilizados en este trabajo: Tabla Plásmidos utilizados en la realización de este trabajo. Plásmido Descripición Referencia pbr322 ori pmb1 ; Ap r, Tc r Bolívar et al., 1977 plg ori psc101 ; Ap r Cunningham et al., 1993 puc19 ori ColE1 ; Ap r Yanisch-Perron et al., 1985 pmc391 lacz' Casadaban et al., 1980 pkd46 orir101, repa101 (ts), arabp-gam-bet-exo (Red helper plasmid, Ts; Ap r ) Datsenko y Wanner, 2000 pkd4 orirγ; Km r, Ap r Datsenko y Wanner, 2000 pcp20 λci857 (ts), ts-rep (Recombinasa FLP, Ts) Cheperanov y Wackernagel, 1995 pet3b Promotor de T7, Ap r Studier et al.,1990 petyfer pet3b + yfer, Ap r Este trabajo petyfer r pet3b + cadena complementaria al ARN m de yfer, Ap r Este trabajo pann pacyc184 + hlyc, hlya, hlyb,hlyd; Cm r Godessart et al.,1988 plgyfer plg yfer, Ap r Este trabajo plgyfehlac plg yfeh::lacz, Ap r plgyfehcat plg yfeh::cat, Ap r Este trabajo Este trabajo placp pla2917 +yfer, Tc r ; Kn r Polo, 2000 pucir puc19 + yfeh + región intergénica yferyfeh + extremo 5 MudJ Este trabajo pko3 Cm r, repa (ts), sacb Link et al., 1997 pkyfeh pko3 + yfeh mutado Este trabajo Tabla Bacteriófagos utilizados en experimentos de transducción generalizada. Bacteriófago Descripción Referencia P22 HT int4 int4 Schmieger, 1972 P22 H5 c2 Torreblanca y Casadesús, 1996
5 Material y métodos MEDIOS DE CULTIVO Y ANTIBIÓTICOS MEDIOS DE CULTIVO Se utilizaron diferentes medios de cultivo líquidos y sólidos, cuya composición y preparación se detallan a continuación: LB (Luria-Bertani; Sambrook et al., 1989): medio de cultivo líquido utilizado de forma habitual para el crecimiento bacteriano. En algunos experimentos se modificó la osmolaridad del medio cambiando la concentración de NaCl. Si en condiciones normales la molaridad del NaCl es de 170 mm se aumentó a 0.5 M (29.22g/l) o bien se disminuyó a 0 M. LB g/l Peptona trípsica de caseína (Schärlau Microbiology) 10 Extracto de levadura (Schärlau Microbiology) 5 NaCl (Panreac) 10 LB agar: es el medio de cultivo sólido utilizado de forma habitual y consiste en añadir 15 g/l de agar para bacteriología (Schärlau Microbiology) al medio LB arriba especificado. LB agar blando: medio de cultivo semisólido utilizado para la titulación de lisados fágicos al permitir el crecimiento en masa de la bacteria. En este caso al medio LB se le añaden de 6-8 g/l de agar para bacteriología (Schärlau Microbiology). EBU: medio sólido utilizado para la obtención de clones libres de pseudolisógenos después de una transducción generalizada con el fago P22. Consiste en el medio LB agar que una vez esterilizado se suplementa el resto de componentes abajo indicados, todos ellos esterilizados por filtración.
6 Material y métodos 44 EBU LB AGAR Glucosa (Panreac) 0,25% (p/v) K 2 HPO 4 (Merck) 0,25% (p/v) Azul de Evans (Sigma) 0,0125 g/l Fluoresceína (Uranina, Sigma) 0,0250 g/l E y NCE: los medios E (Vogel y Bonner, 1956) y NCE (Berkowitz et al., 1968) se usaron en su calidad de medios mínimos. La composición y preparación de ambos medios es prácticamente la misma, salvo que el medio E contiene citrato, fuente de carbono utilizable por Salmonella, además de la glucosa, que es la fuente de carbono habitual. El medio NCE no contiene citrato, por lo que es necesaria una variación en la composición de sales para que el ph sea neutro. Tanto el medio E como el NCE se preparan como soluciones de sales 50 veces concentradas, que se esterilizan con cloroformo. En el momento de preparar el medio se diluyen 50 veces en agua bidestilada estéril para el medio líquido, y en agua con agar 15 g/l para el medio sólido. Posteriormente se añade la fuente de carbono a una concentración de 2 g/l a partir de una solución madre estéril. En el medio NCE el MgSO 4 también se añade a posteriori a partir de una solución madre al 20% (p/v). Como la cepa utilizada mayoritariamente en este trabajo es la TT1704, o cepas isogénicas derivadas de la misma, y la cepa TT1704 es auxótrofa para la histidina, se añadieron 4ml /l de una solución madre de este aminoácido a 10 mg/ml (Miller, 1992). Sales minerales del medio E MgSO 4 (Merck) 0,8 mm Ácido cítrico (Merck) 9,5 mm K 2 HPO 4 (Merck) 57 mm NaNH 4 HPO 4 (Merck) 17 mm Sales minerales del medio NCE KH 2 PO 4 (Merck) 29 mm K 2 HPO 4 (Merck) 28 mm NaNH 4 HPO 4 (Merck) 17 mm MS (Spector y Cubbitt, 1992): medio mínimo resultado de una ligera modificación del medio MOPS (Neidhart et al., 1974), en el que sus componentes están perfectamente controlados, por lo que se puede utilizar para limitar la concentración de determinados nutrientes como el fósforo, el nitrógeno o la glucosa. Este medio se preparó
7 Material y métodos veces concentrado mezclando las soluciones en el orden indicado en la tabla para evitar la precipitación de las sales. El medio se esterilizó por filtración y posteriormente se guardó congelado a -20 º C hasta su utilización. MS MOPS (Sigma) 1M ajustado a ph 7,4 con KOH Tricina (Sigma) 1M ajustada a ph 7,4 con KOH FeSO 4 0,01 M K 2 SO 4 0,276 M CaCl 2 0,5 mm MgCl 2 0,528 M NaCl 5 M Solución de micronutrientes H 2 O bidestilada 1l 400 ml 40 ml 10 ml 10 ml 10 ml 10 ml 100 ml 10 ml 360 ml Solución de micronutrientes (NH 4 ) 6 (MO 7 ) M H 3 BO M CoCl M CuSO M MnCl M ZnSO M El medio 1 x se preparó por dilución de esta solución en agua bidestilada estéril, y se suplementó con cantidades no limitantes de fuentes de fósforo, carbono y nitrógeno (medio MShiPCN, MS high P,C,N ), a una concentración final de: 15 mm KH 2 PO 4 / K 2 HPO 4 (ph 7,4), 0,4% glucosa (p/v) y 15 mm de NH 4 Cl, respectivamente. Cuando se quiso conseguir cantidades limitantes de estos nutrientes la concentración final fue la siguiente: 0,1125 mm KH 2 PO 4 /K 2 HPO 4 (ph 7,4) (MSloP, MS low P ), 0,04% glucosa (p/v) (MSloC, MS low C ), y 0,6 mm de NH 4 Cl (MSloN, MS low N ). Tal y como ya se ha comentado con el medio mínimo E se suplementó el medio con histidina.
8 Material y métodos 46 Caldo de fagos: su composición es la del medio NB (Nutrient Broth, Difco) más 5 g/l de NaCl, sales del medio mínimo E y glucosa al 0,2% (p/v). Para utilizarlo en la obtención de lisados de S. Typhimurium se añaden 0,1 ml de un lisado de P22 crecido en la cepa TT1704 por cada 100 ml de caldo de fagos. SOB (Hanahan et al., 1991): Medio de cultivo líquido utilizado para el crecimiento bacteriano en la mutagénesis de inactivación de genes cromosómicos utilizando fragmentos de PCR (apartado 2.4.1). SOB Peptona trípsica de caseína (Schärlau Microbiology) Extracto de levadura (Schärlau Microbiology) NaCl (Panreac) KCl (Merck) * MgCl 2 + MgSO 4 20 g/l 5 g/l 0,58 g/l 0,19 g/l 20 mm *A partir de una solución madre Mg 2+ 2M (MgCl 2 1 M + MgSO 4 1 M). SOC (Hanahan et al., 1991): Se utiliza como medio de recuperación de las células después de la electroporación en la mutagénesis de genes cromosómicos utilizando fragmentos de PCR (apartado 2.4.1). Se trata del medio SOB suplementado con glucosa 20 mm ANTIBIÓTICOS Cuando fue necesario, los diferentes medios de cultivo anteriormente citados se suplementaron con antibióticos. Estos antibióticos se añadieron al medio estéril a partir de una solución concentrada conservada a -20ºC preparada siguiendo las instrucciones de la bibliografía (Sambrook et al., 1989). En la tabla se muestran las concentraciones finales a las que fueron utilizadas los antibióticos, según la localización del gen de la resistencia o el medio de cultivo. Ampicilina (Ap) (sal sódica, Roche): se preparó una solución concentrada a 100 mg/ml en agua bidestilada y se esterilizó por filtración.
9 Material y métodos 47 Cloranfenicol (Cm) (Fluka): se preparó a una concentración de 100 mg/ml en Etanol absoluto (Prolabo). Estreptomicina (Sm) (Sulfato, Sigma): se preparó a 100 mg/ml en agua bidestilada y se esterilizó por filtración. Kanamicina (Km) (Sulfato ácido, Sigma): la solución concentrada se preparó a 50 mg/ml en agua bidestilada y se esterilizó por filtración. Tetraciclina (Tc) (Hidrocloruro, Fluka): la solución madre se preparó a 12,5 mg/ml en etanol (absoluto) al 50% (v/v), se esterilizó por filtración y se guardó protegida de la luz. Rifampicina (Rf) (Fluka): se preparó a 50 mg/ml en agua bidestilada añadiendo unas gotitas de NaOH 2N hasta su completa disolución. Se esterilizó por filtración pero no se guardó más de dos semanas una vez preparada. Tabla Concentraciones de los antibióticos. Concentración en el medio (µg/ml) ANTIBIÓTICO Medio rico Medio mínimo Ampicilina 25 a - (50-100) b 15 Cloranfenicol 12,5 a -50 b - c Estreptomicina 200 a - c Kanamicina 50 a 125 Rifampicina 75 a - c Tetraciclina 15 a 10 a Para resistencias cromosómicas b Para resistencias contenidas en plásmidos de medio-elevado número de copias. c No utilizado en esta tesis.
10 Material y métodos MÉTODOS DE TRANSFERENCIA GENÉTICA TRANSFORMACIÓN BACTERIANA Para llevar a cabo la transformación de células bacterianas se utilizaron dos métodos diferentes. Normalmente se llevó a cabo la transformación con CaCl 2, pero en algunos casos, especialmente cuando la eficiencia de transformación fue baja, se recurrió a la electroporación Transformación de células competentes obtenidas mediante tratamiento con CaCl 2 (Cohen et al., 1972) Obtención de células competentes de E. coli: a partir de un inóculo crecido durante toda la noche en LB se hizo una dilución 1:50 en medio de cultivo fresco, y se incubó a 37ºC hasta fase exponencial temprana, es decir, hasta una absorbancia de 0,2-0,3 a 600 nm. Se incubó un volumen de cultivo 10 minutos en hielo, y se centrifugó a 3000 x g durante 10 minutos y a una temperatura de 4ºC. A continuación se resuspendieron las células en la mitad de volumen de CaCl 2 50 mm frío, y se centrifugaron de nuevo en las mismas condiciones pero sólo durante 5 minutos. Finalmente se resuspendieron en un volumen de CaCl 2 que representaba 1/20 parte del volumen inicial. Esta suspensión se incubó en hielo desde un mínimo de 30 minutos hasta un máximo de 24 horas antes de la transformación. Obtención de células competentes de Salmonella: esta bacteria presenta una eficiencia de transformación por CaCl 2 50 mm menor que E. coli, por lo que se modificó ligeramente el protocolo de transformación. Si con E. coli el volumen final de resuspensión fue de 1/20 del volumen inicial, en este caso fue de 1/100 del volumen inicial, de forma que las células quedaron mucho más concentradas. En los dos casos alícuotas de 100 µl de células competentes se mezclaron con 1-10 µl de solución de ADN plasmídico. Esta mezcla se incubó 30 minutos en hielo y a continuación se las sometió a un choque térmico a 42ºC durante 40 segundos. Se añadió medio de cultivo LB fresco hasta un volumen de 1 ml y se dejó incubar a 37ºC durante una hora para permitir la expresión de los marcadores fenotípicos transferidos
11 Material y métodos 49 (mayoritariamente genes de resistencia a antibióticos), que permitieron seleccionar las células que adquirieron el plásmido al sembrar en medio sólido con el agente selectivo Electroporación (Dower et al., 1988) Este método permite la captación de ADN mediante la permeabilización de las membranas provocada por una descarga eléctrica. Con este método la eficacia de la transformación aumenta considerablemente, obteniendo entre transformantes por µg de ADN. A partir de un inóculo crecido toda la noche se realiza una dilución 1:50 en medio LB fresco y se incuba a 37ºC hasta una DO a 600 nm de 0,5. Después de enfriar el cultivo en hielo, partiendo de un volumen de 10 ml, se realizan varias centrifugaciones sucesivas a 3000 x g y 4ºC, en las que se va disminuyendo el volumen al resuspender en 1, 0,5, 0,1 y finalmente 0,005 volúmenes de glicerol 10%. De esta forma se disminuye la fuerza iónica de la suspensión celular, al limpiar las sales. Las células se mantienen en hielo hasta el momento de la electroporación: se mezclan 50 µl de células con 1-5 µl de la suspensión que contiene el ADN y a continuación se transfiere a cubetas esterilizadas con luz ultravioleta. El electroporador utilizado en esta tesis fue el Electro Cell Manipulator 600 (BTX Electroporation System), con las condiciones de electroporación que se muestran a continuación: Condiciones de electroporación Temperatura 4ºC Modo 2,5 kv / Resistencia de alto voltaje Capacitancia No utilizar en modo de alto voltaje Resistencia R5 (129 Ω) Voltaje de la descarga 2 kv Fuerza del campo eléctrico aplicado 10 kv / cm (máximo) Duración del pulso 5 ms Una vez realizada la descarga eléctrica se añadió medio LB fresco hasta completar a 1 ml de volumen y se incubó a 37ºC durante una hora para permitir la expresión de los
12 Material y métodos 50 genes de resistencia a antibióticos. Las células transformadas se seleccionaron en medio sólido suplementado con los marcadores correspondientes TRANSDUCCIÓN CON EL BACTERIÓFAGO P22 El bacteriófago P22 HT int4 (Schmieger, 1972) se utilizó en este trabajo para introducir mutaciones en las cepas por recombinación homóloga. Este fago P22 presenta dos mutaciones, HT e int, que le confieren respectivamente una elevada frecuencia de transducción, y una deficiencia en la integración del DNA del bacteriófago en el cromosoma de la cepa receptora, evitando así la lisogenia Obtención de lisados de P22 Un cultivo de toda la noche de la cepa dadora (0,5 ml) se mezcló con 2 ml de caldo de P22 (apartado 2.2.1), y se incubó a 37ºC de 8 a 16 h en agitación a 200 rpm. Aunque durante esta incubación se produce lisis celular la turbidez del cultivo no disminuye. Posteriormente se centrifugó la mezcla (3000 x g, 25 minutos) para eliminar las células. Se recogió el sobrenadante, donde quedan las partículas fágicas, y después de añadir unas gotas de cloroformo, agitar vigorosamente en un vórtex e incubar a temperatura ambiente durante dos horas, se guardó a 4ºC hasta su utilización Titulación del lisado fágico Se mezclaron 200 µl de un cultivo de la cepa hospedadora en fase exponencial (DO 600nm 0,3-0,4), con 100 µl de diluciones seriadas del lisado fágico en MgSO 4 10 mm. Estas mezclas se incubaron a 37ºC durante 20 minutos para permitir la adsorción del fago. Acto seguido se les añadieron 2,5 ml de LB agar blando (mantenido líquido a 55ºC), vertiéndolo todo rápidamente después de mezclar sobre una placa de LB, que se incubó a 37ºC hasta la aparición de calvas de lisis.
13 Material y métodos Transducción con P22 Las transducciones con P22 llevadas a cabo en este trabajo se realizaron en medio líquido. Consistió en mezclar en un tubo de 1,5 ml volúmenes iguales de un cultivo de toda la noche de la cepa receptora y del lisado fágico obtenido sobre la cepa dadora o diluciones del mismo en MgSO 4 10 mm (normalmente 10-1 y 10-2 ). A continuación se incubaron a 37ºC sin agitación durante 30 minutos para permitir la infección del fago, y se sembraron en placas de LB agar conteniendo los marcadores de resistencia a antibióticos transferidos por recombinación homóloga. Es posible que en el proceso de transducción una célula transductante sea infectada de nuevo por el fago P22, dando como resultado un pseudolisógeno, y aunque éste sea deficiente para sus funciones de integración en el cromosoma, puede pasar en este caso que el DNA de P22 se acabe integrando. Estas cepas se vuelven resistentes a subsiguientes infecciones con el fago P22, por lo que no se podrán utilizar en experimentos de transducción. Por esto, después de una transducción con P22 fue necesaria una limpieza de pseudolisógenos, que se realizó sembrando en placas EBU. En estas placas se pueden distinguir las colonias con psedulisógenos, que tienen un color verde oscuro, de las que están libres de ellos, de color verde más claro. El color verde oscuro es debido a la lisis por P22 que libera productos ácidos, haciendo virar un indicador de ph que contiene el medio. Así, se siembran dos o tres estrías sucesivas a partir de colonias verde claras. Tras el paso por placas EBU, se realizó una última prueba para comprobar que los transductantes estuvieran totalmente libres de pseudolisógenos, sometiéndolos a un test de sensibilidad al bacteriófago P22 H5. La mutación en el gen c2 que presenta este derivado virulento de P22 hace que produzca calvas de lisis claras. Para este ensayo se realizó una estría a partir de un lisado de P22 H5 en una placa de LB. En esta misma placa se sembraron la cepa TT1704, como control, y la cepa transductante, haciendo dos estrías paralelas que cruzasen perpendicularmente la estría de P22 H5. Después de incubar la placa h a 37ºC, se observa como las cepas sensibles a la infección, y por lo tanto libres de pseudolisógenos, crecen solamente hasta llegar a la estría de P22 H5, mientras que las cepas con P22 integrado son resistentes a la infección y pueden crecer cruzando la estría de P22 H5.
14 Material y métodos TÉCNICAS DE MUTAGÉNESIS BACTERIANA INACTIVACIÓN DE GENES CROMOSÓMICOS UTILIZANDO FRAGMENTOS DE PCR (Datsenko y Wanner, 2000) Con esta metodología es posible reemplazar una secuencia cromosómica por un gen de resistencia a antibióticos, generado por PCR, utilizando oligonucleótidos con extensiones homólogas correspondientes al gen a delecionar. Estas extensiones permitirán la recombinación mediante la recombinasa Red en las regiones flanqueantes al gen. Después de la selección de mutantes, la resistencia al antibiótico puede ser eliminada utilizando un plásmido auxiliar que expresa la recombinasa FLP, actuando sobre la repetición directa FRT adyacente al gen de la resistencia Generación del framento de PCR La resistencia a Km del plásmido pkd4 se amplificó utilizando oligonucleótidos que en su extremo 5 contenían secuencias homólogas (de entre 35 y 50 nucleótidos) al gen objeto de deleción (H1 y H2), más la secuencia correspondiente a P1 y P2 del plásmido PKD4 (figura 2.4.1). En total los oligonucleótidos midieron entre 56 y 70 nucleótidos. De esta forma, al amplificar la resistencia a Km del plásmido pkd4, se genera un fragmento de PCR que contiene la resistencia al antibiótico flanqueada por los sitios FRT, las secuencias P1 y P2, más las secuencias homólogas al gen que se quiere delecionar. Después de la amplificación el fragmento se purificó por electroelución, y posteriormente se digerió con DpnI para eliminar el ADN molde utilizado en la PCR, ya que este enzima digiere selectivamente el ADN metilado Transformación del plásmido pkd46 El plásmido pkd46 codifica para la recombinasa Red del fago λ bajo el control de un promotor inducible por L-arabinosa, y al tener un origen de replicación termosensible se cura fácilmente a 43ºC. La recombinsa Red, además de favorecer la recombinación inhibe a la exonucleasa V, permitiendo así la entrada a la célula de un fragmento de PCR sin que
15 Material y métodos 53 este sea digerido. El plásmido portador de la recombinasa es resistente a ampicilina (100 µg/ml). Se hizo crecer a la cepa objeto de la mutagénesis, transformada previamente con el plásmido pkd46, a 30ºC en medio SOB suplementado con ampicilina y L-arabinosa 10 mm (Sigma) hasta una DO 600 =0,6. Cuando la cepa llegó a este punto del crecimiento se obtuvieron células electrocompetentes por centrifugación (3000 x g, 4ºC) y lavados con glicerol 10 % (v/v) frío, concentrando las células x 100. Alícuotas de 25 µl de células electrocompetentes se electroporaron con ng de producto de PCR. Una vez sometidas las células al pulso eléctrico se resuspendieron en 1 ml de medio SOC y se incubaron durante 1 hora a 37ºC para favorecer la recombinación y permitir la expresión de la resistencia al antibiótico. La selección de los mutantes se realizó sembrando la mitad de las células en medio sólido LB agar suplementado con kanamicina. El resto de transformantes se incubaron a temperatura ambiente entre horas y se sembraron pasado este tiempo en el mismo medio. Las placas se incubaron a 37ºC 48 horas. Una vez obtenida alguna colonia posible mutante se realizaron amplificaciones por PCR con distintas parejas de oligonucleótidos para comprobar la inserción del antibiótico en el gen en cuestión. Para eliminar el plásmido pkd46 se realizaron estrías en medio sólido sin el antibiótico a 43ºC. En la figura se muestra un esquema del proceso Eliminación de la resistencia al antibiótico Una vez comprobada la mutación por PCR, se transformó con el plásmido pcp20, que codifica para la recombinasa FLP inducible por temperatura (43ºC), resistente a ampicilina y cloranfenicol, y con origen de replicación termosensible. La transformación se sembró en medio sólido con Ap a 30ºC, y se incubó a esta temperatura durante 16 horas. Los transformantes obtenidos se sembraron en medio sólido no selectivo a 43ºC para favorecer la pérdida del plásmido, ya que la mayoría de pérdidas de la resistencia flanqueada por sitios FRT es simultánea a la pérdida del plásmido pcp20. Aún así, se hicieron sucesivas estrías a 43ºC en medio sólido para asegurarnos que realmente se había perdido el plásmido. Para finalizar, se comprobó por PCR que la resistencia a Km del plásmido pkd4 se había perdido y que el gen original estaba delecionado.
16 Material y métodos 54 Paso 1: amplificación por PCR gen resistencia flanqueado por sitios FRT Paso 2: transformación fragmento de PCR a la cepa con plásmido pkd46 Paso 3: selección de los transformantes resistentes al antibiótico Paso 4: eliminación reistencia antibiótico Fig Esquema del proceso de mutagénesis. H1 y H2 se refieren a las regiones de homología del gen, y P1 y P2 a las regiones de homología con el plámido pkd MUTAGÉNESIS DIRIGIDA POR DOBLE RECOMBINACIÓN Este método se utilizó para generar mutaciones cromosómicas en pauta de lectura, evitando así los posibles efectos polares en los genes adyacentes que pueden causar los mutantes por inserción. El sistema utilizado se basó en el plásmido pko3 descrito por Link et al., (1997). El método consistió en sintetizar por PCR una copia del gen que llevara una deleción y al mismo tiempo mantuviese la pauta de lectura. Para ello se diseñaron dos parejas de oligonucleótidos: la pareja A y B amplificó la región inmediatamente anterior al gen y los 5-10 primeros tripletes completos de éste, mientras que la pareja C y D hizo lo mismo pero para el final del gen. Además, los oligonucleótidos B y C fueron diseñados con una cola de 21 pares de bases complementarias que no existían en el gen. Fueron realizadas dos PCRs asimétricas con las parejas A-B y C-D, respectivamente. En estas reacciones de PCR la relación de concentración de los oligonucleótidos era de 10:1: 600 nm los externos (A-D) y 60 nm los internos (B-C). Los productos amplificados fueron utilizados como molde para una última PCR con los oligonucleótidos A y D, de tal forma que se generó un fragmento de PCR con la región anterior al gen a mutar, varios tripletes iniciales del gen, una región de 21 pares de bases aleatorias (que mantenía la pauta de
17 Material y métodos 55 lectura), varios tripletes finales del gen y la región posterior del gen a mutar. En la figura se muestra el proceso de deleción del gen a partir de las reacciones de PCR. Mezclar PCR1 y PCR2 para molde con oligonucleótidos A y D Clonaje en un vector pko3 para sustitución génica Fig Esquema del proceso de deleción del gen utilizando como molde el producto de dos PCRs asimétricas (PCR1+PCR2). Se muestra en color azul las regiones complementarias de los oligonucleótidos B y C, que posteriormente crearán la deleción en pauta del gen objeto de mutagénesis. Las líneas de color verde representan los extremos de los oligonucleótidos A y D diseñados con dianas de restricción para su posterior clonaje en pko3. Esta construcción fue clonada en el plásmido pko3 y se transformó a la cepa objeto de la mutagénesis. La transformación se incubó en LB agar suplementado con cloranfenicol (resistencia del plásmido) a 42ºC, ya que ésta es una temperatura no permisiva de crecimiento para el plásmido. De esta forma las colonias que crecieran a esta temperatura sólo podían ser resultado de una integración del plásmido en el cromosoma por recombinación homóloga. Posteriormente estas colonias se hicieron crecer en LB agar suplementado con sacarosa al 5% (p/v) y se incubó a 30ºC (Blomfield et al., 1991). El plásmido pko3 contiene el gen sacb, que es letal para la bacteria cuando crece en sacarosa. De esta forma las colonias que crecieran a 30ºC y fueran sensibles al cloranfenicol sólo podían ser el resultado de una segunda recombinación homóloga que eliminara el plásmido del cromosoma. Para finalizar, se realizó una PCR para saber si la copia del gen que quedó en el cromosoma bacteriano era la salvaje o la delecionada. Además, la región delecionada fue secuenciada para comprobar que se mantuviera en pauta. En la figura se muestra el esquema del proceso de mutagénesis una vez clonado el gen con la deleción en el plásmido pko3.
18 Material y métodos 56 Transformar cepa objeto de mutagénesis con plásmido pko3 3 con secuencia del gen delecionada in vitro. Seleccionar la integración del plásmido a 42ºC con cloranfenicol Resembrar a 30ºC y seleccionar la pérdida del plásmido Sembrar en réplica para buscar colonias reistentes a sacarosa y sensibles a cloranfenicol sacb mutado? Fig Protocolo utilizado para sustituir secuencias salvajes en el cromosoma con secuencias alteradas in vitro. La línea delgada curvada representa la secuencia del vector pko3; la línea delgada recta la secuencia del cromosoma bacteriano; las cajas representan la secuencia homóloga clonada en el vector (vacía) y localizada en el cromosoma (rayada). La caja negra dentro de la secuencia homóloga clonada en el vector indica la deleción introducida in vitro en el gen objeto de la mutagénesis. 2.5 TÉCNICAS DE ANÁLISIS DE ÁCIDOS NUCLEICOS AISLAMIENTO DE ADN PLASMÍDICO Fueron utilizadas diferentes técnicas, dependiendo de la finalidad que se le quisiera dar al plásmido aislado Aislamiento de ADN plasmídico por lisis alcalina (Birnboim, 1983) Éste fue el método utilizado de forma habitual. El volumen de cultivo bacteriano del que se partió varió en función del ADN plasmídico requerido. El protocolo detallado
19 Material y métodos 57 está diseñado para un cultivo de 5 ml, pero se pueden escalar los volúmenes de las soluciones si partimos de otro volumen de cultivo. Se centrifugaron 5 ml de un cultivo de toda la noche (3000 x g, 10 minutos) y se resuspendieron las células en 0,2 ml de solución I. Tras 15 minutos en hielo se le añadieron 0,4 ml de solución II, se mezcló por inversión y se incubó 10 minutos en hielo. Se añadieron 0,3 ml de solución III, se mezcló de nuevo por inversión y se dejó 15 minutos en hielo. Esta solución se centrifugó ( x g, 15 minutos, 4ºC), y se recogió el sobrenadante, que contenía el ADN plasmídico. A continuación se llevó a cabo una desproteinización del mismo, para la cual se utilizaron dos métodos diferentes: por precipitación con cloruro de litio o por extracción con fenol. El mismo protocolo se adaptó para la extracción de ADN plasmídico de volúmenes más grandes, cambiando los volúmenes de soluciones I, II, y III, pero siempre manteniendo las proporciones entre ellas (1:2:1,5). SOLUCIÓN I Glucosa (Panreac) 55 mm EDTA (Merck) 10 mm Tris (Roche) 25 mm Ajustar a ph 8 SOLUCIÓN II NaOH (Panreac) 0,2 M SDS (Merck) 1% SOLUCIÓN III Acetato sódico (Merck) 3 M Ajustar a ph 4,8 con ácido acético glacial (Panreac) Desproteinización del ADN por precipitación con cloruro de litio (Martínez y de la Cruz, 1988) Este protocolo fue seguido cuando se aisló ADN plasmídico de cultivos de 5 y 10 ml. Al sobrenadante resultante de la lisis alcalina se le añadió el mismo volumen de isopropanol y se incubó en hielo durante 10 minutos. Se centrifugó (12000 x g, temperatura ambiente, 5 minutos), se eliminó el sobrenadante y el precipitado se
20 Material y métodos 58 resuspendió en 0,2 ml de TE (Tris-HCl 10 mm, EDTA 1mM, ph 8). En este punto se realizó el tratamiento con RNasa A para eliminar posibles restos de ARN en la muestra (apartado ). A la solución libre de ARN se le añadieron 2 volúmenes de LiCl 5 M (Merck) y otros 2 volúmenes de cloroformo, se agitó vigorosamente, se centrifugó ( x g, temperatura ambiente, 15 minutos) y se recuperó el sobrenadante. Seguidamente se precipitó el ADN añadiéndole 1/10 parte del volumen de solución III y 2 volúmenes de etanol absoluto frío, y se incubó como mínimo 1 hora a -20ºC o 30 minutos a -70ºC. A continuación se centrifugó el ADN ( x g, 4ºC, 15 minutos), se eliminó el sobrenadante y el precipitado se lavó con etanol al 70% preenfriado a -20ºC para eliminar restos de sales. Finalmente, el precipitado que contenía el ADN plasmídico se secó utilizando un secador de vacío (Speed-Vac, Savant) y se resuspendió en el volumen adecuado de agua bidestilada o TE Desproteinización del ADN por extracción con fenol El ADN plasmídico resultante de la lisis alcalina se concentró por precipitación, añadiéndole dos volúmenes de etanol absoluto frío e incubándolo, como mínimo a -20ºC durante 1 hora, o a -70ºC 30 minutos. A continuación se centrifugó ( x g, 4ºC, 15 minutos) y se eliminó el sobrenadante. El precipitado se secó utilizando un secador de vacío (Speed-Vac, Savant), y se resuspendió en un volumen como mínimo de 0,5 ml de agua bidestilada. En este punto se realizó el tratamiento con RNasa A (apartado ). La fenolización propiamente dicha empezó a partir de este punto: a esta solución se le añadió de forma sucesiva: en un primer paso la ½ de volumen de fenol (Sigma, ph 7,9); ¼ de volumen de fenol más ¼ de volumen de cloroformo:alcohol isoamílico (24:1) en un segundo paso; y finalmente ½ de volumen de cloroformo:alcohol isoamílico (24:1). En cada uno de estos tres pasos se mezcló la fase acuosa y la orgánica hasta conseguir una emulsión, y se centrifugó ( x g, 2 minutos) para separar las dos fases, recogiendo la fase acuosa que es dónde se encuentra el ADN. Generalmente no fueron necesarios más que estos tres pasos para desproteinizar el ADN, pero en algunas muestras en las que se observó mucha suciedad en el segundo paso (en la interfase quedan visibles los restos de proteínas), se repitió éste tantas veces como fue necesario. Una vez desproteinizado el ADN se precipitó con etanol tal y como se ha explicado en el apartado anterior.
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Técnicas de Biología Molecular
 Técnicas de Biología Molecular DNA I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II. Separación electroforética
Técnicas de Biología Molecular DNA I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II. Separación electroforética
DANAGENE RNA PURIFICATION KIT
 DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O. Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua
 C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL
 LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN.
 PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
5. MATERIALES Y MÉTODOS
 5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA. Grado en Medicina
 DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa
 38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
CUESTIONES. 1. Por qué la PCR convencional no es cuantitativa?
 2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR
 PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS
 Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
39. Aislamiento y purificación del DNA de un plásmido recombinante
 39. Aislamiento y purificación del DNA de un plásmido recombinante Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento de Bioquímica
39. Aislamiento y purificación del DNA de un plásmido recombinante Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento de Bioquímica
3. COMPONENTES. Tampón de electroforesis concentrado 2 x 50 ml 10 X (2 envases 500ml)
 PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Práctico: Genética bacteriana 2007.
 Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
3/22/2010. Iván Ferrer Rodríguez, PhD Catedrático. En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR.
 Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz porosa
 INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
PCR gen 18S ARNr humano
 PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES
 PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
2.- MATERIAL Y MÉTODOS
 2.- MATERIAL Y MÉTODOS Materiales y Métodos 45 2.1.- MICROORGANISMOS UTILIZADOS Las cepas bacterianas utilizadas en este trabajo pertenecen a dos especies diferentes, Escherichia coli y Yersinia enterocolitica,
2.- MATERIAL Y MÉTODOS Materiales y Métodos 45 2.1.- MICROORGANISMOS UTILIZADOS Las cepas bacterianas utilizadas en este trabajo pertenecen a dos especies diferentes, Escherichia coli y Yersinia enterocolitica,
I. 15microlitros de agua esteril, perforar el pozo y diluir. II. Se tomaron 2 microlitros para la primera transformación.
 23 de agosto 2007 Se comenzó la elaboración de la bitácora Medio LB Broth, Billar (Luria-Bertani) 25gr/litro Se preparó 500 mililitros agregando 12.5 gramos Se diluyó y esterilizó por autoclave Medio LB
23 de agosto 2007 Se comenzó la elaboración de la bitácora Medio LB Broth, Billar (Luria-Bertani) 25gr/litro Se preparó 500 mililitros agregando 12.5 gramos Se diluyó y esterilizó por autoclave Medio LB
ELECTROFORESIS BASICA
 Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Técnicas descritas en el curso 7.28
 Técnicas descritas en el curso 7.28 Técnica: Clase 1 Experimento de incorporación de dntps Ensayo de extensión del cebador Ensayo de unión en filtro Electroforesis en gel Análisis de ADN Helicasa Polaridad
Técnicas descritas en el curso 7.28 Técnica: Clase 1 Experimento de incorporación de dntps Ensayo de extensión del cebador Ensayo de unión en filtro Electroforesis en gel Análisis de ADN Helicasa Polaridad
Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz
 IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015
 CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
7.012 Serie de ejercicios 5
 Nombre Grupo 7.012 Serie de ejercicios 5 Pregunta 1 Al estudiar los problemas de esterilidad, usted intenta aislar un hipotético gen de conejo que explique la prolífica reproducción de estos animales.
Nombre Grupo 7.012 Serie de ejercicios 5 Pregunta 1 Al estudiar los problemas de esterilidad, usted intenta aislar un hipotético gen de conejo que explique la prolífica reproducción de estos animales.
V MATERIALES Y MÉTODOS
 V MATERIALES Y MÉTODOS 5.1 Aislamiento de bacterias e identificación 5.1.1 Cepas tomadas del cepario de la Universidad de las Américas-Puebla En el cepario del Departamento de Química y Biología hay algunas
V MATERIALES Y MÉTODOS 5.1 Aislamiento de bacterias e identificación 5.1.1 Cepas tomadas del cepario de la Universidad de las Américas-Puebla En el cepario del Departamento de Química y Biología hay algunas
ELECTROFORESIS AVANZADA
 Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
FRAGMENTO OLIGO 5-3 Hebra SECUENCIA 5' - - > 3' TAMAÑO (pb) F1. hmtl 569 L AACCAAACCCCAAAGACACC hmth2982 H CTGATCCAACATCGAGGTCG
 ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a
 Introducción Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a los investigadores verificar la expresión de una proteína, determinar la cantidad relativa
Introducción Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a los investigadores verificar la expresión de una proteína, determinar la cantidad relativa
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR)
 METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
Practica la genética Ficha didáctica del profesorado Bachillerato
 Ficha didáctica del profesorado Bachillerato www.eurekamuseoa.es Extracción de ADN Cuál es la función de cada uno de los ingredientes utilizados para realizar la disolución tampón para la visualización
Ficha didáctica del profesorado Bachillerato www.eurekamuseoa.es Extracción de ADN Cuál es la función de cada uno de los ingredientes utilizados para realizar la disolución tampón para la visualización
TÉCNICAS GENÓMICAS. 2) Cuantificación del número de virus presentes en muestras de sangre o suero: carga vírica
 TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO
 AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO E l estudio del genoma de los seres vivos a sido uno d los principales objetivos de la Biología. Desde los trabajos de Mendel (1866),
AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO E l estudio del genoma de los seres vivos a sido uno d los principales objetivos de la Biología. Desde los trabajos de Mendel (1866),
BIOTECNOLOGÍA. 1.- Ingeniería Genética, ADN recombinante
 BIOTECNOLOGÍA 1.- Ingeniería Genética, ADN recombinante Técnica del ADN recombinante: es la más usada en ingeniería genética, esta basada en el uso de las enzimas de restricción o endonucleasas de restricción
BIOTECNOLOGÍA 1.- Ingeniería Genética, ADN recombinante Técnica del ADN recombinante: es la más usada en ingeniería genética, esta basada en el uso de las enzimas de restricción o endonucleasas de restricción
Preparación de medios de cultivo
 Objetivos Preparación de medios de cultivo Curso: Métodos en fitopatología 24 de abril de 2009 Dr. Pedro Mondino Conocer a que denominamos medio de cultivo en el laboratorio de Fitopatología. Reconocer
Objetivos Preparación de medios de cultivo Curso: Métodos en fitopatología 24 de abril de 2009 Dr. Pedro Mondino Conocer a que denominamos medio de cultivo en el laboratorio de Fitopatología. Reconocer
Clonación molecular. Clonar significa hacer copias idénticas
 INGENIERÍA GENÉTICA Clonación molecular Clonar significa hacer copias idénticas Este término originalmente fue aplicado al procedimiento de aislar una célula y permitirle reproducirse creando una población
INGENIERÍA GENÉTICA Clonación molecular Clonar significa hacer copias idénticas Este término originalmente fue aplicado al procedimiento de aislar una célula y permitirle reproducirse creando una población
DANAGENE SALIVA KIT. Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones
 DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
Reacción en cadena de la polimerasa (PCR)
 Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Facultad de Biología. Departamento de Microbiología
 Facultad de Biología Departamento de Microbiología La Familia de proteínas Hha-YmoA: estudios estructurales y papel regulador en Yersinia enterocolitica Programa de Doctorado: Microbiología Ambiental y
Facultad de Biología Departamento de Microbiología La Familia de proteínas Hha-YmoA: estudios estructurales y papel regulador en Yersinia enterocolitica Programa de Doctorado: Microbiología Ambiental y
LIMPIEZA Y DESINFECCIÓN EN LA INDUSTRIA LÁCTEA
 LIMPIEZA Y EN LA INDUSTRIA LÁCTEA LD EN LAS INDUSTRIAS DE ALIMENTOS La sanitización/higienización es un concepto general que comprende la creación y mantenimiento de las condiciones óptimas de higiene
LIMPIEZA Y EN LA INDUSTRIA LÁCTEA LD EN LAS INDUSTRIAS DE ALIMENTOS La sanitización/higienización es un concepto general que comprende la creación y mantenimiento de las condiciones óptimas de higiene
Soluciones de la serie de ejercicios 4 (Curso 7.012)
 Pregunta 1 Soluciones de la serie de ejercicios 4 (Curso 7.012) Usted está estudiando la síntesis del aminoácido triptófano en las bacterias. Las enzimas TrpA, TrpB, TrpC, TrpD, TrpE and AroH son necesarias
Pregunta 1 Soluciones de la serie de ejercicios 4 (Curso 7.012) Usted está estudiando la síntesis del aminoácido triptófano en las bacterias. Las enzimas TrpA, TrpB, TrpC, TrpD, TrpE and AroH son necesarias
DETECCIÓN DE C. jejuni EN HAMBURGUESA DE POLLO POR PCR TRADICIONAL PRT-712.04-082
 Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Bioquímica III- 2009
 Facultad de Ciencias Exactas, Universidad Nacional de La Plata Bioquímica III- 2009 Trabajo Práctico Nro 4 Extracción de RNA y DNA bacteriano INTRODUCCIÓN El RNA es el ácido nucleico más abundante en la
Facultad de Ciencias Exactas, Universidad Nacional de La Plata Bioquímica III- 2009 Trabajo Práctico Nro 4 Extracción de RNA y DNA bacteriano INTRODUCCIÓN El RNA es el ácido nucleico más abundante en la
Tecnología HaloPlex by Genycell Biotech España
 Paneles de genes custom-made Tecnología HaloPlex by Genycell Biotech España SERVICIO NGS CLÍNICA HALO: OVERVIEW 1. DISEÑO DEL PANEL DE GENES Y DEL KIT DE CAPTURA 2. PREPARACIÓN DE LA MUESTRA EN EL LABORATORIO
Paneles de genes custom-made Tecnología HaloPlex by Genycell Biotech España SERVICIO NGS CLÍNICA HALO: OVERVIEW 1. DISEÑO DEL PANEL DE GENES Y DEL KIT DE CAPTURA 2. PREPARACIÓN DE LA MUESTRA EN EL LABORATORIO
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS.
 NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
Código: IDK-010 Ver: 1. Proteína G C825T. Sistema para la detección de la mutación C825T en el gene de la proteína G.
 C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
DETERMINACIÓN DEL FACTOR Rh por PCR
 Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Guía Práctica 9. Producción de micelio en medio líquido para extracción de ADN. Micelio de hongos. Contenido
 Guía Práctica 9 Micelio de hongos Producción de micelio en medio líquido para extracción de ADN Guillermo Castellanos, Experto en Investigación 2 Carlos Jara, Ing. Agr. M.Sc., Asociado en Investigación
Guía Práctica 9 Micelio de hongos Producción de micelio en medio líquido para extracción de ADN Guillermo Castellanos, Experto en Investigación 2 Carlos Jara, Ing. Agr. M.Sc., Asociado en Investigación
Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala
 Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala IICA/Red SICTA-CIAT INFORME DE AVANCE Gerardo Gallego - Harold
Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala IICA/Red SICTA-CIAT INFORME DE AVANCE Gerardo Gallego - Harold
DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas
 DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas ELEMENTOS BÁSICOS DE LA INVESTIGACIÓN EN GENES Análisis
DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas ELEMENTOS BÁSICOS DE LA INVESTIGACIÓN EN GENES Análisis
MEMORIA DE PRÁCTICAS SILVIA PÉREZ SOLANA
 MEMORIA DE PRÁCTICAS SILVIA PÉREZ SOLANA DATOS DE LA EMPRESA Instituto de Biotecnología y Biomedicina de Cantabria (CSIC-UC- SODERCAN), Avda. Herrera Oria s/n. Santander. El tutor CSIC fue Raúl Fernández
MEMORIA DE PRÁCTICAS SILVIA PÉREZ SOLANA DATOS DE LA EMPRESA Instituto de Biotecnología y Biomedicina de Cantabria (CSIC-UC- SODERCAN), Avda. Herrera Oria s/n. Santander. El tutor CSIC fue Raúl Fernández
Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri
 Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri OBJETIVO Desarrollar un banco de datos a partir de plantas de origen indudable y del uso
Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri OBJETIVO Desarrollar un banco de datos a partir de plantas de origen indudable y del uso
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA"
 P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
PCR en Tiempo Real. Introducción
 Introducción Página 1 de 6 Introducción general La reacción en cadena de la polimerasa, cuyas iniciales en inglés son PCR ("polymerase chain reaction"), es una técnica que fue desarrollada por Kary Mullis
Introducción Página 1 de 6 Introducción general La reacción en cadena de la polimerasa, cuyas iniciales en inglés son PCR ("polymerase chain reaction"), es una técnica que fue desarrollada por Kary Mullis
CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA)
 CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA) PROTOCOLO ADA FUNDAMENTO DEL METODO: La adenosindesaminasa (ADA) es una enzima del catabolismo de las purinas que cataliza la conversión de la adenosina en inosina
CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA) PROTOCOLO ADA FUNDAMENTO DEL METODO: La adenosindesaminasa (ADA) es una enzima del catabolismo de las purinas que cataliza la conversión de la adenosina en inosina
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR
 TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Andrés Ciocchini, Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN) por partición diferencial
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Andrés Ciocchini, Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN) por partición diferencial
TaqMan GMO Maize Quantification Kit. Part No: 4481972
 TaqMan GMO Maize Quantification Kit Part No: 4481972 1. Introducción Los organismos modificados genéticamente (OMG) se encuentran ampliamente distribuidos, siendo la soja y el maíz los vegetales que ocupan
TaqMan GMO Maize Quantification Kit Part No: 4481972 1. Introducción Los organismos modificados genéticamente (OMG) se encuentran ampliamente distribuidos, siendo la soja y el maíz los vegetales que ocupan
PCR. Técnica inventada por Kary Mullis :1987. Premio Nobel 1993. Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN
 PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA
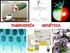
 Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
Enzimas de restricción
 BIOTECNOLOGIA Enzimas de restricción Endonucleasas que reconocen dianas específicas en el ADN Protegen a cada cepa de bacterias de otro ADN que no pertenece al sistema El ADN propio está protegido porque
BIOTECNOLOGIA Enzimas de restricción Endonucleasas que reconocen dianas específicas en el ADN Protegen a cada cepa de bacterias de otro ADN que no pertenece al sistema El ADN propio está protegido porque
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico
 17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
6. Reacciones de precipitación
 6. Reacciones de precipitación Las reacciones de precipitación son aquellas en las que el producto es un sólido; se utilizan en los métodos gravimétricos de análisis y en las titulaciones por precipitación.
6. Reacciones de precipitación Las reacciones de precipitación son aquellas en las que el producto es un sólido; se utilizan en los métodos gravimétricos de análisis y en las titulaciones por precipitación.
UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I
 UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I DRAS. MARÍA LUISA ORTIZ Y ANA PEDREGOSA 1 PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA
UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I DRAS. MARÍA LUISA ORTIZ Y ANA PEDREGOSA 1 PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA
1. Cuáles son las diferencias en los componentes químicos del ADN y ARN?
 ACTIVIDADES TEMA 4 - BIOTECNOLOGÍA 1. Cuáles son las diferencias en los componentes químicos del ADN y ARN? Las cadenas de ADN están formadas por fosfato y desoxirribosa y la del ARN por fosfato y ribosa.
ACTIVIDADES TEMA 4 - BIOTECNOLOGÍA 1. Cuáles son las diferencias en los componentes químicos del ADN y ARN? Las cadenas de ADN están formadas por fosfato y desoxirribosa y la del ARN por fosfato y ribosa.
Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml
 Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml Para la purificación de ADN genómico de todos los kits de colección Oragene y ORAcollect. Visite nuestro sitio
Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml Para la purificación de ADN genómico de todos los kits de colección Oragene y ORAcollect. Visite nuestro sitio
PRACTICA N 8 Cuantificación de nitrógeno total y determinación del contenido de proteína cruda Introducción:
 1 PRACTICA N 8 Cuantificación de nitrógeno total y determinación del contenido de proteína cruda I. Introducción: El nitrógeno es el elemento químico que permite diferenciar las proteínas de otros compuestos,
1 PRACTICA N 8 Cuantificación de nitrógeno total y determinación del contenido de proteína cruda I. Introducción: El nitrógeno es el elemento químico que permite diferenciar las proteínas de otros compuestos,
Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez
 Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez 1 Objetivos Entender la técnica: Reacción de Polimerasa en Cadena (PCR por sus siglas en inglés)
Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez 1 Objetivos Entender la técnica: Reacción de Polimerasa en Cadena (PCR por sus siglas en inglés)
Protocolos de secuenciación de ADN
 1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
AQUAGESTION. ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico.
 AQUAGESTION ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico. Noviembre 2011 ACTUALIDAD Este año Sernapesca confirma
AQUAGESTION ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico. Noviembre 2011 ACTUALIDAD Este año Sernapesca confirma
MANUAL DE PRÁCTICAS DE LABORATORIO
 MANUAL DE PRÁCTICAS DE LABORATORIO INGENIERIA ENBIOTECNOLOGÍA INGENIERÍA GENÉTICA FICHA TECNICA FICHA TÉCNICA SEMINARIO DE BIOTECNOLOGÍA MÉDICO-FARMACÉUTICA Fecha: Nombre del catedrático: 10-Julio-2012
MANUAL DE PRÁCTICAS DE LABORATORIO INGENIERIA ENBIOTECNOLOGÍA INGENIERÍA GENÉTICA FICHA TECNICA FICHA TÉCNICA SEMINARIO DE BIOTECNOLOGÍA MÉDICO-FARMACÉUTICA Fecha: Nombre del catedrático: 10-Julio-2012
EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS
 EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS Objetivos: Mª Jesús González García Mª Amparo Mora Alcácer COLEGIO AVE Mª DE PENYA-ROJA Aprender a trabajar en el laboratorio y apreciar el orden,
EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS Objetivos: Mª Jesús González García Mª Amparo Mora Alcácer COLEGIO AVE Mª DE PENYA-ROJA Aprender a trabajar en el laboratorio y apreciar el orden,
Tema 11. Herramientas de la genética molecular
 Tema 11. Herramientas de la genética molecular Genética CC. Mar 2004-05 Objetivos Técnicas de clonación Construcción de genotecas e identificación de secuencias Mapas de restricción Amplificación (PCR)
Tema 11. Herramientas de la genética molecular Genética CC. Mar 2004-05 Objetivos Técnicas de clonación Construcción de genotecas e identificación de secuencias Mapas de restricción Amplificación (PCR)
MANUAL DE PRÁCTICAS DE LABORATORIO
 MANUAL DE PRÁCTICAS DE LABORATORIO CARRERA Ingeniería en Biotecnología ASIGNATURA: Microbiología Gral. FICHA TECNICA Fecha: Nombre del catedrático: 13-SEPT-2012 FICHA TÉCNICA MICROBIOLOGÍA GENERAL Jesús
MANUAL DE PRÁCTICAS DE LABORATORIO CARRERA Ingeniería en Biotecnología ASIGNATURA: Microbiología Gral. FICHA TECNICA Fecha: Nombre del catedrático: 13-SEPT-2012 FICHA TÉCNICA MICROBIOLOGÍA GENERAL Jesús
AUTOR: JORGE CONTRERAS PINEDA 1. Titulo:
 Página 1 de 1 1. Titulo: Electroforesis de DNA 2. Objetivo Conocer los principios básicos de la electroforesis horizontal en geles de agarosa y aplicarlo para la separación de DNA humano, plasmídico, recombinante
Página 1 de 1 1. Titulo: Electroforesis de DNA 2. Objetivo Conocer los principios básicos de la electroforesis horizontal en geles de agarosa y aplicarlo para la separación de DNA humano, plasmídico, recombinante
Grado en Química. 4º Curso Bioquímica. Guión de Prácticas
 Grado en Química 4º Curso Bioquímica Guión de Prácticas UTILES A TRAER POR EL ALUMNO Bata Gafas de Seguridad Cuaderno de Laboratorio NORMAS DE TRABAJO Antes de empezar Antes de empezar cada práctica, el
Grado en Química 4º Curso Bioquímica Guión de Prácticas UTILES A TRAER POR EL ALUMNO Bata Gafas de Seguridad Cuaderno de Laboratorio NORMAS DE TRABAJO Antes de empezar Antes de empezar cada práctica, el
Informe del trabajo práctico nº7
 Informe del trabajo práctico nº7 Profesora : Lic. Graciela. Lic. Mariana. Alumnas: Romina. María Luján. Graciela. Mariana. Curso: Química orgánica 63.14 turno 1 OBJETIVOS Mostrar las propiedades que presentan
Informe del trabajo práctico nº7 Profesora : Lic. Graciela. Lic. Mariana. Alumnas: Romina. María Luján. Graciela. Mariana. Curso: Química orgánica 63.14 turno 1 OBJETIVOS Mostrar las propiedades que presentan
REACCIONES DE IONES METÁLICOS
 Actividad Experimental 4 REACCIONES DE IONES METÁLICOS Investigación previa -Investigar las medidas de seguridad para trabajar con amoniaco -Investigar las reglas de solubilidad de las sustancias químicas.
Actividad Experimental 4 REACCIONES DE IONES METÁLICOS Investigación previa -Investigar las medidas de seguridad para trabajar con amoniaco -Investigar las reglas de solubilidad de las sustancias químicas.
Extracción sólido-líquido
 Extracción sólido-líquido Objetivos de la práctica! Determinar la concentración de saturación del soluto en el disolvente en un sistema ternario arena-azúcar-agua, estableciendo la zona operativa del diagrama
Extracción sólido-líquido Objetivos de la práctica! Determinar la concentración de saturación del soluto en el disolvente en un sistema ternario arena-azúcar-agua, estableciendo la zona operativa del diagrama
PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN DE LAS COMPETENCIAS PROFESIONALES CUESTIONARIO DE AUTOEVALUACIÓN PARA LAS TRABAJADORAS Y TRABAJADORES
 MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
MATERIALES Y MÉTODOS. Cepas Utilizadas
 MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
INGENIERÍA GENÉTICA 5 GAATTC 3 3 CTTAAG 5
 INGENIERÍA GENÉTICA 1. Fundamentos básicos de la ingeniería genética 2. Desnaturalización e hibridación del ADN 3. Reacción en cadena de la polimerasa (PCR) 4. Nuevas disciplinas surgidas de la ingeniería
INGENIERÍA GENÉTICA 1. Fundamentos básicos de la ingeniería genética 2. Desnaturalización e hibridación del ADN 3. Reacción en cadena de la polimerasa (PCR) 4. Nuevas disciplinas surgidas de la ingeniería
Genómica y transcriptómica para la generación de datos en Evolución
 recuadro Genómica y transcriptómica para la generación de datos en Evolución Gabriela Bedó Genómica. Sus objetivos Compilar todas las secuencias de un organismo Establecer la localización de los genes
recuadro Genómica y transcriptómica para la generación de datos en Evolución Gabriela Bedó Genómica. Sus objetivos Compilar todas las secuencias de un organismo Establecer la localización de los genes
Integrantes: Andrés Felipe Cárdenas Álvarez 2101302 Diana Katherine Carreño Moyano 2100993 Lorena Duarte Peña 2100968. Grupo: 4
 PRÁCTICA 8. DETERMINACIÓN DE CALCIO Y MAGNESIO EN UN LÁCTEO, LECHE ENTERA PARMALAT Integrantes: Andrés Felipe Cárdenas Álvarez 2101302 Diana Katherine Carreño Moyano 2100993 Lorena Duarte Peña 2100968
PRÁCTICA 8. DETERMINACIÓN DE CALCIO Y MAGNESIO EN UN LÁCTEO, LECHE ENTERA PARMALAT Integrantes: Andrés Felipe Cárdenas Álvarez 2101302 Diana Katherine Carreño Moyano 2100993 Lorena Duarte Peña 2100968
Prácticas de Análisis Instrumental
 Prácticas de Análisis Instrumental Asignatura: Análisis Instrumental Alumno: Daniel González Mancebo Practica 1. DETERMINACIÓN DE CONSTANTES DE EQUILIBRIO MEDIANTE ESPECTROFOTOMETRÍA UV- VISIBLE. Lo primero
Prácticas de Análisis Instrumental Asignatura: Análisis Instrumental Alumno: Daniel González Mancebo Practica 1. DETERMINACIÓN DE CONSTANTES DE EQUILIBRIO MEDIANTE ESPECTROFOTOMETRÍA UV- VISIBLE. Lo primero
UNIVERSIDAD NACIONAL ABIERTA Y A DISTANCIA
 UNIVERSIDAD NACIONAL ABIERTA Y A DISTANCIA ESCUELA DE CIENCIAS AGRICOLA, PECUARIAS Y DEL MEDIO AMBIENTE ECAPMA PROTOCOLO DE PRÁCTICAS PARA LA ESCUELA Nombre del Curso: Biotecnología II Código: 203019 Fecha:
UNIVERSIDAD NACIONAL ABIERTA Y A DISTANCIA ESCUELA DE CIENCIAS AGRICOLA, PECUARIAS Y DEL MEDIO AMBIENTE ECAPMA PROTOCOLO DE PRÁCTICAS PARA LA ESCUELA Nombre del Curso: Biotecnología II Código: 203019 Fecha:
ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDS-PAGE. Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE)
 ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDSPAGE Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE) Método rápido, reproducible y de bajo costo Utilizado para cuantificar, comparar
ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDSPAGE Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE) Método rápido, reproducible y de bajo costo Utilizado para cuantificar, comparar
LABORATORIO DE QUÍMICA ANALÍTICA INFORME PRESENTADO A LA PROF. ANGELA SIFONTE
 LABORATORIO DE QUÍMICA ANALÍTICA INFORME PRESENTADO A LA PROF. ANGELA SIFONTE TURBIDIMETRÍA INFORME N 9. PRESENTADO POR LOS BRS.: WILLIAM CHEN CHEN C.I.: 16.113.714 YUSMARY DE ABREU C.I.: 15.914.973 CARACAS,
LABORATORIO DE QUÍMICA ANALÍTICA INFORME PRESENTADO A LA PROF. ANGELA SIFONTE TURBIDIMETRÍA INFORME N 9. PRESENTADO POR LOS BRS.: WILLIAM CHEN CHEN C.I.: 16.113.714 YUSMARY DE ABREU C.I.: 15.914.973 CARACAS,
1. Título. Cuantificacion de ADN por espectrofluorometría mediante el uso de Quant- it PicoGreen dsaadnssay Kit (Invitrogen ).
 1. Título. Cuantificacion de ADN por espectrofluorometría mediante el uso de QuantiT PicoGreen dsaadnssay Kit (Invitrogen ). 2. Finalidad. El presente documento describe el Procedimiento Normalizado de
1. Título. Cuantificacion de ADN por espectrofluorometría mediante el uso de QuantiT PicoGreen dsaadnssay Kit (Invitrogen ). 2. Finalidad. El presente documento describe el Procedimiento Normalizado de
- Clonar un gen (molde ADN o ARN)
 APLICACIONES PCR Aplicaciones de la PCR - Clonar un gen (molde ADN o ARN) - Secuencia conocida - Genes homólogos en especies próximas (primers degenerados) - Información de secuencia proteica (primers
APLICACIONES PCR Aplicaciones de la PCR - Clonar un gen (molde ADN o ARN) - Secuencia conocida - Genes homólogos en especies próximas (primers degenerados) - Información de secuencia proteica (primers
imegen Alfa-1-AT Manual de Usuario Referencia: IMG-211
 Manual de Usuario imegen Alfa-1-AT Genotipado de las mutaciones Glu342Lys (PI-Z) y Glu264Val (PI-S) del gen SERPINA1 mediante PCR a tiempo real Referencia: Fabricado en España Garantías y responsabilidades
Manual de Usuario imegen Alfa-1-AT Genotipado de las mutaciones Glu342Lys (PI-Z) y Glu264Val (PI-S) del gen SERPINA1 mediante PCR a tiempo real Referencia: Fabricado en España Garantías y responsabilidades
23. Hidrólisis ácida y enzimática del glucógeno
 23. Hidrólisis ácida y enzimática del glucógeno Emilia Martínez Galisteo, Carmen Alicia Padilla Peña, Concepción García Alfonso, José Antonio Bárcena Ruiz, Jesús Diez Dapena. Departamento de Bioquímica
23. Hidrólisis ácida y enzimática del glucógeno Emilia Martínez Galisteo, Carmen Alicia Padilla Peña, Concepción García Alfonso, José Antonio Bárcena Ruiz, Jesús Diez Dapena. Departamento de Bioquímica
