Alineamiento múltiple de secuencias
|
|
|
- Manuel Blanco Parra
- hace 6 años
- Vistas:
Transcripción
1 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 11 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
2 1 Alineamiento múltiple de secuencias Introducción Función de puntuación Algoritmos exhaustivos Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
3 Introducción 1 Alineamiento múltiple de secuencias Introducción Función de puntuación Algoritmos exhaustivos Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
4 Introducción Alineamiento múltiple de secuencias Introducción Una extensión natural del alineamiento de pares de secuencias es el alineamiento múltiple, el cual consiste en alinear diversas secuencias relacionadas para lograr la mejor coincidencia entre las secuencias Como vimos la clase pasada la búsqueda de similitud en BD puede devolver como resultado un grupo de secuencias relacionadas con la secuencia consulta (en pares) A menudo es necesario convertir esos númerosos alineamientos de pares en un solo alineamiento (múltiple), el cual permite identificar posiciones evolutivas equivalentes en todas las secuencias Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
5 Introducción Alineamiento múltiple de secuencias Introducción La gran ventaja del alineamiento múltiple de secuencias (AMS) es que permite revelar mucha más información biológica que un grupo de alineamientos de pares El AMS es importante tiene aplicaciones importantes: Análisis filogenético Predicción de la estructura secundaria y terciaria de proteínas... Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
6 Introducción Alineamiento múltiple de secuencias Introducción En teoría es posible usar la programación dinámica para alinear cualquier número de secuencias, sin embargo el tiempo de cálculo y la memoria requerida aumenta exponencialmente En la práctica los enfoques heurísticos son los más utilizados Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
7 Función de puntuación 1 Alineamiento múltiple de secuencias Introducción Función de puntuación Algoritmos exhaustivos Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
8 Función de puntuación Función de puntuación El AMS consiste en acomodar las secuencias de forma tal que el máximo número de residuos de cada secuencia coincidan de acuerdo a una función de puntuación (scoring function) particular La función de puntuación para el AMS está basada en el concepto de suma de pares (SP) Como su nombre lo indica, es la suma de la puntuación de todos los posibles pares de secuencias en un AMS utilizando una matriz de puntuación particular Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
9 Función de puntuación Función de puntuación Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
10 Algoritmos exhaustivos 1 Alineamiento múltiple de secuencias Introducción Función de puntuación Algoritmos exhaustivos Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
11 Algoritmos exhaustivos Algoritmos exhaustivos Estas técnicas implican el alineamiento de todos los posibles alineamientos de manera simultánea y similar a como se realiza en la programación dinámica Por lo tanto, implican la construcción de una matriz multidimensional, e.g. para alinear tres secuencias (k = 3) se requiere de construir una matriz tridimensional Finalmente se debe llevar a cabo un seguimiento a lo largo de las k dimensiones para encontrar el camino que represente el mejor alineamiento posible Suelen limitarse a pequeños conjuntos de secuencias (k < 10) Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
12 Algoritmos exhaustivos Algoritmos exhaustivos La complejidad computacional es O(N k ) (k, núm. de secuencias) N 5 1e+15 Complejidad 9e+14 8e+14 7e+14 6e+14 5e+14 4e+14 3e+14 2e+14 1e N Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
13 Algoritmos exhaustivos Algoritmos exhaustivos Para N = 1000 y k = 5 la complejidad es 1.0E+15 Usando una computadora que ejecute 1 millón de operaciones cada segundo tardaría horas ( días, años) La alternativa es usar métodos heurísticos Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
14 1 Alineamiento múltiple de secuencias Introducción Función de puntuación Algoritmos exhaustivos Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
15 Existen tres tipos de algoritmos heurísticos: Alineamiento progresivo Alineamiento iterativo Alineamiento basado en bloques Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
16 Método de alineamiento progresivo Este método va ensamblando progresivamente alineamientos de pares para formar un AMS Primero se lleva a cabo un alineamiento global de pares de secuencias usando el algoritmo de Needleman-Wunsch Con los resultados se crea una matriz de distancias, está permite ver la relación evolutiva de la secuencia con las demás Se realiza un análisis filogenético simple, dando como resultado la creación de un árbol filogenético (árbol guía) Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
17 Método de alineamiento progresivo Este árbol refleja la proximidad entre todas las secuencias y es empleado para realizar un reajuste de las secuencias Las dos secuencias más relacionadas son realineadas usando el algoritmo de Needleman-Wunsch convirtiéndose en un secuencia (consenso) Este proceso se continúa hasta que todas las secuencias quedan alineadas Clustal ( es un programa que utiliza el método de alineamiento progresivo Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
18 Método de alineamiento progresivo Algorithm 1: Método de alineamiento progresivo Data: N secuencias Result: Alineamiento de las N secuencias 1 begin 2 Construir la matriz de distancias /* Árbol guía */ 3 Construir el árbol filogenético usando Neighbor-Joining 4 while no estén alineadas todas las secuencias do 5 Alinear las secuencias más relacionadas 6 Reducir las secuencias alineadas 7 end 8 end Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
19 Método de alineamiento progresivo (Ejemplo) Se realizan todos los alineamientos de pares de secuencias y se construye la matriz de distancias Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
20 Método de alineamiento progresivo (Ejemplo...) Se calcula un árbol guía con los pares más próximos: C, D y A, B Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
21 Método de alineamiento progresivo (Ejemplo...) Se alinean C, D y A, B por separado usando programación dinámica Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
22 Método de alineamiento progresivo (Ejemplo...) Los alineamientos C, D y A, B se reducen a secuencias consenso las cuales se alinean entre ellas Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
23 Método de alineamiento progresivo (Ejemplo...) Se crea un nuevo consenso para C, D, A, B el cual se alinea con E con lo que se completa el AMS Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
24 Método de alineamiento progresivo (Ejemplo...) Para este ejemplo con Clustal ( se utilizarán las secuencias de proteínas siguientes: NP_ BAB NP_ MUP4_MOUSE P0A901 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
25 Método de alineamiento progresivo, limitantes El método de alineamiento progresivo presenta algunas limitantes: Este método no es adecuado para comparar secuencias de diferentes longitudes (global) El resultado final proporcionado por éste también se ve muy influenciado por el orden de las secuencias Debido a la naturaleza codiciosa (greedy) del método el resultado depende del alineamiento inicial de pares de secuencias (propagación de errores) Si las dos primeras secuencias son muy similares, el alineamiento base contendrá pocos errores Si las dos secuencias son muy divergentes los errores y los huecos se irán propagando Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
26 Método de alineamiento progresivo, T-Coffee T-Coffee (Tree-based Consistency Objective Function for alignment Evaluation, realiza alineamiento progresivo al igual que Clustal La principal diferencia radica en que T-Coffee realiza un alineamiento de pares tanto local como global Para el alineamiento global usa Clustal mientras que para el alineamiento local usa Lalign ( Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
27 Método de alineamiento progresivo, T-Coffee... Los resultados de estas dos alineaciones son almacenadas para formar una biblioteca Para cada par de residuos en cada par de secuencias se calcula una puntuación de consistencia para los alineamientos globales y locales Cada alineamiento de pares se alinea con una tercera posible secuencia Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
28 Método de alineamiento progresivo, T-Coffee... El resultado es usado para refinar el alineamiento de pares original en un proceso llamado extensión de la biblioteca Basado en el refinamiento de alineamiento de pares se construye una matriz de distancias para obtener un árbol guía Finalmente se utiliza este árbol para realizar un AMS mediante el enfoque progresivo Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
29 Método de alineamiento progresivo, T-Coffee... Algorithm 2: Algoritmo T-Coffee Data: N secuencias Result: Alineamiento de las N secuencias 1 begin 2 Alineamiento de pares de secuencias (Clustal y Lalign) 3 Construcción de la librería 4 Calcular la matriz de distancias 5 Crear el árbol guía mediante mediante Neighbor-Joining 6 Construir el alineamiento múltiple siguiendo el árbol 7 end Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
30 Método de alineamiento progresivo, Ejemplo T-Coffee Para este ejemplo usaremos las secuencias de proteínas siguientes y T-Coffee ( NP_ BAB NP_ MUP4_MOUSE P0A901 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
31 Método de alineamiento iterativo Este tipo de métodos se basan en la idea de que la solución óptima a un problema puede ser encontrada mediante la modificación iterativa de soluciones subóptimas existentes El proceso consiste en encontrar un alineamiento de baja calidad y mejorarlo gradualmente hasta que ya no sea posible Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
32 Método de alineamiento iterativo, PRRN PRRN ( es un algoritmo para AMS que emplea una estrategia iterativa doble anidada Efectúa el AMS mediante dos conjuntos de iteraciones: la interna y la externa En la iteración externa, se genera un alineamiento inicial aleatorio que es usado para derivar un árbol UPGMA (Unweighted Pair Group Method with Arithmetic Mean, método de agrupamiento) Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
33 Método de alineamiento iterativo, PRRN... Los pesos son posteriormente aplicados para optimizar el alineamiento En la iteración interna, las secuencias son aleatoriamente divididas en 2 grupos El alineamiento aleatorizado es usado para cada grupo en el ciclo inicial, después de lo cual las posiciones del alineamiento en cada grupo son fijadas Los 2 grupos, cada uno tratado como una sola secuencia, son entonces alineados entre ellos usando programación dinámica global Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
34 Método de alineamiento iterativo, PRRN... El proceso se repite de manera cíclica hasta que la puntuación total SP (suma de pares) no se incremente En este punto, el alineamiento resultante es usado para construir un nuevo árbol UPGMA Los nuevos pesos son aplicados para optimizar las puntuaciones del alineamiento El alineamiento optimizado es sujeto a un realineamiento en la iteración interior Este proceso es repetido durante varios ciclos hasta que no hay más mejora en las puntuaciones globales del alineamiento Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
35 Método de alineamiento iterativo, Ejemplo PRRN Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
36 Método de alineamiento iterativo, Ejemplo PRRN Para este ejemplo usaremos las secuencias de proteínas siguientes y PRRN ( NP_ BAB NP_ MUP4_MOUSE P0A901 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
37 Método basado en bloques Las estrategias progresivas e iterativas se basan en gran medida en alineamiento global Por lo tanto surge la necesidad de generar estrategias para encontrar similitudes locales Las secuencias a alinear pueden compartir bloques más o menos conservados, separados por regiones largas bastante menos conservadas Cada bloque está construido con alineamientos locales de varios fragmentos en cada secuencia Una vez que se han fijado los bloques se utilizan otros métodos de alineamiento para alinear dichas regiones Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
38 Método basado en bloques, DIALIGN2 DIALIGN2 es un programa para encontrar similitudes locales Este método rompe las secuencias en secuencias más pequeñas y realiza todos los alineamientos de pares posibles Los segmentos con alta puntuación son denominados bloques Entre las diferentes secuencias, estos bloques son compilados de manera progresiva Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
39 Método basado en bloques, Ejemplo DIALIGN2 Para este ejemplo usaremos las secuencias de proteínas siguientes y DIALIGN2 ( NP_ BAB NP_ MUP4_MOUSE P0A901 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del / 39
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA. Alineamiento múltiple de secuencias
 TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento múltiple de secuencias Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia Artificial
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento múltiple de secuencias Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia Artificial
Búsqueda de similitud en BD
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 4 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) 4 de junio del 2013 1 / 37 1 Introducción Requerimientos para búsqueda en BD FASTA Comparación
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 4 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) 4 de junio del 2013 1 / 37 1 Introducción Requerimientos para búsqueda en BD FASTA Comparación
DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS
 DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS COMPARACION DE DOS ATPASAS DE PECES El DOT PLOT permite una visualización rápida de la similitud entre dos secuencias Inconvenientes: No identifica
DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS COMPARACION DE DOS ATPASAS DE PECES El DOT PLOT permite una visualización rápida de la similitud entre dos secuencias Inconvenientes: No identifica
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA. Alineamiento múltiple de secuencias
 TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento múltiple de secuencias Mario de J. Pérez Jiménez Luis Valencia Cabrera Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento múltiple de secuencias Mario de J. Pérez Jiménez Luis Valencia Cabrera Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación
Perfiles y modelos ocultos de Markov
 Perfiles y modelos ocultos de Markov Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 13 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Perfiles y modelos ocultos de Markov 13 de junio del 2013
Perfiles y modelos ocultos de Markov Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 13 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Perfiles y modelos ocultos de Markov 13 de junio del 2013
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA. Alineamiento de secuencias de genes/proteínas
 TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento de secuencias de genes/proteínas Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento de secuencias de genes/proteínas Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia
Alineamiento de pares de secuencias
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 30 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento de pares de secuencias 30 de mayo del 2013 1 / 61 1 Alineamiento de pares de secuencias
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 30 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento de pares de secuencias 30 de mayo del 2013 1 / 61 1 Alineamiento de pares de secuencias
Lección 4. Métodos filogenéticos
 Básico La inferencia filogenética es un campo per se del estudio de la evolución, en continuo movimiento y expansión. filogenéticos La inferencia filogenética es un procedimiento de estimación estadística.
Básico La inferencia filogenética es un campo per se del estudio de la evolución, en continuo movimiento y expansión. filogenéticos La inferencia filogenética es un procedimiento de estimación estadística.
Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA
 Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA Martin A. Lozano, Jaime Velasco-Medina Grupo de Bio-nanoelectrónica EIEE, Universidad del Valle, A.A. 25360, Cali, Colombia E-mail:
Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA Martin A. Lozano, Jaime Velasco-Medina Grupo de Bio-nanoelectrónica EIEE, Universidad del Valle, A.A. 25360, Cali, Colombia E-mail:
Andrés M. Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia
 Alineamiento: Análisis computacional de secuencias Andrés M. Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Por qué y para qué... Tengo una secuencia de DNA/Proteína......
Alineamiento: Análisis computacional de secuencias Andrés M. Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Por qué y para qué... Tengo una secuencia de DNA/Proteína......
Práctica 1: Alineamientos
 Práctica 1: Alineamientos Partimos de un archivo de datos que contiene 5 secuencias de mrna asociado a la CFTR http://madma.usc.es/cursoverano/materiales Abrimos el ClustalX en la carpeta C:\ClustalX de
Práctica 1: Alineamientos Partimos de un archivo de datos que contiene 5 secuencias de mrna asociado a la CFTR http://madma.usc.es/cursoverano/materiales Abrimos el ClustalX en la carpeta C:\ClustalX de
Alineamientos Bioinformática. Daniel M. Alberto M. Fernando M.
 Alineamientos Bioinformática Daniel M. Alberto M. Fernando M. Repasando Qué son los alineamientos? Repasando Qué son los alineamientos? Comparación de dos o más secuencias de DNA. Repasando Qué son los
Alineamientos Bioinformática Daniel M. Alberto M. Fernando M. Repasando Qué son los alineamientos? Repasando Qué son los alineamientos? Comparación de dos o más secuencias de DNA. Repasando Qué son los
Algoritmos voraces (greedy)
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 21 de marzo de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Algoritmos voraces 21 de marzo de 2018 1 / 45 1 Algoritmos voraces (greedy) Aplicaciones de
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 21 de marzo de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Algoritmos voraces 21 de marzo de 2018 1 / 45 1 Algoritmos voraces (greedy) Aplicaciones de
BÚSQUEDA DE SECUENCIAS PARECIDAS (Similarity search)
 BÚSQUEDA DE SECUENCIAS PARECIDAS (Similarity search) Cuando los proyectos de secuenciación genómica descubren una nueva secuencia biológica es muy poco lo que se sabe de ella. Por ese motivo, una de las
BÚSQUEDA DE SECUENCIAS PARECIDAS (Similarity search) Cuando los proyectos de secuenciación genómica descubren una nueva secuencia biológica es muy poco lo que se sabe de ella. Por ese motivo, una de las
Introducción al Análisis y Diseño de Algoritmos
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 10 de enero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Introducción al ADA 10 de enero de 2018 1 / 22 1 Introducción al Análisis y Diseño de Algoritmos
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 10 de enero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Introducción al ADA 10 de enero de 2018 1 / 22 1 Introducción al Análisis y Diseño de Algoritmos
obtenido de la fusión de dos rutas y la posterior asignación a un vehículo con capacidad disponible.
 El problema VRP, y por tanto su variante con flota heterogénea, son problemas de optimización combinatoria complejos que caen en la categoría de NP-completo (Choi and Tcha, 2007), lo que significa que
El problema VRP, y por tanto su variante con flota heterogénea, son problemas de optimización combinatoria complejos que caen en la categoría de NP-completo (Choi and Tcha, 2007), lo que significa que
Alineamientos de múltiples secuencias. Rodrigo Santamaría
 Alineamientos de múltiples secuencias Rodrigo Santamaría Alineamientos de múltiples secuencias Introducción Motivación Definición Usos Algoritmos Benchmarking Visualización Bases de Datos 2 Introducción
Alineamientos de múltiples secuencias Rodrigo Santamaría Alineamientos de múltiples secuencias Introducción Motivación Definición Usos Algoritmos Benchmarking Visualización Bases de Datos 2 Introducción
Elvira Mayordomo y Jorge Álvarez. Marzo - Abril de 2016
 TRABAJO DE PRÁCTICAS Elvira Mayordomo y Jorge Álvarez Marzo - Abril de 2016 1 Introducción El trabajo de prácticas de la asignatura consistirá en que cada alumno realice por separado el trabajo que se
TRABAJO DE PRÁCTICAS Elvira Mayordomo y Jorge Álvarez Marzo - Abril de 2016 1 Introducción El trabajo de prácticas de la asignatura consistirá en que cada alumno realice por separado el trabajo que se
Métodos de alineamiento (3) Bioinformática, Elvira Mayordomo
 Métodos de alineamiento (3) Bioinformática, 7-3-16 Elvira Mayordomo Hoy veremos Multialineamiento (MSA): Un problema NP-completo Algoritmo aproximado para Multialineamiento: el método de la estrella Métodos
Métodos de alineamiento (3) Bioinformática, 7-3-16 Elvira Mayordomo Hoy veremos Multialineamiento (MSA): Un problema NP-completo Algoritmo aproximado para Multialineamiento: el método de la estrella Métodos
Filogenética molecular (III)
 Filogenética molecular (III) Bioinformática, 14-3-18 Basado en Kevin Yip-CSE-CUHK (Universidad china de Hong-Kong) HOY 1. Distancia evolutiva y modelos de mutación 2. Árboles: Las estructuras jerárquicas
Filogenética molecular (III) Bioinformática, 14-3-18 Basado en Kevin Yip-CSE-CUHK (Universidad china de Hong-Kong) HOY 1. Distancia evolutiva y modelos de mutación 2. Árboles: Las estructuras jerárquicas
Similitud de Secuencias: de un Par a Todas contra Todas
 Similitud de Secuencias: de un Par a Todas contra Todas Ricardo Baeza-Yates Centro de Investigación de la Web Depto. de Ciencias de la Computación Universidad de Chile ricardo@baeza.cl Resumen Similitud
Similitud de Secuencias: de un Par a Todas contra Todas Ricardo Baeza-Yates Centro de Investigación de la Web Depto. de Ciencias de la Computación Universidad de Chile ricardo@baeza.cl Resumen Similitud
Inteligencia Computacional
 Inteligencia Computacional Búsqueda: sin información otros algoritmos, con información http://blancavg.com/tc3023/ Blanca A. Vargas Govea * blanca.vg@gmail.com * Agosto 17, 2012 B D A A C E B I I F C G
Inteligencia Computacional Búsqueda: sin información otros algoritmos, con información http://blancavg.com/tc3023/ Blanca A. Vargas Govea * blanca.vg@gmail.com * Agosto 17, 2012 B D A A C E B I I F C G
Biotecnología. Alineación de secuencias. Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla
 Biotecnología Alineación de secuencias Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla Motivación Una vez localizado un gen Buscamos parecidos con genes de los que se
Biotecnología Alineación de secuencias Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla Motivación Una vez localizado un gen Buscamos parecidos con genes de los que se
Algoritmos de búsqueda exhaustiva
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 31 de enero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Algoritmos de búsqueda exhaustiva 31 de enero de 2018 1 / 22 1 Algoritmos de búsqueda exhaustiva
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 31 de enero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Algoritmos de búsqueda exhaustiva 31 de enero de 2018 1 / 22 1 Algoritmos de búsqueda exhaustiva
Convertir un AFND a un AFD
 Convertir un AFND a un AFD Existe una equivalencia entre los AFD y AFN, de forma que un autómata M es equivalente a un autómata M' si L(M) ) L(M'). Ejemplo: Los autómatas de la siguiente figura son equivalentes.
Convertir un AFND a un AFD Existe una equivalencia entre los AFD y AFN, de forma que un autómata M es equivalente a un autómata M' si L(M) ) L(M'). Ejemplo: Los autómatas de la siguiente figura son equivalentes.
Tema 3. Estructuras de Datos
 Tema 3! Estructuras de Datos Estructuras Simples, Doble, Anidada y Múltiple En este tema veremos las sentencias selectivas: IF (Si) IF - Then (Si - Entonces) IF - Then - Else ( Si - Entonces - Sino) Switch
Tema 3! Estructuras de Datos Estructuras Simples, Doble, Anidada y Múltiple En este tema veremos las sentencias selectivas: IF (Si) IF - Then (Si - Entonces) IF - Then - Else ( Si - Entonces - Sino) Switch
Algoritmos glotones. mat-151
 Algoritmos glotones (greedy) mat-151 Alonso Ramirez Manzanares Computación y Algoritmos 04.06.2009 Algoritmos glotones Algoritmos utilizados en problemas de optimización. Estos algoritmos siguen típicamente
Algoritmos glotones (greedy) mat-151 Alonso Ramirez Manzanares Computación y Algoritmos 04.06.2009 Algoritmos glotones Algoritmos utilizados en problemas de optimización. Estos algoritmos siguen típicamente
CONGRUENCIA METODOLOGICA EN LOS ANALISIS FILOGENETICOS A PARTIR DE ALINEAMIENTO Y RECONSTRUCCION. Katherine Cuadros
 CONGRUENCIA METODOLOGICA EN LOS ANALISIS FILOGENETICOS A PARTIR DE ALINEAMIENTO Y RECONSTRUCCION Katherine Cuadros 2041379 INTRODUCCIÓN El rápido aumento en la capacidad de generar datos moleculares, y
CONGRUENCIA METODOLOGICA EN LOS ANALISIS FILOGENETICOS A PARTIR DE ALINEAMIENTO Y RECONSTRUCCION Katherine Cuadros 2041379 INTRODUCCIÓN El rápido aumento en la capacidad de generar datos moleculares, y
Métodos de alineamiento. Bioinformática, Elvira Mayordomo
 Métodos de alineamiento Bioinformática, 16-2-17 Elvira Mayordomo Motivación: 2 razones para comparar secuencias biológicas 1. Los errores y omisiones en los datos biológicos producidos en la extracción
Métodos de alineamiento Bioinformática, 16-2-17 Elvira Mayordomo Motivación: 2 razones para comparar secuencias biológicas 1. Los errores y omisiones en los datos biológicos producidos en la extracción
Métodos de alineamiento. Bioinformática, Elvira Mayordomo
 Métodos de alineamiento Bioinformática, 24-2-16 Elvira Mayordomo Motivación: 2 razones para comparar secuencias biológicas 1. Los errores y omisiones en los datos biológicos producidos en la extracción
Métodos de alineamiento Bioinformática, 24-2-16 Elvira Mayordomo Motivación: 2 razones para comparar secuencias biológicas 1. Los errores y omisiones en los datos biológicos producidos en la extracción
Predicción de la estructura terciara de las proteínas
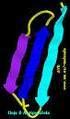 Predicción de la estructura terciara de las proteínas Cuando la estructura terciaria de una proteína no se ha determinado experimentalmente, se puede intentar construir un modelo tridimensional a partir
Predicción de la estructura terciara de las proteínas Cuando la estructura terciaria de una proteína no se ha determinado experimentalmente, se puede intentar construir un modelo tridimensional a partir
Métodos Constructivos. Empiezan desde una solución vacía (a veces pequeña)
 Métodos Constructivos Empiezan desde una solución vacía (a veces pequeña) En repetidas ocasiones, extienden la solución actual hasta que una solución completa se construye. Utiliza una heurística para
Métodos Constructivos Empiezan desde una solución vacía (a veces pequeña) En repetidas ocasiones, extienden la solución actual hasta que una solución completa se construye. Utiliza una heurística para
Por regla general, las búsquedas con BLAST obedecen a uno de estos dos objetivos:
 BLAST en el servidor del NCBI BLAST es la herramienta bioinformática más utilizada en todo el mundo. Compara una secuencia problema (query sequence) de nucleótidos o de proteínas con todas las secuencias
BLAST en el servidor del NCBI BLAST es la herramienta bioinformática más utilizada en todo el mundo. Compara una secuencia problema (query sequence) de nucleótidos o de proteínas con todas las secuencias
CAPÍTULO 4 METODOLOGÍA PROPUESTA
 CAPÍTULO 4 METODOLOGÍA PROPUESTA En este capítulo, se describen los métodos utilizados en el procedimiento propuesto para obtener soluciones del PLIDMC. En la actualidad, muchos de los problemas de optimización
CAPÍTULO 4 METODOLOGÍA PROPUESTA En este capítulo, se describen los métodos utilizados en el procedimiento propuesto para obtener soluciones del PLIDMC. En la actualidad, muchos de los problemas de optimización
Conceptos básicos de filogenética molecular
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 18 de julio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Conceptos básicos de filogenética molecular 18 de julio del 2013 1 / 43 1 Conceptos básicos
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 18 de julio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Conceptos básicos de filogenética molecular 18 de julio del 2013 1 / 43 1 Conceptos básicos
Programación Dinámica
 Programación Dinámica La técnica de programación dinámica se aplica en general a problemas de optimización. l igual que "dividir y conquistar", el problema es dividido en subproblemas de tamaños menores
Programación Dinámica La técnica de programación dinámica se aplica en general a problemas de optimización. l igual que "dividir y conquistar", el problema es dividido en subproblemas de tamaños menores
Decrementa y vencerás II
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 21 de febrero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Decrementa y vencerás II 21 de febrero de 2018 1 / 22 1 Decrementa y vencerás II Dr. Eduardo
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 21 de febrero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Decrementa y vencerás II 21 de febrero de 2018 1 / 22 1 Decrementa y vencerás II Dr. Eduardo
Alineamiento local: búsqueda de homologías
 Alineamiento local: búsqueda de homologías Supongamos que el material de partida para realizar una búsqueda de homologías no es un gen o una proteína completos y bien caracterizados de los que podamos
Alineamiento local: búsqueda de homologías Supongamos que el material de partida para realizar una búsqueda de homologías no es un gen o una proteína completos y bien caracterizados de los que podamos
2008 Pablo Vinuesa, 1
 Introducción a la Inferencia Filogenética y Evolución Molecular 23-26 Junio 2008, Fac. C. Biológicas - UANL Pablo Vinuesa (vinuesa@ccg.unam.mx) Centro de Ciencias Genómicas-UNAM, México http://www.ccg.unam.mx/~vinuesa/
Introducción a la Inferencia Filogenética y Evolución Molecular 23-26 Junio 2008, Fac. C. Biológicas - UANL Pablo Vinuesa (vinuesa@ccg.unam.mx) Centro de Ciencias Genómicas-UNAM, México http://www.ccg.unam.mx/~vinuesa/
Comparación de secuencias
 Comparación de secuencias Por qué nos interesa comparar secuencias de ADN o proteínas de distintos orígenes? Cómo se pueden alinear secuencias? Alineamiento de secuencias Dos tipos de alineamientos Alineamiento
Comparación de secuencias Por qué nos interesa comparar secuencias de ADN o proteínas de distintos orígenes? Cómo se pueden alinear secuencias? Alineamiento de secuencias Dos tipos de alineamientos Alineamiento
Programación Estructurada. Sesión 4:Estructuras de control
 Programación Estructurada Sesión 4:Estructuras de control Contextualización En la sesión anterior vimos las estructuras condicionales, mediante éstas somos capaces de elegir qué expresiones usaremos, pero
Programación Estructurada Sesión 4:Estructuras de control Contextualización En la sesión anterior vimos las estructuras condicionales, mediante éstas somos capaces de elegir qué expresiones usaremos, pero
ASIGNATURA: BIOINFORMÁTICA
 Página 1 de 5 CARACTERÍSTICAS GENERALES* Tipo: Formación básica, Obligatoria, Optativa Trabajo de fin de grado, Prácticas externas Duración: Cuatrimestral Semestre/s: 5 Número de créditos ECTS: 6 Idioma/s:
Página 1 de 5 CARACTERÍSTICAS GENERALES* Tipo: Formación básica, Obligatoria, Optativa Trabajo de fin de grado, Prácticas externas Duración: Cuatrimestral Semestre/s: 5 Número de créditos ECTS: 6 Idioma/s:
CAPÍTULO 3. GRASP (Greedy Randomized Adaptive Search Procedures). Los problemas de optimización surgen de las situaciones de aplicación práctica.
 CAPÍTULO 3 GRASP (Greedy Randomized Adaptive Search Procedures). Los problemas de optimización surgen de las situaciones de aplicación práctica. Estos problemas se aplican en distintas áreas, tales como:
CAPÍTULO 3 GRASP (Greedy Randomized Adaptive Search Procedures). Los problemas de optimización surgen de las situaciones de aplicación práctica. Estos problemas se aplican en distintas áreas, tales como:
Aplicaciones guiadas: Blast. Genome Browsers.
 Curso de Formación UEB Herramientas Bioinformáticas para la Investigación Biomédica 1 r bloque (20/06/2012) Introducción a la Bioinformática y a las Bases de Datos 3 a sesión Aplicaciones guiadas: Blast.
Curso de Formación UEB Herramientas Bioinformáticas para la Investigación Biomédica 1 r bloque (20/06/2012) Introducción a la Bioinformática y a las Bases de Datos 3 a sesión Aplicaciones guiadas: Blast.
Alineamientos de Secuencias. CeCalCULA - C.P.T.M. Mérida. Venezuela.
 Alineamientos de Secuencias Análisis comparativo El alineamiento de secuencias es similar a otros tipos de análisis comparativo. En ambos es necesario cuantificar las similitudes y diferencias (scoring)
Alineamientos de Secuencias Análisis comparativo El alineamiento de secuencias es similar a otros tipos de análisis comparativo. En ambos es necesario cuantificar las similitudes y diferencias (scoring)
Introducción a la Computación Evolutiva
 Introducción a la Computación Evolutiva Departamento de Computación CINVESTAV-IPN Av. IPN No. 2508 Col. San Pedro Zacatenco México, D.F. 07300 email: ccoello@cs.cinvestav.mx http: //delta.cs.cinvestav.mx/~ccoello
Introducción a la Computación Evolutiva Departamento de Computación CINVESTAV-IPN Av. IPN No. 2508 Col. San Pedro Zacatenco México, D.F. 07300 email: ccoello@cs.cinvestav.mx http: //delta.cs.cinvestav.mx/~ccoello
Diseño Computarizado 15023
 Diseño Computarizado 15023 PROGRAMACIÓN: FORTRAN Profesor: Claudio García Herrera Departamento de Ingeniería Mecánica Universidad de Santiago de Chile Índice 1 Introducción 2 Estructura 3 Sentencias y
Diseño Computarizado 15023 PROGRAMACIÓN: FORTRAN Profesor: Claudio García Herrera Departamento de Ingeniería Mecánica Universidad de Santiago de Chile Índice 1 Introducción 2 Estructura 3 Sentencias y
ESTRUCTURA SECUENCIAL ESTRUCTURA SELECTIVA
 ESTRUCTURA SECUENCIAL Es aquélla en la que una acción (instrucción) sigue a otra en secuencia. Las tareas se suceden de tal modo que la salida de una es la entrada de la siguiente y así sucesivamente hasta
ESTRUCTURA SECUENCIAL Es aquélla en la que una acción (instrucción) sigue a otra en secuencia. Las tareas se suceden de tal modo que la salida de una es la entrada de la siguiente y así sucesivamente hasta
UNA MEJOR RESOLUCION: ARBOLES DE ESPECIES O DATOS CONCATENADOS (EVIDENCIA TOTAL)? Angie Tamara Guevara
 Introducción UNA MEJOR RESOLUCION: ARBOLES DE ESPECIES O DATOS CONCATENADOS (EVIDENCIA TOTAL)? Angie Tamara Guevara 2050163 Actualmente las secuencias de genes múltiples se incrementan a una tasa muy alta
Introducción UNA MEJOR RESOLUCION: ARBOLES DE ESPECIES O DATOS CONCATENADOS (EVIDENCIA TOTAL)? Angie Tamara Guevara 2050163 Actualmente las secuencias de genes múltiples se incrementan a una tasa muy alta
Búsqueda Informada. Algoritmos primero el mejor Algoritmos de búsqueda local
 Búsqueda Informada Algoritmos primero el mejor Algoritmos de búsqueda local Algoritmos primero el mejor Búsqueda primero el mejor Búsqueda Voraz A* Algoritmos primero el mejor Familia de algoritmos de
Búsqueda Informada Algoritmos primero el mejor Algoritmos de búsqueda local Algoritmos primero el mejor Búsqueda primero el mejor Búsqueda Voraz A* Algoritmos primero el mejor Familia de algoritmos de
Clasificación Clasific NO SUPERV SUPER ISAD IS A AD AGRUPAMIENTO
 Clasificación NO SUPERVISADA AGRUPAMIENTO Clasificación No Supervisada Se trata de construir clasificadores sin información a priori, o sea, a partir de conjuntos de patrones no etiquetados Objetivo: Descubrir
Clasificación NO SUPERVISADA AGRUPAMIENTO Clasificación No Supervisada Se trata de construir clasificadores sin información a priori, o sea, a partir de conjuntos de patrones no etiquetados Objetivo: Descubrir
Programación Dinámica
 Programación Dinámica Es aplicada típicamente a problemas de optimización, donde puede haber muchas soluciones, cada una tiene un valor asociado y prentendemos obtener la solución con valor óptimo. Al
Programación Dinámica Es aplicada típicamente a problemas de optimización, donde puede haber muchas soluciones, cada una tiene un valor asociado y prentendemos obtener la solución con valor óptimo. Al
Algoritmos de búsqueda en grafos II
 II Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 12 de febrero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Búsqueda en grafos II 12 de febrero de 2018 1 / 26 1 Algoritmos de búsqueda en grafos Dr.
II Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 12 de febrero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Búsqueda en grafos II 12 de febrero de 2018 1 / 26 1 Algoritmos de búsqueda en grafos Dr.
C a l ses P P y y NP C a l se P C a l se N P N P [No N n o -De D te t rmin i i n s i ti t c i Polynomial-tim i e]
![C a l ses P P y y NP C a l se P C a l se N P N P [No N n o -De D te t rmin i i n s i ti t c i Polynomial-tim i e] C a l ses P P y y NP C a l se P C a l se N P N P [No N n o -De D te t rmin i i n s i ti t c i Polynomial-tim i e]](/thumbs/51/28301210.jpg) Análisis y Diseño de Algoritmos Introducción Análisis y Diseño de Algoritmos Concepto de algoritmo Resolución de problemas Clasificación de problemas Algorítmica Análisis de la eficiencia de los algoritmos
Análisis y Diseño de Algoritmos Introducción Análisis y Diseño de Algoritmos Concepto de algoritmo Resolución de problemas Clasificación de problemas Algorítmica Análisis de la eficiencia de los algoritmos
Predicción de estructuras de proteínas y ARN
 Predicción de estructuras de proteínas y ARN Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 19 de julio del 2012 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Predicción de estructuras 19 de julio del 2012
Predicción de estructuras de proteínas y ARN Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 19 de julio del 2012 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Predicción de estructuras 19 de julio del 2012
Teoría de grafos y optimización en redes
 Teoría de grafos y optimización en redes José María Ferrer Caja Universidad Pontificia Comillas Definiciones básicas Grafo: Conjunto de nodos (o vértices) unidos por aristas G = (V,E) Ejemplo V = {,,,,
Teoría de grafos y optimización en redes José María Ferrer Caja Universidad Pontificia Comillas Definiciones básicas Grafo: Conjunto de nodos (o vértices) unidos por aristas G = (V,E) Ejemplo V = {,,,,
Son una clase particular de los algoritmos evolutivos.
 Algoritmos Genéticos (AGs) Los AGs son técnicas de resolución para problemas de Búsqueda Optimización Aprendizaje en máquinas Son una clase particular de los algoritmos evolutivos. Los AGs intentan resolver
Algoritmos Genéticos (AGs) Los AGs son técnicas de resolución para problemas de Búsqueda Optimización Aprendizaje en máquinas Son una clase particular de los algoritmos evolutivos. Los AGs intentan resolver
Problema 9: Diseño de Proteínas
 Problema 9: Diseño de Proteínas Tomado de: The Ten Most Wanted Solutions In Protein Bioinformatics por Anna Tramontano Seminario de Bioinformática Jorge Hernán Victoria Moreno Octubre 23 de 2009 Agenda
Problema 9: Diseño de Proteínas Tomado de: The Ten Most Wanted Solutions In Protein Bioinformatics por Anna Tramontano Seminario de Bioinformática Jorge Hernán Victoria Moreno Octubre 23 de 2009 Agenda
Palabras reservadas de C++ y C. Una palabra reservada no puede declararse como un identificador, esto haría un conflicto entre conectores y funciones.
 Palabras reservadas de C++ y C Una palabra reservada no puede declararse como un identificador, esto haría un conflicto entre conectores y funciones. A continuación se muestra el link del listado de palabras
Palabras reservadas de C++ y C Una palabra reservada no puede declararse como un identificador, esto haría un conflicto entre conectores y funciones. A continuación se muestra el link del listado de palabras
Unidad 3. Estructuras de control en diagrama de Flujo y pseudocódigo Tema Iteración o Repetitivas (repite - mientras) Juan Pablo Cobá Juárez Pegueros
 Unidad 3. Estructuras de control en diagrama de Flujo y pseudocódigo Tema Iteración o Repetitivas (repite - mientras) Juan Pablo Cobá Juárez Pegueros Programación Básica Bioingeniería Médica Facultad de
Unidad 3. Estructuras de control en diagrama de Flujo y pseudocódigo Tema Iteración o Repetitivas (repite - mientras) Juan Pablo Cobá Juárez Pegueros Programación Básica Bioingeniería Médica Facultad de
Tema: Recorrido de Grafos. Ruta más corta
 PED104. Guía N 12 Página 1 Facultad: Ingeniería Escuela: Computación Asignatura: Programación con Estructuras de Datos Tema: Recorrido de Grafos. Ruta más corta Competencia Desarrolla sistemas de información
PED104. Guía N 12 Página 1 Facultad: Ingeniería Escuela: Computación Asignatura: Programación con Estructuras de Datos Tema: Recorrido de Grafos. Ruta más corta Competencia Desarrolla sistemas de información
PARTE II: ALGORÍTMICA
 Programa de teoría Parte I. Estructuras de Datos.. Abstracciones y especificaciones.. Conjuntos y diccionarios.. Representación de conjuntos mediante árboles. 4. Grafos. Parte II. Algorítmica.. Análisis
Programa de teoría Parte I. Estructuras de Datos.. Abstracciones y especificaciones.. Conjuntos y diccionarios.. Representación de conjuntos mediante árboles. 4. Grafos. Parte II. Algorítmica.. Análisis
Uso de herramientas para alineación
 Uso de herramientas para alineación de secuencias y creación de árboles filogenéticos para la determinación de especies Using tools for sequence alignment and outline of phylogenetic trees to determine
Uso de herramientas para alineación de secuencias y creación de árboles filogenéticos para la determinación de especies Using tools for sequence alignment and outline of phylogenetic trees to determine
Construcción y análisis de árboles filogenéticos. Antonio Gómez Tato
 Construcción y análisis de árboles filogenéticos Antonio Gómez Tato Introducción Árboles filogenéticos Surgen a partir de la teoría de la evolución de Darwin. Son representaciones gráficas de las relaciones
Construcción y análisis de árboles filogenéticos Antonio Gómez Tato Introducción Árboles filogenéticos Surgen a partir de la teoría de la evolución de Darwin. Son representaciones gráficas de las relaciones
Sentencia1 Sentencia2 Cuerpo del bucle
 PROGRAMACIÓN 10 Prof. Dolores Cuiñas H. Recuerde que estos son apuntes muy simplificados que deberá completar con la bibliografía recomendada APUNTES Nº 6 ESTRUCTURAS DE CONTROL REPETITIVAS: son aquellas
PROGRAMACIÓN 10 Prof. Dolores Cuiñas H. Recuerde que estos son apuntes muy simplificados que deberá completar con la bibliografía recomendada APUNTES Nº 6 ESTRUCTURAS DE CONTROL REPETITIVAS: son aquellas
Metaheurísticas
 UNIVERSIDAD DE GRANADA E.T.S.I. INFORMÁTICA Y TELECOMUNICACIÓN Departamento de Ciencias de la Computación e Inteligencia Artificial Metaheurísticas http://sci2s.ugr.es/graduatecourses/metaheuristicas https://decsai.ugr.es
UNIVERSIDAD DE GRANADA E.T.S.I. INFORMÁTICA Y TELECOMUNICACIÓN Departamento de Ciencias de la Computación e Inteligencia Artificial Metaheurísticas http://sci2s.ugr.es/graduatecourses/metaheuristicas https://decsai.ugr.es
Programación Estructurada
 Programación Estructurada Técnica de programación que consiste en construir programas de fácil comprensión. Es mucho más sencillo entender la codificación del programa, que se habrá hecho en diferentes
Programación Estructurada Técnica de programación que consiste en construir programas de fácil comprensión. Es mucho más sencillo entender la codificación del programa, que se habrá hecho en diferentes
Estructuras de datos geométricas
 y Half-edge Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 12 de febrero del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) y Half-edge 12 de febrero del 2013 1 / 32 1 Estructuras de datos geométricas
y Half-edge Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 12 de febrero del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) y Half-edge 12 de febrero del 2013 1 / 32 1 Estructuras de datos geométricas
ESTIMACIÓN DE TIEMPO Y COSTO DE PRODUCTOS SOFTWARE
 Análisis y costo de algoritmos Algoritmos Un algoritmo es un conjunto de pasos claramente definidos que a partir de un cierta entrada (input) produce una determinada salida (output) Algoritmos y programas
Análisis y costo de algoritmos Algoritmos Un algoritmo es un conjunto de pasos claramente definidos que a partir de un cierta entrada (input) produce una determinada salida (output) Algoritmos y programas
Investigación en evolución
 Investigación en evolución Análisis de secuencias de: nucleótidos en DNA (genómica) aminoácidos en Proteínas (proteómica) Comparación de secuencias de: nucleótidos aminoácidos Bioinformática Análisis de
Investigación en evolución Análisis de secuencias de: nucleótidos en DNA (genómica) aminoácidos en Proteínas (proteómica) Comparación de secuencias de: nucleótidos aminoácidos Bioinformática Análisis de
Análisis y anotación de una secuencia mediante las herramientas y bases de datos de UCSC Genome Bioinformatics & Galaxy
 Análisis y anotación de una secuencia mediante las herramientas y bases de datos de UCSC Genome Bioinformatics & Galaxy Master de Genética y Evolución 2011/2012 Analisis de Secuencias Michael Hackenberg
Análisis y anotación de una secuencia mediante las herramientas y bases de datos de UCSC Genome Bioinformatics & Galaxy Master de Genética y Evolución 2011/2012 Analisis de Secuencias Michael Hackenberg
Efectos de los alineamientos
 Efectos de los alineamientos Una evaluación empírica mediante el método de parsimonia con alineamientos implícitos generados por POY vs los alineamientos ClustalW en topologías de mamíferos. Claudia Infante
Efectos de los alineamientos Una evaluación empírica mediante el método de parsimonia con alineamientos implícitos generados por POY vs los alineamientos ClustalW en topologías de mamíferos. Claudia Infante
Bioinformática Clásica
 Tema 4: Alineamiento Múltiple y Filogenias (1) Sección 1: Alineamiento Múltiple Dr. Oswaldo Trelles Universidad de Málaga El alineamiento múltiple de secuencias (AM) es con frecuencia el punto de partida,
Tema 4: Alineamiento Múltiple y Filogenias (1) Sección 1: Alineamiento Múltiple Dr. Oswaldo Trelles Universidad de Málaga El alineamiento múltiple de secuencias (AM) es con frecuencia el punto de partida,
Análisis multivariado
 Análisis multivariado -Las técnicas multivariadas permiten establecer relaciones de similitud global (o fenéticas) entre unidades de estudio, sobre la base de la evidencia que brindan sus caracteres -A
Análisis multivariado -Las técnicas multivariadas permiten establecer relaciones de similitud global (o fenéticas) entre unidades de estudio, sobre la base de la evidencia que brindan sus caracteres -A
Introducción a la Bioinformática
 Introducción a la Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 16 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Introducción a la 16 de mayo del 2013 1 / 37 1 Introducción Qué es bioinformática?
Introducción a la Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 16 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Introducción a la 16 de mayo del 2013 1 / 37 1 Introducción Qué es bioinformática?
Unidad 3. Estructuras de control en diagrama de Flujo y pseudocódigo Tema Iteración o Repetitivas (mientras)
 Juan Pablo Cobá Juárez Pegueros Programación Básica Bioingeniería Médica Facultad de Medicina Unidad 3. Estructuras de control en diagrama de Flujo y pseudocódigo Tema Iteración o Repetitivas (mientras)
Juan Pablo Cobá Juárez Pegueros Programación Básica Bioingeniería Médica Facultad de Medicina Unidad 3. Estructuras de control en diagrama de Flujo y pseudocódigo Tema Iteración o Repetitivas (mientras)
Todos los Pares de Rutas más Cortas (All-Pairs Shortest Paths) DR. JESÚS A. GONZÁLEZ BERNAL CIENCIAS COMPUTACIONALES INAOE
 Todos los Pares de Rutas más Cortas (All-Pairs Shortest Paths) DR. JESÚS A. GONZÁLEZ BERNAL CIENCIAS COMPUTACIONALES INAOE Problema de Encontrar todos los Pares de Rutas más Cortas 2 Encontrar las rutas
Todos los Pares de Rutas más Cortas (All-Pairs Shortest Paths) DR. JESÚS A. GONZÁLEZ BERNAL CIENCIAS COMPUTACIONALES INAOE Problema de Encontrar todos los Pares de Rutas más Cortas 2 Encontrar las rutas
Cuadrados fractáureos
 1 Introducción Cuadrados fractáureos J. Romañach y M. Toboso Julio 2016 El número de fractales conocidos crece constantemente, y en este momento se aproxima a los 150 si consideramos tanto los deterministas
1 Introducción Cuadrados fractáureos J. Romañach y M. Toboso Julio 2016 El número de fractales conocidos crece constantemente, y en este momento se aproxima a los 150 si consideramos tanto los deterministas
Práctica 2. Algoritmos de búsqueda local (local search algorithms) y algoritmos avariciosos (greedy algorithms)
 PLANIFICACIÓN Y GESTIÓN DE REDES Grado en Ingeniería Telemática Curso 2012-2013 Práctica 2. Algoritmos de búsqueda local (local search algorithms) y algoritmos avariciosos (greedy algorithms) Autor: Pablo
PLANIFICACIÓN Y GESTIÓN DE REDES Grado en Ingeniería Telemática Curso 2012-2013 Práctica 2. Algoritmos de búsqueda local (local search algorithms) y algoritmos avariciosos (greedy algorithms) Autor: Pablo
Guía práctica de estudio 06: Estructuras de repetición
 Guía práctica de estudio 06: Estructuras de repetición Elaborado por: M.C. M. Angélica Nakayama C. Ing. Jorge A. Solano Gálvez Autorizado por: M.C. Alejandro Velázquez Mena Guía práctica de estudio 06:
Guía práctica de estudio 06: Estructuras de repetición Elaborado por: M.C. M. Angélica Nakayama C. Ing. Jorge A. Solano Gálvez Autorizado por: M.C. Alejandro Velázquez Mena Guía práctica de estudio 06:
Algoritmos y estructuras de datos
 Algoritmos y estructuras de datos Dr. Eduardo A. Rodríguez Tello Laboratorio de Tecnologías de Información Cinvestav Tamaulipas ertello@tamps.cinvestav.mx Cursos de inducción a la MCC Cinvestav Tamaulipas
Algoritmos y estructuras de datos Dr. Eduardo A. Rodríguez Tello Laboratorio de Tecnologías de Información Cinvestav Tamaulipas ertello@tamps.cinvestav.mx Cursos de inducción a la MCC Cinvestav Tamaulipas
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA. Evolución Molecular y Filogenia
 TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Evolución Molecular y Filogenia Ignacio Pérez Hurtado de Mendoza Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia Artificial
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Evolución Molecular y Filogenia Ignacio Pérez Hurtado de Mendoza Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia Artificial
Unidad 4. Estructuras de control Tema. Estructuras de Iteración (do-while)
 Unidad 4. Estructuras de control Tema. Estructuras de Iteración (do-while) Juan Pablo Cobá Juárez Pegueros Programación Avanzada Bioingeniería Médica Facultad de Medicina 05/09/2017 Contenido Qué son las
Unidad 4. Estructuras de control Tema. Estructuras de Iteración (do-while) Juan Pablo Cobá Juárez Pegueros Programación Avanzada Bioingeniería Médica Facultad de Medicina 05/09/2017 Contenido Qué son las
CAPITULO 2: MARCO TEÓRICO. En el desarrollo de este capítulo se presentan descripciones generales,
 CAPITULO 2: MARCO TEÓRICO En el desarrollo de este capítulo se presentan descripciones generales, definiciones y métodos, que nos pueden ayudar a entender con claridad el método que desarrolló en esta
CAPITULO 2: MARCO TEÓRICO En el desarrollo de este capítulo se presentan descripciones generales, definiciones y métodos, que nos pueden ayudar a entender con claridad el método que desarrolló en esta
Algoritmos de fuerza bruta
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 29 de enero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Algoritmos de fuerza bruta 29 de enero de 2018 1 / 26 1 Algoritmos de fuerza bruta Introducción
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 29 de enero de 2018 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Algoritmos de fuerza bruta 29 de enero de 2018 1 / 26 1 Algoritmos de fuerza bruta Introducción
Inteligencia Artificial
 Algoritmos genéticos Bases En la naturaleza todos los seres vivos se enfrentan a problemas que deben resolver con éxito, como conseguir más luz solar o conseguir comida. La Computación Evolutiva interpreta
Algoritmos genéticos Bases En la naturaleza todos los seres vivos se enfrentan a problemas que deben resolver con éxito, como conseguir más luz solar o conseguir comida. La Computación Evolutiva interpreta
Metaheurísticas. Seminario 5. Manejo de restricciones en metaheurísticas. 1. Introducción: Optimización y Restricciones
 Metaheurísticas Seminario 5. Manejo de restricciones en metaheurísticas 1. Introducción: Optimización y Restricciones 2. Manejo de Restricciones en Metaheurísticas 3. Algunos Ejemplos 1 Introducción: Optimización
Metaheurísticas Seminario 5. Manejo de restricciones en metaheurísticas 1. Introducción: Optimización y Restricciones 2. Manejo de Restricciones en Metaheurísticas 3. Algunos Ejemplos 1 Introducción: Optimización
Dadas dos secuencias, X = (x 1 x 2 x m ) e Y = (y 1 y 2 y n ), encontrar la forma de alinearlas con un coste mínimo.
 Análisis y Diseño de Algoritmos Aplicaciones Aplicaciones Bioinformática: Alineación de secuencias de ADN Ingeniería del Software: Detección de clones (fragmentos de código duplicado) Procesamiento del
Análisis y Diseño de Algoritmos Aplicaciones Aplicaciones Bioinformática: Alineación de secuencias de ADN Ingeniería del Software: Detección de clones (fragmentos de código duplicado) Procesamiento del
Procedimientos de búsqueda miopes aleatorizados y adaptativos (GRASP)
 Procedimientos de búsqueda miopes aleatorizados y adaptativos (GRASP) Se basan en la premisa de que soluciones iniciales diversas y de buena calidad juegan un papel importante en el éxito de métodos de
Procedimientos de búsqueda miopes aleatorizados y adaptativos (GRASP) Se basan en la premisa de que soluciones iniciales diversas y de buena calidad juegan un papel importante en el éxito de métodos de
ÁRBOLES FILOGENÉTICOS
 ÁRBOLES FILOGENÉTICOS Por qué usar filogenias? El conocimiento del pasado es importante para poder resolver muchas cuestiones relacionadas con procesos biológicos. Las filogenias nos permiten obtener relaciones
ÁRBOLES FILOGENÉTICOS Por qué usar filogenias? El conocimiento del pasado es importante para poder resolver muchas cuestiones relacionadas con procesos biológicos. Las filogenias nos permiten obtener relaciones
Tema 01: Algoritmia y diagramas de flujo. Estructuras de datos (Prof. Edgardo A. Franco)
 Tema 1 Estructuras de datos (Prof. Edgardo A. Franco) Contenido Algoritmia Qué es un algoritmo? Métodos algorítmicos Diagrama de flujo Símbolos utilizados en los diagramas de flujo Reglas para la construcción
Tema 1 Estructuras de datos (Prof. Edgardo A. Franco) Contenido Algoritmia Qué es un algoritmo? Métodos algorítmicos Diagrama de flujo Símbolos utilizados en los diagramas de flujo Reglas para la construcción
UNIDAD DE CONTROL SEGMENTADA OPTIMIZACIÓN EN LA PLANIFICACIÓN DE PIPES
 Todos los derechos de propiedad intelectual de esta obra pertenecen en exclusiva a la Universidad Europea de Madrid, S.L.U. Queda terminantemente prohibida la reproducción, puesta a disposición del público
Todos los derechos de propiedad intelectual de esta obra pertenecen en exclusiva a la Universidad Europea de Madrid, S.L.U. Queda terminantemente prohibida la reproducción, puesta a disposición del público
Tutorías: 10:00 a 14:00 de lunes a viernes en el L (Solicitud mediante )
 1 José María Font Fernández jm.font@upm.es Tutorías: 10:00 a 14:00 de lunes a viernes en el L-3101. (Solicitud mediante email) http://dl.dropbox.com/u/1074349/computacionevolutiva.pdf 2 o La Computación
1 José María Font Fernández jm.font@upm.es Tutorías: 10:00 a 14:00 de lunes a viernes en el L-3101. (Solicitud mediante email) http://dl.dropbox.com/u/1074349/computacionevolutiva.pdf 2 o La Computación
Cubiertas convexas. Dr. Eduardo A. RODRÍGUEZ TELLO. 18 de enero del CINVESTAV-Tamaulipas
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 18 de enero del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Cubiertas convexas 18 de enero del 2013 1 / 30 1 Cubiertas convexas Introducción El problema
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 18 de enero del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Cubiertas convexas 18 de enero del 2013 1 / 30 1 Cubiertas convexas Introducción El problema
Programa de teoría. Algoritmos y Estructuras de Datos II. 3. Algoritmos voraces. 1. Análisis de algoritmos 2. Divide y vencerás
 Programa de teoría Algoritmos y Estructuras de Datos II 1. Análisis de algoritmos 2. Divide y vencerás 3. Algoritmos voraces 4. Programación dinámica 5. Backtracking 6. Ramificación y poda A.E.D. II 1
Programa de teoría Algoritmos y Estructuras de Datos II 1. Análisis de algoritmos 2. Divide y vencerás 3. Algoritmos voraces 4. Programación dinámica 5. Backtracking 6. Ramificación y poda A.E.D. II 1
INFORMATICA TECNICATURA DE NIVEL SUPERIOR ALGUNOS EJERCICIOS DE SELECCIÓN E ITERACION
 INFORMATICA TECNICATURA DE NIVEL SUPERIOR ALGUNOS EJERCICIOS DE SELECCIÓN E ITERACION DIIAGRAMAS DE FLUJO Un diagrama de flujo es un dibujo que utiliza símbolos estándar de diagramación de algoritmos para
INFORMATICA TECNICATURA DE NIVEL SUPERIOR ALGUNOS EJERCICIOS DE SELECCIÓN E ITERACION DIIAGRAMAS DE FLUJO Un diagrama de flujo es un dibujo que utiliza símbolos estándar de diagramación de algoritmos para
Congruencia- Árboles de consenso Soporte de grupos
 Congruencia- Árboles de consenso Soporte de grupos Los agrupamientos obtenidos a partir de análisis filogenéticos basados en distintos sets o conjuntos de datos son con frecuencia incongruentes. Comparaciones
Congruencia- Árboles de consenso Soporte de grupos Los agrupamientos obtenidos a partir de análisis filogenéticos basados en distintos sets o conjuntos de datos son con frecuencia incongruentes. Comparaciones
Práctica 2 - Ejercicio 2.8
 Algoritmos y Estructura de Datos III Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires 27 de Marzo de 2013 2.8 - Euclides 2.8. a. Escribir el algoritmo de Euclides para calcular el
Algoritmos y Estructura de Datos III Facultad de Ciencias Exactas y Naturales, Universidad de Buenos Aires 27 de Marzo de 2013 2.8 - Euclides 2.8. a. Escribir el algoritmo de Euclides para calcular el
