Por regla general, las búsquedas con BLAST obedecen a uno de estos dos objetivos:
|
|
|
- Lucía Nieto Fernández
- hace 6 años
- Vistas:
Transcripción
1 BLAST en el servidor del NCBI BLAST es la herramienta bioinformática más utilizada en todo el mundo. Compara una secuencia problema (query sequence) de nucleótidos o de proteínas con todas las secuencias de una BD para encontrar regiones de similitud local. Además, calcula la significación estadística de los resultados. BLAST permite inferir relaciones funcionales, estructurales o evolutivas entre dos secuencias de modo que se pueden identificar nuevos miembros de una familia de genes o de proteínas. Por regla general, las búsquedas con BLAST obedecen a uno de estos dos objetivos: Localizar una secuencia dentro de otra, como cuando se quiere encontrar la ubicación de un gen, oligo, cdna o EST en un genoma o cuando se quiere determinar la estructura de un gen (localizar los intrones, los exones y las regiones reguladoras) Explorar las BD en busca de secuencias relacionadas funcional o evolutivamente Hay dos formas de utilizar BLAST: conectados a Internet (on-line). Además de la página del NCBI ( hay muchas otras páginas que ofrecen esta herramienta. En estas condiciones, corremos el peligro de que alguien pueda acceder a los resultados de nuestra búsqueda. sin conexión a Internet. Cuando interesa mantener la confidencialidad de los resultados, lo mejor es descargar el programa e instalarlo en un ordenador personal. El programa se puede descargar gratuitamente desde la dirección: ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/latest/, las BD se pueden descargar desde: ftp://ftp.ncbi.nlm.nih.gov/blast/db/ y las instrucciones de instalación y los comandos de uso se pueden encontrar en la dirección Para utilizar BLAST en el servidor del NCBI hay que: 1.- Ir a la dirección Seleccionar la variante del programa que se va a utilizar 3.- Introducir la secuencia problema. Se puede poner directamente un código de acceso, un código gi (gene identifier), o una secuencia en formato FASTA 4.- Seleccionar la base de datos 5.- Ajustar diversos parámetros de la búsqueda (E-value, word size, scoring parameters: substitution matrix, gap penalties) 6.- BLAST! Versiones del programa BLAST (NCBI) Existen diversas versiones del programa BLAST. Es importante saber cuál es la que mejor se adapta a los objetivos de la búsqueda. Para ello, hay que tener en cuenta 3 factores: (1) la naturaleza de la secuencia problema, (2) el objetivo de la búsqueda y (3) la BD donde se va a llevar a cabo la búsqueda.
2 En la siguiente Tabla se muestran las distintas versiones del programa BLAST: Blastn Compara una secuencia de nucleótidos con una BD que también contiene secuencias de nucleótidos. Se utiliza para: Localizar oligonucleótidos, cdna, EST, productos de PCR o elementos repetitivos en un genoma Identificación de secuencias de DNA y anotación del DNA genómico Localizar secuencias homólogas en especies distintas (genes de RNA o de proteínas, regiones reguladoras, etc.) Generación de contigs a partir de las lecturas más cortas obtenidas durante el proceso de secuenciación Eliminar subsecuencias pertenecientes a vectores Detección de contaminaciones Este tipo de búsqueda no es el más apropiado para encontrar regiones que codifican proteínas homólogas en otros organismos. En este caso es mejor hacer búsquedas a nivel de proteína directamente con blastp o traducir la secuencia problema, la BD, o ambas, según las seis pautas de lectura posibles. Son tres las razones que explican esta circunstancia: (1) la degeneración del código genético, (2) las secuencias proteicas albergan más información que las secuencias de nucleótidos y (3) las matrices de sustitución utilizadas para el alineamiento de secuencias de proteínas son más sofisticadas que las utilizadas para alinear secuencias de nucleótidos. Dentro del programa blastn se pueden seleccionar varios algoritmos: MEGABLAST: diseñado para identificar una secuencia problema (el parecido es del 100%) o para encontrar secuencias muy parecidas (> 95% de residuos idénticos). Es muy rápido porque utiliza un tamaño de palabra (el parámetro w) de 28 residuos. Blastn: Es más sensible que el anterior porque utiliza por defecto un parámetro w = 11, pero es más lento. Está diseñado para encontrar secuencias similares en organismos distintos. Si es preciso, también puede buscar con w = 7, aumentando la sensibilidad pero reduciendo notablemente la velocidad. MEGABLAST discontinuo: también está diseñado para encontrar secuencias similares en organismos distintos. Utiliza w = 11 y, en estas mismas condiciones, es más sensible y eficaz que blastn porque ignora algunas bases (la tercera de cada codón) y porque al buscar las palabras de la secuencia problema
3 en las BD no es necesario que ambas sean idénticas, sino que permite la presencia de discontinuidades. Cuando se introduce una secuencia problema para hacer búsquedas en una BD, BLASTN utiliza las dos hebras de la molécula de DNA. La hebra plus es la que se ha introducido en formato FASTA y la complementaria inversa es la hebra minus. Cuando la región de similitud local está situada en la misma hebra, al representar el alineamiento las dos secuencias (la secuencia problema y la secuencia de la BD) están señaladas como "plus" y las coordenadas de las dos secuencias avanzan en sentido creciente. Cuando la hebra minus de la secuencia problema es similar a una secuencia de la BD, el alineamiento representa la hebra plus de la secuencia problema y la hebra minus de la BD. Por tanto, las coordenadas de la secuencia de la BD están colocadas en sentido decreciente. Blastp Este programa compara una secuencia de aminoácidos con una BD que también contiene secuencias proteicas. Se utiliza para: identificar una secuencia problema: en este caso, el parecido es del 100% y el programa genera un alineamiento global. Para que la identificación sea inequívoca puede ser una buena idea desactivar el filtro de las regiones de poca complejidad (low complexity filter) encontrar secuencias parecidas en una BD de secuencias proteicas. Si el parecido es grande, puede tratarse de proteínas homólogas y es bastante probable que las anotaciones de las secuencias homólogas también sean válidas para la secuencia problema. BLAST permite reunir una colección de secuencias homólogas procedentes de distintos organismos para hacer alineamientos múltiples de secuencias o análisis filogenéticos. localizar regiones de similitud: en este caso el parecido se limita a una región de las secuencias y el programa genera alineamientos locales que pueden corresponder a dominios conservados.
4 Dentro del programa blastp se pueden seleccionar varios algoritmos: Blastp compara una secuencia proteica con una BD de proteínas PSI-BLAST utiliza los resultados de blastp para construir una matriz de puntuación específica de la posición (PSSM) y, a continuación, localizar secuencias con un parentesco remoto PHI-BLAST busca proteínas que contienen un patrón especificado por el usuario y que, además del patrón, presentan otras regiones de similitud con la secuencia problema DELTA-BLAST construye una PSSM basándose en una búsqueda en la BD de dominios conservados y, a continuación, hace una búsqueda en una BD de proteínas Blastx La secuencia problema es una secuencia de nucleótidos. El programa traduce esta secuencia en sus seis posibles marcos de lectura (tres marcos de lecturas por hebra) y compara estas secuencias traducidas con una BD de proteínas. Es un programa lento que se usa cuando se tiene sospecha de que la secuencia problema codifica una proteína pero no se sabe exactamente cuál. Si la secuencia problema corresponde a una región no codificante del DNA, blastx no encontrará nada. Se utiliza para: Localizar genes que codifican proteínas en el DNA genómico Determinar si un transcrito (convertido en cdna o en EST) codifica alguna proteína conocida Definir las regiones codificantes y no codificantes de un mrna A la hora de interpretar los alineamientos generados por blastx hay que tener en cuenta la hebra, la pauta de lectura y las coordenadas. En la hebra plus, las pautas de lectura (frame) se denominan +1, +2 y +3. En la hebra minus, las pautas de lectura se denominan 1, 2 y 3. Las coordenadas de la secuencia problema aumentan de tres en tres (parte a de la figura inferior) porque cada aminoácido corresponde a tres nucleótidos. Si el alineamiento se produce en la hebra minus de la secuencia problema (parte b de la figura inferior, frame = 1), las coordenadas de la secuencia problema aparecen en orden descendente.
5 TBlastn Compara una secuencia proteica con una BD de nucléotidos. Para ello, primero traduce todas las secuencias de nucleótidos de la BD en sus seis marcos de lectura y luego realiza la comparación. TBLASTN es un programa lento que se usa cuando el análisis con Blastp no ha tenido éxito porque la proteína no aparece en las BD. Sin embargo, es posible que las BD de EST o de proyectos genómicos en curso (que carecen de anotaciones) incluyan alguna secuencia que pueda corresponder al transcrito que codifica esa proteína o una similar. Se utiliza para: Localizar una proteína en el DNA genómico, lo que permite ver si existen elementos reguladores cerca de la región codificante del gen y localizar exones Buscar en BD de EST los transcritos que correspondan a la secuencia problema o a una secuencia parecida Hay que tener cuidado con los resultados obtenidos con esta variante de Blast, porque una buena cantidad de las secuencias traducidas no son proteínas que existan en la naturaleza. Tblastx Compara una secuencia de nucleótidos con una BD de nucleótidos, pero primero traduce la secuencia problema y las secuencias de las BD en los seis marcos de lectura posibles. Se aprovecha del hecho de que las secuencias codificantes evolucionan más lentamente que el DNA adyacente. Se trata de una búsqueda más sensible que BLASTP, pero requiere mucho esfuerzo computacional y sólo debería utilizarse como último recurso y, preferentemente, sin conexión a Internet. Se utiliza para: detectar nuevos genes en secuencias genómicas (de la misma especie o de especies distintas), especialmente los que resultan difíciles de encontrar por los métodos tradicionales (genes dentro de otros genes, procesamientos alternativos o genes con bajos niveles de expresión) descubrir transcritos (en forma de cdna o EST) cuyos productos aún no están incluidos en las BD Los alineamientos generados por tblastx son difíciles de interpretar porque hay que tener en cuenta la hebra, la pauta de lectura y las coordenadas, tanto en la secuencia problema como en la secuencia de la BD.
6 PSI-BLAST (PSI = Position Specific Iterative) Si una búsqueda con BLASTP no ha conseguido encontrar proteínas similares o si muchos de los resultados son dudosos ("hypothetical protein", "predicted" o "similar to..."), podemos utilizar PSI-BLAST. Este programa es el más sensible de todos y es muy útil a la hora de (1) encontrar proteínas con parentesco remoto, (2) identificar nuevos miembros de una familia de proteínas, o (3) descubrir proteínas con secuencias muy divergentes pero con una estructura tridimensional parecida. PSI-BLAST se ejecuta en varias etapas: La primera etapa consiste en una búsqueda BLASTP normal utilizando una matriz de sustitución como BLOSUM62 En la segunda etapa, se seleccionan las secuencias con un valor E menor que cierto umbral (por defecto E = 0,005, pero se puede cambiar) y se hace un alineamiento múltiple (AMS) con el que se construye un perfil, también denominado una matriz de puntuación específica de la posición (PSSM, position-specific scoring matrix). Esta PSSM asigna una puntuación distinta a cada posición del AMS: a los residuos conservados en una determinada posición se les asigna una puntuación muy alta, mientras que, en esa misma posición, a los demás residuos se les asigna una puntuación muy negativa. En las regiones no conservadas se asigna una puntuación cercana a cero a todos los residuos. En la tercera etapa se lleva a cabo una nueva búsqueda en la BD utilizando la PSSM en lugar de la matriz de sustitución. Las nuevas secuencias con el valor E apropiado se añaden a las seleccionadas en la segunda etapa para hacer una nueva PSSM con la que iniciar una nueva búsqueda. Esta etapa es iterativa, es decir, se puede repetir las veces que uno quiera o hasta llegar a la convergencia, que es el momento en el que la búsqueda no consigue encontrar nuevas secuencias. El resultado de la búsqueda depende en gran medida de las secuencias seleccionadas en la segunda etapa para construir la PSSM. El usuario puede modificar la selección de secuencias utilizadas para construir la PSSM, eliminando algunas que el programa haya incluido de forma automática o introduciendo otras descartadas por el programa.
BÚSQUEDA DE SECUENCIAS PARECIDAS (Similarity search)
 BÚSQUEDA DE SECUENCIAS PARECIDAS (Similarity search) Cuando los proyectos de secuenciación genómica descubren una nueva secuencia biológica es muy poco lo que se sabe de ella. Por ese motivo, una de las
BÚSQUEDA DE SECUENCIAS PARECIDAS (Similarity search) Cuando los proyectos de secuenciación genómica descubren una nueva secuencia biológica es muy poco lo que se sabe de ella. Por ese motivo, una de las
Investigación en evolución
 Investigación en evolución Análisis de secuencias de: nucleótidos en DNA (genómica) aminoácidos en Proteínas (proteómica) Comparación de secuencias de: nucleótidos aminoácidos Bioinformática Análisis de
Investigación en evolución Análisis de secuencias de: nucleótidos en DNA (genómica) aminoácidos en Proteínas (proteómica) Comparación de secuencias de: nucleótidos aminoácidos Bioinformática Análisis de
Andrés M. Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia
 Alineamiento: Análisis computacional de secuencias Andrés M. Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Por qué y para qué... Tengo una secuencia de DNA/Proteína......
Alineamiento: Análisis computacional de secuencias Andrés M. Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Por qué y para qué... Tengo una secuencia de DNA/Proteína......
Dra. Ing. Agr. Sabrina Costa Tártara Departamento de Tecnología
 Que podemos encontrar en los ácidos nucleicos de los organismos? Megabases de datos inexploradas que nos pueden dar información sobre la diversidad de los organismos. Dra. Ing. Agr. Sabrina Costa Tártara
Que podemos encontrar en los ácidos nucleicos de los organismos? Megabases de datos inexploradas que nos pueden dar información sobre la diversidad de los organismos. Dra. Ing. Agr. Sabrina Costa Tártara
Enfermedad Mendeliana. Búsqueda en bases de datos con proteínas homólogas. Clonaje in silico del gen. Gen candidato
 Enfermedad Mendeliana Defecto determinado por métodos bioquímicos Sin pistas bioquímicas Identificación proteína Clonaje funcional del gen Búsqueda en bases de datos con proteínas homólogas Clonaje in
Enfermedad Mendeliana Defecto determinado por métodos bioquímicos Sin pistas bioquímicas Identificación proteína Clonaje funcional del gen Búsqueda en bases de datos con proteínas homólogas Clonaje in
Existen dos tipos de genes en el genoma humano: codificantes de proteínas y codificantes de ARN.
 EL GEN Es una porción delimitida de ADN con capacidad de codificar una molécula de ARN denominada transcripto primario mediante un proceso llamado transcripción. El ADN contenido en los cromosomas de los
EL GEN Es una porción delimitida de ADN con capacidad de codificar una molécula de ARN denominada transcripto primario mediante un proceso llamado transcripción. El ADN contenido en los cromosomas de los
Análisis y anotación de una secuencia mediante las herramientas y bases de datos de UCSC Genome Bioinformatics & Galaxy
 Análisis y anotación de una secuencia mediante las herramientas y bases de datos de UCSC Genome Bioinformatics & Galaxy Master de Genética y Evolución 2011/2012 Analisis de Secuencias Michael Hackenberg
Análisis y anotación de una secuencia mediante las herramientas y bases de datos de UCSC Genome Bioinformatics & Galaxy Master de Genética y Evolución 2011/2012 Analisis de Secuencias Michael Hackenberg
PRACTICA VI: BUSQUEDA DE SIMILITUDES EN BASES DE DATOS
 Objetivo: PRACTICA VI: BUSQUEDA DE SIMILITUDES EN BASES DE DATOS Ø Conocer el fundamento de los programas BLAST y FASTA y utilizarlos eficientemente para la búsqueda de similitudes de secuencias de DNA
Objetivo: PRACTICA VI: BUSQUEDA DE SIMILITUDES EN BASES DE DATOS Ø Conocer el fundamento de los programas BLAST y FASTA y utilizarlos eficientemente para la búsqueda de similitudes de secuencias de DNA
Perfiles y modelos ocultos de Markov
 Perfiles y modelos ocultos de Markov Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 13 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Perfiles y modelos ocultos de Markov 13 de junio del 2013
Perfiles y modelos ocultos de Markov Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 13 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Perfiles y modelos ocultos de Markov 13 de junio del 2013
Introducción a la Secuenciación Masiva y a la Bioinformática
 Introducción a la Secuenciación Masiva y a la Bioinformática Dietmar Fernández Orth, PhD 24 de Abril de 2014 1 El DNA (Ácido desoxirribonucleico) contiene la información genética usada en el desarrollo
Introducción a la Secuenciación Masiva y a la Bioinformática Dietmar Fernández Orth, PhD 24 de Abril de 2014 1 El DNA (Ácido desoxirribonucleico) contiene la información genética usada en el desarrollo
Predicción computacional de genes (Gene finding)
 Predicción computacional de genes (Gene finding) Predicción de genes (Gene finding) El genoma humano tiene 3.2 GB de nucleótidos, y poco más de 20.000 genes ccgtacgtacgtagagtgctagtctagtcgtagcgccgtagtcgatcgtgtgggt
Predicción computacional de genes (Gene finding) Predicción de genes (Gene finding) El genoma humano tiene 3.2 GB de nucleótidos, y poco más de 20.000 genes ccgtacgtacgtagagtgctagtctagtcgtagcgccgtagtcgatcgtgtgggt
BLAST. Rodrigo Santamaría
 BLAST Rodrigo Santamaría BLAST Introducción Definición Familia BLAST Algoritmo Salida Estrategias Otros programas Introducción BLAST: Basic Local Alignment Search Tool Altschul et al. 1990 (PMID 2231712)
BLAST Rodrigo Santamaría BLAST Introducción Definición Familia BLAST Algoritmo Salida Estrategias Otros programas Introducción BLAST: Basic Local Alignment Search Tool Altschul et al. 1990 (PMID 2231712)
Comparación de secuencias
 Comparación de secuencias Por qué nos interesa comparar secuencias de ADN o proteínas de distintos orígenes? Cómo se pueden alinear secuencias? Alineamiento de secuencias Dos tipos de alineamientos Alineamiento
Comparación de secuencias Por qué nos interesa comparar secuencias de ADN o proteínas de distintos orígenes? Cómo se pueden alinear secuencias? Alineamiento de secuencias Dos tipos de alineamientos Alineamiento
2 Congreso Colombiano de Bioinformática y biología computacional.
 2 Congreso Colombiano de Bioinformática y biología computacional. Presentación y evaluación de ABMS (Automatic Blast for Massive Annotation) Nelson Perez nelsonp@correo.udistrital.edu.co Cristian Rojas
2 Congreso Colombiano de Bioinformática y biología computacional. Presentación y evaluación de ABMS (Automatic Blast for Massive Annotation) Nelson Perez nelsonp@correo.udistrital.edu.co Cristian Rojas
Biotecnología y bioinformática
 IMAGEN: http://4.bp.blogspot.com Biotecnología y bioinformática Opción B 5ª Parte: Bioinformática Tema 9 de Biología NS Diploma BI Curso 2014-2016 Idea Fundamental: La bioinformática consiste en el uso
IMAGEN: http://4.bp.blogspot.com Biotecnología y bioinformática Opción B 5ª Parte: Bioinformática Tema 9 de Biología NS Diploma BI Curso 2014-2016 Idea Fundamental: La bioinformática consiste en el uso
ORGANIZACIÓN, FUNCIÓN Y VARIABILIDAD DEL GENOMA 2010 PRÁCTICO: ANOTACIÓN GENÓMICA UTILIZANDO ARTEMIS
 1 ORGANIZACIÓN, FUNCIÓN Y VARIABILIDAD DEL GENOMA 2010 Objetivos PRÁCTICO: ANOTACIÓN GENÓMICA UTILIZANDO ARTEMIS Profundizar en el análisis de secuencias nucleotídicas/aa utilizando la base de datos del
1 ORGANIZACIÓN, FUNCIÓN Y VARIABILIDAD DEL GENOMA 2010 Objetivos PRÁCTICO: ANOTACIÓN GENÓMICA UTILIZANDO ARTEMIS Profundizar en el análisis de secuencias nucleotídicas/aa utilizando la base de datos del
Predicción de la estructura terciara de las proteínas
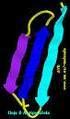 Predicción de la estructura terciara de las proteínas Cuando la estructura terciaria de una proteína no se ha determinado experimentalmente, se puede intentar construir un modelo tridimensional a partir
Predicción de la estructura terciara de las proteínas Cuando la estructura terciaria de una proteína no se ha determinado experimentalmente, se puede intentar construir un modelo tridimensional a partir
Estructura y partes de un gen.
 UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Estructura y partes de un gen. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira 2. Eduardo Vargas 2. 1. Responsable
UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Estructura y partes de un gen. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira 2. Eduardo Vargas 2. 1. Responsable
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA. Alineamiento múltiple de secuencias
 TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento múltiple de secuencias Mario de J. Pérez Jiménez Luis Valencia Cabrera Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento múltiple de secuencias Mario de J. Pérez Jiménez Luis Valencia Cabrera Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación
GRADO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR. ASIGNATURA: BIOINFORMÁTICA (6 Créditos)
 GRADO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR ASIGNATURA: BIOINFORMÁTICA (6 Créditos) OBJETIVOS 1.- Familiarizar al alumno con los recursos disponibles en los principales portales bioinformáticos disponibles
GRADO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR ASIGNATURA: BIOINFORMÁTICA (6 Créditos) OBJETIVOS 1.- Familiarizar al alumno con los recursos disponibles en los principales portales bioinformáticos disponibles
GRADO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR. ASIGNATURA: BIOINFORMÁTICA (6 Créditos)
 GRADO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR ASIGNATURA: BIOINFORMÁTICA (6 Créditos) OBJETIVOS 1.- Familiarizar al alumno con los recursos disponibles en los principales portales bioinformáticos disponibles
GRADO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR ASIGNATURA: BIOINFORMÁTICA (6 Créditos) OBJETIVOS 1.- Familiarizar al alumno con los recursos disponibles en los principales portales bioinformáticos disponibles
ÍNDICE GENERAL ÍNDICE DE FIGURAS ÍNDICE DE TABLAS INDICE DE GRÁFICOS RESUMEN SUMMARY I. INTRODUCCIÓN 1
 ÍNDICE GENERAL Página ÍNDICE GENERAL ÍNDICE DE FIGURAS ÍNDICE DE TABLAS INDICE DE GRÁFICOS RESUMEN SUMMARY i iv vii viii ix x I. INTRODUCCIÓN 1 I.1 Consideraciones Generales 1 I.1.1 Flavonoides 1 I.2 Biosíntesis
ÍNDICE GENERAL Página ÍNDICE GENERAL ÍNDICE DE FIGURAS ÍNDICE DE TABLAS INDICE DE GRÁFICOS RESUMEN SUMMARY i iv vii viii ix x I. INTRODUCCIÓN 1 I.1 Consideraciones Generales 1 I.1.1 Flavonoides 1 I.2 Biosíntesis
Búsqueda de secuencias en Bases de Datos.
 Búsqueda de secuencias en Bases de Datos. Existe una amplia red de bases de datos en diferentes servidores científicos que permiten acceder a una gran cantidad de información científica. Y entre ella,
Búsqueda de secuencias en Bases de Datos. Existe una amplia red de bases de datos en diferentes servidores científicos que permiten acceder a una gran cantidad de información científica. Y entre ella,
Aplicaciones guiadas: Blast. Genome Browsers.
 Curso de Formación UEB Herramientas Bioinformáticas para la Investigación Biomédica 1 r bloque (20/06/2012) Introducción a la Bioinformática y a las Bases de Datos 3 a sesión Aplicaciones guiadas: Blast.
Curso de Formación UEB Herramientas Bioinformáticas para la Investigación Biomédica 1 r bloque (20/06/2012) Introducción a la Bioinformática y a las Bases de Datos 3 a sesión Aplicaciones guiadas: Blast.
BLAST EJERCICIOS PNLHGLFGRKTG
 BLAST EJERCICIOS Ejercicio 1 Haz una búsqueda blastp en el NCBI usando la siguiente secuencia de búsqueda: PNLHGLFGRKTG Por defecto, los parámetros se van a reajustar para búsquedas de secuencias cortas.
BLAST EJERCICIOS Ejercicio 1 Haz una búsqueda blastp en el NCBI usando la siguiente secuencia de búsqueda: PNLHGLFGRKTG Por defecto, los parámetros se van a reajustar para búsquedas de secuencias cortas.
BASES DE DATOS DE INTERÉS EN BIOQUÍMICA
 BASES DE DATOS DE INTERÉS EN BIOQUÍMICA Las técnicas de alto rendimiento desarrolladas en las últimas décadas han permitido la adquisición masiva de información de biología molecular que se depositan en
BASES DE DATOS DE INTERÉS EN BIOQUÍMICA Las técnicas de alto rendimiento desarrolladas en las últimas décadas han permitido la adquisición masiva de información de biología molecular que se depositan en
La síntesis de proteínas
 La síntesis de proteínas La Transcripción La información para fabricar todas las proteínas está almacenada en las moléculas de ADN de los cromosomas. La sucesión de bases en las moléculas de ADN es un
La síntesis de proteínas La Transcripción La información para fabricar todas las proteínas está almacenada en las moléculas de ADN de los cromosomas. La sucesión de bases en las moléculas de ADN es un
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA. Alineamiento de secuencias de genes/proteínas
 TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento de secuencias de genes/proteínas Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Alineamiento de secuencias de genes/proteínas Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia
Elvira Mayordomo y Jorge Álvarez. Marzo - Abril de 2016
 TRABAJO DE PRÁCTICAS Elvira Mayordomo y Jorge Álvarez Marzo - Abril de 2016 1 Introducción El trabajo de prácticas de la asignatura consistirá en que cada alumno realice por separado el trabajo que se
TRABAJO DE PRÁCTICAS Elvira Mayordomo y Jorge Álvarez Marzo - Abril de 2016 1 Introducción El trabajo de prácticas de la asignatura consistirá en que cada alumno realice por separado el trabajo que se
Alineamiento múltiple de secuencias
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 11 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del 2013 1 / 39 1 Alineamiento múltiple de
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 11 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento múltiple de secuencias 11 de junio del 2013 1 / 39 1 Alineamiento múltiple de
Guía de actividades. Trabajo colaborativo 2
 1. Objetivo Guía de actividades Trabajo colaborativo 2 Familiarizar al estudiante con las herramientas bioinformáticas a través del desarrollo de ejercicios prácticos. 2. Actividades Investigadores que
1. Objetivo Guía de actividades Trabajo colaborativo 2 Familiarizar al estudiante con las herramientas bioinformáticas a través del desarrollo de ejercicios prácticos. 2. Actividades Investigadores que
BASES DE DATOS DE INTERÉS EN BIOQUÍMICA
 BASES DE DATOS DE INTERÉS EN BIOQUÍMICA Las técnicas de alto rendimiento desarrolladas en las últimas décadas han permitido la adquisición masiva de información de biología molecular que se depositan en
BASES DE DATOS DE INTERÉS EN BIOQUÍMICA Las técnicas de alto rendimiento desarrolladas en las últimas décadas han permitido la adquisición masiva de información de biología molecular que se depositan en
INGENIERÍA GENÉTICA. Sinónimos: Manipulación genética Clonaje génico Tecnología del DNA recombinante
 INGENIERÍA GENÉTICA Es una revolución metodológica que ha puesto a nuestra disposición gran número de técnicas procedentes de diversos campos de la ciencia con un objetivo común muy importante: el acceso
INGENIERÍA GENÉTICA Es una revolución metodológica que ha puesto a nuestra disposición gran número de técnicas procedentes de diversos campos de la ciencia con un objetivo común muy importante: el acceso
+ Expresión de Genes
 + Expresión de Genes Si todas las células de un organismo contienen el mismo genoma, cómo y por qué las células de la piel son diferentes de las células del cerebro o de las células del hígado? + Expresión
+ Expresión de Genes Si todas las células de un organismo contienen el mismo genoma, cómo y por qué las células de la piel son diferentes de las células del cerebro o de las células del hígado? + Expresión
CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO
 Alianza para el Aprendizaje de Ciencias y Matemáticas CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO GUÍA DEL MAESTRO ESTÁNDARES ATENDIDOS: 1. LA NATURALEZA
Alianza para el Aprendizaje de Ciencias y Matemáticas CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO GUÍA DEL MAESTRO ESTÁNDARES ATENDIDOS: 1. LA NATURALEZA
Métodos de alineamiento (2) Bioinformática, Elvira Mayordomo
 Métodos de alineamiento (2) Bioinformática, 26-2-16 Elvira Mayordomo Hemos visto 1. Bioinformática y Biología Molecular 2. Algoritmos para strings A. Clásicos B. Árboles de sufijos C. Vectores de sufijos
Métodos de alineamiento (2) Bioinformática, 26-2-16 Elvira Mayordomo Hemos visto 1. Bioinformática y Biología Molecular 2. Algoritmos para strings A. Clásicos B. Árboles de sufijos C. Vectores de sufijos
UPR RECINTO DE RÍO PIEDRAS FACULTAD DE CIENCIAS NATURALES DEPARTAMENTO DE BIOLOGÍA LABORATORIO DE BIOTECNOLOGÍA (BIOL. 3365)
 UPR RECINTO DE RÍO PIEDRAS FACULTAD DE CIENCIAS NATURALES DEPARTAMENTO DE BIOLOGÍA LABORATORIO DE BIOTECNOLOGÍA (BIOL. 3365) Análisis del gen AmyE mediante el uso de herramientas de bioinformática A. Objetivos
UPR RECINTO DE RÍO PIEDRAS FACULTAD DE CIENCIAS NATURALES DEPARTAMENTO DE BIOLOGÍA LABORATORIO DE BIOTECNOLOGÍA (BIOL. 3365) Análisis del gen AmyE mediante el uso de herramientas de bioinformática A. Objetivos
Introducción a la Bioinformática
 Introducción a la Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 16 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Introducción a la 16 de mayo del 2013 1 / 37 1 Introducción Qué es bioinformática?
Introducción a la Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 16 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Introducción a la 16 de mayo del 2013 1 / 37 1 Introducción Qué es bioinformática?
DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS
 DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS COMPARACION DE DOS ATPASAS DE PECES El DOT PLOT permite una visualización rápida de la similitud entre dos secuencias Inconvenientes: No identifica
DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS COMPARACION DE DOS ATPASAS DE PECES El DOT PLOT permite una visualización rápida de la similitud entre dos secuencias Inconvenientes: No identifica
ACIDOS NUCLEICOS. Índice. RNA Diferencias entre DNA Y RNA Tipos de RNA Traspaso del material genético: RNA polimerasa Transcripción Traducción Resumen
 Índice ACIDOS NUCLEICOS RNA Diferencias entre DNA Y RNA Tipos de RNA Traspaso del material genético: RNA polimerasa Transcripción Traducción Resumen ARN: Ácido ribunucleico Polímero lineal de nucleótidos
Índice ACIDOS NUCLEICOS RNA Diferencias entre DNA Y RNA Tipos de RNA Traspaso del material genético: RNA polimerasa Transcripción Traducción Resumen ARN: Ácido ribunucleico Polímero lineal de nucleótidos
GENOMICA Y PROTEOMICA
 GENOMICA Y PROTEOMICA Dr. José A Gutiérrez Bustamante Area de Genética y Biología Molecular y Celular Depto. de Morfología Humana Fac. de Medicina - UNT Célula ADN gen A gen B gen C ARN Tecnología Bioinformática
GENOMICA Y PROTEOMICA Dr. José A Gutiérrez Bustamante Area de Genética y Biología Molecular y Celular Depto. de Morfología Humana Fac. de Medicina - UNT Célula ADN gen A gen B gen C ARN Tecnología Bioinformática
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
Tema 8. Mapas físicos Genómica estructural
 Tema 8 Mapas físicos Genómica estructural Genoma Genómica Genómica estructural Genómica funcional Mapas genéticos Mapas físicos EXPRESIÓN Transcriptoma Proteoma Interactoma FUNCIÓN Genética inversa Secuenciacion
Tema 8 Mapas físicos Genómica estructural Genoma Genómica Genómica estructural Genómica funcional Mapas genéticos Mapas físicos EXPRESIÓN Transcriptoma Proteoma Interactoma FUNCIÓN Genética inversa Secuenciacion
Bioinformática Clásica
 Bioinformática Clásica Dr. Oswaldo Trelles Universidad de Málaga Esta presentación contiene información sobre la organización del curso de Bioinformática Clásica. En ella se describe para cada tema su
Bioinformática Clásica Dr. Oswaldo Trelles Universidad de Málaga Esta presentación contiene información sobre la organización del curso de Bioinformática Clásica. En ella se describe para cada tema su
comprende todos aquellos del gen a nivel de traducción o transcripción, regulando los productos funcionales de un gen. Los niveles de regulación de la
 La regulación genética comprende todos aquellos procesos que afectan la acción del gen a nivel de traducción o transcripción, regulando los productos funcionales de un gen. Los niveles de regulación de
La regulación genética comprende todos aquellos procesos que afectan la acción del gen a nivel de traducción o transcripción, regulando los productos funcionales de un gen. Los niveles de regulación de
Mapeo, Ensamble y Comparación del Genoma
 José Antonio Molinet Berenguer CINVESTAV 30 de julio del 2013 José A. MOLINET B. (CINVESTAV) Mapeo, Ensamble y Comparación Genómica 30 de julio del 2013 1 / 45 1 Mapeo, Ensamble y Comparación del Genoma
José Antonio Molinet Berenguer CINVESTAV 30 de julio del 2013 José A. MOLINET B. (CINVESTAV) Mapeo, Ensamble y Comparación Genómica 30 de julio del 2013 1 / 45 1 Mapeo, Ensamble y Comparación del Genoma
Alineamiento de pares de secuencias
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 30 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento de pares de secuencias 30 de mayo del 2013 1 / 61 1 Alineamiento de pares de secuencias
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 30 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Alineamiento de pares de secuencias 30 de mayo del 2013 1 / 61 1 Alineamiento de pares de secuencias
Bases moleculares de la herencia. Código genético y Traducción.
 UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Código genético y Traducción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira
UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Código genético y Traducción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira
BIOINFORMÁTICA. Vicente Arnau Llombart. Técnicas Avanzadas de Inteligencia Artificial.
 BIOINFORMÁTICA Vicente Arnau Llombart Técnicas Avanzadas de Inteligencia Artificial. http://www.uv.es/~varnau/taia_2011-12.htm E-mail: Vicente.Arnau@uv.es BIOINFORMÁTICA Clase 2ª: Análisis de secuencias
BIOINFORMÁTICA Vicente Arnau Llombart Técnicas Avanzadas de Inteligencia Artificial. http://www.uv.es/~varnau/taia_2011-12.htm E-mail: Vicente.Arnau@uv.es BIOINFORMÁTICA Clase 2ª: Análisis de secuencias
Introducción a la Biología Molecular
 Introducción a la Biología Molecular Gonzalo Gómez ggomez@cnio.es Unidad de Bioinformática (CNIO) http://bioinfo.cnio.es/ Facultad de Informática, Ourense Mayo 2009 1 Niveles de organización DNA El ácido
Introducción a la Biología Molecular Gonzalo Gómez ggomez@cnio.es Unidad de Bioinformática (CNIO) http://bioinfo.cnio.es/ Facultad de Informática, Ourense Mayo 2009 1 Niveles de organización DNA El ácido
Alineamiento local: búsqueda de homologías
 Alineamiento local: búsqueda de homologías Supongamos que el material de partida para realizar una búsqueda de homologías no es un gen o una proteína completos y bien caracterizados de los que podamos
Alineamiento local: búsqueda de homologías Supongamos que el material de partida para realizar una búsqueda de homologías no es un gen o una proteína completos y bien caracterizados de los que podamos
Definición:Bioinformática
 Bioinformática Definición:Bioinformática La bioinformática es el estudio del contenido y flujo de la información en sistemas y procesos biológicos. Requieren el uso o el desarrollo de diferentes técnicas
Bioinformática Definición:Bioinformática La bioinformática es el estudio del contenido y flujo de la información en sistemas y procesos biológicos. Requieren el uso o el desarrollo de diferentes técnicas
Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA
 Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA Martin A. Lozano, Jaime Velasco-Medina Grupo de Bio-nanoelectrónica EIEE, Universidad del Valle, A.A. 25360, Cali, Colombia E-mail:
Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA Martin A. Lozano, Jaime Velasco-Medina Grupo de Bio-nanoelectrónica EIEE, Universidad del Valle, A.A. 25360, Cali, Colombia E-mail:
Alineamientos de Secuencias. CeCalCULA - C.P.T.M. Mérida. Venezuela.
 Alineamientos de Secuencias Análisis comparativo El alineamiento de secuencias es similar a otros tipos de análisis comparativo. En ambos es necesario cuantificar las similitudes y diferencias (scoring)
Alineamientos de Secuencias Análisis comparativo El alineamiento de secuencias es similar a otros tipos de análisis comparativo. En ambos es necesario cuantificar las similitudes y diferencias (scoring)
2. La figura representa un segmento de la fibra básica de cromatina.
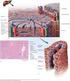 Bloque IV: Base molecular de la herencia 1. a. El esquema representa un cromosoma eucariótico en metafase mitótica. Qué denominación reciben los elementos indicados por los números 1 a 4? b. Dibuje un
Bloque IV: Base molecular de la herencia 1. a. El esquema representa un cromosoma eucariótico en metafase mitótica. Qué denominación reciben los elementos indicados por los números 1 a 4? b. Dibuje un
Introducción a la Bioinformática
 Introducción a la Bioinformática Genómica y bioinformática: Nuevas áreas de biotecnología Genómica-Ciencia que se encarga de las estrategias de clonación, secuenciación y análisis de genes. Cómo los científicos
Introducción a la Bioinformática Genómica y bioinformática: Nuevas áreas de biotecnología Genómica-Ciencia que se encarga de las estrategias de clonación, secuenciación y análisis de genes. Cómo los científicos
Tutorial Uso y manejo básico de EupathDB. TritrypDB como modelo.
 Tutorial Uso y manejo básico de EupathDB. TritrypDB como modelo. Dr. Luis Miguel de Pablos Torró FEBS associated Postdoctoral Researcher. University of Cambridge. EuPathDB (hfp://eupathdb.org) es una base
Tutorial Uso y manejo básico de EupathDB. TritrypDB como modelo. Dr. Luis Miguel de Pablos Torró FEBS associated Postdoctoral Researcher. University of Cambridge. EuPathDB (hfp://eupathdb.org) es una base
TRANSCRIPCION Y TRADUCCION
 TRANSCRIPCION Y TRADUCCION BibliograJa: CurKs. 7ma edición. Capítulo 10. (Código fotocopiadora 2160) htp://www.curksbiologia.com/indice_figuras_animadas Asignatura: Biología para IRNR Profesora Responsable:
TRANSCRIPCION Y TRADUCCION BibliograJa: CurKs. 7ma edición. Capítulo 10. (Código fotocopiadora 2160) htp://www.curksbiologia.com/indice_figuras_animadas Asignatura: Biología para IRNR Profesora Responsable:
Análisis de proteínas
 Análisis de proteínas Qué determina su estructura? Composición de las proteínas Las proteínas son polímeros de aminoácidos que se unen mediante una unión peptídica Todos los aminoácidos tienen un grupo
Análisis de proteínas Qué determina su estructura? Composición de las proteínas Las proteínas son polímeros de aminoácidos que se unen mediante una unión peptídica Todos los aminoácidos tienen un grupo
Estas prácticas tienen por objeto aprender a manejar la información contenida en el NCBI de una forma más o menos sencilla o elemental.
 NCBI. Bases de Datos: Pubmed, Nucleotide, Protein, Structure A lo largo de los últimos 15 o 20 años, se ha ido acumulando una gran cantidad de información de naturaleza molecular (secuencias de genes,
NCBI. Bases de Datos: Pubmed, Nucleotide, Protein, Structure A lo largo de los últimos 15 o 20 años, se ha ido acumulando una gran cantidad de información de naturaleza molecular (secuencias de genes,
Temas actuales: Next Generation Sequencing (NGS) Bioinformática Elvira Mayordomo
 Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Secuenciación del ARN
 Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
REPLICACIÓN DEL ADN. Procesos de replicación, transcripción y traducción.
 REPLICACIÓN DEL ADN Procesos de replicación, transcripción y traducción. La transmisión de información implica que el ADN es capaz de duplicarse de manera de obtener dos moléculas iguales a partir de la
REPLICACIÓN DEL ADN Procesos de replicación, transcripción y traducción. La transmisión de información implica que el ADN es capaz de duplicarse de manera de obtener dos moléculas iguales a partir de la
tuberosus (Fairmaire & Germain)
 Caracterización y análisis de variabilidad genética en poblaciones de Calvertius tuberosus (Fairmaire & Germain) (Coleoptera: Curculionidae) Ramón Rebolledo, Luis Huala,, Rubén Carrillo, Alejandro Espinoza,
Caracterización y análisis de variabilidad genética en poblaciones de Calvertius tuberosus (Fairmaire & Germain) (Coleoptera: Curculionidae) Ramón Rebolledo, Luis Huala,, Rubén Carrillo, Alejandro Espinoza,
Índice de contenidos INTRODUCCIÓN... 1 OBJETIVOS... 5 HIPÓTESIS... 5 MARCO TEÓRICO... 6. Genes superpuestos... 6
 Índice de contenidos INTRODUCCIÓN... 1 OBJETIVOS... 5 HIPÓTESIS... 5 MARCO TEÓRICO... 6 Genes superpuestos... 6 Marcos de lectura abiertos (Open Reading Frame, ORFs) alternativos... 7 MATERIALES Y MÉTODOS...
Índice de contenidos INTRODUCCIÓN... 1 OBJETIVOS... 5 HIPÓTESIS... 5 MARCO TEÓRICO... 6 Genes superpuestos... 6 Marcos de lectura abiertos (Open Reading Frame, ORFs) alternativos... 7 MATERIALES Y MÉTODOS...
Estructura del genoma humano
 Estructura del genoma humano Definiremos genes y genomas. Analizaremos los tipos de secuencias presentes en el genoma humano. Analizaremos su distribución y función. Describiremos algunas aplicaciones
Estructura del genoma humano Definiremos genes y genomas. Analizaremos los tipos de secuencias presentes en el genoma humano. Analizaremos su distribución y función. Describiremos algunas aplicaciones
Tema 18. Traducción I. Código genético y RNA de transferencia
 Tema 18 Traducción I. Código genético y RNA de transferencia Código genético Cómo se traduce la información contenida en el ARNm a proteínas? 4 nucleótidos 20 aminoácidos Código Código genético * Es el
Tema 18 Traducción I. Código genético y RNA de transferencia Código genético Cómo se traduce la información contenida en el ARNm a proteínas? 4 nucleótidos 20 aminoácidos Código Código genético * Es el
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS. Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida
 SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida La secuencia de aminoácidos de un polipéptido está programada en una unidad heredable denominada gen.
SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida La secuencia de aminoácidos de un polipéptido está programada en una unidad heredable denominada gen.
ALGORITMOS PARA PREDICCIÓN DE LA FUNCIÓN DE PROTEÍNAS
 ALGORITMOS PARA PREDICCIÓN DE LA FUNCIÓN DE PROTEÍNAS Estefanía Prior Cano Ing. Telecomunicación 100055190@alumnos.uc3m.es Sergio Tejeda Pastor Ing. Telecomunicación 100061107@alumnos.uc3m.es RESÚMEN Este
ALGORITMOS PARA PREDICCIÓN DE LA FUNCIÓN DE PROTEÍNAS Estefanía Prior Cano Ing. Telecomunicación 100055190@alumnos.uc3m.es Sergio Tejeda Pastor Ing. Telecomunicación 100061107@alumnos.uc3m.es RESÚMEN Este
A qué da lugar el mensaje del ADN?
 A qué da lugar el mensaje del ADN? Cómo se lee el mensaje? FLUJO DE INFORMACIÓN DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR (enunciado por F. Crick) DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR ACTUALIZADO TRANSCRIPCIÓN
A qué da lugar el mensaje del ADN? Cómo se lee el mensaje? FLUJO DE INFORMACIÓN DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR (enunciado por F. Crick) DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR ACTUALIZADO TRANSCRIPCIÓN
1. Escribir la secuencia de bases de las cadenas complementarias de las siguientes: 5' AGCCTAGCAA 3' 5 GATCAATCGA 3 5 TACCATACGC 3
 DEPARTAMENTO DE BIOLOGIA MOLECULAR FACULTAD DE MEDICINA Isabel Andrés PROBLEMAS TEMA 10. INFORMACION GENETICA. 1. Escribir la secuencia de bases de las cadenas complementarias de las siguientes: 5' AGCCTAGCAA
DEPARTAMENTO DE BIOLOGIA MOLECULAR FACULTAD DE MEDICINA Isabel Andrés PROBLEMAS TEMA 10. INFORMACION GENETICA. 1. Escribir la secuencia de bases de las cadenas complementarias de las siguientes: 5' AGCCTAGCAA
Yolanda Loarce Tejada. Universidad de Alcalá Patrimonio de la Humanidad
 Yolanda Loarce Tejada GENOMA: conjunto de genes que llevan los organismos (1920) GENÓMICA: el estudio de los genomas Estructural Funcional Comparativa El significado de una PALABRA depende de la secuencia
Yolanda Loarce Tejada GENOMA: conjunto de genes que llevan los organismos (1920) GENÓMICA: el estudio de los genomas Estructural Funcional Comparativa El significado de una PALABRA depende de la secuencia
PRACTICA V: ALINEAMIENTO POR METODOS HEURISTICOS Y ANÁLISIS ESTADÍSTICO DE LAS PUNTACIONES DE LOS ALINEAMIENTOS.
 PRACTICA V: ALINEAMIENTO POR METODOS HEURISTICOS Y ANÁLISIS ESTADÍSTICO DE LAS PUNTACIONES DE LOS ALINEAMIENTOS. Objetivo general: Ø Conocer y aplicar las técnicas de alineamiento heurístico así como las
PRACTICA V: ALINEAMIENTO POR METODOS HEURISTICOS Y ANÁLISIS ESTADÍSTICO DE LAS PUNTACIONES DE LOS ALINEAMIENTOS. Objetivo general: Ø Conocer y aplicar las técnicas de alineamiento heurístico así como las
Genómica comparada. Brown 2002, págs
 Genómica comparada 1. Teoría neutralista de la evolución molecular 2. Sustituciones nucleotídicas sinónimas y no-sinónimas. Interpretación de la razón Ka/Ks. 3. Duplicaciones y el destino de los genes
Genómica comparada 1. Teoría neutralista de la evolución molecular 2. Sustituciones nucleotídicas sinónimas y no-sinónimas. Interpretación de la razón Ka/Ks. 3. Duplicaciones y el destino de los genes
Señales en el ADN. Bioinformática, Kevin Yip-CSE-CUHK (Universidad china de Hong-Kong)
 Señales en el ADN Bioinformática, 5-4-17 Kevin Yip-CSE-CUHK (Universidad china de Hong-Kong) Señales en el ADN Hemos visto cómo determinar y comparar secuencias de ADN Cómo podemos averiguar el significado
Señales en el ADN Bioinformática, 5-4-17 Kevin Yip-CSE-CUHK (Universidad china de Hong-Kong) Señales en el ADN Hemos visto cómo determinar y comparar secuencias de ADN Cómo podemos averiguar el significado
Distintos tipos de ARN en el citoplasma. El dogma central. Nomenclatura transcripcional. Que es un gen? Una hebra de ADN sirve de molde (hebra molde)
 El dogma central Distintos tipos de ARN en el citoplasma La principal función de los genes es la manufactura de proteinas. La hipótesis de la secuencia : El orden de las bases en una porcion del ADN representa
El dogma central Distintos tipos de ARN en el citoplasma La principal función de los genes es la manufactura de proteinas. La hipótesis de la secuencia : El orden de las bases en una porcion del ADN representa
RNA-Seq: Introducción al ensamblado de novo de transcriptomas
 RNA-Seq: Introducción al ensamblado de novo de transcriptomas Diego Nicolás de Panis Instituto de Ecología Genética y Evolución de Buenos Aires (IEGEBA, CONICET-UBA) RNAseq y Anotación Funcional FCEN UBA
RNA-Seq: Introducción al ensamblado de novo de transcriptomas Diego Nicolás de Panis Instituto de Ecología Genética y Evolución de Buenos Aires (IEGEBA, CONICET-UBA) RNAseq y Anotación Funcional FCEN UBA
Código de barras del ADN. Dra. Analía A. Lanteri División Entomología- Museo de La Plata
 Código de barras del ADN Dra. Analía A. Lanteri División Entomología- Museo de La Plata CÓDIGO DE BARRAS DEL ADN Los genes están formados por EXONES (traducen a proteínas) y los INTRONES (no codificantes)
Código de barras del ADN Dra. Analía A. Lanteri División Entomología- Museo de La Plata CÓDIGO DE BARRAS DEL ADN Los genes están formados por EXONES (traducen a proteínas) y los INTRONES (no codificantes)
EJERCICIOS DE BIOINFORMÁTICA
 EJERCICIOS DE BIOINFORMÁTICA Estos ejercicios pueden ser muy complicados. Sin embargo, debe seguir cada uno de los pasos, tal y como se detalla. Si en algún momento, tiene dudas o no puede realizar los
EJERCICIOS DE BIOINFORMÁTICA Estos ejercicios pueden ser muy complicados. Sin embargo, debe seguir cada uno de los pasos, tal y como se detalla. Si en algún momento, tiene dudas o no puede realizar los
CURSO DE BIOLOGÍA MOLECULAR y GENÓMICA PARA PROFESORES DE ENSEÑANZA MEDIA Y FORMACIÓN EN EDUCACIÓN. Maldonado, Uruguay 16 al 20 de diciembre, 2013
 CURSO DE BIOLOGÍA MOLECULAR y GENÓMICA PARA PROFESORES DE ENSEÑANZA MEDIA Y FORMACIÓN EN EDUCACIÓN Maldonado, Uruguay 16 al 20 de diciembre, 2013 ÍNDICE PARTE I Introducción. 3 Objetivos del Curso. 3 Programa.
CURSO DE BIOLOGÍA MOLECULAR y GENÓMICA PARA PROFESORES DE ENSEÑANZA MEDIA Y FORMACIÓN EN EDUCACIÓN Maldonado, Uruguay 16 al 20 de diciembre, 2013 ÍNDICE PARTE I Introducción. 3 Objetivos del Curso. 3 Programa.
Transcripción. - Generalidades - DNA RNA. - Mecanismo. - Modelos Protein. - Moléculas. - Métodos de detección de RNA. Chromosome RNA DNA
 TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
TEMA 1. ADN, GENES Y GENOMAS. 2.- Naturaleza química y estructura del DNA 3.- Tipos de DNA eucariótico. 5.- Genomas: Tamaño y número de genes
 TEMA 1. ADN, GENES Y GENOMAS 1.- Definición i ió y conceptos 2.- Naturaleza química y estructura del DNA 3.- Tipos de DNA eucariótico 4.- Genes: Intrones y exones 5.- Genomas: Tamaño y número de genes
TEMA 1. ADN, GENES Y GENOMAS 1.- Definición i ió y conceptos 2.- Naturaleza química y estructura del DNA 3.- Tipos de DNA eucariótico 4.- Genes: Intrones y exones 5.- Genomas: Tamaño y número de genes
Práctico prueba de ensamblaje con Galaxy
 Práctico prueba de ensamblaje con Galaxy taller de Herramientas para el Análisis de Secuenias y proteínas 26 de octubre de 2016 1 Introducción La tuberculosis bovina es causada por la bacteria Mycobacterium
Práctico prueba de ensamblaje con Galaxy taller de Herramientas para el Análisis de Secuenias y proteínas 26 de octubre de 2016 1 Introducción La tuberculosis bovina es causada por la bacteria Mycobacterium
b) Quién es el ancestro común más cercano de la especie H y las del género F?
 1. La gráfica muestra el árbol filogenético de las especies de un reino a partir de un antepasado común, en las que las letras indican representantes conocidos, actuales o fósiles, de las correspondientes
1. La gráfica muestra el árbol filogenético de las especies de un reino a partir de un antepasado común, en las que las letras indican representantes conocidos, actuales o fósiles, de las correspondientes
Predicción de Promotores y Elementos Reguladores
 Pedro Eduardo TORRES JIMÉNEZ CINVESTAV-Zacatenco 09 de julio del 2013 Pedro Eduardo Torres Jiménez (CINVESTAV) Predicción de Promotores y Elementos Reguladores 09 de julio del 2013 1 / 60 1 Predicción
Pedro Eduardo TORRES JIMÉNEZ CINVESTAV-Zacatenco 09 de julio del 2013 Pedro Eduardo Torres Jiménez (CINVESTAV) Predicción de Promotores y Elementos Reguladores 09 de julio del 2013 1 / 60 1 Predicción
TRANSCRIPCIÓN CONCEPTO DE OPERÓN Y PROMOTOR. González Pérez Ana Karen Robledo Sarmiento Danely
 TRANSCRIPCIÓN CONCEPTO DE OPERÓN Y PROMOTOR González Pérez Ana Karen Robledo Sarmiento Danely Transcripción Es la copia de un gen en un ARNm Ribosa Uracilo Cadena sencilla Operón Se define como una unidad
TRANSCRIPCIÓN CONCEPTO DE OPERÓN Y PROMOTOR González Pérez Ana Karen Robledo Sarmiento Danely Transcripción Es la copia de un gen en un ARNm Ribosa Uracilo Cadena sencilla Operón Se define como una unidad
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ
 REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
El Dogma Central de la Biología Molecular v.1. Manuel J. Gómez Laboratorio de Bioinformática Centro de Astrobiología INTA- CSIC
 El Dogma Central de la Biología Molecular v.1 Manuel J. Gómez Laboratorio de Bioinformática Centro de Astrobiología INTA- CSIC Flujo de información Material genético Proteínas Replicación Transcripción
El Dogma Central de la Biología Molecular v.1 Manuel J. Gómez Laboratorio de Bioinformática Centro de Astrobiología INTA- CSIC Flujo de información Material genético Proteínas Replicación Transcripción
Alineamiento de pares de secuencias. Rodrigo Santamaría
 Alineamiento de pares de secuencias Rodrigo Santamaría Alineamiento de pares de secuencias Introducción Definiciones Ejemplo Algoritmos Matrices de puntuación 2 Objetivo Determinar si una secuencia de
Alineamiento de pares de secuencias Rodrigo Santamaría Alineamiento de pares de secuencias Introducción Definiciones Ejemplo Algoritmos Matrices de puntuación 2 Objetivo Determinar si una secuencia de
Guía de Actividad de clases biología IV medio Mutaciones
 Guía de Actividad de clases biología IV medio Mutaciones Nombre: Curso: Fecha: Instrucciones.- Desarrolla la siguiente guía siguiendo la instrucción que se da en cada actividad. En aquellas donde se indique
Guía de Actividad de clases biología IV medio Mutaciones Nombre: Curso: Fecha: Instrucciones.- Desarrolla la siguiente guía siguiendo la instrucción que se da en cada actividad. En aquellas donde se indique
MicroRNAs. Michael Hackenberg
 MicroRNAs Michael Hackenberg (hackenberg@ugr.es) Introducción Un microrna es un RNA corto de entre 19 y 25 nt de longitud. Están implicados en la regulación génica post-transcripcional y probablemente
MicroRNAs Michael Hackenberg (hackenberg@ugr.es) Introducción Un microrna es un RNA corto de entre 19 y 25 nt de longitud. Están implicados en la regulación génica post-transcripcional y probablemente
Regulación de la Expresión genética. Curso: Biología Celular y Molecular Oscar Nolasco Cárdenas MSc. Noviembre
 Regulación de la Expresión genética Curso: Biología Celular y Molecular Oscar Nolasco Cárdenas MSc. oscarnol@hotmail.com Noviembre - 2013 Una célula puede cambiar la expresión de sus genes en respuesta
Regulación de la Expresión genética Curso: Biología Celular y Molecular Oscar Nolasco Cárdenas MSc. oscarnol@hotmail.com Noviembre - 2013 Una célula puede cambiar la expresión de sus genes en respuesta
Repaso de PCR y qpcr
 Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
TRANSCRIPCIÓN. Mathews C.K.; Van Holde K.E.; Ahern, K. G (2002). Bioquímica, 3ª ed. Addison Wesley
 TRANSCRIPCIÓN Nelson DL y Cox MM (2000) Lehninger Principios de Bioquímica 3ª ed Ed Omega Nelson DL y Cox MM. (2000). Lehninger Principios de Bioquímica, 3ª ed. Ed. Omega. Mathews C.K.; Van Holde K.E.;
TRANSCRIPCIÓN Nelson DL y Cox MM (2000) Lehninger Principios de Bioquímica 3ª ed Ed Omega Nelson DL y Cox MM. (2000). Lehninger Principios de Bioquímica, 3ª ed. Ed. Omega. Mathews C.K.; Van Holde K.E.;
ADN. Transcripción. ARN transcrito primario afuncional. Modificación Post-transcripcional. ARNs maduros funcionales
 Introducción Modificación del ARN transcrito para ser un ARNm del que se puedan obtener proteínas. ARN transcrito primario sin función, a no ser que madure y se transforme en un ARNm Nunca un ARNr transcrito
Introducción Modificación del ARN transcrito para ser un ARNm del que se puedan obtener proteínas. ARN transcrito primario sin función, a no ser que madure y se transforme en un ARNm Nunca un ARNr transcrito
CAPÍTULO 2 FLUJO DE LA INFORMACIÓN BIOLÓGICA FACULTAD DE AGRONOMÍA CURSO DE BIOQUÍMICA
 CAPÍTULO 2 FLUJO DE LA INFORMACIÓN BIOLÓGICA FACULTAD DE AGRONOMÍA CURSO DE BIOQUÍMICA Contenido 2.1 Información biológica e interacciones no covalentes. 2.2 Almacenamiento de la información biológica
CAPÍTULO 2 FLUJO DE LA INFORMACIÓN BIOLÓGICA FACULTAD DE AGRONOMÍA CURSO DE BIOQUÍMICA Contenido 2.1 Información biológica e interacciones no covalentes. 2.2 Almacenamiento de la información biológica
1.2. MOVERSE DE UN DOCUMENTO A OTRO BUSCAR TEXTO.
 9 1.2. MOVERSE DE UN DOCUMENTO A OTRO BUSCAR TEXTO. Word nos permite buscar un texto de varias formas: a través del panel Navegación o con la página Buscar del cuadro de diálogo Buscar y reemplazar. La
9 1.2. MOVERSE DE UN DOCUMENTO A OTRO BUSCAR TEXTO. Word nos permite buscar un texto de varias formas: a través del panel Navegación o con la página Buscar del cuadro de diálogo Buscar y reemplazar. La
