UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN
|
|
|
- Francisca Escobar Núñez
- hace 6 años
- Vistas:
Transcripción
1 COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN OBJETIVO: el presente trabajo práctico tiene como objetivo principal que el alumno adquiera destreza en el análisis de electroferogramas generados por equipos automatizados de secuenciación. I. Introducción Secuenciación se define como el método que permite determinar el orden de los nucleótidos (G, A, T/U, C) en un fragmento de ADN o ARN. El término también hace referencia al orden de aminoácidos de la proteína. El fragmento a secuenciar puede ser pequeño (~500pb) como también puede ser el genoma entero de un organismo. El orden de A, C, G y T dentro de la cadena de ADN determina los codones START y STOP, cuando el gen es expresado y, lo mas importante, provee la información que especifica la secuencia de aminoácidos de la proteína, cuando el gen es codificante. El proceso detallado de secuenciación se realiza en el laboratorio y permite detectar pequeños cambios dentro del gen, los cuales pueden determinar un polimorfismo poblacional, una patología clínica, una nueva especie, etc. Uno de los métodos de secuenciación mas utilizado es el conocido como secuenciación de terminación de cadena (chain termination sequencing) o secuenciación enzimática o simplemente secuenciación de Sanger, en honor a su inventor el bioquímico Frederick Sanger (quien ganó su segundo premio Nobel por la técnica). El método de Sanger se basa en el principio de que las cadenas de ADN normalmente están formadas a partir de nucleótidos deoxi (dntps), aunque emplea también dideoxinucleótidos (didntps o simplemente ddntps). La diferencia entre los dos tipos de moléculas (Figura 1) se encuentra en la posición 3', lugar donde normalmente se establece la unión entre dos nucleótidos consecutivos para formar la cadena de ADN. Si no existe un grupo OH en esa posición, el enlace que une el nucleótido entrante no puede realizarse, interrumpiendo así la elongación de la cadena. Figura 1: Representación esquemática de la diferencia entre un dideoxinucleótido (llamado también stopnucleotide) y un deoxinucleótido (nucleótido normal).
2 La mezcla de reacción de la PCR de secuenciación contiene una miscelánea de nucleótidos deoxy (dntp s normales) juntamente con una pequeña cantidad de nucleótidos dideoxy (o terminadores). Durante este proceso de amplificación cada nucleótido incorporado es complementario al nucleótido existente (C si hay G o A si hay T, y viceversa) pero al azar (por ej. datp o didatp) en el extremo terminal 3' de la nueva cadena en crecimiento. Si la base incorporada es un dideoxinucleótido (por ej. didatp), la polimerasa no puede continuar elongando la cadena en crecimiento porque el extremo 3' posee un H. La nueva cadena deja de ser elongada en ese punto. Por el contrario, si la base incorporada es un deoxinucleótido (por ej. datp) la cadena continuará creciendo hasta que otro dideoxinucleótido se incorpore. Debido a que los dideoxy se encuentran en menor proporción, cada vez que al azar uno de ellos es incorporado, se aborta la síntesis de esa cadena. Esto genera una mezcla de cadenas de diferente longitud entre las varias cadenas que se sintetizan simultáneamente durante el proceso de amplificación de la PCR. Inicialmente, se usaba PAGE para ordenar las cadenas sintetizadas desde la mayor (parte superior del gel) hasta la de menor tamaño (parte inferior del gel) y posteriormente, mediante detección por rayos-x se determinaba el codon marcado radiactivamente en cada cadena. (Figura 2). Desde 1986, el proceso de lectura de la secuencia de las bases C, T, G y A de una molécula de ADN puede ser realizado en un secuenciador automático de fluorescencia (no radiactivo). Este aparato utiliza los mismos principios que el método de Sanger, o sea los nucleótidos terminadores, pero con la diferencia de que un láser escanea continuamente la región final del gel, detectando las bandas que migran. Mientras que el método manual usaba marcado radiactivo, los secuenciadores automáticos usan marcado fluorescente en los didntps. Esto hace posible que los 4 nucleótidos (dgtp, datp, dctp, y dttp) corran al mismo tiempo en el gel (y no en 4 reacciones separadas como en el método Sanger original) aumentando así en 4 veces la velocidad de la secuenciación. Posteriormente, aparecieron secuenciadores automáticos donde los geles fueron reemplazados por capilares, haciendo el método extremadamente eficiente y aplicable a cualquier tipo de fragmento de ADN. La resolución es tan alta que un simple nucleótido de más es suficiente para separar esa cadena de la próxima más larga y de la anterior mas corta. Al ser iluminados por un láser, cada uno de los 4 dideoxinucleótidos fluoresce con un color diferente que es detectado por un scanner automático el cual realiza la lectura de esa intensidad de luz, que es interpretada por un software que construye la secuencia impresa (Figura 3).
3 Figura 2: Ejemplificación gráfica de cómo se realizaba la secuenciación por el método Sanger original. Este método separa las moléculas por su tamaño en PAGE y posteriormente identificando los didntps radioactivos mediante rayos-x.
4 Figura 3: Ejemplo de cuatro corridas de secuenciación automática. En A y B se muestra en la parte superior el electrofenograma de la secuencia alterada (paciente afectado) y en la parte inferior se muestra la secuencia patrón (paciente normal). Animaciones complementarias en:
5 Protocolo para secuenciación automática Paso 1: Primera PCR Realizar una reacción de PCR estándar del fragmento que se desea secuenciar (ver prácticos anteriores) Paso 2: Purificar la muestra de ADN amplificado Este paso es extremadamente importante ya que permite eliminar todos los componentes que sobran de la 1º PCR (restos de primers, dntp, enzima Taq, etc.) los cuales podrían inhibir o contaminar los pasos siguientes. Los métodos de purificación son varios y dependen del tipo de secuenciador que se disponga (Megabase, ABI, ROCHE etc.) una de las opciones utiliza sucesivos pasos de centrifugación con kits de columnas descartables que tienen la capacidad de retener el ADN amplificado pero dejando pasar las impurezas. Después de realizar el proceso de purificación, independientemente de cual sea, se procede a constatar la concentración de la muestra del ADN. Se toman 2 µl de la muestra (con 1 µl de buffer de carga) y se aplica juntamente con un patrón de intensidad de bandas, en un gel de chequeo de agarosa al 2%. La comparación de la intensidad de la banda de nuestra muestra con el patrón (Figura 4) nos permite determinar visualmente la concentración (en ng) del ADN. Con la concentración establecida podemos ahora determinar cuántos µl de ADN usaremos para realizar la segunda PCR. Paso 3: Reacción de secuenciación Para secuenciar un fragmento de cadena simple de DNA, su cadena complementaria es sintetizada usando una segunda reacción de PCR especial cuyo mix contiene: Una mezcla de los 4 nucleótidos normales (deoxi) en grandes cantidades o datp + dgtp + dctp + dttp una mezcla de los 4 dideoxinucleótidos, cada uno presente en cantidades limitadas y marcado con un "tag" fluorescente de diferente color o didatp + didgtp + didctp + didttp ADN polimerasa el fragmento de ADN a secuenciar (previamente amplificado) primers # agua ultra pura (o Milli-Q) autoclavada
6 Todos los componentes se colocan en un tubo que va a la máquina de PCR (ver practico anterior), debidamente identificadas com nombre o número e indicando el tipo de primer. 100ng 60ng 40ng 20ng 10ng 5ng Figura 4: En la imagen se muestra el patrón Low DNA mass ladder (Invitrogen) usado para cuantificar la concentración del ADN en agarosa al 2%. Los nanogramos (ng) de cada banda corresponden a 2µl de del marcador sembrado. # ATENCION: el secuenciador no puede leer ambas cadenas (molde y complementaria) simultáneamente. Por eso cada cadena se secuencia por separado. Para la 2º PCR un tubo contiene el cóctel de PCR y solo un primer (e.g. únicamente forward) y en otro tubo se coloca el mismo mix de PCR pero con el primer restante (e.g. únicamente reverse). Paso 4: Precipitación con etanol Algunos secuenciadores reciben la muestra a secuenciar en seco y solamente en los minutos previos a colocar la muestras para la lectura, resuspenden el material en formamida (ABI) o en otros loadings especificos (Megabase). Por este motivo explicaremos la precipitación con etanol. Al tubo que contiene la muestra se le agrega 28µl de etanol 100% helado y se centrifuga el tubo a 15000g por 15 min.
7 Posteriomente, se aspira y descarta con la pipeta todo el sobrenadante con cuidado de no tocar las paredes del tubo donde el ADN se encuentra adherido. Por segunda vez se coloca etanol 75%, pero un volumen mayor (100µl). La mezcla se resuspende y centrifuga igual que en el paso anterior. Finalmente, se aspira y descarta todo el sobrenadante (sin tocar las paredes o el fondo del tubo) y se deja secar el tubo por 24hs a temperatura ambiente y protegido de la luz (porque los didntps son fluorescentes y por tanto fotosensibles). Las muestras son sometidas al secuenciador, juntamente con una ficha identificando cada tubo. La identificación no sólo se refiere al origen de la muestra (e.g. nombre o número de la muestra), sino que también información referida al primer que se encuentra en cada tubo (forward o reverse); esto significa que serán entregados dos tubos por cada paciente/muestra.
Caracteristicas del ADN. Se rompen con las siguientes condiciones -Temperaturas >90 - ph >10.5
 PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
Protocolos de secuenciación de ADN
 1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
PCR Múltiplex. Listeria monocytogenes
 PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
3. COMPONENTES. Tampón de electroforesis concentrado 2 x 50 ml 10 X (2 envases 500ml)
 PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
3/22/2010. Iván Ferrer Rodríguez, PhD Catedrático. En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR.
 Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática.
 Departamento de Bioquímica Médica y Biología Molecular Práctica 3 Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática. Curso 1 Medicina (Abril) 2012 BIOQUÍMICA
Departamento de Bioquímica Médica y Biología Molecular Práctica 3 Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática. Curso 1 Medicina (Abril) 2012 BIOQUÍMICA
Purinas (R); Pirimidinas (Y)
 Replicación del ADN La doble hélice Purinas (R); Pirimidinas (Y) Tautómeros Puentes de H normales a) Una vez que la replicación comienza, todo el ADN es replicado. b) Como regla general, cuando la replicación
Replicación del ADN La doble hélice Purinas (R); Pirimidinas (Y) Tautómeros Puentes de H normales a) Una vez que la replicación comienza, todo el ADN es replicado. b) Como regla general, cuando la replicación
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO
 SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA"
 P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
La primera pregunta: semiconservativa o conservativa? Experimento de Meselson y Stahl
 REPLICACIÓN DE ADN La primera pregunta: semiconservativa o conservativa? Experimento de Meselson y Stahl Replicación semiconservativa La copia de DNA original fue marcada con N15 (isótopo pesado). Este
REPLICACIÓN DE ADN La primera pregunta: semiconservativa o conservativa? Experimento de Meselson y Stahl Replicación semiconservativa La copia de DNA original fue marcada con N15 (isótopo pesado). Este
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
Técnicas moleculares.
 Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006
 IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
Usos de Herramientas Moleculares en Agricultura: Marcadores Moleculares. Técnicas Moleculares. Técnicas Moleculares 8/13/13
 8/13/13 Usos de Herramientas Moleculares en Agricultura: Marcadores Moleculares CAF516 O Recursos Genéticos y Biotecnología 14 Agosto 2013 Técnicas Moleculares Aplicaciones en muchas disciplinas Generan
8/13/13 Usos de Herramientas Moleculares en Agricultura: Marcadores Moleculares CAF516 O Recursos Genéticos y Biotecnología 14 Agosto 2013 Técnicas Moleculares Aplicaciones en muchas disciplinas Generan
Bases moleculares de la herencia. Transcripción.
 UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Transcripción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira 2. Eduardo
UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Transcripción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira 2. Eduardo
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez
 Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez 1 Objetivos Entender la técnica: Reacción de Polimerasa en Cadena (PCR por sus siglas en inglés)
Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez 1 Objetivos Entender la técnica: Reacción de Polimerasa en Cadena (PCR por sus siglas en inglés)
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Definición de PCR Es la amplificación enzimática de un
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Definición de PCR Es la amplificación enzimática de un
CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA. Grado en Medicina
 DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015
 CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR)
 METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
Bases Moleculares. Efrén Santos
 Bases Moleculares Efrén Santos ADN, ARN, proteínas ADN, contiene la información genética ARN, muy similar al ADN. (1) Copia temporal del ADN (2) Parte funcional y estructural del aparato traductor Proteinas,
Bases Moleculares Efrén Santos ADN, ARN, proteínas ADN, contiene la información genética ARN, muy similar al ADN. (1) Copia temporal del ADN (2) Parte funcional y estructural del aparato traductor Proteinas,
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA
 UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
DOGMA CENTRAL DE LA BIOLOGIA
 La única diferencia entre las moléculas de ADN de distintos individuos es el orden en el que se disponen sus nucleótidos; lo que se denomina secuencia. Los nucleótidos se ordenan a modo de palabras que
La única diferencia entre las moléculas de ADN de distintos individuos es el orden en el que se disponen sus nucleótidos; lo que se denomina secuencia. Los nucleótidos se ordenan a modo de palabras que
 Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Bases moleculares de la herencia. Código genético y Traducción.
 UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Código genético y Traducción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira
UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Código genético y Traducción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira
Técnica de PCR. Beatriz Bellosillo. Servei de Patologia, Hospital del Mar, Barcelona, Spain
 Técnica de PCR Beatriz Bellosillo Servei de Patologia, Hospital del Mar, Barcelona, Spain Biología Molecular Estudio de los procesos que se desarrollan en los seres vivos desde un punto de vista molecular
Técnica de PCR Beatriz Bellosillo Servei de Patologia, Hospital del Mar, Barcelona, Spain Biología Molecular Estudio de los procesos que se desarrollan en los seres vivos desde un punto de vista molecular
INFORMACIÓN AL USUARIO
 INFORMACIÓN AL USUARIO 1. SERVICIOS QUE SE OFRECEN 2. CONDICIONES QUE DEBEN CUMPLIR LAS MUESTRAS 3. INFORMACIÓN ADICIONAL 4. CÓMO HACER USO DEL SERVICIO 5. RESULTADOS 1.- SERVICIOS QUE SE OFRECEN Nuestro
INFORMACIÓN AL USUARIO 1. SERVICIOS QUE SE OFRECEN 2. CONDICIONES QUE DEBEN CUMPLIR LAS MUESTRAS 3. INFORMACIÓN ADICIONAL 4. CÓMO HACER USO DEL SERVICIO 5. RESULTADOS 1.- SERVICIOS QUE SE OFRECEN Nuestro
PCR gen 18S ARNr humano
 PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
PRÁCTICAS DE MICROBIOLOGIA CLINICA
 PRÁCTICAS DE MICROBIOLOGIA CLINICA Juan Carlos Rodríguez Hospital General Universitario de Alicante E-mail: rodriguez_juadia@gva.es http://microbiologia-alicante.umh.es CASO CLINICO: Rubéola Evolución
PRÁCTICAS DE MICROBIOLOGIA CLINICA Juan Carlos Rodríguez Hospital General Universitario de Alicante E-mail: rodriguez_juadia@gva.es http://microbiologia-alicante.umh.es CASO CLINICO: Rubéola Evolución
Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz
 IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
La síntesis de proteínas
 La síntesis de proteínas La Transcripción La información para fabricar todas las proteínas está almacenada en las moléculas de ADN de los cromosomas. La sucesión de bases en las moléculas de ADN es un
La síntesis de proteínas La Transcripción La información para fabricar todas las proteínas está almacenada en las moléculas de ADN de los cromosomas. La sucesión de bases en las moléculas de ADN es un
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Índice. 4. Protocolo de envío de muestras 9 5. Problemas más frecuentes durante la secuenciación 10 6. Algunos datos y formulas de utilidad 17
 Índice Introducción 1 1. Modalidades de secuenciación 2 a. Secuenciación de una sola cadena 2 b. Secuenciación analítica 3 c. Secuenciación diagnóstica 3 2. Oligonucleótidos disponibles 5 3. Protocolo
Índice Introducción 1 1. Modalidades de secuenciación 2 a. Secuenciación de una sola cadena 2 b. Secuenciación analítica 3 c. Secuenciación diagnóstica 3 2. Oligonucleótidos disponibles 5 3. Protocolo
Proyecto de Fin de Cursos
 Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS
 Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Organización del genoma eucariotico. 1. Complejidad del genoma eucariota
 Organización del genoma eucariotico Procariotas: Prácticamente todo el ADN existe en forma de copia única y codifica productos génicos (proteínas y ARNs) Eucariotas: La mayor parte del ADN es NO codificante.
Organización del genoma eucariotico Procariotas: Prácticamente todo el ADN existe en forma de copia única y codifica productos génicos (proteínas y ARNs) Eucariotas: La mayor parte del ADN es NO codificante.
DETERMINACIÓN DEL FACTOR Rh por PCR
 Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL EXAMEN DHE MOLECULARES EN EL EXAMEN DHE
 X Curso de Formación sobre la Protección de las Obtenciones Vegetales para Países Iberoamericanos Montevideo (URUGUAY) 12 al 16 de Diciembre de 2011 EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL
X Curso de Formación sobre la Protección de las Obtenciones Vegetales para Países Iberoamericanos Montevideo (URUGUAY) 12 al 16 de Diciembre de 2011 EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL
CUESTIONES. 1. Por qué la PCR convencional no es cuantitativa?
 2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
12.359,04 IMPORTE MAXIMO LOTE 1: ,92 IMPORTE MAXIMO LOTE 2: IMPORTE MAXIMO LOTE 3: ,54 REACTIVOS PARA AISLAMIENTO DE ACIDOS NUCLEICOS
 HOSPITAL RAMON Y CAJAL RAZON SOCIAL: SUMINISTROS - PETICION DE OFERTAS C.I.F.: SERVICIO DE GENETICA MEDICA DOMICILIO: LOCALIDAD: TRAMITA: HOSPITAL RAMON Y CAJAL TELEFONO/FAX: Nº DE COMPRA: CONCURSO ABIERTO
HOSPITAL RAMON Y CAJAL RAZON SOCIAL: SUMINISTROS - PETICION DE OFERTAS C.I.F.: SERVICIO DE GENETICA MEDICA DOMICILIO: LOCALIDAD: TRAMITA: HOSPITAL RAMON Y CAJAL TELEFONO/FAX: Nº DE COMPRA: CONCURSO ABIERTO
Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS
 Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS DICIEMBRE 2006 Tratamiento de Imágenes por Computadora Proyecto
Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS DICIEMBRE 2006 Tratamiento de Imágenes por Computadora Proyecto
DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas
 DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas ELEMENTOS BÁSICOS DE LA INVESTIGACIÓN EN GENES Análisis
DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas ELEMENTOS BÁSICOS DE LA INVESTIGACIÓN EN GENES Análisis
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR).
 Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
BIOLOGIA CELULAR Y MOLECULAR. LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR)
 BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR) 1. INTRODUCCIÓN La Reacción en Cadena de la Polimerasa (PCR) es una técnica in Vitro que imita la habilidad natural
BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR) 1. INTRODUCCIÓN La Reacción en Cadena de la Polimerasa (PCR) es una técnica in Vitro que imita la habilidad natural
PCR. Técnica inventada por Kary Mullis :1987. Premio Nobel 1993. Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN
 PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
LA NUEVA BIOTECNOLOGÍA
 LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN
 INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
Técnicas moleculares para
 Técnicas moleculares para detectar enfermedades vasculares en cítricos Isidro Humberto Almeyda-León Mario Alberto Rocha-Peña Fermín n Orona-Castro María a Magdalena Iracheta-Cárdenas rdenas Reyna Xochitl
Técnicas moleculares para detectar enfermedades vasculares en cítricos Isidro Humberto Almeyda-León Mario Alberto Rocha-Peña Fermín n Orona-Castro María a Magdalena Iracheta-Cárdenas rdenas Reyna Xochitl
Transcripción. - Generalidades - DNA RNA. - Mecanismo. - Modelos Protein. - Moléculas. - Métodos de detección de RNA. Chromosome RNA DNA
 TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
Dogma Central de la Biología Molecular
 Replicación Dogma Central de la Biología Molecular Ciclo celular Modelo semiconservador Comprobación Polimerización Procariontes (Modelo Theta) Procariontes y Eucariontes Mitocondrias Origen de la replicación
Replicación Dogma Central de la Biología Molecular Ciclo celular Modelo semiconservador Comprobación Polimerización Procariontes (Modelo Theta) Procariontes y Eucariontes Mitocondrias Origen de la replicación
El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN.
 PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
Polymerase Chain Reaction (PCR)
 Polymerase Chain Reaction (PCR) Un poco de historia La técnica de PCR fue inventada por Kary B. Mullis en 1983. La primer publicación sobre PCR apareció en 1985, aunque el principio básico de replicar
Polymerase Chain Reaction (PCR) Un poco de historia La técnica de PCR fue inventada por Kary B. Mullis en 1983. La primer publicación sobre PCR apareció en 1985, aunque el principio básico de replicar
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Secuenciación del ARN
 Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
Tecnologías basadas en la PCR
 Tecnologías de marcadores moleculares para estudios de diversidad genética de plantas: Módulo de aprendizaje Tecnologías basadas en el ADN Tecnologías basadas en la PCR Fundamentos de la PCR Derechos de
Tecnologías de marcadores moleculares para estudios de diversidad genética de plantas: Módulo de aprendizaje Tecnologías basadas en el ADN Tecnologías basadas en la PCR Fundamentos de la PCR Derechos de
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
3. MATERIALES Y MÉTODOS
 3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
 EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri
 Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri OBJETIVO Desarrollar un banco de datos a partir de plantas de origen indudable y del uso
Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri OBJETIVO Desarrollar un banco de datos a partir de plantas de origen indudable y del uso
1. Contaminación de agua y alimentos por microorganismos patógenos 1
 INTRODUCCIÓN GENERAL 1. Contaminación de agua y alimentos por microorganismos patógenos 1 2. Papel del agua en la transmisión de enfermedades infecciosas 2 2.1. Las aguas residuales 2 2.2. El agua potable
INTRODUCCIÓN GENERAL 1. Contaminación de agua y alimentos por microorganismos patógenos 1 2. Papel del agua en la transmisión de enfermedades infecciosas 2 2.1. Las aguas residuales 2 2.2. El agua potable
Replicación del ADN - Replicación de la doble hélice: Biosíntesis de ADN en células procariotas y
 Replicación del ADN - Replicación de la doble hélice: Biosíntesis de ADN en células procariotas y eucariotas. Corrección de errores. (poner especial atención, es muy importante entenderla, pues en este
Replicación del ADN - Replicación de la doble hélice: Biosíntesis de ADN en células procariotas y eucariotas. Corrección de errores. (poner especial atención, es muy importante entenderla, pues en este
SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS. Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida
 SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida La secuencia de aminoácidos de un polipéptido está programada en una unidad heredable denominada gen.
SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida La secuencia de aminoácidos de un polipéptido está programada en una unidad heredable denominada gen.
GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA
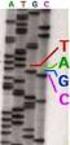 Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
Código: IDK-010 Ver: 1. Proteína G C825T. Sistema para la detección de la mutación C825T en el gene de la proteína G.
 C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
Informe técnico del proyecto. de diagnóstico genético de pacientes con. síndrome de Dravet y espectros asociados
 Informe técnico del proyecto de diagnóstico genético de pacientes con síndrome de Dravet y espectros asociados Introducción El objetivo principal de este proyecto es realizar un diagnóstico genético a
Informe técnico del proyecto de diagnóstico genético de pacientes con síndrome de Dravet y espectros asociados Introducción El objetivo principal de este proyecto es realizar un diagnóstico genético a
La Biotecnología como Tema de Integración Curricular y su Relevancia en el Desarrollo de las Destrezas del Pensamiento
 La Biotecnología como Tema de Integración Curricular y su Relevancia en el Desarrollo de las Destrezas del Pensamiento Gerardo Arroyo Cruzado, Ph.D. Departamento de Ciencias Biológicas, Facultad de Ciencias
La Biotecnología como Tema de Integración Curricular y su Relevancia en el Desarrollo de las Destrezas del Pensamiento Gerardo Arroyo Cruzado, Ph.D. Departamento de Ciencias Biológicas, Facultad de Ciencias
DETECCIÓN DE C. jejuni EN HAMBURGUESA DE POLLO POR PCR TRADICIONAL PRT-712.04-082
 Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Reacción en cadena de la polimerasa (PCR)
 Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
PLANIFICACIÓN DE ASIGNATURA
 PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
La Patología a Molecular y el Control de Calidad
 UNIDAD DIDÁCTICA 7: LOS PROCESOS EN ANATOMÍA PATOLÓGICA La Patología a Molecular y el Control de Calidad Asunción n Olmo Sevilla Master Diagnóstica S. L. Patología Molecular: Patología del futuro X Personal:
UNIDAD DIDÁCTICA 7: LOS PROCESOS EN ANATOMÍA PATOLÓGICA La Patología a Molecular y el Control de Calidad Asunción n Olmo Sevilla Master Diagnóstica S. L. Patología Molecular: Patología del futuro X Personal:
ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS
 ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS Luis Sáenz Mateos R4 Análisis Clínicos- Hospital General Universitario de Ciudad Real Rotación Externa Hospital Morales Meseguer-Hospital
ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS Luis Sáenz Mateos R4 Análisis Clínicos- Hospital General Universitario de Ciudad Real Rotación Externa Hospital Morales Meseguer-Hospital
CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL
 LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
FRAGMENTO OLIGO 5-3 Hebra SECUENCIA 5' - - > 3' TAMAÑO (pb) F1. hmtl 569 L AACCAAACCCCAAAGACACC hmth2982 H CTGATCCAACATCGAGGTCG
 ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
Técnicas de Biología Molecular. Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Extracción de ácidos nucleicos o o DNA RNA Tipos de Muestras Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Extracción de ácidos nucleicos o o DNA RNA Tipos de Muestras Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
ADN: USOS FORENSES. Juan Antiñolo Borrego 11ºC Nº3. Ensayo de C.C.M.C
 ADN: USOS FORENSES Juan Antiñolo Borrego 11ºC Nº3 Ensayo de C.C.M.C Dra. Rosario García Repetto, INTCF, Departamento Sevilla: 23/04/2013 1 ÍNDICE Qué es el INTCF? Pág. 3 Bases Genéticas para la identificación
ADN: USOS FORENSES Juan Antiñolo Borrego 11ºC Nº3 Ensayo de C.C.M.C Dra. Rosario García Repetto, INTCF, Departamento Sevilla: 23/04/2013 1 ÍNDICE Qué es el INTCF? Pág. 3 Bases Genéticas para la identificación
Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato
 Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
FACULTAD REGIONAL ROSARIO
 FACULTAD REGIONAL ROSARIO CATEDRA BIOTECNOLOGIA ROFESOR: Ing. Eduardo Santambrosio JTP: Ing. Marta Ortega AUX 1ª : Ing. Pablo Garibaldi PCR (Polymerase chain reaction) Reacción en cadena de la polimerasa
FACULTAD REGIONAL ROSARIO CATEDRA BIOTECNOLOGIA ROFESOR: Ing. Eduardo Santambrosio JTP: Ing. Marta Ortega AUX 1ª : Ing. Pablo Garibaldi PCR (Polymerase chain reaction) Reacción en cadena de la polimerasa
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR
 PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
Diagnóstico Virológico PCR y pruebas rápidas: Gripe A. Carlos Artaza Alvarez MIR 4 Hospital Universitario Fundación Alcorcón
 Diagnóstico Virológico PCR y pruebas rápidas: Gripe A. Carlos Artaza Alvarez MIR 4 Hospital Universitario Fundación Alcorcón Reacción en Cadena de la INTRODUCCION: Polimerasa (RCP) Década de los 70: Grandes
Diagnóstico Virológico PCR y pruebas rápidas: Gripe A. Carlos Artaza Alvarez MIR 4 Hospital Universitario Fundación Alcorcón Reacción en Cadena de la INTRODUCCION: Polimerasa (RCP) Década de los 70: Grandes
5.2. NITRÓGENO Y AZUFRE.
 5.2. NITRÓGENO Y AZUFRE. 5.2.1. PROTEÍNAS. Si hidratos de carbono y lípidos están formados únicamente por átomos de carbono, oxígeno e hidrógeno, las proteínas tienen en su composición, además, nitrógeno
5.2. NITRÓGENO Y AZUFRE. 5.2.1. PROTEÍNAS. Si hidratos de carbono y lípidos están formados únicamente por átomos de carbono, oxígeno e hidrógeno, las proteínas tienen en su composición, además, nitrógeno
La PCR. La Reacción en Cadena de la Polimerasa es la herramienta más utilizada
 La PCR La Reacción en Cadena de la Polimerasa es la herramienta más utilizada PCR- Ventajas y Usos Amplificación ilimitada de secuencias de interés. Rápida, Sencilla y Eficaz. Técnica altamente sensible
La PCR La Reacción en Cadena de la Polimerasa es la herramienta más utilizada PCR- Ventajas y Usos Amplificación ilimitada de secuencias de interés. Rápida, Sencilla y Eficaz. Técnica altamente sensible
Dogma central de la Biología Molecular. Replicación ADN. Transcripción ARN. Traducción. Proteínas
 Dogma central de la Biología Molecular ADN ARN Proteínas Replicación Transcripción Traducción Replicación En la horquilla se encuentra enzimas y proteínas con funciones diferentes Primasa ADN polimerasa
Dogma central de la Biología Molecular ADN ARN Proteínas Replicación Transcripción Traducción Replicación En la horquilla se encuentra enzimas y proteínas con funciones diferentes Primasa ADN polimerasa
Epidemiología molecular Clostridium difficile
 Epidemiología molecular Clostridium difficile Dra Paola Pidal Laboratorio biomédico Nacional y de Referencia Clostridium difficile: Epidemiología molecular Disciplina que resulta de la aplicación e integración
Epidemiología molecular Clostridium difficile Dra Paola Pidal Laboratorio biomédico Nacional y de Referencia Clostridium difficile: Epidemiología molecular Disciplina que resulta de la aplicación e integración
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
41. Comprobación de colonias transformantes mediante PCR y electroforesis
 41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
