Modelos de Markov ocultos Predicción de genes. Alex Sánchez i Mireia Vilardell. Departament d Estadística U.B. 1. Estadística i Bioinformàtica
|
|
|
- María Dolores Reyes Ferreyra
- hace 7 años
- Vistas:
Transcripción
1 Modelos de Markov ocultos Predicción de genes i Mireia Vilardell Departament d Estadística U.B. Estadística i Bioinformàtica Esquema del tema Introducción: Genes y predicción de genes Predicción con modelos tradicionales: Glimmer, geneid Predicción con HMM (1): Conceptos básicos Extensiones del modelo: SemiHMM y Genscan Comparación de programas de predicción Departament d Estadística U.B. 1
2 1. Introducción 1.1. El problema de la identificación de genes El problema de la identificación de los genes se puede describir como el problema de deducir la secuencia de aminoácidos codificada por una determinada región de ADN Es un problema difícil pero muy relevante puesto que... Es necesario para anotar los datos procedentes de los proyectos de secuenciación Ayuda a entender los mecanismos implicados en la codificación decodificación de la información biológica El problema es más simple en organismos inferiores (procariotas) que en los superiores (eucariotas) cuya estructura genómica es más compleja Departament d Estadística U.B. 2 Figura 1: Modelos de transcripción y traslación en procariotas y eucariotas Departament d Estadística U.B. 3
3 1.2. Estructura de los genes en procariotas El genoma de los procariotas ( sin nucleo celular ) suele ser rico en genes: El 80 % 90 % de la secuencia es codificante De forma simplificada un gen procariota es una secuencia de codones que Empieza con un codon de inicio, (ATG), Continua con un número múltiplo de tres de nucleótidos Acaba con un codon de stop (TAA/TAG/TGA) Departament d Estadística U.B. 4 Figura 2: Un gen procariota Departament d Estadística U.B. 5
4 1.3. Estructura de los genes en eucariotas En los organismos superiores los genes no son ni contínuos ni contiguos Los genes suelen estar fragmentados en cierto número de fragmentos codificantes conocidos como exones separados por grandes fragmentos no codificantes conocidos como intrones. Existen una diversidad de señales, algunas más claras que otras, que es preciso localizar e identificar para la predicción de los genes Departament d Estadística U.B. 6 Figura 3: Estructura de un gen eucariota Departament d Estadística U.B. 7
5 1.4. Las señales de especificación de los genes Durante el camino del ADN a la secuencia de aminoácidos los genes son ensamblados por un proceso en tres etapas conocido como splicing Durante este proceso se eliminan los intrones antes de traducir el ADN a proteínas Distintas señales que indican como debe actuar la maquinaria celular que regula el proceso se hallan codificadas en la secuencia original del ADN 1. En la transcripcion intervienen elementos promotores y señales de fin de transcripción 2. En el splicing participan los sitios dadores y aceptores 3. En la traducción intervienen los codones de iniciación ode parada Departament d Estadística U.B. 8 Figura 4: De la secuencia de ADN a la de Aminoácidos Departament d Estadística U.B. 9
6 2. Predicción de genes (1) 2.1. Predicción en procariotas El problema principal suele ser identificar cual de dos o más pautas abiertas de lectura contiene un gen (se supone que sólo una) Una pauta abierta de lectura es una secuencia de codones que empieza con un codon de inicio (ATG) y acaba en un codon de stop (TAA / TAG / TGA) sin que haya ningún otro codon de stop entre ellos Existen señales de inicio y final que es preciso identificar y distinguir del ruido de fondo Departament d Estadística U.B Predicciónconmodelosdemarkov El programa GeneMark, (Borodovsky et al., 1993) utiliza cadenas de Markov de orden 5 para identificar genes microbianos. Esto representa analizar 2 codones cada vez Los genomas bacterianos suelen ser lo bastante largos como para proporcionar buenos estimadores de 4 6 = 4096 probabilidades de transición necesarias Un modelo de orden ocho seria preferible, pero el número de probabilidades a estimar es excesivo Departament d Estadística U.B. 11
7 ALgoritmo simplificado de GenMark De forma simplificada el algoritmo que utiliza GeneMark es el siguiente: 1. Entrenar un modelo de orden 5 con genes conocidos (=pautas de lectura largas, hits en bases de datos) 2. Entrenar un modelo de orden 0 como modelo nulo 3. Puntuar cada pauta abierta de lectura siguiendo las 6 posibles pautas de lectura (3 forward, 3backward) 4. Si la pauta de lectura con mayor puntuación es la pauta abierta, llamésele un gen 5. Si hay pautas abiertas superpuestas puntúese las regiones superpuestas separadamente. Departament d Estadística U.B Predicción en eucariotas (1) Identificacion de genes mediante señales Un método habitual de predicción consiste en: Construir un conjunto de exones potenciales, identificados a traves de señales de inicio/aceptores ydedonores/stop Puntuarlos mediante un modelo estadístico apropiado Ensamblar los genes mediante programación dinámica Se elegirán como candidatos aquellos genes cuya puntuación total sea más elevada Departament d Estadística U.B. 13
8 2.4. Modelos estadísticos de puntuación En análisis de secuencias biológicas son comunes los sistemas de puntuación en donde se compara la puntuación que se asigna a una secuencia bajo un modelo concreto con la que le asigna un modelo nulo o background. Por motivos computacionales dichas puntuaciones suelen expresarse como logaritmos de razones de verosimilitudes ( log-likelihood ratios scores, LLR scores o LODs ) Aparecen sistemas de puntuación basados en LLRs en: Matrices PAM o BLOSUM Identificación de islas CpG Identificación de motivos mediante matrices de pesos posicionales (PWM) Departament d Estadística U.B El sistema de puntuación de geneid El programa de predicción de genes geneid utiliza LLRs en la puntuación de los exones potenciales Un gen, concebido como una sucesión de exones e intrones alternados, puede representarse de forma simplificada como: S = e 1 i 1 e 2 i 2 e 3 i 3 e 4 i 4...e N Sea e i = s i 1s i 2s i 3...s i n i un exon potencial que consta de tres partes diferenciadas: } {{ } e ia :Inicio/Aceptor } {{ } e im :P arte codificante } {{ } e id :Stop/Donor geneid puntua cada parte separadamente utilizando un modelo para los extremos y otro para la parte codificante. Departament d Estadística U.B. 15
9 Modelo de puntuación de un exon Sea e ia el punto de inicio o un acceptor site y e id el punto de stop o un donor site. El exon potencial se puntua: = L E (e i )=L A (e ia )+L D (e id )+L M (e i ) n A n D A s i j j + D s i j j + LI l ( n ) i 5 e i1..,5 + LF l ( ) e ij...j+5, j=1 j=1 j=1 L A (e ia )yl D (e id ) son las puntuaciones de los extremos del exon, que se obtienen mediante LLRs basados en matrices de pesos posicionales para los sitios dadores o aceptores L M (e i ) es el potencial de codificación, que se calcula mediante un modelo de Markov de orden 5 Departament d Estadística U.B Modelo de puntuación (1) Sitios aceptores y donores El calculo L A (e ia )yl D (e id ) esta basado en matrices de pesos posicionales A s i j j,d s i j j son elementos de esta PWM, determinadas a partir de secuencias en las que se conocen las posiciones de los genes (y por tanto de los aceptores, donores, y sitios de start y stop). Se definen como: A ij =log P ij A Q A, (respectivament, D ij,b ij ) ij Departament d Estadística U.B. 17
10 Matrices de pesos posicionales Pij A (respectivamente, P ij D, P ij B ) representan la probabilidad de observar el nucleótido i (i A, C, T, G) en la posición j (j 3, 2,..., 5) en un acceptor site (respectivament, donor o start),yportantoseestimaapartirdelafrecuenciarelativa de nucleótids i que ocupen la posición j en los acceptor sites reales, es decir conocidos (respectivament, donor o start). Q A ij (respectivamente, QD ij, QS ij ) representan la probabilidad de observar el nucleótido i (i A, C, T, G) en la posición j (j 3, 2,..., 5) entorno de cualquier dinucleótido AG (respectivament GT para los donors o AT G para los start codons). Representa pues el modelo nulo, o más exactamente background. Departament d Estadística U.B. 18 Figura 5: Matrices de pesos posicionales Departament d Estadística U.B. 19
11 Modelo de puntuación (2) Potencial de codificación El potencial de codificación consta de dos componentes: F j (h) =F j (s 1 s 2 s 3 s 4 s 5 s 6 ) es la probabilidad (de transición) de observar dentro de un exon el hexámero h = s 1 s 2 s 3 s 4 s 5 s 6 con el nucleótido s 1 en la posición j (j =1, 2, 3 correspondiente a las tres posibles pautas de lectura) suponiendo que s 1 se encuentre en la posición j en el pentámero s 1 s 2 s 3 s 4 s 5. I j (p) es la probabilidad inicial para cada pentámero p en cada posición dentro de los exones para las pautas de lectura 1,2,3. F 0 (h) ii 0 (p) son las probabilidades de transición iniciales correspondientes a los intrones Departament d Estadística U.B. 20 Ensamblado de los genes El modelo anterior permite puntuar cada uno de los posibles exones de un gen Como las señales son muy débiles el número de exones potenciales es muy alto, la mayoria de ellos superpuestos entre si Para escoger un conjunto óptimo que configura un gen se utiliza un algoritmo de programación dinámica que realiza el ensamblado maximizando la suma de las puntuaciones de conjuntos de exones compatibles con un gen (i.e. sin superposición, sin stop codons en medio etc...) Departament d Estadística U.B. 21
12 Departament d Estadística U.B. 22 Figura 6: El número de exones potenciales es muy alto Departament d Estadística U.B. 23
13 3. Predicción de genes con MOM Los MOM resultan especialmente adecuados para la predicción de genes por su capacidad para modelizar estructuras gramaticales, es decir, estructuras en las que aparecen restricciones relativas al tipo de elementos que las constituyen al orden en que aparecen estos elementos Los genes tienen una estructura gramatical sencilla: No se trata tan sólo de conjuntos de caracteres (nucleótidos), palabras (exones /intrones) o frases (genes): Hay una estructura en el sentido que ciertas expresiones no tienen sentido, no son posibles. Por ejemplo, en genes eucariotas 1. Las frases nunca acaban en un intron 2. Un exon nunca sigue a otro exon Departament d Estadística U.B MOM para predicción de genes procariotas Los genes procariotas tienen una gramática particularmente sencilla Codon de inicio Region codificante Codon de parada Un MOM para predecir genes de tal tipo deberá contemplar estados para los tres tipos de regiones Departament d Estadística U.B. 25
14 Figura 7: Un MMO para genes procariotas Departament d Estadística U.B MOM para predicción de genes eucariotas La estructura de los genes eucariotas es compleja. De forma simplificada contempla Codon de inicio Region codificante: Un cierto número de exones ( 1) e intrones ( 0) terminados por un exón Codon de parada Los MOM desarrollados para genes eucariotas suelen constar de varios modelos encadenados, unos para modelizar las señales de inicio o finalización y otros para la región codificante. Departament d Estadística U.B. 27
15 Figura 8: Para los sitios aceptores se construye un MMO sencillo. Excepto en casos raros el intron acaba con un AG, sombreado. El modelo contemplará no tan sólo estos dos nucleótidos con probabilidades de emisión 1, sino 16 bases anteriores y tres bases siguientes. Puesto que no hay huecos el modelo será equivalente a una matriz de pesos. Departament d Estadística U.B. 28 Departament d Estadística U.B. 29
16 Figura 9: Para las regiones codificantes se construye otro MMO. Los estados uno, dos y tres del modelo representan respectivamente el primer, segundo y tercer codon Cualquier región codificante puede ser representada por este modelo porque del estado tres se puede volver al uno En la parte inferior se muestra un modelo sencillo en el que los tres primeros estados coinciden con un codon de inicio, los tres siguientes con el modelo de región codificante de la parte superior y los tres últimos con un codon de parada (solo se muestra uno de los tres posibles estados de parada) Departament d Estadística U.B. 30 Figura 10: Los modelos se encadenan en un modelo general. Una x indica un estado para DNA no codificante y una c un estado para DNA codificante (solo se muestra uno de los tres posibles estados de parada) Departament d Estadística U.B. 31
17 Figura 11: Un modelo combinado que contempla el splicing Departament d Estadística U.B Identificación de genes con MMO Los MMO como los anteriores implican una estructura determinada para el gen Una secuencia que no cumpla las restricciones impuestas recibirá probabilidad cero bajo este modelo Si se desea localizar los genes en un fragmento de genoma Aplicar el algoritmo de Viterbi a la secuencia Identificar como genes aquellas sucesiones de observaciones del camino más probable que cumplan las reglas gramaticales impuestas por el modelo: ATG Ex Int Ex Int... TAA Fin Departament d Estadística U.B. 33
18 Figura 12: Predicción de genes: Dada una secuencia observada la predicción del gen se obtiene aplicaandole el Algoritmo de Viterbi Departament d Estadística U.B. 34 En la practica Los MMO que se utilizan en los programas reales de predicción de genes son mucho más complejos que el ejemplo anterior. 1. VEIL utiliza un modelo simple con muchos estados 2. HMMGene Utiliza CHMM: MMO con clases 3. Genie usa GHMM: MMO generalizados: Los estados del modelo general son, a su vez MMO completos 4. GENSCAN (Burge & Karlin) usa SHMM: MMO con capacidad de incluir la longitud de los exones e intrones... Departament d Estadística U.B. 35
Tema 1: Breve Lección de biología (2)
 Tema 1: Breve Lección de biología (2) Genes y Proteínas Dr. Oswaldo Trelles Universidad de Málaga La función primordial de los genes es proporcionar la información necesaria para la síntesis de proteínas.
Tema 1: Breve Lección de biología (2) Genes y Proteínas Dr. Oswaldo Trelles Universidad de Málaga La función primordial de los genes es proporcionar la información necesaria para la síntesis de proteínas.
Predicción computacional de genes (Gene finding)
 Predicción computacional de genes (Gene finding) Predicción de genes (Gene finding) El genoma humano tiene 3.2 GB de nucleótidos, y poco más de 20.000 genes ccgtacgtacgtagagtgctagtctagtcgtagcgccgtagtcgatcgtgtgggt
Predicción computacional de genes (Gene finding) Predicción de genes (Gene finding) El genoma humano tiene 3.2 GB de nucleótidos, y poco más de 20.000 genes ccgtacgtacgtagagtgctagtctagtcgtagcgccgtagtcgatcgtgtgggt
Existen dos tipos de genes en el genoma humano: codificantes de proteínas y codificantes de ARN.
 EL GEN Es una porción delimitida de ADN con capacidad de codificar una molécula de ARN denominada transcripto primario mediante un proceso llamado transcripción. El ADN contenido en los cromosomas de los
EL GEN Es una porción delimitida de ADN con capacidad de codificar una molécula de ARN denominada transcripto primario mediante un proceso llamado transcripción. El ADN contenido en los cromosomas de los
Cadenas de Markov en la Investigación del Genoma
 Máster en Técnicas Estadísticas - Proyecto Fin de Máster Cadenas de Markov en la Investigación del Genoma Autora: Sara Prada Alonso Directora de proyecto: María de los Ángeles Casares de Cal 8 de Julio
Máster en Técnicas Estadísticas - Proyecto Fin de Máster Cadenas de Markov en la Investigación del Genoma Autora: Sara Prada Alonso Directora de proyecto: María de los Ángeles Casares de Cal 8 de Julio
Modelos de Markov ocultos Profile HMM. Alex Sánchez. Departament d Estadística U.B. 1. Estadística i Bioinformàtica. MMO en Biologia Computacional
 Modelos de Markov ocultos Profile HMM Departament d Estadística U.B. Estadística i Bioinformàtica Esquema del tema Alineamientos múltiples de secuencias (AMS) Representaciones de AMS: Patrones y Perfiles
Modelos de Markov ocultos Profile HMM Departament d Estadística U.B. Estadística i Bioinformàtica Esquema del tema Alineamientos múltiples de secuencias (AMS) Representaciones de AMS: Patrones y Perfiles
Maestría en Bioinformática Probabilidad y Estadística: Clase 13
 Maestría en Bioinformática Probabilidad y Estadística: Clase 13 Gustavo Guerberoff gguerber@fing.edu.uy Facultad de Ingeniería Universidad de la República Mayo de 2010 Contenidos 1 Hidden Markov Models
Maestría en Bioinformática Probabilidad y Estadística: Clase 13 Gustavo Guerberoff gguerber@fing.edu.uy Facultad de Ingeniería Universidad de la República Mayo de 2010 Contenidos 1 Hidden Markov Models
Predicción de Genes. Jesús Alejandro Hernández Mejía. Cinvestav-Zacatenco. 2 de Julio de 2013
 Predicción de Genes Jesús Alejandro Hernández Mejía Cinvestav-Zacatenco 2 de Julio de 2013 Jesús Alejandro Hernández Mejía (Cinvestav) Predicción de Genes 2 de Julio de 2013 1 / 52 1 Predicción de genes
Predicción de Genes Jesús Alejandro Hernández Mejía Cinvestav-Zacatenco 2 de Julio de 2013 Jesús Alejandro Hernández Mejía (Cinvestav) Predicción de Genes 2 de Julio de 2013 1 / 52 1 Predicción de genes
Introducción a los Modelos de Markov ocultos. Alex Sánchez. Departament d Estadística U.B. 1. Estadística i Bioinformàtica
 Introducción a los Modelos de Markov ocultos Departament d Estadística U.B. Estadística i Bioinformàtica Esquema de la exposición Introducción a los MMO Tipos de problemas que podemos resolver Aplicaciones
Introducción a los Modelos de Markov ocultos Departament d Estadística U.B. Estadística i Bioinformàtica Esquema de la exposición Introducción a los MMO Tipos de problemas que podemos resolver Aplicaciones
Usando Modelos de Markov para buscar genes
 Usando Modelos de Markov para buscar genes Anotando un genoma Una vez que tenemos la secuencia de un genoma, lo siguiente es ver qué es lo que está escrito ahí. A eso se le llama anotar el genoma. Qué
Usando Modelos de Markov para buscar genes Anotando un genoma Una vez que tenemos la secuencia de un genoma, lo siguiente es ver qué es lo que está escrito ahí. A eso se le llama anotar el genoma. Qué
Tema V: Regulación génica
 SIMULACIÓN Y ANÁLISIS COMPUTACIONAL EN BIOLOGÍA DE SISTEMAS Tema V: Regulación génica Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia
SIMULACIÓN Y ANÁLISIS COMPUTACIONAL EN BIOLOGÍA DE SISTEMAS Tema V: Regulación génica Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia
GENE FINDING El problema de identificar genes en secuencias de DNA
 GENE FINDING El problema de identificar genes en secuencias de DNA Ricardo Graña Montes Mòdul de Genòmica i Proteòmica Curs 2009/2010 Necesidad de Gene Finding Algorithms Rápida Acumulación de Secuencias
GENE FINDING El problema de identificar genes en secuencias de DNA Ricardo Graña Montes Mòdul de Genòmica i Proteòmica Curs 2009/2010 Necesidad de Gene Finding Algorithms Rápida Acumulación de Secuencias
Perfiles y modelos ocultos de Markov
 Perfiles y modelos ocultos de Markov Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 13 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Perfiles y modelos ocultos de Markov 13 de junio del 2013
Perfiles y modelos ocultos de Markov Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 13 de junio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Perfiles y modelos ocultos de Markov 13 de junio del 2013
UTILIZACIÓN DE MODELOS DE INTERPOLACIÓN DE MARKOV PARA LA
 UTILIZACIÓN DE MODELOS DE INTERPOLACIÓN DE MARKOV PARA LA IDENTIFICACIÓN DE SECUENCIAS DE GENES Marco Gerardo Torres Andrade Resumen Uno de los principales problemas a resolver en la bioinformática es
UTILIZACIÓN DE MODELOS DE INTERPOLACIÓN DE MARKOV PARA LA IDENTIFICACIÓN DE SECUENCIAS DE GENES Marco Gerardo Torres Andrade Resumen Uno de los principales problemas a resolver en la bioinformática es
DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR (Francis Crick 1970) (Excepción de la transcriptasa inversa) ADN Transcripción ARN traducción PROTEINAS
 DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR (Francis Crick 1970) (Excepción de la transcriptasa inversa) ADN Transcripción ARN traducción PROTEINAS TRANSCRIPCIÓN DEL CÓDIGO: BIOSÍNTESIS DE ARN Se sintetiza
DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR (Francis Crick 1970) (Excepción de la transcriptasa inversa) ADN Transcripción ARN traducción PROTEINAS TRANSCRIPCIÓN DEL CÓDIGO: BIOSÍNTESIS DE ARN Se sintetiza
Cadenas de Markov y aplicaciones en biología computacional. Alex Sánchez. Departament d Estadística U.B. 1. Estadística i Bioinformàtica
 Cadenas de Markov y aplicaciones en biología computacional Departament d Estadística U.B. Estadística i Bioinformàtica Esquema del tema Modelos de secuencias biológicas Cadenas de Markov Definición y conceptos
Cadenas de Markov y aplicaciones en biología computacional Departament d Estadística U.B. Estadística i Bioinformàtica Esquema del tema Modelos de secuencias biológicas Cadenas de Markov Definición y conceptos
EXAMEN DE LA PRIMERA PARTE (30%) NOMBRE CÓDIGO FECHA. El examen consta de un total de 20 puntos y el tiempo máximo para contestar es de 1 hora.
 1 PONTIFICIA UNIVERSIDAD JAVERIANA MAESTRÍA EN INFORMÁTICA CURSO DE BIOINFORMÁTICA EXAMEN DE LA PRIMERA PARTE (30%) NOMBRE CÓDIGO FECHA El examen consta de un total de 20 puntos y el tiempo máximo para
1 PONTIFICIA UNIVERSIDAD JAVERIANA MAESTRÍA EN INFORMÁTICA CURSO DE BIOINFORMÁTICA EXAMEN DE LA PRIMERA PARTE (30%) NOMBRE CÓDIGO FECHA El examen consta de un total de 20 puntos y el tiempo máximo para
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA
 TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Agustín Riscos Núñez (Coord.) Carmen Graciani Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA Agustín Riscos Núñez (Coord.) Carmen Graciani Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad
Ácidos nucleicos. 3ª y 4ª Parte: Transcripción y traducción I & II. Tema 12 de Biología NS Diploma BI Curso 2012-2014
 Ácidos nucleicos 3ª y 4ª Parte: Transcripción y traducción I & II Tema 12 de Biología NS Diploma BI Curso 2012-2014 Expresión de la información genética Ya se ha visto cómo la información genética se conserva
Ácidos nucleicos 3ª y 4ª Parte: Transcripción y traducción I & II Tema 12 de Biología NS Diploma BI Curso 2012-2014 Expresión de la información genética Ya se ha visto cómo la información genética se conserva
Tecnología de ADN recombinante. Técnicas fundamentales: - Restricción - Clonado - Hibridización -PCR
 Tecnología de ADN recombinante Técnicas fundamentales: - Restricción - Clonado - Hibridización -PCR 1 Bibliotecas de ADN = Genotecas = bancos de secuencias = libraries colección de clones celulares (bacterias,
Tecnología de ADN recombinante Técnicas fundamentales: - Restricción - Clonado - Hibridización -PCR 1 Bibliotecas de ADN = Genotecas = bancos de secuencias = libraries colección de clones celulares (bacterias,
Curso de Inteligencia Artificial
 Curso de Inteligencia Artificial Modelos Ocultos de Markov Gibran Fuentes Pineda IIMAS, UNAM Redes Bayesianas Representación gráfica de relaciones probabilísticas Relaciones causales entre variables aleatorias
Curso de Inteligencia Artificial Modelos Ocultos de Markov Gibran Fuentes Pineda IIMAS, UNAM Redes Bayesianas Representación gráfica de relaciones probabilísticas Relaciones causales entre variables aleatorias
Proceso conservativo (El ADN utilizado va a permanecer intacto) y selectivo (se selecciona la parte de información genética que se transcribe)
 Características generales de la transcripción Proceso conservativo (El ADN utilizado va a permanecer intacto) y selectivo (se selecciona la parte de información genética que se transcribe) Selección del
Características generales de la transcripción Proceso conservativo (El ADN utilizado va a permanecer intacto) y selectivo (se selecciona la parte de información genética que se transcribe) Selección del
Genes. Estructura. Promotores. Intrones y exones. Pseudogenes. Genética 1 er Curso. Facultad de Medicina TEMA 0-2
 Facultad de Medicina Genética 1 er Curso TEMA 0-2 EXPRESIÓN GÉNICA:FLUJO DE INFORMACIÓN GENÉTICA Genes. El código genético. Dogma central de la Biología Molecular.. Genes Apartados Estructura Promotores
Facultad de Medicina Genética 1 er Curso TEMA 0-2 EXPRESIÓN GÉNICA:FLUJO DE INFORMACIÓN GENÉTICA Genes. El código genético. Dogma central de la Biología Molecular.. Genes Apartados Estructura Promotores
Traducción en Procariotas. en los procariotas la traducción se produce junto con la transcripción
 Traducción Traducción en Procariotas en los procariotas la traducción se produce junto con la transcripción Traducción en Eucariotas en los eucariotas la traducción se produce en el citoplasma Iniciación
Traducción Traducción en Procariotas en los procariotas la traducción se produce junto con la transcripción Traducción en Eucariotas en los eucariotas la traducción se produce en el citoplasma Iniciación
Examen de Teoría de (Introducción al) Reconocimiento de Formas
 Examen de Teoría de (Introducción al) Reconocimiento de Formas Facultad de Informática, Departamento de Sistemas Informáticos y Computación Universidad Politécnica de Valencia, Enero de 007 Apellidos:
Examen de Teoría de (Introducción al) Reconocimiento de Formas Facultad de Informática, Departamento de Sistemas Informáticos y Computación Universidad Politécnica de Valencia, Enero de 007 Apellidos:
LA TRANSCRIPCIÓN El paso de la información del ADN al ARN. Realizado por José Mayorga Fernández
 LA TRANSCRIPCIÓN El paso de la información del ADN al ARN Realizado por José Mayorga Fernández Diferencias entre la transcripción en eucariotas y en procariotas. Transcripción en procariotas En procariotas
LA TRANSCRIPCIÓN El paso de la información del ADN al ARN Realizado por José Mayorga Fernández Diferencias entre la transcripción en eucariotas y en procariotas. Transcripción en procariotas En procariotas
El Dogma Central de la Biología Molecular v.1. Manuel J. Gómez Laboratorio de Bioinformática Centro de Astrobiología INTA- CSIC
 El Dogma Central de la Biología Molecular v.1 Manuel J. Gómez Laboratorio de Bioinformática Centro de Astrobiología INTA- CSIC Flujo de información Material genético Proteínas Replicación Transcripción
El Dogma Central de la Biología Molecular v.1 Manuel J. Gómez Laboratorio de Bioinformática Centro de Astrobiología INTA- CSIC Flujo de información Material genético Proteínas Replicación Transcripción
Elementos de control de la expresión génica en ADN eucariota: el promotor. Elementos iniciadores de la transcripción (promotores) en el ADN eucariota
 Introducción El genoma eucariota es mucho más complejo que el procariota y requiere mecanismos de regulación más complejos Diferencias entre genoma eucariota y procariota PROCARIOTAS Un solo cromosoma
Introducción El genoma eucariota es mucho más complejo que el procariota y requiere mecanismos de regulación más complejos Diferencias entre genoma eucariota y procariota PROCARIOTAS Un solo cromosoma
Cadenas de Markov Ocultas
 Nicolás Carrère Análisis Inteligente de Datos Departamento de Informática Universidad Técnica Federico Santa María Valparaíso, 24 de Noviembre de 2005 1 2 3 4 Temario Las cadenas de Markov pueden ser representadas
Nicolás Carrère Análisis Inteligente de Datos Departamento de Informática Universidad Técnica Federico Santa María Valparaíso, 24 de Noviembre de 2005 1 2 3 4 Temario Las cadenas de Markov pueden ser representadas
TRANSCRIPCIÓN CONCEPTO DE OPERÓN Y PROMOTOR. González Pérez Ana Karen Robledo Sarmiento Danely
 TRANSCRIPCIÓN CONCEPTO DE OPERÓN Y PROMOTOR González Pérez Ana Karen Robledo Sarmiento Danely Transcripción Es la copia de un gen en un ARNm Ribosa Uracilo Cadena sencilla Operón Se define como una unidad
TRANSCRIPCIÓN CONCEPTO DE OPERÓN Y PROMOTOR González Pérez Ana Karen Robledo Sarmiento Danely Transcripción Es la copia de un gen en un ARNm Ribosa Uracilo Cadena sencilla Operón Se define como una unidad
Por regla general, las búsquedas con BLAST obedecen a uno de estos dos objetivos:
 BLAST en el servidor del NCBI BLAST es la herramienta bioinformática más utilizada en todo el mundo. Compara una secuencia problema (query sequence) de nucleótidos o de proteínas con todas las secuencias
BLAST en el servidor del NCBI BLAST es la herramienta bioinformática más utilizada en todo el mundo. Compara una secuencia problema (query sequence) de nucleótidos o de proteínas con todas las secuencias
Genética 1 er Curso CONCEPTOS
 Grado Medicina Genética 1 er Curso TEMA 6 MUTACIONES SIMPLES COMO CAUSA DE ENFERMEDAD 6.1 Características generales de las mutaciones. 6.2 Mutaciones simples, tipos. 6.3 Potencial patogénico de las mutaciones
Grado Medicina Genética 1 er Curso TEMA 6 MUTACIONES SIMPLES COMO CAUSA DE ENFERMEDAD 6.1 Características generales de las mutaciones. 6.2 Mutaciones simples, tipos. 6.3 Potencial patogénico de las mutaciones
SISTEMAS DE NUMERACIÓN
 SISTEMAS DE NUMERACIÓN BINARIO, OCTAL Y HEXADECIMAL EDICIÓN: 091113 LUIS GONZÁLEZ DEPARTAMENTO DE TECNOLOGÍA I.E.S. SANTA EUGENIA SISTEMAS DE NUMERACIÓN Un sistema de numeración es un conjunto de símbolos
SISTEMAS DE NUMERACIÓN BINARIO, OCTAL Y HEXADECIMAL EDICIÓN: 091113 LUIS GONZÁLEZ DEPARTAMENTO DE TECNOLOGÍA I.E.S. SANTA EUGENIA SISTEMAS DE NUMERACIÓN Un sistema de numeración es un conjunto de símbolos
Qué es un gen? Helen Pearson : Genetics: What is a gene? (Nature 441, )
 Qué es un gen? 'Gene' is not a typical four-letter word. It is not offensive. It is never bleeped out of TV shows. And where the meaning of most four-letter words is all too clear, that of gene is not.
Qué es un gen? 'Gene' is not a typical four-letter word. It is not offensive. It is never bleeped out of TV shows. And where the meaning of most four-letter words is all too clear, that of gene is not.
TRADUCCION SINTESIS DE PROTEÍNAS
 TRADUCCION SINTESIS DE PROTEÍNAS Traducción = síntesis de proteínas. Proceso que involucra la participación ordenada de más de 100 macromoléculas, Ribosomas (ARNr y proteínas) ARN mensajero ARN de transferencia
TRADUCCION SINTESIS DE PROTEÍNAS Traducción = síntesis de proteínas. Proceso que involucra la participación ordenada de más de 100 macromoléculas, Ribosomas (ARNr y proteínas) ARN mensajero ARN de transferencia
315 M/R Versión 1 Integral 1/13 2009/1 UNIVERSIDAD NACIONAL ABIERTA VICERRECTORADO ACADÉMICO ÁREA INGENIERÍA
 35 M/R Versión Integral /3 29/ UNIVERSIDAD NACIONAL ABIERTA VICERRECTORADO ACADÉMICO ÁREA INGENIERÍA MODELO DE RESPUESTA (VERSION.2) ASIGNATURA: Investigación de Operaciones I CÓDIGO: 35 MOMENTO: Prueba
35 M/R Versión Integral /3 29/ UNIVERSIDAD NACIONAL ABIERTA VICERRECTORADO ACADÉMICO ÁREA INGENIERÍA MODELO DE RESPUESTA (VERSION.2) ASIGNATURA: Investigación de Operaciones I CÓDIGO: 35 MOMENTO: Prueba
ÁRBOLES FILOGENÉTICOS
 ÁRBOLES FILOGENÉTICOS Por qué usar filogenias? El conocimiento del pasado es importante para poder resolver muchas cuestiones relacionadas con procesos biológicos. Las filogenias nos permiten obtener relaciones
ÁRBOLES FILOGENÉTICOS Por qué usar filogenias? El conocimiento del pasado es importante para poder resolver muchas cuestiones relacionadas con procesos biológicos. Las filogenias nos permiten obtener relaciones
Una población es el conjunto de todos los elementos a los que se somete a un estudio estadístico.
 Introducción a la Melilla Definición de La trata del recuento, ordenación y clasificación de los datos obtenidos por las observaciones, para poder hacer comparaciones y sacar conclusiones. Un estudio estadístico
Introducción a la Melilla Definición de La trata del recuento, ordenación y clasificación de los datos obtenidos por las observaciones, para poder hacer comparaciones y sacar conclusiones. Un estudio estadístico
EVOLUCIÓN Y GENÉTICA Fernando Tuya, Departamento Biología, Planta 1, oficina b-107,
 Bloque Temático III EVOLUCIÓN Y GENÉTICA Fernando Tuya, Departamento Biología, Planta 1, oficina b-107, E-mail: fernando.tuya@ulpgc.es, www.fernandotuya.org Tema 10. Introducción a la Genética. Conceptos
Bloque Temático III EVOLUCIÓN Y GENÉTICA Fernando Tuya, Departamento Biología, Planta 1, oficina b-107, E-mail: fernando.tuya@ulpgc.es, www.fernandotuya.org Tema 10. Introducción a la Genética. Conceptos
Biotecnología. Anotación de genes. Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla
 Biotecnología Anotación de genes Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla Localizar los genes Segundo paso en el análisis del genoma ab initio: propiedades estadísticas
Biotecnología Anotación de genes Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla Localizar los genes Segundo paso en el análisis del genoma ab initio: propiedades estadísticas
A qué da lugar el mensaje del ADN?
 A qué da lugar el mensaje del ADN? Cómo se lee el mensaje? FLUJO DE INFORMACIÓN DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR (enunciado por F. Crick) DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR ACTUALIZADO TRANSCRIPCIÓN
A qué da lugar el mensaje del ADN? Cómo se lee el mensaje? FLUJO DE INFORMACIÓN DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR (enunciado por F. Crick) DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR ACTUALIZADO TRANSCRIPCIÓN
GLOSARIO DE ANÁLISIS DE REDES SOCIALES
 GLOSARIO DE ANÁLISIS DE REDES SOCIALES Sunbelt XXI. Budapest 25-28 de Abril de 2001 LISTA REDES ÍNDICE INTRODUCCIÓN... 3 GLOSARIO... 4 EJEMPLO DE TRADUCCIÓN... 6 2 INTRODUCCIÓ N En este documento se propone
GLOSARIO DE ANÁLISIS DE REDES SOCIALES Sunbelt XXI. Budapest 25-28 de Abril de 2001 LISTA REDES ÍNDICE INTRODUCCIÓN... 3 GLOSARIO... 4 EJEMPLO DE TRADUCCIÓN... 6 2 INTRODUCCIÓ N En este documento se propone
Características e importancia del código genético
 Características e importancia del código genético Estudio del ADN como portador de la información genética. Concepto de gen. Mecanismos responsables de su transmisión y variación. Los procesos de transcripción-traducción
Características e importancia del código genético Estudio del ADN como portador de la información genética. Concepto de gen. Mecanismos responsables de su transmisión y variación. Los procesos de transcripción-traducción
Introducción a la Bioinformática Gene prediction
 Introducción a la Bioinformática Gene prediction Bioinformática - curso de posgrado Instituto de Investigaciones Biotecnológicas UNSAM Gene prediction Qué significa buscar/predecir genes? Dada una secuencia
Introducción a la Bioinformática Gene prediction Bioinformática - curso de posgrado Instituto de Investigaciones Biotecnológicas UNSAM Gene prediction Qué significa buscar/predecir genes? Dada una secuencia
descripción del argumento identificador tipo longitud condición restricción
 Recuerde que estos son apuntes muy simplificados que deberá completar con la bibliografía PROGRAMACIÓN 0 Prof. Dolores Cuiñas H. recomendada APUNTES Nº. METODOLOGÍA PARA LA CONSTRUCCIÓN DE PROGRAMAS. Presentaremos
Recuerde que estos son apuntes muy simplificados que deberá completar con la bibliografía PROGRAMACIÓN 0 Prof. Dolores Cuiñas H. recomendada APUNTES Nº. METODOLOGÍA PARA LA CONSTRUCCIÓN DE PROGRAMAS. Presentaremos
Microbiología Clínica 2006-2007. Tema 4: Transmisión de la información genética
 Microbiología Clínica 2006-2007 Tema 4: Transmisión de la información genética Transmisión de la información genética Reparto del material genético en procariontes y eucariontes. Transferencia horizontal
Microbiología Clínica 2006-2007 Tema 4: Transmisión de la información genética Transmisión de la información genética Reparto del material genético en procariontes y eucariontes. Transferencia horizontal
Muchas de las células eucariotas son especializadas: el ser humano tiene más de 200 tipos de células.
 Muchas de las células eucariotas son especializadas: el ser humano tiene más de 200 tipos de células. Si todas tienen el mismo genoma, qué las hace diferentes? Las proteínas: no todas las células fabrican
Muchas de las células eucariotas son especializadas: el ser humano tiene más de 200 tipos de células. Si todas tienen el mismo genoma, qué las hace diferentes? Las proteínas: no todas las células fabrican
DEL ADN A LAS PROTEÍNAS
 Trabajo Práctico 2.1 DEL ADN A LAS PROTEÍNAS La mayoría de los genomas de todas las formas de vida celular están compuestos por ADN (ácido desoxirribonucleico), mientras que algunos pocos virus tienen
Trabajo Práctico 2.1 DEL ADN A LAS PROTEÍNAS La mayoría de los genomas de todas las formas de vida celular están compuestos por ADN (ácido desoxirribonucleico), mientras que algunos pocos virus tienen
Unidad 1 Números. Los números naturales son aquellos que se utilizan para contar los elementos de un conjunto.
 Unidad 1 Números 1.- Números Naturales Los números naturales son aquellos que se utilizan para contar los elementos de un conjunto. El conjunto de números naturales se representa por la letra N Operaciones
Unidad 1 Números 1.- Números Naturales Los números naturales son aquellos que se utilizan para contar los elementos de un conjunto. El conjunto de números naturales se representa por la letra N Operaciones
Tecnología del ADN recombinante.
 Tecnología del ADN recombinante. Genética molecular procariota Principios de regulación. Ejemplos. La célula procariótica es más sencilla Las células de plantas y animales son diferentes La mayoría de
Tecnología del ADN recombinante. Genética molecular procariota Principios de regulación. Ejemplos. La célula procariótica es más sencilla Las células de plantas y animales son diferentes La mayoría de
Evolución de secuencias de DNA
 Evolución de secuencias de DNA 4 transiciones (α) 8 transversiones (β) Proporción esperada Transiciones/Transversiones = 1/2 DESDE HASTA ATCTAGATCTAGTGCATAGCATGCA * * * * * * ACCTAAATTTAGTGAATATCATCCA
Evolución de secuencias de DNA 4 transiciones (α) 8 transversiones (β) Proporción esperada Transiciones/Transversiones = 1/2 DESDE HASTA ATCTAGATCTAGTGCATAGCATGCA * * * * * * ACCTAAATTTAGTGAATATCATCCA
PROBLEMAS MATEMATICOS EN SEXTO DE EDUCACIÓN PRIMARIA. (2006/2007: Comienzo del curso)
 COLEGIO VIRGEN DE ATOCHA PP. DOMINICOS MADRID ORIENTACIÓN AV. CIUDAD DE BARCELONA, 1 28007 MADRID TELF. 91 552 48 04 FAX 91 552 96 43 http:// www.cvatocha.com PROBLEMAS MATEMATICOS EN SEXTO DE EDUCACIÓN
COLEGIO VIRGEN DE ATOCHA PP. DOMINICOS MADRID ORIENTACIÓN AV. CIUDAD DE BARCELONA, 1 28007 MADRID TELF. 91 552 48 04 FAX 91 552 96 43 http:// www.cvatocha.com PROBLEMAS MATEMATICOS EN SEXTO DE EDUCACIÓN
REGRESIÓN LINEAL CON SPSS
 ESCUELA SUPERIOR DE INFORMÁTICA Prácticas de Estadística REGRESIÓN LINEAL CON SPSS 1.- INTRODUCCIÓN El análisis de regresión lineal es una técnica estadística utilizada para estudiar la relación entre
ESCUELA SUPERIOR DE INFORMÁTICA Prácticas de Estadística REGRESIÓN LINEAL CON SPSS 1.- INTRODUCCIÓN El análisis de regresión lineal es una técnica estadística utilizada para estudiar la relación entre
Aplicaciones de la programación
 Tema 3 Aplicaciones de la programación dinámica 3.1. Problemas de Inventario Ejemplo 3.1. Supóngase que una empresa sabe que la demanda de un determinado producto durante cada uno de los próximos cuatro
Tema 3 Aplicaciones de la programación dinámica 3.1. Problemas de Inventario Ejemplo 3.1. Supóngase que una empresa sabe que la demanda de un determinado producto durante cada uno de los próximos cuatro
Procesamiento de voz - Reconocimiento de voz II
 Procesamiento de voz - Reconocimiento de voz II Marc S. Reßl Roxana Saint-Nom 2009 Ingeniería Electrónica Instituto Tecnológico de Buenos Aires Reconocimiento de voz Las técnicas que vimos hasta ahora
Procesamiento de voz - Reconocimiento de voz II Marc S. Reßl Roxana Saint-Nom 2009 Ingeniería Electrónica Instituto Tecnológico de Buenos Aires Reconocimiento de voz Las técnicas que vimos hasta ahora
2. La figura representa un segmento de la fibra básica de cromatina.
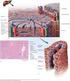 Bloque IV: Base molecular de la herencia 1. a. El esquema representa un cromosoma eucariótico en metafase mitótica. Qué denominación reciben los elementos indicados por los números 1 a 4? b. Dibuje un
Bloque IV: Base molecular de la herencia 1. a. El esquema representa un cromosoma eucariótico en metafase mitótica. Qué denominación reciben los elementos indicados por los números 1 a 4? b. Dibuje un
Instituto tecnológico de Minatitlán. Investigación de operaciones Ing. Erika Lissette Minaya mortera Unidad 3: programación no lineal
 Instituto tecnológico de Minatitlán Investigación de operaciones Ing. Erika Lissette Minaya mortera Unidad 3: programación no lineal Alejandra de la cruz francisco Ingeniería en sistemas computacionales
Instituto tecnológico de Minatitlán Investigación de operaciones Ing. Erika Lissette Minaya mortera Unidad 3: programación no lineal Alejandra de la cruz francisco Ingeniería en sistemas computacionales
SÍNTESIS DE PROTEÍNAS
 SÍNTESIS DE PROTEÍNAS SINTESIS DE PROTEÍNAS FLUJO DE INFORMACIÓN ESTRUCTURA DEL RNA TRANSCRIPCIÓN CAMBIOS POST TRANSCRIPCIONALES CÓDIGO GENÉTICO TRADUCCIÓN POLIRRIBOSOMAS MUTACIONES MODIFICACIONES POSTRADUCCIONALES
SÍNTESIS DE PROTEÍNAS SINTESIS DE PROTEÍNAS FLUJO DE INFORMACIÓN ESTRUCTURA DEL RNA TRANSCRIPCIÓN CAMBIOS POST TRANSCRIPCIONALES CÓDIGO GENÉTICO TRADUCCIÓN POLIRRIBOSOMAS MUTACIONES MODIFICACIONES POSTRADUCCIONALES
PRÁCTICA 1. Mediciones
 PRÁCTICA 1 Mediciones Objetivo General El alumno determinará la incertidumbre de las mediciones. Objetivos particulares 1. El alumno determinará las incertidumbres a partir de los instrumentos de medición..
PRÁCTICA 1 Mediciones Objetivo General El alumno determinará la incertidumbre de las mediciones. Objetivos particulares 1. El alumno determinará las incertidumbres a partir de los instrumentos de medición..
Análisis genético mediante secuenciación masiva. Biocomputación Grado en Bioquímica
 Análisis genético mediante secuenciación masiva Biocomputación Grado en Bioquímica Motivación Existen diferentes situaciones que requieren la determinación del genotipo de un individuo Asesoramiento genético:
Análisis genético mediante secuenciación masiva Biocomputación Grado en Bioquímica Motivación Existen diferentes situaciones que requieren la determinación del genotipo de un individuo Asesoramiento genético:
REPLICACIÓN DEL ADN. Procesos de replicación, transcripción y traducción.
 REPLICACIÓN DEL ADN Procesos de replicación, transcripción y traducción. La transmisión de información implica que el ADN es capaz de duplicarse de manera de obtener dos moléculas iguales a partir de la
REPLICACIÓN DEL ADN Procesos de replicación, transcripción y traducción. La transmisión de información implica que el ADN es capaz de duplicarse de manera de obtener dos moléculas iguales a partir de la
Expresión del material hereditario. Regulación en procariontes. Regulación en Eucariontes.
 Expresión del material hereditario. Regulación en procariontes. Regulación en Eucariontes. Genética Moderna. Griffiths, Gelbart, Miller, Lewontin. Capítulo 3. La Función de los Genes. Capítulo 14. Regulación
Expresión del material hereditario. Regulación en procariontes. Regulación en Eucariontes. Genética Moderna. Griffiths, Gelbart, Miller, Lewontin. Capítulo 3. La Función de los Genes. Capítulo 14. Regulación
Biotecnología de células procarióticas. Profesor: Javier Cabello Schomburg, MS
 Biotecnología de células procarióticas Profesor: Javier Cabello Schomburg, MS Reflexión Cuando amamos una persona conocemos sus defectos, igual o mejor que los nuestros, y la amamos con ellos. Si yo quiero
Biotecnología de células procarióticas Profesor: Javier Cabello Schomburg, MS Reflexión Cuando amamos una persona conocemos sus defectos, igual o mejor que los nuestros, y la amamos con ellos. Si yo quiero
Colegio Beato Carlos Manuel Rodríguez Departamento de Matemáticas. Mapa curricular Algebra I 8 vo grado
 Colegio Beato Carlos Manuel Rodríguez Departamento de Matemáticas Mapa curricular Algebra I 8 vo grado Colegio Beato Carlos Manuel Rodríguez Mapa curricular Algebra I 8 vo grado periodo 11 al 22 de agosto
Colegio Beato Carlos Manuel Rodríguez Departamento de Matemáticas Mapa curricular Algebra I 8 vo grado Colegio Beato Carlos Manuel Rodríguez Mapa curricular Algebra I 8 vo grado periodo 11 al 22 de agosto
TRABAJOS PRÁCTICOS DE BIOLOGÍA C
 TRABAJOS PRÁCTICOS DE BIOLOGÍA C TRABAJO PRÁCTICO N 1 Actividad N 1 1) En la naturaleza las células pueden ser de dos tipos: procariotas o eucariotas. Señale con una X las características de cada una según
TRABAJOS PRÁCTICOS DE BIOLOGÍA C TRABAJO PRÁCTICO N 1 Actividad N 1 1) En la naturaleza las células pueden ser de dos tipos: procariotas o eucariotas. Señale con una X las características de cada una según
Herramientas de Bioinformática en NGS
 Herramientas de Bioinformática en NGS Ing. Sergio Gonzalez CICVyA - Instituto de Biotecnología INTA gonzalez.sergio@inta.gob.ar Temario Introducción NGS Análisis de calidad Ensamblado Anotación Estructural
Herramientas de Bioinformática en NGS Ing. Sergio Gonzalez CICVyA - Instituto de Biotecnología INTA gonzalez.sergio@inta.gob.ar Temario Introducción NGS Análisis de calidad Ensamblado Anotación Estructural
Introducción. Reversibilidad de la diferenciación. Experiencias
 Introducción Control de la expresión génica En un organismo multicelular existen muchos tipos celulares (diferenciación). Tienen la misma información genética, pero poseen morfología y fisiología distintas.
Introducción Control de la expresión génica En un organismo multicelular existen muchos tipos celulares (diferenciación). Tienen la misma información genética, pero poseen morfología y fisiología distintas.
Tema 7.- Genética Molecular. Biología y Geología 4º ESO: Genética Molecular
 Tema 7.- Genética Molecular 1 El ADN, la molécula de la herencia El ADN (ácido desoxirribonucléico), es el portador de la información genética y es el responsable de las características biológicas de un
Tema 7.- Genética Molecular 1 El ADN, la molécula de la herencia El ADN (ácido desoxirribonucléico), es el portador de la información genética y es el responsable de las características biológicas de un
Estadística de secuencias genómicas. Rodrigo Santamaría
 Estadística de secuencias genómicas Rodrigo Santamaría Estadística de secuencias genómicas Objetivo Modelos probabilísticos Significación estadística Modelos ocultos de Markov Objetivo Encontrar estructuras
Estadística de secuencias genómicas Rodrigo Santamaría Estadística de secuencias genómicas Objetivo Modelos probabilísticos Significación estadística Modelos ocultos de Markov Objetivo Encontrar estructuras
Modelos de input-output y cadenas de Markov
 MaMaEuSch Management Mathematics for European Schools http://www.mathematik.unikl.de/~mamaeusch/ Modelos de input-output y cadenas de Markov Ao. Univ.-Prof. Werner Peschek El proyecto MaMaEuSch ha sido
MaMaEuSch Management Mathematics for European Schools http://www.mathematik.unikl.de/~mamaeusch/ Modelos de input-output y cadenas de Markov Ao. Univ.-Prof. Werner Peschek El proyecto MaMaEuSch ha sido
REPRESENTACIÓN SÍMBOLICA DE UNIONES SOLDADAS UNE-EN ISO 2553:2014
 REPRESENTACIÓN SÍMBOLICA DE UNIONES SOLDADAS UNE-EN ISO 2553:2014 1. INTRODUCCIÓN La soldadura es uno de los procedimientos más habituales para la obtención de uniones fijas (permanentes), o no desmontables,
REPRESENTACIÓN SÍMBOLICA DE UNIONES SOLDADAS UNE-EN ISO 2553:2014 1. INTRODUCCIÓN La soldadura es uno de los procedimientos más habituales para la obtención de uniones fijas (permanentes), o no desmontables,
Pasos en el Método Simplex
 Pontificia Universidad Católica Escuela de Ingeniería Departamento de Ingeniería Industrial y de Sistemas Clase 20 El Método Simplex ICS 1102 Optimización Profesor : Claudio Seebach 16 de octubre de 2006
Pontificia Universidad Católica Escuela de Ingeniería Departamento de Ingeniería Industrial y de Sistemas Clase 20 El Método Simplex ICS 1102 Optimización Profesor : Claudio Seebach 16 de octubre de 2006
REFUERZO DE BIOLOGÍA 4º ESO: TEMA 1, 2 Y 3. 2. Nombra las principales partes de la siguiente célula procariota indicando sus funciónes.
 C.M. MIGUEL CASTILLEJO Fundación VERA-CRUZ Ciencias experimentales BIOLOGÍA CURSO 4º /GRUPO A Código PC.04.4-EXM-30 REFUERZO DE BIOLOGÍA 4º ESO: TEMA 1, 2 Y 3 1. Indica el nivel de organización de cada
C.M. MIGUEL CASTILLEJO Fundación VERA-CRUZ Ciencias experimentales BIOLOGÍA CURSO 4º /GRUPO A Código PC.04.4-EXM-30 REFUERZO DE BIOLOGÍA 4º ESO: TEMA 1, 2 Y 3 1. Indica el nivel de organización de cada
Introducción a la Bioinformática
 Introducción a la Bioinformática Genómica y bioinformática: Nuevas áreas de biotecnología Genómica-Ciencia que se encarga de las estrategias de clonación, secuenciación y análisis de genes. Cómo los científicos
Introducción a la Bioinformática Genómica y bioinformática: Nuevas áreas de biotecnología Genómica-Ciencia que se encarga de las estrategias de clonación, secuenciación y análisis de genes. Cómo los científicos
F.I.G.: Experimento de Volkin and Astrachan, 1956
 F.I.G.: Experimento de Volkin and Astrachan, 1956 Pi 32 Ultracentrifugación en CsCl y BrEt DNA 5 Bacterias más fagos RNA Aislamiento de ácidos nucleicos Cómo seguiría el experimento? Qué esperaría? F.I.G.:
F.I.G.: Experimento de Volkin and Astrachan, 1956 Pi 32 Ultracentrifugación en CsCl y BrEt DNA 5 Bacterias más fagos RNA Aislamiento de ácidos nucleicos Cómo seguiría el experimento? Qué esperaría? F.I.G.:
Introducción a las imágenes digitales. Segunda parte
 Introducción a las imágenes digitales Segunda parte Introducción a las imágenes digitales Herramientas matemáticas. Transformaciones de intensidad. Histograma de una imagen. Imágenes a color. Modelos de
Introducción a las imágenes digitales Segunda parte Introducción a las imágenes digitales Herramientas matemáticas. Transformaciones de intensidad. Histograma de una imagen. Imágenes a color. Modelos de
APLICACIÓN DE ÁRBOLES BINARIOS
 PRÁCTICA Nº 6: 2 sesiones (del 22 al 31 de Mayo de 2002) APLICACIÓN DE ÁRBOLES BINARIOS A LA COMPRESIÓN DE FICHEROS DE TEXTO MEDIANTE LA UTILIZACIÓN DE LOS CÓDIGOS DE HUFFMAN 0.- OBJETIVOS El objetivo
PRÁCTICA Nº 6: 2 sesiones (del 22 al 31 de Mayo de 2002) APLICACIÓN DE ÁRBOLES BINARIOS A LA COMPRESIÓN DE FICHEROS DE TEXTO MEDIANTE LA UTILIZACIÓN DE LOS CÓDIGOS DE HUFFMAN 0.- OBJETIVOS El objetivo
Diferencia entre Procariotas y Eucariotas
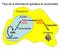 Diferencia entre Procariotas y Eucariotas TRANSCRIPCION Proceso encargado de la síntesis de una molécula de ARN a partir de la información genética contenida en una molécula d ADN Da lugar a una copia
Diferencia entre Procariotas y Eucariotas TRANSCRIPCION Proceso encargado de la síntesis de una molécula de ARN a partir de la información genética contenida en una molécula d ADN Da lugar a una copia
TRANSCRIPCION Y TRADUCCION
 TRANSCRIPCION Y TRADUCCION BibliograJa: CurKs. 7ma edición. Capítulo 10. (Código fotocopiadora 2160) htp://www.curksbiologia.com/indice_figuras_animadas Asignatura: Biología para IRNR Profesora Responsable:
TRANSCRIPCION Y TRADUCCION BibliograJa: CurKs. 7ma edición. Capítulo 10. (Código fotocopiadora 2160) htp://www.curksbiologia.com/indice_figuras_animadas Asignatura: Biología para IRNR Profesora Responsable:
1. Cuál es el complemento cromosómico diploide de la especie?: a. 2n=3 b. n=3 c. n=6 d. 2n=6
 Prototipo 1er parcial BCM-2016 PROBLEMA 1 El dibujo muestra un esquema del complemento cromosómico de tres individuos de una especie de monos. La célula dibujada de cada individuo, es una célula somática
Prototipo 1er parcial BCM-2016 PROBLEMA 1 El dibujo muestra un esquema del complemento cromosómico de tres individuos de una especie de monos. La célula dibujada de cada individuo, es una célula somática
Tema II: Programación Lineal
 Tema II: Programación Lineal Contenido: Solución a problemas de P.L. por el método gráfico. Objetivo: Al finalizar la clase los alumnos deben estar en capacidad de: Representar gráficamente la solución
Tema II: Programación Lineal Contenido: Solución a problemas de P.L. por el método gráfico. Objetivo: Al finalizar la clase los alumnos deben estar en capacidad de: Representar gráficamente la solución
DaVinciTEXTIL. Codificación de artículos
 Codificación de artículos El código de los artículos, consta de un máximo de 10 caracteres alfanuméricos Antes de empezar a codificar los artículos/materiales se debe tener en cuenta que el sistema permite
Codificación de artículos El código de los artículos, consta de un máximo de 10 caracteres alfanuméricos Antes de empezar a codificar los artículos/materiales se debe tener en cuenta que el sistema permite
CUESTIONES DE SELECTIVIDAD GENÉTICA MOLECULAR 1. REPLICACION DEL ADN
 CUESTIONES DE SELECTIVIDAD GENÉTICA MOLECULAR 1. REPLICACION DEL ADN 1.- Con relación al proceso de replicación del ADN: a) Qué es la replicación del ADN? (0,5 b) Cuál es su significado biológico? (0,5
CUESTIONES DE SELECTIVIDAD GENÉTICA MOLECULAR 1. REPLICACION DEL ADN 1.- Con relación al proceso de replicación del ADN: a) Qué es la replicación del ADN? (0,5 b) Cuál es su significado biológico? (0,5
SÍNTESIS DE PROTEINAS DEL ADN A LAS PROTEÍNAS
 SÍNTESIS DE PROTEINAS DEL ADN A LAS PROTEÍNAS La molécula de ADN contiene toda la información hereditaria de los seres vivos es decir en ella se encuentran los genes que determinan las características
SÍNTESIS DE PROTEINAS DEL ADN A LAS PROTEÍNAS La molécula de ADN contiene toda la información hereditaria de los seres vivos es decir en ella se encuentran los genes que determinan las características
EJEMPLO DE SIMPLEX PARA PROBLEMA DE PROGRAMACIÓN LINEAL CASO DE MAXIMIZAR Prof.: MSc. Julio Rito Vargas Avilés
 EJEMPLO DE SIMPLEX PARA PROBLEMA DE PROGRAMACIÓN LINEAL CASO DE MAXIMIZAR Prof.: MSc. Julio Rito Vargas Avilés CONSTRUCCION DE LA TABLA INICIAL DEL MÉTODO SIMPLEX Una vez que el alumno ha adquirido la
EJEMPLO DE SIMPLEX PARA PROBLEMA DE PROGRAMACIÓN LINEAL CASO DE MAXIMIZAR Prof.: MSc. Julio Rito Vargas Avilés CONSTRUCCION DE LA TABLA INICIAL DEL MÉTODO SIMPLEX Una vez que el alumno ha adquirido la
Representación de números enteros: el convenio complemento a uno
 Representación de números enteros: el convenio complemento a uno Apellidos, nombre Martí Campoy, Antonio (amarti@disca.upv.es) Departamento Centro Informàtica de Sistemes i Computadors Escola Tècnica Superior
Representación de números enteros: el convenio complemento a uno Apellidos, nombre Martí Campoy, Antonio (amarti@disca.upv.es) Departamento Centro Informàtica de Sistemes i Computadors Escola Tècnica Superior
Revisión de opción múltiple- Genes
 Revisión de opción múltiple- Genes 1. Los nucleótidos del ácido desoxirribonucleico están compuestos de: a. Azúcar ribosa, un grupo fosfato y uno de cuatro bases (adenina, citosina, timina y guanina) b.
Revisión de opción múltiple- Genes 1. Los nucleótidos del ácido desoxirribonucleico están compuestos de: a. Azúcar ribosa, un grupo fosfato y uno de cuatro bases (adenina, citosina, timina y guanina) b.
TEMA 3: Expresión Génica
 TEMA 3: Expresión Génica Genómica Estructural: composición de los Genomas ADN Génico y Relacionado: 37% (1.5% CODIFICANTE, EXONES!!) ADN No Codificante: 63% (44 % ELEMENTOS TRANSPONIBLES) 1.5% 44% CONCEPTO
TEMA 3: Expresión Génica Genómica Estructural: composición de los Genomas ADN Génico y Relacionado: 37% (1.5% CODIFICANTE, EXONES!!) ADN No Codificante: 63% (44 % ELEMENTOS TRANSPONIBLES) 1.5% 44% CONCEPTO
11 knúmero de publicación: 2 154 624. 51 kint. Cl. 6 : C12N 15/54 DE PATENTE EUROPEA
 k 19 OFICINA ESPAÑOLA DE PATENTES Y MARCAS ESPAÑA 11 knúmero de publicación: 2 154 624 51 kint. Cl. 6 : C12N 15/54 C12N 15/82, C12N 15/70 C12N 15/11, C12N 9/10 C12N 5/10, A01H 5/00 12 k TRADUCCION DE REIVINDICACIONES
k 19 OFICINA ESPAÑOLA DE PATENTES Y MARCAS ESPAÑA 11 knúmero de publicación: 2 154 624 51 kint. Cl. 6 : C12N 15/54 C12N 15/82, C12N 15/70 C12N 15/11, C12N 9/10 C12N 5/10, A01H 5/00 12 k TRADUCCION DE REIVINDICACIONES
e-mail: mtoledo@dgrfsa.gov.ar
 Arreglos Introducción a las Estructuras de Datos. Arreglos Unidimensionales: Vectores Vectores Estáticos en VB Vectores Dinámicos en VB Ordenando un Vector Introducción a las Estructuras de Datos. Una
Arreglos Introducción a las Estructuras de Datos. Arreglos Unidimensionales: Vectores Vectores Estáticos en VB Vectores Dinámicos en VB Ordenando un Vector Introducción a las Estructuras de Datos. Una
Dogma central de la Biología Molecular. Replicación ADN. Transcripción ARN. Traducción. Proteínas
 Dogma central de la Biología Molecular ADN ARN Proteínas Replicación Transcripción Traducción Replicación En la horquilla se encuentra enzimas y proteínas con funciones diferentes Primasa ADN polimerasa
Dogma central de la Biología Molecular ADN ARN Proteínas Replicación Transcripción Traducción Replicación En la horquilla se encuentra enzimas y proteínas con funciones diferentes Primasa ADN polimerasa
Los fenómenos psicológicos (por ejemplo, la comunicación verbal) se puede analizar desde distintos puntos de vista:
 Los fenómenos psicológicos (por ejemplo, la comunicación verbal) se puede analizar desde distintos puntos de vista: (1) Cuáles son los procesos mentales que realizamos para construir una frase? (por ejemplo,
Los fenómenos psicológicos (por ejemplo, la comunicación verbal) se puede analizar desde distintos puntos de vista: (1) Cuáles son los procesos mentales que realizamos para construir una frase? (por ejemplo,
EJERCICIOS PAU (Castilla y León) Fuente:
 Temas 14 y 15. Genética Molecular (Transcripción, traducción, mutaciones) Preguntas anteriores a 2010?? EJERCICIOS PAU (Castilla y León) Fuente: http://www.usal.es/node/28881 1. Si la hebra codificante
Temas 14 y 15. Genética Molecular (Transcripción, traducción, mutaciones) Preguntas anteriores a 2010?? EJERCICIOS PAU (Castilla y León) Fuente: http://www.usal.es/node/28881 1. Si la hebra codificante
Conversión Analógica a Digital
 Índice Conversión analógica a digital Señales básicas de tiempo discreto Relación Exponencial Discreta con sinusoides Relación Exponencial discreta con sinusoides Propiedades exponenciales complejas continuas
Índice Conversión analógica a digital Señales básicas de tiempo discreto Relación Exponencial Discreta con sinusoides Relación Exponencial discreta con sinusoides Propiedades exponenciales complejas continuas
Acido ribonucleico RNA En RNA U aparea con A
 Metabolismo del RNA Transcripcion, enzimas, localizacion, inhibicion En procariotas, factor sigma En eucariotas, sequencias cis-regulatorios, TATA, inr, enhancer In eucariotas, Pol II, TF, iniciacion,
Metabolismo del RNA Transcripcion, enzimas, localizacion, inhibicion En procariotas, factor sigma En eucariotas, sequencias cis-regulatorios, TATA, inr, enhancer In eucariotas, Pol II, TF, iniciacion,
CURSOSO. Aritmética: Númerosnaturalesyenteros. Númerosracionalesyfraciones. MATEMÁTICAS. AntonioF.CostaGonzález
 CURSOSO CURSOSO MATEMÁTICAS Aritmética: Númerosnaturalesyenteros. Númerosracionalesyfraciones. AntonioF.CostaGonzález DepartamentodeMatemáticasFundamentales FacultaddeCiencias Índice 1 Introducción y objetivos
CURSOSO CURSOSO MATEMÁTICAS Aritmética: Númerosnaturalesyenteros. Númerosracionalesyfraciones. AntonioF.CostaGonzález DepartamentodeMatemáticasFundamentales FacultaddeCiencias Índice 1 Introducción y objetivos
Tema 11: Intervalos de confianza.
 Tema 11: Intervalos de confianza. Presentación y Objetivos. En este tema se trata la estimación de parámetros por intervalos de confianza. Consiste en aproximar el valor de un parámetro desconocido por
Tema 11: Intervalos de confianza. Presentación y Objetivos. En este tema se trata la estimación de parámetros por intervalos de confianza. Consiste en aproximar el valor de un parámetro desconocido por
II. SECCIONES PRINCIPALES 1-2-3... Figura1: Partes principales de un Informe Técnico
 Formato del Informe El informe técnico debe ser un documento profesional. Para clarificar el texto deben usarse Figuras, tablas y dibujos. Las tres partes principales de un informe técnico se muestran
Formato del Informe El informe técnico debe ser un documento profesional. Para clarificar el texto deben usarse Figuras, tablas y dibujos. Las tres partes principales de un informe técnico se muestran
SERIE INFORMES TÉCNICOS Nº. 2 / 93 UTILIZACIÓN DE LA ESPECTROSCOPIA EN EL INFRARROJO CERCANO PARA EL ANÁLISIS DE FORRAJES Y MATERIAS PRIMAS
 UTILIZACIÓN DE LA ESPECTROSCOPIA EN EL INFRARROJO CERCANO PARA EL ANÁLISIS DE FORRAJES Y MATERIAS PRIMAS SERIE INFORMES TÉCNICOS Nº. 2 / 93 Instituto de Experimentación y Promoción Agraria. UTILIZACIÓN
UTILIZACIÓN DE LA ESPECTROSCOPIA EN EL INFRARROJO CERCANO PARA EL ANÁLISIS DE FORRAJES Y MATERIAS PRIMAS SERIE INFORMES TÉCNICOS Nº. 2 / 93 Instituto de Experimentación y Promoción Agraria. UTILIZACIÓN
