Luis Alfredo Baquero Suárez
|
|
|
- Juan Francisco Miguélez Salinas
- hace 6 años
- Vistas:
Transcripción
1 Identificación y Caracterización del Antígeno de Micronemas con Anclaje a GPI de Plasmodium vivax (PvGAMA) y Determinación de la Unión a Reticulocitos Humanos. Luis Alfredo Baquero Suárez Universidad Nacional de Colombia Posgrado Interfacultades de Microbiología Bogotá, Colombia 2017
2 Identificación y Caracterización del Antígeno de Micronemas con Anclaje a GPI de Plasmodium vivax (PvGAMA) y Determinación de la Unión a Reticulocitos Humanos. Luis Alfredo Baquero Suárez Trabajo de grado presentado como requisito para optar al título de: Magíster en Ciencias, Microbiología Director: Darwin Andrés Moreno Pérez M.Sc., Dr.Sc. (c) Codirector: Manuel Alfonso Patarroyo Gutiérrez M.D., Dr.Sc. Línea de Investigación: Biología Molecular de Agentes Infecciosos Grupo de Investigación: Biología Molecular Malaria (FIDIC) Universidad Nacional de Colombia Posgrado Interfacultades de Microbiología Bogotá, Colombia 2017
3 A mis padres, por su lucha incansable de sacarme adelante. A mi hermano por estar siempre acompañándome y a DIOS porque sin él, nada se logra.
4 Agradecimientos Infinitas gracias a Darwin Andrés Moreno Pérez, mi director de tesis, por todos los aportes realizados a este trabajo, por ser un gran maestro en mi formación y por su lucha constante para sacar adelante este proyecto. Muchas gracias al Doctor Manuel Alfonso Patarroyo, mi jefe, por sus valiosos aportes al desarrollo del trabajo, su codirección y por permitirme hacer parte de su grupo de investigación. Muchas gracias al Doctor Manuel Elkin Patarroyo, mi otro jefe, por su ejemplo de persistencia para lograr lo que se quiere. A todos mis compañeros de grupo, por su ayuda constante, infinitas gracias. Al posgrado interfacultades en Microbiología de la Universidad Nacional de Colombia, en especial a las Profesoras Marta Raquel Fontanilla y María Teresa Reguero, por sus consejos y ayuda en mi formación profesional. También a Socorro Prieto por su acompañamiento incondicional.
5 V Resumen Las proteínas tipo adhesinas son usadas por parásitos del género Plasmodium para unirse e invadir sus células diana. Por lo tanto, caracterizar las moléculas que participan en la interacción con los reticulocitos humanos es un punto clave para entender la base molecular del mecanismo de invasión utilizado por el parásito Plamodium vivax. Este estudio describe por primera vez la caracterización y la actividad de unión a reticulocitos del homólogo al Antígeno de Micronemas con Anclaje a GPI de Plasmodium falciparum (PfGAMA del inglés, GPI-Anchored Micronemal Antigen) en la cepa VCG-I de P. vivax. El gen pvgama se transcribe en esquizontes de la cepa VCG-I de P. vivax, codifica para una proteína de 771 aa la cual está compuesta de un péptido señal, una secuencia de anclaje a GPI y una región repetitiva exclusiva de la especie. PvGAMA se expresa en esquizontes maduros, presenta un patrón de localización apical y desencadena una respuesta antigénica durante una infección natural por P. vivax. Dos regiones conservadas de la proteína presentan actividad de unión a reticulocitos con fenotipo duffy Fya-Fyb+. Este estudio describe la caracterización funcional y antigénica de GAMA en P. vivax. La proteína es altamente antigénica y está compuesta por dominios de unión que interactúan con reticulocitos humanos. Estos hallazgos sugieren que PvGAMA tiene un importante papel como adhesina en merozoitos de P. vivax. Palabras claves: proteínas de adhesión, Plasmodium vivax, caracterización, unión a reticulocitos.
6 VI Abstract. Adhesin proteins are used by Plasmodium parasites to bind to and invade target cells. Hence, characterizing such molecules or part of them participating in human reticulocyte interaction is a key point for understanding the molecular basis of Plasmodium vivax mechanism invasion. This study thus describes for the first time the characterization and the reticulocyte binding activity of the new homologous protein to Plasmodium falciparum GPI-Anchored Micronemal Antigen in the Plasmodium vivax VCG-I strain. The pvgama gene was transcribed in P. vivax VCG-I strain schizonts, encoding 771 amino acids which include a secretion signal, a GPI-anchor site and a repeat region exclusive to the species. PvGAMA was expressed on mature schizonts, had an apical localization pattern and could trigger an antigen response during natural infection. Two conserved regions of the protein displayed reticulocyte binding activity to cells having the Fya-Fyb+ duffy phenotype. This study describes the functional and antigenic characterization of GAMA in P. vivax. The protein is highly antigenic and is composed of binding domains that interact with human reticulocytes. These findings suggest that GAMA plays an important role as adhesin in merozoites of P. vivax. Key words: adhesion proteins, Plasmodium vivax, characterization, reticulocyte binding.
7 VII Contenido Pág Resumen... V Lista de figuras... IX Lista de tablas... X Lista de abreviaturas... XI Introducción Hipótesis Objetivos Marco Teórico La malaria Ciclo de vida de Plasmodium Fase eritrocítica Mecanismos y fases de la invasión Candidatos a vacuna en P. vivax Vacunas contra la malaria Materiales y métodos Tipo de investigación Análisis bioinformático, diseño de cebadores y péptidos sintéticos Aislamiento y enriquecimiento de esquizontes de P. vivax Extracción de material biológico Clonación y secuenciación Análisis de polimorfismo de PvGAMA Expresión y purificación de los fragmentos recombinantes... 41
8 VIII Extracción denaturante Extracción en condiciones no detaturantes Purificación de las proteínas Obtención de anticuerpos policlonales Western blot de las proteínas recombinantes y del parásito Localización de la proteína por Inmunofluorescencia indirecta (IFI) Colección y procesamiento de muestras de sangre Ensayo por inmunoadsorción ligado a enzimas (ELISA) Procesamiento de muestras de sangre de cordón umbilical Ensayo de unión a reticulocitos humanos Análisis estadístico Resultados Identificación in silico de proteínas relacionadas con la invasión de P. vivax El gen pvgama se transcribe en esquizontes de la cepa VCG-I de P. vivax PvGAMA se expresa en esquizontes maduros y se localiza en el polo apical del parásito Diversidad genética y señales de selección en el antígeno GAMA PvGAMA es antigénica durante una infección natural por P. vivax PvGAMA se une a reticulocitos humanos Discusión Conclusiones Perspectivas Aspectos éticos Consentimiento informado Manejo y regulaciones para el cuidado animal Anexos Referencias... 89
9 IX Lista de figuras Figura 3-1. Distribución global de la malaria causada por especies del género Plasmodium.. 20 Figura 3-2. Ciclo de vida de Plasmodium Figura 3-3. Estructura del merozoito Figura 3-4. Identificación de GAMA en P. falciparum Figura 3.5. Fases de la invasión al glóbulo rojo Figura 3-6. Fases de la invasión a reticulocitos por P. vivax Figura 3-7. Candidatos identificados en P. falciparum y P. vivax Figura 5-8. Caracterización de PvGAMA Figura 5-9. Ventana deslizante de índice ω Figura Amplificación por PCR de los fragmentos de GAMA Figura Detección de las proteínas recombinantes Figura Antigenicidad de PvGAMA durante una infección natural por malaria Figura Obtención de las regiones conservadas y variables de PvGAMA en su forma soluble Figura Análisis de citometría de flujo Figura Actividad de unión de PvGAMA a reticulocitos humanos Pág.
10 X Lista de tablas Tabla 4-1. Fragmentos recombinantes de GAMA Tabla 4-2. Epítopes lineales de células B diseñados de acuerdo al análisis de ANTHEPROT.. 43 Pág
11 XI Lista de abreviaturas Abreviatura Término GPI Glicosilfosfatidilinositol EDTA Ácido Etilendiaminotetraacético. VCG-I Vivax Colombia Guaviare I (Cepa de P. vivax) Sal-I Salvador I (Cepa de referencia de P. vivax) PE R-ficoeritrina APC Aloficocianina rpvgama GAMA de Plasmodium vivax recombinante RT-PCR Reacción en cadena de la polimerasa transcriptasa reversa. SDS-PAGE Electroforesis en Gel de Poliacrilamida con Dodecilsulfato Sódico. DS Desviación estándar DAPI DAPI: Diclorhidrato de 4',6-diamidino-2-fenilindol ADN Ácido desoxirribonucleico ARN Ácido ribonucleico ADNg ADN genómico ADNc ADN complementario
12 12 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
13 13 Introducción La malaria es una de las enfermedades infecciosas de mayor importancia en salud pública, debido a las altas tasas de mortalidad que genera, principalmente en países en vías de desarrollo. Es la principal enfermedad en humanos generada por protozoos y afecta severamente a individuos que viven en comunidades endémicas. Esta patología es causada en humanos por 5 especies del género Plasmodium: Plasmodium falciparum, Plasmodium vivax, Plasmodium malariae, Plasmodium ovale y Plasmodium knowlesi, de las cuales, P. falciparum y P. vivax, generan el 90% de los casos reportados en el mundo, lo que hace que su control sea prioritario en la agenda de salud internacional (1). Cerca de millones de personas se encuentran en riesgo de contraer la infección por las dos especies más prevalentes causantes de malaria (P. falciparum y P. vivax). P. vivax se caracteriza por presentar mayor distribución geográfica y es responsable de causar alta morbilidad principalmente en los continentes Asiático y Americano, donde el 91% de los individuos en riesgo viven en el centro y sureste de Asia y el 5,5% en América (2). Se ha estimado que P. vivax puede generar entre 70 a 390 millones de casos anualmente (3). Aunque se pensaba que P. vivax era una especie que causaba malaria benigna, recientes estudios muestran un incremento durante los últimos años de complicaciones clínicas y muertes causadas por esta especie, principalmente en la India, Indonesia y Pakistán (4, 5). A pesar de la implementación de los mecanismos de control establecidos por la Organización Mundial de la Salud (OMS) como el uso de pesticidas, el uso de mosquiteros impregnados con insecticidas y los fármacos antimaláricos, éstos no han sido suficientes para controlar o prevenir la enfermedad (1, 6). La malaria continúa expandiéndose en gran medida por la resistencia que han desarrollado los parásitos a los fármacos antimaláricos (7, 8) y los vectores a los insecticidas (9). Frente a este problema, varios grupos de investigación han
14 14 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax enfocado sus esfuerzos en desarrollar una vacuna, al considerarla una alternativa eficiente que ha demostrado ser útil en el control y erradicación de otras enfermedades infecciosas (10, 11). A pesar de la exhaustiva investigación, no existe en la actualidad una vacuna totalmente efectiva contra ninguna de las especies de Plasmodium causantes de malaria en humanos, con lo cual hay que continuar realizando estudios. Diversos trabajos han sugerido que las vacunas deben estar compuestas por aquellas proteínas o sus fragmentos que utilizan los agentes infecciosos para unirse a su célula diana (12). En este sentido, para el caso de la malaria, el conocimiento de las moléculas que expresa el parásito al final del ciclo de vida intra-eritrocítico, y en especial aquellas que interactúan con el glóbulo rojo, son las adecuadas para evaluar su utilidad como componentes de una vacuna. Actualmente, los esfuerzos para desarrollar una vacuna contra la malaria se han centrado principalmente en P. falciparum, debido a la disponibilidad de un cultivo in vitro estandarizado para este parásito, el cual ha permitido la identificación a gran escala de genes (13), transcritos (14) y proteínas (15, 16). Con base en la caracterización de estas moléculas, se ha podido avanzar en la determinación del rol que desempeñan algunas de ellas en el proceso de invasión de los merozoitos de P. falciparum a los eritrocitos; a la fecha, se han descrito varias moléculas tipo adhesinas localizadas en los micronemas que favorecen la adhesión e internalización del parásito a los eritrocitos tales como el antígeno apical de merozoito (AMA-1, del inglés Apical Merozoite Antigen 1), los antígenos de unión a eritrocito -175, -181, -160 y -140 (EBA, del inglés Erythrocyte Binding Antigen), el ligando de unión a eritrocitos 1 (EBL 1, del inglés Erythrocyte Binding Ligand 1), la proteína apical de merozoito relacionada a trombospondina (TRAMP, del inglés Thrombospondin-Related Apical Merozoite Protein) (17) y recientemente, el antígeno de micronemas anclado a GPI (GAMA, por sus siglas en inglés GPI-Anchored Micronemal Antigen) (18, 19) como también su homólogo en Toxoplasma gondii (20). Varias de estas moléculas son blanco de respuesta inmune y por ende, están siendo evaluadas como candidatas a vacuna en estudios clínicos (21). El antígeno de micronemas con anclaje a GPI de P. falciparum (PfGAMA) es una molécula de 738 aminoácidos que contiene una región rica en asparagina, un péptido señal y un anclaje tipo GPI. Los hallazgos muestran que el gen pfgama se transcribe y traduce para una proteína localizada en los micronemas de los esquizontes maduros del parásito (18, 19). PfGAMA posee una región ubicada al extremo C-terminal de la proteína que se une fuertemente a eritrocitos
15 15 de manera independiente a ácido siálico (18). La antigenicidad de la proteína, dada por la habilidad de desencadenar una respuesta inmune frente a la infección natural por P. falciparum, así como la inhibición de la invasión del parásito in vitro cuando se usan anticuerpos contra PfGAMA generados en conejo (18), ponen de manifiesto la importancia de caracterizar este antígeno en P. vivax. Desafortunadamente, el estudio de la biología de P. vivax ha estado retrasado por las complicaciones que se presentan al propagar el parásito in vitro (22, 23). Debido a esta dificultad, nuestro grupo de investigación ha tomado un enfoque bioinformático para identificar in silico proteínas candidatas a vacuna en P. vivax mediante análisis comparativos, teniendo en cuenta las proteínas descritas de otras especies de Plasmodium. En este sentido, la información proveniente de estudios de genómica (24), transcriptómica (25) y proteómica (26, 27) de P. vivax, es útil para el análisis a gran escala de la composición de genes, transcritos y proteínas del parásito, lo que junto con la adaptación de cepas de P. vivax a distintos primates (principalmente Saimiri y Aotus) (28) ha sido de gran ayuda para la caracterización de varias moléculas parasitarias. En el momento de iniciar esta investigación, se habían descrito tan solo 4 proteínas de micronemas: AMA-1, la proteína de unión a Duffy (DBP, por sus siglas en inglés Duffy Binding Protein) (29), la proteína de superficie de merozoito 5 (MSP-5, del inglés Merozoite Surface Protein-5) y TRAMP (23, 30). Además de ellas, se han descrito 12 moléculas que interactúan con los reticulocitos aparte de DBP como la proteína de unión a reticulocitos 1 (RBP-1, por sus siglas en inglés Reticulocyte Binding Protein-1), la cual hace parte de la ruta de invasión selectiva del parásito a dicho tipo de células (31), 10 antígenos ricos en triptófano (TRAg, del inglés Tryptophan Rich Antigen) (32, 33) y el antígeno del cuello de las roptrias 5 (RON5, del inglés Rhoptry Neck Antigen 5) (34). Este escenario muestra que la cantidad de moléculas de P. vivax que tienen un rol en adhesión celular es bajo comparado con la información disponible para P. falciparum, lo cual resalta que se requieren de estudios adicionales para suplementar el actual conocimiento de las proteínas tipo adhesinas que contiene el parásito. Basados en la importancia que tienen las proteínas de micronemas del parásito en el proceso de invasión y considerando las características reportadas para el antígeno PfGAMA, el presente estudio se centró en la identificación y caracterización del Antígeno de Micronemas con
16 16 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Anclaje a GPI en la cepa VCG-1 de P. vivax y en la determinación de la unión a reticulocitos humanos.
17 17 1. Hipótesis P. vivax tiene un gen homólogo a gama de P. falciparum, el cual se transcribe y traduce para una molécula que se localiza apicalmente en esquizontes maduros y se une a reticulocitos humanos. 2. Objetivos 2.1 Objetivo General Identificar y caracterizar el Antígeno de Micronemas con Anclaje a GPI en la cepa VCG-I de Plasmodium vivax (PvGAMA) y determinar la unión a los reticulocitos humanos. 2.2 Objetivos Específicos Evaluar la presencia del gen pvgama y su transcripción en esquizontes de la cepa VCG- I de P. vivax. Determinar la expresión de la proteína GAMA y su localización subcelular en parásitos maduros (esquizontes) de la cepa VCG-I de P. vivax. Evaluar la antigenicidad de la proteína PvGAMA. Estudiar la unión de PvGAMA recombinante a reticulocitos humanos.
18 18 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
19 19 3. Marco Teórico 3.1. La malaria La malaria es la enfermedad parasitaria más importante en el mundo, debido a que tiene un impacto global significativo en la salud humana y es prevalente en aproximadamente 100 países (Figura 3-1); cerca de 198 millones de casos se presentan anualmente cobrando la vida de personas, principalmente niños menores de 5 años y mujeres embarazadas (35, 36). El 85% de los casos y el 95% de las muertes ocurren en los países africanos. Se estima que la mitad de la población mundial (cerca de millones de personas) se encuentran en riesgo de contraer la infección (1). La enfermedad es causada por parásitos intracelulares obligados del género Plasmodium, los cuales hacen parte del filo Apicomplexa, orden Haemosporidia. Existen varias especies dentro del género que infectan reptiles, aves y mamíferos. Para el caso del humano, cinco especies son las responsables de causar la infección: P. falciparum, P. vivax, P. malariae, P. ovale y P. knowlesi. De estas cinco especies, P. falciparum y P. vivax son las más prevalentes a nivel mundial. A P. falciparum se le atribuyen las mayores tasas de mortalidad por su asociación con casos de malaria severa, causada por la adhesión de eritrocitos infectados a las células endoteliales. P. vivax posee la distribución geográfica más alta principalmente en zonas tropicales de Asia, Latinoamérica y el norte de África (37, 38). Aunque se pensaba que P. vivax era una especie causante de malaria benigna, estudios recientes muestran un incremento durante los últimos 15 años de complicaciones clínicas asociadas con disfunción hepática, renal, anemia severa y malaria cerebral (39, 40). Sin embargo, las interacciones hospedero-parásito que causan este tipo de disfunción multiorgánica por P. vivax son desconocidas (41). P. malariae y P. ovale son especies que causan manifestaciones leves de la enfermedad y es común encontrarlos en
20 20 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax coinfecciones con otras especies como P. falciparum o P. vivax (35). Por su parte, P. knowlesi es un parásito que causa malaria en simios e infecta humanos que habitan principalmente en áreas boscosas del sudeste Asiático en donde residen primates del género Macaca (38). Figura 3-1. Distribución global de la malaria causada por especies del género Plasmodium. Las áreas rojas representan zonas de alta trasmisión, las rosadas zonas de trasmisión moderada y las grises sin riesgo de trasmisión o muy bajo. Modificado de: En América, cerca de 112 millones de personas distribuidas en 21 países se encuentran en riesgo de contraer la malaria. P. falciparum es la especie responsable del 30% de los casos, mientras que el 70% restante es causado por P. vivax. Algunos países se encuentran en fases de pre-eliminación como: Ecuador, Republica dominicana, El Salvador, México y en fase de eliminación Argentina, Costa Rica y Paraguay. Sin embargo, países como Brasil, Colombia y Venezuela continúan presentado alta prevalencia, con un 77% de los casos reportados en América para el año 2013 (1). Colombia no es ajena a las infecciones por parásitos del género Plasmodium. Se estima que el 23% de la población se encuentra en riesgo de contraer esta enfermedad con un 15% en zonas de alta trasmisión y un 8% en zonas de baja trasmisión (1). El reporte de malaria para Colombia por la OMS para el año 2016, confirmó casos de malaria no complicada, de los cuales (40%) correspondieron a P. vivax, (60%)
21 21 a P. falciparum. Con respecto a la malaria complicada, se reportaron 18 muerte (42). Merece la pena resaltar que, en los últimos años, en Colombia se han venido reportando alrededor de casos de malaria anualmente, y de estos, el 58,2% de los casos son confirmados para P. vivax. Las regiones más afectadas debido a sus condiciones climáticas y geográficas son Antioquia, Chocó, Córdoba, Bolívar y Amazonas, donde se reporta el 90% de los casos en el país (1, 42) Ciclo de vida de Plasmodium El filo Apicomplexa está compuesto por un grupo de parásitos intracelulares obligados que infectan reptiles, aves y mamíferos, dentro de los cuales se encuentra el agente causal de la malaria, Plasmodium. Este parásito se caracteriza por poseer un motor actina-miosina y un conjunto de organelos ubicados en las zonas apicales que se conocen como roptrias y micronemas, además de los gránulos densos distribuidos en el citoplasma. Estos organelos, contienen proteínas que son secretadas por el parásito para llevar a cabo el proceso de invasión a la célula hospedera (43, 44). El ciclo de vida de Plasmodium spp comprende dos fases: la fase sexual en un hospedero invertebrado (hospedero definitivo) que se lleva a cabo en mosquitos hembra del género Anopheles y una fase asexual que ocurre en un hospedero vertebrado (hospedero intermediario) (Figura 3-2). En esta última fase se pueden diferenciar dos sub-fases, una hepática y otra intra-eritrocítica. Es importante destacar que durante el ciclo de vida, el parásito experimenta diversos cambios morfológicos en su desarrollo y además hay una expresión diferencial de proteínas que le van a facilitar la interacción con las células diana en el hospedero (45, 46). El ciclo inicia cuando un mosquito hembra del género Anopheles se alimenta del hospedero sano e inocula por medio de su probóscide, formas del parásito denominadas esporozoitos que se encuentran en sus glándulas salivales y alcanzan el tejido cutáneo para luego avanzar hasta torrente sanguíneo. Los esporozoitos viajan rápidamente por el torrente sanguíneo hasta llegar al hígado en un tiempo estimado de 2 a 60 minutos, penetran los sinusoides hepáticos a través de las células de Kupffer e invaden los hepatocitos. Estos parásitos pueden invadir varios hepatocitos hasta encontrar el adecuado para poder desarrollarse. En
22 22 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax aproximadamente 6 días, los esporozoitos se desarrollan hasta formar esquizontes hepáticos maduros, que al romperse liberan entre a merozoitos uninucleados que se encargan de invadir eritrocitos sanos (47). Algunas especies de parásito como P. vivax y P. ovale tienen la capacidad de generar formas latentes que se conocen como hipnozoitos, los cuales pueden persistir en el individuo infectado durante meses e incluso años y están asociados con las recaídas de la enfermedad (48). Los mecanismos por los cuales se activan estas formas latentes son desconocidos pero varias hipótesis han sido planteadas: la presencia de proteínas en la saliva del mosquito posiblemente reconocen los hipnozoitos y median su activación (49) o los picos febriles podrían estimular la activación de estas formas latentes durante la infección por otros patógenos como bacterias o virus en el individuo afectado (50). Una vez los merozoitos se distribuyen por el torrente sanguíneo, estos invaden los eritrocitos sanos lo que da inicio al estadio eritrocítico. Una vez dentro del eritrocito, el merozoito se desarrolla en aproximadamente dos días generando las formas de anillo, trofozoito y finalmente esquizonte maduro, cada uno de los cuales puede liberar en promedio 16 merozoitos infectivos. Alternativamente, los parásitos en la etapa eritrocítica pueden diferenciarse en gametocitos (microgametocitos y macrogametocitos), que son las formas sexuales del parásito (47). Al momento en que un nuevo mosquito se alimenta del individuo infectado, toma dichos gametocitos masculinos y femeninos, quienes se fusionan en su intestino para dar origen al cigoto. En aproximadamente 18 a 24 horas, el cigoto se transforma en un ooquinete que atraviesa la pared del intestino medio, pasando a través de las células epiteliales hasta llegar a la lámina de células basales, allí se diferencia en un oocisto, el cual sufre varios ciclos de reproducción asexual hasta convertirse en esporozoito; este proceso se lleva a cabo en 12 días hasta que los esporozoitos son liberados en la hemolinfa del mosquito y migran hacia las glándulas salivales, donde se convierten en infectivos para los seres humanos y de esta forma se continúa el ciclo de infección (35, 43).
23 23 Figura 3-2. Ciclo de vida de Plasmodium. En el ciclo de vida del parásito se diferencian dos fases, una fase sexual que se desarrolla en el hospedero invertebrado (mosquito) y otra fase asexual que se lleva a cabo en hospedero vertebrado (humanos). Modificado de: Fase eritrocítica Esta etapa del ciclo de vida del parásito es la más importante dado a que está relacionada con las manifestaciones clínicas de la enfermedad y su severidad. Cabe destacar que durante la liberación de los merozoitos al torrente sanguíneo, se desencadena una respuesta inmune por parte del hospedero, la cual va dirigida contra las proteínas parasitarias. Esta respuesta a menudo logra inhibir la invasión de parásitos a los glóbulos rojos y por ende se considera importante estudiar aquellas moléculas que están implicadas en el proceso invasivo para evaluarlas como componentes de una vacuna (51). Dado lo anterior, esta fase ha sido ampliamente estudiada principalmente en P. falciparum debido a que se tiene establecido un cultivo in vitro para la especie (52, 53). En el caso de P. vivax, el estudio de esta fase está retrasado por causa de las limitaciones de propagar continuamente el parásito in vitro dado su
24 24 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax tropismo por invadir glóbulos rojos inmaduros (reticulocitos) (22). En este sentido, nuestro grupo de investigación se ha centrado en estudiar proteínas del merozoito en P. vivax con ayuda de herramientas bioinformáticas y confirmación experimental usando técnicas de biología molecular (ver más adelante) El merozoito y organelos apicales Los merozoitos son células eucariotas que presentan un tamaño aproximado de 1,5 x 1,0 micras de diámetro y poseen orgánulos como núcleo, mitocondrias, ribosomas, retículo endoplásmico, complejo de Golgi, entre otros (Figura 3-3) (51). En la superficie, los merozoitos contienen una gran variedad de filamentos gruesos (complejos de proteínas de superficie) y delgados (proteínas individuales de superficie) que le facilitan el reconocimiento a los glóbulos rojos; estas proteínas son procesadas por proteasas del parásito a medida que éste se internaliza en la célula (54, 55). Una característica especial de estos microorganismos es la presencia de organelos propios del filo como micronemas y roptrias (localizados en el polo apical) y gránulos densos (distribuidos en el citoplasma); estos organelos son de gran importancia para el parásito ya que en ellos están contenidas un conjunto de proteínas que son secretadas durante el proceso de invasión a la célula diana (56). Las roptrias son organelos localizados en el polo apical del merozoito que tienen una longitud aproximada de 550 x 250 nm (57). Están compuestas por dos regiones distintas en la cuales se encuentran proteínas con diferentes roles en la invasión; una primera región es el ducto apical conocido como el cuello de las roptrias y una región basal rica en lípidos conocida como el bulbo (58). Las proteínas presentes en el cuello de las roptrias están involucradas principalmente en la adhesión parásito-célula diana antes y durante la invasión, y se ha demostrado que tienen orígenes evolutivos compartidos con proteínas de otros Apicomplexa (59, 60). Un ejemplo de las moléculas de P. falciparum presentes en esta región de las roptrias son las proteínas homologas a las de P. vivax que se unen a reticulocitos: Rh1, Rh2a, Rh2b, Rh4 y Rh5 (Rh, del inglés Reticulocyte-binding homologs) que son adhesinas involucradas en establecer la interacción entre el merozoito y el glóbulo rojo por medio del reconocimiento de receptores celulares (61).
25 25 Figura 3-3. Estructura del merozoito El merozoito es la forma del parásito involucrada en la invasión de glóbulos rojos o de la fase eritrocítica asociada con las manifestaciones clínicas de la malaria. En su estructura presenta organelos típicos de una célula eucariota como mitocondrias, núcleo y ribosomas pero además, organelos propios del filo como roptrias, micronemas y gránulos densos. Modificado de (43). Adicionalmente, el complejo de las proteínas del cuello de roptrias (RONs, del inglés Rhoptry neck proteins) tales como RON-2, -4 y -5 interactúan con el antígeno apical de membrana 1 (AMA-1, del inglés Apical Membrane Antigen 1) y ayudan a la formación de la unión fuerte entre el parásito y el eritrocito (62). Con respecto a las proteínas del bulbo de las roptrias, a diferencia de las presentes en el cuello, no comparten homología con proteínas de roptrias de otros Apicomplexa, lo que sugiere que son una adaptación de este organelo para invadir a su célula diana (63). Dos complejos de proteínas se han caracterizado en el bulbo de las roptrias de P. falciparum; las proteínas de las roptrias de bajo peso molecular -1, -2 y -3 (RAP, del inglés Rhoptry Associated Protein) y el complejo de alto peso molecular (RhopH1, -2 y -3, del inglés High molecular mass Rhoptry Protein) (64, 65). Sus roles en la invasión son importantes debido a que anticuerpos dirigidos a RAP-1 de P. falciparum bloquean parcialmente la invasión a glóbulos rojos in vitro (57). El rol inmunológico de una de estas proteínas se estudió en P. vivax; la inoculación de la proteína recombinante RAP-2 emulsificada con adyuvante de Freund
26 26 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax en monos Aotus, mostró una protección parcial de los animales, ya que disminuyó el porcentaje de parasitemia luego del reto experimental con la cepa VCG-1 de P. vivax (66). Los micronemas son organelos apicales del merozoito al igual que las roptrias, pero presentan un tamaño menor; tienen una longitud aproximada de 120 nm con apariencia de sacos (67). Estos organelos son importantes en el proceso de invasión debido a que contienen proteínas que están implicadas en la interacción inicial del parásito con receptores de superficie del glóbulo rojo (68). Dos familias de proteínas juegan un papel central en dicho proceso: los ligandos de unión a Duffy (DBL, del inglés Duffy Binding Ligand), los cuales tienen un dominio rico en cisteínas en la región II (69). Dentro de las DBL se encuentran las proteínas de unión a Duffy (DBPs, del inglés Duffy Binding Proteins) presentes en P. knowlesi (70) y P. vivax (29), los antígenos de unión a eritrocitos (EBA, del inglés Erythrocyte Binding Antigen) conformadas por EBA-140, -175 y 181 y el ligando de unión a eritrocitos-1 (EBL-1, del inglés Erythrocyte Binding Ligand-1) presente en P. falciparum (71). AMA-1, también presente en este organelo, está involucrada en la formación de la unión fuerte (tight junction) en complejo con algunas proteínas RONs (62). Otra familia de antígenos que hacen parte de los micronemas son las proteínas de unión a reticulocitos 1 y 2 (RBPs, del inglés Reticulocyte Binding Proteins), las cuales le confieren la especificidad a P. vivax de invadir reticulocitos; (31) (72). En P. vivax, solo se han descrito seis proteínas presentes en este organelo: DBP, AMA-1, MSP-5, PvTRAMP y RBP-1a, 1b. En 2011, Arumugam y colaboradores identificaron el antígeno PfGAMA, el cual es una proteína de 738 aminoácidos con una masa molecular aproximada de 85,24 kda, compuesta por un péptido señal en los residuos 1-24, una región rica en asparagina entre los residuos y un dominio transmembrana del aminoácido (Figura 3-4A). Dada la co-localización de GAMA con AMA-1 por inmunofluorescencia en P. falciparum, se sugirió que se encuentra en micronemas, lo cual se demostró posteriormente por microscopía inmunoelectrónica (Figura 3-4B). De particular interés, la región C-terminal de la proteína contiene un dominio que se une fuertemente a eritrocitos y además, anticuerpos dirigidos contra la proteína completa son capaces de inhibir la invasión del parásito in vitro hasta un 72% (Figura 3-4C). PfGAMA es expuesta al sistema inmune debido a que anticuerpos generados en una infección natural por P. falciparum reconocen la proteína recombinante por ELISA (Figura 3-4D) (18). Recientemente, Huynh y colaboradores identificaron el homólogo de PfGAMA en Toxoplasma
27 27 gondii como una proteína que se localiza en micronemas y se procesa en múltiples fragmentos. Además, la proteína se une a varios tipos celulares in vitro y parásitos Knockout para TgGAMA, muestran una reducción significativa en su capacidad para unirse e invadir fibroblastos humanos (20). Dada la evidencia funcional, se sugiere que éste es un antígeno a tener en cuenta a la hora de utilizarlo como componente de una vacuna. Los gránulos densos son vesículas membranosas de forma esférica con un tamaño aproximado de 80 nm de diámetro y se localizan en el citoplasma del merozoito en el espacio comprendido entre las roptrias y el núcleo. Después de la invasión a los glóbulos rojos, dichos gránulos se translocan a la superficie del merozoito y descargan su contenido por exocitosis dentro de la vacuola parasitófora, generando unas protuberancias que se extienden en el citoplasma de la célula invadida (73, 74). Varias proteínas presentes en los gránulos densos han sido descritas en P. falciparum, entre ellas, el antígeno de superficie del eritrocito infectado con anillos (RESA, del inglés Ring- Infected Erythrocyte Surface Antigen), el cual, una vez secretado, hace interacción con la espectrina del glóbulo rojo presumiblemente para protegerla y evitar cambios conformacionales que afecten la integridad de la membrana del eritrocito (74, 75). Otra proteína presente en gránulos densos es el antígeno de membrana de anillo (RIMA, del inglés Ring Membrane Antigen) que se expresa en estadios de esquizontes maduros y merozoitos libres, pero su función es desconocida (76). Por último, dos serin-proteasas tipo subtilisina hacen parte de las proteínas contenidas en los gránulos densos (SUB-1 y SUB-2, del inglés Subtilisine). La SUB-1 está asociada con el procesamiento de varias moléculas asociadas a la superficie del merozoito como la proteína de superficie de merozoito -1, -6 y -7 (MSP, del inglés Merozoite Surface Protein) a las cuales se les atribuye las interacciones de baja afinidad entre el merozoito y la célula (77). En P. vivax, a la fecha no se han descrito proteínas presentes en estos organelos.
28 28 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Figura 3-4. Identificación de GAMA en P. falciparum. En la imagen se muestra la identificación y caracterización de PfGAMA. A) Representación gráfica de la composición de la proteína, B) localización subcelular de PfGAMA en el parásito por IFI y microscopía inmunoelectrónica, C) ensayo de inhibición de la invasión in vitro del parásito a glóbulos rojos y D) antigenicidad de la proteína recombinante con sueros humanos por ELISA. Figura modificada de (18) Mecanismos y fases de la invasión La invasión de merozoitos de Plasmodium a los glóbulos rojos es un proceso complejo que involucra múltiples pasos y es probablemente similar para todas las especies del parásito (Figura 3-5) (78). El proceso se lleva a cabo en tres etapas importantes, las cuales son: unión reversible del merozoito a la superficie de los eritrocitos y reorientación apical, la formación de la unión fuerte e irreversible (conocida como tight junction (TJ) en inglés) y por último la formación de una vacuola parasitófora, que le facilita la entrada a la célula por invaginación de su membrana (79, 80). La unión inicial del merozoito al glóbulo rojo es reversible y de baja afinidad pero es un paso importante ya que el parásito reconoce eritrocitos competentes para poder invadirlos; cabe
29 29 destacar que este contacto puede ocurrir en cualquier parte de la superficie del merozoito (81). En este contacto inicial, están involucradas varias proteínas de superficie que se encuentran ancladas a la membrana vía glicosilfosfatidilinositol (GPI, del inglés Glycosylphosphatidylinositol) tales como MSP-1, -2, -4, -5, -8 y -10, mientras que otras como MSP-3, -6, -7 y -9 son solubles. En P. vivax sólo se han descrito MSP-1, -3, -7, -8, -9 y -10 (23). Las MSPs se caracterizan por tener uno o dos dominios relacionados con el factor de crecimiento epidérmico (EGF, del inglés Epidermal Growth Factor) que puede estar mediando interacciones proteína-proteína, pero su función exacta es desconocida (78, 82). Figura 3.5. Fases de la invasión al glóbulo rojo En esta figura se puede observar: 1) Contacto inicial de baja afinidad, 2) reorientación del polo apical y formación del tight junction, 3) secreción del contenido de micronemas y roptrias para formar la vacuola parasitófora, 4) invasión del merozoito mediado por la acción del motor actina-miosina, 5) internalización completa del parásito y liberación de adhesinas por proteasas del parásito, 6, 7 y 8) parásito desarrollándose en el eritrocito y alimentándose de la hemoglobina citoplasmática a través del citostoma. Figura modificada de: MSP-1 es el antígeno más abundante en la superficie del merozoito. La MSP-1 de P. falciparum se procesa en cuatro fragmentos proteolíticos que tienen una masa molecular de 83, 30, 38 y
30 30 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 42 kda, los cuales tienen la capacidad de hacer complejos no covalentes con otras proteínas como MSP-6, -7 y -9 para unirse a la proteína banda 3 de la superficie del eritrocito. Como consecuencia, este antígeno ha sido considerado como el determinante en el reconocimiento y unión inicial al eritrocito (83-85). Una vez hecho el contacto inicial entre el merozoito y el glóbulo rojo, el parásito se reorienta para disponer la maquinaria apical en la membrana del eritrocito. La proteína AMA-1, la cual es altamente conservada en el filo, está involucrada en la reorientación del parásito (86, 87); esto se ha comprobado dado que anticuerpos específicos contra PfAMA-1 no afectan la unión inicial del parásito, pero si bloquen la reorientación del merozoito (88). Si bien hacen falta varios estudios para elucidar completamente el rol de AMA-1 en la invasión del merozoito, se ha sugerido que esta proteína se requiere para establecer la interacción apical entre las adhesinas del cuello de las roptrias y los micronemas (43). Después de la reorientación del parásito se activa el proceso de invasión, el cual está mediado por la interacción directa de ligandos del polo apical del merozoito con receptores del eritrocito. El mecanismo preciso por el cual se da la liberación del contenido de las roptrias y micronemas es desconocido (89). En merozoitos de P. falciparum, el aumento intracelular en la concentración de iones calcio está relacionado con la liberación del contenido de las roptrias, la cual es crítica para la invasión (90). Con respecto a las proteínas de las roptrias, los mecanismos que subyacen detrás de su secreción son aún desconocidos. Las proteínas que se secretan de las roptrias y micronemas son de tipo adhesinas que reconocen receptores en el glóbulo rojo e invasinas que no tienen receptores específicos pero su papel es crítico para el proceso de invasión (91). Entre las adhesinas más importantes vinculadas con este proceso están las EBAs (localizadas en los micronemas), que incluyen EBA-140, -165, -175, -181 y EBL- 1, las cuales comparten un dominio rico en cisteínas en el N-terminal que les permite interactuar con sus receptores (43, 92). Además de ellas, se han descrito las Rhs (localizadas en roptrias) que incluyen Rh1, Rh2a, Rh2b, Rh4 y Rh5, las cuales se encargan de reconocer las células adecuadas para invadirlas mediante la unión a los receptores de membrana del eritrocito (93, 94). A la fecha, hay algunos receptores de la membrana del eritrocito identificados, a los cuales se unen moléculas de P. falciparum. Por ejemplo, la glicoforina C es el receptor de EBA-140, la glicoforina A interactúa con EBA-175, CR1 es el receptor de Rh4 y la basigina es el receptor de Rh5 (95).
31 31 En P. vivax, el proceso de invasión está mediado por la interacción específica de la proteína de unión a Duffy (PvDBP) y el antígeno Duffy presente en la superficie del eritrocito (DARC, del inglés Duffy Antigen Receptor for Chemokines). El antígeno Duffy está compuesto de 236 aminoácidos que conforman siete dominios transmembranales; su función en eritrocitos está relacionada con la unión a quimiocinas para impedir su difusión en órganos y espacios de tejidos periféricos, regulando así la inflamación (96). La proteína DBP es secretada de los micronemas de P. vivax y la unión al receptor Duffy está mediada por la región II de la proteína, que se caracteriza por la presencia de un dominio rico en cisteínas similar al encontrado en EBA-175 de P. falciparum (Figura 3-6) (97, 98). Adicionalmente, P. vivax expresa varias proteínas de unión a reticulocitos dentro de las cuales, PvRBP-1 y PvRBP-2 están implicadas en la especificidad por invadir los reticulocitos (31). Como los eritrocitos maduros también expresan el antígeno Duffy, se ha propuesto que las RBPs están involucradas en la unión inicial y reorientación del merozoito, de manera que censan la célula adecuada (los reticulocitos en el caso de P. vivax) antes de que se produzca la interacción entre DBP-DARC y la formación de la unión estrecha (31). PvRBP-1 y PvRBP-2 se expresan en el polo apical del merozoito, tienen una masa molecular de 325 y 330 kda, respectivamente, poseen dominios transmembranales localizados en el extremo carboxilo terminal y dominos citoplasmáticos cortos. Además, PvRBP-1 es una proteína dimérica que hace complejo con PvRBP-2 a través de interacciones no covalentes. A la fecha, los receptores presentes en los reticulocitos a los cuales se unen las RPBs son desconocidos (78). AMA-1 como se mencionó anteriormente está asociada a la reorientación del merozoito y se relaciona con la posterior descarga de proteínas de los organelos apicales (99). También juega un papel crítico en la formación de la unión estrecha, debido a que es translocada a la membrana del merozoito donde actúa como ligando de unión a los eritrocitos (99, 100). Los receptores de la membrana a los cuales se une AMA-1 es un complejo formado por las propias proteínas del parasito denominadas RON2, -4 y -5 en P. falciparum, mientras que para P. vivax dicha interacción es aún desconocida. Esta interacción entre la proteína AMA-1 con el complejo RONs es importante, ya que se ha demostrado que en ausencia de éstas, la invasión del parásito a su célula diana es inhibida (101, 102). Por último, luego de la secreción de las proteínas de micronemas y roptrias, se inicia la internalización del parásito gracias a que la unión estrecha se torna móvil por la acción del
32 32 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax motor actina-miosina. El contenido del bulbo de las roptrias (lípidos y proteínas) participa en la formación de la vacuola parasitófora, mientras que algunas proteasas ayudan a liberar las adhesinas del parásito (56, 77). Una vez la maquinaria del parásito se encuentra en su parte posterior, se cierra la vacuola parasitófora y se inicia el desarrollo asexual dentro del glóbulo rojo para producir merozoitos infectivos (43). Figura 3-6. Fases de la invasión a reticulocitos por P. vivax. La imagen muestra las etapas de la invasión de P. vivax a reticulocitos. 1) Contacto inicial del parásito en cualquier parte del merozoito. 2) Reorientación del merozoito para disponer el polo apical en la membrana del reticulocito (células con fenotipo Duffy positivo). 3) Formación del tight junction mediado por el complejo AMA-1-RONs y secreción del contenido de roptrias y micronemas. 4) Invasión del merozoito y formación de la vacuola parasitófora. 5) Parásito internalizado y cierre de la membrana del reticulocito. Figura modificada de (96).
33 Candidatos a vacuna en P. vivax A diferencia de P. falciparum, en P. vivax la identificación y caracterización de candidatos a vacuna ha estado retrasada por la dificultad de desarrollar un sistema de cultivo in vitro para esta especie de parásito, dado principalmente por su preferencia de invadir reticulocitos (103). Sin embargo, gracias a la adaptación de cepas de P. vivax en distintos primates y a la información disponible de los estudios de genómica (24), transcriptómica (25), proteómica (26, 27, 104) e inmunómica (105) de la especie, se han identificado varias moléculas del parásito que posiblemente presentan un potencial rol en el proceso de invasión. El análisis de candidatos a vacuna en P. falciparum ha permitido encontrar varias características que se deben tener en cuenta a la hora de seleccionar una proteína para ser incluida en estudios de caracterización como: 1) el gen codificante de la proteína debe transcribirse y expresarse al final del ciclo de vida intraeritrocítico, 2) la proteína debe al menos presentar una secuencia señal de secreción, tener dominios de unión o secuencias transmembrana o de anclaje a GPI, 3) la molécula debe ser capaz de generar una respuesta inmune en infecciones naturales o experimentales, 4) anticuerpos dirigidos contra la proteína deben inhibir la invasión del parásito a la célula y 5) que no sea posible generar parásitos knock out para el gen que codifica el antígeno, lo que implica que la proteína producto es esencial (106). En P. vivax se han descrito mediante herramientas bioinformáticas y confirmación experimental por biología molecular, varias proteínas que son homologas a las de P. falciparum y que están implicadas en la invasión celular. Algunas de dichas moléculas presentan un patrón de localización de superficie tales como PvMSP-7, -8, -10, Pv41 y Pv12, mientras que otras tienen patrón de localización apical como Pv34, Pv38, PvTRAMP y PvClag7 (23). Algunos de esos antígenos como Pv34 inducen la proliferación de células mononucleares de sangre periférica aisladas de individuos con antecedentes de malaria por P. vivax. Otros son blancos de respuesta inmune como PvTRAMP y PvClag7 durante la infección natural por la misma especie de parásito, indicando la importancia de usarlos para el desarrollo de una vacuna contra P. vivax. Por ende, se requiere confirmar su inmunogenicidad y realizar ensayos de protección en modelos experimentales como monos Aotus (Figura 3-7) (23).
34 34 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Figura 3-7. Candidatos identificados en P. falciparum y P. vivax. En la figura se presentan los candidatos a vacunas identificados en merozoitos de P. falciparum y P. vivax hasta el Modificado a partir de (23). Recientemente, se evaluó la actividad de unión de 36 proteínas ricas en triptófano en P. vivax que pertenecen a la familia Pv-fam-a; diez de ellas presentan capacidad de unión a reticulocitos: PvTRAg35.2, PvTRAg38, PvTRAg36, PvTRAg34, PvTRAg, PvTRAg26.3, PvTRAg36.6, PvTRAg33.5, PvTRAg74 y PvTRAg69.4 (32, 33). Aunque sus funciones no se conocen con certeza, parece que interactúan con espectrina en los glóbulos rojos. Esta interacción puede estar mediando funciones biológicas importantes para el parásito como en P. falciparum tales como: transporte de proteínas, salida del merozoito y modulación del citoesqueleto de la célula roja, presumiblemente para proporcionar rigidez en la membrana del reticulocito infectado (107, 108). El antígeno PvTRAg38 tiene la capacidad de unirse al receptor de eritrocitos banda 3 por medio de la interacción específica con los ectodominios 1, 3 y 6. Además, la proteína es capaz de inhibir el crecimiento in vitro de P. falciparum, probablemente por competir con ligandos de esta especie de parásito por el receptor banda 3 (109). Otro antígeno descrito recientemente en P. vivax es la proteína del cuello de las roptrias 5 (RON-5), la cual fue expresada en E. coli para obtenerla de forma recombinante y se evaluó su capacidad de unión a células de cordón umbilical por ensayos de inmunoprecipitación e inmunofluorescencia. En estos ensayos, se observó que la proteína se une preferentemente a reticulocitos marcados con anticuerpos anti-cd71 (receptor de transferrina que se expresa en
35 35 fases tempranas del desarrollo eritroide), lo que soporta que PvRON-5 presumiblemente está involucrada en el proceso de adhesión del merozoito a glóbulos rojos inmaduros (34) Vacunas contra la malaria La complejidad del ciclo de vida de las especies de Plasmodium y los mecanismos involucrados en la evasión de la respuesta inmune, continúan siendo el mayor obstáculo para el desarrollo de una vacuna totalmente efectiva (35). Algunas vacunas están siendo ensayadas actualmente en ensayos clínicos, principalmente contra P. falciparum. Entre ellas se destaca la RTS,S, la cual está en ensayos clínicos de fase 3 y está basada en la región C- terminal de la proteína circumsporozoito (CSP, del inglés Circumsporozoite Protein). Sin embargo, la protección de esta vacuna en niños contra la malaria clínica es de 46% y del 27% en neonatos, después de 18 meses de la vacunación (110, 111). Un estudio reciente mostró, que la protección con la vacuna en neonatos con malaria severa es cercana al 17% con una dosis de refuerzo y un 10% en aquellos infantes que no recibieron refuerzo, después de 38 meses de vacunación (112). Otras vacunas también se encuentran en ensayos clínicos y están basadas en antígenos de esporozoitos como TRAP (del inglés, Thrombospondin-Related Adhesive Protein) (113) y CelTOS (del inglés, Cell-Traversal protein for Ookinetes and Sporozoites) (114). Con respecto a vacunas de la fase eritrocítica se han probado proteínas del merozoito como MSP-1 y AMA-1 (115, 116). Dentro de las vacunas que contienen componentes tanto de la fase pre-eritrocítica como de la intra-eritrocítica, se destaca la SPf66, un polipéptido sintético que mostró eficacia protectiva alrededor del 40% en diversos estudios en América Latina y África cuando se inmunizaban niños mayores de 1 año (117, 118). En contraste con las 12 vacunas en ensayos clínicos para P. falciparum, existen en la actualidad solo dos vacunas en fase clínica 1 y 2 para P. vivax (35). Una primera vacuna denominada proteína 001 de la malaria por P. vivax (VMP001, del inglés P. vivax Malaria Protein 001) que incorpora la región amino y carboxilo terminal de la proteína CSP y una región repetitiva que contiene secuencias de las dos formas alélicas de CSP expresadas en E. coli. La proteína quimérica ha sido evaluada en ensayos pre-clínicos en monos Aotus nancymaae e induce inmunidad protectiva en el 66% de los animales ante el reto experimental con esporozoitos de P. vivax (119). Sin embargo, los datos de protección de los ensayos clínicos de fase 1 y 2 para esta vacuna formulada en adyuvante AS01B aún no están disponibles (35). La segunda vacuna,
36 36 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax en fase 1 de ensayos clínicos está compuesta por la proteína DBP, la cual es crítica en la invasión gracias a su interacción con el antígeno Duffy de los glóbulos rojos. En esta vacuna, la proteína ha sido incorporada a vectores virales, generados del adenovirus o virus de Ankara y de igual manera que con VMP001, no hay datos disponibles de protección (29, 35). Lo anterior pone en manifiesto la importancia de identificar y caracterizar antígenos de Plasmodium debido a que la inmunidad contra la malaria requiere del bloqueo de todas las moléculas que participan en la invasión. Por ejemplo, en P. falciparum se han encontrado más de 50 proteínas que se unen a la célula diana. En P. vivax, el conocimiento de proteínas de micronemas es escaso y a la fecha solo se han identificado seis proteínas presentes en este orgánulo, por lo que se hace necesario continuar con el estudio de dichas moléculas, principalmente aquellas que presentan función de unión a sus células diana. Por ende y tomando en cuenta la evidencia del antígeno PfGAMA y su posible rol en la invasión del merozoito a eritrocitos y la importancia que representa la identificación de nuevos antígenos para el conocimiento de la biología del parásito P. vivax, en este trabajo se propone la identificación y caracterización del Antígeno de Micronemas con Anclaje a GPI en la cepa VCG- 1 de P. vivax y determinar su habilidad de unión a los reticulocitos humanos.
37 37 4. Materiales y métodos 4.1. Tipo de investigación Este estudio es de tipo descriptivo experimental Análisis bioinformático, diseño de cebadores y péptidos sintéticos. La información publicada en los estudios de proteoma de P. vivax (27, 104, 120), se empleó para analizar in silico, proteínas potenciales candidatas a vacuna, teniendo en cuenta los siguientes criterios: expresión dominante de los genes codificantes mayor a 35 horas postinvasión según el estudio del transcriptoma del ciclo de vida intraeritrocítico de P. vivax (requerido) (25), 2) predicción positiva usando los programas SignalP 4.0 (121), BaCelLo (122), FragAnchor (123) y la base de datos InterPro (124) de una secuencia señal de secreción (requerido), localización extracelular (requerido), secuencias de anclaje a GPI o la presencia de dominios putativos sobre la secuencia de aminoácidos, respectivamente, y 3) evidencia previa del rol en la invasión o adhesión celular determinado experimentalmente en otras especies de Plasmodium (requerido). La secuencia del gen codificante de la proteína seleccionada se obtuvo de la base de datos PlasmoDB (125) y luego se usó para el diseño de cebadores específicos en el programa Gene Runner versión 3,05 (Tabla 4-1), al igual que epítopes de células B prediciendo los valores promedios más altos de hidrofilicidad, accesibilidad al solvente y Antigenicidad de Parker con el software ANTHEPROT (Tabla 4-2) (126).
38 38 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 4.3. Aislamiento y enriquecimiento de esquizontes de P. vivax La cepa del parásito VCG-I (Vivax Colombia Guaviare-I) se propagó hace seis años y se usó como fuente de material biológico (descrito más adelante). Brevemente, un mono Aotus mantenido en la estación de primates de FIDIC fue inoculado con 2.5x10 6 reticulocitos infectados con P. vivax, de acuerdo a una metodología previamente descrita (28). El mono fue supervisado continuamente por un primatólogo, quien monitoreo diariamente el progreso de la infección durante los días del estudio (aproximadamente 18 días), mediante tinción con naranja de acridina. Por último, el primate fue tratado con dosis pediátricas de cloroquina (10 mg/kg en el día primero y 7.5 mg/kg al quinto día de tratamiento) y primaquina (0.25 mg/kg en el tercer día de tratamiento) con la finalidad de eliminar por completo los parásitos en sangre. Una vez finalizado el estudio, CorpoAmazonía supervisó el retorno de los animales a su hábitat natural. Formas maduras del parásito, principalmente esquizontes, se enriquecieron a partir de 3 ml de sangre del mono Aotus infectado, usando un gradiente discontinuo de Percoll (GE Healthcare, Uppsala, Sweden) (127). El parásito fue obtenido de estas muestras enriquecidas con esquizontes por incubación durante 5 min en buffer de lisis que contiene 0.02 mm de saponina, 7 mm de K 2HPO 4, 1 mm de NaH 2PO 4, 11 mm de NaHCO 3, 58 mm de KCl, 56 mm de NaCl, 1 mm de MgCl 2, 14 mm de glucosa, ph 7.5 y posteriormente, la muestra fue lavada varias veces con PBS ph 7.0. Los parásitos se usaron como fuente de ARN, ADN genómico (ADNg) y proteínas totales en los procedimientos que siguen a continuación Extracción de material biológico El ARN total se extrajo de la muestra enriquecida con esquizontes usando el método del Trizol y tratado con DNasa RQ1 libre de RNasa (Promega, Wisconsin, USA), de acuerdo con las instrucciones del fabricante. Brevemente, se usó 1 µg de ARN para sintetizar el ADN complementario (ADNc) por RT-PCR, con la enzima SuperScript III enzyme (RT+) (Invitrogen, California, USA). La síntesis se realizó con las siguientes condiciones: 65 C por 5 min, 50 C por 1 hora y 70 C por 15 min. Una reacción adicional sin la enzima SuperScript III se usó como control negativo (RT-). Los productos sintetizados se trataron con RNasa por 15min (Promega, USA). Por otra parte, el ADN genómico (ADNg) se extrajo con el kit Wizard Genomic purification (Promega, USA). Por último, para extraer las proteínas totales, el parásito se
39 39 homogenizó en buffer de lisis que contiene 5% de SDS, 10 mm de PMSF, 10 mm de iodoacetamida y 1 mm de EDTA; el lisado se recuperó por centrifugación a rpm por 5 minutos. Las proteínas de la fracción soluble obtenidas, se cuantificaron usando el kit BCA protein assay (Thermo Scientific). El ARN, ADNc, ADNg y proteínas totales del parásito, se almacenaron a -70 C hasta su uso Clonación y secuenciación Las muestras de ADNg, ADNc RT (+) y RT (-), se usaron como plantilla en reacciones de PCR de 25 µl que contienen: 1X de la enzima KAPA HiFi HotStart ReadyMix 2X (KAPABiosystems), 0.3 µm de los cebadores y agua ultrapura hasta completar el volumen de reacción. Los cebadores específicos previamente diseñados, se utilizaron para amplificar el gen completo de pvgama en P. vivax o fragmentos de la molécula según un análisis de polimorfismo realizado (Tabla 4-1) el cual se describe a continuación. Fragmento $Secuencia del primer (5 3 ) TM Tamaño del producto (pb) Masa molecular (kda) pvgama* Dir: ATGAAGTGCAACGCCTCC Rev: AAAAATGAATAGGAGCAACG pvgama -Nt Dir: ATACGGAATGGAAACAACC Rev: AGTCGGTTCGTTATTCTCG 58 ºC pvgama -Ct Dir: CTGCTCAAGAACACGAAC Rev: GCTTCCACTCTGCAATTC pvgama RC-1 Dir: GACGATCATCTGTGTTCAAAAA Rev: GACCTCATTTTTGGACTTCTC Dir: GGCGCCTTCCTGCAGTC pvgama RV Rev: CATTAACATGGTGTTGTCGCT 60 ºC Dir: CAGGCGGCCATCTTACTAA pvgama RC Rev: GCTCCCGTTGACGCCCTT pvgama RV-2 Dir: GCCGCAAACGCAGACGCC Rev: GTTTGCCGAGAAGCTTCCAC Tabla 4-1. Fragmentos recombinantes de GAMA. Nt y Ct: región amino y carboxilo terminal; RC: región conservada; RV: región variable; Dir: Directo; Rev: Reverso; $: El codón de inicio para la expresión de la proteína se incluyó en todos los cebadores directos en el extremo 5 ; TM: temperatura de fusión; pb: pares de bases; kda: kilodaltons; *: indica que la PCR se usó para obtener solo la secuencia del gen completo.
40 40 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax La PCR para amplificar el gen completo o los fragmentos de pvgama empezó con un paso de denaturación a 95 C por 5 min, seguido por 35 ciclos a 98 C por 20 s, 58 C por 15 s y 72 C por 1.5 min de acuerdo con el tamaño del producto; finalmente, un paso de extensión final a 72 C por 5 min. Para purificar los productos de PCR a partir de RT+ y ADNg, se usó el kit Wizard PCR Clean-Up System (Promega, USA) y se evaluó la calidad de los amplicones por geles de agarosa. Los productos purificados se ligaron al vector de expresión pexp-ct/topo (Invitrogen) o al vector de clonación pgem (para el gen completo de la proteína) y se transformaron en células de E. coli TOP10 (Invitrogen). Varios clones recombinantes se crecieron para hacer la extracción de ADN plasmídico con UltraClean mini plasmid prep purification Kit (MO BIO laboratories, California, USA). La integridad del inserto y su orientación correcta, se confirmó por secuenciación usando el método de Sanger (MACROGEN, Seoul, South Korea). La comparación entre las secuencias del gen gama de la cepa de referencia Sal-I y la cepa VCG-I adaptada a primates se hizo usando el software ClustalW NPS@ (128). La secuencia de nucleótidos y aminoácidos de la cepa VCG-I se reportó en la base de datos GenBank bajo el código de acceso KT Análisis de polimorfismo de PvGAMA Los métodos evolutivos comparan la tasa de mutaciones no sinónimas (dn, mutaciones que alteran la secuencia de proteínas) con la tasa de mutaciones sinónimas (ds, las que codifican el mismo aminoácido), en la búsqueda de señales de selección natural. Las mutaciones deletéreas suelen ser removidas de las poblaciones por selección natural negativa (dn<ds o ω < 1). Las regiones que experimentan este tipo de selección pueden tener importancia funcional/estructural, manteniendo alta conservación de secuencias entre especies (129). Por otro lado, las mutaciones que tienen una ventaja adaptativa (un papel beneficioso) se fijan en una población por selección natural positiva (dn > ds or ω > 1). Teniendo en cuenta lo anterior, se predijeron las regiones funcionales de pvgama mediante enfoques evolutivos (130). Secuencias del gen pvgama de 6 cepas de P. vivax (VCG-I, Sal-I, Brasil-I, India-VII, Mauritania-I y Korea del Norte (131)) y 5 especies filogenéticamente relacionadas (P. cynomolgi, P. inui, P. fragile, P. knowlesi y P. coatneyi) (132) se obtuvieron por tblastn (excepto para VCG-I) usando la base de datos NCBI, para evaluar la diversidad genética y las fuerzas evolutivas en relación con GAMA. El algoritmo MUSCLE (133) se usó para alinear la secuencias y los alineamientos se corrigieron manualmente. La diversidad de nucleótidos por sitio (π) se estimó a partir de las
41 41 secuencias de P. vivax y el método de Nei-Gojobori modificado (134) se usó para evaluar la señales de selección natural mediante el cálculos de la diferencia entre las tasas de sustituciones no sinónimas y sinónimas (dn-ds). La selección natural también se evaluó mediante la estimación de la diferencia entre las tasas de divergencia sinónima y no sinónima (KN-KS), utilizando secuencias de P. vivax y especies relacionadas con el método de Nei- Gojobori modificado y la corrección Jukes-Cantor (135). Los codones específicos bajo selección natural entre las especies, se identificaron usando métodos de probabilidad Bayesiana y de máxima verosimilitud (SLAC, FEL, REL (136), MEME (137) y FUBAR (138)), seguido por el método GARD para evaluar eventos de recombinación (139). Los métodos basados en codones estiman la tasa evolutiva (ω) en cada codón usando una prueba estadística, para determinar si ω es significativamente diferente a 1 (evolución neutral). El algoritmo Branch-site REL (140) se utilizó para identificar linajes bajo selección positiva episódica (selección ocasionalmente con períodos transitorios de evolución adaptativa enmascarados por la selección negativa o evolución neutral). El servidor web Datamonkey se utilizó para realizar estos análisis (141) Expresión y purificación de los fragmentos recombinantes Los plásmidos recombinantes pexp-pvgama una vez confirmados por secuenciación, se transformaron en células de E. coli BL21-DE3 (Invitrogen), siguiendo las recomendaciones del fabricante. Las células se crecieron toda la noche a 37 C en agitación constante y luego se inocularon en medio Luria Bertani (LB) suplementado con 0.1 mg/ml de ampicilina. Al día siguiente, se escaló la expresión a 1L de LB bajo las mismas condiciones de crecimiento. Una vez las células alcanzaron una DO 600 de 0.5, los cultivos se incubaron en hielo por 30 min y luego se adicionó 1mM de IPTG para inducir la expresión de la molécula por 16h a temperatura ambiente y con agitación a 200 rpm. Las bacterias se recuperaron por centrifugación a rpm por 20 min y el pellet celular se usó para realizar extracción de proteínas en condiciones nativas y denaturantes Extracción denaturante El pellet celular correspondiente a la expresión de los fragmentos PvGAMA-Nt y Ct se homogenizó en buffer de extracción denaturante (6 M de urea, 20 mm de imidazol, 10 mm de
42 42 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Tris-Cl, 100 mm de NaH 2PO 4), suplementado con un cóctel de inhibidores de proteasas SIGMAFAST (Sigma Aldrich) y luego lisado con 0.1 mg/ml de lisozima toda la noche a 4 C y agitación a 10 rpm. La recuperación del sobrenadante se logró por centrifugación a rpm por 30 min Extracción en condiciones no detaturantes PvGAMA Región conservada 1 (RC-1), Región variable 1 (RV-1), Región conservada 2 (RC-2) y Región variable 2 (RV-2) se extrajeron usando un nuevo método para obtener moléculas en su forma soluble, igual que para el control positivo y negativo (DBP región II y DBP región III/IV, respectivamente). En resumen, el pellet celular se congeló/descongeló durante tres ciclos y se homogenizó en buffer de extracción nativa que contiene (50 mm de Tris-Cl, 300 de mm NaCl, 25mM de Imidazol, 0.1 mm de EGTA y 0,25% Tween-20, ph 8.0). La mezcla se incubó por 1 hora a 4 C y agitación a 10 rpm. El sobrenadante se recuperó por centrifugación a rpm por 30 min Purificación de las proteínas Los sobrenadantes derivados del lisado total de las células se incubaron con la resina Ni 2+ -NTA (Qiagen, California, USA) toda la noche a 4 C para purificarlos por cromatografía de afinidad en fase sólida, previo a la verificación de la expresión de las proteínas por Western blot. La mezcla de toda la noche se vertió en una columna para cromatografía y luego se lavó con 20 ml de buffer que contiene 0.1% de Triton X-114, seguido de 50 ml del mismo buffer sin detergente, para eliminar las proteínas débilmente unidas a la resina (contaminantes). Las proteínas extraídas bajo condiciones denaturantes se dializaron en la columna a través de varios pases en buffer urea en concentraciones decrecientes (6M, 3M, 1.5M, 0.75M y PBS). Luego, la elución de las proteínas se hizo con PBS que contenía concentraciones crecientes de imidazol (50, 100, 250 y 500 mm) en 2 ml cada una. Todas las fracciones colectadas se analizaron por SDS-PAGE al 12% y Western blot, aquellas que presentaron una sola banda se dializaron en PBS ph 7.2. Las proteínas ultrafiltradas y concentradas con Amicom Ultra-4 centrifugal filters (Merck Millipore) se cuantificaron con el kit micro BCA protein assay (Thermo scientific).
43 Obtención de anticuerpos policlonales Cuatro péptidos de 20 aminoácidos de longitud de PvGAMA de la cepa VCG-I de P. vivax, predichos como epítopes de células B (Tabla 4-2), y un péptido de 6 histidinas, se sintetizaron de acuerdo a una metodología previamente establecida por el grupo de química de nuestro instituto (142). Número Posición (aa) Sequence * APR HID AS Reactividad CG-RTRNILKKINEKSKNEVLN-GC 18,4 5, CG-LREIEQNRKALKNSKGTLSV-GC 27,2 4,6 174, CG-VTQGDVSVDEKGDRATTAGG-GC 44,2 4,3 129, CG-DLNAATEEDDLINEFSSRYN-GC 15,8 2,1 130,4 +++ Tabla 4-2. Epítopes lineales de células B diseñados de acuerdo al análisis de ANTHEPROT. *: residuos CG se añadieron en el extremo de cada péptido para permitir su polimerización; APR: antigenicidad de Parker; HID: hidrofibicidad; AS: accesibilidad al solvente; aa: aminoácidos; (- ó +): describe la reactividad del suero contra PvGAMA por Western blot. Los péptidos polimerizados se liofilizaron y caracterizaron por RP-HPLC y MALDI-TOF MS. 500 µg de cada péptido emulsificados con adyuvante completo de Freund (FCA del inglés Freund s complete adyuvant) (Sigma, Missouri, USA) se inocularon en conejos de raza Nueva Zelanda en el día cero, mientras que dosis iguales de cada péptido se formularon con adyuvante incompleto de Freund e inocularon en los días 21 y 42. Se colectaron sueros antes de la primera inmunización (suero pre-inmune) y 20 días después de la tercera dosis (suero post-tercera). Anticuerpos específicos dirigidos contra el péptido se purificaron por cromatografía de afinidad en una columna de Sefarosa CNBr 4B (Amersham, Biosciences AB), dado que fue el péptido que mostró mayor reactividad contra la proteína en comparación con los otros (Tabla 2). Brevemente, 5 µmoles de péptido se diluyeron en buffer de acople (0.1 M de NaHCO 3, 0.5 M de NaCl, ph 8.3) y luego se incubaron 16 horas a 4 C con resina de Sefarosa. Después de varios lavados para eliminar el exceso de ligando con 5 volúmenes de buffer de acople, los grupos libres de la resina se bloquearon con buffer Tris-HCl 0.1 M por dos horas sin agitación a temperatura ambiente y finalmente, la resina se lavó tres veces con soluciones de diferentes ph (0.1 M de buffer acetato con 0.5 M de NaCl, ph 4.0 y 0.1 M de Tris-HCl con 0.5 M de NaCl, ph 8.0). Cinco ml de suero de conejo (inmunizados con el péptido 39472) diluido en relación 1:1 con buffer de acople, se pasaron por la resina después de haber sido homogenizada en PBS. Los anticuerpos no unidos se lavaron con 10 ml de buffer de acople mientras que los
44 44 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax anticuerpos fuertemente unidos se eluyeron con 1 ml de buffer de elución en ph decreciente (0.1M de Glicina ph 7.0, 6.0, 5.0, 3.9 y 2.9) y se neutralizaron con 1mM de Tris ph 8.0 en relación 1:9 (buffer elución:buffer neutralización). Los anticuerpos se incubaron con 45% de sulfato de amonio por 1h en hielo con agitación constante y luego por 16h a 4 C sin agitación. Después de centrifugar a rpm por 15 min, el pellet se homogenizó en 100 µl de PBS y la muestra se dializó y almacenó a -20 C hasta su uso Western blot de las proteínas recombinantes y del parásito Cada rpvgama purificada (5 µg) y las proteínas totales del parásito (50 µg) se separaron en SDS-PAGE al 12% y luego se transfirieron a una membrana de nitrocelulosa. Después del bloqueo con 5% de leche descremada en TBS-0.05% Tween por una hora a temperatura ambiente (TA), cada membrana se cortó en tiras que se incubaron durante una hora con sueros pre-inmune e hiper-inmune anti-pvgama en dilución 1:100. Seguido a tres lavados, las tiras se incubaron nuevamente durante 1h (TA) con un anticuerpo producido en cabra anti-conejo acoplado a fosfatasa alcalina (9503F, ICN) como anticuerpo secundario en dilución 1: Una de las tiras se incubó con un anticuerpo monoclonal anti-histidinas (A7058, Sigma) acoplado a peroxidasa (1:4.500) como control positivo que reconoce la secuencia de histidinas de cada recombinante. Los blots se revelaron con el kit de sustrato peroxidasa (Vector Laboratories, Burlingame, Canadá) o de fosfatasa (BCIP/NBT) (Promega, USA) de acuerdo con las recomendaciones del fabricante. Las masas moleculares para las bandas esperadas se determinaron por regresión lineal usando el marcador de peso XL-OptiProtein (New England Biolabs) como referencia de masa molecular Localización de la proteína por Inmunofluorescencia indirecta (IFI). Reticulocitos de monos Aotus infectados con la cepa de parásito VCG-I de P. vivax se lavaron tres veces con PBS y luego se diluyeron hasta obtener de 5 a 7 esquizontes por campo con tinción por naranja de acridina. Posteriormente, las células se fijaron y permeabilizaron en un portaobjeto de vidrio de ocho pocillos (Biomedicals, Inc), durante 5 min a TA con formaldehído al 4% (v/v) y con 0.1% (v/v) de Tritón X-100 preparados en PBS, respectivamente.
45 45 Posteriormente, se procedió al bloqueo de los portaobjetos con una solución de BSA 5% en PBS 0.1% - Tritón X-100 a TA durante 1 hora. Seguido, los portaobjetos se incubaron con sueros de conejo en dilución 1:30 y luego con anticuerpo secundario anti-conejo Alexa Fluor 633 producido en cabra (Life Technologies); tanto los anticuerpos primarios como secundarios, se incubaron a TA durante 1 hora. Los núcleos de los parásitos se tiñeron con DAPI (0.5 mg/ml) durante 10 min a TA y luego se hicieron varios lavados con PBS para eliminar el exceso de reactivo. Por último, los portaobjetos se observaron en un microscopio de fluorescencia (Olympus BX51) con aceite de inmersión en un aumento de 100X Colección y procesamiento de muestras de sangre Individuos con antecedentes clínicos de malaria por P. vivax (37 pacientes) y P. falciparum (30 pacientes), entre 18 y 50 años de edad y que viven en áreas endémicas de malaria en Colombia (Chocó, Nariño, Córdoba, Vichada y Guaviare) se seleccionaron para este estudio. Los sueros de individuos sanos (16 pacientes) que nunca han sido afectados por la enfermedad y que viven en zonas no endémicas, se utilizaron como controles negativos. Las muestras de sangre colectadas en tubos Vacutainer BD sin anticoagulante por el personal de FIDIC, desde el mes Octubre de 2006 a Marzo de 2011 (para P. vivax) y el mes de Junio a Octubre de 1993 (para P. falciparum), se almacenaron a 4 C para su transporte a Bogotá y procesamiento final. La sangre total se centrifugó a 5.000g durante 5 min, el suero se recuperó y se almacenó a -80 C en el banco de sueros de FIDIC hasta la fecha Ensayo por inmunoadsorción ligado a enzimas (ELISA) La antigenicidad de PvGAMA se evaluó por ensayos de ELISA utilizando sueros de pacientes que han sufrido episodios de malaria por P. vivax. Para los controles negativos, se usaron pacientes sanos que no han tenido episodios de malaria. Brevemente, en cajas de ELISA de 96 pozos, se fijaron cada uno de los fragmentos de PvGAMA (amino y carboxilo terminal) e incubaron durante toda la noche a 4 C y posteriormente a 37 C por 1 hora. Las placas se bloquearon con 200 µl leche descremada al 5% en PBS Tween 0.05% a 37 C durante 1 hora. La reactividad de los anticuerpos contra las proteínas recombinantes, se evaluó por incubación con sueros de humano en dilución 1:100 por una hora a 37 C. Las placas se incubaron con un
46 46 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax anticuerpo secundario anti-humano producido en cabra conjugado con peroxidasa (Biomedicals, Inc.) en dilución 1:5.000 usando las mismas condiciones que los sueros humanos. El ensayo se reveló con una solución de sustrato para peroxidasa (KPL Laboratories, WA, USA), de acuerdo con las indicaciones del fabricante. Finalmente, a las placas se les midió DO 620 en un MultiskanGo (Thermo scientific). El valor de DO se calculó restando el valor obtenido para cada pocillo menos el pocillo control sin antígeno. El umbral de corte para determinar positividad correspondió a la media de las DO más tres veces la desviación estándar de la reactividad de los sueros de individuos sanos. Los ensayos se realizaron por triplicado Procesamiento de muestras de sangre de cordón umbilical Las muestras de sangre de cordón umbilical de recién nacidos utilizadas en esta investigación, colectadas por el personal del Hemocentro Distrital (Bogotá D.C.), se procesaron en el sistema de procesamiento de células (SEPAX, Biosafe) para reducir las células nucleadas de acuerdo con las recomendaciones del fabricante. Las muestras se almacenaron a 4ºC y el receptor del Antígeno Duffy para Quimiocinas (DARC) se determinó por ensayo de aglutinación usando anticuerpos dirigidos contra la fracción Fya o Fyb de la molécula. El porcentaje de células nucleadas se determinó por el conteo de 20 campos con una ampliación de 100x, utilizando la tinción de Wright, antes de llevar a cabo el ensayo de unión Ensayo de unión a reticulocitos humanos La unión de los fragmentos recombinantes de PvGAMA a reticulocitos humanos se evaluó por citometría de flujo en tres ensayos independientes. En resumen, 5 µl de una muestra de cordón umbilical con fenotipo duffy Fya-Fyb+ se incubó con 25 µg de cada proteína recombinante (PvGAMA RC-1, RV-1, RC-2 y RV-2) por 16 horas a 4 C y con agitación a 4 rpm. De igual manera, 25 µg de DBP-II y DBP-III/IV se utilizaron en el ensayo como controles. Las células se incubaron con un anticuerpo monoclonal de ratón anti-his-pe (1:40) (MACS molecular-miltenyi Biotec) por 30 min en la oscuridad previo a lavados con solución de PBS- BSA 1%. Células blancas y reticulocitos se marcaron incubando con anticuerpos monoclonales anti-cd45 clon 2D1 (1:80) (Becton Dickinson) y anti-cd71 APC-H7 clon M-A712 (1:80) (Becton Dickinson) por 20 min en oscuridad. Finalmente, la unión a reticulocitos se cuantificó
47 47 analizando 1 millón de eventos en el citómetro FACSCanto II (BD, San Diego, CA, USA.) y el software FlowJo V10. Las células CD71 low (CD71 lo ) y high (CD71 hi ) se determinaron considerando las señales de intensidad de PE como una función de CD Análisis estadístico Los valores de las medias y desviaciones estándar se calcularon de las mediciones de tres experimentos independientes. La significancia estadística se evaluó por comparación de medias usando un nivel de significancia de 0.05 para probar la hipótesis planteada. La prueba t-student y el análisis de varianza (ANOVA) se utilizaron para comparar cada grupo experimental y el grupo control. La prueba de Tukey s se utilizó para comparaciones múltiples de las medias entre el grupo experimental y el control. GradhPad Software (San Diego, CA) se usó para calcular todos los análisis estadísticos.
48 48 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
49 49 5. Resultados 5.1. Identificación in silico de proteínas relacionadas con la invasión de P. vivax Con los criterios establecidos en la metodología, se identificaron varios genes que codifican moléculas de P. vivax localizadas en organelos apicales, las cuales desempeñan un rol importante en la unión a células como las previamente reportadas RBPs (31, 143) y RONs (34). GAMA fue otra molécula identificada y está presente en otras especies de Plasmodium que causan malaria en humanos (P. falciparum y P. knowlesi), simios (P. cynomolgi) y roedores (P. yoelii, P. chabaudi y P. berghei) de acuerdo con la información disponible en la base de datos PlasmoDB (ID: PVX_088910). Respecto al gen gama de la cepa de referencia Sal-I de P. vivax, éste codifica para una proteína de 771 aa de longitud con una masa molecular aproximada de 82.7 kda; la molécula es 33 residuos más larga que su homólogo en P. falciparum (PfGAMA: 738 aa). PvGAMA tiene una secuencia señal de secreción con un sitio de escisión probable en el aminoácido 20 y una secuencia de anclaje a GPI situada entre los residuos de acuerdo con la predicción hecha con los software SignalP 4.0 y FragAnchor (Fig 5-8A), coincidiendo con sus homólogos PfGAMA (18) y en TgGAMA (Toxoplasma gondii) (20). El análisis de la secuencia en la base de datos Interpro reveló la presencia de una repetición en tándem degenerada, que consiste de 21 copias del pentapétido (A/L)AN(A/G)(N/D) y está localizada entre los residuos 591 y 695 que contienen varios epítopes de células B de acuerdo con la predicción de Antheprot. Tomando en cuenta las características predichas in silico para GAMA y la evidencia de su rol en la invasión en parásitos del filo Apicomplexa (Plasmodium y Toxoplasma) descritas en una de las secciones anteriores, se escogió dicha proteína para ser caracterizada en la cepa VCG-I de P. vivax.
50 50 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Figura 5-8. Caracterización de PvGAMA (A) Predicción in silico de características de PvGAMA. El diagrama muestra la localización del péptido señal, la región repetitiva de pentapéptidos y la secuencia de anclaje a GPI. Las barras en colores representan los péptidos seleccionados para el ensayo de inmunización en conejos: amarillo (péptido 39236); azul (péptido 39470); rojo (péptido 39472) y verde (péptido 39238). Los fragmentos recombinantes usados para la ELISA (rpvgama-nt (aa ), rpvgama-ct (aa )) y para los ensayos de citometría (RC-1 (aa ); RV-1 (aa ); RC-2 (aa ) y RV-2 (aa )) también se muestran. (B a D) Transcripción del gen gama y expresión en esquizontes. (B) Línea 1 indica el marcador de masa molecular. Líneas 2-4 muestra la amplificación del gen usando ADNc RT+, RT- y ADNg, respectivamente. (C) Reconocimiento de PvGAMA usando anticuerpos producidos en conejos. Línea 1 indica marcador de masa molecular (MW). Líneas 2 y 3 muestran el reconocimiento de GAMA usando suero pre-inmune y suero post-iii (anti-pvgama39472), respectivamente. (D) Localización celular de PvGAMA en esquizontes maduros y merozoitos libres. La imagen muestra el reconocimiento de la proteína (verde), núcleos (azul) y reticulocitos infectados (RETi) en luz blanca.
51 El gen pvgama se transcribe en esquizontes de la cepa VCG-I de P. vivax La presencia del gen gama en el genoma de la cepa VCG-I de P. vivax y su transcripción se confirmó por PCR usando cebadores específicos (diseñados usando la secuencia del gen en Sal- I), ADNc y ADNg de esquizontes como molde. La Figura 5-8B muestra un producto de amplificación de aproximadamente pb usando ADNc (RT+) y ADNg (líneas 2 y 4, respectivamente) el cual corresponde al gen pvgama completo, mientras que no se obtuvo producto en la muestra control (RT-), lo que indica que el ADNc no está contaminado con ADNg (Fig 5-8B, carril 3). Comparando la secuencia obtenida de ADNc (depositada en el NCBI bajo el código de acceso KT248546) y el ADNg, se pudo evidenciar que el gen es codificado por un único exón. Gracias a los alineamientos de la secuencia del gen de la cepa VCG-I con la cepa de referencia Sal-I, se identificaron tres mutaciones sinónimas c.258 T>C, c.1926 T>A y c.1929 T>C. La amplificación del gen pvgama a partir del ADNc sintetizado, sugiere que éste se transcribe en esquizontes, coincidiendo así con el análisis de la transcripción de tres aislados clínicos de P. vivax, donde gama tiene un perfil de expresión prominente durante tiempos TP7- TP9, correspondientes a parásitos que se desarrollan durante el estadio maduro (esquizonte temprano y maduro) del ciclo de vida intra-reticulocito (25) y con los estudios en P. falciparum y T. gondii (18, 20) PvGAMA se expresa en esquizontes maduros y se localiza en el polo apical del parásito. Anticuerpos dirigidos contra el péptido sintético 39472, diseñado con base en la secuencia de PvGAMA (Fig. 5-8A), se usaron para evaluar la presencia de la proteína y localización en esquizontes maduros del parásito. Los anticuerpos se purificaron para mejorar la reactividad contra la proteína GAMA, dado que mostraron mayor reactividad por Western blot (Tabla 4-2, Anexo 2). Anticuerpos específicos anti-pvgama detectaron bandas por Western blot en el lisado de parásito de VCG-I tratado en condiciones reducidas, correspondientes a productos de ~56 kda, ~44 kda y ~30 kda; únicamente la banda de 56 kda fue reconocida débilmente con el suero pre-inmune (Fig 5-8C, línea 2), lo cual indica un efecto de reactividad cruzada. Desafortunadamente, los péptidos y no mostraron reactividad contra el lisado de parásito (Tabla 4-2, anexo 2), por lo que no fue posible obtener anticuerpos para verificar
52 52 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax los productos proteolíticos generados hacia la región amino terminal de PvGAMA. Los resultados encontrados, sugieren que PvGAMA presenta dos procesamientos proteolíticos que corresponden a los fragmentos de ~44 kda y ~30 kda (Fig 5-8C), el primero de ellos es similar al observado en el fragmento carboxilo terminal de PfGAMA de ~49 kda, generando uno de ~42 kda (19). La identificación de la proteína GAMA en lisado de esquizontes de la cepa VCG- I de P. vivax, concuerda con la de un estudio del proteoma de P. vivax de 10 aislados de pacientes sintomáticos en Tailandia, donde un péptido de GAMA se identificó (99% de certeza) por espectrometría de masas (27). Usando anticuerpos anti- PvGAMA para localizar la proteína, se detectó una señal punteada de fluorescencia cerca de los núcleos del parásito en esquizontes maduros (Fig 5-8D), lo cual es característico de proteínas localizadas en organelos apicales (roptrias o micronemas) y es coherente con el patrón de expresión apical para PfGAMA y TgGAMA (18, 20). La doble localización usando un marcador de roptrias o micronemas no fue exitosa, por lo que se sugiere que la microscopía inmunoelectrónica es fundamental para validar la localización orgánulo-específica de PvGAMA. 5.4 Diversidad genética y señales de selección en el antígeno GAMA Las secuencias de pvgama obtenidas a partir de 5 diferentes genomas se alinearon con la secuencia de la cepa VCG I y secuencias ortólogas de 5 especies filogenéticamente relacionadas. El alineamiento mostró la existencia de un polimorfismo de tamaño en pvgama debido a la repetición [C/T]C[G/C]C[A/T]AA[C/T][C/G][A/G/C][G/A]AC[G/C/A], la cual no está presente en P. cynomolgi, P. inui, P. fragile, P. knowlesi o P. coaneti (Anexo 3). En P. vivax se observaron 5 sitios segregantes y una π de Al evaluar las tasas de sustitución sinónima y no sinónima, no se encontraron valores significativos (d N-d S = (0.001), p > 0.1). Sin embargo, la divergencia sinónima fue mayor a la divergencia no sinónima (p < ) cuando se compararon las secuencias de pvgama con cada una de las especies relacionadas (K N-K S P. vivax/p. cynomolgi = (0.006); K N-K S P. vivax/p. inui = (0.008); K N-K S P. vivax/p. fragile = (0.006); K N-K S P. vivax/p. knowlesi = (0.009); K N-K S P. vivax/p. coaneti = (0.007)). La ventana deslizante de la tasa ω (d N/d S y K N/K S) mostró dos regiones altamente conservadas entre especies (ω < 0.5)
53 53 entre los codones y (Figura 5-9). Adicionalmente, se observaron 308 sitios seleccionados negativamente entre especies (Figura 5-9). El algoritmo Branch-site REL identifico señales de selección positiva episódica en los linajes que dieron origen a P. knowlesi y P. coaneti, así como al linaje formado por P. cynomolgi y P. fragile (Anexo 1). 22 sitios mostraron evidencia de selección positiva entre especies (Figura 5-9). Figura 5-9. Ventana deslizante de índice ω Valores ω intra-especies (dn/ds) son representados en azul, mientras que los valores ω inter-especies (KN/KS entre P. vivax y parásitos de malaria que infectan primates) se muestra en púrpura. Un diagrama del gen se observa debajo de la ventana deslizante. Sitios (codones) inter-especies seleccionados negativamente se marcaron en verde mientras los sitios seleccionados positivamente en rojo. Numeración basada en el alineamiento del anexo PvGAMA es antigénica durante una infección natural por P. vivax Fragmentos de pb y 951 pb codificantes de la región amino terminal (rpvgama-nt) y carboxilo terminal (rpvgama-ct) de GAMA, respectivamente (Fig 5-10), se amplificaron por PCR, se clonaron y exitosamente se expresaron en el sistema E. coli. Después de la purificación de los fragmentos por cromatografía de afinidad en fase sólida, su integridad fue evaluada por SDS-PAGE; rpvgama-nt presentó un leve procesamiento proteolítico debido posiblemente a proteasas de la bacteria, mientras se observó una sola banda para rpvgama-ct (Figura 5-11A y B, línea 4). La correcta traducción de la proteína se verificó con un anticuerpo monoclonal
54 54 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax anti-tag de histidinas (Fig. 5-11A y B, línea 5) y contra los péptidos de PvGAMA (Fig 5-11C) que reconocen cada fragmento recombinante. Figura Amplificación por PCR de los fragmentos de GAMA. Líneas 1 y 5 indican marcador de masa molecular. Líneas 2 y 6 muestran el producto de PCR de rpvgama-nt y rpvgama-ct amplificado a partir de la muestra RT+; estos se usaron para la clonación en el vector de expresión pexp-ct/topo. Líneas 3 y 7 corresponden a la PCR usando como templado la muestra RT-. Líneas 4 y 8 muestran la amplificación de rpvgama-nt y rpvgama-ct a partir de ADNg. Figura Detección de las proteínas recombinantes. (A y B) Expresión y purificación de los fragmentos recombinantes de GAMA. Línea 1 indica el marcador de masa molecular (MW). Líneas 2 y 3 muestran el lisado celular no-inducido e inducido respectivamente. Líneas 4 y 5 los fragmentos purificados rpvgama-nt y rpvgama-ct teñidos con azul de Coomassie o Western blot, respectivamente, usando anticuerpos monoclonales anti-histidinas en el último caso. (C) anticuerpos policlonales de conejo son capaces de reconocer rpvgama por Western blot. Línea 1 indica marcador de masa molecular (MW). Líneas 2 y 4 muestran el reconocimiento con suero pre-inmune. Líneas 3 y 5 indican el reconocimiento de rpvgama-nt y rpvgama-ct, respectivamente.
55 55 De acuerdo con el enfoque clásico, las proteínas antigénicas deben tenerse en cuenta para el desarrollo de vacunas; por lo tanto, se evaluó la antigenicidad de rpvgama-nt y Ct con sueros de 37 pacientes que sufrieron malaria por P. vivax e individuos que nunca han tenido episodios de la enfermedad como controles. rpvgama-nt reaccionó con el 64.8% de los sueros evaluados (punto de corte de 0.26) mientras que el fragmento rpvgama-ct fue reconocido por el 67.5% de ellos (punto de corte de 0.47) (Fig 5-12A). Estos datos concuerdan con un estudio del perfil de respuesta inmune humoral para P. vivax en el cual rpvgama fue reconocida por el 54.5% con los sueros usados en el array (144). Figura Antigenicidad de PvGAMA durante una infección natural por malaria. El diagrama muestra la distribución en la densidad óptica (OD) (Eje Y) para la detección rpvgama-nt y rpvgama-ct por P. vivax (A) o rpvgama-ct por P. falciparum (B) con sueros de individuos infectados y no infectados (Eje X). Las diferencias significativas entre las medias se muestran, así como los valores p correspondientes al análisis estadístico. El análisis estadístico para rpvgama-nt mostró una diferencia significativa entre las medias (m) de los grupos (p=0.035), obteniendo una m=0.38 para el grupo de pacientes infectados y m=0.12 para el grupo control (Figura 5-12A). Así mismo, hubo una diferencia estadísticamente significativa entre las medias para el reconocimiento de los sueros por rpvgama-ct (p<0.0001), donde la media para el grupo de pacientes infectados fue de 0.67 y para el grupo control 0.14 (Figura 5-12A). Cuando se analizaron las medias entre rpvgama-nt y rpvgama- Ct, hubo de igual manera diferencias significativas (p=0.0002). Teniendo en cuenta que la respuesta fue mayor frente a rpvgama-ct, se decidió confirmar si los anticuerpos generados
56 56 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax en una infección natural por P. falciparum eran capaces de detectar este fragmento. No se observó diferencia estadísticamente significativa (p=0.6962) para el reconocimiento de rpvgama-ct por estos sueros. La significativa reactividad de los sueros de individuos infectados contra las recombinantes, indica que se generó una respuesta inmune frente a la proteína durante una infección natural, la cual fue más alta y específica de la especie frente a la región carboxilo terminal PvGAMA se une a reticulocitos humanos. Eritrocitos con fenotipo duffy Fya- Fyb+ (Uno de los fenotipos más prevalente en Colombia) de sangre de cordón umbilical, se incubaron con las regiones variables y conservadas de PvGAMA, exitosamente extraídas en su forma soluble (Fig 5-13) y posteriormente se analizaron por citometría de flujo para cuantificar la interacción. El porcentaje de unión de cada recombinante (marcada con el anticuerpo anti-his-pe) se calculó de la población de células CD71+CD45- (reticulocitos) o CD71-CD45- (eritrocitos maduros), usando una estrategia de exclusión de glóbulos blancos (Figura 5-14). Todas las proteínas mostraron un desplazamiento en la curva del histograma cuando se comparó la señal de PE con el control (células no incubadas con proteína recombinante) (Figura 5-14). Figura Obtención de las regiones conservadas y variables de PvGAMA en su forma soluble. (A y B) Expresión y purificación de los fragmentos recombinantes de GAMA. Las líneas 1 indican el marcador de masa molecular (MW). Líneas 2 y 4 muestra la tinción con azul de Coomassie. Líneas 3 y 5 muestra el Western blot usando anticuerpos monoclonales anti-histidinas para cada una de la regiones conservadas y variables. (C) Expresión y purificación del control positivo (DBP-RII) y negativo (DBP- RIII/IV). Se indica la tinción con azul de Coomassie (carril 2 y 4) y el Western blot (carril 3 y 5).
57 Figura Análisis de citometría de flujo. El diagrama muestra la estrategia de selección para las poblaciones maduras e inmaduras de eritrocitos. Se excluyeron los dobletes enfrentando la señal para FSC-H contra FSC-A. Luego, las células se seleccionaron por su complejidad interna, analizando SSC-A contra FSC-A. La señal CD45 y CD71 se utilizó para escoger las poblaciones de eritrocitos maduros (CD71-CD45-) y reticulocitos (CD71+CD45- ) y omitir linfocitos activados (CD71+CD45+). El porcentaje de células que tienen la proteína unida se calculó usando la señal de PE (CD71+CD45-PE+). También se muestra un histograma representativo de tres experimentos independientes que muestran la señal de PE para el ensayo de unión de RC-2, en comparación con el control. 57
58 58 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Es importante destacar, que los fragmentos de GAMA se unen en mayor porcentaje a reticulocitos comparado con glóbulos rojos maduros (Fig 5-15A). Las regiones conservadas mostraron mayor interacción con reticulocitos comparado con los controles negativos (DBP- III/V: X ±DS = 2.0±0.34; péptido de 6 histidinas: X ±DS = 0.49±0.18) (p<0.05). RC-2 se unió a un 10.11% (DS=1.33) de reticulocitos, lo cual fue muy similar a lo observado en el control positivo DBP-RII (X ±SD = 11.8±1.15) (p>0.05), mientras que RC-1 fue capaz de unirse a un 6.36% de las células (DS=0.30) (Fig 5-15A). Respecto a las regiones variables, GAMA región variable 1 se unió un 3.08% de los reticulocitos (DS=0.54) mientras que RV-2 se unió un 5.64% (DS=0.37) (Figura 5-15A). Por otro lado, cuando el porcentaje de unión se analizó en función de la señal CD71 APC-H7, los fragmentos RC-1, RC-2 y RV-2 mostraron alta interacción con reticulocitos CD71 hi en contraste con RV-1 y DBP-RII. Además, se encontró una diferencia significativa con un valor p<0.05 entre el porcentaje de unión de las recombinantes RC-1, RC-2 y RV-2 con el control DBP-RII en cada grupo (CD71 low y CD71 hi ) (Figura 5-15B). Estos hallazgos son una fuerte evidencia para sugerir que GAMA en P. vivax tiene un rol funcional en la interacción con reticulocitos, principalmente de las etapas más inmaduras. Figura Actividad de unión de PvGAMA a reticulocitos humanos. El análisis de citometría de flujo muestra el porcentaje de unión de los fragmentos recombinantes a las células CD71-CD45- y CD71+CD45- (A), como también la señal CD71- (solo para reticulocitos CD71+CD45-) (B). Se muestran los controles usados en el ensayo, DBP-RII (control positivo) y DBP- RIII/IV y un péptido de 6 histidinas (controles negativos). (C) ensayo de inhibición de la unión a reticulocitos con las regiones C1 y C2 usado sueros humanos (Anticuerpos generados en una infección natural). El porcentaje de unión en ambos análisis se expresó como la media más las desviaciones estándar de tres experimentos independientes. ** indica una diferencia estadísticamente significativa con un valor P<0.05.
59 59 Por último, se realizó un ensayo de inhibición de la adhesión por citometría con sueros de individuos que habían tenido episodios de malaria por P. vivax, para determinar si los anticuerpos producidos en una infección natural podrían inhibir la interacción de las regiones funcionales conservadas (RC-1 y RC-2) con reticulocitos. La Figura 5-15C, muestra que los fragmentos recombinantes conservados pre-incubados con sueros humanos, fueron capaces de unirse a su célula diana (CR1: X ±SD = 6.21±0.27; CR2: X ±SD = 9.83±0.09) con un porcentaje similar a los controles del ensayo (CR1: X ±SD = 6.5±0.08; CR2: X ±SD = 10.01±0.95) (P>0.05), lo que sugiere que la respuesta inmune está dirigida contra las regiones que no están implicadas en la unión a las células.
60 60 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
61 61 6. Discusión La invasión de merozoitos a eritrocitos implica la participación de varias moléculas del parásito expresadas al final del ciclo de vida intra-eritrocítico, principalmente aquellas que están presentes en los organelos apicales como roptrias y micronemas (91). La mayoría de las proteínas apicales identificadas hasta la fecha, desempeñan un papel clave en la adhesión del parásito para invadir las células diana de invasión; por ejemplo, la proteína AMA-I es una de las proteínas mejor caracterizadas, debido a que participa en la formación de la unión fuerte con el eritrocito en complejo con las proteínas RONs (101), mientras que los antígenos de unión a eritrocitos (EBL) son proteínas integrales de membrana que poseen dominios de unión, que les permiten hacer interacción proteína-proteína, promoviendo así la entrada del parásito a la célula (91). Este estudio tuvo como objetivo principal identificar y caracterizar una proteína en P. vivax (diferente a las ya identificadas), con un papel potencial en la adhesión celular. Un enfoque para identificar proteínas en P. vivax emplea la proteómica en combinación con análisis in silico, el cual ha permitido avanzar en el conocimiento de la maquinaria proteica que utiliza el parásito en el proceso de invasión, junto con la predicción de la función que estas moléculas puedan tener (27, 120). Gracias a la información disponible de los estudios de proteoma de P. vivax, se realizó una predicción de candidatos a vacuna, teniendo en cuenta los siguientes parámetros: un perfil de transcripción en el ciclo intraeritrocítico, predicción de una secuencia señal de secreción o secuencias de anclaje a GPI y dominios putativos. Con la revisión se logró identificar GAMA cuyo homólogo en P. falciaprum había sido descrito como un candidato potencial a vacuna (18,
62 62 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 19). Encontrar varios homólogos en diferentes especies de Plasmodium apoyan la idea de que GAMA, posiblemente podría tener una función específica dentro del género. A pesar de la dificultad en la investigación básica con respecto a P. vivax, dadas las características intrínsecas de su biología (22), hemos sido capaces de identificar la proteína GAMA debido a la adaptación de la cepa VCG-I de P. vivax en primates (28). Los resultados mostraron que el gen pvgama se transcribe y codifica una proteína que sufre procesamiento proteolítico y muestra un patrón punteado propio de proteínas apicales en esquizontes maduros de la cepa VCG-I de P. vivax (Fig 5-8). Se ha encontrado, que los transcritos del parásito están estrictamente controlados durante el desarrollo del ciclo de vida intraeritrocítico (14, 25) y que los productos que codifican se correlacionan con los que ejercen una función especializada. Por ejemplo, más de 50 secuencias que codifican para proteínas en P. falciparum tienen una expresión máxima durante etapas maduras (>35 horas post-invasión) y desempeñan un papel importante durante la invasión celular (145). Lo anterior, sumado a que GAMA se transcribe y se expresa en esquizontes de P. vivax, sugiere que la proteína podría tener una función durante la invasión a reticulocitos. Recientemente Cornejo et., al (146) y Garzón-Ospina et., al (130) identificaron señales de selección natural en genes de P. vivax al analizar las secuencias de 5 genomas provenientes de diferentes regiones del mundo (131). Esos resultados fueron concordantes con los obtenidos al aumentar el número de secuencias analizadas ( ), por lo tanto este tipo de análisis pueden ser una aproximación para seleccionar antígenos conservados y con restricciones funcionales con el fin de diseñar una vacuna completamente efectiva. El antígeno de micronemas con anclaje a GPI (GAMA), mostro una baja diversidad genética, comparable a la observada en msp-4 (150, 151), msp-7a/-7k/-7f/-7l (152, 153), msp-8 (154), msp-10 (152, 154), pv12, pv38 (147, 149), pv41 (148, 149) y rap-1/-2 (155) los cuales parecen estar involucrados en el proceso de invasión a la célula huésped. Se encontró evidencia de selección positiva episódica en algunos linajes, lo cual puede ser una consecuencia de la adaptación en estos parásitos a diferentes hospederos (156, 157) durante su historia evolutiva. A pesar de que no se observó valores estadísticamente significativos en la diferencia d N-d S, se observó que entre especies, la divergencia K S fue mayor a la divergencia K N, encontrándose un alto número de sitios bajo selección negativa, sugiriendo que ésta juega un papel importante en la evolución del antígeno GAMA. Dos regiones a lo largo del antígeno
63 63 fueron altamente conservadas entre especies mostrando una tasa evolutiva (ω) <0.5. Teniendo en cuenta que las regiones funcionales suelen evolucionar más lentamente y que la selección natural tiende a mantener estas regiones conservadas entre las especies (129), nuestros resultados sugieren que las regiones entre los residuos (40 % de los sitios seleccionados negativamente) y (64.5 % de los sitios seleccionados negativamente) podrían estar restringidas funcional/estructuralmente y por lo tanto el diseño de una vacuna podría enfocarse en dichos fragmentos. Basados en el estudio de polimorfismo de PvGAMA, decidimos determinar la unión de dos regiones conservadas (RC-1 RC-2) y dos regiones variables (RV-1 RV-2) a las células, con el fin de validar la nueva estrategia de predicción in silico de regiones funcionales y elucidar la función de la proteína. Además, se utilizó una muestra de reticulocitos con un fenotipo Duffy positivo, dado que PvGAMA tiene actividad de unión independiente de la expresión de dicho antígeno (158). En este análisis, encontramos que los fragmentos de PvGAMA se unen en una menor proporción a eritrocitos maduros, comparado con reticulocitos (CD71+CD45-) (Fig 5-15A), lo que permite sugerir que la proteína interactúa preferentemente con este último tipo de células. Los fragmentos localizados en la región carboxilo terminal (RC-2 y RV-2) presentaron mayor unión a los reticulocitos, a diferencia de los fragmentos presentes en la región amino terminal (RC-1 y RV-1) (Fig 5-15A); esto es consistente con los resultados observados para el fragmento C-terminal en P. falciparum (denominado Tr3 en el estudio); sin embrago, la región amino terminal de PfGAMA (Tr1) no tiene actividad de unión a eritrocitos (18). Estas diferencias pueden explicarse por la sensibilidad de las diferentes técnicas utilizadas para los ensayos de unión, debido a que posiblemente PfGAMA (Tr1) presenta una muy baja actividad de unión que no alcanza a ser detectada por Western blot (como también ocurre con TgGAMA-Ct (20)), pero si puede ser detectada por citometría de flujo como se mostró en este estudio. De acuerdo con la literatura, los merozoitos de P. vivax tienen tropismo por reticulocitos en estadios muy inmaduros que se caracterizan por expresar el receptor CD71 (159); por tanto, se utilizó un anticuerpo monoclonal anti-cd71 para identificar la preferencia de unión de las regiones de GAMA (variables y conservadas) a los reticulocitos inmaduros. Hubo un alto porcentaje de unión para RC-1, RC-2 y RV-2 a reticulocitos CD71 hi (Fig 5-15B), sugiriendo que PvGAMA interactúa principalmente con la etapa más inmadura de este tipo celular. Se ha
64 64 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax reportado que durante la maduración de los reticulocitos disminuyen las proteínas integrales de membrana (160). Por tanto, los hallazgos encontrados sugieren que el receptor de PvGAMA es menos abundante en células CD71 lo a diferencia de CD71 hi, como consecuencia de la maduración celular. El hecho de que más del 69% de las células CD71+CD45- fueron CD71 lo (X ±SD = 69.3±3.3) puede ser la explicación del porqué los fragmentos de PvGAMA no se unieron al 100% de los reticulocitos CD71+. Algunas moléculas descritas a la fecha de P. vivax presentan el mismo perfil de unión como: DBP (161), MSP-1 (162), RBP-1 (143), EBP (del inglés, Erythrocyte Binding Protein) (163) y las RBP-1a y 1b (72). Con respecto a la proteína EBP, se une preferentemente a reticulocitos CD71 hi Fy +, similar a lo encontrado con PvGAMA, lo que sugiere, que los receptores para estos ligandos son más abundantes en reticulocitos jóvenes que están confinados principalmente en médula ósea (159), y que la selección de reticulocitos por P. vivax no solo está gobernada por las RBPs sino también por otras proteínas como DBP, MSP-1, EBP y ahora GAMA. La antigenicidad es otro de los parámetros clásicos para la selección de moléculas que pueden servir para el desarrollo de una vacuna, ya que se ha visto que una respuesta inmune inducida durante la infección, está relaciona con la inmunidad adquirida de forma natural (164). Los resultados obtenidos en este estudio, indican que PvGAMA es capaz de desencadenar una respuesta inmune durante una infección natural por P. vivax, debido a que anticuerpos generados en los individuos afectados reconocen los fragmentos recombinantes por ELISA (Fig 5-12A). Se observó un reconocimiento mayor para rpvgama-ct, probablemente debido a la presencia de la secuencia repetitiva localizada en esta región (aa 591 a 695), compuesta por residuos de [A/L]AN[A/G][N/D]. Estas inserciones podrían ser una adaptación de P. vivax a los seres humanos ya que regiones repetitivas suelen actuar como cortinas de humo para evadir las respuestas inmunes del hospedero (45, 165). Esta característica ha sido encontrada en algunos antígenos descritos en la cepa VCG-I de P. vivax localizados en la superficie del parásito como Pv12 (166), ARP (167) y en el polo apical como Pv34 (168), RON 1 (169), RON 2 (170) y RON 4 (171). Aunque hubo una respuesta inmune frente a PvGAMA (Fig 5-12A), ésta no fue suficiente para inhibir la unión de las regiones conservadas a los reticulocitos (Fig 5-15C). Se ha observado que las regiones conservadas de las proteínas de P. falciparum (implicadas en la unión a eritrocitos) no desencadenan una respuesta inmune cuando se usan como vacunas en el modelo Aotus, mientras que las regiones variables activan respuestas alelo específicas frente
65 65 al reto con el parásito (172). Por consiguiente, los anticuerpos contra PvGAMA inducidos en una infección natural están dirigidos frente a epítopes inmunodominantes que no tienen importancia en la actividad de unión. Estos hallazgos soportan que PvGAMA posee algunas regiones que facilitan la actividad de unión a reticulocitos humanos. Teniendo en cuenta que GAMA es un antígeno inmunorreactivo y habiendo confirmado que las regiones conservadas se unen a los reticulocitos, se postulan RC-1 y RC-2 como posibles candidatos para el desarrollo de una vacuna contra P. vivax, las cuales podrían ser críticas para prevenir el problema de la enorme variabilidad del parásito en el desarrollo de una vacuna.
66 66 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
67 67 7. Conclusiones El gen pvgama se transcribe y traduce para un producto proteico que sufre procesamiento proteolítico y presenta un patrón punteado típico de antígenos apicales en esquizontes de la cepa VCG-I de P. vivax; esta característica posiblemente se relaciona con su función en la adherencia de los merozoitos de P. vivax a los reticulocitos. PvGAMA es una proteína antigénica, ya que es capaz de desencadenar una respuesta inmune durante una infección natural por P. vivax; esto se corroboró debido a que anticuerpos generados en los individuos con episodios de malaria reconocieron los fragmentos recombinantes por ELISA. GAMA en P. vivax, posee algunas regiones a lo largo de la secuencia de aminoácidos que se unen a reticulocitos, principalmente CD71 hi, que corresponden a la etapa más inmadura de este tipo celular. Este hallazgo pone de manifiesto sus propiedades funcionales como una adhesina usada por el parásito para adherirse a su célula diana.
68 68 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
69 69 8. Perspectivas Con la finalidad de conocer en detalle la interacción entre la molécula GAMA y el reticulocito, en relación a determinar la especificidad de la unión, la identidad del receptor, la inmunogenicidad y protección, se requiere adelantar diferentes experimentos: Con respecto a determinar la especificidad de la unión y evaluar si esta es hacia el mismo receptor o hay redundancia por otros receptores, se beben realizar ensayos de saturación de la unión para evaluar si es dependiente de concentración, y ensayos de competencia con los fragmentos recombinantes de PvGAMA marcados y no marcados. Para conocer la identidad del receptor en la membrana de los reticulocitos al cual se une GAMA, es necesario realizar ensayos como inmunoprecipitación usando anticuerpos policlonales anti-pvgama y posteriores análisis por espectrometría de masas de los complejos receptor-ligando aislados. Adicionalmente, para poder usar PvGAMA como posible candidato a vacuna, es necesario realizar estudios de inmunogenicidad y protección en un modelo experimental como los monos Aotus, utilizando las regiones conservadas aquí identificadas.
70 70 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
71 71 9. Aspectos éticos 9.1 Consentimiento informado Suero de 37 pacientes obtenidos en áreas endémicas de malaria en Colombia, quienes sufrieron episodios de malaria por P. vivax, como también de 6 individuos sanos que nunca han sufrido la enfermedad fueron usados para el estudio. Además, muestras de cordón umbilical empleadas para los ensayos de unión fueron colectadas y proporcionadas por el Hemocentro distrital de Bogotá (Colombia). Todos los individuos (incluyendo las progenitoras respecto a las muestras de cordón umbilical) firmaron un documento de consentimiento donde se les informaba detalladamente de los objetivos del estudio. Todos los procedimientos fueron aprobados por el comité de ética de FIDIC. 9.2 Manejo y regulaciones para el cuidado animal La manipulación experimental de animales usados en el estudio se realizó hace más de 6 años (estudio con Monos Aotus spp) de acuerdo con la Ley 84 de 1989 y la Resolución 504 de 1996 de la República de Colombia. Monos Aotus spp mantenidos en la estación de primates de la Fundación Instituto de Inmunología de Colombia - FIDIC (Leticia, Amazonas) y conejos de la raza Nueva Zelanda se manipularon siguiendo las guías de cuidados para animales de laboratorio (National Institute of Health, USA) bajo la supervisión constante de un médico veterinario. Los procedimientos de inmunización y sangría de los monos Aotus y conejos, fueron previamente aprobados por el comité de ética de FIDIC y se realizaron de acuerdo con las condiciones estipuladas por CorpoAmazonía (Resolución del 13 de septiembre de 2006). Vale la pena destacar que se hicieron todos los esfuerzos para minimizar el sufrimiento de los animales utilizados en este estudio.
72 72 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
73 Anexos Anexo 1. Selección positiva linaje específica. Ramas bajo selección diversificación fueron identificadas por el método REL-site Branch. La filogenia fue inferida en MEGA v6 por el método de Máxima verosimilitud utilizando el modelo GTR+G. ω+: valores de la tasa ω; Pr [ω = ω +]: proporción de sitios evolucionando a la tasa ω+; valor P corregido para múltiples pruebas utilizando el método de Holm-Bonferroni.
74 74 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Anexo 2. Reactividad de los anticuerpos policlonales. Reconocimiento de la proteína en un lisado de P. vivax, usando suero de conejos dirigidos contra los péptidos 39326, 39328, y PI: suero pre-inmune, HI: suero híper-inmune. Anexo 3. Alineamientos del antígeno GAMA. Secuencias del gen pvgama de 6 cepas de P. vivax fueron alineadas con las secuencias ortólogas de P. cynomolgi, P. inui, P. fragile, P. coatneyi y P. knowlesi. (A) alineamientos de las secuencias de ADN y (B) alineamientos deducidos de la secuencia de aminoácidos. Las secuencias fueron obtenidas del GenBank bajo los siguientes códigos de acceso: India-VII AFBK AFBK , North Korean AFNJ , Brazil-I AFMK AFMK , Mauritania-I AFNI AFNI , P. inui NW_ , P. fragile NW_ , P. cynomolgi BAEJ , P. coatneyi CM y P. knowlesi NC_ (Ver páginas siguientes).
75 75 A. Alineamiento deducido de la secuencia de ADN * 20 * 40 * 60 * P.coatneyi_CM : ATGAAGTGCAACGGCTCCTTGCTAGTCCTACTTAGCGCAATACTCAGCGCTACGAACGCTTTGATACGAAATGGGAACAA : 80 P.knowlesi_NC_ : ATGAAGTGCAACGGCTCCTTGCTAGTCCTACTTAGCGCAATACTCAGCGCCACGAACGCTTTGATACGAAATGGGAACAA : 80 P.inui_NW_ : ATGAAGTGCAACACGGCCTTGCTAGTACTACTTAGCGCAATACTCAGCGCTGCAAACGCTTTGATACGAAATGGGAACAC : 80 P.fragile_NW_ : ATGAAGTATAACGCCACCTTGCTTGTCCTACTTAGCGCAATATTGAGCGCTGGGAAAGCTTTGATACGAAATGGGAACAA : 80 P.cynomolgi_BAEJ : ATGAAGTGCAACGCCGCCTTGCTAGTCCTACTTAGCGCATTACTCAGCGCTTCGAACGCTTTGATACGAAATGGGAACAA : 80 Brazil-I_AFMK _A : ATGAAGTGCAACGCCTCCTTGCTAGTCCTACTTAGCGCACTACTCAGCGCTGCGAACGCTTTGATACGGAATGGAAACAA : 80 VCG-I : ATGAAGTGCAACGCCTCCTTGCTAGTCCTACTTAGCGCACTACTCAGCGCTGCGAACGCTTTGATACGGAATGGAAACAA : 80 Sal-1_PVX_ : ATGAAGTGCAACGCCTCCTTGCTAGTCCTACTTAGCGCACTACTCAGCGCTGCGAACGCTTTGATACGGAATGGAAACAA : 80 Mauritania_I_AFNI : ATGAAGTGCAACGCCTCCTTGCTAGTCCTACTTAGCGCACTACTCAGCGCTGCGAACGCTTTGATACGGAATGGAAACAA : 80 NKorean_AFNJ : ATGAAGTGCAACGCCTCCTTGCTAGTCCTACTTAGCGCACTACTCAGCGCTGCGAACGCTTTGATACGGAATGGAAACAA : 80 India-VII_AFBK _AFBK : ATGAAGTGCAACGCCTCCTTGCTAGTCCTACTTAGCGCACTACTCAGCGCTGCGAACGCTTTGATACGGAATGGAAACAA : 80 ATGAAGTgcAACgcc CCTTGCTaGTcCTACTTAGCGCA TAcTcAGCGCt cgaacgctttgatacg AATGG AACAa * 100 * 120 * 140 * 160 P.coatneyi_CM : TCCGCAGGCATTAGTTCCGGAAAAGAGCGATGACTCGAGTGGGGGCCAGAACAAACCCTCCGGTGACAACCAAGACACGT : 160 Pknowlesi_NC_ : TCCGCAGGCATTAGTTCCGGACAATAGTGGTGACCCGAATGCGGGCCAGAACAAAACGTCCAGTGACAACCAAGACACAT : 160 Pinui_NW_ : TACGCAGGCATTAGTTCCGGAAAAGAGCAATGACCCGAGTGGGGGCCAGAACAACCCCTCCGGTGACAACCGAGAGACTT : 160 Pfragile_NW_ : TACGCAGGCCTTAGTTCCTGAAAATGGCGGTGACCCTAGTGGGGGACAGAACAACCCCTCCGGTGAAAACCAAGATACGT : 160 Pcynomolgi_BAEJ : TACGCAAGCATTAGTTCCGGAAAAGAGCGGTGACCCGAGTGGGGGCCAGAACAACCCCTCCGGTGATAACCAAGACACGT : 160 Brazil-I_AFMK _A : CCCGCAGGCATTAGTTCCTGAAAAGGGCGCTGACCCGAGTGGGGGCCAGAACAACCGCTCCGGAGAAAACCAAGACACGT : 160 VCG-I : CCCGCAGGCATTAGTTCCTGAAAAGGGCGCTGACCCGAGTGGGGGCCAGAACAACCGCTCCGGAGAAAACCAAGACACGT : 160 Sal-1_PVX_ : CCCGCAGGCATTAGTTCCTGAAAAGGGCGCTGACCCGAGTGGGGGCCAGAACAACCGCTCCGGAGAAAACCAAGACACGT : 160 Mauritania_I_AFNI : CCCGCAGGCATTAGTTCCTGAAAAGGGCGCTGACCCGAGTGGGGGCCAGAACAACCGCTCCGGAGAAAACCAAGACACGT : 160 NKorean_AFNJ : CCCGCAGGCATTAGTTCCTGAAAAGGGCGCTGACCCGAGTGGGGGCCAGAACAACCGCTCCGGAGAAAACCAAGACACGT : 160 India-VII_AFBK _AFBK : CCCGCAGGCATTAGTTCCTGAAAAGGGCGCTGACCCGAGTGGGGGCCAGAACAACCGCTCCGGAGAAAACCAAGACACGT : 160 CGCAgGCaTTAGTTCC GAaAAg Gcg TGACcCgAgTGgGGGcCAGAACAAcc ctccgg GA AACCaAGAcACgT * 180 * 200 * 220 * 240 P.coatneyi_CM : GCGAAATTCAAAAGATGGCGGAAGAAATGATGGAAAAAATGGTGAACGAAAAAGATGTGTTTACCTCCATCATGGAACCT : 240 Pknowlesi_NC_ : GTGAAATTCAAAAGATGGCCGAAGAAATGATGGAAAAAATGATGAAAGAAAAAGACGTGTTTAGCTCCATTATGGAACCT : 240 Pinui_NW_ : GTGAAATTCAACAGATGGCCCAAGAAATGATGGAAAAAATGATGAAAGAAAAAGACGTGTTTAGCTCCATCATGGAACCC : 240 Pfragile_NW_ : GCGAAGTTCAAAAGATGGCTGAAGAAATGATGGGAAAAATGATGAAAGAAAAAGACGTGTTTAGCTCCATTATGGAACCT : 240 Pcynomolgi_BAEJ : GTGAAATTCAAAAGATGGCCGAAGAAATGATGGAAAAAATGATGAAAGAAAAAGACATGTTTAGCTCCATCATGGAACCT : 240 Brazil-I_AFMK _A : GCGAAATTCAAAAGATGGCCGAAGAAATGATGGAAAAAATGATGAAGGAAAAAGACGTGTTTAGCTCCATCATGGAACCT : 240 VCG-I : GCGAAATTCAAAAGATGGCCGAAGAAATGATGGAAAAAATGATGAAGGAAAAAGACGTGTTTAGCTCCATCATGGAACCT : 240 Sal-1_PVX_ : GCGAAATTCAAAAGATGGCCGAAGAAATGATGGAAAAAATGATGAAGGAAAAAGACGTGTTTAGCTCCATCATGGAACCT : 240 Mauritania_I_AFNI : GCGAAATTCAAAAGATGGCCGAAGAAATGATGGAAAAAATGATGAAGGAAAAAGACGTGTTTAGCTCCATCATGGAACCT : 240 NKorean_AFNJ : GCGAAATTCAAAAGATGGCCGAAGAAATGATGGAAAAAATGATGAAGGAAAAAGACGTGTTTAGCTCCATCATGGAACCT : 240 India-VII_AFBK _AFBK : GCGAAATTCAAAAGATGGCCGAAGAAATGATGGAAAAAATGATGAAGGAAAAAGACGTGTTTAGCTCCATCATGGAACCT : 240 G GAAaTTCAAaAGATGGCcgAAGAAATGATGGaAAAAATGaTGAA GAAAAAGAcgTGTTTAgCTCCATcATGGAACCt
76 76 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax * 260 * 280 * 300 * 320 P.coatneyi_CM : CTCCAGAGCAAATTAACCGATGATCACCTCTGTTCAAAACTAAAATATACGAACGTTTGTCTTCACGAAAAGGACAAAAC : 320 Pknowlesi_NC_ : CTCCAGAGCAAATTAACCGATGATCGCCTCTGTTCTAAAATGAAATATACAAATATTTGTCTTCACGAAAAGGACAAAAC : 320 Pinui_NW_ : CTCCAGAGCAAATTAGCCGACGATCACCTCTGTTCAGAAGTAAAATATAAGAACATTTGTCTTCAAGAAAAGGACAATAA : 320 Pfragile_NW_ : CTCCAGAGCAAATTATCCGACAATCATCTCTGTTCAACAGTGAAATATACGAACATTTGTCTTCACGAAAAGGATAAAAC : 320 Pcynomolgi_BAEJ : CTCCAGAGCAAATTACCCGACGATCACCTCTGTTCAAAACTGAAATATACGAACATTTGTCTTCAGGAAAAGGACAAAAC : 320 Brazil-I_AFMK _A : CTCCAGAGCAAATTAACCGACGATCATCTGTGTTCAAAAATGAAATATACGAACATTTGTCTTCACGAAAAGGACAAAAC : 320 VCG-I : CTCCAGAGCAAATTAACCGACGATCATCTGTGTTCAAAAATGAAATATACGAACATTTGTCTTCACGAAAAGGACAAAAC : 320 Sal-1_PVX_ : CTCCAGAGCAAATTAACTGACGATCATCTGTGTTCAAAAATGAAATATACGAACATTTGTCTTCACGAAAAGGACAAAAC : 320 Mauritania_I_AFNI : CTCCAGAGCAAATTAACCGACGATCATCTGTGTTCAAAAATGAAATATACGAACATTTGTCTTCACGAAAAGGACAAAAC : 320 NKorean_AFNJ : CTCCAGAGCAAATTAACCGACGATCATCTGTGTTCAAAAATGAAATATACGAACATTTGTCTTCACGAAAAGGACAAAAC : 320 India-VII_AFBK _AFBK : CTCCAGAGCAAATTAACCGACGATCATCTGTGTTCAAAAATGAAATATACGAACATTTGTCTTCACGAAAAGGACAAAAC : 320 CTCCAGAGCAAATTA CcGAcgATCa CT TGTTCaaaA TgAAATATAcgAAcaTTTGTCTTCAcGAAAAGGAcAAaAc * 340 * 360 * 380 * 400 P.coatneyi_CM : TCCCTTGACCTTCCCCTGCACAAGTCCGGAGTACGAAAAATTAATTCAGCAATTTACTTATCAAAAGTTATGCAACTCCA : 400 Pknowlesi_NC_ : TCCCTTGACCTTCCCATGCACAAATCCACAATACGAACAATTAATTCAGCAATTTACTTATAAAAAGTTATGCAACTCCA : 400 Pinui_NW_ : TTCCTTGACCTTCCCCTGCACAAGTCCGGAGTACGAACAATTAATTCACGAATTTACTTATAAAAAGTTATGCAATTCCA : 400 Pfragile_NW_ : TCCCTTGACCTTCCCCTGTACAAGTCCGCAGTACGAACAATTAATTCACCACTTTACTTATAAAAAGTTGTGTAACTCTC : 400 Pcynomolgi_BAEJ : TCCCTTGACCTTACCCTGCACAAGTCCGCAGTACGAACAATTAATTCAACACTTTACTTATAAAAAGTTATGCAACTCCC : 400 Brazil-I_AFMK _A : TCCCTTGACCTTCCCCTGCACAAGTCCGCAGTACGAACAGCTAATTCATCGCTTCACTTATAAAAAGTTGTGCAACTCCA : 400 VCG-I : TCCCTTGACCTTCCCCTGCACAAGTCCGCAGTACGAACAGCTAATTCATCGCTTCACTTATAAAAAGTTGTGCAACTCCA : 400 Sal-1_PVX_ : TCCCTTGACCTTCCCCTGCACAAGTCCGCAGTACGAACAGCTAATTCATCGCTTCACTTATAAAAAGTTGTGCAACTCCA : 400 Mauritania_I_AFNI : TCCCTTGACCTTCCCCTGCACAAGTCCGCAGTACGAACAGCTAATTCATCGCTTCACTTATAAAAAGTTGTGCAACTCCA : 400 NKorean_AFNJ : TCCCTTGACCTTCCCCTGCACAAGTCCGCAGTACGAACAGCTAATTCATCGCTTCACTTATAAAAAGTTGTGCAACTCCA : 400 India-VII_AFBK _AFBK : TCCCTTGACCTTCCCCTGCACAAGTCCGCAGTACGAACAGCTAATTCATCGCTTCACTTATAAAAAGTTGTGCAACTCCA : 400 TcCCTTGACCTTcCCcTGcACAAgTCCgcAgTACGAAcA TAATTCA c TT ACTTATaAAAAGTT TGcAAcTCca * 420 * 440 * 460 * 480 P.coatneyi_CM : AGGTGGCCTTCAGCAACGTGTTGCTGAAATCCTTCATCGATAAGAAGAACGAAGAAAACACATTCAACGCGATCATACAG : 480 Pknowlesi_NC_ : AGGTGGCCTTCAGCAACGTGTTGCTGAAATCCTTCATCGATAAAAAGAACGAAGAAAACACATTTAACGCGATCATACAG : 480 Pinui_NW_ : AGGTGGCCTTTAGCAACGTGTTGCTCAAATCGTTCATCGATAAAAAAAACGAAGAAAACACATTTAACACGATCATAGAG : 480 Pfragile_NW_ : AAGTGGCCTTTAACAACGTGTTGCTCAAATCCTTCATCAATAAAAACAACGAAGAAAACACATTTAACACGATTATACAG : 480 Pcynomolgi_BAEJ : AGGTGGCCTTTAGTAACGTGTTGCTCAAATCCTTCATCAATAAAAAAAACGAAGAAAATACATTTAATACGATCATACAG : 480 Brazil-I_AFMK _A : AGGTGGCCTTTAGCAACGTCTTGCTCAAATCCTTCATCGATAAAAAAAATGAAGAAAACACATTTAACACGATCATACAG : 480 VCG-I : AGGTGGCCTTTAGCAACGTCTTGCTCAAATCCTTCATCGATAAAAAAAATGAAGAAAACACATTTAACACGATCATACAG : 480 Sal-1_PVX_ : AGGTGGCCTTTAGCAACGTCTTGCTCAAATCCTTCATCGATAAAAAAAATGAAGAAAACACATTTAACACGATCATACAG : 480 Mauritania_I_AFNI : AGGTGGCCTTTAGCAACGTCTTGCTCAAATCCTTCATCGATAAAAAAAATGAAGAAAACACATTTAACACGATCATACAG : 480 NKorean_AFNJ : AGGTGGCCTTTAGCAACGTCTTGCTCAAATCCTTCATCGATAAAAAAAATGAAGAAAACACATTTAACACGATCATACAG : 480 India-VII_AFBK _AFBK : AGGTGGCCTTTAGCAACGTCTTGCTCAAATCCTTCATCGATAAAAAAAATGAAGAAAACACATTTAACACGATCATACAG : 480 AgGTGGCCTTtAgcAACGT TTGCTcAAATCcTTCATCgATAAaAA AA GAAGAAAAcACATTtAAcaCGATcATAcAG * 500 * 520 * 540 * 560 P.coatneyi_CM : AATTACAAAGTTCTGTCCACTTGTATTGATGAAGATTTGAAGGATATTTACAATGCATCTATAGAATTATTCTCCGACAT : 560 Pknowlesi_NC_ : AATTACAAAGTTTTGTCCACTTGTATTGATGAAGATTTGAAGGACATTTATAATGCATCCATAGAGTTATTTTCCGATCT : 560 Pinui_NW_ : AATTACAAAGTTCTGTCCACTTGTATTGATGCAGATTTGAAGGACATTTATAGTGCATCCATACAGTTATTCTCCGACCT : 560
77 77 Pfragile_NW_ : AATTACAAAATTCTGTCCACTTGTATTGATGAAGATTTGAAGGACATTTATGATGCGTCCATACAGTTATTCTCCGACAT : 560 Pcynomolgi_BAEJ : AATTACAAACTTCTGTCCACTTGTATTGATGAAGATTTGAAGGACATTTATAATGCATCCATAGATTTATTCTCCGATAT : 560 Brazil-I_AFMK _A : AATTACAAAGTTCTGTCCACTTGCATTGACGATGATTTGAAGGACATTTATAATGCATCCATAGAGTTATTCTCCGACAT : 560 VCG-I : AATTACAAAGTTCTGTCCACTTGCATTGACGATGATTTGAAGGACATTTATAATGCATCCATAGAGTTATTCTCCGACAT : 560 Sal-1_PVX_ : AATTACAAAGTTCTGTCCACTTGCATTGACGATGATTTGAAGGACATTTATAATGCATCCATAGAGTTATTCTCCGACAT : 560 Mauritania_I_AFNI : AATTACAAAGTTCTGTCCACTTGCATTGACGATGATTTGAAGGACATTTATAATGCATCCATAGAGTTATTCTCCGACAT : 560 NKorean_AFNJ : AATTACAAAGTTCTGTCCACTTGCATTGACGATGATTTGAAGGACATTTATAATGCATCCATAGAGTTATTCTCCGACAT : 560 India-VII_AFBK _AFBK : AATTACAAAGTTCTGTCCACTTGCATTGACGATGATTTGAAGGACATTTATAATGCATCCATAGAGTTATTCTCCGACAT : 560 AATTACAAAgTTcTGTCCACTTG ATTGA Ga GATTTGAAGGAcATTTAtaaTGCaTCcATAgAgTTATTcTCCGAcaT * 580 * 600 * 620 * 640 P.coatneyi_CM : TAGAAGCTCCGTCACAGAAATCACGGAAAAGTTGTGGTCCAAAAATATGATCGATGTTTTAAAAACAAGAGAGCAAGCCA : 640 Pknowlesi_NC_ : TAGAACATCTGTCAGAGAAATTACGGAAAAATTGTGGTCCAAAAATATGATCGAGGTTTTAAAAACAAGAGAGCAAGCGA : 640 Pinui_NW_ : TAGAAGCGCCGTCACAGAAATCTCGGAAAGGTTGTGGTCCAAGAATATGATCGATGTTTTAAAGACAAGAGAGGAAACCA : 640 Pfragile_NW_ : TAGAGTGTCCGTCACAGAAATCACAGAAAAGCTGTGGTCTAAAAATATGATCGACGTTTTAAAGACAAGAGAGCAAACCA : 640 Pcynomolgi_BAEJ : TAGAACCTCCGTCACGGAAATCACGGAAAAGTTGTGGTCCAAAAATATGATCGAGGTTTTAAAGGCAAGAGAGCAAACCA : 640 Brazil-I_AFMK _A : AAGAACCTCCGTCACAGAAATTACCGAAAAGTTGTGGTCCAAAAATATGATCGAAGTTTTAAAGACAAGAGAGCAAACCA : 640 VCG-I : AAGAACCTCCGTCACAGAAATTACCGAAAAGTTGTGGTCCAAAAATATGATCGAAGTTTTAAAGACAAGAGAGCAAACCA : 640 Sal-1_PVX_ : AAGAACCTCCGTCACAGAAATTACCGAAAAGTTGTGGTCCAAAAATATGATCGAAGTTTTAAAGACAAGAGAGCAAACCA : 640 Mauritania_I_AFNI : AAGAACCTCCGTCACAGAAATTACCGAAAAGTTGTGGTCCAAAAATATGATCGAAGTTTTAAAGACAAGAGAGCAAACCA : 640 NKorean_AFNJ : AAGAACCTCCGTCACAGAAATTACCGAAAAGTTGTGGTCCAAAAATATGATCGAAGTTTTAAAGACAAGAGAGCAAACCA : 640 India-VII_AFBK _AFBK : AAGAACCTCCGTCACAGAAATTACCGAAAAGTTGTGGTCCAAAAATATGATCGAAGTTTTAAAGACAAGAGAGCAAACCA : 640 AGAa ctccgtcacagaaat ac GAAAagtTGTGGTCcAAaAATATGATCGA GTTTTAAAgaCAAGAGAGcAAaCcA * 660 * 680 * 700 * 720 P.coatneyi_CM : TTGCAGGCATTTTATGTGAGTTAAGAAACGGAAACAATTCTCCCCTAGTATCAAACAGTTTGTCGTATGAAAATTTTGGA : 720 Pknowlesi_NC_ : TTGCAGGTATTTTATGTGAGTTAAGAAACGGAAATAATTCTACCCTAGTATCAAATAGTTTGTCGTATGAAAATTTTGGA : 720 Pinui_NW_ : TTACAGGCATTTTATGTGAGTTAAGAAAGGGAAATAATTCTACTCTAGTATCGAACAGTTTTTCGTATGACAATTTTGGA : 720 Pfragile_NW_ : TTGCAGGTATTTTATGTGAGTTACGAAATGGAAATAAATCAACCCTAGTATCAAACAGTTTGTCATATGAAAATTTTGGG : 720 Pcynomolgi_BAEJ : TTGCAGGCATTTTATGTGAGTTAAGAAACGGAAATAATTCTACCCTAGTATCCAACAGTTTGTCGTATGAAAATTTTGGA : 720 Brazil-I_AFMK _A : TTGCAGGCATTTTATGTGAGTTAAGAAATGGAAATAATTCTCCCCTAGTATCGAACAGTTTTTCCTATGAAAATTTTGGA : 720 VCG-I : TTGCAGGCATTTTATGTGAGTTAAGAAATGGAAATAATTCTCCCCTAGTATCGAACAGTTTTTCCTATGAAAATTTTGGA : 720 Sal-1_PVX_ : TTGCAGGCATTTTATGTGAGTTAAGAAATGGAAATAATTCTCCCCTAGTATCGAACAGTTTTTCCTATGAAAATTTTGGA : 720 Mauritania_I_AFNI : TTGCAGGCATTTTATGTGAGTTAAGAAATGGAAATAATTCTCCCCTAGTATCGAACAGTTTTTCCTATGAAAATTTTGGA : 720 NKorean_AFNJ : TTGCAGGCATTTTATGTGAGTTAAGAAATGGAAATAATTCTCCCCTAGTATCGAACAGTTTTTCCTATGAAAATTTTGGA : 720 India-VII_AFBK _AFBK : TTGCAGGCATTTTATGTGAGTTAAGAAATGGAAATAATTCTCCCCTAGTATCGAACAGTTTTTCCTATGAAAATTTTGGA : 720 TTgCAGGcATTTTATGTGAGTTAaGAAA GGAAAtAAtTCt CcCTAGTATC AAcAGTTT TC TATGAaAATTTTGGa * 740 * 760 * 780 * 800 P.coatneyi_CM : ATCCTCAAGGTTAATTATGAAGCCTTAATGAACCAGGCGTATAAAGCTTTTTCAGACTACTATTCATACTTTCCCGCTTT : 800 Pknowlesi_NC_ : ATCCTTAAAGTGAATTATGAAGGCTTAATAAACCAGGCGTATAAAGCCTTTTCAGACTACTATTCTTACTTTCCCGCGTT : 800 Pinui_NW_ : ATCCTCAAGGTTAATTATGAAGGGTTACTAAACCAGGCGTATGTGGCATTTTCAGACTACTATTCATTCTTTCCCCTTTT : 800 Pfragile_NW_ : ATCCTCAAGGTTAATTATGAAGGGTTAATGAACCAGGCGTACAAGGCATTTTCAGACTACTATTCATACTTTCCCGATTT : 800 Pcynomolgi_BAEJ : ATCCTCAAGGTTAATTATGAAGAGTTACTAAACCAGGCGTATGCGGCGTTTTCAGACTACTATTCATATTTCCCCGCTTT : 800 Brazil-I_AFMK _A : ATTCTCAAGGTTAATTATGAGGGATTACTAAACCAGGCGTATGCGGCCTTTTCAGACTACTATTCATACTTTCCCGCTTT : 800 VCG-I : ATTCTCAAGGTTAATTATGAGGGATTACTAAACCAGGCGTATGCGGCCTTTTCAGACTACTATTCATACTTTCCCGCTTT : 800
78 78 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Sal-1_PVX_ : ATTCTCAAGGTTAATTATGAGGGATTACTAAACCAGGCGTATGCGGCCTTTTCAGACTACTATTCATACTTTCCCGCTTT : 800 Mauritania_I_AFNI : ATTCTCAAGGTTAATTATGAGGGATTACTAAACCAGGCGTATGCGGCCTTTTCAGACTACTATTCATACTTTCCCGCTTT : 800 NKorean_AFNJ : ATTCTCAAGGTTAATTATGAGGGATTACTAAACCAGGCGTATGCGGCCTTTTCAGACTACTATTCATACTTTCCCGCTTT : 800 India-VII_AFBK _AFBK : ATTCTCAAGGTTAATTATGAGGGATTACTAAACCAGGCGTATGCGGCCTTTTCAGACTACTATTCATACTTTCCCGCTTT : 800 AT CTcAAgGTtAATTATGA Gg TTA TaAACCAGGCGTAt ggc TTTTCAGACTACTATTCaTacTTtCCCgctTT * 820 * 840 * 860 * 880 P.coatneyi_CM : TGCCATTAAATTGTTAGAAAAGGGAGGGTTCGTCGAGCGATTGGTCGCTATCCATGAAAGCTTGACCAACTACAGGACGA : 880 Pknowlesi_NC_ : TGCCATTAGACTTTTAGAAAAGGATGGGTTGGTCGAGCGGTTGGTCGCTATCCATGAAAGCTTGACCAACTACAGGACGA : 880 Pinui_NW_ : TGCCATGCGCTTGTTAGAAAATGGCGGATTGGTCGAGCGATTGGTCGCTATCCATGAGAAATTGACCAACTACAGGACGA : 880 Pfragile_NW_ : TGCCATTAAATTGTTGGAGAAAGGTGGGTTGGTCGAGCGGTTGGTCGCTATCCATGAGAGCTTGACCAACTACAGGACGA : 880 Pcynomolgi_BAEJ : TGCCATTAAATTGTTAGAAAAGGGCGGGTTGGTCGAGCGGTTGGTCGCTATCCATGAGAGCTTAACCAACTACAGGACCA : 880 Brazil-I_AFMK _A : TGCCATTAGCATGTTAGAAAAGGGAGGGTTGGTCGACCGCTTGGTCGCCATCCATGAGAGCTTGACCAACTACAGGACGA : 880 VCG-I : TGCCATTAGCATGTTAGAAAAGGGAGGGTTGGTCGACCGCTTGGTCGCCATCCATGAGAGCTTGACCAACTACAGGACGA : 880 Sal-1_PVX_ : TGCCATTAGCATGTTAGAAAAGGGAGGGTTGGTCGACCGCTTGGTCGCCATCCATGAGAGCTTGACCAACTACAGGACGA : 880 Mauritania_I_AFNI : TGCCATTAGCATGTTAGAAAAGGGAGGGTTGGTCGACCGCTTGGTCGCCATCCATGAGAGCTTGACCAACTACAGGACGA : 880 NKorean_AFNJ : TGCCATTAGCATGTTAGAAAAGGGAGGGTTGGTCGACCGCTTGGTCGCCATCCATGAGAGCTTGACCAACTACAGGACGA : 880 India-VII_AFBK _AFBK : TGCCATTAGCATGTTAGAAAAGGGAGGGTTGGTCGACCGCTTGGTCGCCATCCATGAGAGCTTGACCAACTACAGGACGA : 880 TGCCATta TgTTaGAaAAgGg GGgTTgGTCGA CG TTGGTCGC ATCCATGAgAgcTTgACCAACTACAGGACgA * 900 * 920 * 940 * 960 P.coatneyi_CM : AAAATATTCTCAAGAAGATCAATGAGAAGTCCAAAAATGAGGTCCTGAATAACGAGGATATTATGCACAGCTTGAGCAGT : 960 Pknowlesi_NC_ : GAAATATTCTCAAGAAGATCAATGAGAAGTCCAAAAATGAGGTCCTGAATAATGAGGATATTATGCACAGCTTGAGCAGT : 960 Pinui_NW_ : GAAATATTCTCAAGAAGATCAATGACAAGTCCAAAAATGAGGTCCTGAATAACGAGGATATTATGCACAGCTTGAGCAGT : 960 Pfragile_NW_ : AAAATATTCTCCAGAAAATCAATGAGAAGTCAAAAAATGAAGTCCTGAATAACGAGGATATTATGCACAGCTTGAGCAGT : 960 Pcynomolgi_BAEJ : GAAATATTCTCAAGAAGATCAATGAAAAGTCCAAAAATGAGGTCCTGAATAATGGGGATATTATGCACAGCTTGAGCAGT : 960 Brazil-I_AFMK _A : GAAATATTCTCAAGAAGATCAATGAGAAGTCCAAAAATGAGGTCCTCAATAATGAAGAAATTATGCACAGCTTGAGCAGT : 960 VCG-I : GAAATATTCTCAAGAAGATCAATGAGAAGTCCAAAAATGAGGTCCTCAATAATGAAGAAATTATGCACAGCTTGAGCAGT : 960 Sal-1_PVX_ : GAAATATTCTCAAGAAGATCAATGAGAAGTCCAAAAATGAGGTCCTCAATAATGAAGAAATTATGCACAGCTTGAGCAGT : 960 Mauritania_I_AFNI : GAAATATTCTCAAGAAGATCAATGAGAAGTCCAAAAATGAGGTCCTCAATAATGAAGAAATTATGCACAGCTTGAGCAGT : 960 NKorean_AFNJ : GAAATATTCTCAAGAAGATCAATGAGAAGTCCAAAAATGAGGTCCTCAATAATGAAGAAATTATGCACAGCTTGAGCAGT : 960 India-VII_AFBK _AFBK : GAAATATTCTCAAGAAGATCAATGAGAAGTCCAAAAATGAGGTCCTCAATAATGAAGAAATTATGCACAGCTTGAGCAGT : 960 gaaatattctcaagaagatcaatgagaagtccaaaaatgaggtcct AATAA Ga GA ATTATGCACAGCTTGAGCAGT * 980 * 1000 * 1020 * 1040 P.coatneyi_CM : TACAAGCACCATGCCGGGGGCACACGTGGCTCCTTCGTGCAGTCCAGAGGTGTGAGCCAAGTTACGCAAGGCGTCCTGAG : 1040 Pknowlesi_NC_ : TATAAGCACCACGCCGGGGGTACTCGTGGCTCCTTCATGCAGTACAGAGGTGTGGGCCAACTTTCGAAGGGCGGCCTCAG : 1040 Pinui_NW_ : TACAAGCACCATGCCACGGGTACGCGTGGCTCCTTCCTGCAGTCCAGAGGTGTGCGCCAAGTTACGAACAGCAACGTGAG : 1040 Pfragile_NW_ : TACAAGCACCGTGCCGGGAGTACGCGTGGTTCCTTCCTGCAGTCCAGAGGTGCGCACAATGTTACAAAAGTCGATGTGAA : 1040 Pcynomolgi_BAEJ : TACAAGCACCATGCTGGGGGCACGCGTGGCTCCTTCCTGCAGTCCAGAGTTGCGCGCGAAGTTAGGCAAGGCGACGTGAG : 1040 Brazil-I_AFMK _A : TACAAGCACCATGCCGGGGGCACGCGTGGCGCCTTCCTGCAGTCCAGAGATGTGCGCGAAGTTACGCAAGGAGATGTGAG : 1040 VCG-I : TACAAGCACCATGCCGGGGGCACGCGTGGCGCCTTCCTGCAGTCCAGAGATGTGCGCGAAGTTACGCAAGGAGATGTGAG : 1040 Sal-1_PVX_ : TACAAGCACCATGCCGGGGGCACGCGTGGCGCCTTCCTGCAGTCCAGAGATGTGCGCGAAGTTACGCAAGGAGATGTGAG : 1040 Mauritania_I_AFNI : TACAAGCACCATGCCGGGGGCACGCGTGGCGCCTTCCTGCAGTCCAGAGATGTGCGCGAAGTTACGCAAGGAGATGTGAG : 1040 NKorean_AFNJ : TACAAGCACCATGCCGGGGGCACGCGTGGCGCCTTCCTGCAGTCCAGAGATGTGCGCGAAGTTACGCAAGGAGATGTGAG : 1040
79 79 India-VII_AFBK _AFBK : TACAAGCACCATGCCGGGGGCACGCGTGGCGCCTTCCTGCAGTCCAGAGATGTGCGCGAAGTTACGCAAGGAGATGTGAG : 1040 TAcAAGCACCatGCcggGgG ACgCGTGGc CCTTCcTGCAGTcCAGAG TGtGcgC AagTTacg Aagg ga gtgag * 1060 * 1080 * 1100 * 1120 P.coatneyi_CM : CGTCGACGAGAAGGGTGACCCGTCCTCCACCGCGGGG---GGCAACCAAAGCGCAAACATGGCCACGGCGGCCCCCAAC- : 1116 Pknowlesi_NC_ : CGTTGACGAGAAGGGTGGCCAGCAGATTGCCTCCGCGGTGGGCAACCAAAGCGCAAACATGGTCGCGCCGGCTCCTAAGG : 1120 Pinui_NW_ : CGTTGACCAGAAGGGCGATCAGACCGCCACCGCGGGG---GGCAACGAAAGCGAAAACGTGCCCGCGGCGGTCCCCAAGG : 1117 Pfragile_NW_ : GGTTGACGAG ACCTCCACCACGGGG---GGCAACCAAAGCGCAAACATGGCCGCTGCGGCGCCTAACA : 1105 Pcynomolgi_BAEJ : CGTTGACGAGAAGGGTGACCAGACTGCCACCACAGGG---GGCAACCAAAGCGCAAACATGGCCGCGGCGGCCCCCAAGG : 1117 Brazil-I_AFMK _A : CGTTGATGAGAAGGGCGACCGGGCCACCACCGCGGGG---GGCAACCAAAGCGCAAGCGTGGCTGCGGCGGCCCCGAAGG : 1117 VCG-I : CGTTGATGAGAAGGGCGACCGGGCCACCACCGCGGGG---GGCAACCAAAGCGCAAGCGTGGCTGCGGCGGCCCCGAAGG : 1117 Sal-1_PVX_ : CGTTGATGAGAAGGGCGACCGGGCCACCACCGCGGGG---GGCAACCAAAGCGCAAGCGTGGCTGCGGCGGCCCCGAAGG : 1117 Mauritania_I_AFNI : CGTTGATGAGAAGGGCGACCGGGCCACCACCGCGGGG---GGCAACCAAAGCGCAAGCGTGGCTGCGGCGGCCCCGAAGG : 1117 NKorean_AFNJ : CGTTGATGAGAAGGGCGACCGGGCCACCACCGCGGGG---GGCAACCAAAGCGCAAGCGTGGCTGCGGCGGCCCCGAAGG : 1117 India-VII_AFBK _AFBK : CGTTGATGAGAAGGGCGACCGGGCCACCACCGCGGGG---GGCAACCAAAGCGCAAGCGTGGCTGCGGCGGCCCCGAAGG : 1117 cgttga gagaaggg gacc g cc ccacc CgGgG GGCAACcAAAGCGcAA C TGgc gcggcggcccc AAgg * 1140 * 1160 * 1180 * 1200 P.coatneyi_CM : ACGGTTCCTGCTACCAACACGGCTGCTGCTACCACTGCTACT : 1158 Pknowlesi_NC_ : ATTCCTCCCCTACAATG GCTGCTCCTTCCACCGACACTGTTACTAACAACATGGCTACC : 1179 Pinui_NW_ : ATGACGCGCCTACTAGGGTTGCTTCTCCCGGCCCTGCTGCTTTTCCCAACACGGTTGCTTCCAACCCTACTGCTGTTTCC : 1197 Pfragile_NW_ : ATTCGGGCCCTACCACCGCTGCTGCTGCAAACAGTGCTGCTTCCCCAAACACGGCAGCAACTACCAACACTGCGGCAACT : 1185 Pcynomolgi_BAEJ : ATGCGAGCCCTACCACG---GCTGCTTCCAACGCTGCTGCTTCCCCCGACACTACTGCTGCGAACACTGCTGCTGCGAAC : 1194 Brazil-I_AFMK _A : ATGCGGGCCCAACCGTG GCTGCTCCTAACACTGCTGCTACGCTCAAAACGGCT : 1170 VCG-I : ATGCGGGCCCAACCGTG GCTGCTCCTAACACTGCTGCTACGCTCAAAACGGCT : 1170 Sal-1_PVX_ : ATGCGGGCCCAACCGTG GCTGCTCCTAACACTGCTGCTACGCTCAAAACGGCT : 1170 Mauritania_I_AFNI : ATGCGGGCCCAACCGTG GCTGCTCCTAACACTGGTGCTACGCTCAAAACGGCT : 1170 NKorean_AFNJ : ATGCGGGCCCAACCGTG GCTGCTCCTAACACTGCTGCTACGCTCAAAACGGCT : 1170 India-VII_AFBK _AFBK : ATGCGGGCCCAACCGTG GCTGCTCCTAACACTGCTGCTACGCTCAAAACGGCT : 1170 at c ccc ac g gct ctcc acac g gct c ca ac gct * 1220 * 1240 * 1260 * 1280 P.coatneyi_CM : ACTACGAACACGGCAACTAATACCAACACGGCA ACTAATACCAACACGGCTACT---AATAC : 1217 Pknowlesi_NC_ : ACTACAACCGCGCCTGCTACTGCCAACATGGCTGCTCCTTCCACCAACACTGCTACTGCCAACATGGCTGCT---CCTTC : 1256 Pinui_NW_ : AACGCTACTGCTGCTTCCAACGCTACTGCTGCTTCCAACGCTACTGCTGCTTCCAACGCTACTGAT GCTTC : 1268 Pfragile_NW_ : ACCAGC GCTTC : 1196 Pcynomolgi_BAEJ : ACCGCTGCTGCGAACACCGCTGCTGCTCCGAACACTGCTTCTGCTTCCAACGCTGCTGCTACTCCCAACCCTGCTGCTGC : 1274 Brazil-I_AFMK _A : GCTTC : 1175 VCG-I : GCTTC : 1175 Sal-1_PVX_ : GCTTC : 1175 Mauritania_I_AFNI : GCTTC : 1175 NKorean_AFNJ : GCTTC : 1175 India-VII_AFBK _AFBK : GCTTC : 1175 gcttc
80 80 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax * 1300 * 1320 * 1340 * 1360 P.coatneyi_CM : CAACAGTGCAGCTACTACCAACACGGCCGCT ACTACCAACACGGCCGCTACTACCAACACGA : 1279 Pknowlesi_NC_ : CACCAACACTGCTACTGCCAACATGGCTGCTCCTTCCACCGACACTGTAACTACCAACATGGCTACCACTACAACCGTGC : 1336 Pinui_NW_ : CAACGCTACTGCTGCTTCCAACCCTGCTGCT C : 1300 Pfragile_NW_ : CAACGATACTAATGCTTCTCCCCACACGAAT A : 1228 Pcynomolgi_BAEJ : TCCGAACACTGCTGCTACTCCCAACTCTGCT A : 1306 Brazil-I_AFMK _A : CCCCAACGCGGCTGCTACTAACACTGCTGCT C : 1207 VCG-I : CCCCAACGCGGCTGCTACTAACACTGCTGCT C : 1207 Sal-1_PVX_ : CCCCAACGCGGCTGCTACTAACACTGCTGCT C : 1207 Mauritania_I_AFNI : CCCCAACGCGGCTGCTACTAACACTGCTGCT C : 1207 NKorean_AFNJ : CCCCAACGCGGCTGCTACTAACACTGCTGCT C : 1207 India-VII_AFBK _AFBK : CCCCAACGCGGCTGCTACTAACACTGCTGCT C : 1207 c caa C gctgct C aaca gctgct * 1380 * 1400 * 1420 * 1440 P.coatneyi_CM : CCGCTACTACCAGTGTAGCTACTTCCCCCCTGAACACC CCCCTGTACGGCACCAGCTCC : 1338 Pknowlesi_NC_ : CTGCTACTGCCAACACGGCTGCTTCCCCC---GACACTAATACTGATTCTTCTACTTACCCCCTGTATGGCACGGAATCC : 1413 Pinui_NW_ : CTCCCAACACTGCTGCT---ACTTCCCAA---CTGAGCAAC CCCCTGTATGGCATCAGCTCC : 1356 Pfragile_NW_ : CTTCCAACGAGGGTGCTTCCACCAGTCCC---CTGAGCAAC CCCCTGTACGGCACCCGCTAC : 1287 Pcynomolgi_BAEJ : CCCCCAACGCTGCTGCT---ACTTCCCCA---CTGAGCACC CCCCTGTACGACACAAGCTCC : 1362 Brazil-I_AFMK _A : CCCCCAACATGGGTGCC---ACCTCCCCG---CTGAGCAAC CCCCTGTACGGCACCAGCTCC : 1263 VCG-I : CCCCCAACATGGGTGCC---ACCTCCCCG---CTGAGCAAC CCCCTGTACGGCACCAGCTCC : 1263 Sal-1_PVX_ : CCCCCAACATGGGTGCC---ACCTCCCCG---CTGAGCAAC CCCCTGTACGGCACCAGCTCC : 1263 Mauritania_I_AFNI : CCCCCAACATGGGTGCC---ACCTCCCCG---CTGAGCAAC CCCCTGTACGGCACCAGCTCC : 1263 NKorean_AFNJ : CCCCCAACATGGGTGCC---ACCTCCCCG---CTGAGCAAC CCCCTGTACGGCACCAGCTCC : 1263 India-VII_AFBK _AFBK : CCCCCAACATGGGTGCC---ACCTCCCCG---CTGAGCAAC CCCCTGTACGGCACCAGCTCC : 1263 C CcAac g tgc ac tcccc ctgagcaac CCCCTGTAcGgCAccagcTcC * 1460 * 1480 * 1500 * 1520 P.coatneyi_CM : TCGAGGACAAAGGACGTCGTAGTGCTGGTCAGAGATCTGCTCAAAAACACAAACATCATAAAATTCGAGAAGAACGAACC : 1418 Pknowlesi_NC_ : TCGAAGACAAAGGACGTCGTAGTTCTAGTCAGAGATCTTCTCAAAGACACAAACATCATCAAGTTCGAGAAGAACGAACC : 1493 Pinui_NW_ : TTGAATACAAAGGACGTCGTAGTGCTGGTCAGAGATCTGCTCAAGAACACAAACATCATCAAGTTCGACAAGGACGAACC : 1436 Pfragile_NW_ : TTGAAGACAAAGGACGTCGTAGTGCTCATCAGAGATCTGCTCAAAAACACAAACATCATCAAGTTCGAGAAGGACGAACC : 1367 Pcynomolgi_BAEJ : TTGAAGACAAAGGACGTCGTAGTGCTCGTCAGAGATCTGCTCAAAAACACAAACATCATCAAGTTCGAGAAGGACGAACC : 1442 Brazil-I_AFMK _A : CTGCAGCCAAAGGACGTCGCGGTGCTGGTCAGAGATCTGCTCAAGAACACGAACATCATCAAGTTCGAGAATAACGAACC : 1343 VCG-I : CTGCAGCCAAAGGACGTCGCGGTGCTGGTCAGAGATCTGCTCAAGAACACGAACATCATCAAGTTCGAGAATAACGAACC : 1343 Sal-1_PVX_ : CTGCAGCCAAAGGACGTCGCGGTGCTGGTCAGAGATCTGCTCAAGAACACGAACATCATCAAGTTCGAGAATAACGAACC : 1343 Mauritania_I_AFNI : CTGCAGCCAAAGGACGTCGCGGTGCTGGTCAGAGATCTGCTCAAGAACACGAACATCATCAAGTTCGAGAATAACGAACC : 1343 NKorean_AFNJ : CTGCAGCCAAAGGACGTCGCGGTGCTGGTCAGAGATCTGCTCAAGAACACGAACATCATCAAGTTCGAGAATAACGAACC : 1343 India-VII_AFBK _AFBK : CTGCAGCCAAAGGACGTCGCGGTGCTGGTCAGAGATCTGCTCAAGAACACGAACATCATCAAGTTCGAGAATAACGAACC : 1343 tg ag CAAAGGACGTCG GTgCT gtcagagatctgctcaa aacac AACATCATcAAgTTCGAgAA ACGAACC * 1540 * 1560 * 1580 * 1600 P.coatneyi_CM : GACTAGCCAAATAGACGATGAAGGAATTAAGAAGCTCATAGAGAGTTCCTTCTTCGACCTAAGCGACAACACCATGTTAA : 1498 Pknowlesi_NC_ : GACTAGCCAAATAGACGATGAAGGAATTAAGAAGCTCATAGAGAGTTCGTTCTTCGACTTGAGCGACAACACCATGTTAA : 1573
81 81 Pinui_NW_ : GACTAGTCAAATGGACGATGAAGGAATTAAGAAGCTCATAGAGAGTTCCTTCTTCGACTTGAGCGACAACACCATGTTAA : 1516 Pfragile_NW_ : AACTGGCCAAATTGACCATGAAGGAATTAAAAAACTCATAGAAAGTTCCTTCTTCGACCTGAGCGACAACACCATGTTAA : 1447 Pcynomolgi_BAEJ : GACTAGCCAAATAGACGAGGAAGGAATTAAGAAGCTCATAGAGAGTTCCTTCTTCGACTTGAGCGACAACACCATGTTAA : 1522 Brazil-I_AFMK _A : GACTAGCCAAATGGACGATGAAGAAATTAAGAAGCTCATTGAGAGCTCCTTTTTCGACTTGAGCGACAACACCATGTTAA : 1423 VCG-I : GACTAGCCAAATGGACGATGAAGAAATTAAGAAGCTCATTGAGAGCTCCTTTTTCGACTTGAGCGACAACACCATGTTAA : 1423 Sal-1_PVX_ : GACTAGCCAAATGGACGATGAAGAAATTAAGAAGCTCATTGAGAGCTCCTTTTTCGACTTGAGCGACAACACCATGTTAA : 1423 Mauritania_I_AFNI : GACTAGCCAAATGGACGATGAAGAAATTAAGAAGCTCATTGAGAGCTCCTTTTTCGACTTGAGCGACAACACCATGTTAA : 1423 NKorean_AFNJ : GACTAGCCAAATGGACGATGAAGAAATTAAGAAGCTCATTGAGAGCTCCTTTTTCGACTTGAGCGACAACACCATGTTAA : 1423 India-VII_AFBK _AFBK : GACTAGCCAAATGGACGATGAAGAAATTAAGAAGCTCATTGAGAGCTCCTTTTTCGACTTGAGCGACAACACCATGTTAA : 1423 gactagccaaat GACgAtGAAG AATTAAgAAgCTCAT GAgAG TCcTT TTCGACtTgAGCGACAACACCATGTTAA * 1620 * 1640 * 1660 * 1680 P.coatneyi_CM : TGCGCTTGCTCATAAAGCCGCAGGCAGCCATCCTATTCATCATTCAGTCCTTCATTATGATGACGCCGTCCCCCACAAGA : 1578 Pknowlesi_NC_ : TGCGATTGATCATAAAGCCGCAGGCTTCCATCTTATTCATTATCCAGTCCTTCATTATGATGACGCCATCCCCCACGAGA : 1653 Pinui_NW_ : TGCAGTTGCTCATAAAGCCGCAGGCAGCCATGTTATTCATCATTCAGTCCTTCATTATGATGACACCCTCCCCCACGCGA : 1596 Pfragile_NW_ : TGAGGTTGCTCATAAAGCCGCAGGCAGCCATCTTATTCATCATTCAGTCCTTTATCATGATGACACCGTCGCCCACAAGA : 1527 Pcynomolgi_BAEJ : TGCGATTGCTCATAAAGCCGCAGGCAGCCATCTTATTCATCATTCAGTCCTTCATTATGATGACGCCGTCCCCCACGCGA : 1602 Brazil-I_AFMK _A : TGCGGTTGCTCATAAAGCCGCAGGCGGCCATCTTACTAATCATTGAGTCCTTCATTATGATGACGCCCTCCCCCACGAGG : 1503 VCG-I : TGCGGTTGCTCATAAAGCCGCAGGCGGCCATCTTACTAATCATTGAGTCCTTCATTATGATGACGCCCTCCCCCACGAGG : 1503 Sal-1_PVX_ : TGCGGTTGCTCATAAAGCCGCAGGCGGCCATCTTACTAATCATTGAGTCCTTCATTATGATGACGCCCTCCCCCACGAGG : 1503 Mauritania_I_AFNI : TGCGGTTGCTCATAAAGCCGCAGGCGGCCATCTTACTAATCATTGAGTCCTTCATTATGATGACGCCCTCCCCCACGAGG : 1503 NKorean_AFNJ : TGCGGTTGCTCATAAAGCCGCAGGCGGCCATCTTACTAATCATTGAGTCCTTCATTATGATGACGCCCTCCCCCACGAGG : 1503 India-VII_AFBK _AFBK : TGCGGTTGCTCATAAAGCCGCAGGCGGCCATCTTACTAATCATTGAGTCCTTCATTATGATGACGCCCTCCCCCACGAGG : 1503 TGcg TTGcTCATAAAGCCGCAGGC gccatctta T ATcATt AGTCCTTcATtATGATGACgCC TCcCCCACgaG * 1700 * 1720 * 1740 * 1760 P.coatneyi_CM : GACGCCAAAATGTATTGCAAGAAAAAGCTAGTTAATGGCCAGCTAATAGAAAACTCCGACTTAAAAGCTGCGACGGAGGA : 1658 Pknowlesi_NC_ : GACGCCAGAATGTATTGCAAGAAAAAACTAGTTAATGGCCAGTTAATAGAAAACAACGATTTAAAGGCTGAGACAGAGGA : 1733 Pinui_NW_ : GACGCCAAAACCTATTGCAACAAAAGCCTAGTTAATGGCAAGCTAATCGAAACCTCCGATATAAAGGCTTCGACGGAGGA : 1676 Pfragile_NW_ : GACGCCAAAATTTATTGCAAGAAAAATCTAGTTAATGGCCAGCTAGTACAAACCTCTGATTTCAGCGATGCGCACGAGGA : 1607 Pcynomolgi_BAEJ : GACGCCAAAATCTATTGCAAGAAAGTCCTAGTTAATGGTCAGCTAGTCGAAACCTCCGATTTAAGCGCTGCGACGGAGGA : 1682 Brazil-I_AFMK _A : GACGCCAAGACCTATTGCAAGAAAGCCCTAGTTAATGGCCAGCTAATCGAAACCTCAGATTTAAACGCGGCGACGGAGGA : 1583 VCG-I : GACGCCAAGACCTATTGCAAGAAAGCCCTAGTTAATGGCCAGCTAATCGAAACCTCAGATTTAAACGCGGCGACGGAGGA : 1583 Sal-1_PVX_ : GACGCCAAGACCTATTGCAAGAAAGCCCTAGTTAATGGCCAGCTAATCGAAACCTCAGATTTAAACGCGGCGACGGAGGA : 1583 Mauritania_I_AFNI : GACGCCAAGACCTATTGCAAGAAAGCCCTAGTTAATGGCCAGCTAATCGAAACCTCAGATTTAAACGCGACGACGGAGGA : 1583 NKorean_AFNJ : GACGCCAAGACCTATTGCAAGAAAGCCCTAGTTAATGGCCAGCTAATCGAAACCTCAGATTTAAACGCGGCGACGGAGGA : 1583 India-VII_AFBK _AFBK : GACGCCAAGACCTATTGCAAGAAAGCCCTAGTTAATGGCCAGCTAATCGAAACCTCAGATTTAAACGCGGCGACGGAGGA : 1583 GACGCCAa A TATTGCAAgAAA CTAGTTAATGGccAGcTAaT gaaacctc GAttTaAa Gc gcgacggagga * 1780 * 1800 * 1820 * 1840 P.coatneyi_CM : AGAAGACATGATAACCGAGTTTTCCAGCAAGTACAATTTATTCTACGAAAGGCTGAAACTGGAGGAGTTGCGCGAAATTG : 1738 Pknowlesi_NC_ : AGAAGATATGATAAACGAATTTTCCAGTAAGTACAATTTATTCTATGAAAGGCTTAAGATGGAGGAGTTGCGCGAAATTG : 1813 Pinui_NW_ : AGAAGACCTGATAACCGAGTTTTCCAGCAAGTACAATTTATTCTACGAAAAGCTGAAGATGGAAGAGTTGCGAGAAATTG : 1756 Pfragile_NW_ : AGAAGATCTGATAAACGAATTTTCCAGCAAATACAATTTATTCTACGAAAGGTTGAAGCTGGAGGAGTTGCGCGAAATTG : 1687 Pcynomolgi_BAEJ : AGAAGATCTGATAAATGAGTTTTCCAGCAAGTACAATTTATTCTACGAAAGGCTGAAACTGGAGGAGTTGCGCGAAATTG : 1762
82 82 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Brazil-I_AFMK _A : AGACGACCTCATAAACGAGTTTTCCAGCAGGTACAATTTATTCTACGAGAGGCTCAAGCTGGAGGAGTTGCGCGAAATTG : 1663 VCG-I : AGACGACCTCATAAACGAGTTTTCCAGCAGGTACAATTTATTCTACGAGAGGCTCAAGCTGGAGGAGTTGCGCGAAATTG : 1663 Sal-1_PVX_ : AGACGACCTCATAAACGAGTTTTCCAGCAGGTACAATTTATTCTACGAGAGGCTCAAGCTGGAGGAGTTGCGCGAAATTG : 1663 Mauritania_I_AFNI : AGACGACCTCATAAACGAGTTTTCCAGCAGGTACAATTTATTCTACGAGAGGCTCAAGCTGGAGGAGTTGCGCGAAATTG : 1663 NKorean_AFNJ : AGACGACCTCATAAACGAGTTTTCCAGCAGGTACAATTTATTCTACGAGAGGCTCAAGCTGGAGGAGTTGCGCGAAATTG : 1663 India-VII_AFBK _AFBK : AGACGACCTCATAAACGAGTTTTCCAGCAGGTACAATTTATTCTACGAGAGGCTCAAGCTGGAGGAGTTGCGCGAAATTG : 1663 AGA GA ct ATAAacGAgTTTTCCAGcA gtacaatttattctacga AgGcT AAgcTGGAgGAGTTGCGcGAAATTG * 1860 * 1880 * 1900 * 1920 P.coatneyi_CM : AACAGAACAGGAAAGCGCTCAAGAATTCGAAGGGAAGTCTGTCCGTGCTGCAGGTCGGCAACTCGCAGAATGGTCCAGAC : 1818 Pknowlesi_NC_ : AACAGGACAGAAAATCGCTAAAGAATTCCAAGGGCAACTTGTCTGTGTTGGAGGTGCGCAACTCCCAGAATGGTCCCGAC : 1893 Pinui_NW_ : AAGAGAACAAGAAAGCTATCAAGAATTCGAAGGGCAGCCTGTCCGTGCTGGAGGTCGCCAACTCGCAAAATAGTCCCGAC : 1836 Pfragile_NW_ : AACAGAACAGGAAAGCGCTCAAGAATGCGAAGGGTAGCCTCTCCGTGCTGCAGGTCAGCAATTCGCAGAATGGTACCCAC : 1767 Pcynomolgi_BAEJ : AACAGAACAGGAAAGCGCTCAAGAATTCGAAGGGCAGCCTGTCCGTGCTGGAGGTAGGCAACTCGCAGAATGGTCCAGAC : 1842 Brazil-I_AFMK _A : AACAGAACAGGAAAGCGCTCAAGAATTCGAAGGGCACCCTGTCCGTGCTGGAGGTCGCCAACTCGCAGAACGCCCCCGAC : 1743 VCG-I : AACAGAACAGGAAAGCGCTCAAGAATTCGAAGGGCACCCTGTCCGTGCTGGAGGTCGCCAACTCGCAGAACGCCCCCGAC : 1743 Sal-1_PVX_ : AACAGAACAGGAAAGCGCTCAAGAATTCGAAGGGCACCCTGTCCGTGCTGGAGGTCGCCAACTCGCAGAACGCCCCCGAC : 1743 Mauritania_I_AFNI : AACAGAACAGGAAAGCGCTCAAGAATTCGAAGGGCACCCTGTCCGTGCTGGAGGTCGCCAACTCGCAGAACGCCCCCGAC : 1743 NKorean_AFNJ : AACAGAACAGGAAAGCGCTCAAGAATTCGAAGGGCACCCTGTCCGTGCTGGAGGTCGCCAACTCGCAGAACGCCCCCGAC : 1743 India-VII_AFBK _AFBK : AACAGAACAGGAAAGCGCTCAAGAATTCGAAGGGCACCCTGTCCGTGCTGGAGGTCGCCAACTCGCAGAACGCCCCCGAC : 1743 AAcAGaACAggAAAgCgcTcAAGAATtCgAAGGGcA cctgtccgtgctggaggtcg CAAcTCgCAgAA g cccgac * 1940 * 1960 * 1980 * 2000 P.coatneyi_CM : GGGAAGGAGGCCAACGGAAGTGGAAACGCCGCAAATGGA : 1857 Pknowlesi_NC_ : GGGAAGGAAGTCAACGGAAGCGGAGATGCCGCAAATGGA : 1932 Pinui_NW_ : GGGAAGAGGGTCAACGGAAGCGGAAACGACGCGAATGGA : 1875 Pfragile_NW_ : GGGAACGGGGTCAATCAAGGTGGACACGCTGCAAATGAA : 1806 Pcynomolgi_BAEJ : GGGAGCGGAAACAACGGAAGCGGAAACGCCGCAAATGGA : 1881 Brazil-I_AFMK _A : GGGAAGGGCGTCAACGGGAGCGGAAACGCAGCAAATGCGAACGCCGCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 1823 VCG-I : GGGAAGGGCGTCAACGGGAGCGGAAACGCAGCAAATGCGAACGCCGCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 1823 Sal-1_PVX_ : GGGAAGGGCGTCAACGGGAGCGGAAACGCAGCAAATGCGAACGCCGCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 1823 Mauritania_I_AFNI : GGGAAGGGCGTCAACGGGAGCGGAAACGCAGCAAATGCC : 1782 NKorean_AFNJ : GGGAAGGGCGTCAACGGGAGCGGAAACGCAGCAAATGCGAACGCCGCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 1823 India-VII_AFBK _AFBK : GGGAAGGGCGTCAACGGGAGCGGAAACGCAGCAAATGCGAACGCCGCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 1823 GGGAaggg gtcaacgg agcggaaacgc GCaAATG * 2020 * 2040 * 2060 * 2080 P.coatneyi_CM : : - Pknowlesi_NC_ : : - Pinui_NW_ : : - Pfragile_NW_ : : - Pcynomolgi_BAEJ : : - Brazil-I_AFMK _A : TGCAAACGCCGCAAATGGGAACCTCGCAAATGCAAACCTCGCAAACGCAAAC : 1875 VCG-I : TGCAAACGCCGCAAATGGGAACCTCGCAAATGCAAACCTCGCAAACGCAAACGCCGCAAACGCAGACGCCGCAAACGCAA : 1903 Sal-1_PVX_ : TGCAAACGCCGCAAATGGGAACCTCGCAAATGCAAACCTCGCAAACGCAAACGCCGCAAACGCAGACGCCGCAAACGCAA : 1903
83 83 Mauritania_I_AFNI : : - NKorean_AFNJ : TGCAAACGCCGCAAATGNNN AAATGCAAACCTCGCAAACGCAAACGCCGCAAACGCAAACGCCGCAAACGCAA : 1893 India-VII_AFBK _AFBK : TGCAAACGCCGCAAATNNGAACCTCGCAAATGCAAACCTCGCAAACGCAAACGCCGCA : 1879 * 2100 * 2120 * 2140 * 2160 P.coatneyi_CM : : - Pknowlesi_NC_ : : - Pinui_NW_ : : - Pfragile_NW_ : : - Pcynomolgi_BAEJ : : - Brazil-I_AFMK _A : : - VCG-I : ACGCCGCAAACGCAAACGCCGCAAACGCAAACGCCGCAAATGCAAACCTCGCAAATGCAAACCTCGCAAATGCAAACCTC : 1983 Sal-1_PVX_ : ACGCCGCAAACGCAAACGCCGCTAATGCAAACGCCGCAAATGCAAACCTCGCAAATGCAAACCTCGCAAATGCAAACCTC : 1983 Mauritania_I_AFNI : : - NKorean_AFNJ : ACGCCGCAAACGCAAACGCCGCTAATGCAAACGCCGCAAATGGG : 1937 India-VII_AFBK _AFBK : AATGGG : 1885 * 2180 * 2200 * 2220 * 2240 P.coatneyi_CM : : - Pknowlesi_NC_ : : - Pinui_NW_ : : - Pfragile_NW_ : AG : 1808 Pcynomolgi_BAEJ : AACAACGCAAATGGGAACAACGCAAA : 1907 Brazil-I_AFMK _A : GCAAATGCAAACCTCGCAAATGCAAACCTCGCAAATGCAAACCTCGCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 1955 VCG-I : GCAAATGCAAACCTCGCAAATGCAAACCTCGCAAATGCAAACCTCGCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 2063 Sal-1_PVX_ : GCAAATGCAAACCTCGCAAATGCAAACCTCGCAAATGCAAACCTCGCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 2063 Mauritania_I_AFNI : GCAAACGCAAACGCCGCAAACGCAAACGCCGCTAA : 1817 NKorean_AFNJ : : - India-VII_AFBK _AFBK : : - * 2260 * 2280 * 2300 * 2320 P.coatneyi_CM : AACGCCCAAAATGGAAACGGCGCAAATGTAAATGCTCAAAATGAAAACGCCCCAAATGGAA : 1918 Pknowlesi_NC_ : AACAACATGAATGGA GGTA : 1951 Pinui_NW_ : AACGCCGCAAATGGAA : 1891 Pfragile_NW_ : CGGACATGCGGCAAATGAC AACGCACATGCTGCAA : 1843 Pcynomolgi_BAEJ : TGGAAACAACGCAAATGGA AACGCCGCAAATGGAA : 1942 Brazil-I_AFMK _A : TGCAAACGCCGCAAATGGG AACGCCCCAAATAGCA : 1990 VCG-I : TGCAAACGCCGCAAATGGG AACGCCCCAAATAGCA : 2098 Sal-1_PVX_ : TGCAAACGCCGCAAATGGG AACGCCCCAAATAGCA : 2098 Mauritania_I_AFNI : TGCAAACGCCGCAAATGGG AACGCCCCAAATAGCA : 1852 NKorean_AFNJ : AACGCCCCAAATAGCA : 1953 India-VII_AFBK _AFBK : AACGCCCCAAATAGCA : 1901 aacgcc caaat g A
84 84 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax * 2340 * 2360 * 2380 * 2400 P.coatneyi_CM : ACAACGGAAGCGCCTCCCCCCTCATCGTAGTGGTAGGGGACGAGCTCGGAGAAAAAACCGACGATATCATCAAAAAAAAT : 1998 Pknowlesi_NC_ : ACAACGGAAGCGCCTCCTCCCTCATCGTAGTAGTAAGGGACGATCTCGCTGAAAAAACTGATGACATCATCAAAAATAAT : 2031 Pinui_NW_ : ACAACGGAAGCGGCTCCCCCCTCCTCGTAGTGGTAGGGGCCGATCTCGGCGAAAAAACCGAAGACATCATCAAGAACAAT : 1971 Pfragile_NW_ : ATGACNNN : 1848 Pcynomolgi_BAEJ : ACAACGGAAACGGATCCCCCCTCATCGCAGTGGTAGGGGCCGATCTCGGCGAAAAAACTGACGACATCATCAAGAACAAT : 2022 Brazil-I_AFMK _A : ACAACGGAAGCGGATCCCCCCTCATCGTAGTGGTAGGGGCCGATCTCGGCGAAAAAACCGAGGACATCATCAAGAACAAC : 2070 VCG-I : ACAACGGAAGCGGATCCCCCCTCATCGTAGTGGTAGGGGCCGATCTCGGCGAAAAAACCGAGGACATCATCAAGAACAAC : 2178 Sal-1_PVX_ : ACAACGGAAGCGGATCCCCCCTCATCGTAGTGGTAGGGGCCGATCTCGGCGAAAAAACCGAGGACATCATCAAGAACAAC : 2178 Mauritania_I_AFNI : ACAACGGAAGCGGATCCCCCCTCATCGTAGTGGTAGGGGCCGATCTCGGCGAAAAAACCGAGGACATCATCAAGAACAAC : 1932 NKorean_AFNJ : ACAACGGAAGCGGATCCCCCCTCATCGTAGTGGTAGGGGCCGATCTCGGCGAAAAAACCGAGGACATCATCAAGAACAAC : 2033 India-VII_AFBK _AFBK : ACAACGGAAGCGGATCCCCCCTCATCGTAGTGGTAGGGGCCGATCTTGGCGAAAAAACCGAGGACATCATCAAGAACAAC : 1981 AcaACggaagcg tcccccctcatcgtagtggtagggg cgatctcgg gaaaaaac ga gacatcatcaa aa aa * 2420 * 2440 * 2460 * 2480 P.coatneyi_CM : GTGGACGTGGAAGCCTTGACAGCCGACGTGGAACAAGCCTTCAAAAGCTTCGAATACCAAAGTGGAAGTTTCTCTGCAAA : 2078 Pknowlesi_NC_ : GTGGACCTGGAATCCTTAAAAGCCGACGTGGAACAAGCCTTTAGAAACTTCGAATACCAAAGTGGAAGTTTCTCGGCAAA : 2111 Pinui_NW_ : GTGGACGTGGCAACCTTGACAGAAGGCGTGGAACAAGCCTTTAAAAACCTCTCATTGGAAGGTGGAAGTTTCTCGGCAAA : 2051 Pfragile_NW_ : : - Pcynomolgi_BAEJ : GTGGACGTGGCAGCCTTGGCAGCCGACGTGGAACAAGCCTTTAGAAACCTCGAATTGAAAAGTGGAAGCTTCTCGGCAAA : 2102 Brazil-I_AFMK _A : GTGGACGTGGCAGCCTTGACAGCCGACGTGGAACAAGCCTTTAAAAACCTCGAATTGCAGAGTGGAAGCTTCTCGGCAAA : 2150 VCG-I : GTGGACGTGGCAGCCTTGACAGCCGACGTGGAACAAGCCTTTAAAAACCTCGAATTGCAGAGTGGAAGCTTCTCGGCAAA : 2258 Sal-1_PVX_ : GTGGACGTGGCAGCCTTGACAGCCGACGTGGAACAAGCCTTTAAAAACCTCGAATTGCAGAGTGGAAGCTTCTCGGCAAA : 2258 Mauritania_I_AFNI : GTGGACGTGGCAGCCTTGACAGCCGACGTGGAACAAGCCTTTAAAAACCTCGAATTGCAGAGTGGAAGCTTCTCGGCAAA : 2012 NKorean_AFNJ : GTGGACGTGGCAGCCTTGACAGCCGACGTGGAACAAGCCTTTAAAAACCTCGAATTGCAGAGTGGAAGCTTCTCGGCAAA : 2113 India-VII_AFBK _AFBK : GTGGACGTGGCAGCCTTGACAGCCGACGTGGAACAAGCCTTTAAAAACCTCGAATTGCAGAGTGGAAGCTTCTCGGCAAA : 2061 gtggacgtgg a ccttgacagccgacgtggaacaagccttta aaac tcgaat a agtggaag ttctcggcaaa * 2500 * 2520 * P.coatneyi_CM : CTTATCTCACGCCCTGGTTCTCCTCTCCTCCATTGCGTTGCTCTTATTCATTTGT : 2133 Pknowlesi_NC_ : CTTATCTCACGCCCTGGTTCTCCTATCCTCCATTGCGTTAGTCCTATTCATTTGT : 2166 Pinui_NW_ : CTTATCGCACGCGCTGGTTCTCCTCTCTTCCATTGCTTTACTCCTATTCATTTGT : 2106 Pfragile_NW_ : : - Pcynomolgi_BAEJ : CTTATCGCACGCGCTGGTTCTCCTCTCTTCCATTGCGTTACTCCTATTCATTTTT : 2157 Brazil-I_AFMK _A : CTTATCGCACGCGCTGGTTCTCCTCTCCTCCATTGCGTTGCTCCTATTCATTTTT : 2205 VCG-I : CTTATCGCACGCGCTGGTTCTCCTCTCCTCCATTGCGTTGCTCCTATTCATTTTT : 2313 Sal-1_PVX_ : CTTATCGCACGCGCTGGTTCTCCTCTCCTCCATTGCGTTGCTCCTATTCATTTTT : 2313 Mauritania_I_AFNI : CTTATCGCACGCGCTGGTTCTCCTCTCCTCCATTGCGTTGCTCCTATTCATTTTT : 2067 NKorean_AFNJ : CTTATCGCACGCGCTGGTTCTCCTCTCCTCCATTGCGTTGCTCCTATTCATTTTT : 2168 India-VII_AFBK _AFBK : CTTATCGCACGCGCTGGTTCTCCTCTCCTCCATTGCGTTGCTCCTATTCATTTTT : 2116 cttatc cacgc ctggttctcctctc tccattgcgtt ctcctattcattt t
85 85 B. Alineamiento deducido de la secuencia de aminoácidos * 20 * 40 * 60 * 80 P.coatneyi_CM : MKCNGSLLVLLSAILSATNALIRNGNNPQALVPEKSDDSSGGQNKPSGDNQDTCEIQKMAEEMMEKMVNEKDVFTSIMEPL : 81 Pknowlesi_NC_ : MKCNGSLLVLLSAILSATNALIRNGNNPQALVPDNSGDPNAGQNKTSSDNQDTCEIQKMAEEMMEKMMKEKDVFSSIMEPL : 81 Pinui_NW_ : MKCNTALLVLLSAILSAANALIRNGNTTQALVPEKSNDPSGGQNNPSGDNRETCEIQQMAQEMMEKMMKEKDVFSSIMEPL : 81 Pfragile_NW_ : MKYNATLLVLLSAILSAGKALIRNGNNTQALVPENGGDPSGGQNNPSGENQDTCEVQKMAEEMMGKMMKEKDVFSSIMEPL : 81 Pcynomolgi_BAEJ : MKCNAALLVLLSALLSASNALIRNGNNTQALVPEKSGDPSGGQNNPSGDNQDTCEIQKMAEEMMEKMMKEKDMFSSIMEPL : 81 Brazil-I_AFMK _A : MKCNASLLVLLSALLSAANALIRNGNNPQALVPEKGADPSGGQNNRSGENQDTCEIQKMAEEMMEKMMKEKDVFSSIMEPL : 81 VCG-I : MKCNASLLVLLSALLSAANALIRNGNNPQALVPEKGADPSGGQNNRSGENQDTCEIQKMAEEMMEKMMKEKDVFSSIMEPL : 81 Sal-1_PVX_ : MKCNASLLVLLSALLSAANALIRNGNNPQALVPEKGADPSGGQNNRSGENQDTCEIQKMAEEMMEKMMKEKDVFSSIMEPL : 81 Mauritania_I_AFNI : MKCNASLLVLLSALLSAANALIRNGNNPQALVPEKGADPSGGQNNRSGENQDTCEIQKMAEEMMEKMMKEKDVFSSIMEPL : 81 N.Korean_AFNJ : MKCNASLLVLLSALLSAANALIRNGNNPQALVPEKGADPSGGQNNRSGENQDTCEIQKMAEEMMEKMMKEKDVFSSIMEPL : 81 India-VII_AFBK _AFBK : MKCNASLLVLLSALLSAANALIRNGNNPQALVPEKGADPSGGQNNRSGENQDTCEIQKMAEEMMEKMMKEKDVFSSIMEPL : 81 MKcN LLVLLSA6LSA nalirngnn QALVPek DpsgGQNn Sg NqdTCE6QkMA2EMMeKM6kEKD6F3SIMEPL * 100 * 120 * 140 * 160 P.coatneyi_CM : QSKLTDDHLCSKLKYTNVCLHEKDKTPLTFPCTSPEYEKLIQQFTYQKLCNSKVAFSNVLLKSFIDKKNEENTFNAIIQNY : 162 Pknowlesi_NC_ : QSKLTDDRLCSKMKYTNICLHEKDKTPLTFPCTNPQYEQLIQQFTYKKLCNSKVAFSNVLLKSFIDKKNEENTFNAIIQNY : 162 Pinui_NW_ : QSKLADDHLCSEVKYKNICLQEKDNNSLTFPCTSPEYEQLIHEFTYKKLCNSKVAFSNVLLKSFIDKKNEENTFNTIIENY : 162 Pfragile_NW_ : QSKLSDNHLCSTVKYTNICLHEKDKTPLTFPCTSPQYEQLIHHFTYKKLCNSQVAFNNVLLKSFINKNNEENTFNTIIQNY : 162 Pcynomolgi_BAEJ : QSKLPDDHLCSKLKYTNICLQEKDKTPLTLPCTSPQYEQLIQHFTYKKLCNSQVAFSNVLLKSFINKKNEENTFNTIIQNY : 162 Brazil-I_AFMK _A : QSKLTDDHLCSKMKYTNICLHEKDKTPLTFPCTSPQYEQLIHRFTYKKLCNSKVAFSNVLLKSFIDKKNEENTFNTIIQNY : 162 VCG-I : QSKLTDDHLCSKMKYTNICLHEKDKTPLTFPCTSPQYEQLIHRFTYKKLCNSKVAFSNVLLKSFIDKKNEENTFNTIIQNY : 162 Sal-1_PVX_ : QSKLTDDHLCSKMKYTNICLHEKDKTPLTFPCTSPQYEQLIHRFTYKKLCNSKVAFSNVLLKSFIDKKNEENTFNTIIQNY : 162 Mauritania_I_AFNI : QSKLTDDHLCSKMKYTNICLHEKDKTPLTFPCTSPQYEQLIHRFTYKKLCNSKVAFSNVLLKSFIDKKNEENTFNTIIQNY : 162 N.Korean_AFNJ : QSKLTDDHLCSKMKYTNICLHEKDKTPLTFPCTSPQYEQLIHRFTYKKLCNSKVAFSNVLLKSFIDKKNEENTFNTIIQNY : 162 India-VII_AFBK _AFBK : QSKLTDDHLCSKMKYTNICLHEKDKTPLTFPCTSPQYEQLIHRFTYKKLCNSKVAFSNVLLKSFIDKKNEENTFNTIIQNY : 162 QSKL D1hLCSk6KYtN6CLhEKDktpLTfPCTsP2YEqLI FTYkKLCNSkVAFsNVLLKSFI1KkNEENTFNtII2NY * 180 * 200 * 220 * 240 P.coatneyi_CM : KVLSTCIDEDLKDIYNASIELFSDIRSSVTEITEKLWSKNMIDVLKTREQAIAGILCELRNGNNSPLVSNSLSYENFGILK : 243 Pknowlesi_NC_ : KVLSTCIDEDLKDIYNASIELFSDLRTSVREITEKLWSKNMIEVLKTREQAIAGILCELRNGNNSTLVSNSLSYENFGILK : 243 Pinui_NW_ : KVLSTCIDADLKDIYSASIQLFSDLRSAVTEISERLWSKNMIDVLKTREETITGILCELRKGNNSTLVSNSFSYDNFGILK : 243 Pfragile_NW_ : KILSTCIDEDLKDIYDASIQLFSDIRVSVTEITEKLWSKNMIDVLKTREQTIAGILCELRNGNKSTLVSNSLSYENFGILK : 243 Pcynomolgi_BAEJ : KLLSTCIDEDLKDIYNASIDLFSDIRTSVTEITEKLWSKNMIEVLKAREQTIAGILCELRNGNNSTLVSNSLSYENFGILK : 243 Brazil-I_AFMK _A : KVLSTCIDDDLKDIYNASIELFSDIRTSVTEITEKLWSKNMIEVLKTREQTIAGILCELRNGNNSPLVSNSFSYENFGILK : 243 VCG-I : KVLSTCIDDDLKDIYNASIELFSDIRTSVTEITEKLWSKNMIEVLKTREQTIAGILCELRNGNNSPLVSNSFSYENFGILK : 243 Sal-1_PVX_ : KVLSTCIDDDLKDIYNASIELFSDIRTSVTEITEKLWSKNMIEVLKTREQTIAGILCELRNGNNSPLVSNSFSYENFGILK : 243 Mauritania_I_AFNI : KVLSTCIDDDLKDIYNASIELFSDIRTSVTEITEKLWSKNMIEVLKTREQTIAGILCELRNGNNSPLVSNSFSYENFGILK : 243 N.Korean_AFNJ : KVLSTCIDDDLKDIYNASIELFSDIRTSVTEITEKLWSKNMIEVLKTREQTIAGILCELRNGNNSPLVSNSFSYENFGILK : 243 India-VII_AFBK _AFBK : KVLSTCIDDDLKDIYNASIELFSDIRTSVTEITEKLWSKNMIEVLKTREQTIAGILCELRNGNNSPLVSNSFSYENFGILK : 243 K6LSTCID DLKDIY ASI LFSD6R svtei3e4lwsknmi VLKtRE2tIaGILCELRnGNnS LVSNS SYeNFGILK
86 86 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax * 260 * 280 * 300 * 320 P.coatneyi_CM : VNYEALMNQAYKAFSDYYSYFPAFAIKLLEKGGFVERLVAIHESLTNYRTKNILKKINEKSKNEVLNNEDIMHSLSSYKHH : 324 Pknowlesi_NC_ : VNYEGLINQAYKAFSDYYSYFPAFAIRLLEKDGLVERLVAIHESLTNYRTRNILKKINEKSKNEVLNNEDIMHSLSSYKHH : 324 Pinui_NW_ : VNYEGLLNQAYVAFSDYYSFFPLFAMRLLENGGLVERLVAIHEKLTNYRTRNILKKINDKSKNEVLNNEDIMHSLSSYKHH : 324 Pfragile_NW_ : VNYEGLMNQAYKAFSDYYSYFPDFAIKLLEKGGLVERLVAIHESLTNYRTKNILQKINEKSKNEVLNNEDIMHSLSSYKHR : 324 Pcynomolgi_BAEJ : VNYEELLNQAYAAFSDYYSYFPAFAIKLLEKGGLVERLVAIHESLTNYRTRNILKKINEKSKNEVLNNGDIMHSLSSYKHH : 324 Brazil-I_AFMK _A : VNYEGLLNQAYAAFSDYYSYFPAFAISMLEKGGLVDRLVAIHESLTNYRTRNILKKINEKSKNEVLNNEEIMHSLSSYKHH : 324 VCG-I : VNYEGLLNQAYAAFSDYYSYFPAFAISMLEKGGLVDRLVAIHESLTNYRTRNILKKINEKSKNEVLNNEEIMHSLSSYKHH : 324 Sal-1_PVX_ : VNYEGLLNQAYAAFSDYYSYFPAFAISMLEKGGLVDRLVAIHESLTNYRTRNILKKINEKSKNEVLNNEEIMHSLSSYKHH : 324 Mauritania_I_AFNI : VNYEGLLNQAYAAFSDYYSYFPAFAISMLEKGGLVDRLVAIHESLTNYRTRNILKKINEKSKNEVLNNEEIMHSLSSYKHH : 324 N.Korean_AFNJ : VNYEGLLNQAYAAFSDYYSYFPAFAISMLEKGGLVDRLVAIHESLTNYRTRNILKKINEKSKNEVLNNEEIMHSLSSYKHH : 324 India-VII_AFBK _AFBK : VNYEGLLNQAYAAFSDYYSYFPAFAISMLEKGGLVDRLVAIHESLTNYRTRNILKKINEKSKNEVLNNEEIMHSLSSYKHH : 324 VNYEgL6NQAY AFSDYYS5FPaFA6 6LEkgGlV RLVAIHEsLTNYRT4NILkKINeKSKNEVLNNe IMHSLSSYKHh * 340 * 360 * 380 * 400 P.coatneyi_CM : AGGTRGSFVQSRGVSQVTQGVLSVDEKGDPSSTAG-GNQSANMATAAPN TVPATNTAAATTATTTNTA : 391 Pknowlesi_NC_ : AGGTRGSFMQYRGVGQLSKGGLSVDEKGGQQIASAVGNQSANMVAPAPKDSSPTM AAPSTDTVTNNMATTTTAP : 398 Pinui_NW_ : ATGTRGSFLQSRGVRQVTNSNVSVDQKGDQTATAG-GNESENVPAAVPKDDAPTRVASPGPAAFPNTVASNPTAVSNATAA : 404 Pfragile_NW_ : AGSTRGSFLQSRGAHNVTKVDVKVDE----TSTTG-GNQSANMAAAAPNNSGPTTAAAANSAASPNTAATTNTAATTS--- : 397 Pcynomolgi_BAEJ : AGGTRGSFLQSRVAREVRQGDVSVDEKGDQTATTG-GNQSANMAAAAPKDASPTT-AASNAAASPDTTAANTAAANTAAAN : 403 Brazil-I_AFMK _A : AGGTRGAFLQSRDVREVTQGDVSVDEKGDRATTAG-GNQSASVAAAAPKDAGPTV AAPNTAATLKTA : 390 VCG-I : AGGTRGAFLQSRDVREVTQGDVSVDEKGDRATTAG-GNQSASVAAAAPKDAGPTV AAPNTAATLKTA : 390 Sal-1_PVX_ : AGGTRGAFLQSRDVREVTQGDVSVDEKGDRATTAG-GNQSASVAAAAPKDAGPTV AAPNTAATLKTA : 390 Mauritania_I_AFNI : AGGTRGAFLQSRDVREVTQGDVSVDEKGDRATTAG-GNQSASVAAAAPKDAGPTV AAPNTGATLKTA : 390 N.Korean_AFNJ : AGGTRGAFLQSRDVREVTQGDVSVDEKGDRATTAG-GNQSASVAAAAPKDAGPTV AAPNTAATLKTA : 390 India-VII_AFBK _AFBK : AGGTRGAFLQSRDVREVTQGDVSVDEKGDRATTAG-GNQSASVAAAAPKDAGPTV AAPNTAATLKTA : 390 AggTRG F6QsR v 6 g 6sVD2kgd t g GN2Sa 6aaaaPk pt a P T a ta * 420 * 440 * 460 * 480 P.coatneyi_CM : TNTNTA------TNTNTAT-NTNSAATTNTAA------TTNTAATTNTTATTSVATSPLNT PLYGTSSSRTKDV : 452 Pknowlesi_NC_ : ATANMAAPSTNTATANMAA-PSTNTATANMAAPSTDTVTTNMATTTTVPATANTAASP-DTNTDSSTYPLYGTESSKTKDV : 477 Pinui_NW_ : SNATAASNATAASNATD---ASNATAASNPAA PPNTAA-TSQ-LSN------PLYGISSLNTKDV : 458 Pfragile_NW_ : ASNDTNASPHTN TSNEGASTSP-LSN------PLYGTRYLKTKDV : 435 Pcynomolgi_BAEJ : TAAAPNTASASNAAATPNPAAAPNTAATPNSA TPNAAA-TSP-LST------PLYDTSSLKTKDV : 460 Brazil-I_AFMK _A : ASPNAAATNTAA PPNMGA-TSP-LSN------PLYGTSSLQPKDV : 427 VCG-I : ASPNAAATNTAA PPNMGA-TSP-LSN------PLYGTSSLQPKDV : 427 Sal-1_PVX_ : ASPNAAATNTAA PPNMGA-TSP-LSN------PLYGTSSLQPKDV : 427 Mauritania_I_AFNI : ASPNAAATNTAA PPNMGA-TSP-LSN------PLYGTSSLQPKDV : 427 N.Korean_AFNJ : ASPNAAATNTAA PPNMGA-TSP-LSN------PLYGTSSLQPKDV : 427 India-VII_AFBK _AFBK : ASPNAAATNTAA PPNMGA-TSP-LSN------PLYGTSSLQPKDV : 427 a aa n aa n a tsp l3n PLYgtssl KDV
87 87 * 500 * 520 * 540 * 560 P.coatneyi_CM : VVLVRDLLKNTNIIKFEKNEPTSQIDDEGIKKLIESSFFDLSDNTMLMRLLIKPQAAILFIIQSFIMMTPSPTRDAKMYCK : 533 Pknowlesi_NC_ : VVLVRDLLKDTNIIKFEKNEPTSQIDDEGIKKLIESSFFDLSDNTMLMRLIIKPQASILFIIQSFIMMTPSPTRDARMYCK : 558 Pinui_NW_ : VVLVRDLLKNTNIIKFDKDEPTSQMDDEGIKKLIESSFFDLSDNTMLMQLLIKPQAAMLFIIQSFIMMTPSPTRDAKTYCN : 539 Pfragile_NW_ : VVLIRDLLKNTNIIKFEKDEPTGQIDHEGIKKLIESSFFDLSDNTMLMRLLIKPQAAILFIIQSFIMMTPSPTRDAKIYCK : 516 Pcynomolgi_BAEJ : VVLVRDLLKNTNIIKFEKDEPTSQIDEEGIKKLIESSFFDLSDNTMLMRLLIKPQAAILFIIQSFIMMTPSPTRDAKIYCK : 541 Brazil-I_AFMK _A : AVLVRDLLKNTNIIKFENNEPTSQMDDEEIKKLIESSFFDLSDNTMLMRLLIKPQAAILLIIESFIMMTPSPTRDAKTYCK : 508 VCG-I : AVLVRDLLKNTNIIKFENNEPTSQMDDEEIKKLIESSFFDLSDNTMLMRLLIKPQAAILLIIESFIMMTPSPTRDAKTYCK : 508 Sal-1_PVX_ : AVLVRDLLKNTNIIKFENNEPTSQMDDEEIKKLIESSFFDLSDNTMLMRLLIKPQAAILLIIESFIMMTPSPTRDAKTYCK : 508 Mauritania_I_AFNI : AVLVRDLLKNTNIIKFENNEPTSQMDDEEIKKLIESSFFDLSDNTMLMRLLIKPQAAILLIIESFIMMTPSPTRDAKTYCK : 508 N.Korean_AFNJ : AVLVRDLLKNTNIIKFENNEPTSQMDDEEIKKLIESSFFDLSDNTMLMRLLIKPQAAILLIIESFIMMTPSPTRDAKTYCK : 508 India-VII_AFBK _AFBK : AVLVRDLLKNTNIIKFENNEPTSQMDDEEIKKLIESSFFDLSDNTMLMRLLIKPQAAILLIIESFIMMTPSPTRDAKTYCK : 508 VL6RDLLK1TNIIKFe 1EPTsQ6DdE IKKLIESSFFDLSDNTMLMrL6IKPQAa6L II2SFIMMTPSPTRDA4 YCk * 580 * 600 * 620 * 640 P.coatneyi_CM : KKLVNGQLIENSDLKAATEEEDMITEFSSKYNLFYERLKLEELREIEQNRKALKNSKGSLSVLQVGNSQNGPDGKEANGSG : 614 Pknowlesi_NC_ : KKLVNGQLIENNDLKAETEEEDMINEFSSKYNLFYERLKMEELREIEQDRKSLKNSKGNLSVLEVRNSQNGPDGKEVNGSG : 639 Pinui_NW_ : KSLVNGKLIETSDIKASTEEEDLITEFSSKYNLFYEKLKMEELREIEENKKAIKNSKGSLSVLEVANSQNSPDGKRVNGSG : 620 Pfragile_NW_ : KNLVNGQLVQTSDFSDAHEEEDLINEFSSKYNLFYERLKLEELREIEQNRKALKNAKGSLSVLQVSNSQNGTHGNGVNQGG : 597 Pcynomolgi_BAEJ : KVLVNGQLVETSDLSAATEEEDLINEFSSKYNLFYERLKLEELREIEQNRKALKNSKGSLSVLEVGNSQNGPDGSGNNGSG : 622 Brazil-I_AFMK _A : KALVNGQLIETSDLNAATEEDDLINEFSSRYNLFYERLKLEELREIEQNRKALKNSKGTLSVLEVANSQNAPDGKGVNGSG : 589 VCG-I : KALVNGQLIETSDLNAATEEDDLINEFSSRYNLFYERLKLEELREIEQNRKALKNSKGTLSVLEVANSQNAPDGKGVNGSG : 589 Sal-1_PVX_ : KALVNGQLIETSDLNAATEEDDLINEFSSRYNLFYERLKLEELREIEQNRKALKNSKGTLSVLEVANSQNAPDGKGVNGSG : 589 Mauritania_I_AFNI : KALVNGQLIETSDLNATTEEDDLINEFSSRYNLFYERLKLEELREIEQNRKALKNSKGTLSVLEVANSQNAPDGKGVNGSG : 589 N.Korean_AFNJ : KALVNGQLIETSDLNAATEEDDLINEFSSRYNLFYERLKLEELREIEQNRKALKNSKGTLSVLEVANSQNAPDGKGVNGSG : 589 India-VII_AFBK _AFBK : KALVNGQLIETSDLNAATEEDDLINEFSSRYNLFYERLKLEELREIEQNRKALKNSKGTLSVLEVANSQNAPDGKGVNGSG : 589 K LVNGqL62tsD a tee D6InEFSS4YNLFYE4LK6EELREIE214Ka6KNsKG LSVL2V NSQN pdgk vngsg * 660 * 680 * 700 * 720 P.coatneyi_CM : NAANG : 619 Pknowlesi_NC_ : DAANG : 644 Pinui_NW_ : NDANG : 625 Pfragile_NW_ : HAANE : 602 Pcynomolgi_BAEJ : NAANG : 627 Brazil-I_AFMK _A : NAANANAANANAANANAANANAANGNLANANLANAN ANANLANAN : 634 VCG-I : NAANANAANANAANANAANANAANGNLANANLANANAANADAANANAANANAANANAANANLANANLANANLANANLANAN : 670 Sal-1_PVX_ : NAANANAANANAANANAANANAANGNLANANLANANAANADAANANAANANAANANAANANLANANLANANLANANLANAN : 670 Mauritania_I_AFNI : NAANA : 594 N.Korean_AFNJ : NAANANAANANAANANAANANAANX--XNANLANANAANANAANANAANANAANANAANG : 645 India-VII_AFBK _AFBK : NAANANAANANAANANAANANAANXNLANANLANANAA NG : 628 aan
88 88 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax * 740 * 760 * 780 * 800 * P.coatneyi_CM : NAQNGNGANVNAQNENAPNGNNGSASPLIVVVGDELGEKTDDIIKKNVDVEALTADV : 676 Pknowlesi_NC_ : NNMNG GNNGSASSLIVVVRDDLAEKTDDIIKNNVDLESLKADV : 687 Pinui_NW_ : NAANGNNGSGSPLLVVVGADLGEKTEDIIKNNVDVATLTEGV : 667 Pfragile_NW_ : SGHAAND NAHAAND : 616 Pcynomolgi_BAEJ : NNANGNNANGNNANG NAANGNNGNGSPLIAVVGADLGEKTDDIIKNNVDVAALAADV : 684 Brazil-I_AFMK _A : LANANLANANAANANAANANAANG NAPNSNNGSGSPLIVVVGADLGEKTEDIIKNNVDVAALTADV : 700 VCG-I : LANANLANANAANANAANANAANG NAPNSNNGSGSPLIVVVGADLGEKTEDIIKNNVDVAALTADV : 736 Sal-1_PVX_ : LANANLANANAANANAANANAANG NAPNSNNGSGSPLIVVVGADLGEKTEDIIKNNVDVAALTADV : 736 Mauritania_I_AFNI : ANANAANANAANANAANG NAPNSNNGSGSPLIVVVGADLGEKTEDIIKNNVDVAALTADV : 654 N.Korean_AFNJ : NAPNSNNGSGSPLIVVVGADLGEKTEDIIKNNVDVAALTADV : 687 India-VII_AFBK _AFBK : NAPNSNNGSGSPLIVVVGADLGEKTEDIIKNNVDVAALTADV : 670 na n N1gs spl vvvg dlgekt diiknnvd l adv 820 * 840 P.coatneyi_CM : EQAFKSFEYQSGSFSANLSHALVLLSSIALLLFIC : 711 Pknowlesi_NC_ : EQAFRNFEYQSGSFSANLSHALVLLSSIALVLFIC : 722 Pinui_NW_ : EQAFKNLSLEGGSFSANLSHALVLLSSIALLLFIC : 702 Pfragile_NW_ : : - Pcynomolgi_BAEJ : EQAFRNLELKSGSFSANLSHALVLLSSIALLLFIF : 719 Brazil-I_AFMK _A : EQAFKNLELQSGSFSANLSHALVLLSSIALLLFIF : 735 VCG-I : EQAFKNLELQSGSFSANLSHALVLLSSIALLLFIF : 771 Sal-1_PVX_ : EQAFKNLELQSGSFSANLSHALVLLSSIALLLFIF : 771 Mauritania_I_AFNI : EQAFKNLELQSGSFSANLSHALVLLSSIALLLFIF : 689 N.Korean_AFNJ : EQAFKNLELQSGSFSANLSHALVLLSSIALLLFIF : 722 India-VII_AFBK _AFBK : EQAFKNLELQSGSFSANLSHALVLLSSIALLLFIF : 705 eqaf n e sgsfsanlshalvllssial lfi
89 Referencias 1. WHO. World Malaria Report Guerra CA, Howes RE, Patil AP, Gething PW, Van Boeckel TP, Temperley WH, et al. The international limits and population at risk of Plasmodium vivax transmission in PLoS Negl Trop Dis. 2010;4(8):e Kumar R, Saravu K. Severe vivax malaria: a prospective exploration at a tertiary healthcare centre in Southwestern India. Pathogens and global health. 2017: Rahimi BA, Thakkinstian A, White NJ, Sirivichayakul C, Dondorp AM, Chokejindachai W. Severe vivax malaria: a systematic review and meta-analysis of clinical studies since Malar J. 2014;13: Aashish A, Manigandan G. Complicated vivax malaria, an often underestimated condition - Case Report. J Family Community Med. 2015;22(3): Ridley RG. Malaria: to kill a parasite. Nature. 2003;424(6951): Auburn S, Serre D, Pearson RD, Amato R, Sriprawat K, To S, et al. Genomic Analysis Reveals a Common Breakpoint in Amplifications of the Plasmodium vivax Multidrug Resistance 1 Locus in Thailand. J Infect Dis. 2016;214(8): Wang Z, Cabrera M, Yang J, Yuan L, Gupta B, Liang X, et al. Genome-wide association analysis identifies genetic loci associated with resistance to multiple antimalarials in Plasmodium falciparum from China-Myanmar border. Sci Rep. 2016;6: Riveron JM, Osae M, Egyir-Yawson A, Irving H, Ibrahim SS, Wondji CS. Multiple insecticide resistance in the major malaria vector Anopheles funestus in southern Ghana: implications for malaria control. Parasit Vectors. 2016;9(1): WHO. State of the art of new vaccine research and development Baldo V, Bonanni P, Castro M, Gabutti G, Franco E, Marchetti F, et al. Combined hexavalent diphtheria-tetanus-acellular pertussis-hepatitis B-inactivated poliovirus-
90 90 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax Haemophilus influenzae type B vaccine; Infanrix hexa: twelve years of experience in Italy. Hum Vaccin Immunother. 2014;10(1): Patarroyo ME, Bermudez A, Patarroyo MA. Structural and immunological principles leading to chemically synthesized, multiantigenic, multistage, minimal subunit-based vaccine development. Chem Rev. 2011;111(5): Gardner MJ, Hall N, Fung E, White O, Berriman M, Hyman RW, et al. Genome sequence of the human malaria parasite Plasmodium falciparum. Nature. 2002;419(6906): Bozdech Z, Llinas M, Pulliam BL, Wong ED, Zhu J, DeRisi JL. The transcriptome of the intraerythrocytic developmental cycle of Plasmodium falciparum. PLoS biology. 2003;1(1):E Lasonder E, Ishihama Y, Andersen JS, Vermunt AM, Pain A, Sauerwein RW, et al. Analysis of the Plasmodium falciparum proteome by high-accuracy mass spectrometry. Nature. 2002;419(6906): Florens L, Washburn MP, Raine JD, Anthony RM, Grainger M, Haynes JD, et al. A proteomic view of the Plasmodium falciparum life cycle. Nature. 2002;419(6906): Thompson J, Cooke RE, Moore S, Anderson LF, Janse CJ, Waters AP. PTRAMP; a conserved Plasmodium thrombospondin-related apical merozoite protein. Mol Biochem Parasitol. 2004;134(2): Arumugam TU, Takeo S, Yamasaki T, Thonkukiatkul A, Miura K, Otsuki H, et al. Discovery of GAMA, a Plasmodium falciparum merozoite micronemal protein, as a novel bloodstage vaccine candidate antigen. Infect Immun. 2011;79(11): Hinds L, Green JL, Knuepfer E, Grainger M, Holder AA. Novel putative glycosylphosphatidylinositol-anchored micronemal antigen of Plasmodium falciparum that binds to erythrocytes. Eukaryot Cell. 2009;8(12): Huynh MH, Carruthers VB. A Toxoplasma gondii Ortholog of Plasmodium GAMA Contributes to Parasite Attachment and Cell Invasion. msphere. 2016;1(1). 21. Richards JS, Beeson JG. The future for blood-stage vaccines against malaria. Immunol Cell Biol. 2009;87(5): Moreno-Perez DA, Ruiz JA, Patarroyo MA. Reticulocytes: Plasmodium vivax target cells. Biology of the cell / under the auspices of the European Cell Biology Organization. 2013;105(6): Patarroyo MA, Calderon D, Moreno-Perez DA. Vaccines against Plasmodium vivax: a research challenge. Expert Rev Vaccines. 2012;11(10):
91 Carlton JM, Adams JH, Silva JC, Bidwell SL, Lorenzi H, Caler E, et al. Comparative genomics of the neglected human malaria parasite Plasmodium vivax. Nature. 2008;455(7214): Bozdech Z, Mok S, Hu G, Imwong M, Jaidee A, Russell B, et al. The transcriptome of Plasmodium vivax reveals divergence and diversity of transcriptional regulation in malaria parasites. Proceedings of the National Academy of Sciences of the United States of America. 2008;105(42): Acharya P, Pallavi R, Chandran S, Chakravarti H, Middha S, Acharya J, et al. A glimpse into the clinical proteome of human malaria parasites Plasmodium falciparum and Plasmodium vivax. Proteomics Clinical applications. 2009;3(11): Roobsoong W, Roytrakul S, Sattabongkot J, Li J, Udomsangpetch R, Cui L. Determination of the Plasmodium vivax schizont stage proteome. J Proteomics. 2011;74(9): Pico de Coana Y, Rodriguez J, Guerrero E, Barrero C, Rodriguez R, Mendoza M, et al. A highly infective Plasmodium vivax strain adapted to Aotus monkeys: quantitative haematological and molecular determinations useful for P. vivax malaria vaccine development. Vaccine. 2003;21(25-26): Wertheimer SP, Barnwell JW. Plasmodium vivax interaction with the human Duffy blood group glycoprotein: identification of a parasite receptor-like protein. Experimental parasitology. 1989;69(4): Black CG, Barnwell JW, Huber CS, Galinski MR, Coppel RL. The Plasmodium vivax homologues of merozoite surface proteins 4 and 5 from Plasmodium falciparum are expressed at different locations in the merozoite. Mol Biochem Parasitol. 2002;120(2): Galinski MR, Medina CC, Ingravallo P, Barnwell JW. A reticulocyte-binding protein complex of Plasmodium vivax merozoites. Cell. 1992;69(7): Zeeshan M, Tyagi RK, Tyagi K, Alam MS, Sharma YD. Host-parasite interaction: selective Pv-fam-a family proteins of Plasmodium vivax bind to a restricted number of human erythrocyte receptors. The Journal of infectious diseases. 2015;211(7): Tyagi RK, Sharma YD. Erythrocyte Binding Activity Displayed by a Selective Group of Plasmodium vivax Tryptophan Rich Antigens Is Inhibited by Patients' Antibodies. PLoS One. 2012;7(12):e Arevalo-Pinzon G, Bermudez M, Curtidor H, Patarroyo MA. The Plasmodium vivax rhoptry neck protein 5 is expressed in the apical pole of Plasmodium vivax VCG-1 strain schizonts and binds to human reticulocytes. Malaria journal. 2015;14:106.
92 92 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 35. Moreno A, Joyner C. Malaria vaccine clinical trials: what's on the horizon. Curr Opin Immunol. 2015;35: Hoffman SL, Vekemans J, Richie TL, Duffy PE. The March Toward Malaria Vaccines. Am J Prev Med. 2015;49(6 Suppl 4):S Singh B, Daneshvar C. Human infections and detection of Plasmodium knowlesi. Clin Microbiol Rev. 2013;26(2): Antinori S, Galimberti L, Milazzo L, Corbellino M. Plasmodium knowlesi: the emerging zoonotic malaria parasite. Acta Trop. 2013;125(2): Amireh S, Shaaban H, Guron G. Severe Plasmodium vivax cerebral malaria complicated by hemophagocytic lymphohistiocytosis treated with artesunate and doxycycline. Hematol Oncol Stem Cell Ther Demissie Y, Ketema T. Complicated malaria symptoms associated with Plasmodium vivax among patients visiting health facilities in Mendi town, Northwest Ethiopia. BMC Infect Dis. 2016;16(1): Mehndiratta S, Rajeshwari K, Dubey AP. Multiple-organ dysfunction in a case of Plasmodium vivax malaria. J Vector Borne Dis. 2013;50(1): Grupo ETV INdS. Boletín epidemiológico semana Cowman AF, Crabb BS. Invasion of red blood cells by malaria parasites. Cell. 2006;124(4): Gonzalez V, Combe A, David V, Malmquist NA, Delorme V, Leroy C, et al. Host cell entry by apicomplexa parasites requires actin polymerization in the host cell. Cell Host Microbe. 2009;5(3): Ferreira MU, da Silva Nunes M, Wunderlich G. Antigenic diversity and immune evasion by malaria parasites. Clin Diagn Lab Immunol. 2004;11(6): Prudencio M, Rodriguez A, Mota MM. The silent path to thousands of merozoites: the Plasmodium liver stage. Nat Rev Microbiol. 2006;4(11): Hoffman SL, Vekemans J, Richie TL, Duffy PE. The march toward malaria vaccines. Vaccine Markus MB. Do hypnozoites cause relapse in malaria? Trends Parasitol. 2015;31(6): Hulden L. Activation of the hypnozoite: a part of Plasmodium vivax life cycle and survival. Malar J. 2011;10:90.
93 Shanks GD, White NJ. The activation of vivax malaria hypnozoites by infectious diseases. Lancet Infect Dis. 2013;13(10): Bannister LH, Hopkins JM, Fowler RE, Krishna S, Mitchell GH. A brief illustrated guide to the ultrastructure of Plasmodium falciparum asexual blood stages. Parasitol Today. 2000;16(10): Oakley MS, Gerald N, McCutchan TF, Aravind L, Kumar S. Clinical and molecular aspects of malaria fever. Trends Parasitol. 2011;27(10): Trager W, Jensen JB. Human malaria parasites in continuous culture. Science. 1976;193(4254): Aikawa M, Miller LH, Johnson J, Rabbege J. Erythrocyte entry by malarial parasites. A moving junction between erythrocyte and parasite. J Cell Biol. 1978;77(1): Bannister LH, Butcher GA, Dennis ED, Mitchell GH. Structure and invasive behaviour of Plasmodium knowlesi merozoites in vitro. Parasitology. 1975;71(3): Preiser P, Kaviratne M, Khan S, Bannister L, Jarra W. The apical organelles of malaria merozoites: host cell selection, invasion, host immunity and immune evasion. Microbes Infect. 2000;2(12): Kats LM, Black CG, Proellocks NI, Coppel RL. Plasmodium rhoptries: how things went pear-shaped. Trends Parasitol. 2006;22(6): Counihan NA, Kalanon M, Coppel RL, de Koning-Ward TF. Plasmodium rhoptry proteins: why order is important. Trends Parasitol. 2013;29(5): Harvey KL, Gilson PR, Crabb BS. A model for the progression of receptor-ligand interactions during erythrocyte invasion by Plasmodium falciparum. Int J Parasitol. 2012;42(6): Proellocks NI, Coppel RL, Waller KL. Dissecting the apicomplexan rhoptry neck proteins. Trends Parasitol. 2010;26(6): Crosnier C, Bustamante LY, Bartholdson SJ, Bei AK, Theron M, Uchikawa M, et al. Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum. Nature. 2011;480(7378): Zuccala ES, Gout AM, Dekiwadia C, Marapana DS, Angrisano F, Turnbull L, et al. Subcompartmentalisation of proteins in the rhoptries correlates with ordered events of erythrocyte invasion by the blood stage malaria parasite. PLoS One. 2012;7(9):e46160.
94 94 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 63. Bradley PJ, Ward C, Cheng SJ, Alexander DL, Coller S, Coombs GH, et al. Proteomic analysis of rhoptry organelles reveals many novel constituents for host-parasite interactions in Toxoplasma gondii. J Biol Chem. 2005;280(40): Kaneko O, Tsuboi T, Ling IT, Howell S, Shirano M, Tachibana M, et al. The high molecular mass rhoptry protein, RhopH1, is encoded by members of the clag multigene family in Plasmodium falciparum and Plasmodium yoelii. Mol Biochem Parasitol. 2001;118(2): Kaneko O, Yim Lim BY, Iriko H, Ling IT, Otsuki H, Grainger M, et al. Apical expression of three RhopH1/Clag proteins as components of the Plasmodium falciparum RhopH complex. Mol Biochem Parasitol. 2005;143(1): Rojas-Caraballo J, Mongui A, Giraldo MA, Delgado G, Granados D, Millan-Cortes D, et al. Immunogenicity and protection-inducing ability of recombinant Plasmodium vivax rhoptryassociated protein 2 in Aotus monkeys: a potential vaccine candidate. Vaccine. 2009;27(21): Bannister LH, Mitchell GH. The fine structure of secretion by Plasmodium knowlesi merozoites during red cell invasion. J Protozool. 1989;36(4): Kappe SH, Curley GP, Noe AR, Dalton JP, Adams JH. Erythrocyte binding protein homologues of rodent malaria parasites. Mol Biochem Parasitol. 1997;89(1): Rydzak J, Kaczmarek R, Czerwinski M, Lukasiewicz J, Tyborowska J, Szewczyk B, et al. The baculovirus-expressed binding region of Plasmodium falciparum EBA-140 ligand and its glycophorin C binding specificity. PLoS One. 2015;10(1):e Adams JH, Hudson DE, Torii M, Ward GE, Wellems TE, Aikawa M, et al. The Duffy receptor family of Plasmodium knowlesi is located within the micronemes of invasive malaria merozoites. Cell. 1990;63(1): Adams JH, Blair PL, Kaneko O, Peterson DS. An expanding ebl family of Plasmodium falciparum. Trends Parasitol. 2001;17(6): Han JH, Lee SK, Wang B, Muh F, Nyunt MH, Na S, et al. Identification of a reticulocytespecific binding domain of Plasmodium vivax reticulocyte-binding protein 1 that is homologous to the PfRh4 erythrocyte-binding domain. Sci Rep. 2016;6: Torii M, Adams JH, Miller LH, Aikawa M. Release of merozoite dense granules during erythrocyte invasion by Plasmodium knowlesi. Infect Immun. 1989;57(10): Culvenor JG, Day KP, Anders RF. Plasmodium falciparum ring-infected erythrocyte surface antigen is released from merozoite dense granules after erythrocyte invasion. Infect Immun. 1991;59(3):
95 Foley M, Tilley L, Sawyer WH, Anders RF. The ring-infected erythrocyte surface antigen of Plasmodium falciparum associates with spectrin in the erythrocyte membrane. Mol Biochem Parasitol. 1991;46(1): Trager W, Rozario C, Shio H, Williams J, Perkins ME. Transfer of a dense granule protein of Plasmodium falciparum to the membrane of ring stages and isolation of dense granules. Infect Immun. 1992;60(11): O'Donnell RA, Blackman MJ. The role of malaria merozoite proteases in red blood cell invasion. Curr Opin Microbiol. 2005;8(4): Gaur D, Mayer DC, Miller LH. Parasite ligand-host receptor interactions during invasion of erythrocytes by Plasmodium merozoites. Int J Parasitol. 2004;34(13-14): Sibley LD. Intracellular parasite invasion strategies. Science. 2004;304(5668): Harrison T, Samuel BU, Akompong T, Hamm H, Mohandas N, Lomasney JW, et al. Erythrocyte G protein-coupled receptor signaling in malarial infection. Science. 2003;301(5640): Bannister LH, Dluzewski AR. The ultrastructure of red cell invasion in malaria infections: a review. Blood Cells. 1990;16(2-3):257-92; discussion Sanders PR, Gilson PR, Cantin GT, Greenbaum DC, Nebl T, Carucci DJ, et al. Distinct protein classes including novel merozoite surface antigens in Raft-like membranes of Plasmodium falciparum. J Biol Chem. 2005;280(48): Goel VK, Li X, Chen H, Liu SC, Chishti AH, Oh SS. Band 3 is a host receptor binding merozoite surface protein 1 during the Plasmodium falciparum invasion of erythrocytes. Proc Natl Acad Sci U S A. 2003;100(9): Li X, Chen H, Oo TH, Daly TM, Bergman LW, Liu SC, et al. A co-ligand complex anchors Plasmodium falciparum merozoites to the erythrocyte invasion receptor band 3. J Biol Chem. 2004;279(7): Pachebat JA, Ling IT, Grainger M, Trucco C, Howell S, Fernandez-Reyes D, et al. The 22 kda component of the protein complex on the surface of Plasmodium falciparum merozoites is derived from a larger precursor, merozoite surface protein 7. Mol Biochem Parasitol. 2001;117(1): Triglia T, Healer J, Caruana SR, Hodder AN, Anders RF, Crabb BS, et al. Apical membrane antigen 1 plays a central role in erythrocyte invasion by Plasmodium species. Mol Microbiol. 2000;38(4):
96 96 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 87. Mital J, Meissner M, Soldati D, Ward GE. Conditional expression of Toxoplasma gondii apical membrane antigen-1 (TgAMA1) demonstrates that TgAMA1 plays a critical role in host cell invasion. Mol Biol Cell. 2005;16(9): Mitchell GH, Thomas AW, Margos G, Dluzewski AR, Bannister LH. Apical membrane antigen 1, a major malaria vaccine candidate, mediates the close attachment of invasive merozoites to host red blood cells. Infect Immun. 2004;72(1): Singh S, Alam MM, Pal-Bhowmick I, Brzostowski JA, Chitnis CE. Distinct external signals trigger sequential release of apical organelles during erythrocyte invasion by malaria parasites. PLoS Pathog. 2010;6(2):e Vaid A, Thomas DC, Sharma P. Role of Ca2+/calmodulin-PfPKB signaling pathway in erythrocyte invasion by Plasmodium falciparum. J Biol Chem. 2008;283(9): Cowman AF, Berry D, Baum J. The cellular and molecular basis for malaria parasite invasion of the human red blood cell. J Cell Biol. 2012;198(6): Gilberger TW, Thompson JK, Triglia T, Good RT, Duraisingh MT, Cowman AF. A novel erythrocyte binding antigen-175 paralogue from Plasmodium falciparum defines a new trypsin-resistant receptor on human erythrocytes. J Biol Chem. 2003;278(16): Duraisingh MT, Triglia T, Ralph SA, Rayner JC, Barnwell JW, McFadden GI, et al. Phenotypic variation of Plasmodium falciparum merozoite proteins directs receptor targeting for invasion of human erythrocytes. EMBO J. 2003;22(5): Gunalan K, Gao X, Yap SS, Huang X, Preiser PR. The role of the reticulocyte-binding-like protein homologues of Plasmodium in erythrocyte sensing and invasion. Cell Microbiol. 2013;15(1): Weiss GE, Gilson PR, Taechalertpaisarn T, Tham WH, de Jong NW, Harvey KL, et al. Revealing the sequence and resulting cellular morphology of receptor-ligand interactions during Plasmodium falciparum invasion of erythrocytes. PLoS Pathog. 2015;11(2):e Zimmerman PA, Ferreira MU, Howes RE, Mercereau-Puijalon O. Red blood cell polymorphism and susceptibility to Plasmodium vivax. Adv Parasitol. 2013;81: Chitnis CE, Sharma A. Targeting the Plasmodium vivax Duffy-binding protein. Trends Parasitol. 2008;24(1): Chitnis CE, Miller LH. Identification of the erythrocyte binding domains of Plasmodium vivax and Plasmodium knowlesi proteins involved in erythrocyte invasion. J Exp Med. 1994;180(2):
97 Howell SA, Withers-Martinez C, Kocken CH, Thomas AW, Blackman MJ. Proteolytic processing and primary structure of Plasmodium falciparum apical membrane antigen-1. J Biol Chem. 2001;276(33): Healer J, Crawford S, Ralph S, McFadden G, Cowman AF. Independent translocation of two micronemal proteins in developing Plasmodium falciparum merozoites. Infect Immun. 2002;70(10): Lamarque M, Besteiro S, Papoin J, Roques M, Vulliez-Le Normand B, Morlon-Guyot J, et al. The RON2-AMA1 interaction is a critical step in moving junction-dependent invasion by apicomplexan parasites. PLoS Pathog. 2011;7(2):e Srinivasan P, Yasgar A, Luci DK, Beatty WL, Hu X, Andersen J, et al. Disrupting malaria parasite AMA1-RON2 interaction with a small molecule prevents erythrocyte invasion. Nat Commun. 2013;4: Roobsoong W, Tharinjaroen CS, Rachaphaew N, Chobson P, Schofield L, Cui L, et al. Improvement of culture conditions for long-term in vitro culture of Plasmodium vivax. Malar J. 2015;14: Moreno-Perez DA, Degano R, Ibarrola N, Muro A, Patarroyo MA. Determining the Plasmodium vivax VCG-1 strain blood stage proteome. J Proteomics. 2014;113C: Chen JH, Chen SB, Wang Y, Ju C, Zhang T, Xu B, et al. An immunomics approach for the analysis of natural antibody responses to Plasmodium vivax infection. Mol Biosyst. 2015;11(8): Richie TL, Saul A. Progress and challenges for malaria vaccines. Nature. 2002;415(6872): Favaloro JM, Coppel RL, Corcoran LM, Foote SJ, Brown GV, Anders RF, et al. Structure of the RESA gene of Plasmodium falciparum. Nucleic Acids Res. 1986;14(21): Shi H, Liu Z, Li A, Yin J, Chong AG, Tan KS, et al. Life cycle-dependent cytoskeletal modifications in Plasmodium falciparum infected erythrocytes. PLoS One. 2013;8(4):e Alam MS, Choudhary V, Zeeshan M, Tyagi RK, Rathore S, Sharma YD. Interaction of Plasmodium vivax Tryptophan-rich Antigen PvTRAg38 with Band 3 on Human Erythrocyte Surface Facilitates Parasite Growth. J Biol Chem. 2015;290(33): Stoute JA, Slaoui M, Heppner DG, Momin P, Kester KE, Desmons P, et al. A preliminary evaluation of a recombinant circumsporozoite protein vaccine against Plasmodium falciparum malaria. RTS,S Malaria Vaccine Evaluation Group. N Engl J Med. 1997;336(2):86-91.
98 98 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 111. Efficacy and safety of the RTS,S/AS01 malaria vaccine during 18 months after vaccination: a phase 3 randomized, controlled trial in children and young infants at 11 African sites. PLoS Med. 2014;11(7):e Rosenthal PJ. The RTS,S/AS01 vaccine continues to show modest protection against malaria in African infants and children. Evid Based Med. 2015;20(5): Kimani D, Jagne YJ, Cox M, Kimani E, Bliss CM, Gitau E, et al. Translating the immunogenicity of prime-boost immunization with ChAd63 and MVA ME-TRAP from malaria naive to malaria-endemic populations. Mol Ther. 2014;22(11): Bergmann-Leitner ES, Mease RM, De La Vega P, Savranskaya T, Polhemus M, Ockenhouse C, et al. Immunization with pre-erythrocytic antigen CelTOS from Plasmodium falciparum elicits cross-species protection against heterologous challenge with Plasmodium berghei. PLoS One. 2010;5(8):e Sheehy SH, Duncan CJ, Elias SC, Choudhary P, Biswas S, Halstead FD, et al. ChAd63- MVA-vectored blood-stage malaria vaccines targeting MSP1 and AMA1: assessment of efficacy against mosquito bite challenge in humans. Mol Ther. 2012;20(12): Polhemus ME, Magill AJ, Cummings JF, Kester KE, Ockenhouse CF, Lanar DE, et al. Phase I dose escalation safety and immunogenicity trial of Plasmodium falciparum apical membrane protein (AMA-1) FMP2.1, adjuvanted with AS02A, in malaria-naive adults at the Walter Reed Army Institute of Research. Vaccine. 2007;25(21): Alonso PL, Smith T, Schellenberg JR, Masanja H, Mwankusye S, Urassa H, et al. Randomised trial of efficacy of SPf66 vaccine against Plasmodium falciparum malaria in children in southern Tanzania. Lancet. 1994;344(8931): Valero MV, Amador LR, Galindo C, Figueroa J, Bello MS, Murillo LA, et al. Vaccination with SPf66, a chemically synthesised vaccine, against Plasmodium falciparum malaria in Colombia. Lancet. 1993;341(8847): Yadava A, Hall CE, Sullivan JS, Nace D, Williams T, Collins WE, et al. Protective efficacy of a Plasmodium vivax circumsporozoite protein-based vaccine in Aotus nancymaae is associated with antibodies to the repeat region. PLoS Negl Trop Dis. 2014;8(10):e Acharya P, Pallavi R, Chandran S, Dandavate V, Sayeed SK, Rochani A, et al. Clinical proteomics of the neglected human malarial parasite Plasmodium vivax. PLoS One. 2011;6(10):e Petersen TN, Brunak S, von Heijne G, Nielsen H. SignalP 4.0: discriminating signal peptides from transmembrane regions. Nat Methods. 2011;8(10):785-6.
99 Pierleoni A, Martelli PL, Fariselli P, Casadio R. BaCelLo: a balanced subcellular localization predictor. Bioinformatics. 2006;22(14):e Poisson G, Chauve C, Chen X, Bergeron A. FragAnchor: a large-scale predictor of glycosylphosphatidylinositol anchors in eukaryote protein sequences by qualitative scoring. Genomics Proteomics Bioinformatics. 2007;5(2): Hunter S, Apweiler R, Attwood TK, Bairoch A, Bateman A, Binns D, et al. InterPro: the integrative protein signature database. Nucleic Acids Res. 2009;37(Database issue):d Aurrecoechea C, Brestelli J, Brunk BP, Dommer J, Fischer S, Gajria B, et al. PlasmoDB: a functional genomic database for malaria parasites. Nucleic Acids Res. 2009;37(Database issue):d Deleage G, Combet C, Blanchet C, Geourjon C. ANTHEPROT: an integrated protein sequence analysis software with client/server capabilities. Comput Biol Med. 2001;31(4): Andrysiak PM, Collins WE, Campbell GH. Concentration of Plasmodium ovale- and Plasmodium vivax-infected erythrocytes from nonhuman primate blood using Percoll gradients. Am J Trop Med Hyg. 1986;35(2): Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Res. 1994;22(22): Graur D, Zheng Y, Price N, Azevedo RB, Zufall RA, Elhaik E. On the immortality of television sets: "function" in the human genome according to the evolution-free gospel of ENCODE. Genome Biol Evol. 2013;5(3): Garzon-Ospina D, Forero-Rodriguez J, Patarroyo MA. Inferring natural selection signals in Plasmodium vivax-encoded proteins having a potential role in merozoite invasion. Infection, genetics and evolution : journal of molecular epidemiology and evolutionary genetics in infectious diseases. 2015;33: Neafsey DE, Galinsky K, Jiang RH, Young L, Sykes SM, Saif S, et al. The malaria parasite Plasmodium vivax exhibits greater genetic diversity than Plasmodium falciparum. Nature genetics. 2012;44(9): Escalante AA, Cornejo OE, Freeland DE, Poe AC, Durrego E, Collins WE, et al. A monkey's tale: the origin of Plasmodium vivax as a human malaria parasite. Proceedings of the National Academy of Sciences of the United States of America. 2005;102(6):
100 100 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 133. Edgar RC. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic acids research. 2004;32(5): Zhang J, Rosenberg HF, Nei M. Positive Darwinian selection after gene duplication in primate ribonuclease genes. Proceedings of the National Academy of Sciences of the United States of America. 1998;95(7): Jukes TH. Recent advances in studies of evolutionary relationships between proteins and nucleic acids. Space life sciences. 1969;1(4): Kosakovsky Pond SL, Frost SD. Not so different after all: a comparison of methods for detecting amino acid sites under selection. Molecular biology and evolution. 2005;22(5): Murrell B, Wertheim JO, Moola S, Weighill T, Scheffler K, Kosakovsky Pond SL. Detecting individual sites subject to episodic diversifying selection. PLoS genetics. 2012;8(7):e Murrell B, Moola S, Mabona A, Weighill T, Sheward D, Kosakovsky Pond SL, et al. FUBAR: a fast, unconstrained bayesian approximation for inferring selection. Molecular biology and evolution. 2013;30(5): Kosakovsky Pond SL, Posada D, Gravenor MB, Woelk CH, Frost SD. Automated phylogenetic detection of recombination using a genetic algorithm. Molecular biology and evolution. 2006;23(10): Kosakovsky Pond SL, Murrell B, Fourment M, Frost SD, Delport W, Scheffler K. A random effects branch-site model for detecting episodic diversifying selection. Molecular biology and evolution. 2011;28(11): Delport W, Poon AF, Frost SD, Kosakovsky Pond SL. Datamonkey 2010: a suite of phylogenetic analysis tools for evolutionary biology. Bioinformatics. 2010;26(19): Houghten RA. General method for the rapid solid-phase synthesis of large numbers of peptides: specificity of antigen-antibody interaction at the level of individual amino acids. Proc Natl Acad Sci U S A. 1985;82(15): Urquiza M, Patarroyo MA, Mari V, Ocampo M, Suarez J, Lopez R, et al. Identification and polymorphism of Plasmodium vivax RBP-1 peptides which bind specifically to reticulocytes. Peptides. 2002;23(12): Lu F, Li J, Wang B, Cheng Y, Kong DH, Cui L, et al. Profiling the humoral immune responses to Plasmodium vivax infection and identification of candidate immunogenic rhoptry-associated membrane antigen (RAMA). J Proteomics. 2014;102:66-82.
101 Rodriguez LE, Curtidor H, Urquiza M, Cifuentes G, Reyes C, Patarroyo ME. Intimate molecular interactions of P. falciparum merozoite proteins involved in invasion of red blood cells and their implications for vaccine design. Chemical reviews. 2008;108(9): Cornejo OE, Fisher D, Escalante AA. Genome-Wide Patterns of Genetic Polymorphism and Signatures of Selection in Plasmodium vivax. Genome Biol Evol. 2014;7(1): Forero-Rodriguez J, Garzon-Ospina D, Patarroyo MA. Low genetic diversity and functional constraint in loci encoding Plasmodium vivax P12 and P38 proteins in the Colombian population. Malar J. 2014;13: Forero-Rodriguez J, Garzon-Ospina D, Patarroyo MA. Low genetic diversity in the locus encoding the Plasmodium vivax P41 protein in Colombia's parasite population. Malar J. 2014;13: Wang Y, Ma A, Chen SB, Yang YC, Chen JH, Yin MB. Genetic diversity and natural selection of three blood-stage 6-Cys proteins in Plasmodium vivax populations from the China- Myanmar endemic border. Infect Genet Evol. 2014;28: Martinez P, Suarez CF, Gomez A, Cardenas PP, Guerrero JE, Patarroyo MA. High level of conservation in Plasmodium vivax merozoite surface protein 4 (PvMSP4). Infect Genet Evol. 2005;5(4): Putaporntip C, Jongwutiwes S, Ferreira MU, Kanbara H, Udomsangpetch R, Cui L. Limited global diversity of the Plasmodium vivax merozoite surface protein 4 gene. Infect Genet Evol. 2009;9(5): Garzon-Ospina D, Romero-Murillo L, Tobon LF, Patarroyo MA. Low genetic polymorphism of merozoite surface proteins 7 and 10 in Colombian Plasmodium vivax isolates. Infect Genet Evol. 2011;11(2): Garzon-Ospina D, Forero-Rodriguez J, Patarroyo MA. Heterogeneous genetic diversity pattern in Plasmodium vivax genes encoding merozoite surface proteins (MSP) -7E, -7F and - 7L. Malar J. 2014;13: Pacheco MA, Elango AP, Rahman AA, Fisher D, Collins WE, Barnwell JW, et al. Evidence of purifying selection on merozoite surface protein 8 (MSP8) and 10 (MSP10) in Plasmodium spp. Infect Genet Evol. 2012;12(5): Garzon-Ospina D, Romero-Murillo L, Patarroyo MA. Limited genetic polymorphism of the Plasmodium vivax low molecular weight rhoptry protein complex in the Colombian population. Infect Genet Evol. 2010;10(2):261-7.
102 102 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax 156. Sawai H, Otani H, Arisue N, Palacpac N, de Oliveira Martins L, Pathirana S, et al. Lineagespecific positive selection at the merozoite surface protein 1 (msp1) locus of Plasmodium vivax and related simian malaria parasites. BMC Evol Biol. 2010;10: Muehlenbein MP, Pacheco MA, Taylor JE, Prall SP, Ambu L, Nathan S, et al. Accelerated diversification of nonhuman primate malarias in Southeast Asia: adaptive radiation or geographic speciation? Mol Biol Evol. 2015;32(2): Cheng Y, Lu F, Wang B, Li J, Han JH, Ito D, et al. Plasmodium vivax GPI-anchored micronemal antigen (PvGAMA) binds human erythrocytes independent of Duffy antigen status. Scientific reports. 2016;6: Malleret B, Li A, Zhang R, Tan KS, Suwanarusk R, Claser C, et al. Plasmodium vivax: restricted tropism and rapid remodeling of CD71-positive reticulocytes. Blood. 2015;125(8): Wilson MC, Trakarnsanga K, Heesom KJ, Cogan N, Green C, Toye AM, et al. Comparison of the Proteome of Adult and Cord Erythroid Cells, and Changes in the Proteome Following Reticulocyte Maturation. Molecular & cellular proteomics : MCP. 2016;15(6): Ocampo M, Vera R, Eduardo Rodriguez L, Curtidor H, Urquiza M, Suarez J, et al. Plasmodium vivax Duffy binding protein peptides specifically bind to reticulocytes. Peptides. 2002;23(1): Rodriguez LE, Urquiza M, Ocampo M, Curtidor H, Suarez J, Garcia J, et al. Plasmodium vivax MSP-1 peptides have high specific binding activity to human reticulocytes. Vaccine. 2002;20(9-10): Ntumngia FB, Thomson-Luque R, Torres Lde M, Gunalan K, Carvalho LH, Adams JH. A Novel Erythrocyte Binding Protein of Plasmodium vivax Suggests an Alternate Invasion Pathway into Duffy-Positive Reticulocytes. MBio. 2016;7(4) Dent AE, Nakajima R, Liang L, Baum E, Moormann AM, Sumba PO, et al. Plasmodium falciparum Protein Microarray Antibody Profiles Correlate With Protection From Symptomatic Malaria in Kenya. J Infect Dis. 2015;212(9): Schofield L. On the function of repetitive domains in protein antigens of Plasmodium and other eukaryotic parasites. Parasitol Today. 1991;7(5): Moreno-Perez DA, Areiza-Rojas R, Florez-Buitrago X, Silva Y, Patarroyo ME, Patarroyo MA. The GPI-anchored 6-Cys protein Pv12 is present in detergent-resistant microdomains of Plasmodium vivax blood stage schizonts. Protist. 2013;164(1):37-48.
103 Moreno-Perez DA, Saldarriaga A, Patarroyo MA. Characterizing PvARP, a novel Plasmodium vivax antigen. Malar J. 2013;12: Mongui A, Angel DI, Gallego G, Reyes C, Martinez P, Guhl F, et al. Characterization and antigenicity of the promising vaccine candidate Plasmodium vivax 34kDa rhoptry antigen (Pv34). Vaccine. 2009;28(2): Moreno-Perez DA, Montenegro M, Patarroyo ME, Patarroyo MA. Identification, characterization and antigenicity of the Plasmodium vivax rhoptry neck protein 1 (PvRON1). Malar J. 2011;10: Arevalo-Pinzon G, Curtidor H, Patino LC, Patarroyo MA. PvRON2, a new Plasmodium vivax rhoptry neck antigen. Malar J. 2011;10: Arevalo-Pinzon G, Curtidor H, Abril J, Patarroyo MA. Annotation and characterization of the Plasmodium vivax rhoptry neck protein 4 (PvRON4). Malar J. 2013;12: Patarroyo ME, Patarroyo MA. Emerging rules for subunit-based, multiantigenic, multistage chemically synthesized vaccines. Accounts of chemical research. 2008;41(3):
104 104 Identificación y caracterización de PvGAMA en la cepa VCG-I de P. vivax
105 105 Como producto de la investigación, se tiene sometido un artículo en la revista Parásitos y Vectores, del cual estamos esperando respuesta a los comentarios menores de los revisores asignados.
UNIVERSIDAD DE LA SABANA
 UNIVERSIDAD DE LA SABANA Identificación y caracterización de la proteína del cuello de las roptrias 5 (RON5) en Plasmodium falciparum y determinación de las regiones de unión a glóbulos rojos humanos HERNANDO
UNIVERSIDAD DE LA SABANA Identificación y caracterización de la proteína del cuello de las roptrias 5 (RON5) en Plasmodium falciparum y determinación de las regiones de unión a glóbulos rojos humanos HERNANDO
GUIA DE APOYO. 1.- Complete el siguiente diagrama anotando los organelos y estructuras celulares correspondientes a cada tipo de célula.
 Royal American School Asignatura: Biología Profesor Mario Navarrete GUIA DE APOYO NOMBRE. CURSO 1º Medio. 1.- Complete el siguiente diagrama anotando los organelos y estructuras celulares correspondientes
Royal American School Asignatura: Biología Profesor Mario Navarrete GUIA DE APOYO NOMBRE. CURSO 1º Medio. 1.- Complete el siguiente diagrama anotando los organelos y estructuras celulares correspondientes
MALARIA VACUNAS. Alejandro Llanos Cuentas, MD, PhD Universidad Peruana Cayetano Heredia
 MALARIA VACUNAS Alejandro Llanos Cuentas, MD, PhD Universidad Peruana Cayetano Heredia Curso de Vacunología, Lima, 21-23 Octubre 2013 Malaria: Etiología 4 especies del género Plasmodium infectan humanos.
MALARIA VACUNAS Alejandro Llanos Cuentas, MD, PhD Universidad Peruana Cayetano Heredia Curso de Vacunología, Lima, 21-23 Octubre 2013 Malaria: Etiología 4 especies del género Plasmodium infectan humanos.
Antígeno completo (inmunógeno)= inmunogenicidad+ antigenicidad
 TEMA 5. Antígenos e inmunógenos. Definición de: Antígeno, Inmunógeno, Hapteno, Tolerógeno, Alergeno, Vacuna, Toxoide. Epitopos o determinantes antigénicos. Factores que afectan a la inmunogenicidad. Antígenos
TEMA 5. Antígenos e inmunógenos. Definición de: Antígeno, Inmunógeno, Hapteno, Tolerógeno, Alergeno, Vacuna, Toxoide. Epitopos o determinantes antigénicos. Factores que afectan a la inmunogenicidad. Antígenos
La célula. Unidad Los seres vivos están formados por células
 La célula Unidad Los seres vivos están formados por células Definición de célula. Es la unidad anatómico y funcional de todo ser vivo. Tiene función de autoconservación y autorreproducción. Es por esto,
La célula Unidad Los seres vivos están formados por células Definición de célula. Es la unidad anatómico y funcional de todo ser vivo. Tiene función de autoconservación y autorreproducción. Es por esto,
Guía para el educador sobre la malaria. (Información sobre herramientas) FINANCIADO POR AUTORES
 Guía para el educador sobre la malaria (Información sobre s) AUTORES FINANCIADO POR Índice Índice... 2 I. Introducción... 3 II. Las s... 3 1. Juego en vídeo: Ponle freno a la malaria... 4 2. Vídeo 1 Trabajar
Guía para el educador sobre la malaria (Información sobre s) AUTORES FINANCIADO POR Índice Índice... 2 I. Introducción... 3 II. Las s... 3 1. Juego en vídeo: Ponle freno a la malaria... 4 2. Vídeo 1 Trabajar
Malaria III Prof. Zilka
 Malaria III Prof. Zilka DIAGNÓSTICO DE MALARIA Gota gruesa y extensión de sangre teñido por Giemsa: es el más conocido y el más utilizado y no ha podido ser igualado. En estos métodos se utilizan 100 µl
Malaria III Prof. Zilka DIAGNÓSTICO DE MALARIA Gota gruesa y extensión de sangre teñido por Giemsa: es el más conocido y el más utilizado y no ha podido ser igualado. En estos métodos se utilizan 100 µl
Unidad básica estructural y funcional de todos los seres vivos.
 Unidad básica estructural y funcional de todos los seres vivos. Robert Hooke Siglo XVII Observó que el corcho y otras materias vegetales aparecían constituidas por celdillas. De allí viene el nombre de
Unidad básica estructural y funcional de todos los seres vivos. Robert Hooke Siglo XVII Observó que el corcho y otras materias vegetales aparecían constituidas por celdillas. De allí viene el nombre de
ABREVIATURAS... XI I. INTRODUCCIÓN EL TOMATE... 3
 ÍNDICE ABREVIATURAS... XI I. INTRODUCCIÓN... 1 1 EL TOMATE... 3 1.1 Taxonomía... 3 1.2 Características generales... 3 1.3 La flor... 4 1.4 Características del fruto... 5 2 CUAJADO Y DESARROLLO DEL FRUTO...
ÍNDICE ABREVIATURAS... XI I. INTRODUCCIÓN... 1 1 EL TOMATE... 3 1.1 Taxonomía... 3 1.2 Características generales... 3 1.3 La flor... 4 1.4 Características del fruto... 5 2 CUAJADO Y DESARROLLO DEL FRUTO...
UNIDAD II ANATOMÍA Y FISIOLOGÍA DE LA CÉLULA. Prof. Glamil Acevedo Anatomía y Fisiología
 UNIDAD II ANATOMÍA Y FISIOLOGÍA DE LA CÉLULA Prof. Glamil Acevedo Anatomía y Fisiología La Célula Es la unidad funcional y estructural más pequeña de los organismos vivos. Se compone de partes características,
UNIDAD II ANATOMÍA Y FISIOLOGÍA DE LA CÉLULA Prof. Glamil Acevedo Anatomía y Fisiología La Célula Es la unidad funcional y estructural más pequeña de los organismos vivos. Se compone de partes características,
PREGUNTAS DE SELECTIVIDAD POR TEMAS
 BIOMOLÉCULAS PREGUNTAS DE SELECTIVIDAD POR TEMAS A. Defina los siguientes términos: a. Polisacáridos. (1 punto) b. Lípidos saponificables. (1 punto) B. Dada la siguiente secuencia de ADN: 3' TACCTACACAGATCTTGC
BIOMOLÉCULAS PREGUNTAS DE SELECTIVIDAD POR TEMAS A. Defina los siguientes términos: a. Polisacáridos. (1 punto) b. Lípidos saponificables. (1 punto) B. Dada la siguiente secuencia de ADN: 3' TACCTACACAGATCTTGC
Apoptosis. Término que hace referencia a los procesos morfológicos que llevan a la autodestrucción celular controlada
 APOPTOSIS Apoptosis Término que hace referencia a los procesos morfológicos que llevan a la autodestrucción celular controlada Esta muerte es necesaria y parte integral del ciclo de vida de los organismos
APOPTOSIS Apoptosis Término que hace referencia a los procesos morfológicos que llevan a la autodestrucción celular controlada Esta muerte es necesaria y parte integral del ciclo de vida de los organismos
La célula. En que se diferencia una célula animal de una vegetal? Dónde se localiza el material hereditario?
 La célula En que se diferencia una célula animal de una vegetal? Dónde se localiza el material hereditario? Definición de célula. Es la unidad anatómico y funcional de todo ser vivo. Tiene función de autoconservación
La célula En que se diferencia una célula animal de una vegetal? Dónde se localiza el material hereditario? Definición de célula. Es la unidad anatómico y funcional de todo ser vivo. Tiene función de autoconservación
2. La Influenza A/H1N1
 2. La Influenza A/H1N1 Haemagglutinin (HA) Influenza Virus Neuraminidase (NA) El gráfico representa una partícula viral completa del virus (virión) de influenza. El virus posee una envoltura externa que
2. La Influenza A/H1N1 Haemagglutinin (HA) Influenza Virus Neuraminidase (NA) El gráfico representa una partícula viral completa del virus (virión) de influenza. El virus posee una envoltura externa que
UNIDAD 1. D. Borja Blanco Vives. Profesor de Biología y Geología 4ºESO
 UNIDAD 1. D. Borja Blanco Vives. Profesor de Biología y Geología 4ºESO 1. LA COMPOSICIÓN DE LOS SERES VIVOS. Todos los seres vivos están formados por: - materia inorgánica: agua y sales minerales - materia
UNIDAD 1. D. Borja Blanco Vives. Profesor de Biología y Geología 4ºESO 1. LA COMPOSICIÓN DE LOS SERES VIVOS. Todos los seres vivos están formados por: - materia inorgánica: agua y sales minerales - materia
AGRADECIMIENTOS... VII RESUMEN... XIII RESUM... XV SUMMARY... XVII ABREVIATURAS... XIX ÍNDICE DE CONTENIDO... XXIII ÍNDICE DE FIGURAS...
 ÍNDICE AGRADECIMIENTOS... VII RESUMEN... XIII RESUM... XV SUMMARY... XVII ABREVIATURAS... XIX ÍNDICE DE CONTENIDO... XXIII ÍNDICE DE FIGURAS... XXXI ÍNDICE DE TABLAS... XXXVII INTRODUCCIÓN... 1 1. La mitocondria...
ÍNDICE AGRADECIMIENTOS... VII RESUMEN... XIII RESUM... XV SUMMARY... XVII ABREVIATURAS... XIX ÍNDICE DE CONTENIDO... XXIII ÍNDICE DE FIGURAS... XXXI ÍNDICE DE TABLAS... XXXVII INTRODUCCIÓN... 1 1. La mitocondria...
Composición Líquido Elementos formes
 Composición Líquido Elementos formes Plasma Glóbulos blancos Glóbulos rojos Plaquetas Transporte de sustancias Homeostasis de los líquidos l corporales Protección TRANSPORTE O 2 desde los pulmones a los
Composición Líquido Elementos formes Plasma Glóbulos blancos Glóbulos rojos Plaquetas Transporte de sustancias Homeostasis de los líquidos l corporales Protección TRANSPORTE O 2 desde los pulmones a los
Acerca del libro Nada en la biología tiene sentido, si no se considera a la luz de la evolución. El origen de la vida.
 Acerca del libro Los contenidos de Biología 2 responden a la nueva currícula del programa de biología para segundo año del Gobierno de la Ciudad de Buenos Aires. La evolución constituye el hilo conductor
Acerca del libro Los contenidos de Biología 2 responden a la nueva currícula del programa de biología para segundo año del Gobierno de la Ciudad de Buenos Aires. La evolución constituye el hilo conductor
Desarrollo de vacunas contra parásitos
 ciencia Julio Vladimir Cruz Chan, Miguel Enrique Rosado Vallado y Eric Dumonteil n n nnn n n Desarrollo de vacunas contra parásitos Aún no existen vacunas contra enfermedades como la malaria, leishmaniosis,
ciencia Julio Vladimir Cruz Chan, Miguel Enrique Rosado Vallado y Eric Dumonteil n n nnn n n Desarrollo de vacunas contra parásitos Aún no existen vacunas contra enfermedades como la malaria, leishmaniosis,
Estructura y dinámica del citoesqueleto
 Estructura y dinámica del citoesqueleto Dr. Julio Tapia Pineda Programa de Biología Celular y Molecular ICBM, Facultad de Medicina TERAPIA OCUPACIONAL Abril 2012 Compartimentalización de una célula eucarionte
Estructura y dinámica del citoesqueleto Dr. Julio Tapia Pineda Programa de Biología Celular y Molecular ICBM, Facultad de Medicina TERAPIA OCUPACIONAL Abril 2012 Compartimentalización de una célula eucarionte
Selectividad: ANATOMÍA CELULAR
 Selectividad: ANATOMÍA CELULAR Jun09.3.- La figura siguiente muestra un orgánulo celular importante. a.- Cómo se llama este orgánulo? b.- En qué tipo de células se encuentra? c.- Indica qué estructura
Selectividad: ANATOMÍA CELULAR Jun09.3.- La figura siguiente muestra un orgánulo celular importante. a.- Cómo se llama este orgánulo? b.- En qué tipo de células se encuentra? c.- Indica qué estructura
Nadia Isabel Hornquist Hurtarte Química Bióloga Clínica de Enfermedades Infecciosas Hospital Roosevelt
 Nadia Isabel Hornquist Hurtarte Química Bióloga Clínica de Enfermedades Infecciosas Hospital Roosevelt Fase aguda: Entre el 40% a 90% sintomáticos (similar mononucleosis) Fase crónica: asintomaticos El
Nadia Isabel Hornquist Hurtarte Química Bióloga Clínica de Enfermedades Infecciosas Hospital Roosevelt Fase aguda: Entre el 40% a 90% sintomáticos (similar mononucleosis) Fase crónica: asintomaticos El
2. ESTRATEGIA PARA EFECTUAR LA CLONACION Y EXPRESION DEL GEN
 1. CONTROL FINAL Nombre Internacional y nombre de la vacuna Nombre del propietario Nombre y dirección del fabricante Número de Lote Fecha de fabricación Fecha de caducidad Temperatura de almacenamiento
1. CONTROL FINAL Nombre Internacional y nombre de la vacuna Nombre del propietario Nombre y dirección del fabricante Número de Lote Fecha de fabricación Fecha de caducidad Temperatura de almacenamiento
- Microtúbulos - Microfilamentos - Filamentos intermedios
 Citoesqueleto Cómo se mantiene la forma de una célula? Cómo se transportan proteínas y otras sustancias a través del citoplasma? Cómo se ubican las distintas organelas? La respuesta a estas y otras preguntas
Citoesqueleto Cómo se mantiene la forma de una célula? Cómo se transportan proteínas y otras sustancias a través del citoplasma? Cómo se ubican las distintas organelas? La respuesta a estas y otras preguntas
LA NUEVA BIOTECNOLOGÍA
 LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
Preguntas de Selectividad sobre inmunología.
 Preguntas de Selectividad sobre inmunología. 1. Existen virus que producen en los humanos enfermedades mortales por inmunodeficiencia. Sin embargo, la muerte del individuo no es provocada directamente
Preguntas de Selectividad sobre inmunología. 1. Existen virus que producen en los humanos enfermedades mortales por inmunodeficiencia. Sin embargo, la muerte del individuo no es provocada directamente
Estructura celular. Teoría Celular. Cap. 4. Los organismos se componen de una o más células. La célula es la unidad más pequeña de la vida.
 Estructura celular Cap. 4 Teoría Celular Los organismos se componen de una o más células. Procesos metabólicos y de la herencia ocurren dentro de la célula. La célula es la unidad más pequeña de la vida.
Estructura celular Cap. 4 Teoría Celular Los organismos se componen de una o más células. Procesos metabólicos y de la herencia ocurren dentro de la célula. La célula es la unidad más pequeña de la vida.
Nombres alternativos de la Respuesta Inmune Adaptativa
 Nombres alternativos de la Respuesta Inmune Adaptativa Inmunidad Adaptativa: Porque se produce como respuesta a la infección y se adapta a esta Inmunidad Específica: Porque es capaz de distinguir entre
Nombres alternativos de la Respuesta Inmune Adaptativa Inmunidad Adaptativa: Porque se produce como respuesta a la infección y se adapta a esta Inmunidad Específica: Porque es capaz de distinguir entre
Microorganismos. Microbiología General 11-P
 Microorganismos Microbiología General 11-P Microorganismo Los microorganismos también llamados microbios son seres diminutos que individualmente son demasiado pequeños para ser observados a simple vista.
Microorganismos Microbiología General 11-P Microorganismo Los microorganismos también llamados microbios son seres diminutos que individualmente son demasiado pequeños para ser observados a simple vista.
Definiciones de Inmunología
 Definiciones de Inmunología Son sustancias extrañas a nuestro organismo que desencadenan la formación de Anticuerpos (Ac). La unión entre Ag y Ac es de naturaleza no covalente. Hay complementariedad entre
Definiciones de Inmunología Son sustancias extrañas a nuestro organismo que desencadenan la formación de Anticuerpos (Ac). La unión entre Ag y Ac es de naturaleza no covalente. Hay complementariedad entre
El descubrimiento de la célula
 República Bolivariana de Venezuela Ministerio del Poder Popular para la Educación U.E. Colegio Santo Tomás de Villanueva Departamento de Ciencias Cátedra: Ciencias Biológicas 3 Año Tema III: La Célula
República Bolivariana de Venezuela Ministerio del Poder Popular para la Educación U.E. Colegio Santo Tomás de Villanueva Departamento de Ciencias Cátedra: Ciencias Biológicas 3 Año Tema III: La Célula
Plataforma para la monitorización de la Gripe en España
 Plataforma para la monitorización de la Gripe en España La gripe es una de las enfermedades más contagiosas que existen y cada año es responsable de la muerte de cientos de miles de personas en todo el
Plataforma para la monitorización de la Gripe en España La gripe es una de las enfermedades más contagiosas que existen y cada año es responsable de la muerte de cientos de miles de personas en todo el
LA CÉLULA. Hecho por: Alba García Murillo 1º B
 LA CÉLULA Hecho por: Alba García Murillo 1º B dhñ Xf R La célula es la unidad más pequeña dotada de vida. Todos los seres vivos están formados por células: -Una célula : unicelulares Ejem: La salmonella
LA CÉLULA Hecho por: Alba García Murillo 1º B dhñ Xf R La célula es la unidad más pequeña dotada de vida. Todos los seres vivos están formados por células: -Una célula : unicelulares Ejem: La salmonella
dentro y hacia afuera de la célula (secreción) Metabolismo de lípidos.
 BIOLOGÍA GUÍA DE EJERCITACIÓN 1 RESPUESTAS PREGUNTA 1 Nombre Función 1 Nucléolo Síntesis de ribosomas 2 Núcleo Almacena la información genética (ADN en la forma de cromosomas). Lugar donde ocurre la síntesis
BIOLOGÍA GUÍA DE EJERCITACIÓN 1 RESPUESTAS PREGUNTA 1 Nombre Función 1 Nucléolo Síntesis de ribosomas 2 Núcleo Almacena la información genética (ADN en la forma de cromosomas). Lugar donde ocurre la síntesis
USO DE ALERGENOS EN LA CLÍNICA Escuela Superior de Medicina.
 USO DE ALERGENOS EN LA CLÍNICA Escuela Superior de Medicina. QUÉ ES UN ALERGENO? Cualquier antígeno que induce una respuesta mediada por IgE. - El término involucra las moléculas antigénicas y/o las fuentes
USO DE ALERGENOS EN LA CLÍNICA Escuela Superior de Medicina. QUÉ ES UN ALERGENO? Cualquier antígeno que induce una respuesta mediada por IgE. - El término involucra las moléculas antigénicas y/o las fuentes
Capítulo 12 REGULACIÓN DE LA EXPRESIÓN GÉNICA. Factores de Transcripción. Metilación. Procesamiento del ARN. Post-traduccional
 REGULACIÓN DE LA EXPRESIÓN GÉNICA - Mecanismos de Regulación Regulación Procariontes Eucariontes Operón Lactosa Operón Triptofano Transcripcional Procesamiento del ARN Traduccional Post-traduccional Factores
REGULACIÓN DE LA EXPRESIÓN GÉNICA - Mecanismos de Regulación Regulación Procariontes Eucariontes Operón Lactosa Operón Triptofano Transcripcional Procesamiento del ARN Traduccional Post-traduccional Factores
Insuficiencia de hierro...
 Insuficiencia de hierro... TFR Un diagnóstico eficaz de su Receptor Soluble de Transferrina. Qué es el Receptor Soluble de Transferrina (TFR)? Es una proteína de transmembrana presente en todas las células.
Insuficiencia de hierro... TFR Un diagnóstico eficaz de su Receptor Soluble de Transferrina. Qué es el Receptor Soluble de Transferrina (TFR)? Es una proteína de transmembrana presente en todas las células.
Colegio Marista San José - Ampliación de Biología y Geología Tema 5: Del átomo a la célula.
 La teoría celular. La idea de que los seres vivos están constituidos por células y que cada una de ellas proviene de otra, que ya existía con anterioridad, es relativamente reciente. Es en la segunda mitad
La teoría celular. La idea de que los seres vivos están constituidos por células y que cada una de ellas proviene de otra, que ya existía con anterioridad, es relativamente reciente. Es en la segunda mitad
1 sesión: Presentación de la asignatura y criterios de evaluación. 1 sesión: Prueba inicial. UNIDAD DIDÁCTICA I LAS MOLÉCULAS DE LA VIDA
 PRIMER TRIMESTRE TOTAL TRIMESTRE 42 SESIONES 1 sesión: Presentación de la asignatura y criterios de evaluación. 1 sesión: Prueba inicial. UNIDAD DIDÁCTICA I LAS MOLÉCULAS DE LA VIDA TEMA 1. BIOELEMENTOS
PRIMER TRIMESTRE TOTAL TRIMESTRE 42 SESIONES 1 sesión: Presentación de la asignatura y criterios de evaluación. 1 sesión: Prueba inicial. UNIDAD DIDÁCTICA I LAS MOLÉCULAS DE LA VIDA TEMA 1. BIOELEMENTOS
Técnicas de Estudio de las células
 Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO
 Alianza para el Aprendizaje de Ciencias y Matemáticas CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO GUÍA DEL MAESTRO ESTÁNDARES ATENDIDOS: 1. LA NATURALEZA
Alianza para el Aprendizaje de Ciencias y Matemáticas CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO GUÍA DEL MAESTRO ESTÁNDARES ATENDIDOS: 1. LA NATURALEZA
Estructura y Función de la Célula
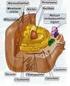 Estructura y Función de la Célula Analisis de Video 1. Que es un celula? 2. Mencione un ejemplo de una celular procariota? Que significa procariota? 3. Nombre del organismo celular que no posee ADN. 4.
Estructura y Función de la Célula Analisis de Video 1. Que es un celula? 2. Mencione un ejemplo de una celular procariota? Que significa procariota? 3. Nombre del organismo celular que no posee ADN. 4.
LABORATORIO No8 PROTOZOOARIOS TISULARES Y HEMATICOS
 LABORATORIO No8 PROTOZOOARIOS TISULARES Y HEMATICOS Plasmodium (malaria): Son cuatro las especies de plasmodium que parasitan al hombre: Plasmodium vivax Plasmodium falciparum Plasmodium ovale Plasmodium
LABORATORIO No8 PROTOZOOARIOS TISULARES Y HEMATICOS Plasmodium (malaria): Son cuatro las especies de plasmodium que parasitan al hombre: Plasmodium vivax Plasmodium falciparum Plasmodium ovale Plasmodium
Biotecnología y su aplicación a las ciencias agropecuarias. Oris Sanjur Instituto Smithsonian de Investigaciones Tropicales
 Biotecnología y su aplicación a las ciencias agropecuarias Oris Sanjur Instituto Smithsonian de Investigaciones Tropicales Niveles de organización en seres vivos Organismo Organo Tejido Celula La Célula:
Biotecnología y su aplicación a las ciencias agropecuarias Oris Sanjur Instituto Smithsonian de Investigaciones Tropicales Niveles de organización en seres vivos Organismo Organo Tejido Celula La Célula:
Generalidades de Protozoos
 Generalidades de Protozoos Protozoos: Organismos unicelulares (microscópicos, formados por una sola célula) eucariontes (material genético protegido por una membrana nuclear). Estructura y Metabolismo
Generalidades de Protozoos Protozoos: Organismos unicelulares (microscópicos, formados por una sola célula) eucariontes (material genético protegido por una membrana nuclear). Estructura y Metabolismo
GENÉTICA MOLECULAR. Unidad 1: Introducción a la genética molecular
 GENÉTICA MOLECULAR CONTENIDOS CONCEPTUALES COMPETENCIAS Unidad 1: Introducción a la genética molecular Concepto de genética molecular y aplicaciones a diferentes ramas de la ciencia. Dogma central de la
GENÉTICA MOLECULAR CONTENIDOS CONCEPTUALES COMPETENCIAS Unidad 1: Introducción a la genética molecular Concepto de genética molecular y aplicaciones a diferentes ramas de la ciencia. Dogma central de la
GUÍA DE ESTUDIO VEGETALISTA. Proyecto Vegetalista Guía de Estudio para el alumno
 Proyecto Vegetalista Guía de Estudio para el alumno Indicaciones al docente Estimado (a) profesor (a): El siguiente material, ha sido preparado para complementar y profundizar temas vistos en clases y
Proyecto Vegetalista Guía de Estudio para el alumno Indicaciones al docente Estimado (a) profesor (a): El siguiente material, ha sido preparado para complementar y profundizar temas vistos en clases y
Vigilancia de Malaria. Chile, 2011 2014.
 1 Vol. 5, No. 2, febrero 2015. Vigilancia de Malaria. Chile, 2011 2014. 1. Antecedentes La Malaria o paludismo, es una enfermedad potencialmente mortal causada por parásitos que se transmiten al ser humano
1 Vol. 5, No. 2, febrero 2015. Vigilancia de Malaria. Chile, 2011 2014. 1. Antecedentes La Malaria o paludismo, es una enfermedad potencialmente mortal causada por parásitos que se transmiten al ser humano
La célula. Por Pedro Álvarez Maíllo. 1. Marca las características comunes a todos los seres vivos. Todos poseen grupos de moléculas muy semejantes.
 La célula Por Pedro Álvarez Maíllo Identifica la respuesta/as correcta/as 1. Marca las características comunes a todos los seres vivos. Todos poseen grupos de moléculas muy semejantes. Todos están formados
La célula Por Pedro Álvarez Maíllo Identifica la respuesta/as correcta/as 1. Marca las características comunes a todos los seres vivos. Todos poseen grupos de moléculas muy semejantes. Todos están formados
Soluciones de la serie de ejercicios 7 (7.012)
 Nombre AT Grupo Pregunta 1 Soluciones de la serie de ejercicios 7 (7.012) a) Mi gata, Sophie, está enfadada porque paso demasiado tiempo trabajando como profesor auxiliar. Una noche, en un ataque de celos,
Nombre AT Grupo Pregunta 1 Soluciones de la serie de ejercicios 7 (7.012) a) Mi gata, Sophie, está enfadada porque paso demasiado tiempo trabajando como profesor auxiliar. Una noche, en un ataque de celos,
Tema 7. La clasificación de los seres vivos. Microorganismos
 La clasificación de los seres vivos Tema 7 La clasificación de los seres vivos. Microorganismos Biología y Geología Número de especies Número de grupos La clasificación de los seres vivos Los criterios
La clasificación de los seres vivos Tema 7 La clasificación de los seres vivos. Microorganismos Biología y Geología Número de especies Número de grupos La clasificación de los seres vivos Los criterios
BLOQUE II. ORGANIZACIÓN Y FISIOLOGÍA CELULAR
 I.E.S. Flavio Irnitano El Saucejo (Sevilla) Curso 2.015 2.016 Departamento de Biología y Geología NIVEL: 2º Bachillerato MATERIA: BIOLOGÍA BLOQUE II. ORGANIZACIÓN Y FISIOLOGÍA CELULAR 3. CÉLULA EUCARIÓTICA.
I.E.S. Flavio Irnitano El Saucejo (Sevilla) Curso 2.015 2.016 Departamento de Biología y Geología NIVEL: 2º Bachillerato MATERIA: BIOLOGÍA BLOQUE II. ORGANIZACIÓN Y FISIOLOGÍA CELULAR 3. CÉLULA EUCARIÓTICA.
PREGUNTAS DE LA PRUEBA DE ACCESO A LA UNIVERSIDAD
 CÉLULA III CITOESQUELETO Y ORGÁNULOS NO MEMBRANOSOS 1. Indique qué es el citoesqueleto [0,4]. Describa los elementos del mismo [0,6] y las funciones que desempeñan relacionándolas con el elemento correspondiente
CÉLULA III CITOESQUELETO Y ORGÁNULOS NO MEMBRANOSOS 1. Indique qué es el citoesqueleto [0,4]. Describa los elementos del mismo [0,6] y las funciones que desempeñan relacionándolas con el elemento correspondiente
Facultad de Ciencias Agropecuarias Universidad Nacional de Córdoba. Biología Celular. Práctico N 3 Sistema de endomembranas
 Facultad de Ciencias Agropecuarias Universidad Nacional de Córdoba Biología Celular Práctico N 3 OBJETIVOS Identificar los distintos componentes del sistema de endomembranas en la célula. Comprender la
Facultad de Ciencias Agropecuarias Universidad Nacional de Córdoba Biología Celular Práctico N 3 OBJETIVOS Identificar los distintos componentes del sistema de endomembranas en la célula. Comprender la
EPIDEMIOLOGÍA DE LA MALARIA CICLO BIOLÓGICO DEL PLASMODIUM
 EPIDEMIOLOGÍA DE LA MALARIA CICLO BIOLÓGICO DEL PLASMODIUM Trinidad Sabalete Moya Fundación IO Centro de Enfermedades Infecciosas y Salud Internacional Granada PROBLEMA DE SALUD PÚBLICA MUNDIAL ENFERMEDAD
EPIDEMIOLOGÍA DE LA MALARIA CICLO BIOLÓGICO DEL PLASMODIUM Trinidad Sabalete Moya Fundación IO Centro de Enfermedades Infecciosas y Salud Internacional Granada PROBLEMA DE SALUD PÚBLICA MUNDIAL ENFERMEDAD
COMPLEJO EDUCACIONAL JOAQUIN EDWARDS BELLO GUÍA REFORZAMIENTO 1 MEDIO
 COMPLEJO EDUCACIONAL JOAQUIN EDWARDS BELLO GUÍA REFORZAMIENTO 1 MEDIO NOMBRE: PJE. TOTAL: FECHA: CURSO: 1 MEDIO PJE. OBTENIDO: CALIFICACION: ASIGNATURA: BIOLOGIA UNIDAD: I - II OBJETIVO (S): CONTENIDO
COMPLEJO EDUCACIONAL JOAQUIN EDWARDS BELLO GUÍA REFORZAMIENTO 1 MEDIO NOMBRE: PJE. TOTAL: FECHA: CURSO: 1 MEDIO PJE. OBTENIDO: CALIFICACION: ASIGNATURA: BIOLOGIA UNIDAD: I - II OBJETIVO (S): CONTENIDO
Células pequeñas 1 10 µm Células de mayor tamaño; µm
 La Célula REPASO La Célula - Repaso Diferencias entre célula procariota y eucariota Diferencias entre célula animal y vegetal Membrana celular Estructura Funciones Citoplasma Hialoplasma (composición)
La Célula REPASO La Célula - Repaso Diferencias entre célula procariota y eucariota Diferencias entre célula animal y vegetal Membrana celular Estructura Funciones Citoplasma Hialoplasma (composición)
UD 2. CÉLULAS Y ESTRUCTURAS SUBCELULARES
 PRUEBAS DE ACCESO A LA UNIVERSIDAD. BACHILLERATO LOGSE. CANTABRIA. BIOLOGÍA (2001-2011). UD 2. CÉLULAS Y ESTRUCTURAS SUBCELULARES Introducción. Clasificación 1. Dibuja una célula eucariótica y una procariótica,
PRUEBAS DE ACCESO A LA UNIVERSIDAD. BACHILLERATO LOGSE. CANTABRIA. BIOLOGÍA (2001-2011). UD 2. CÉLULAS Y ESTRUCTURAS SUBCELULARES Introducción. Clasificación 1. Dibuja una célula eucariótica y una procariótica,
TECNOLOGÍA EN SALUD DE INTERÉS Deferasirox
 TECNOLOGÍA EN SALUD DE INTERÉS Deferasirox 1. RESUMEN Título del reporte: Evaluación de efectividad y seguridad de deferasirox en Hemosiderosis Transfusional Información general de la tecnología: deferasirox
TECNOLOGÍA EN SALUD DE INTERÉS Deferasirox 1. RESUMEN Título del reporte: Evaluación de efectividad y seguridad de deferasirox en Hemosiderosis Transfusional Información general de la tecnología: deferasirox
ESTRUCTURA Y FUNCION CELULAR
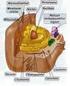 ESTRUCTURA Y FUNCION CELULAR Sumario Historia de la teoría celular Estructura y función celular 1. El núcleo 2. Los organelos citoplásmicos 3. Los organelos de células vegetales Transporte celular Métodos
ESTRUCTURA Y FUNCION CELULAR Sumario Historia de la teoría celular Estructura y función celular 1. El núcleo 2. Los organelos citoplásmicos 3. Los organelos de células vegetales Transporte celular Métodos
7.012 Serie de ejercicios 7
 Nombre AT Grupo 7.012 Serie de ejercicios 7 Pregunta 1 a) Mi gata, Sophie, está enfadada porque paso demasiado tiempo trabajando como profesor auxiliar. Una noche, en un ataque de celos, me arañó un dedo.
Nombre AT Grupo 7.012 Serie de ejercicios 7 Pregunta 1 a) Mi gata, Sophie, está enfadada porque paso demasiado tiempo trabajando como profesor auxiliar. Una noche, en un ataque de celos, me arañó un dedo.
Desarrollo de nuevas líneas celulares permisivas a PRRSV, y sus implicaciones próximas. Paloma Suárez, DVM, Ph.D New Products Marketing Madison, NJ
 Desarrollo de nuevas líneas celulares permisivas a PRRSV, y sus implicaciones próximas Paloma Suárez, DVM, Ph.D New Products Marketing Madison, NJ 1 Introducción Primera descripción de la enfermedad a
Desarrollo de nuevas líneas celulares permisivas a PRRSV, y sus implicaciones próximas Paloma Suárez, DVM, Ph.D New Products Marketing Madison, NJ 1 Introducción Primera descripción de la enfermedad a
Biologia Celular CBI 111 Instituto de Ciencias Naturales. Estructura y función de la Membrana Citoplasmática. Clase 6
 Biologia Celular CBI 111 Instituto de Ciencias Naturales Estructura y función de la Membrana Citoplasmática. Clase 6 MATERIAL PROPIEDAD DE UDLA. AUTORIZADA SU UTILIZACIÓN SÓLO PARA FINES ACADÉMICOS. Objetivos
Biologia Celular CBI 111 Instituto de Ciencias Naturales Estructura y función de la Membrana Citoplasmática. Clase 6 MATERIAL PROPIEDAD DE UDLA. AUTORIZADA SU UTILIZACIÓN SÓLO PARA FINES ACADÉMICOS. Objetivos
1. Citosol 2. Citoesqueleto 3. Cilios y flagelos 4. El centrosoma. 1. El Citosol
 1. Citosol 2. Citoesqueleto 3. Cilios y flagelos 4. El centrosoma 1. El Citosol El Citosol, también denominado hialoplasma es el medio acuoso del citoplasma en el que se encuentran inmersos los orgánulos
1. Citosol 2. Citoesqueleto 3. Cilios y flagelos 4. El centrosoma 1. El Citosol El Citosol, también denominado hialoplasma es el medio acuoso del citoplasma en el que se encuentran inmersos los orgánulos
Rotavirus: pequeños individuos con grandes
 Rotavirus: pequeños individuos con grandes repercusiones Edgar Reyna Rosas Los rotavirus son algunos de esos virus con los que convivimos diariamente. Debido a que las primeras veces que se observaron
Rotavirus: pequeños individuos con grandes repercusiones Edgar Reyna Rosas Los rotavirus son algunos de esos virus con los que convivimos diariamente. Debido a que las primeras veces que se observaron
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
La célula y sus estructuras. Profesor Mauricio Hernández F Biología 1 Medio
 La célula y sus estructuras INTRODUCCIÓN Toda célula posee Membrana plasmática: límite Matriz coloidal: contiene las estructuras intracelulares Material genético (ADN): dirige el funcionamiento celular;
La célula y sus estructuras INTRODUCCIÓN Toda célula posee Membrana plasmática: límite Matriz coloidal: contiene las estructuras intracelulares Material genético (ADN): dirige el funcionamiento celular;
Tabla: Tasas de incidencia por año 2005 hasta semana 34 de 2010
 VIGILANCIA EPIDEMIOLOGICA DE LAS ENFERMEDADES TRANSMITIDAS POR VECTORES (Dengue, Leishmaniasis, Malaria) VIGILANCIA DEL DENGUE CLASICO Y DENGUE GRAVE En la actualidad el Dengue en nuestra Región es la
VIGILANCIA EPIDEMIOLOGICA DE LAS ENFERMEDADES TRANSMITIDAS POR VECTORES (Dengue, Leishmaniasis, Malaria) VIGILANCIA DEL DENGUE CLASICO Y DENGUE GRAVE En la actualidad el Dengue en nuestra Región es la
LA CÉLULA Y SUS ORGANELOS. Colegio Internacional Montessori Ciencias Naturales-1ro Básico Walter Moscoso Zschech
 LA CÉLULA Y SUS ORGANELOS Colegio Internacional Montessori Ciencias Naturales-1ro Básico Walter Moscoso Zschech LA CÉLULA Es la unidad básica de la vida. Según la teoría celular todos los organismos vivos:
LA CÉLULA Y SUS ORGANELOS Colegio Internacional Montessori Ciencias Naturales-1ro Básico Walter Moscoso Zschech LA CÉLULA Es la unidad básica de la vida. Según la teoría celular todos los organismos vivos:
CURSO CONVOCATORIA: JUNIO BIOLOGIA
 PRUEBAS DE ACCESO A LA UNIVERSIDAD L.O.G.S.E. / L.O.C.E. CURSO 00 005 CONVOCATORIA: JUNIO MATERIA: BIOLOGIA ACLARACIONES PREVIAS El alumno debe elegir una de las dos opciones, A o B, de que consta la prueba,
PRUEBAS DE ACCESO A LA UNIVERSIDAD L.O.G.S.E. / L.O.C.E. CURSO 00 005 CONVOCATORIA: JUNIO MATERIA: BIOLOGIA ACLARACIONES PREVIAS El alumno debe elegir una de las dos opciones, A o B, de que consta la prueba,
Celulas eucariotas. Cáncer de pulmon
 QUE ES CELULA? Unidad fundamental de todo ser vivo. Fue descubierta por ROBERT HOOKE observando un corte de corcho en un microscopio de la época. Dio el nombre de CELULA a las estructuras regulares en
QUE ES CELULA? Unidad fundamental de todo ser vivo. Fue descubierta por ROBERT HOOKE observando un corte de corcho en un microscopio de la época. Dio el nombre de CELULA a las estructuras regulares en
Barriga Mejia Brenda Paloma López Alavez Ana Maria Martinez Romero Jorge Manuel Grupo: 2405 BACTERIAS
 Barriga Mejia Brenda Paloma López Alavez Ana Maria Martinez Romero Jorge Manuel Grupo: 2405 BACTERIAS Bacterias Las bacterias son microorganismos procariotas que pertenecen al reino monera. Presentan un
Barriga Mejia Brenda Paloma López Alavez Ana Maria Martinez Romero Jorge Manuel Grupo: 2405 BACTERIAS Bacterias Las bacterias son microorganismos procariotas que pertenecen al reino monera. Presentan un
SESIÓN 3. Describir las características distintivas de los seres vivos a partir de modelos naturales.
 SESIÓN 3. Características distintivas de los seres vivos OBJETIVO DE LA SESIÓN Describir las características distintivas de los seres vivos a partir de modelos naturales. INTRODUCCIÓN Independientemente
SESIÓN 3. Características distintivas de los seres vivos OBJETIVO DE LA SESIÓN Describir las características distintivas de los seres vivos a partir de modelos naturales. INTRODUCCIÓN Independientemente
X-Plain El paludismo (la malaria) Sumario
 X-Plain El paludismo (la malaria) Sumario El paludismo (o la malaria) es una de las infecciones parasíticas más comunes en el mundo. Cada año aparecen unos 500 millones de casos nuevos, causando cerca
X-Plain El paludismo (la malaria) Sumario El paludismo (o la malaria) es una de las infecciones parasíticas más comunes en el mundo. Cada año aparecen unos 500 millones de casos nuevos, causando cerca
Procariota y Eucariota
 Célula Todas las células comparten dos características esenciales. La primera es una membrana externa, la membrana celular -o membrana plasmática- que separa el citoplasma de la célula de su ambiente externo.
Célula Todas las células comparten dos características esenciales. La primera es una membrana externa, la membrana celular -o membrana plasmática- que separa el citoplasma de la célula de su ambiente externo.
Repaso: Química celular (biomoléculas)
 Repaso: Química celular (biomoléculas) Hay 4 tipos principales de biomoléculas: 1) glúcidos o hidratos de carbono, 2) lípidos o grasas, 3) proteínas y 4) ácidos nucleicos. Las biomoléculas más grandes,
Repaso: Química celular (biomoléculas) Hay 4 tipos principales de biomoléculas: 1) glúcidos o hidratos de carbono, 2) lípidos o grasas, 3) proteínas y 4) ácidos nucleicos. Las biomoléculas más grandes,
La célula: organelos
 La célula: organelos Teoría celular ( 1839 ) Teoría celular: Historia y antecedentes Robert Hooke 1665 Observó paredes celulares usando un microscopio, en una lámina de corcho. Acuñó el concepto de célula
La célula: organelos Teoría celular ( 1839 ) Teoría celular: Historia y antecedentes Robert Hooke 1665 Observó paredes celulares usando un microscopio, en una lámina de corcho. Acuñó el concepto de célula
UNIDAD 4: LIVING THINGS (LOS SERES VIVOS)
 REPASO EN ESPAÑOL C.E.I.P. GLORIA FUERTES NATURAL SCIENCE 6 UNIDAD 4: LIVING THINGS (LOS SERES VIVOS) Nuestro mundo está formado por seres vivos y seres inertes. Muchos de estos seres vivos son muy pequeños,
REPASO EN ESPAÑOL C.E.I.P. GLORIA FUERTES NATURAL SCIENCE 6 UNIDAD 4: LIVING THINGS (LOS SERES VIVOS) Nuestro mundo está formado por seres vivos y seres inertes. Muchos de estos seres vivos son muy pequeños,
Biosíntesis de Ácidos nucleicos (ARN) y Proteínas
 Biosíntesis de Ácidos nucleicos (ARN) y Proteínas Núcleo celular Dogma central de la Biología Molecular Repasando los Ácidos nucleicos. Empaquetamiento de ADN 46 cromosomas en las células somáticas del
Biosíntesis de Ácidos nucleicos (ARN) y Proteínas Núcleo celular Dogma central de la Biología Molecular Repasando los Ácidos nucleicos. Empaquetamiento de ADN 46 cromosomas en las células somáticas del
Comparando las células procariotas y eucariotas
 OpenStax-CNX module: m53336 1 Comparando las células procariotas y eucariotas OpenStax College Based on Comparing Prokaryotic and Eukaryotic Cells by OpenStax College This work is produced by OpenStax-CNX
OpenStax-CNX module: m53336 1 Comparando las células procariotas y eucariotas OpenStax College Based on Comparing Prokaryotic and Eukaryotic Cells by OpenStax College This work is produced by OpenStax-CNX
5.2. NITRÓGENO Y AZUFRE.
 5.2. NITRÓGENO Y AZUFRE. 5.2.1. PROTEÍNAS. Si hidratos de carbono y lípidos están formados únicamente por átomos de carbono, oxígeno e hidrógeno, las proteínas tienen en su composición, además, nitrógeno
5.2. NITRÓGENO Y AZUFRE. 5.2.1. PROTEÍNAS. Si hidratos de carbono y lípidos están formados únicamente por átomos de carbono, oxígeno e hidrógeno, las proteínas tienen en su composición, además, nitrógeno
Diferenciaciones de la membrana plasmática. Microvellosidades
 Diferenciaciones de la membrana plasmática Son regiones de la membrana plasmática que presentan modificaciones estructurales especializadas para cumplir diferentes funciones. Un típico ejemplo lo ofrecen
Diferenciaciones de la membrana plasmática Son regiones de la membrana plasmática que presentan modificaciones estructurales especializadas para cumplir diferentes funciones. Un típico ejemplo lo ofrecen
MEMBRANA CELULAR: Modelo de organización, Transporte a través de membrana
 MEMBRANA CELULAR: Modelo de organización, Transporte a través de membrana Componentes de la membrana celular Modelo Mosaico Fluido Glúcidos Proteínas Colesterol Fosfolípidos Componentes de la membrana
MEMBRANA CELULAR: Modelo de organización, Transporte a través de membrana Componentes de la membrana celular Modelo Mosaico Fluido Glúcidos Proteínas Colesterol Fosfolípidos Componentes de la membrana
Unidad 3: Organización molecular de la célula Proteínas
 Proteínas Las proteínas son las biomoléculas más abundantes de las células, constituyendo el 50% de su peso seco aproximadamente. Estructuralmente, son polímeros de aminoácidos. Existe una enorme variedad
Proteínas Las proteínas son las biomoléculas más abundantes de las células, constituyendo el 50% de su peso seco aproximadamente. Estructuralmente, son polímeros de aminoácidos. Existe una enorme variedad
Avances en el desarrollo de vacunas contra la malaria
 61 Rev Biomed 2008; 19:61-79 Revisión Avances en el desarrollo de vacunas contra la malaria Mario Henry Rodríguez-López Instituto Nacional de Salud Pública, Cuernavaca, Morelos, México RESUMEN Las vacunas
61 Rev Biomed 2008; 19:61-79 Revisión Avances en el desarrollo de vacunas contra la malaria Mario Henry Rodríguez-López Instituto Nacional de Salud Pública, Cuernavaca, Morelos, México RESUMEN Las vacunas
Aspectos generales de farmacodinamia Parte I. Dr. Mario Acosta Mejía
 Aspectos generales de farmacodinamia Parte I Dr. Mario Acosta Mejía Objetivos Analizar el concepto general y actualizado de farmacodinamia Entender los mecanismos de acción por los cuales actúan los fármacos
Aspectos generales de farmacodinamia Parte I Dr. Mario Acosta Mejía Objetivos Analizar el concepto general y actualizado de farmacodinamia Entender los mecanismos de acción por los cuales actúan los fármacos
BIOACTIVIDADES BACTERIAS, VIRUS
 FUNDACION EDUCACIONAL COLEGIO CARMELA ROMERO DE ESPINOSA MADRES DOMINICAS- CONCEPCION Prof. María Eugenia Muñoz Jara Biología 4 Medios BIOACTIVIDADES BACTERIAS, VIRUS Morfología y estructura Las bacterias
FUNDACION EDUCACIONAL COLEGIO CARMELA ROMERO DE ESPINOSA MADRES DOMINICAS- CONCEPCION Prof. María Eugenia Muñoz Jara Biología 4 Medios BIOACTIVIDADES BACTERIAS, VIRUS Morfología y estructura Las bacterias
Capítulo 1 Biología: ciencia de la vida Qué es la biología? Qué es la vida?... 12
 ÍNDICE UNIDAD 1 Qué es la biología? Capítulo 1 Biología: ciencia de la vida... 4 1.1 Qué es la biología?... 6 1.2 Qué es la vida?... 12 Capítulo 2 Métodos científicos en biología... 24 2.1 Métodos para
ÍNDICE UNIDAD 1 Qué es la biología? Capítulo 1 Biología: ciencia de la vida... 4 1.1 Qué es la biología?... 6 1.2 Qué es la vida?... 12 Capítulo 2 Métodos científicos en biología... 24 2.1 Métodos para
PALUDISMO O MALARIA Departamento de Control de Vectores y Patología Regional Area Epidemiología Ministerio de Bienestar Social Provincia de Jujuy
 PALUDISMO O MALARIA Departamento de Control de Vectores y Patología Regional Area Epidemiología Ministerio de Bienestar Social Provincia de Jujuy Enfermedad infecciosa de origen parasitario, producida
PALUDISMO O MALARIA Departamento de Control de Vectores y Patología Regional Area Epidemiología Ministerio de Bienestar Social Provincia de Jujuy Enfermedad infecciosa de origen parasitario, producida
Profesor(a): C.D. María Isabel Pérez Aguilar
 Área Académica: Biología Básica Tema: Bioelementos Profesor(a): C.D. María Isabel Pérez Aguilar Periodo: Enero- Julio 2012 Abstract Bioelements Bioelements are the essential components of life. This topic
Área Académica: Biología Básica Tema: Bioelementos Profesor(a): C.D. María Isabel Pérez Aguilar Periodo: Enero- Julio 2012 Abstract Bioelements Bioelements are the essential components of life. This topic
Introducción. Expresión génica. Regulación de la expresión génica en procariotas
 Introducción La secuencia genética siempre es la misma en todas las células y siempre está presente en organismos unicelulares, pero no se expresa igual en cada célula y en cada momento. Expresión génica
Introducción La secuencia genética siempre es la misma en todas las células y siempre está presente en organismos unicelulares, pero no se expresa igual en cada célula y en cada momento. Expresión génica
CUESTIONES SELECTIVIDAD: MICROBIOLOGÍA. 1) Qué es un virus?(0,5).describa el ciclo lítico de un bacteriófago. (JUNIO 2007)
 CUESTIONES SELECTIVIDAD: MICROBIOLOGÍA 1) Qué es un virus?(0,5).describa el ciclo lítico de un bacteriófago. (JUNIO 2007) ) Realice una clasificación de los cinco tipos principales de microorganismos indicando
CUESTIONES SELECTIVIDAD: MICROBIOLOGÍA 1) Qué es un virus?(0,5).describa el ciclo lítico de un bacteriófago. (JUNIO 2007) ) Realice una clasificación de los cinco tipos principales de microorganismos indicando
INSTITUCIÓN EDUCATIVA CASD ARMENIA Q GESTIÓN ACADÉMICA DISEÑO PEDAGÓGICO- PLAN DE ÁREA
 INSTITUCIÓN EDUCATIVA CASD ARMENIA Q GESTIÓN ACADÉMICA DISEÑO PEDAGÓGICO- PLAN DE ÁREA GA-DP-R20 ÁREA: CIENCIAS NATURALES ASIGNATURA: BIOLOGÍA GRADO: 11 CICLO: FECHA: ENERO 21 A MAYO 17 DE DOCENTES RESPONSABLES:
INSTITUCIÓN EDUCATIVA CASD ARMENIA Q GESTIÓN ACADÉMICA DISEÑO PEDAGÓGICO- PLAN DE ÁREA GA-DP-R20 ÁREA: CIENCIAS NATURALES ASIGNATURA: BIOLOGÍA GRADO: 11 CICLO: FECHA: ENERO 21 A MAYO 17 DE DOCENTES RESPONSABLES:
Ontogenia. R3AIC Dra. Patricia María O Farrill Romanillos México, D.F a 10 de Abril del 2012
 Ontogenia R3AIC Dra. Patricia María O Farrill Romanillos México, D.F a 10 de Abril del 2012 Definición Ontogenia (del griego οντος, ser, estar y génesiv: origen, generación). 1. f. Biol. Desarrollo del
Ontogenia R3AIC Dra. Patricia María O Farrill Romanillos México, D.F a 10 de Abril del 2012 Definición Ontogenia (del griego οντος, ser, estar y génesiv: origen, generación). 1. f. Biol. Desarrollo del
BLOQUE I. Reproducción Celular
 BLOQUE I. Reproducción Celular Tipos de reproducción La importancia de la reproducción Principales actores de la reproducción Anomalías de la reproducción ESQUEMA DE CONTENIDOS REPRODUCCIÓN Es fundamental
BLOQUE I. Reproducción Celular Tipos de reproducción La importancia de la reproducción Principales actores de la reproducción Anomalías de la reproducción ESQUEMA DE CONTENIDOS REPRODUCCIÓN Es fundamental
CARACTERIZACIÓN DE LA PROTEÍNA DEL CUELLO DE LAS ROPTRIAS 5 EN Plasmodium falciparum (PfRON5).
 CARACTERIZACIÓN DE LA PROTEÍNA DEL CUELLO DE LAS ROPTRIAS 5 EN Plasmodium falciparum (PfRON5). LILIANA CATHERINE PATIÑO MOLANO UNIVERSIDAD DEL ROSARIO FACULTAD DE MEDICINA UNIDAD DE GENÉTICA Bogotá, Junio
CARACTERIZACIÓN DE LA PROTEÍNA DEL CUELLO DE LAS ROPTRIAS 5 EN Plasmodium falciparum (PfRON5). LILIANA CATHERINE PATIÑO MOLANO UNIVERSIDAD DEL ROSARIO FACULTAD DE MEDICINA UNIDAD DE GENÉTICA Bogotá, Junio
GENÉTICA MOLECULAR. El ADN, LA MOLÉCULA DE LA HERENCIA
 GENÉTICA MOLECULAR El ADN, LA MOLÉCULA DE LA HERENCIA INTRODUCCIÓN: COMENCEMOS PREGUNTÁNDONOS Qué significan estas dos palabras? GENÉTICA MOLECULAR Estudio de la genética (genes) desde el punto de vista
GENÉTICA MOLECULAR El ADN, LA MOLÉCULA DE LA HERENCIA INTRODUCCIÓN: COMENCEMOS PREGUNTÁNDONOS Qué significan estas dos palabras? GENÉTICA MOLECULAR Estudio de la genética (genes) desde el punto de vista
EL A.D.N. Existen 2 tipos de Acidos Nucleicos : ADN (Acido Desoxirribonucleico) y ARN (Acido Ribonucleico) Diferencias entre ADN y ARN
 EL A.D.N Existen 2 tipos de Acidos Nucleicos : ADN (Acido Desoxirribonucleico) y ARN (Acido Ribonucleico) Diferencias entre ADN y ARN Hay tres tipos netamente diferenciados de ARN, tanto en su estructura
EL A.D.N Existen 2 tipos de Acidos Nucleicos : ADN (Acido Desoxirribonucleico) y ARN (Acido Ribonucleico) Diferencias entre ADN y ARN Hay tres tipos netamente diferenciados de ARN, tanto en su estructura
CURSO DE PREPARACIÓN DE LAS PRUEBAS LIBRES PARA LA OBTENCIÓN DEL TÍTULO DE BACHILLER PARA PERSONAS MAYORES DE VEINTE AÑOS BIOLOGÍA
 CURSO DE PREPARACIÓN DE LAS PRUEBAS LIBRES PARA LA OBTENCIÓN DEL TÍTULO DE BACHILLER PARA PERSONAS MAYORES DE VEINTE AÑOS BIOLOGÍA DISTRIBUCIÓN DE LOS CONTENIDOS POR BLOQUES TEMÁTICOS Y CRITERIOS DE EVALUACIÓN
CURSO DE PREPARACIÓN DE LAS PRUEBAS LIBRES PARA LA OBTENCIÓN DEL TÍTULO DE BACHILLER PARA PERSONAS MAYORES DE VEINTE AÑOS BIOLOGÍA DISTRIBUCIÓN DE LOS CONTENIDOS POR BLOQUES TEMÁTICOS Y CRITERIOS DE EVALUACIÓN
