SECUENCIACIÓN SANGER Purificación mediante columnas Protocolos manuales.
|
|
|
- Marcos José Miguel Fidalgo Gutiérrez
- hace 6 años
- Vistas:
Transcripción
1 SECUENCIACIÓN SANGER En la Unidad de Genómica se realiza la secuenciación automática del DNA mediante electroforesis capilar y mediante el uso de la química BigDye Terminator v3.1 de Applied Biosystems. Todas las muestras son purificadas con el fin de eliminar el ruido de fondo y los terminadores sobrantes colaborando en la obtención de unos resultados satisfactorios. Los cebadores utilizados para la reacción de secuenciación pueden ser universales o específicos. 1. PREPARACIÓN DE LAS MUESTRAS La calidad de los datos obtenidos depende en gran medida de la pureza y correcta cuantificación de las muestras de ADN proporcionadas. Se recomienda utilizar protocolos de extracción, purificación y cuantificación fiables que permitan verificar la integridad y concentración de la muestra. Es muy importante que el usuario prepare siempre LAS MUESTRAS SIEMPRE DILUIDAS EN AGUA. Evitar el tampón TE y los buffers de elución del ADN de los kits comerciales porque la presencia de EDTA en la muestra inhibe la reacción de secuenciación ADN clonado Pueden utilizarse cualquier protocolo de obtención de ADN, pero todos tienen que producir un ADN libre de ARN, fenol, sales, etanol, EDTA, proteínas, etc... Cada usuario puede elegir el protocolo que más se adapte a sus necesidades pero funcionan mejor los que conllevan métodos de purificación con minicolumnas Purificación mediante columnas. Por lo general todos los kits comercializados que comprenden una etapa de purificación en columna dan plásmidos de buena calidad para la secuenciación (Quiagen, Roche, Eppendorf, Biorad, etc...), a excepción de algunas sílicas/resinas comerciales que dejan impurezas. Se recomienda centrifugar la columna dos veces después del paso de lavado y a continuación eluir el DNA con agua. Para asegurar la eliminación del etanol presente en la solución de lavado de la muestra Protocolos manuales. - Los métodos tipo "boiling" pueden dar buenos resultados si se realiza tratamiento posterior con fenol/cloroformo, cloroformo, precipitación con isopropanol y lavados con etanol
2 - Los métodos de "lisis alcalina" no son recomendable a no ser que vaya seguido de una purificación posterior con un kit específico o mediante precipitación. - Si en la purificación del DNA se ha utilizado algún solvente orgánico como fenol o cloroformo, el DNA debe purificarse al menos dos veces mediante precipitación con etanol, para eliminar cualquier traza del solvente orgánico. - Si se utiliza etanol o isopropanol para precipitar el DNA, realizar la precipitación utilizando acetato de amonio en lugar de acetato de sodio. Lavar el precipitado después de la centrifugación al menos una vez con etanol al 70%. Secar el precipitado el máximo posible y resuspender en agua. La presencia de trazas de etanol en el molde es una de las principales causas de fallo de la secuenciación. En cualquier caso, el ADN siempre debe ser resuspendido en agua-miliq estéril. Evitar cualquier tampón que contenga EDTA pues éste interfiere en la reacción de secuenciación Productos de PCR La mezcla de PCR debe ser purificada (precipitación, kits comerciales...) con objeto de eliminar subproductos, cebadores y dntps contaminantes que impidan la secuenciación. La secuenciación de fragmentos de pequeño tamaño es algo problemática por lo que se recomienda que los productos sean de al menos 150 pb. Los fragmentos más pequeños se pueden comportar como primers y son difíciles de purificar con buen rendimiento Purificación mediante Columna. Con cualquiera de los kits existentes en el mercado (Quiaquick, Quiagen, Millipore, Amersham, Roche, etc...). Este método sólo puede utilizarse si la banda de interés es producto único de amplificación y no una PCR múltiple, en tal caso la secuenciación será fallida ( lectura mezclada) Precipitación. Existen varios protocolos: a) con acetato amonico sólo si la concentración total de cebadores en la PCR no es superior a 5 pmol; b) También se puede usar Etanol y acetato sódico, etc Aplicables todos solo si el producto de amplificación es único, aunque se puede realizar una precipitación selectiva, pero no es recomendable Dilución directa del producto de PCR. Este método implica diluir una alícuota del producto de PCR entre 5 y 10 veces en agua y emplearlo directamente en la secuenciación automática. Para ello, es necesario emplear bajas concentraciones de dntps y primers en la reacción de PCR, de tal manera que, las concentraciones finales de los primers sean de 0,2 μm o menos y las concentraciones de dntps sean de 100 μm o menos. La principal desventaja de este método es que se requiere una optimización individual de las condiciones para cada producto de PCR
3 Purificación del producto de PCR de un gel de agarosa. Cuando existan artefactos o más de un producto de amplificación, es necesario el aislamiento de la banda de interés mediante electroforesis en gel de agarosa Además, permite la eliminación de primers y dntps. Se trata de correr el producto de PCR en un gel de agarosa de bajo punto de fusión, cortar la banda correspondiente (procurar no contaminar la banda con oligos y cortar la menor cantidad de agarosa posible) y fundir la agarosa. Finalmente, extraer y purificar el DNA con sucesivos pasos de fenol:cloroformo:alcohol isoamilico (25:24:1) y precipitación con etanol y sales. Alternativamente, se puede emplear para la purificación kits comerciales Purificación enzimática del producto de PCR. Consiste en el tratamiento del producto de PCR con Exonucleasa I y SAP. Recomendado para productos pequeños de PCR. No puede aplicarse en el caso de productos de PCR contaminados con subproductos secundarios. El tratamiento con exonucleasa I degrada los primers de PCR residuales mientras que la SAP (Shrimp Alkaline Phosphatase) defosforila los dntps restantes. Tras la inactivación por calor de ambas enzimas puede utilizarse el producto de PCR para secuenciación automática Lambda, Cósmidos, BACs Recomendamos para la extracción de ADN maxipreps obtenidas por gradiente en ClCs. Para cualquier consulta en relación a este tema ponerse en contacto con la Unidad ya que requieren de cantidades de ADN y primer diferentes así como de un tratamiento previo antes de someterlas a secuenciación. 2. CUANTIFICACIÓN DE LAS MUESTRAS Otro factor determinante en el éxito de las reacciones de secuenciación es la correcta cuantificación del ADN. Si la cantidad de ADN presente en la muestra es inferior a la necesaria, casi con toda probabilidad la reacción de secuenciación fallará produciendo datos con baja señal, ruido de fondo, errores y ambigüedades. Demasiado DNA molde también puede producir efectos similares. Se puede realizar mediante espectrofotometría a 260 nm. Puesto que la contaminación del DNA molde con RNA o proteínas pueden afectar negativamente a los resultados de la secuenciación, hay que medir también la absorbancia de la muestra a 280 y 230 nm y comprobar las ratio A260/A280 y A260/A230. El DNA molde debe tener una ratio entre A260 y A280 nm de Aun así, debido al amplio margen de error de los espectrofotómetros, nosotros recomendamos realizar la cuantificación mediante electroforesis en gel de agarosa con un control de concentración conocida. De esta forma se comprueba tanto la cantidad como la calidad del ADN a secuenciar
4 3. CEBADORES DISPONIBLES El Servicio suministra los cebadores universales siguientes: T7 T3 Universal M13 forward -20 Reverso SP6 T 25 (A,G o C) 5 -TAA TAC GAC TCA CTA TAG GG-3 5 -ATT AAC CCT CAC TAA AGG GA-3 5 -TGT AAA ACG ACG GCC AGT-3 5 -CAG GAA ACA GCT ATG ACC-3 5 -GAT TTA GGT GAC ACT ATA G-3 5 -TTT TTT TTT TTT TTT TTT TTT TTT T(A,G o C)-3 Además de los cebadores descritos se puede utilizar cualquier cebador sintetizado por el usuario. Para un correcto diseño de cebadores que vayan a ser utilizados en secuenciación, recomendamos lo siguiente: Los cebadores deberán tener, al menos, 18 bases para asegurarnos que hibriden con el ADN a secuenciar y evitar hibridaciones secundarias en el vector o en el inserto. Deben tener una Tm entre 50 ºC-65 ºC. Se puede utilizar la siguiente fórmula para calcular la Tm. Tm= x(%GC)-650/longitud del primer Debe evitarse la utilización de cebadores con secuencias que formen estructuras secundarias, especialmente en el extremo 3, o puedan hibridar formando dímeros intercatenarios. Para ello, evitar en lo posible la utilización de cebadores que tengan en su secuencia repeticiones de más de 3 ó 4 Gs ó Cs seguidas y % GC entre %. No deben emplearse cebadores con errores, ni cebadores que hibriden en más de un sitio en la secuencia o que puedan formar dímeros. Diseñar el primer al menos 50 bases upstream de la secuencia de interés. La secuencia inmediatamente posterior al primer o no se obtiene o puede ser bastante imprecisa. 3. PROGRAMAS El formato ABI es compatible con los visores habituales disponibles en las siguientes direcciones: - EditView en Mac: - Chromas en PC En la siguiente dirección web: /3100/conversion.cfm el usuario puede descargarse el programa de conversión de los ficheros AVI de PC a MAC
5 4. REPETICIONES 4.1. Secuencias fallidas Los procedimientos empleados en la Unidad se han diseñado para minimizar la variabilidad y la frecuencia de secuencias fallidas. No obstante se pueden producir fallos debido a tres tipos de causas: a. Características de las muestras, bien del molde bien de los cebadores. b. Información insuficiente por parte del usuario. c. Error operativo del funcionamiento interno del servicio. Las secuencias fallidas se facturan cuando se estima que las causas de error son del tipo a o b. Las producidas por errores del tipo c se repiten. Si la repetición vuelve a fallar se asumirá que no se trataba de un error operativo. Para cualquier duda o consulta ponerse en contacto con la Unidad. Cuando las muestras enviadas sean repeticiones indicarlo en el Formulario de Solicitud que se adjunta con las muestras para hacer los cambios oportunos con el fin de conseguir un resultado satisfactorio Causas de error Las causas de error más habituales que se asocian con el DNA de partida y que afectan, principalmente, a la calidad del mismo, son las siguientes: a. Para muestras en vectores de clonación, son dos los factores más importantes: Las condiciones de crecimiento del cultivo. Los cultivos bacterianos para la purificación de plásmidos deben obtenerse de colonias frescas crecidas en medios selectivos o de pequeños precultivos obtenidos de stocks de glicerol o de agar. La cepa bacteriana donde se propaga el molde puede afectar a su calidad. Se obtienen resultados fiables con DH1, DH5α, DH10B, XL1-blue y C600. Las cepas MV1190, HB101 y JM101 no son recomendables puesto que contienen una gran cantidad de carbohidratos y poseen el locus enda con lo que producen relativamente grandes cantidades de nucleasa. b. Características del ADN. Formación de estructuras en la clonación que impiden una correcta secuenciación, clones o pcrs múltiples, polit, presencia de microsatélites, etc En general, características del fragmento u organismo a secuenciar de las cuales no se tiene conocimiento hasta un primer paso de secuenciación. Una vez conocidas tales características, se requieren uno o varios pasos de optimización para conseguir resultados óptimos. c. Cantidad insuficiente de DNA molde. Se recomienda cuantificar el DNA mediante electroforesis, empleando diluciones y comparando con marcadores de masa conocida y comprobar la concentración. d. Calidad del molde. Aunque para su preparación se hayan utilizado kits comerciales pueden permanecer impurezas que interfieran con la secuenciación: - 5 -
6 Para productos de PCR las impurezas más habituales son los fragmentos que copurifican con el molde como por ejemplo primer-dimers, que contienen sitios de unión para los cebadores de secuenciación. Contaminación por nucleasas: por ejemplo la DNasa causa un cambio de la forma del plásmido superenrollada a lineal lo que reduce gradualmente la intensidad de la señal y la longitud de la secuencia obtenida. Por lo tanto hay que evitar las cepas hospedadoras que produzcan nucleasas. Contaminación por RNA: esto ocurre más frecuentemente cuando los cultivos han crecido demasiado tiempo y el tampón de lisis es insuficiente para lisar todas las células. Las muestras contaminadas con RNA muestran un ruido de fondo alto. El tratamiento del DNA molde con RNasa (libre de DNasa) degradará el RNA con lo que se pueden mejorar los resultados de las secuenciación. Por otro lado, la presencia de RNA falsea la cuantificación del ADN. Contaminación por sales: la presencia de sales inhibe la reacción de secuenciación (disminuye la procesividad de la polimerasa). Sin embargo el tipo de sal influye en la severidad del efecto: por ejemplo el cloruro de sodio o de potasio tiene un efecto mayor que la misma concentración de acetato de sodio. Un concentración de acetato de sodio mayor de 20mM inhibe severamente la reacción de secuenciación. Las causas más comunes de contaminación por sales son: la coprecipitación de éstas con el etanol en la precipitación del DNA, sobre todo si esta se realiza a baja temperatura; una eliminación insuficiente del sobrenadante; o un lavado con etanol 70% insuficiente. Por lo tanto la precipitación del DNA molde y los lavados deben realizarse muy cuidadosamente y a temperatura ambiente. Contaminación por etanol: se produce por la resuspensión del DNA molde tras la precipitación sin que se haya secado suficientemente. La presencia de pequeñas cantidades de etanol (5%) puede causar la terminación prematura de las cadenas y por lo tanto la obtención de secuencias cortas. Una contaminación con etanol superior al 10% tiene como consecuencia normalmente el fallo total de la reacción de secuenciación. Contaminación con fenol: algunos métodos de preparación de DNA plasmídico utilizan un paso de extracción con fenol. La contaminación con este reactivo tiene como resultado la degradación severa de la secuencia resultante, especialmente en lecturas largas. Más de un 1,4% de fenol en el DNA molde produce secuencias totalmente inutilizables. e. Problemas con los cebadores específicos del molde. La muestra puede contener distintos sitios de unión, o el cebador no unirse al molde o cantidad insuficiente del cebador, error en el cálculo de la Tm, DNA subclonado que arrastre polilinker y existan varios sitios diana para cebadores universales, etc 5. PROBLEMAS MÁS FRECUENTES DURANTE LA SECUENCIACIÓN A) No se produce reacción o esta decae poco después de comenzar - 6 -
7 No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria. El primer puede estar degradado El cebador no encuentra el sitio de hibridación El ADN presenta contaminación El cebador está mal diseñado o la temperatura de melting no es la adecuada El sitio de unión del cebador en el vector se ha perdido o dañado. Durante la clonación pueden crearse artefactos con una deleción cerca del sitio de inserción del DNA a clonar o deleciones que eliminan la secuencia del primer del vector. En los productos de PCR puede que los amplificados no sean el producto de la amplificación con los dos cebadores. Si el amplificado se genera a partir de uno de los dos primers sólo se obtendrá secuencia con éste. Este efecto se puede producir cuando se utilizan para secuenciar los mismos primer que se han utilizado en la amplificación del DNA molde y alguno de ellos no funciona correctamente. Aumentar la cantidad de ADN Aumentar la concentración o cantidad de cebador, Cambiar la alícuota. Cambiar el cebador. Asegurarse del diseño Volver a preparar el ADN Rediseñar el cebador Volver a clonar en el vector Cambiar de cebador B) Señal de baja intensidad. Unión débil del cebador. Mala calidad del ADN ADN presenta estructura secundaria en el sitio de hibridación del cebador La concentración del ADN inferior a la necesaria Volver a purificar el ADN Cambiar de cebador Aumentar la concentración - 7 -
8 El cebador utilizado en la secuenciación no es el adecuado: - Estructura secundaria (afecta más en extremo 3 ) - Tm del cebador demasiado baja (muy corto, poco contenido GC, etc ) Cambiar el cebador C) El cromatograma es correcto pero presenta mucho ruido de fondo Existe un sitio secundario de unión del cebador que da lugar a picos extra La cantidad de ADN es insuficiente y la señal es demasiado baja El ADN presenta contaminación 1 La PCR no fue especifica y existen amplicones contaminantes en la muestra Cebador mal purificado 2 o degradado Fragmento de PCR mal purificado Intentar eliminar añadiendo DMSO en la secuenciación o cambiar de cebador Aumentar la cantidad de ADN Purificar el ADN Cambiar de cebador Cambiar de cebador o purificar Eliminar los cebadores de la amplificación 1. En la secuenciación en capilares este problema (contaminación del ADN) es aún mayor al ser más sensible. 2. Puede verse una secuencia sombra (N-1). El cebador está compuesto por cadenas completas (N), pero una proporción de ellas contiene una base menos (N-1), esto da lugar a un cromatograma ilegible al tener picos superpuestos y desfasados. D) El cromatograma presenta dos secuencias superpuestas El cebador tiene varias dianas Primer drimer: Esto puede ocurrir cuando los primers empleados en la amplificación forman dímeros y están presentes en la reacción de Cambiar de cebador Volver a purificar el producto de PCR - 8 -
9 secuenciación debido a una mala purificación del producto. Cuando termina la secuencia de los dímeros la lectura es correcta. Cebador degenerado ADN subclonado existiendo dos sitios diana para el cebador universal utilizado 1 PCR inespecífica: diana del cebador en ambos extremos, es decir, amplicón inespecífico generado por un solo cebador Hay más de un ADN molde en la muestra 3 Cambiar de cebador Secuenciar con otro cebador Cambiar de cebador Repreparar el ADN 1. Comprobar, si se ha arrastrado polilinker durante la subclonación porque esto implicaría que el/los cebadores universales tienen sitio de hibridación duplicado y no se pueden utilizar en la secuenciación de esa muestra. Aseguraros de que partimos de una única colonia o que no es PCR múltiple con amplificaciones secundarias inespecíficas. -Primer Drimer Si el producto de PCR no está purificado o en la purificación no se ha eliminado totalmente los restos de primer que no se utilizaron en la amplificación, se pueden formar dimeros de primer que actúan como molde en la reacción de secuenciación. Esto genera lecturas superpuestas. El rango o longitud de la superposición dependerá del tamaño del molde contaminante. - En el caso de productos de PCR: la mezcla de lecturas comienza desde el principio. Si a partir de las pb de bases la mezcla desaparece es indicativo de primer drimer. - En el caso de un clon múltiple: la mezcla de lecturas comienza a partir de las pb de bases, es decir, la lectura de ambas colonias coincide solo en el vector. Una vez comienza el inserto, si son distintos, se mezclan las lecturas. Ejemplo: La reacción comienza de forma normal pero existe doble lectura a partir de un determinado punto (30-90 pb aprox.) -Inserción/Deleción La reacción comienza de forma normal pero existe doble lectura a partir de un determinado punto (más de 150 pb aprox.) - 9 -
10 Región heterocigótica para ese fragmento Mutaciones frame shift Digestión para eliminarlo E) La secuencia pierde bruscamente la señal y cae Efecto de una estructura secundaria (presencia de secuencias repetidas, palindromicas, etc ) La cantidad entre el ADN y el cebador no es la equilibrada Formación de estructuras en la clonación que impiden una correcta secuenciación Presencia de compuestos contaminantes en la muestra (EDTA, sales, etanol, fenol, etc.) ADN cortado, digerido a ese nivel o finalización de producto de pcr Añadir DMSO, secuenciar la cadena complementaria, digerir el molde, subclonarlo y secuenciar los subclones o utilizar un kit alternativo para las reacciones de secuenciación cómo el kit dgtp Big Dye de ABI. Respetar las concentraciones y cantidades indicadas en las tablas de entrega de muestras Realizar PCR sobre el clon (cebadores universales o propios) y mandar a secuenciar el amplicón resultante y no el clon No resuspender nunca el ADN en solución que contenga EDTA F) Presencia de picos saturados y fuera de escala en el cromatograma Demasiada cantidad de ADN Reducir la cantidad de ADN ( según figura en las tablas de entrega de muestras) G) La secuencia es normal pero aparece un pico de forma repentina
11 Terminadores no incorporados (entre pb) Repetir la reacción de secuenciación H) Aparición de un pico anómalo con los colores correspondientes a todas las bases Burbuja producida por el capilar Volver a analizar la muestra o repetir la reacción de secuenciación I) Aparición de picos muy abiertos con la consecuente pérdida de resolución Exceso de ADN (capilar) Presencia de sales o contaminantes en la muestra Problema del capilar Respetar la cantidad y concentración de ADN Volver a purificar la muestra Repetir la secuencia J) Secuenciación de regiones ricas en GCs, GAs
12 Formación de dúplex que alteran estabilidad del cebador Usar temperaturas de secuenciación más bajas que la estándar Composición nucleotídica (Repeticiones dinucleótidos GT, GC, GA.) Estructura secundaria, efecto de la alta Tm del tratamiento del ADN Secuenciar cadena complementaria Repetir la secuenciación con un kit alternativo (dgtp Big Dyes). Añadir DMSO en la secuenciación Cambiar condiciones de PCR: aumentar la tª de desnaturalización a 98º, el nº de ciclos de PCR o la cantidad de Taq; incluir un paso de predesnaturalización de 5 min Linearizar el vector y secuenciar productos tras subclonarlos Cuando se secuencien organismos con un alto contenido GC indicarlo en las tablas que se adjuntan con la muestras a secuenciar 1. Las repeticiones de AG pueden ser problemáticas pues la Taq produce una señal débil de G después de A cuando se utilizan terminadores marcados. En este caso conviene secuenciar la cadena complementaria. K) Presencia de microsatélites Presencia Microsatélites: SSRs, VNTRs, en el caso de productos de PCR son más problemáticos que en productos clonados, sobre todo si son de elevada longitud. Se puede producir el deslizamiento de la polimerasa y no se puede obtener secuencia de esa región. Repeticiones dinucleótidos GC Secuenciar a partir del plásmido no del producto de PCR y añadir DMSO en la reacción de secuenciación Crear delecciones seriadas en esas zonas y secuenciar Cambios ya recomendados
13 L) Presencia de largos homopolímeros Presencia de polia/polit 1 Secuenciar cadena complementaria Empleando cebador degenerado del tipo T25(A, G ó C) 2 si no se conoce la base siguiente a la región polia/polit o los cebadores individuales específicos T25A, T25G ó T25C. La base 3 del cebador permite anclar este a la secuencia al final del homopolímero, mejorando los resultados obtenidos 1. La polimersa patina dando lugar a una lectura ilegible después de la región homopolimérica, aunque los datos de antes de esa región sean de buena calidad. Este problema se puede producir en la reacción de secuenciación o en la de amplificación, aunque en este último caso no son aparentes cuando el fragmento se visualiza en un gel de agarosa. 2. Se denominan primer anclado: Son primers que se unen al homopolímero, tipo T25-(N) y que terminan en una base degenerada para obligar al oligo a pegarse en la posición 3. Homopolímero de Gs o Cs (causa deslizamiento de la polimerasa generándose una secuencia ilegible) Secuenciar cadena complementaria Usar un cebador más interno que hibride bases antes del homopolímero para forzar a la polimerasa a pasar sobre él Realizar deleciones seriadas y secuenciarlas En los productos de PCRs pueden existir saltos, problemas que no existen en el material clonado Secuenciar el material clonado, además del amplificado. Si es posible, es mejor verificar la especificidad de la secuencia a partir de otros clones
14 M)Primer N-1 Secuencia sombra N-1 El cebador está compuesto por cadenas completas (N), pero una proporción de ellas contiene una base menos (N-1), esto da lugar a un cromatograma ilegible, en algunos casos, al tener picos superpuestos y desfasados. Puede verse una secuencia sombra (N-1) de menor altura, donde cada pico sombra es igual que el pico siguiente de mas altura y/o intensidad de fluorescencia. Prepara una nueva alícuota del primer N) Solo terminadores Aparecen solo los terminadores debido a que la cantidad de ADN es insuficiente Poner mas cantidad de ADN o de mejor calidad
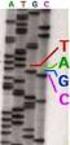
15 O) No inserto Inserto no clonado correctamente Falso positivo Clonar de nuevo Seleccionar otra colonia P)Heterocigosis (No fallo) G C + T C A A Heterozigot o: GT
16 SECUENCIACION MASIVA ( NGS) La Unidad de Genómica tiene disponible la tecnología Ion Torrent que utiliza chips de sensores de tipo semiconductor que miden la liberación de protones durante la reacción de polimerización del ADN. La detección se realiza en todos los sensores en paralelo y en tiempo real, por lo que la duración de las carreras es de solo unas horas. Los equipos disponibles son el Ion PGM y el Ion S5 (Life Technologies-Thermo Fisher) además de los equipos Ion Chef y Ion One Touch 2, para la preparaciión automática de las muestras. El flujo de trabajo es el siguiente: Reunión previa con el usuario para valorar el tipo de ensayo que quiere realizar y elección de la mejor química y soporte para su experimento ( Kits de librerias, chips, eleccion de secuenciador, ). Entrega de la muestra y preparación de la librería (específica de aplicación). También se puede entregar la librería ya preparada siempre que sea compatible con el sistema de secuenciación Ion Torrent. En ambos casos se examinará la calidad y requisitos de las muestras/librerías antes de comenzar (Bioanalyzer, Fluorimetría-Qubit). Preparación del molde de secuenciación: Amplificación clonal y enriquecimiento de las muestras. Secuenciación en el equipo ION PGM o en el ION S5. Análisis primario de los datos. Uso de los programas Torrent Suite y Ion Reporter. Para un estudio completo y detallado de los resultados contactar con la Unidad de Bioinformática. INFORMACIÓN ADICIONAL El Servicio pone a disposición de los usuarios la asesoria científica-técnica necesaria para ser usuarios de este Servicio. También prestamos la asesoría informática necesaria, para realizar los análisis relacionados con el ensamblaje, edición, alineamiento, búsqueda de secuencias homólogas, relaciones filogenéticas, detección de motivos, análisis de uso de codones, comparación de la secuencia problema con las publicadas en las bases de datos, etc.. En caso de dudas sobre cualquier punto, pueden ponerse en contacto con la Unidad a través de nuestro teléfono o o genomica@uco.es
17 - 17 -
SECUENCIACIÓN. 1.1.1. Purificación mediante columnas. 1.1.2. Protocolos manuales.
 SECUENCIACIÓN En la Unidad se realiza la secuenciación automática del DNA mediante electroforesis capilar y mediante el uso de la química BigDye Terminator v3.1 de Applied Biosystems. Todas las muestras
SECUENCIACIÓN En la Unidad se realiza la secuenciación automática del DNA mediante electroforesis capilar y mediante el uso de la química BigDye Terminator v3.1 de Applied Biosystems. Todas las muestras
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES
 PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA
 Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE ADN
 Servicio de Secuenciación de ADN UNIVERSIDAD DE LA LAGUNA Edf. Servicios Generales - IUBO Avda. Francisco Sánchez s/n Tfno.: 922 318 644 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE ADN SERVICIO DE SECUENCIACIÓN
Servicio de Secuenciación de ADN UNIVERSIDAD DE LA LAGUNA Edf. Servicios Generales - IUBO Avda. Francisco Sánchez s/n Tfno.: 922 318 644 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE ADN SERVICIO DE SECUENCIACIÓN
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR
 PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
Índice. 4. Protocolo de envío de muestras 9 5. Problemas más frecuentes durante la secuenciación 10 6. Algunos datos y formulas de utilidad 17
 Índice Introducción 1 1. Modalidades de secuenciación 2 a. Secuenciación de una sola cadena 2 b. Secuenciación analítica 3 c. Secuenciación diagnóstica 3 2. Oligonucleótidos disponibles 5 3. Protocolo
Índice Introducción 1 1. Modalidades de secuenciación 2 a. Secuenciación de una sola cadena 2 b. Secuenciación analítica 3 c. Secuenciación diagnóstica 3 2. Oligonucleótidos disponibles 5 3. Protocolo
DANAGENE BLOOD DNA KIT
 Ref. 0601 100 ml Ref. 0602 200 ml DANAGENE BLOOD DNA KIT 1.INTRODUCCION DANAGENE BLOOD DNA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de sangre total o médula ósea.
Ref. 0601 100 ml Ref. 0602 200 ml DANAGENE BLOOD DNA KIT 1.INTRODUCCION DANAGENE BLOOD DNA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de sangre total o médula ósea.
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS. Es un ácido capaz de formar sales con iones cargados
 Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
PLANIFICACIÓN DE ASIGNATURA
 PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
 EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN.
 PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
SECUENCIACIÓN SANGER DE ADN: GUÍA DE SOLUCIONES TÉCNICAS
 SECUENCIACIÓN SANGER DE ADN: GUÍA DE SOLUCIONES TÉCNICAS Secugen recomienda que para la visualización de las secuencias se usen programas que permitan ver el dato crudo, ya que a partir de ese dato se
SECUENCIACIÓN SANGER DE ADN: GUÍA DE SOLUCIONES TÉCNICAS Secugen recomienda que para la visualización de las secuencias se usen programas que permitan ver el dato crudo, ya que a partir de ese dato se
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO
 SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
- Clonar un gen (molde ADN o ARN)
 APLICACIONES PCR Aplicaciones de la PCR - Clonar un gen (molde ADN o ARN) - Secuencia conocida - Genes homólogos en especies próximas (primers degenerados) - Información de secuencia proteica (primers
APLICACIONES PCR Aplicaciones de la PCR - Clonar un gen (molde ADN o ARN) - Secuencia conocida - Genes homólogos en especies próximas (primers degenerados) - Información de secuencia proteica (primers
INFORMACIÓN AL USUARIO
 INFORMACIÓN AL USUARIO 1. SERVICIOS QUE SE OFRECEN 2. CONDICIONES QUE DEBEN CUMPLIR LAS MUESTRAS 3. INFORMACIÓN ADICIONAL 4. CÓMO HACER USO DEL SERVICIO 5. RESULTADOS 1.- SERVICIOS QUE SE OFRECEN Nuestro
INFORMACIÓN AL USUARIO 1. SERVICIOS QUE SE OFRECEN 2. CONDICIONES QUE DEBEN CUMPLIR LAS MUESTRAS 3. INFORMACIÓN ADICIONAL 4. CÓMO HACER USO DEL SERVICIO 5. RESULTADOS 1.- SERVICIOS QUE SE OFRECEN Nuestro
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015
 CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR).
 Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
Protocolos de secuenciación de ADN
 1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497.
 Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497. Extracción de DNA genómico El DNA de A. brasilense CBG 497 se extrajo siguiendo el método descrito por la casa comercial distribuidora
Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497. Extracción de DNA genómico El DNA de A. brasilense CBG 497 se extrajo siguiendo el método descrito por la casa comercial distribuidora
 Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
La primera parte del proyecto consistió en la obtención de un control positivo mediante
 7. DISCUSIÓN La primera parte del proyecto consistió en la obtención de un control positivo mediante técnicas de clonación molecular. E. coli, a diferencia de otras bacterias que presentan competencia
7. DISCUSIÓN La primera parte del proyecto consistió en la obtención de un control positivo mediante técnicas de clonación molecular. E. coli, a diferencia de otras bacterias que presentan competencia
PCR. Técnica inventada por Kary Mullis :1987. Premio Nobel 1993. Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN
 PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Definición de PCR Es la amplificación enzimática de un
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Definición de PCR Es la amplificación enzimática de un
5. MATERIALES Y MÉTODOS
 5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA
 UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS
 ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS Luis Sáenz Mateos R4 Análisis Clínicos- Hospital General Universitario de Ciudad Real Rotación Externa Hospital Morales Meseguer-Hospital
ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS Luis Sáenz Mateos R4 Análisis Clínicos- Hospital General Universitario de Ciudad Real Rotación Externa Hospital Morales Meseguer-Hospital
Técnicas de Estudio de las células
 Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
Purificación de proteínas
 Purificación de proteínas Purificación Proceso que cuenta con varias etapas cuyo objetivo es lograr la concentración diferencial de la proteína o molécula de interés Condiciones generales a evaluar Calidad
Purificación de proteínas Purificación Proceso que cuenta con varias etapas cuyo objetivo es lograr la concentración diferencial de la proteína o molécula de interés Condiciones generales a evaluar Calidad
Secuenciación del ARN
 Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
PRÁCTICA III. AISLAMIENTO DE MITOCONDRIAS: ACTIVIDAD MALATO DESHIDROGENASA
 PRÁCTICA III. AISLAMIENTO DE MITOCONDRIAS: ACTIVIDAD MALATO DESHIDROGENASA 1. Fundamento Teórico La mayoría de los procesos bioquímicos que producen energía química se realizan en orgánulos intracelulares
PRÁCTICA III. AISLAMIENTO DE MITOCONDRIAS: ACTIVIDAD MALATO DESHIDROGENASA 1. Fundamento Teórico La mayoría de los procesos bioquímicos que producen energía química se realizan en orgánulos intracelulares
Caracteristicas del ADN. Se rompen con las siguientes condiciones -Temperaturas >90 - ph >10.5
 PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
CUESTIONES. 1. Por qué la PCR convencional no es cuantitativa?
 2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR)
 METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
Técnicas de Biología Molecular
 Técnicas de Biología Molecular DNA I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II. Separación electroforética
Técnicas de Biología Molecular DNA I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II. Separación electroforética
Clonación molecular. Clonar significa hacer copias idénticas
 INGENIERÍA GENÉTICA Clonación molecular Clonar significa hacer copias idénticas Este término originalmente fue aplicado al procedimiento de aislar una célula y permitirle reproducirse creando una población
INGENIERÍA GENÉTICA Clonación molecular Clonar significa hacer copias idénticas Este término originalmente fue aplicado al procedimiento de aislar una célula y permitirle reproducirse creando una población
Manual de Uso (Ref /2)
 Fabricado por: BIOTOOLS B&M Labs, S.A. Valle de Tobalina - 52 - Nave 39 28021 Madrid España Tel. (34) 91 710 00 74 Fax (34) 93 843 78 84 e-mail: info@biotools.eu www.biotools.eu Diseñado para la purificación
Fabricado por: BIOTOOLS B&M Labs, S.A. Valle de Tobalina - 52 - Nave 39 28021 Madrid España Tel. (34) 91 710 00 74 Fax (34) 93 843 78 84 e-mail: info@biotools.eu www.biotools.eu Diseñado para la purificación
1. Contaminación de agua y alimentos por microorganismos patógenos 1
 INTRODUCCIÓN GENERAL 1. Contaminación de agua y alimentos por microorganismos patógenos 1 2. Papel del agua en la transmisión de enfermedades infecciosas 2 2.1. Las aguas residuales 2 2.2. El agua potable
INTRODUCCIÓN GENERAL 1. Contaminación de agua y alimentos por microorganismos patógenos 1 2. Papel del agua en la transmisión de enfermedades infecciosas 2 2.1. Las aguas residuales 2 2.2. El agua potable
ABREVIATURAS... XI I. INTRODUCCIÓN EL TOMATE... 3
 ÍNDICE ABREVIATURAS... XI I. INTRODUCCIÓN... 1 1 EL TOMATE... 3 1.1 Taxonomía... 3 1.2 Características generales... 3 1.3 La flor... 4 1.4 Características del fruto... 5 2 CUAJADO Y DESARROLLO DEL FRUTO...
ÍNDICE ABREVIATURAS... XI I. INTRODUCCIÓN... 1 1 EL TOMATE... 3 1.1 Taxonomía... 3 1.2 Características generales... 3 1.3 La flor... 4 1.4 Características del fruto... 5 2 CUAJADO Y DESARROLLO DEL FRUTO...
SERVICIO DE SECUENCIACION DE ACIDOS NUCLEICOS
 CORPORACIÓN CORPOGEN Carrera 5 No 66A-34 Bogotá, Colombia Tels: 8050118/19 Ext. 104 Fax: 3484607 E-mail: servicios@corpogen.org SERVICIO DE SECUENCIACION DE ACIDOS NUCLEICOS El servicio de secuenciación
CORPORACIÓN CORPOGEN Carrera 5 No 66A-34 Bogotá, Colombia Tels: 8050118/19 Ext. 104 Fax: 3484607 E-mail: servicios@corpogen.org SERVICIO DE SECUENCIACION DE ACIDOS NUCLEICOS El servicio de secuenciación
PCR gen 18S ARNr humano
 PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
3/22/2010. Iván Ferrer Rodríguez, PhD Catedrático. En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR.
 Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA"
 P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA
 BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA PROFESORADO CURSO 1994-1995: Dr. J. Ariño (Unidad de Bioquímica) Dra. F. Bosch (Unidad de Bioquímica) Dr. A. Sánchez (Unidad de Genética y Mejora)
BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA PROFESORADO CURSO 1994-1995: Dr. J. Ariño (Unidad de Bioquímica) Dra. F. Bosch (Unidad de Bioquímica) Dr. A. Sánchez (Unidad de Genética y Mejora)
TÉCNICAS GENÓMICAS. 2) Cuantificación del número de virus presentes en muestras de sangre o suero: carga vírica
 TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
TEMA 14. Métodos inmunológicos para la identificación microbiana
 TEMA 14 Métodos inmunológicos para la identificación microbiana Tema 14. Métodos inmunológicos para la identificación microbiana 1. Introducción 2. Detección de antígenos 2.1. Obtención de anticuerpos
TEMA 14 Métodos inmunológicos para la identificación microbiana Tema 14. Métodos inmunológicos para la identificación microbiana 1. Introducción 2. Detección de antígenos 2.1. Obtención de anticuerpos
DIRECTRICES QUALANOD HOJA DE ACTUALIZACIÓN Nº 16 Edición Página 1 de 5 INSPECCIONES RUTINARIAS A LICENCIATARIOS
 Página 1 de 5 INSPECCIONES RUTINARIAS A LICENCIATARIOS Propuesta: Comité Técnico Decisión de QUALANOD: Reunión noviembre 2012 Fecha de aplicación: 1 de julio de 2013 Parte de las Directrices a la que afecta:
Página 1 de 5 INSPECCIONES RUTINARIAS A LICENCIATARIOS Propuesta: Comité Técnico Decisión de QUALANOD: Reunión noviembre 2012 Fecha de aplicación: 1 de julio de 2013 Parte de las Directrices a la que afecta:
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006
 IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
DANAGENE RNA PURIFICATION KIT
 DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
ANEXOS. Anexo 1 Transformación mediante electroporación
 Anexo 1 Transformación mediante electroporación ANEXOS En este método de transformación de E. coli se requieren células electrocompetentes. La preparación de estas células consta de los siguientes pasos:
Anexo 1 Transformación mediante electroporación ANEXOS En este método de transformación de E. coli se requieren células electrocompetentes. La preparación de estas células consta de los siguientes pasos:
Para aumentar la eficiencia de un producto, se pueden trabajar diferentes variables dentro de ciertos límites:
 EL LAVADO DE LA ROPA LA EFICIENCIA DE UN PRODUCTO Existen diferentes tipos y precios de productos para el lavado de la ropa, con nombres genéricos tales como Jabones para lavar o Sistemas de limpieza.
EL LAVADO DE LA ROPA LA EFICIENCIA DE UN PRODUCTO Existen diferentes tipos y precios de productos para el lavado de la ropa, con nombres genéricos tales como Jabones para lavar o Sistemas de limpieza.
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
AGRADECIMIENTOS... VII RESUMEN... XIII RESUM... XV SUMMARY... XVII ABREVIATURAS... XIX ÍNDICE DE CONTENIDO... XXIII ÍNDICE DE FIGURAS...
 ÍNDICE AGRADECIMIENTOS... VII RESUMEN... XIII RESUM... XV SUMMARY... XVII ABREVIATURAS... XIX ÍNDICE DE CONTENIDO... XXIII ÍNDICE DE FIGURAS... XXXI ÍNDICE DE TABLAS... XXXVII INTRODUCCIÓN... 1 1. La mitocondria...
ÍNDICE AGRADECIMIENTOS... VII RESUMEN... XIII RESUM... XV SUMMARY... XVII ABREVIATURAS... XIX ÍNDICE DE CONTENIDO... XXIII ÍNDICE DE FIGURAS... XXXI ÍNDICE DE TABLAS... XXXVII INTRODUCCIÓN... 1 1. La mitocondria...
Proyecto de Fin de Cursos
 Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
TÍTULO: Determinación colorimétrica de fenoles en agua por el método de la 4- aminoantipirina
 Página 1 de 6 1.- INTRODUCCIÓN Desde el punto de vista analítico el término fenol engloba este producto y sus homólogos inmediatamente superiores. El fenol se emplea como patrón y el resultado obtenido
Página 1 de 6 1.- INTRODUCCIÓN Desde el punto de vista analítico el término fenol engloba este producto y sus homólogos inmediatamente superiores. El fenol se emplea como patrón y el resultado obtenido
LA NUEVA BIOTECNOLOGÍA
 LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
PCR en Tiempo Real. Introducción
 Introducción Página 1 de 6 Introducción general La reacción en cadena de la polimerasa, cuyas iniciales en inglés son PCR ("polymerase chain reaction"), es una técnica que fue desarrollada por Kary Mullis
Introducción Página 1 de 6 Introducción general La reacción en cadena de la polimerasa, cuyas iniciales en inglés son PCR ("polymerase chain reaction"), es una técnica que fue desarrollada por Kary Mullis
DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas
 DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas ELEMENTOS BÁSICOS DE LA INVESTIGACIÓN EN GENES Análisis
DNA RECONBINANTE Depende de enzimas capaces de: Cortar Unir Replicar Transcribir inversamente el RNA Otra herramienta es el uso de Sondas específicas ELEMENTOS BÁSICOS DE LA INVESTIGACIÓN EN GENES Análisis
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN
 INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
PCR Múltiplex. Listeria monocytogenes
 PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
EXTRACCIÓN Y CUANTIFICACION DEL ARN DE LA LEVADURA
 EXTRACCIÓN Y CUANTIFICACION DEL ARN DE LA LEVADURA Objetivos: Al finalizar el trabajo práctico los cursantes estarán en capacidad de: - Extraer ARN a partir de células de levadura. - Realizar la hidrólisis
EXTRACCIÓN Y CUANTIFICACION DEL ARN DE LA LEVADURA Objetivos: Al finalizar el trabajo práctico los cursantes estarán en capacidad de: - Extraer ARN a partir de células de levadura. - Realizar la hidrólisis
3. MATERIALES Y MÉTODOS
 3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
Biología Molecular y Filogenia en Micología
 Biología Molecular y Filogenia en Micología Biól. Nicolás Pastor Micología 2014 Por qué usar herramientas moleculares? Sistemática - Delimitación de especies Concepto filogenético de especie Diagnosis
Biología Molecular y Filogenia en Micología Biól. Nicolás Pastor Micología 2014 Por qué usar herramientas moleculares? Sistemática - Delimitación de especies Concepto filogenético de especie Diagnosis
Para obtener más información sobre esta función, haga clic en un tema:
 Cuando envíe un trabajo a la impresora, puede especificar en el controlador que desea que la impresora retenga el trabajo en la memoria. Cuando esté preparado para imprimir el trabajo, debe ir a la impresora
Cuando envíe un trabajo a la impresora, puede especificar en el controlador que desea que la impresora retenga el trabajo en la memoria. Cuando esté preparado para imprimir el trabajo, debe ir a la impresora
PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN DE LAS COMPETENCIAS PROFESIONALES CUESTIONARIO DE AUTOEVALUACIÓN PARA LAS TRABAJADORAS Y TRABAJADORES
 MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo
 Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo ESQUEMA GENERAL Sesión 1. Extracción y precipitación con sulfato de amonio. Sesión 2. Purificación por cromatografía Sesión
Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo ESQUEMA GENERAL Sesión 1. Extracción y precipitación con sulfato de amonio. Sesión 2. Purificación por cromatografía Sesión
Anexo Conservar a 4 o C.
 Anexo 1 Purificación de ADN viral 1. Partir de un sobrenadante de un cultivo celular infectado y clarificado. 2. Concentrar las formas brotantes de los baculovirus por centrifugación (centrifugrar 1,5
Anexo 1 Purificación de ADN viral 1. Partir de un sobrenadante de un cultivo celular infectado y clarificado. 2. Concentrar las formas brotantes de los baculovirus por centrifugación (centrifugrar 1,5
Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato
 Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
TERMINOLOGÍA ANALÍTICA - PROCESO ANALÍTICO - TÉCNICA ANALÍTICA - MÉTODO ANALÍTICO - PROCEDIMIENTO ANALÍTICO - PROTOCOLO ANALÍTICO
 TERMINOLOGÍA ANALÍTICA - PROCESO ANALÍTICO - TÉCNICA ANALÍTICA - MÉTODO ANALÍTICO - PROCEDIMIENTO ANALÍTICO - PROTOCOLO ANALÍTICO PROCESO ANALÍTICO Conjunto de operaciones analíticas intercaladas que se
TERMINOLOGÍA ANALÍTICA - PROCESO ANALÍTICO - TÉCNICA ANALÍTICA - MÉTODO ANALÍTICO - PROCEDIMIENTO ANALÍTICO - PROTOCOLO ANALÍTICO PROCESO ANALÍTICO Conjunto de operaciones analíticas intercaladas que se
Detección de mutaciones en el gen rpob de M. tuberculosis 8 MATERIALES Y MÉTODOS
 8 MATERIALES Y MÉTODOS 8.2 CEPAS DE M. TUBERCULOSIS. Se utilizó 1 cepa control y se analizaron un total de 13 extracciones diferentes de DNA de cepas de M. tuberculosis previamente aisladas de muestras
8 MATERIALES Y MÉTODOS 8.2 CEPAS DE M. TUBERCULOSIS. Se utilizó 1 cepa control y se analizaron un total de 13 extracciones diferentes de DNA de cepas de M. tuberculosis previamente aisladas de muestras
Anexo A: Métodos de extracción de RNA
 Anexo A: Métodos de extracción de RNA Anexo A.1: Extracción de RNA total con Trizol (Invitrogen) Anexo A.1.1 Extracción: 1. Pulverizar 100mg de material vegetal con Nitrógeno líquido. 2. Homogenizar el
Anexo A: Métodos de extracción de RNA Anexo A.1: Extracción de RNA total con Trizol (Invitrogen) Anexo A.1.1 Extracción: 1. Pulverizar 100mg de material vegetal con Nitrógeno líquido. 2. Homogenizar el
Ruptura y Disrupción celular
 Utilización de las reglas de oro de la purificación REGLA 1 Elegir procesos de separación basados en diferentes propiedades físicas, químicas o bioquímicas REGLA 2 Separar las impurezas más abundantes
Utilización de las reglas de oro de la purificación REGLA 1 Elegir procesos de separación basados en diferentes propiedades físicas, químicas o bioquímicas REGLA 2 Separar las impurezas más abundantes
DANAGENE GENOMIC DNA KIT
 Ref. 0603.1 Ref. 0603.2 Ref. 0603.3 DANAGENE GENOMIC DNA KIT 1.INTRODUCCION 1.1 Descripción del producto Este kit está designado para la extracción de ADN genómico de alta calidad a partir de una amplia
Ref. 0603.1 Ref. 0603.2 Ref. 0603.3 DANAGENE GENOMIC DNA KIT 1.INTRODUCCION 1.1 Descripción del producto Este kit está designado para la extracción de ADN genómico de alta calidad a partir de una amplia
Biotecnología - Bioprocesos II. INTRODUCCION A LOS PROCESOS BIOTECNOLOGICOS Separación y purificación en Biotecnología ( Downstream processing )
 Biotecnología - Bioprocesos II INTRODUCCION A LOS PROCESOS BIOTECNOLOGICOS Separación y purificación en Biotecnología ( Downstream processing ) La comercialización de nuevos productos obtenidos a través
Biotecnología - Bioprocesos II INTRODUCCION A LOS PROCESOS BIOTECNOLOGICOS Separación y purificación en Biotecnología ( Downstream processing ) La comercialización de nuevos productos obtenidos a través
ANÁLISIS CUANTITATIVO POR WDFRX
 ANÁLISIS CUANTITATIVO POR WDFRX El análisis cuantitativo se obtiene mediante la medida de las intensidades de las energías emitidas por la muestra. Siendo la intensidad de la emisión (número de fotones)
ANÁLISIS CUANTITATIVO POR WDFRX El análisis cuantitativo se obtiene mediante la medida de las intensidades de las energías emitidas por la muestra. Siendo la intensidad de la emisión (número de fotones)
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
EXTRACCIÓN Y DETERMINACIÓN DE ÁCIDOS NUCLEICOS DE LA LEVADURA
 EXTRACCIÓN Y DETERMINACIÓN DE ÁCIDS NUCLEICS DE LA LEVADURA bjetivos: Al finalizar el trabajo práctico los cursantes estarán en capacidad de: - Extraer ARN a partir de células de levadura. - Realizar la
EXTRACCIÓN Y DETERMINACIÓN DE ÁCIDS NUCLEICS DE LA LEVADURA bjetivos: Al finalizar el trabajo práctico los cursantes estarán en capacidad de: - Extraer ARN a partir de células de levadura. - Realizar la
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA. Grado en Medicina
 DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
Tema 11. Herramientas de la genética molecular
 Tema 11. Herramientas de la genética molecular Genética CC. Mar 2004-05 Objetivos Técnicas de clonación Construcción de genotecas e identificación de secuencias Mapas de restricción Amplificación (PCR)
Tema 11. Herramientas de la genética molecular Genética CC. Mar 2004-05 Objetivos Técnicas de clonación Construcción de genotecas e identificación de secuencias Mapas de restricción Amplificación (PCR)
Requisitos de las muestras y condiciones de análisis
 Requisitos de las muestras y condiciones de análisis Página 1 INTRODUCCION A continuación se indican los requisitos generales que han de cumplir las muestras para poder ser analizadas en el Servicio de
Requisitos de las muestras y condiciones de análisis Página 1 INTRODUCCION A continuación se indican los requisitos generales que han de cumplir las muestras para poder ser analizadas en el Servicio de
PRÁCTICA 1: MÉTODOS DE RUPTURA CELULAR
 PRÁCTICA 1: MÉTODOS DE RUPTURA CELULAR FUNDAMENTO TEÓRICO Para estudiar la estructura y/o las funciones metabólicas de los distintos compartimentos celulares hay que aislarlos del resto de los orgánulos.
PRÁCTICA 1: MÉTODOS DE RUPTURA CELULAR FUNDAMENTO TEÓRICO Para estudiar la estructura y/o las funciones metabólicas de los distintos compartimentos celulares hay que aislarlos del resto de los orgánulos.
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS. DOCENTES: Dr. Andrés Ciochinni, Dra. Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR
 TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Dr. Andrés Ciochinni, Dra. Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN). El ADN es un polímero
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Dr. Andrés Ciochinni, Dra. Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN). El ADN es un polímero
Protocolo de laboratorio para purificación manual de ADN a partir de una muestra entera
 Protocolo de laboratorio para purificación manual de ADN a partir de una muestra entera Para la purificación de ADN genómico de todos los estuches de colección Oragene y ORAcollect. Visite nuestro sitio
Protocolo de laboratorio para purificación manual de ADN a partir de una muestra entera Para la purificación de ADN genómico de todos los estuches de colección Oragene y ORAcollect. Visite nuestro sitio
Técnicas de Biología Molecular. Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Extracción de ácidos nucleicos o o DNA RNA Tipos de Muestras Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Extracción de ácidos nucleicos o o DNA RNA Tipos de Muestras Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
7/13/2009. Breve introducción a la biología molecular y sus aplicaciones. Victoria koala. Queensland koala. Morfología vs. ADN? Phascolarctus cinereus
 Morfología vs. ADN? Phascolarctus cinereus Queensland koala Victoria koala New South Wales koala 3 subespecies Morfológicamente: Diferencias en tamaño y color principalmente Molecularmente: Un grupo dividido
Morfología vs. ADN? Phascolarctus cinereus Queensland koala Victoria koala New South Wales koala 3 subespecies Morfológicamente: Diferencias en tamaño y color principalmente Molecularmente: Un grupo dividido
CURSO ACADÉMICO 2009 2010
 TITULACIÓN: BIOLOGÍA CURSO ACADÉMICO 2009 2010 GENÉTICA APLICADA CÓDIGO: 200810419 Departamento de adscripción: Parasitología, Ecología y Genética Área de conocimiento: Genética Ciclo: 2º Curso: 4º Tipo:
TITULACIÓN: BIOLOGÍA CURSO ACADÉMICO 2009 2010 GENÉTICA APLICADA CÓDIGO: 200810419 Departamento de adscripción: Parasitología, Ecología y Genética Área de conocimiento: Genética Ciclo: 2º Curso: 4º Tipo:
