CARACTERIZACION DE LAS PROTEINAS: PESO MOLECULAR. DETERMINACION DE LA ESTRUCTURA PRIMARIA.
|
|
|
- Vicenta Valdéz San Segundo
- hace 6 años
- Vistas:
Transcripción
1 CARACTERIZACION DE LAS PROTEINAS: PESO MOLECULAR. DETERMINACION DE LA ESTRUCTURA PRIMARIA.
2 ELECTROFORESIS. V = E z / f v: velocidad de migración de la molécula en un campo eléctrico. E: intensidad de campo eléctrico, z: carga neta de la proteína. f = 6phr es el coeficiente friccional. h (eta) es la viscosidad del medio y r es el radio de la partícula. Electroforesis en gel de poliacrilamida: (PAGE) o en presencia de SDS (SDS-PAGE). en condiciones nativas Isoelectroenfocado (IEF): Separación por punto isoeléctrico (pi). Electroforesis bidimensional en gel de poliacrilamida (2D -PAGE): IEF en unaprimera dimensión, SDS-PAGE en la segunda.
3
4
5
6 ISOELECTROENFOCADO: Separación por pi. Las proteínas se corren en un gradiente de ph preformado, que se obtiene sometiendo a electroforesis previa una mezcla de polianfolitos (polímeros pequeños con múltiple carga) con diferentes valores de pi. En A se carga la muestra y se aplica el voltaje, con lo cual las proteínas migrarán hasta llegar al valor de su pi, donde, al no tener carga neta, quedarán enfocadas, lo que se observa en B.
7
8 DETERMINACIÓN DEL PESO MOLECULAR DE LAS PROTEINAS. Mr = masa molecular relativa (por ejemplo, 50,000) Masa molecular: Por ejemplo 50,000daltons (50 kda). A) Proteína nativa: 1) Ultracentrifugación. 2) Filtración por gel. 3) Espectrometría de masa. B) Subunidad: 1) Electroforesis en gel de poliacrilamida en presencia de dodecil sulfato de sodio (SDS-PAGE).
9 ULTRACENTRIFUGACION. Fc= fuerza centrífuga. r = radio del rotor. w = velocidad angular. w 2 r = campo centrífugo. = volumen específico parcial (inversa de la densidad de la proteína). m (masa efectiva) es menor que m (masa real) porque el flúido desplazado ejerce una fuerza opuesta (1 r). f= coeficiente friccional de la partícula = 6phr, donde h es la viscosidad del medio y r el radio de la partícula. v = Fc / f = m(1 r) w 2 r / f s = coeficiente de sedimentación (unidades seg = 1 Svedberg). s = v / w 2 r = m (1 r) / f La velocidad de sedimentación (v) depende en parte de la masa de la partícula; tambien depende de su forma (una partícula compacta tiene un valor de f menor que el de una alargada); una partícula densa se mueve mas rápido que una menos densa porque su valor de (1 r) es menor; y depende tambien de la densidad de la solución (r): si r es < 1, se hunde; si es > 1, flota y si es = 1 no se desplaza.
10
11 Ultracentrifugación zonal (en gradiente) La solución de proteína se corre en un gradiente de densidad (por ejemplo, sacarosa), con proteínas de s conocido como marcadoras.
12
13
14 Peso molecular de subunidad: Determinacion por SDS-PAGE
15
16 ESPECTROMETRIA DE MASA: SUS APLICACIONES EN LA DETERMINACION DE PESOS MOLECULARES DE PROTEINAS Y EN LA SECUENCIACION DE PEPTIDOS.
17 Espectrometría de Masa La Espectrometría de Masa es una tecnica que permite determinar la masa de moléculas por medio de la medida de la relacion masa/carga (m/z). La medida de masas moleculares involucra la produccion, separación y detección de iones moleculares en fase gaseosa. Cada molécula puede originar iones moleculares con distinto número de cargas ( y distintas m/z).
18 Un espectrómetro de masa consiste de tres módulos: 1) Fuente de iones. 2) Analizador de masas. 3) Sistema de detección y adquisición de datos. Técnicas actuales para trabajo biológico: 1) MALDI-ToF MS (desorción/ionización por laser asistida por matriz, combinada con análisis de masas por tiempo de vuelo) 2) ES/MS (ionización por electrospray combinada con análisis de masas por cuadrupolo)
19 ES/MS IONIZACION POR ELECTROSPRAY GENERACION DE IONES MOLECULARES (ESI MS) La solución a analizar se pasa a través de una aguja hipodérmica mantenida a un alto potencial. Se forma un cono de gotitas muy finas, altamente cargadas, que irradian de la punta de la aguja. Se evaporan rápidamente, pasando las moléculas a analizar a la fase gaseosa. Si son moléculas grandes se producen iones con cargas múltiples.
20 Aqueous solution 1914, John Zeleny dripping, burst, pulsating, cone-jet Sir Geoffrey Taylor (1964). "Disintegration of Water Droplets in an Electric Field". Proc. Roy. Soc. London. Ser. A 280 (1382): 383. This ionization method arises in the attempt to couple the LC to MS The effect of vacuum, temperature and curtain gas causes the formation of increasingly smaller drops until the molecule is fully desolvated in vacuum.
21 What is the ionization mechanism in ESI? Lord Rayleigh, 1882 No tolerant to the presence of salts, surfactants, inorganic acids, TFA no so good for sensitivity, (regularly desalting steps are required, acetic and formic acids are used in the mobile phase).
22 ANALISIS DE LA MASA DE LOS IONES ANALISIS DE LA MASA DE LOS IONES MOLECULARES. MOLECULARES Los iones se dirigen al vacío del espectrómetro de masa de cuadrupolo a través de un orificio o capilar calentado. El cuadrupolo consta de cuatro barras metálicas paralelas que generan un campo eléctrico oscilatorio. Los iones se separan en este campo: sólo los de una determinada m/z se dejan pasar a una determinada amplitud y frecuencia de oscilación de los potenciales eléctricos aplicados a las barras, y llegan al detector.
23 Quadrupole Mass Analyzer radiofrequency DC: Direct current RF: Radio frequency Limited mass range (4 000 Da). From: Ion transmitted along the quadrupole in an stable trajectory Ions do not have an stable trajectory and is ejected from the quadrupole Were the first mass analyzer to be coupled to ESI. Excellent mass filters. Acquisition every Da.
24
25 MALDI-ToF MS GENERACION DE IONES MOLECULARES Pseudocristales mixtos de una matriz (p.ej.: ácido 3,5- dimetoxi-4-hidroxicinámico). Se los irradia con un pulso de laser. La sublimación rápida de los cristales lleva a la formación de iones moleculares protonados en fase gaseosa. SeANALISIS aceleran ade una LA energía MASA cinética DE LOS de IONES aprox. MOLECULARES. 25 KeV y se los deja volar a través de una distancia fija sin campo eléctrico. Teniendo energía cinética idéntica, los iones chicos se mueven más rápido que los grandes y llegan antes al detector. M/z se determina a partir del tiempo de vuelo, comparando con standards.
26 ESQUEMA GENERAL DE UN ESPECTROMETRO DE MASA MALDI-TOF Sample plate Extraction grids Laser Timed ion selector Reflector detector Reflector Linear detector Beam guide Pumping Camera Pumping
27 Espectrometro de masa, MALDI-ToF
28 Sample mixed with a strong UV absorbing matrix. The matrix absorbs energy from the laser causing the sample and matrix to be volatilized. Ionized matrix molecules transfer a proton to the sample. Sample ions are then accelerated down the flight tube for mass analysis (TOF MS). MALDI is a pulsed ionization source
29 MALDI: Matrix Assisted Laser Desorption Ionization. Excess of Matrix Matrix : Analyte 1000: :1 Ionization by laser (pulses 1-5nsec but.high potency: kw MW). Nd:YAG (355nm) N 2 laser (337 nm) Easy Sample prep: Dried droplet Layered method Compatible with TOF analyzers Highly efficient ionization method
30 Matrices absorb strongly l of the laser irradiation cold matrix hot matrix
31 La relación masa a carga de un ion es proporcional al cuadrado de su tiempo de vuelo. m z 2t 2 L 2 K t = Tiempo de vuelo L = Longitud recorrida por los iones dentro del analizador m = Masa K = Energía cinética del ion z = Número de cargas en el ion
32 MALDI-MS 1. MALDI-MS spectra are very simple, only singly-charged ions are observed: (M+H) + or (M-H) Very efficient ionization method. High sensitivity in the analysis of biomolecules and biopolimers. 3. Ideal for the analysis of mixture of tryptic peptides (ID of proteins by PMF, peptide mass fingerprinting). 4. High throughput analysis. 5. Compatible with Time of Flight mass analyzer. 6. Laser intensity may help to induce fragmentations and obtain structural information.
33
34
35 SECUENCIACION DE PEPTIDOS Y PROTEINAS
36 Cuales son lo objetivos de la secuenciación de péptidos? Comparar estructuras primarias de diferentes proteínas para establecer relaciones evolutivas (actualmente, se hace mayormente usando secuencias traducidas de secuencias de nucleótidos). Obtener una secuencia de péptido de una proteína desconocida proveniente de un organismo cuyo genoma no está secuenciado; esto permitirá la síntesis de oligonuclótidos degenerados para identificar y clonar el gen que la codifica. Confirmar la identidad de una proteína sospechada a partir de un experimento de PMF. Identificar modificaciones post-traduccionales. Cont.
37 SECUENCIACION DE PROTEINAS Y PEPTIDOS. Para secuenciar una proteína siempre se debe partir de la proteína purificada. La purificación puede consistir en obtener la proteína pura en un tubo, a través de los métodos de purificación ya mencionados, o, aún si no está completamente homogénea, como una banda discreta en un gel de SDS-PAGE. Para secuenciar una proteína siempre se la debe fragmentar para obtener péptidos de menor tamaño, secuenciables por distintos procedimientos. Esta fragmentación se hace enzimáticamente (proteasas) o químicamente (p.ej.: BrCN).
38 Pasos a seguir para secuenciar una proteína pura por el método de Edman. 1) Desnaturalizar, con urea por ejemplo, y reducir los puentes disulfuro. 2) Bloquear los grupos SH, con iodoacetamida por ejemplo. 3) Fragmentar la proteína en péptidos mas pequeños, usando dos métodos diferentes (por ejemplo, digestiones con tripsina y BrCN) 4) Separar los péptidos obtenidos por el primer método por HPLC en fase reversa (RP-HPLC). 5) Tratar cada péptido purificado con el reactivo de Edman, isotiocianato de fenilo, en un secuenciador automático. 6) Identificar los residuos de aminoácidos por RP-HPLC de los derivados de feniltiohidantoína (PTH-aminoácido) on line en el secuenciador. 7) Hacer lo mismo con los péptidos obtenidos por el segundo método de ruptura de la proteína. 8) Armar la secuencia por superposición de los péptidos obtenidos por ambos métodos.
39
40
41 SECUENCIACION DE PROTEINAS Y PEPTIDOS. 1) Método de Edman: Previa separación de los péptidos por HPLC en fase reversa, secuenciarlos químicamente por reacción con el reactivo de Edman (isotiocianato de fenilo), en un secuenciador automático.
42 LAS MALATO DEHIDROGENASAS DE TRYPANOSOMA CRUZI MDH-1 MDH-2
43
44 El Procise Protein Sequencing System de Applied Biosystems Está compuesto por 4 módulos integrados: el Secuenciador propiamente dicho, el Microgradient Delivery System, el detector de UV de longitud de onda variable, y la computadora equipada con Procise control software y SequencePro software. El sistema controla la entrega precisa de hasta 12 solventes y reactivos diferentes. Incluye el cartucho de reacción, donde se obtiene el primer derivado, el frasco de conversión donde se lo transforma en el PTH-derivado, que luego se transfiere a la columna de HPLC en fase reversa, donde se separan por el gradiente de solvente, se lo detecta en el detector UV/VIS por su absorbancia y se lo identifica por el tiempo de retención en la columna. La calibración se hace en base a una corrida con los PTH derivados de los 20 aminoácidos proteicos, además en algún caso de un derivado correspondiente a una modificación post-traduccional que se quiere detectar.
45
46 1er.Ciclo 1er. Ciclo 2 2o. Ciclo DEGRADACI ON DE EDMAN Secuenciación automática por REACCION DEL RESIDUO N- el TERMINAL Método de CON ISOTIOCIANATO Edman DE FENILO (N-terminal de la cruzipaína) 3er. 3er. Ciclo
47
48 2) Espectrometría de masa: Puede utilizarse para secuenciar péptidos o para identificar proteínas. 2a) Secuenciación: Seleccionar un péptido determinado por MS y fragmentarlo, obteniendo su secuencia a partir de las masas de los fragmentos resultantes (CID, fragmentación en cámara de colisión, o PSD, decaimiento después de la fuente de iones) 2b) Identificación: Sin separar los péptidos, determinar la masa de cierto número de los mismos por MS e identificar a la proteína en los bancos de datos, por comparación con las masas de digeridos virtuales de todas las proteínas ya secuenciadas. Se conoce como peptide mass fingerprinting (PMF). Es óptimo cuando el genoma del organismo de origen está completamente secuenciado.
49 SECUENCIACION POR ESPECTROMETRIA DE MASA FRAGMENTACION DE LOS PEPTIDOS Un péptido cargado puede ser fragmentado en dos trozos de tres maneras, que pueden producir un par de iones a y x, un par de iones b e y, o un par de iones c y z. Teóricamente una fragmentación puede tener lugar en cualquier lugar de un péptido, y se espera un espectro que contenga todos los posibles picos de iones. En la práctica, debido a la fortaleza irregular de las ligaduras en las diferentes posiciones, los diferentes iones aparecen con distintas frecuencias. Los mas abundantes son los iones y, los cuales a menudo forman la serie completa en un espectro. Los siguientes en frecuencia son los iones a y b, de los cuales muchos no se observan. Los iones c, x y z se presentan mucho menos frecuentemente. Adicionalmente, estos iones pueden formar nuevos iones por pérdida de agua o de amonio. La introducción de un grupo sulfónico en el N-terminal de un péptido tríptico favorece la fragmentación que da lugar a los iones y, y suprime a los de la serie b, dando un espectro simplificado, que facilita la determinación de la secuencia.
50 The backbone fragmentation are the most intense signals in the ESI-MSMS spectra of peptides fragmented by CID. x n y n z n C-terminal ions O H N C NH 2 C N COOH N C N-terminal ions a n b n c n Roepstorff, P., and Fohlmann J. Biomed. Mass Spectrom. 11, 601 (1984) Jhonson R. S., Martin S. A., Biemann K., Int. J. Mass Spectrom. Ion Processes. 86, 137 (1988).
51 Peptide fragmentation (2)
52 Collision-induced dissociation (CID) Es una técnica de fragmentación usada para obtener información estructural. Se utiliza la colisión con las moléculas de un gas para obtener la fragmentación de un ión seleccionado por su masa. La ES/MS puede aplicarse directamente a la secuenciación de péptidos, utilizando un ES MS/MS, es decir un espectrómetro de masa de electrospray en tandem, que tiene dos analizadores de masa separados por una celda de colisión, donde el péptido seleccionado se fragmenta y da iones hijos que se separan en elsegundo analizador y permiten determinar la secuencia.
53
54 Post-source decay (PSD) Es una técnica de fragmentación usada con MALDI-ToF MS para obtener información estructural, típicamente a partir de péptidos menores que 2 kda. El decaimiento de un ion precursor seleccionado por su masa tiene lugar después que el ión deja la fuente de iones, en el tubo de vuelo, y los fragmentos iónicos son separados por el reflectrón.
55 La trayectoria de vuelo de un ion puede ser incrementada incorporando un espejo iónico o reflectron al final del tubo de vuelo. Tal dispositivo retarda y luego revierte las velocidades iónicas para corregir diferencias menores en energía cinética entre iones de la misma relación m/z, y así minimiza variaciones en los tiempos de vuelo y mejora la resolución y la precisión de los valores de masa. Reflectron (ion mirror): compensates for the kinetic energy spread of ions from the source (100 ev to 1 kev) by reflecting the ions back along the same flight path prior to detection. Ions of equal m/z with different velocities Detector
56 EL FUNDAMENTO DE PSD Los fragmentos se obtienen por descomposición de los iones originales, que ocurre en el tubo de vuelo, es decir, después de que los iones salieron de la fuente. La descomposición se debe a la energía interna del ión precursor. Sólo una pequeña fracción de los iones precursores tiene suficiente energía para fragmentarse. Esta energía puede aumentarse usando una intensidad del laser mas elevada, o usando una celda de colisión. No. de iones Cuando los iones precursores se fragmentan, la velocidad de los fragmentos es igual a la del precursor, por lo cual viajarán todos juntos en el tubo de vuelo. Esto se debe a que la velocidad es determinada por la aceleración inicial; si la energía inicial es 20 kev, las energías de la unión química están alrededor de 10 ev, de modo que la ruptura de una unión tendrá un efecto mínimo sobre la velocidad. Energía interna Los aparatos usados tienen un selector de velocidad (Bradbury-Neilson gate), que permite seleccionar al ión precursor y a sus fragmentos para que pasen a su través y lleguen al reflectrón. El ión molecular precursor intacto tiene una energía cinética traslacional KE M = ½ Mv 2, siendo M su masa y v su velocidad. Un fragmento del ión precursor tiene una energía cinética traslacional KE m = KE M (m/m), siendo M la masa del precursor y m la del fragmento. Estas diferencias hacen que los iones precursor e hijos sigan diferentes caminos en el reflectrón. Cómo se hace para que los fragmentos que están viajando a la misma velocidad que el precursor pero tienen una energía cinética reducida lleguen enfocados al detector? Variando la pendiente del gradiente de voltaje del reflectrón a través del rango de masas de los fragmentos.
57 Los iones precursores y los fragmentos iónicos de PSD siguen diferentes caminos en el reflectrón normal Detector del reflectrón Ion precursor intacto + + Fragment formado por PSD 0 V. Reflectrón +20 kv
58 A una relación de espejo = 1.00 BH + AH+ MH + MH+ ( 1,000) correctamente enfocado AH+ (700) Pobremente enfocado BH+ (300) Pobremente enfocado
59 A una relación de espejo = 0.7 BH + AH + MH + MH+ ( 1,000) no enfocado AH+ (700) correctamente enfocado BH+ (300) pobremente enfocado
60 A una relación de espejo = 0.3 BH + AH + & MH + MH+ ( 1,000) no enfocado AH+ (700) no enfocado BH+ (300) correctamente enfocado
61 AA sequencing using Post Source Decay MALDI has in principle been possible for many years, buthas not been much used due to difficult interpretation. A dramatic improvement was the development of derivatizations of the peptide prior to PSD: Acidification by sulfonation directs fragmentation to the b-y bond and as the b-fragments become neutral, a unique series of y-ion results. Two common reagents are CAF or SPITC shown below: O O N O O O S O OH + H 2 N NHS-ester of 3-Sulfopropionic acid anhydride H R 2 Pep RT, ph>9 O O HO S O NH Mass addition, +136 Da H R 2 Pep Adds 215 Da at the N-terminal AND free Lys side chain
62 How can sulfonation at the N-terminus of tryptic peptides improve PSD performance? 1. It favours bond breakage at the b/y position (the peptide bond) 2. It neutralizes the b-fragments (negatively charged sulfogroup + proton); therefore the b-fragments never leave the target. 3. The resulting series of y-ions is easily interpreted the distance betwen adjacent peaks represents one amino acid. The sulfonation reaction derivatizes the N-termini of the tryptic peptides (and the side chain of Lys in approximately half of the peptides) To avoid the Lys sulfonation, Lys is first blocked by an imidazole reaction, adding 68 Da/Lys. This is specific for the Lys side arm and does not affect the N-terminus.
63 A tryptic digest (probably from an SDS-PAGE- or a 2-D-gel) is analyzed by PMF. The Lys blocking (when CAF was used) and the sulfonation reactions areperformed. (One can ignore the Lys blocking and only look for Arg-peptides). The modified peptide mixture is analyzed again by MALDI. Look for masses representing Arg or Lys peptides. (I. e. those that increased by 136 or 204 Da) Switch the MALDI to PSD and select THAT peptide mass. Perform the PSD analysis MALDI - this takes approximately 2 in a TOF/TOF
64 Peptide mass fingerprinting of Trypanosoma cruzi aconitase (- / + sulfonation)
65 Post Source Decay (PSD) sequencing of aconitase tryptic peptides
66 IDENTIFICACIÓN DE PROTEÍNAS POR PEPTIDE MASS FINGERPRINTING (PMF) La muestra reducida y alquilada (en un fragmento de gel o mancha) se digiere con una proteasa (tripsina?). La mezcla de péptidos resultante se analiza por MALDI-ToF-MS. La lista de masas de péptidos se utiliza para escanear un digerido in-silica de una base de datos de secuencias por Internet. El resultado de la búsqueda se presenta como probability scores y otras medidas de confianza La homogeneidad de la muestra y la precisión de la medida son factores muy improtantes para una interpretación segura de los resultados.
67 Intens. [a.u.] x10 4 PMF of band # m/z
68
69
70
71
72 LAS MALATO DEHIDROGENASAS DE TRYPANOSOMA CRUZI MDH-1 MDH-2
73 MDH-1 MDH-2
74 EDMAN
75
76 SINTESIS QUIMICA DE PEPTIDOS La sintesis quimica de peptidos es muy importante actualmente, pues permite a) estudiar el plegamiento de regiones individuales de una proteína; b) aislar, usandolos como ligandos para cromatografía de afinidad, a receptores y otras proteínas, c) usarlos como drogas, p.ej.: la 1-desamino-8-D-Arg-vasopresina, hormona antidiurética sin efectos sobre la presión sanguinea; d) para generar anticuerpos específicos. La sintesis se realiza en fase sólida, aplicando el método desarrollado por Merrifield, que le permitió sintetizar interferon (155 residuos) y ribonucleasa (124residuos), ambos activos Los aminoácidos que se introducen deben estar bloqueados en los sitios en que no debe producirse reacción, y luego desbloquearlos para continuar la etapa siguiente. Se efectúa con el residuo C- terminal ligado a una resina, lo que permite eliminar facilmente subproductos.
77
78
79 INTRODUCCION A LA PROTEOMICA
80 GENOMA Y PROTEOMA GENOME: GENes and ChromosOMEs (Winkler, 1920): Conjunto completo de los cromosomas y sus genes. PROTEOME: Conjunto completo de las proteínas de un organismo (el conjunto de proteínas codificadas por un genoma, Wilkins y Williams, 1994; Wasinger et al., 1995).
81 La secuenciación de los genomas se hizo posible con la introducción de los métodos de secuenciación del ADN, a partir del establecimiento del método utilizando dideoxynucleótidos por Frederick Sanger, en 1975, que le permitió secuenciar el genoma completo del bacteriófago X174, con 5,375 pares de bases, en La primera secuenciación completa del genoma de un organismo de vida libre fué la de Haemophilus influenzae (1.8 Mb), en El Human Genome Project comenzó en 1990 y se completó en 2003; la estimación del número total de genes en el genoma humano se estima entre 20,000 y 25,000. Al 28 de Marzo de 2017 la Kyoto Encyclopedia of Genes and Genomes (KEGG) registraba ya 360 genomas eucarióticos completos, además de 4128 genomas de bacterias y 248 de Archaea.
82 DETERMINACIÓN DE LA FUNCIÓN DE LOS GENES Determinación de las características químicas, bioquímicas y biológicas de las proteínas. 1) Estructura molecular. 2) Modificaciones post-traduccionales. 3) Capacidad de unir ligandos. 4) Velocidad de síntesis y degradación. Concentracion intracelular. 5) Especificidad de tejido, célula y organela. 6) Especificidad de sexo. 7) Especificidad de estadío de desarrollo embrionario, post natal y de envejecimiento. Se estima en 252 el número de tipos celulares diferentes que se encuentran en el hombre. Todas esas células tienen el mismo DNA, pero todas ellas presentan diferencias cuali y cuantitativas en sus proteínas. El DNAalmacena información; lasproteínas hacen todo lo demás.
83 HAY IGUAL NUMERO DE PROTEINAS QUE DE GENES? NO. gen. Hay frecuentemente mas de una proteína proveniente de un único Causas principales: 1) Splicing alternativo: A nivel de la maduración del mrna. Diferentes proteínas armadas usando diferentes combinaciones de exones. 2) Modificación post-traduccional: La proteína es modificada después de la traducción, ya sea procesándola proteolíticamente, o modificando quimicamente residuos de aminoácidos, o de ambas maneras.
84 ADN Pre-mARN Transcripción marn 1 marn 2 marn 3 Proteína 1 Proteína 2 Proteína 3 Splicing alternativo Traducción Modificación Post-traduccional y/o Proteólisis Prot. 1 Prot. 1 Prot. 2 Prot. 2 Prot. 3 Prot. 3
85 LA NATURALEZA POLIGENICA DE LAS PROTEINAS. Para la funcionalidad adecuada de una proteína se requiere mucho mas que la integridad del gen estructural que la codifica. Si este está mutado, y el producto es inactivo, naturalmente la proteína no será funcional. Pero puede suceder algo parecido si el gen estructural está intacto, pero hay mutaciones en otros genes que participan en lo que realmente le interesa a la célula, que es la proteína perfectamente plegada, con las modificaciones posttraduccionales requeridas y localizada en el compartimento subcelular correcto. genes: Se requerirá la integridad y funcionalidad correcta de otros
86 a) Los que codifican a enzimas que introducen modificaciones post-traduccionales. b) Los que codifican proteinasas de procesamiento. c) Los que codifican proteínas requeridas para el transporte e integración de la proteína en el compartimento correcto. d) Los que codifican enzimas que catalizan la degradación de la proteína. e) Los genes regulatorios que codifican factores de transcripción necesarios para la expresión de la proteína. f) Secuencias regulatorias, como enhancers, etc. g) Genes involucrados en metilación del DNA, estructura de la cromatina, etc.
87 Gen estructural Genes que codifican factores de transcripción. Genes que codifican proteínas regulatorias. Genes que codifican proteínas que modifican la estructura de la cromatina. Secuencias regulatorias en el ADN ( enhancers ) Genes que codifican chaperonas necesarias para el plegamiento correcto. Genes que codifican enzimas que modifican post-traduccionalmente a la proteína. Genes que codifican proteasas de procesamiento. Genes que codifican proteínas de transporte e integración a organelas. Genes que codifican proteasas que degradan a la proteína Proteína madura, correctamente localizada, en la concentración necesaria
88 DESAFIOS TECNICOS DE LA PROTEOMICA. 1) Si suponemos alrededor de 22,000 genes en el genoma humano, y que cada gen da origen a entre 3 y 6 o más proteínas, el número total de proteínas en un organismo superior puede variar entre 50,000 y 500,000. 2) Las proteínas tienen una heterogeneidad mucho mayor que los ácidos nucleicos. La composición y secuencia de aminoácidos de una proteína determina: a) su masa molecular; b) su carga total a diferentes valores de ph; c) su hidrofobicidad; d) su forma, es decir el plegamiento de la proteína nativa. Esto a su vez determina la solubilidad de las proteínas en diferentes solventes, su comportamiento en los métodos de separación y purificación, y su vida media in vitro. La diversidad de propiedades fisicoquímicas es muchísimo mayor quela de los ácidos nucleicos. 3) Los valores de pi de las proteínas varían entre menos de 3 y más de 12, con dos grandes agrupamientos, uno alrededor de 5.5 y el otro alrededor de 8.5. Los ácidos nucleicos tienen un rango estrecho de pi, en la zona ácida.
89 4) Los ácidos nucleicos son muy solubles en soluciones acuosas, en tanto que muchas proteínas son muy hidrofóbicas, y por ello insolubles en agua. Se estima que más del 15 % de las proteínas totales nunca entrarán en un gel de 2D-PAGE por su extrema hidrofobicidad. 5) La masa molecular de las proteínas varía entre unos pocos miles y varios millones de Daltons, y su tamaño no puede predecirse con certeza a partir de la secuencia de aminoácidos derivada de la secuencia dedna, porlas modificaciones post-traduccionales. 6) La concentración de las proteínas varía dentro de un rango muy amplio, probablemente mayor que el rango de los mrnas. La diferencia en concentración de proteínas abundantes y raras en los flúidos corporales es mayor que 12 órdenes de magnitud.
90 MACROMOLECULAS DE UNA CELULA TIPICA HUMANA: 1) DNA: unos 22,000 genes. 2) mrnas: Alrededor de 40,000 moléculas en todo momento, pertenecientes a alrededor de 5,000 especies moleculares diferentes. Un 80 % están en bajos niveles. 3) Proteínas: Unos de moléculas, de las cuales las 100 proteínas más abundantes constituyen alrededor del 90 % de la masa proteica total celular. La diferencia entre las proteínas demayor abundancia, como la actina, y las de menor abundancia, como proteínas regulatorias, es de alrededor de de veces.
91 LAS HERRAMIENTAS PARA EL ESTUDIO DEL PROTEOMA La técnica clásica, y hasta hace unos pocos años, la principal utilizada, es la separación de las proteínas por electroforesis bidimensional en geles de poliacrilamida, seguida de identificación de dichas proteínas por secuenciación o, en general actualmente, por espectrometría de masa, después de hidrólisis enzimatica parcial. Actualmente lo mas utilizado para estudios amplios de proteómica son otras combinaciones de técnicas, por ejemplo cromatografía de alta resolución (HPLC) combinada con espectrometría de masa, o electroforesis capilar tambien combinada con espectrometría de masa. Este tipo de técnicas permite la identificación de proteínas en mezclas complejas, previa hidrólisis enzimática parcial.
92 ELECTROFORESIS BIDIMENSIONAL Electrophoresis 2000, 21,
93 VENTAJAS Y DESVENTAJAS DE LA ELECTROFORESIS BIDIMENSIONAL EN EL ESTUDIO DEL PROTEOMA. Ventajas: Excelente resolución: pueden identificarse a veces hasta 10,000 proteínas en un solo gel. Ninguna otra técnica es capaz de analizar simultáneamente miles de proteínas. Desventajas: 1)Pueden ser necesarios varios geles, corridos en diferentes condiciones de rango de ph y de concentración de acrilamida, para resolver las mezclas muy complejas. 2) Es difícil detectar proteínas muy grandes o muy chicas, muy ácidas o muy básicas, o proteínas de membrana (muy hidrofóbicas). 3) Se detectan sólo las proteínas de alta y mediana abundancia en el material en estudio. En cualquier célula, sus proteínas pueden variar en concentración en seis órdenes de magnitud. Para ver proteínas minoritarias, es necesario en general purificar la organela en que se encuentran y trabajar con ese material. Tambien se puede trabajar previa marcación radioactiva, para aumentar la sensibilidad de detección.
94 NUMERO DE MANCHAS PROTEICAS REVELADAS EN GELES GRANDES DE 2-D A PARTIR DE TRES ORGANOS DE RATON. Fracción tisular Número de manchas Hígado Cerebro Corazón (A) Sobrenadante (buffer) 9,204 8,458 4,790 (B) Extracto del pellet (urea, CHAPS) 1,975 1,692 1,470 (C) Pellet suspendido, DNA digerido Manchas totales por órgano 11,252 10,200 6,300 NOTA: Las manchas contadas en (A) no están en (B) ni en (C), y así sucesivamente. (A) son probablemente proteínas citosólicas; (B) de organelas, y (C) proteínas cromosomales, como lashistonas. LOS EXTRACTOS CONCENTRADOS DE ORGANELAS REVELAN MUCHAS PROTEINAS MINORITARIAS.
95 ID OF PROTEINS BY EDMAN vs MALDI-ToF-MS EDMAN DEGRAD. MALDI-ToF-MS Sensitivity Low pmole (6x10 11 moléculas; 67 ng albúmina) Low fmole (6x10 8 moléculas; 67 pg albúmina) Running cost US$ 150/peptide < 1 /analysis Analysis time Overnight A few min # Cell culture flasks needed
96 DIFFERENCE GEL ELECTROPHORESIS (DIGE). Fluorescent tagging of the two protein samples to be compared with two different dyes, amino reactive, and designed to keep the same relative mobility. The tagged protein samples are mixed and run in the same 2D gel. After the run fluorescence imaging of the gel is used to create two images, which are superimposed to identify pattern differences. This technique makes unnecessary to compare different gels. It is commercially available.
97 Isotope-coded affinity tags (ICAT) The ICAT method for measuring differential protein expression. (A) Structure of the ICAT reagent. ICAT consists of a biotin affinity group, a linker region that can incorporate heavy (deuterium) or light (hydrogen) atoms, and a thiol-reactive end group for linkage to cysteines. (B) ICAT strategy. Proteins are harvested from two different cell states and labeled on cysteine residues with either the light or heavy form of the ICAT reagent. Following labeling, the two protein samples are mixed and digested with a protease such as trypsin. Peptides labeled with the ICAT reagent can be purified by virtue of the biotin tag by using avidin chromatography. Following puri fication, ICAT-labeled peptides can be analyzed by MS to quantitate the peak ratios and proteins can be identified by sequencing the peptides with MS/MS.
98 Si bien la idea última detrás de los estudios de proteómica es llegar algún día al conocimiento completo de las proteínas de un organismo, lo mas frecuente actualmente es hacer proteómicas parciales: la proteómica deun tipo celular determinado; la proteómica de una organela aislada; la proteómica de las proteínas blanco de una determinada modificación posttraduccional; las diferencias en la expresión de proteínas antes y después de la diferenciación de un estadío a otro en un organismo que tiene un ciclo de vida complejo; las diferencias en la expresión de proteínas de una misma célula antes y después de un determinado tratamiento. Veremos a continuación dos ejemplos.
99 Identificación de proteínas potencialmente SUMOiladas en Trypanosoma cruzi Cromatografía líquida en 2 dimensiones acoplada a espectrometría de masa (2D LC MS/MS) Resuspensión en Urea Reducción con DTT+ Alquilación con IAA Digestión con Tripsina Desalado en Columna fase reversa C18 (DSC-18, Supelco, Sigma-Aldrich) Lleva muestra a sequedad Resuspende en 2% ACN/0.1%FA 20 μg proteína Columna intercambio ionico (SCX 5 ul opti-pak) Eluidos 0,25,50,100,200 y 500 mm NaCl Sistema de nano-hplc Trap loading C18-column Trap separation C18-column Elución en gradiente 5 40% ACN/0.1% FA en 100 min Análisis de péptidos en línea con un espectrómetro de masa (LTQ XL/ETD, Thermo Fisher Scientific).
100 Proteínas potencialmente SUMOiladas Se identificaron 7178 péptidos en ambas muestras. Corresponden a 2686 proteínas. 236 resultaron enriquecidas en la Muestra vs. Control Proteínas potencialmente sumoiladas Distribución por categoria funcional
101 Metacaspasa 3 in vivo Validación de proteínas SUMOiladas
102 Protein expression profiling by 2-DE. Whole-cell lysates from nontransformed and Abelson murine leukemia virus (AMuLV)-transformed mouse fibroblasts were resolved by 2-DE, and proteins were visualized by silver staining. Differentially expressed proteins were excised from the gel and identified by MS.
ANALIZADORES. Facultad de Ciencias Exactas y Naturales UBA
 ANALIZADORES 1 Analizadores Cuadrupolo (Q) Trampa de iones (IT) Sector magnético-eléctrico (BE) Tiempo de vuelo (TOF) Resonancia iónica ciclotrónica (ICR) 2 Analizadores Analizador de Tiempo de Vuelo (TOF)
ANALIZADORES 1 Analizadores Cuadrupolo (Q) Trampa de iones (IT) Sector magnético-eléctrico (BE) Tiempo de vuelo (TOF) Resonancia iónica ciclotrónica (ICR) 2 Analizadores Analizador de Tiempo de Vuelo (TOF)
Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo
 Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo ESQUEMA GENERAL Sesión 1. Extracción y precipitación con sulfato de amonio. Sesión 2. Purificación por cromatografía Sesión
Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo ESQUEMA GENERAL Sesión 1. Extracción y precipitación con sulfato de amonio. Sesión 2. Purificación por cromatografía Sesión
OFERTA DE ANÁLISIS. Tinción con coomassie coloidal
 Tinción con coomassie coloidal OFERTA DE ANÁLISIS La tinción de geles con Coomassie es una tinción lista para usar basada en el colorante coomassie coloidal G-250 que permite la detección a nivel de nanogramos
Tinción con coomassie coloidal OFERTA DE ANÁLISIS La tinción de geles con Coomassie es una tinción lista para usar basada en el colorante coomassie coloidal G-250 que permite la detección a nivel de nanogramos
Proteínas: Métodos para su Estudio
 Proteínas: Métodos para su Estudio Propiedades Espectroscópicas Todos los aminoácidos absorben luz en la región infraroja Sólo Phe, Tyr, & Trp absorben UV Absorbancia a 280 nm es un buen diagnóstico para
Proteínas: Métodos para su Estudio Propiedades Espectroscópicas Todos los aminoácidos absorben luz en la región infraroja Sólo Phe, Tyr, & Trp absorben UV Absorbancia a 280 nm es un buen diagnóstico para
IDENTIFICACIÓN DE PROTEÍNAS POR ESPECTROMETRÍA DE MASAS. EQUIPAMIENTO
 IDENTIFICACIÓN DE PROTEÍNAS POR ESPECTROMETRÍA DE MASAS. EQUIPAMIENTO La Espectrometría de Masas es una tecnología analítica esencial en el contexto de la proteómica actual debido a su alta capacidad de
IDENTIFICACIÓN DE PROTEÍNAS POR ESPECTROMETRÍA DE MASAS. EQUIPAMIENTO La Espectrometría de Masas es una tecnología analítica esencial en el contexto de la proteómica actual debido a su alta capacidad de
Espectrometría de masas Instrumentación y aplicaciones
 Clase 02 Espectroscopía de Compuestos Orgánicos MALDI-MS ESI-MS Espectrometría de masas Instrumentación y aplicaciones Espectrometría de Masa Fluid (no salt) + _ cyano-hydroxy cinnamic acid Gold tip needle
Clase 02 Espectroscopía de Compuestos Orgánicos MALDI-MS ESI-MS Espectrometría de masas Instrumentación y aplicaciones Espectrometría de Masa Fluid (no salt) + _ cyano-hydroxy cinnamic acid Gold tip needle
DESARROLLO COGNOSCITIVO Y APRENDIZAJE
 Prof. Jhonis Valbuena Cátedra: Análisis Instrumental La Electrofóresis es un método de la Química Analítica que utiliza corriente eléctrica controlada con la finalidad de separar biomolèculas según su
Prof. Jhonis Valbuena Cátedra: Análisis Instrumental La Electrofóresis es un método de la Química Analítica que utiliza corriente eléctrica controlada con la finalidad de separar biomolèculas según su
PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS. Es un ácido capaz de formar sales con iones cargados
 Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
ESI-MALDI-TOF. El análisis por espectrometría de masas se realiza en cuatro etapas básicas:
 ESI-MALDI-TOF Introducción Espectrometría de masas La espectrometría de masas es una técnica analítica en la que los átomos o moléculas de una muestra son ionizados, con mayor frecuencia positivamente,
ESI-MALDI-TOF Introducción Espectrometría de masas La espectrometría de masas es una técnica analítica en la que los átomos o moléculas de una muestra son ionizados, con mayor frecuencia positivamente,
Purificación de proteínas
 Purificación de proteínas Purificación Proceso que cuenta con varias etapas cuyo objetivo es lograr la concentración diferencial de la proteína o molécula de interés Condiciones generales a evaluar Calidad
Purificación de proteínas Purificación Proceso que cuenta con varias etapas cuyo objetivo es lograr la concentración diferencial de la proteína o molécula de interés Condiciones generales a evaluar Calidad
Técnicas de Estudio de las células
 Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
Capítulo 12 REGULACIÓN DE LA EXPRESIÓN GÉNICA. Factores de Transcripción. Metilación. Procesamiento del ARN. Post-traduccional
 REGULACIÓN DE LA EXPRESIÓN GÉNICA - Mecanismos de Regulación Regulación Procariontes Eucariontes Operón Lactosa Operón Triptofano Transcripcional Procesamiento del ARN Traduccional Post-traduccional Factores
REGULACIÓN DE LA EXPRESIÓN GÉNICA - Mecanismos de Regulación Regulación Procariontes Eucariontes Operón Lactosa Operón Triptofano Transcripcional Procesamiento del ARN Traduccional Post-traduccional Factores
ESPECTROMETRIA DE MASAS
 ESPECTROMETRIA DE MASAS Se puede sub-dividir en dos áreas de aplicación: Espectrometría de masas atómica EMA: determinar cuali y cuantitativamente los elementos presentes en una muestra. Espectrometría
ESPECTROMETRIA DE MASAS Se puede sub-dividir en dos áreas de aplicación: Espectrometría de masas atómica EMA: determinar cuali y cuantitativamente los elementos presentes en una muestra. Espectrometría
ESTRUCTURA Y PROPIEDADES DE PROTEÍNAS
 ESTRUCTURA Y PROPIEDADES DE PROTEÍNAS Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo PARTE I. EXTRACCIÓN Y PRECIPITACIÓN POR SALADO. Las proteínas se purifican mediante
ESTRUCTURA Y PROPIEDADES DE PROTEÍNAS Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo PARTE I. EXTRACCIÓN Y PRECIPITACIÓN POR SALADO. Las proteínas se purifican mediante
Secuencia de Aminoácidos en las Proteínas. Tema 7 SECUENCIACIÓN PEPTÍDICA. Etapas para el conocimiento de una secuencia peptídica (I)
 Secuencia de Aminoácidos en las Proteínas Es característica para cada proteína Tema 7 SECUENCIACIÓN PEPTÍDICA Es codificada por una secuencia de nucleótidos del DNA Por lo tanto, es una forma de información
Secuencia de Aminoácidos en las Proteínas Es característica para cada proteína Tema 7 SECUENCIACIÓN PEPTÍDICA Es codificada por una secuencia de nucleótidos del DNA Por lo tanto, es una forma de información
Cuantificación y análisis químico de proteínas
 Cuantificación y análisis químico de proteínas Enlace peptídico R 1 R 2 Secuencia: AlaGluGlyLys o AEGK Puentes disulfuro COO- COO- COO- NH 2 NH 2 NH 2 Modificaciones Post-traduccionales I Análisis químico
Cuantificación y análisis químico de proteínas Enlace peptídico R 1 R 2 Secuencia: AlaGluGlyLys o AEGK Puentes disulfuro COO- COO- COO- NH 2 NH 2 NH 2 Modificaciones Post-traduccionales I Análisis químico
ELECTROFORESIS. Electroforesis en gel de poliacrilamida: en condiciones nativas (PAGE) o en presencia de SDS (SDS-PAGE).
 ELECTROFORESIS. V = E z / f v: velocidad de migración de la molécula en un campo eléctrico. E: intensidad de campo eléctrico, z: carga neta de la proteína. f = 6πηr es el coeficiente friccional. η es la
ELECTROFORESIS. V = E z / f v: velocidad de migración de la molécula en un campo eléctrico. E: intensidad de campo eléctrico, z: carga neta de la proteína. f = 6πηr es el coeficiente friccional. η es la
CROMATOGRAFÍA. En la siguiente tabla se observan diferentes fases móviles y fases estacionarias en diferentes técnicas de cromatografía:
 CROMATOGRAFÍA La palabra cromatografía significa gráfica de colores y fue diseñada por Michael Tswett en el 1903. Tswett llevó a cabo una extracción de una mezcla de pigmentos de hojas verdes y luego pasó
CROMATOGRAFÍA La palabra cromatografía significa gráfica de colores y fue diseñada por Michael Tswett en el 1903. Tswett llevó a cabo una extracción de una mezcla de pigmentos de hojas verdes y luego pasó
PROTEÓMICA Una herramienta o una utopía?
 PROTEÓMICA Una herramienta o una utopía? Blanca Gómez Guerrero Departamento de Cereales Laboratorio Tecnológico del Uruguay Contenido Que es la proteómica? Metodologías utilizadas Aplicaciones en el área
PROTEÓMICA Una herramienta o una utopía? Blanca Gómez Guerrero Departamento de Cereales Laboratorio Tecnológico del Uruguay Contenido Que es la proteómica? Metodologías utilizadas Aplicaciones en el área
Modificaciones de Proteínas
 Modificaciones de Proteínas Bibliografía: M. en C. Elva Carolina Chávez Hernández Alberts Molecular Biology of the Cell. 4a Ed disponible online. (2002, 2008). Lehninger Principles of Biochemistry 4ª,
Modificaciones de Proteínas Bibliografía: M. en C. Elva Carolina Chávez Hernández Alberts Molecular Biology of the Cell. 4a Ed disponible online. (2002, 2008). Lehninger Principles of Biochemistry 4ª,
Espectrometría de Masas. Instrumentación y métodos de ionización. Preguntas y ejercicios.
 Espectrometría de Masas. Instrumentación y métodos de ionización. Preguntas y ejercicios. 1- Construya un diagrama de bloques donde especifique los principales componentes de un espectrómetro de masas
Espectrometría de Masas. Instrumentación y métodos de ionización. Preguntas y ejercicios. 1- Construya un diagrama de bloques donde especifique los principales componentes de un espectrómetro de masas
Analizadores en EM: tiempo de vuelo (TOF), cuadrupolo (Q), trampa iónica (IT; TQ) Segundo Cuatrimestre de 2010
 Rosa Erra-Balsells Analizadores en EM: tiempo de vuelo (TOF), cuadrupolo (Q), trampa iónica (IT; TQ) Segundo Cuatrimestre de 2010 V L zv = mv 2 /2 mv/r = zb m/z = B 2 r 2 /2V (EB) A Two Dimensional Quadrupole
Rosa Erra-Balsells Analizadores en EM: tiempo de vuelo (TOF), cuadrupolo (Q), trampa iónica (IT; TQ) Segundo Cuatrimestre de 2010 V L zv = mv 2 /2 mv/r = zb m/z = B 2 r 2 /2V (EB) A Two Dimensional Quadrupole
CROMATOGRAFIA DE GASES
 CROMATOGRAFIA DE GASES Prof. Jorge Mendoza C. Dpto. Química Inorgánica y Analítica Definición: La cromatografía es un método de separación física en la cual los componentes aser separados son distribuidos
CROMATOGRAFIA DE GASES Prof. Jorge Mendoza C. Dpto. Química Inorgánica y Analítica Definición: La cromatografía es un método de separación física en la cual los componentes aser separados son distribuidos
Electroforesis de proteínas en geles de poliacrilamida
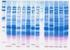 Electroforesis de proteínas en geles de poliacrilamida Electroforesis de proteínas en geles de poliacrilamida: Introducción y técnica básica La electroforesis de proteínas en geles con una matriz de poliacrilamida,
Electroforesis de proteínas en geles de poliacrilamida Electroforesis de proteínas en geles de poliacrilamida: Introducción y técnica básica La electroforesis de proteínas en geles con una matriz de poliacrilamida,
Transcriptomics and Proteomics
 Transcriptomics and Proteomics Tipificación de la expresión: permite el estudio de la funcion de los genes Tipificación del RNA depende de la hibridizacion de los ácidos nucleicos Northern blot Producir
Transcriptomics and Proteomics Tipificación de la expresión: permite el estudio de la funcion de los genes Tipificación del RNA depende de la hibridizacion de los ácidos nucleicos Northern blot Producir
ESPECTROMETRÍA DE MASAS DE TIPO ELECTROSPRAY (ES/MS)
 ESPECTROMETRÍA DE MASAS DE TIPO ELECTROSPRAY (ES/MS) La ionización mediante electrospray se basa en una elegante idea propuesta por Dole en los años 60 que fue puesta a punto por Fenn et al en 1988, hecho
ESPECTROMETRÍA DE MASAS DE TIPO ELECTROSPRAY (ES/MS) La ionización mediante electrospray se basa en una elegante idea propuesta por Dole en los años 60 que fue puesta a punto por Fenn et al en 1988, hecho
CROMATOGRAFÍA LÍQUIDA DE ALTA RESOLUCIÓN
 CROMATOGRAFÍA LÍQUIDA DE ALTA RESOLUCIÓN Curso: Química Analítica II Loreto Ascar 2012 Esquema de las bases de la separación en cromatografía de: (a)absorción, (b) partición, (c) Intercambio iónico y (d)
CROMATOGRAFÍA LÍQUIDA DE ALTA RESOLUCIÓN Curso: Química Analítica II Loreto Ascar 2012 Esquema de las bases de la separación en cromatografía de: (a)absorción, (b) partición, (c) Intercambio iónico y (d)
GENÉTICA MOLECULAR. Unidad 1: Introducción a la genética molecular
 GENÉTICA MOLECULAR CONTENIDOS CONCEPTUALES COMPETENCIAS Unidad 1: Introducción a la genética molecular Concepto de genética molecular y aplicaciones a diferentes ramas de la ciencia. Dogma central de la
GENÉTICA MOLECULAR CONTENIDOS CONCEPTUALES COMPETENCIAS Unidad 1: Introducción a la genética molecular Concepto de genética molecular y aplicaciones a diferentes ramas de la ciencia. Dogma central de la
MEDIDAS DE POTENCIAL ZETA EN LA SERIE ZETASIZER NANO. Enrique Mazarrón
 MEDIDAS DE POTENCIAL ZETA EN LA SERIE ZETASIZER NANO Enrique Mazarrón Medida de Potencial Zeta Usando Electroforesis Doppler con Láser Es una técnica usada para medir el movimiento de las partículas cargadas
MEDIDAS DE POTENCIAL ZETA EN LA SERIE ZETASIZER NANO Enrique Mazarrón Medida de Potencial Zeta Usando Electroforesis Doppler con Láser Es una técnica usada para medir el movimiento de las partículas cargadas
Metodos de purificación
 Metodos de purificación Basado en las siguientes propiedades proteicas: Tamaño Solubilidad Carga Adsorción Afinidad CAROLINA VITA Característica Tamaño Solubidad Polaridad Carga Procedimientos Dialisis
Metodos de purificación Basado en las siguientes propiedades proteicas: Tamaño Solubilidad Carga Adsorción Afinidad CAROLINA VITA Característica Tamaño Solubidad Polaridad Carga Procedimientos Dialisis
Transcripción. - Generalidades - DNA RNA. - Mecanismo. - Modelos Protein. - Moléculas. - Métodos de detección de RNA. Chromosome RNA DNA
 TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
PLANIFICACIÓN DE ASIGNATURA
 PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
Secuenciación del ARN
 Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
CENTRO NACIONAL DE INVESTIGACIONES ONCOLÓGICAS RED NACIONAL DE CENTROS DE CÁNCER PROGRAMA DE GENÓMICA
 CENTRO NACIONAL DE INVESTIGACIONES ONCOLÓGICAS RED NACIONAL DE CENTROS DE CÁNCER PROGRAMA DE GENÓMICA CATÁLOGO DE OFERTA DE SERVICIOS GENÓMICOS I. INFORMACIÓN GENERAL: NOMBRE DE UNIDAD: Unidad de Genómica
CENTRO NACIONAL DE INVESTIGACIONES ONCOLÓGICAS RED NACIONAL DE CENTROS DE CÁNCER PROGRAMA DE GENÓMICA CATÁLOGO DE OFERTA DE SERVICIOS GENÓMICOS I. INFORMACIÓN GENERAL: NOMBRE DE UNIDAD: Unidad de Genómica
AMINOÁCIDOS SE PUEDEN UNIR POR ENLACES PEPTÍDICOS
 Tema 4. Proteínas: funciones biológicas y estructura primaria. Enlace peptídico. Péptidos y proteínas. Diversidad de funciones biológicas. Niveles de organización estructural de las proteínas. Separación
Tema 4. Proteínas: funciones biológicas y estructura primaria. Enlace peptídico. Péptidos y proteínas. Diversidad de funciones biológicas. Niveles de organización estructural de las proteínas. Separación
PROGRAMA DE ESTUDIO. 2. CICLO O AREA: División de Ciencias e Ingeniería/Ingeniería Ambiental.
 PROGRAMA DE ESTUDIO 1. NOMBRE DE LA ASIGNATURA: BIOQUÍMICA. 2. CICLO O AREA: División de Ciencias e Ingeniería/Ingeniería Ambiental. 3. CLAVE: 4. SERIACION: Química Orgánica. 5. H.T.S. H.P.S. T.H.S. C.
PROGRAMA DE ESTUDIO 1. NOMBRE DE LA ASIGNATURA: BIOQUÍMICA. 2. CICLO O AREA: División de Ciencias e Ingeniería/Ingeniería Ambiental. 3. CLAVE: 4. SERIACION: Química Orgánica. 5. H.T.S. H.P.S. T.H.S. C.
Composición química de los seres vivos
 Composición química de los seres vivos Elementos químicos de la materia viva BIOELEMENTOS Se clasifican por su abundancia En proporcion superior al 96% PRIMARIOS En una proporción aprox. 3,9% SECUNDARIOS
Composición química de los seres vivos Elementos químicos de la materia viva BIOELEMENTOS Se clasifican por su abundancia En proporcion superior al 96% PRIMARIOS En una proporción aprox. 3,9% SECUNDARIOS
Espectrometría de masas Instrumentación y aplicaciones
 Clase 01 Espectroscopía de Compuestos Orgánicos MALDI-MS ESI-MS Espectrometría de masas Instrumentación y aplicaciones Espectrometría de Masa Espectrometría de Masa (MS: Mass spectrometry) Espectroscopía
Clase 01 Espectroscopía de Compuestos Orgánicos MALDI-MS ESI-MS Espectrometría de masas Instrumentación y aplicaciones Espectrometría de Masa Espectrometría de Masa (MS: Mass spectrometry) Espectroscopía
Protocolo de Análisis por Espectrometría de Masas de Alta Resolución. Código LEM-PR-01 Fecha 15/Octubre/2015 Versión 1
 OBJETIVO. Establecer los mecanismos y requisitos necesarios para realizar análisis de muestras tanto externas como internas en el laboratorio de espectrometría de masas. ALCANCE. Aplica al Laboratorio
OBJETIVO. Establecer los mecanismos y requisitos necesarios para realizar análisis de muestras tanto externas como internas en el laboratorio de espectrometría de masas. ALCANCE. Aplica al Laboratorio
Análisis Funcional Orgánico. Cromatografía Líquida de Alta Resolución (HPLC/CLAR)
 Análisis Funcional Orgánico Cromatografía Líquida de Alta Resolución (HPLC/CLAR) PARTE A Introducción Técnicas instrumentales cromatográficas 1. Gaseosa líquida (CGL) o sólida (CGS) 2. Líquida de Alta
Análisis Funcional Orgánico Cromatografía Líquida de Alta Resolución (HPLC/CLAR) PARTE A Introducción Técnicas instrumentales cromatográficas 1. Gaseosa líquida (CGL) o sólida (CGS) 2. Líquida de Alta
LA PROTEÓMICA, UN RETO CONSTANTE EN BIOMEDICINA
 LA PROTEÓMICA, UN RETO CONSTANTE EN BIOMEDICINA Mª Luisa Hernáez Sánchez Responsable Técnico Unidad de Proteómica UCM-PCM Concha Gil García Catedrática de la UCM. Responsable Científico Unidad de Proteómica
LA PROTEÓMICA, UN RETO CONSTANTE EN BIOMEDICINA Mª Luisa Hernáez Sánchez Responsable Técnico Unidad de Proteómica UCM-PCM Concha Gil García Catedrática de la UCM. Responsable Científico Unidad de Proteómica
Unidad de Espectrometría de masas. Servicios Científico-Técnicos. Universidad de Oviedo
 Unidad de Espectrometría de masas Servicios Científico-Técnicos. Universidad de Oviedo Ubicación Edificio Severo Ochoa. Campus del Cristo (plantas baja y 1ª) Servicios Científico-Técnicos: Unidad de Espectrometría
Unidad de Espectrometría de masas Servicios Científico-Técnicos. Universidad de Oviedo Ubicación Edificio Severo Ochoa. Campus del Cristo (plantas baja y 1ª) Servicios Científico-Técnicos: Unidad de Espectrometría
TRANSPORTE A TRAVÉS DE LA MEMBRANA CELULAR
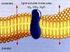 TRANSPORTE A TRAVÉS DE LA MEMBRANA CELULAR Introducción Compartimientos Acuosos separados por Barreras Biológicas Organelos selectivos, autocatalíticos, autoreproductivos, competitivos, adaptativos...
TRANSPORTE A TRAVÉS DE LA MEMBRANA CELULAR Introducción Compartimientos Acuosos separados por Barreras Biológicas Organelos selectivos, autocatalíticos, autoreproductivos, competitivos, adaptativos...
Material elaborado por F. Agius, O. Borsani, P.Díaz, S. Gonnet, P. Irisarri, F. Milnitsky y J. Monza. Bioquímica. Facultad de Agronomía.
 AMINOACIDOS Las proteínas de bacterias, hongos, plantas y animales están constituidas a partir de las mismas 20 unidades o monómeros: los á-aminoácidos. Los aminoácidos tienen un grupo amino y un grupo
AMINOACIDOS Las proteínas de bacterias, hongos, plantas y animales están constituidas a partir de las mismas 20 unidades o monómeros: los á-aminoácidos. Los aminoácidos tienen un grupo amino y un grupo
FÍSICA Y QUÍMICA. 3º ESO. IES TEROR. EXAMEN FINAL
 NOMBRE Y APELLIDOS: 11.06.2013 1.- El amoníaco se descompone dando gas hidrógeno (molecular) y gas nitrógeno (molecular). Calcula cuántos gramos de amoníaco se deben descomponer para obtener 7 gramos de
NOMBRE Y APELLIDOS: 11.06.2013 1.- El amoníaco se descompone dando gas hidrógeno (molecular) y gas nitrógeno (molecular). Calcula cuántos gramos de amoníaco se deben descomponer para obtener 7 gramos de
Theory Espanol (Colombia) El Gran Colisionador de Hadrones (Large Hadron Collider) (10 puntos)
 Q3-1 El Gran Colisionador de Hadrones (Large Hadron Collider) (10 puntos) Por favor asegúrese de leer las instrucciones generales dentro del sobre adjunto antes de comenzar a resolver este problema. En
Q3-1 El Gran Colisionador de Hadrones (Large Hadron Collider) (10 puntos) Por favor asegúrese de leer las instrucciones generales dentro del sobre adjunto antes de comenzar a resolver este problema. En
Requisitos de las muestras y condiciones de análisis
 Requisitos de las muestras y condiciones de análisis Página 1 INTRODUCCION A continuación se indican los requisitos generales que han de cumplir las muestras para poder ser analizadas en el Servicio de
Requisitos de las muestras y condiciones de análisis Página 1 INTRODUCCION A continuación se indican los requisitos generales que han de cumplir las muestras para poder ser analizadas en el Servicio de
La síntesis de proteínas
 La síntesis de proteínas La Transcripción La información para fabricar todas las proteínas está almacenada en las moléculas de ADN de los cromosomas. La sucesión de bases en las moléculas de ADN es un
La síntesis de proteínas La Transcripción La información para fabricar todas las proteínas está almacenada en las moléculas de ADN de los cromosomas. La sucesión de bases en las moléculas de ADN es un
Espectrometría de Masa
 Espectrometría de Masa (ESI-IT) Aplicación al estudio de proteínas Lanais-Pro Lic. Susana Linskens Abril 2010 Esquema básico de un MS Medida de Masas Moleculares con un error de 0.001 % - 0.01 % Ionización
Espectrometría de Masa (ESI-IT) Aplicación al estudio de proteínas Lanais-Pro Lic. Susana Linskens Abril 2010 Esquema básico de un MS Medida de Masas Moleculares con un error de 0.001 % - 0.01 % Ionización
CLASIFICACIÓN DE LA MATERIA
 1. Clasificación de la materia por su aspecto CLASIFICACIÓN DE LA MATERIA La materia homogénea es la que presenta un aspecto uniforme, en la cual no se pueden distinguir a simple vista sus componentes.
1. Clasificación de la materia por su aspecto CLASIFICACIÓN DE LA MATERIA La materia homogénea es la que presenta un aspecto uniforme, en la cual no se pueden distinguir a simple vista sus componentes.
ELECTROFORESIS Fundamento.
 ELECTROFORESIS La electroforesis es un método de laboratorio en el que se utiliza una corriente eléctrica controlada con la finalidad de separar biomoleculas según su tamaño y carga eléctrica a través
ELECTROFORESIS La electroforesis es un método de laboratorio en el que se utiliza una corriente eléctrica controlada con la finalidad de separar biomoleculas según su tamaño y carga eléctrica a través
EL CONTROL DE LAS ACTIVIDADES CELULARES
 EL CONTROL DE LAS ACTIVIDADES CELULARES LAS REACCIONES CELULARES BÁSICAS Todas las células llevan a cabo funciones vitales: Ingestión de nutrientes Eliminación de desperdicios Crecimiento Reproducción
EL CONTROL DE LAS ACTIVIDADES CELULARES LAS REACCIONES CELULARES BÁSICAS Todas las células llevan a cabo funciones vitales: Ingestión de nutrientes Eliminación de desperdicios Crecimiento Reproducción
Bioelementos y Biomoléculas 1-Moléculas inorganicas : Agua
 Bioelementos y Biomoléculas 1-Moléculas inorganicas : Agua Técnico Professor: Verónica en masoterapia Pantoja. Lic. MSP. Objetivos Reconocer los bioelementos en los seres vivos Reconocer el agua como biomolécula
Bioelementos y Biomoléculas 1-Moléculas inorganicas : Agua Técnico Professor: Verónica en masoterapia Pantoja. Lic. MSP. Objetivos Reconocer los bioelementos en los seres vivos Reconocer el agua como biomolécula
Proteínas y Ácidos Nucleicos
 Proteínas y Ácidos Nucleicos Mapa conceptual Biomoléculas. Biomoléculas inorgánicas: Moléculas que no presentan carbono en su estructura. Biomoléculas orgánicas: Moléculas que presentan carbono en su estructura.
Proteínas y Ácidos Nucleicos Mapa conceptual Biomoléculas. Biomoléculas inorgánicas: Moléculas que no presentan carbono en su estructura. Biomoléculas orgánicas: Moléculas que presentan carbono en su estructura.
CROMATOGRAFIA LIQUIDA DE ALTA RESOLUCION
 CROMATOGRAFIA LIQUIDA DE ALTA RESOLUCION Componentes de un Cromatógrafo Suministro de Solvente Procesador de Muestras Columna Detector Procesador de Datos Ubicación GASES I.C.-H.P.L.C. L.C.-G.P.C. 50 500
CROMATOGRAFIA LIQUIDA DE ALTA RESOLUCION Componentes de un Cromatógrafo Suministro de Solvente Procesador de Muestras Columna Detector Procesador de Datos Ubicación GASES I.C.-H.P.L.C. L.C.-G.P.C. 50 500
DIPLOMADO DE CROMATOGRAFIA
 DIPLOMADO DE CROMATOGRAFIA Objetivos 1.-Brindar elementos teórico-prácticos de la cromatografía partiendo desde los principios fundamentales hasta la aplicación de las técnicas cualitativas y cuantitativas.
DIPLOMADO DE CROMATOGRAFIA Objetivos 1.-Brindar elementos teórico-prácticos de la cromatografía partiendo desde los principios fundamentales hasta la aplicación de las técnicas cualitativas y cuantitativas.
TÉCNICAS DE MUESTREO, ANÁLISIS E INTERPRETACIÓN. Ingeniería Ambiental
 TÉCNICAS DE MUESTREO, ANÁLISIS E INTERPRETACIÓN Ingeniería Ambiental Problema Elección del método analítico Toma de muestra Tratamiento de la muestra Proceso de medida Tratamiento de los datos Valoración
TÉCNICAS DE MUESTREO, ANÁLISIS E INTERPRETACIÓN Ingeniería Ambiental Problema Elección del método analítico Toma de muestra Tratamiento de la muestra Proceso de medida Tratamiento de los datos Valoración
Proyecto de Fin de Cursos
 Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS
 Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS DICIEMBRE 2006 Tratamiento de Imágenes por Computadora Proyecto
Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS DICIEMBRE 2006 Tratamiento de Imágenes por Computadora Proyecto
METODOLOGÍA DE EXTRACCIÓN Y PURIFICACIÓN DE INHIBIDORES DE PROTEASAS EN Opuntia streptacantha
 METODOLOGÍA DE EXTRACCIÓN Y PURIFICACIÓN DE INHIBIDORES DE PROTEASAS EN Opuntia streptacantha RESUMEN Estudiante: Jannet Alejandra Vargas Sánchez Instituto Tecnológico de Roque jannettitajm@hotmail.com
METODOLOGÍA DE EXTRACCIÓN Y PURIFICACIÓN DE INHIBIDORES DE PROTEASAS EN Opuntia streptacantha RESUMEN Estudiante: Jannet Alejandra Vargas Sánchez Instituto Tecnológico de Roque jannettitajm@hotmail.com
Biosíntesis de Ácidos nucleicos (ARN) y Proteínas
 Biosíntesis de Ácidos nucleicos (ARN) y Proteínas Núcleo celular Dogma central de la Biología Molecular Repasando los Ácidos nucleicos. Empaquetamiento de ADN 46 cromosomas en las células somáticas del
Biosíntesis de Ácidos nucleicos (ARN) y Proteínas Núcleo celular Dogma central de la Biología Molecular Repasando los Ácidos nucleicos. Empaquetamiento de ADN 46 cromosomas en las células somáticas del
Niveles de organización biológica. La composición de los seres vivos
 Niveles de organización biológica. La composición de los seres vivos Imagen de Fondo bajo licencia Creative Commons, autor: René Ehrtardt. Animación ADN de dominio público, fuente:wikipedia Con este tema
Niveles de organización biológica. La composición de los seres vivos Imagen de Fondo bajo licencia Creative Commons, autor: René Ehrtardt. Animación ADN de dominio público, fuente:wikipedia Con este tema
6 APENDICE. A. Curvas de Calibración
 6 APENDICE A. Curvas de Calibración Las muestras colectadas en las hidrólisis contenían básicamente carbohidratos como, glucosa, xilosa y arabinosa, entre otros. Se realizaron curvas de calibración para
6 APENDICE A. Curvas de Calibración Las muestras colectadas en las hidrólisis contenían básicamente carbohidratos como, glucosa, xilosa y arabinosa, entre otros. Se realizaron curvas de calibración para
FÍSICA Y QUÍMICA 3º ESO. OBJETIVOS, CONTENIDOS Y CRITERIOS DE EVALUACIÓN 1ª Evaluación: Unidad 1. La medida y el método científico.
 FÍSICA Y QUÍMICA 3º ESO. OBJETIVOS, CONTENIDOS Y CRITERIOS DE EVALUACIÓN 1ª Evaluación: Unidad 1. La medida y el método científico. OBJETIVOS 1. Reconocer las etapas del trabajo científico y elaborar informes
FÍSICA Y QUÍMICA 3º ESO. OBJETIVOS, CONTENIDOS Y CRITERIOS DE EVALUACIÓN 1ª Evaluación: Unidad 1. La medida y el método científico. OBJETIVOS 1. Reconocer las etapas del trabajo científico y elaborar informes
PROTEINAS RECOMBINANTES DISEÑO DE CLONES SISTEMAS DE EXPRESION CONTROL DE CALIDAD
 PROTEINAS RECOMBINANTES DISEÑO DE CLONES SISTEMAS DE EXPRESION CONTROL DE CALIDAD I Congreso de Biotecnología Merck Junio 2009 INDUSTRIA BIOTECNOLOGICA Mercado farmaceutico mundial 2008: US$ 750 Billion
PROTEINAS RECOMBINANTES DISEÑO DE CLONES SISTEMAS DE EXPRESION CONTROL DE CALIDAD I Congreso de Biotecnología Merck Junio 2009 INDUSTRIA BIOTECNOLOGICA Mercado farmaceutico mundial 2008: US$ 750 Billion
MÉTODOS CROMATOGRÁFICOS: 1. Exclusión molecular 2. Intercambio iónico 3. Afinidad SSN
 MÉTODOS CROMATOGRÁFICOS: 1. Exclusión molecular 2. Intercambio iónico 3. Afinidad SSN Propiedades de una proteína que son útiles para su purificación por cromatografía Peso molecular Carga iónica Hidrofobicidad
MÉTODOS CROMATOGRÁFICOS: 1. Exclusión molecular 2. Intercambio iónico 3. Afinidad SSN Propiedades de una proteína que son útiles para su purificación por cromatografía Peso molecular Carga iónica Hidrofobicidad
MASS ESPECTROMETRY DR. SERGIO ALEMANO. Como procesar el material vegetal? Identificación y cuantificación de fitohormonas // -UNRC
 MASS ESPECTRMETRY R. SERGI ALEMAN Como procesar el material vegetal? Identificación y cuantificación de fitohormonas // -UNRC 2 1 LFV UNRC 2013 Cromatografía 3 Separación basada en polaridad Historia de
MASS ESPECTRMETRY R. SERGI ALEMAN Como procesar el material vegetal? Identificación y cuantificación de fitohormonas // -UNRC 2 1 LFV UNRC 2013 Cromatografía 3 Separación basada en polaridad Historia de
Colegio San Lorenzo - Copiapó - Región de Atacama Per Laborem ad Lucem
 TEMARIO EXAMENES QUIMICA 2012 7º BASICO Descubrimiento del átomo: Quién lo descubrió y su significado Estructura atómica: Partes del átomo, características del núcleo y la corteza, cálculo del protón,
TEMARIO EXAMENES QUIMICA 2012 7º BASICO Descubrimiento del átomo: Quién lo descubrió y su significado Estructura atómica: Partes del átomo, características del núcleo y la corteza, cálculo del protón,
1.5 Métodos de separación de mezclas Decantación, filtración, extracción, cristalización, destilación, cromatografía.
 1.5 Métodos de separación de mezclas Decantación, filtración, extracción, cristalización, destilación, cromatografía. 1 Para tomar apuntes MÉTODO DE SEPARACIÓN PROPIEDAD(ES) EN LAS QUE SE BASA MEZCLAS
1.5 Métodos de separación de mezclas Decantación, filtración, extracción, cristalización, destilación, cromatografía. 1 Para tomar apuntes MÉTODO DE SEPARACIÓN PROPIEDAD(ES) EN LAS QUE SE BASA MEZCLAS
Las funciones de los seres vivos. Fisiología celular
 Las funciones de los seres vivos Fisiología celular La célula, como unidad funcional de los seres vivos, está capacitada para llevar a cabo las funciones características de éstos: nutrición, reproducción
Las funciones de los seres vivos Fisiología celular La célula, como unidad funcional de los seres vivos, está capacitada para llevar a cabo las funciones características de éstos: nutrición, reproducción
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
La Célula. Membrana celular. UNIVERSIDAD NACIONAL DE GRAL. SAN MARTÍN ECyT BIOLOGÍA. Biología I
 La Célula. Membrana celular UNIVERSIDAD NACIONAL DE GRAL. SAN MARTÍN ECyT BIOLOGÍA Biología I Los seres vivos están formados por células Organismos unicelulares: bacterias, plantas (algas unicelulares),
La Célula. Membrana celular UNIVERSIDAD NACIONAL DE GRAL. SAN MARTÍN ECyT BIOLOGÍA Biología I Los seres vivos están formados por células Organismos unicelulares: bacterias, plantas (algas unicelulares),
DEPARTAMENTO DE INGENIERIA QUÍMICA CÁTEDRA DE FISICOQUÍMICA TRABAJO PRÁCTICO DE LABORATORIO Nº 4
 Universidad Tecnológica Nacional Facultad Regional La Plata DEPARTAMENTO DE INGENIERIA QUÍMICA CÁTEDRA DE FISICOQUÍMICA TRABAJO PRÁCTICO DE LABORATORIO Nº 4 Descenso crioscópico Objeto de la experiencia:
Universidad Tecnológica Nacional Facultad Regional La Plata DEPARTAMENTO DE INGENIERIA QUÍMICA CÁTEDRA DE FISICOQUÍMICA TRABAJO PRÁCTICO DE LABORATORIO Nº 4 Descenso crioscópico Objeto de la experiencia:
II.- Espectrometría de masas
 II.- Espectrometría de masas Generalidades de espectrometría de masas La espectrometría de masas en una técnica analítica que nos permite identificar y determinar estructuras, así como analizar trazas
II.- Espectrometría de masas Generalidades de espectrometría de masas La espectrometría de masas en una técnica analítica que nos permite identificar y determinar estructuras, así como analizar trazas
Introducción n a la. Algunas aplicaciones de la Espectrometría de Masa en biología y biomedicina
 Introducción n a la Espectrometría a de Masa Algunas aplicaciones de la Espectrometría de Masa en biología y biomedicina Espectrometría de Masa La Espectrometría de Masa es una técnica que permite determinar
Introducción n a la Espectrometría a de Masa Algunas aplicaciones de la Espectrometría de Masa en biología y biomedicina Espectrometría de Masa La Espectrometría de Masa es una técnica que permite determinar
Bloque II: Las moléculas forman estructuras: Célula
 Bloque II: Las moléculas forman estructuras: Célula 1. a. Indique dos tipos de moléculas presentes en todas las membranas celulares. Indique una diferencia entre la membrana plasmática y otras membranas
Bloque II: Las moléculas forman estructuras: Célula 1. a. Indique dos tipos de moléculas presentes en todas las membranas celulares. Indique una diferencia entre la membrana plasmática y otras membranas
Componentes químicos de
 Componentes químicos de las célulasc Componentes químicos Las células están compuestas por una enorme cantidad y variedad de moléculas que pueden clasificarse en: Componentes inorgánicos Componentes orgánicos
Componentes químicos de las célulasc Componentes químicos Las células están compuestas por una enorme cantidad y variedad de moléculas que pueden clasificarse en: Componentes inorgánicos Componentes orgánicos
Espectrometría de Masa
 Espectrometría de Masa ESI-IT Identificación de proteínas Lanais-Pro Marzo 2009 1 Esquema básico de un MS Medida de Masas Moleculares con un error de ± 0.01 % Ionización Analizador Detector 2 Ionización
Espectrometría de Masa ESI-IT Identificación de proteínas Lanais-Pro Marzo 2009 1 Esquema básico de un MS Medida de Masas Moleculares con un error de ± 0.01 % Ionización Analizador Detector 2 Ionización
Repaso: Química celular (biomoléculas)
 Repaso: Química celular (biomoléculas) Hay 4 tipos principales de biomoléculas: 1) glúcidos o hidratos de carbono, 2) lípidos o grasas, 3) proteínas y 4) ácidos nucleicos. Las biomoléculas más grandes,
Repaso: Química celular (biomoléculas) Hay 4 tipos principales de biomoléculas: 1) glúcidos o hidratos de carbono, 2) lípidos o grasas, 3) proteínas y 4) ácidos nucleicos. Las biomoléculas más grandes,
Niveles de Organización Conceptos, átomos y enlaces
 Niveles de Organización Conceptos, átomos y enlaces Cómo se organiza la materia viva? NIVELES DE ORGANIZACIÓN ÁTOMO MOLÉCULA (ELEMENTO- COMPUESTO) MACROMOLÉCULA CÉLULA TEJIDO ÓRGANO SISTEMA ORGANISMO MULTICELULAR
Niveles de Organización Conceptos, átomos y enlaces Cómo se organiza la materia viva? NIVELES DE ORGANIZACIÓN ÁTOMO MOLÉCULA (ELEMENTO- COMPUESTO) MACROMOLÉCULA CÉLULA TEJIDO ÓRGANO SISTEMA ORGANISMO MULTICELULAR
Química Biológica TP 1: ESPECTROFOTOMETRIA.
 TP 1: ESPECTROFOTOMETRIA. Introducción Al observar una solución acuosa de un colorante a trasluz, observamos una leve coloración, la cual se debe a la interacción entre las moléculas del colorante y la
TP 1: ESPECTROFOTOMETRIA. Introducción Al observar una solución acuosa de un colorante a trasluz, observamos una leve coloración, la cual se debe a la interacción entre las moléculas del colorante y la
Perfiles metabólicos por cromatografía gaseosa acoplada a espectrometría de masas (GC-MS) en plantas. Dra. Mariana G. López
 Perfiles metabólicos por cromatografía gaseosa acoplada a espectrometría de masas (GC-MS) en plantas Dra. Mariana G. López mglopez@cnia.inta.gov.ar PROTOCOLO PARA LA OBTENCIÓN DE PERFILES METABOLICOS EN
Perfiles metabólicos por cromatografía gaseosa acoplada a espectrometría de masas (GC-MS) en plantas Dra. Mariana G. López mglopez@cnia.inta.gov.ar PROTOCOLO PARA LA OBTENCIÓN DE PERFILES METABOLICOS EN
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
Una mezcla es un compuesto formado por varias sustancias con distintas propiedades
 COMPOSICIÓN DE LA MATERIA Mezclas homogéneas y heterogéneas Una mezcla es un compuesto formado por varias sustancias con distintas propiedades Algunos sistemas materiales como la leche a simple vista parecen
COMPOSICIÓN DE LA MATERIA Mezclas homogéneas y heterogéneas Una mezcla es un compuesto formado por varias sustancias con distintas propiedades Algunos sistemas materiales como la leche a simple vista parecen
Mediante HPLC podemos analizar un rango más amplio de compuestos en comparación con GC.
 CURSO TEÓRICO-PRÁCTICO DE ESPECTROMETRÍA DE MASAS Dra. Pilar Blasco Dr. Pablo Candela CURSO FORMACIÓN ESPECÍFICA PAS - 2012 HPLC/MS Por qué acoplar un HPLC y un MS? Mediante HPLC podemos analizar un rango
CURSO TEÓRICO-PRÁCTICO DE ESPECTROMETRÍA DE MASAS Dra. Pilar Blasco Dr. Pablo Candela CURSO FORMACIÓN ESPECÍFICA PAS - 2012 HPLC/MS Por qué acoplar un HPLC y un MS? Mediante HPLC podemos analizar un rango
Theory Spanish (Costa Rica) El Gran Colisionador de Hadrones (Large Hadron Collider LHC) (10 puntos)
 Q3-1 El Gran Colisionador de Hadrones (Large Hadron Collider LHC) (10 puntos) Por favor asegúrese de leer las instrucciones generales del sobre adjunto antes de comenzar a resolver este problema. En este
Q3-1 El Gran Colisionador de Hadrones (Large Hadron Collider LHC) (10 puntos) Por favor asegúrese de leer las instrucciones generales del sobre adjunto antes de comenzar a resolver este problema. En este
Compartimentos intracelulares y distribución de proteínas
 Compartimentos intracelulares y distribución de proteínas 1 2 3 regulado 4 Péptido y zona de señal 5 6 7 8 9 Las proteínas con señales de localización nuclear vuelven a entrar al núcleo 10 11 Retículo
Compartimentos intracelulares y distribución de proteínas 1 2 3 regulado 4 Péptido y zona de señal 5 6 7 8 9 Las proteínas con señales de localización nuclear vuelven a entrar al núcleo 10 11 Retículo
Proteómica - Identificación de proteínas y fosfoproteómica (Javier Muñoz, PhD clase, Nuria Ibarz Ferrer, nibarz@cnio.es, práctica).
 Proteómica - Identificación de proteínas y fosfoproteómica (Javier Muñoz, PhD clase, Nuria Ibarz Ferrer, nibarz@cnio.es, práctica). Por Mauricio Lema Medina 02.2013 Nota de quien resume Pocas clases del
Proteómica - Identificación de proteínas y fosfoproteómica (Javier Muñoz, PhD clase, Nuria Ibarz Ferrer, nibarz@cnio.es, práctica). Por Mauricio Lema Medina 02.2013 Nota de quien resume Pocas clases del
Biotecnología - Bioprocesos II. INTRODUCCION A LOS PROCESOS BIOTECNOLOGICOS Separación y purificación en Biotecnología ( Downstream processing )
 Biotecnología - Bioprocesos II INTRODUCCION A LOS PROCESOS BIOTECNOLOGICOS Separación y purificación en Biotecnología ( Downstream processing ) La comercialización de nuevos productos obtenidos a través
Biotecnología - Bioprocesos II INTRODUCCION A LOS PROCESOS BIOTECNOLOGICOS Separación y purificación en Biotecnología ( Downstream processing ) La comercialización de nuevos productos obtenidos a través
ELECTROFORESIS GENERALIDADES Y APLICACIONES CLINICAS
 ELECTROFORESIS GENERALIDADES Y APLICACIONES CLINICAS Marcelo Castillo Navarrrete, T.M. MsCs (C) Facultad de Medicina Carrera Tecnología Médica 18 de Noviembre de 2004 Fundamentos de Electroforesis Alta
ELECTROFORESIS GENERALIDADES Y APLICACIONES CLINICAS Marcelo Castillo Navarrrete, T.M. MsCs (C) Facultad de Medicina Carrera Tecnología Médica 18 de Noviembre de 2004 Fundamentos de Electroforesis Alta
UNIDAD 1 La materia y sus cambios
 UNIDAD 1 La materia y sus cambios Tema 1.2 Composición de la materia. Sustancias y mezclas. Mezclas homogéneas, heterogéneas y coloides. Introducción a las disoluciones: no saturadas, saturadas y sobresaturadas.
UNIDAD 1 La materia y sus cambios Tema 1.2 Composición de la materia. Sustancias y mezclas. Mezclas homogéneas, heterogéneas y coloides. Introducción a las disoluciones: no saturadas, saturadas y sobresaturadas.
Universidad Nacional de la Patagonia San Juan Bosco Facultad de Ciencias Naturales Química Biológica Introducción
 Química Biológica TP 4 PROTEINAS,. Introducción Las proteínas son biomoléculas formados por 20 L-α-aminoácidos distintos, unidos linealmente entre sí por enlaces peptídicos, en un número que varía entre
Química Biológica TP 4 PROTEINAS,. Introducción Las proteínas son biomoléculas formados por 20 L-α-aminoácidos distintos, unidos linealmente entre sí por enlaces peptídicos, en un número que varía entre
IV. B. 3. 8. DETERMINACIÓN DE LOS PUENTES DISULFURO DEL QUINTO INTERMEDIARIO DE PLEGAMIENTO DEL PCI, Xe.
 IV. B. 3. 8. DETERMINACIÓN DE LOS PUENTES DISULFURO DEL QUINTO INTERMEDIARIO DE PLEGAMIENTO DEL PCI, Xe. La caracterización estructural por digestión con proteasas de la especie scrambled Xe no se realizó
IV. B. 3. 8. DETERMINACIÓN DE LOS PUENTES DISULFURO DEL QUINTO INTERMEDIARIO DE PLEGAMIENTO DEL PCI, Xe. La caracterización estructural por digestión con proteasas de la especie scrambled Xe no se realizó
TEMA 1 1.CONSIDERACIONES SOBRE LA QUIMICA 1.1 QUE ES LA QUIMICA? 1.2 CLASIFICACIÓN DE LA QUÍMICA 1.3 PROCESOS QUIMICOS: SINTESIS Y ANÁLISIS
 TEMA 1 1.CONSIDERACIONES SOBRE LA QUIMICA 1.1 QUE ES LA QUIMICA? 1.2 CLASIFICACIÓN DE LA QUÍMICA 1.3 PROCESOS QUIMICOS: SINTESIS Y ANÁLISIS 2.CONCEPTOS BASICOS 1.1 ELEMENTOS, ISOTOPOS, U.M.A. Y PESOS ATOMICOS
TEMA 1 1.CONSIDERACIONES SOBRE LA QUIMICA 1.1 QUE ES LA QUIMICA? 1.2 CLASIFICACIÓN DE LA QUÍMICA 1.3 PROCESOS QUIMICOS: SINTESIS Y ANÁLISIS 2.CONCEPTOS BASICOS 1.1 ELEMENTOS, ISOTOPOS, U.M.A. Y PESOS ATOMICOS
UNIVERSIDAD SANTO TOMAS SECCIONAL BUCARAMANGA. División de Ingenierías - Facultad de Química Ambiental
 UNIVERSIDAD SANTO TOMAS SECCIONAL BUCARAMANGA División de Ingeniería Facultad de Química Ambiental Nombre de Asignatura: QUÍMICA INSTRUMENTAL II Àrea: Básicas de Química / Química Analítica Créditos: 4
UNIVERSIDAD SANTO TOMAS SECCIONAL BUCARAMANGA División de Ingeniería Facultad de Química Ambiental Nombre de Asignatura: QUÍMICA INSTRUMENTAL II Àrea: Básicas de Química / Química Analítica Créditos: 4
QUÉ ES LA TEMPERATURA?
 1 QUÉ ES LA TEMPERATURA? Nosotros experimentamos la temperatura todos los días. Cuando estamos en verano, generalmente decimos Hace calor! y en invierno Hace mucho frío!. Los términos que frecuentemente
1 QUÉ ES LA TEMPERATURA? Nosotros experimentamos la temperatura todos los días. Cuando estamos en verano, generalmente decimos Hace calor! y en invierno Hace mucho frío!. Los términos que frecuentemente
INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR
 INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR - Componentes químicos de la célula - Un poco de química 1 Un poco de química El 99% del peso de una célula corresponde a C, H, N y O. La sustancia más abundante
INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR - Componentes químicos de la célula - Un poco de química 1 Un poco de química El 99% del peso de una célula corresponde a C, H, N y O. La sustancia más abundante
Licenciatura Ingeniería Bioquímica Industrial MANUAL DE PRÁCTICAS DEL LABORATORIO DE INGENIERÍA ENZIMÁTICA
 División de Ciencias Biológicas y de la Salud Departamento de Biotecnología Licenciatura Ingeniería Bioquímica Industrial MANUAL DE PRÁCTICAS DEL LABORATORIO DE INGENIERÍA ENZIMÁTICA Dr. Sergio Huerta
División de Ciencias Biológicas y de la Salud Departamento de Biotecnología Licenciatura Ingeniería Bioquímica Industrial MANUAL DE PRÁCTICAS DEL LABORATORIO DE INGENIERÍA ENZIMÁTICA Dr. Sergio Huerta
Bioenergética e introducción al metabolismo Departamento de Bioquímica Noviembre de 2005
 U.T.I. Biología Celular Bioenergética e introducción al metabolismo Departamento de Bioquímica Noviembre de 2005 Definiciones LA TERMODINAMICA ES LA CIENCIA QUE ESTUDIA LA ENERGIA Y SUS TRANSFORMACIONES
U.T.I. Biología Celular Bioenergética e introducción al metabolismo Departamento de Bioquímica Noviembre de 2005 Definiciones LA TERMODINAMICA ES LA CIENCIA QUE ESTUDIA LA ENERGIA Y SUS TRANSFORMACIONES
Física y Química 3º ESO
 1. Física y Química. Ciencias de la medida forman parte de las necesitan Ciencias de la naturaleza medir las propiedades de los cuerpos que se dividen en para lo cual se emplean lo que siempre conlleva
1. Física y Química. Ciencias de la medida forman parte de las necesitan Ciencias de la naturaleza medir las propiedades de los cuerpos que se dividen en para lo cual se emplean lo que siempre conlleva
