Trabajo práctico n 5 ENZIMAS DE RESTRICCIÓN
|
|
|
- Esperanza Lozano Domínguez
- hace 7 años
- Vistas:
Transcripción
1 Facultad de Ciencias Exactas, Químicas y Naturales Universidad Nacional de Misiones Trabajo práctico n 5 ENZIMAS DE RESTRICCIÓN Cátedra de Genética Molecular GRUPO 2 Alumnos o Briñoccoli Yanina o Cortese Julieta o Olexen Cinthia o Rodríguez Ana Laura o Soria Florencia o Zerda Moreira Andrea
2 TRABAJO PRÁCTICO Nº5 ENZIMAS DE RESTRICCIÓN (ER) PARTE PRÁCTICA A) Antes de efectuar una digestión de ADN genómico total es necesario conocer algunas características de la enzima de restricción a utilizar. Con la ayuda de catálogos identificar los siguientes aspectos de HaeIII: -Fuente de donde es extraída la enzima: Haemophilus aegyptius -Sitio diana (apunte la secuencia): GG CC CC GG -Temperatura a la que opera la enzima: 37 C -Buffer en el que actúa: Buffer C (ver Tabla 1 para especificación) -Costo de la enzima: 2500 USD + IVA: (Promega) -Detalle: Qué resultados espera obtener luego de la digestión con la ER? Cómo se observaría a luz UV el ADN digerido luego de una electroforesis en gel de agarosa? Luego de la digestión con la enzima se espera obtener fragmentos de diferente peso molecular, dependiendo de la cantidad de sitios diana que la secuencia en análisis presenta. Los fragmentos obtenidos de la digestión luego se someten a electroforesis en gel y al revelar con la luz UV se observarán los distintos fragmentos brillantes, en la distancia corrida según su peso molecular. Se debe considerar que si se utiliza ADN genómico total es posible observar smear en la corrida electroforética dada la cantidad de fragmentos que se generarían, muy por encima de la resolución del gel de agarosa. B) A continuación se le presentarán una serie de fotografías que corresponden a fragmentos de ADN digeridos con: TaqI, HinfI, AluI, MboI, HaeIII y DdeI, con el objeto de determinar diferencias intra (entre sexos) e interespecíficas. Las corridas fueron realizadas en geles de agarosa al 2%. Determinar: TaqI 1. Buscar en catálogos las características de las diferentes enzimas a utilizar de igual forma que en el punto A. 2
3 -Fuente de donde es extraída la enzima: Thermus aquaticus YTI. -Sitio diana (apunte la secuencia): T CG A A GC T -Temperatura a la que opera la enzima: 65 C (exhibe una actividad <10% a 37 C) -Buffer en el que actúa: Buffer E (ver Tabla 1 para especificación) -Costo de la enzima: 1000u USD: (Promega) Tabla 1: Proporciona composiciones de Buffers para la reacción de enzimas de restricción, que figuran como concentraciones 1X. Buffer ph (a 37 C) Tris-HCl (mm) De MgCl 2 (mm) NaCl (mm) KCl (mm) TDT (mm) B C D E HinfI -Fuente de donde es extraída la enzima: Haemophilus influenzae Rf. -Sitio diana (apunte la secuencia): G ANT C C TNA G -Temperatura a la que opera la enzima: 37 C -Buffer en el que actúa: Buffer B (ver Tabla 1 para especificación) -Costo de la enzima: 1000u USD + IVA:85,80 (Promega) AluI 3
4 -Fuente de donde es extraída la enzima: Arthrobacter luteus. -Sitio diana (apunte la secuencia): AG CT TC GA -Temperatura a la que opera la enzima: 37 C -Buffer en el que actúa: Buffer B (ver Tabla 1 para especificación) -Costo de la enzima: 500u USD + IVA: (Promega) MboI -Fuente de donde es extraída la enzima: Cepa E. coli que lleva el gen MboI de Moraxella bovis (ATCC 10900). -Sitio diana (apunte la secuencia): GATC CTAG -Temperatura a la que opera la enzima: 37 C -Buffer en el que actúa: 1X CutSmart Buffer: 50 mm acetato de potasio,20 mm Tris-acetato,10 mm Acetato de magnesio,100 mg / ml de BSA -Costo de la enzima: 500u --> USD: 69 (NEB) DdeI -Fuente de donde es extraída la enzima: Desulfovibrio desulfuricans. -Sitio diana (apunte la secuencia): C TNA G G ANT C -Temperatura a la que opera la enzima: 37 C -Buffer en el que actúa: Buffer D (ver Tabla 1 para especificación) 4
5 -Costo de la enzima: 200u USD + IVA:59.40 (Promega) 2. Reconocer, con la ayuda de catálogos, qué marcador de PM se utilizó en las diferentes corridas electroforéticas. Detallar el tamaño de las diferentes bandas del mismo. Con la ayuda del catálogo de Genbiotech, el marcador de PM que se utilizó en las diferentes corridas electroforéticas fue una escalera de ADN de 100bp (Fig.1), de 11 fragmentos que varían en tamaño desde 100 pb a 1000 pb en incrementos de 100 con una banda adicional en 1,500bp. El fragmento de 500 pb está presente en mayor intensidad para facilitar su identificación. En el presente trabajo se consideró de 600pb a la banda de mayor intensidad. Fig 1. Marcador de peso molecular. Escalera de ADN de 100pb 3. Estimar el tamaño de las diferentes bandas producto de la digestión de una enzima cualquiera. En la tabla siguiente se presentan el tamaño, en pares de bases, de las diferentes bandas producto de la digestión de cada una de las enzimas propuestas, en cada especie y discriminada por sexo: 5
6 Especies Enzima sexo AluI Machos Hembras DdeI Machos Hembras HaeIII Machos Hembras Himfl Machos Hembras Mbol Machos Hembras Machos TaqI Hembras
7 El cálculo del tamaño de las bandas obtenidas por las diferentes enzimas de restricción se realizó a partir de la medición de las imágenes de las corridas electroforéticas y mediante una curva estándar, la cual consiste en: la distancia de corrida de las bandas del marcador de peso molecular en el gel en milímetros, versus el tamaño en pares de bases. Tal curva se realizó para cada gel presentado, para así, luego de medir la distancia de la/las banda/s de cada especie en dicho gel, ubicar tal distancia en la curva y poder determinar el peso molecular correspondiente: Tamaño en pares de bases Distancia de migración en mm 4. Elaborar un mapa de restricción tentativo para las diferentes enzimas utilizadas. 7
8 En el mapa tentativo presentado se debe considerar que los puntos de corte han sido estimados de mediciones de corridas electroforéticas que, en algunos casos, pueden haberse perdido algunas bandas, por lo que el tamaño del fragmento total y el número de sitios podría no reflejar la verdadera acción enzimática. Asimismo, el orden de los fragmentos es arbitrario. 5. Determinar si con alguna de las enzimas se detectaron diferencias intra o interespecíficas. La enzima Alu I no presentó diferencias intra e interespecíficas. DdeI presentó diferencias intra e interespecíficas. En el macho 2 se observan 5 fragmentos, obtenidos por 4 cortes realizados por la enzima. Por otra parte los machos de las especies y 7 presentan una banda adicional. Los machos 2 y 3 también tiene una banda de 130 pb ausente en las demás especies. En cuanto a las hembras en la especies 8 y 9 está ausente la banda de 270 pb. Mientras que las especie 3 y 6 presentan una banda extra de 100 pb. La enzima TaqI, evidenció en la corrida electroforética, diferencias intra e interespecificas en los sitios de corte para esta enzima. Los machos de la especie 1 presentaron 4 bandas (3 sitios de corte) en comparación con las hembras de la especie 1 que presentaron 3 bandas (2 sitios de corte). La misma situación puede observarse para los machos y hembras de las especies 2, 3, 5,6. Los machos y hembras de las especies 4 y 7, presentaron 3 bandas (2 sitios de corte). Con respecto a la especie 9, para los machos se observaron 3 bandas ( 2 sitios de corte) mientras que para las hembras se observaron solo 2(un sitio de corte). En todos los casos la longitud de las bandas entre machos y hembras fue diferente con excepción de los machos de las especies 1,2,5 y 6 que comparten una banda de igual longitud con las hembras de las especies 1,2, 4,5, 6 y 7. Algunas bandas fueron de tamaño muy similar entre machos y hembras de la misma especie. 8
9 Por otro lado, se observan diferentes patrones de bandas de distintas longitudes entre las especies, pudiendo decir que la enzima TaqI presenta 1, 2 y 3 sitios de corte, dependiendo de la especie observada. HaeIII presentó diferencias intra e interespecíficas. Los machos y las hembras de las especies 1, 3, 4, 5, 6, 7, 8 y 9 presentan patrones de bandas distintivos, en cuanto al tamaño y, en algunos casos al número, presentando ninguno, 1 a 2 sitios de corte (quizás más). La especie 2 parecería no tener diferencias intraespecíficas. Asimismo, son evidentes las diferencias entre las especies, mostrando diferentes o igual número de bandas, y en el último caso, con diferente tamaño. La enzima HaeIII parecería presentar diferentes sitios de corte en las diferentes especies y entre los distintos sexos. En la enzima MboI se observó tanto diferencias intra como interespecificas. Dentro de las primeras observamos que las hembras presentaban mayor longitud (PM) de los fragmentos obtenidos y que podían diferir en el número de bandas con respecto a los machos de una misma especie. En cuanto a las diferencias interespecificas se puede mencionar 3 patrones de bandas diferentes 1, 2 o 3 bandas donde algunas especies comparten el mismo patrón como en el caso de los machos de las especies 2, 3, 4, 5, 9 y 10, que a su vez difieren de la especie 7. Al digerir con la enzima Hinf I, y luego del análisis de las fotografías de las corridas electroforéticas, se pone de manifiesto que existen diferencias tanto intraespecificas como interespecificas. Siendo más acentuadas las diferencias interespecificas. Respecto a las diferencias intraespecificas: solo en la especie 9 se observa que los machos presentan dos bandas (1 sitio de corte), mientras que en las hembras presentan 3 bandas (2 sitios de corte). Los individuos, tanto machos como hembras, de las especies 1, 2, y 5 presentan el mismo patrón de bandas, dos fragmentos (1 sitio de corte). Los representantes de las especies 3, 4, 6, 7, y 8 presentan a su vez el mismo patrón de bandas, 3 fragmentos (2 sitios de corte). Pero en las hembras de las especies 6 y 7 aparte existen otros tres fragmentos en los que no se puede determinar el peso molecular de los mismos, debido a que se encuentran por fuera del rango del marcador de peso molecular. No sucede lo mismo en los machos de las mismas especies en donde solo existen los tres fragmentos mencionados anteriormente. 6. Discutir los resultados. En los resultados obtenidos resaltamos que, al no poder determinar el tamaño del fragmento total tratado con diferentes enzimas, los puntos de cortes propuestos son sólo sugerentes y representan los sitios tentativos de acción de las enzimas. Esto se debe a que el número de bandas presentadas 9
10 en las corridas no implica, necesariamente, un número de sitios de cortes determinado. Por otro lado, existe la dificultad de la determinación del tamaño de los fragmentos más pequeños que caen fuera de la escala del marcador. C) Utilizando el siguiente número de acceso M del NCBI rescate la secuencia del genzfy- 2 de Mardon and Page (1994). Ubique los primers (AK1 y AK2) con la herramienta del NCBI. Verifique el tamaño del amplicón. Levante la secuencia delimitada por los primers y traslade al programa NEB CUTTER ( Proceda a efectuar la digestión con las ER detalladas en el punto B. Determine: (a) cuántos sitios de reconocimiento para cada ER posee el fragmento amplificado? (b) la posición de los diferente sitio diana para las diferentes ER. (c) Genere una corrida electroforética electrónica que le permita discriminar los fragmentos discretos producto de la digestión con las diferentes enzimas. (d) Compare con los datos de las digestiones realizadas en las fotografías entregadas. Utilizando el número de acceso M del NCBI identificamos la secuencia del gen Zfy-2 de Mardon and Page (1994) y ubicamos los primers (AK1 y AK2): LOCUS MUSZFY2A 2794 bp mrna linear Mus musculus, proteína de dedos de zinc (Zfy-2) mrna. Mus musculus (ratón doméstico) Clasificación (NCBI) Mus musculus Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Glires; Rodentia; Sciurognathi; Muroidea; Muridae; Murinae; Mus; Mus. La secuencia delimitada por los primer es: 10
11 GTCTATCCTTGCATGTTTTGTGGGAAAAAATTTAAGACCAAAAGGTTTTTGAAAAGACACATAAAAAACCAT CCTGAATACCTTGCTAATAAAAAATATCACTGTACTGAGTGTGATTACAGTACCAACAAGAAGATAAGCTTA CATAATCACATGGAGAGCCACAAGCTAACCATTAAGACAGAAAAGACCACCGAATGTGATGACTGTAGGAA GAATCTTTCTCATGCTGGGACTTTGTGTACTCACAAAACAATGCATACAGAAAAAGGAGTCAACAAAACATG TAAGTGTAAGTTCTGTGACTATGAAACAGCTGAACAGACATTATTGAATCACCACCTTTTGGTGGTCCACAG GAAGAAATTTCCTCACATTTGTGGAGAATGTGGTAAAGGTTTCCGTCACCCATCAGCACTCAAAAAGCACAT ACGAGTTCACACAGGAGAGAAGCCCTATGAATGTCAGTATTGTGAGTACAAGTCTGCAGACTCTTCCAACTT GAAAACTCATATAAAATCTAAGCATAGTAAAGAGATACCACTGAAGTGTGACATCTGTCTCCTGACTTTCTC AGATACCAAAGAGGCTCAGCAACATGCCGTTCTGCACCAAGAAAGCAGAACACATCAATGTTCACATTGCA ACCATAAGAGTTCAAACTCAAGTGATTTAAAGCGACACATAATTTCCGTTCACACAAAGGCGTATCCTCATA AATGTGACATGTGCAGCAAAGGATTTCATAGGCCTTCAGAACTCAAGAAGCATGTGGCTACCCATAAAAGT AAAAAAATGCACCAATGTAGACACTGTGACTTTAATAGTCCAGATCCATTTCTGCTTAGTCACCATATTCTCT CAGCTCACACAAAGAATGTTCCATTCAAGTGTAAGAGATGTAAAAAGGAATTTCAACAACAGTGTGAGCTT CAAACGCATATGAAGACCCACAGTAGCCGAAAAGTCTATCAGTGTGAGTACTGTGAATATAGCACCAAAGA TGCCTCAGGTTTTAAGCGTCACGTTATCTCCATTCATACGAAAGACTATCCTCACCGCTGTGACTTCTGCAAG AAAGGATTCCGGAGACCCTCGGAAAAGAATCAACACATAATGCGACAT Luego, levantamos la secuencia delimitada por los primers al programa NEB CUTTER ( y continuamos con la digestión con las ER detalladas en el punto anterior. Así determinamos que: a) Los sitios de reconocimiento para las enzimas de restricción que posee el fragmento amplificado son: En la Tabla 1, se observa en la fila Cuts, los sitios de reconocimiento para las enzimas de restricción. Para la enzima TaqI, no se detectaron sitios de reconocimiento en la secuencia amplificada: 11
12 b) La posición de los diferentes sitio diana para las diferentes ER son: Sequence digested with: AluI, DdeI, HaeIII, HinfI, MboI Sequence digested with: AluI, DdeI, HaeIII, HinfI, MboI ALu I Ddel 12
13 HaeIII HinfI Mbol e) Corridas electroforéticas electrónicas para cada ER: Alu I 13
14 Ddel 14
15 Hae III 15
16 HinfI 16
17 Mbol I 17
18 d) Compare con los datos de las digestiones realizadas en las fotografías entregadas. Al comparar las corridas electroforéticas electrónicas con las fotografías entregadas observamos que: La enzima HaeIII presenta un patrón de bandas muy similar, en la especie 7, al presentado en la electroforesis electrónica para ésta. Asimismo, la enzima MboI presenta un patrón equivalente al de la electrónica en aquellas especies con dos bandas. La enzima AluI exhibe un patrón de bandas análogo al de su electroforesis electrónica pero, sin embargo, los tamaños de las bandas difieren. HinfI y DdeI no son parecidas a sus equivalentes electrónicos. La enzima TaqI no exhibe sitio de corte en la corrida electrónica. 18
19 Bibliografía Sitios de internet consultados: (Página de biodynamics donde se encuentran las enzimas de restricción especificando cantidad (u) y precio en dólares más IVA) (página donde se encuentra la composición de los buffers de reacción que se especifican para cada enzima) 19
3. MATERIALES Y MÉTODOS
 3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
RESULTADOS Y DISCUSIÓN
 RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
Digestión de DNA usando Enzimas de Restricción
 Digestión de DNA usando Enzimas de Restricción Objetivos El cortar el DNA con enzimas de restricción es muchas veces el primer paso de los investigadores para estudiar un gen. En este laboratorio se hará
Digestión de DNA usando Enzimas de Restricción Objetivos El cortar el DNA con enzimas de restricción es muchas veces el primer paso de los investigadores para estudiar un gen. En este laboratorio se hará
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR
 TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR Microbiología a General Área Micología Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las
TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR Microbiología a General Área Micología Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80
 AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
VARIACIÓN INTER-INDIVIDUAL
 Facultad de Medicina Genética 1 er Curso TEMA 2 LA GEOGRAFÍA DEL GENOMA HUMANO 2.1 Estructura del genoma humano y variación inter-individual. 2.2 El ADN repetitivo. 2.1 ESTRUCTURA DEL GENOMA HUMANO Y VARIACIÓN
Facultad de Medicina Genética 1 er Curso TEMA 2 LA GEOGRAFÍA DEL GENOMA HUMANO 2.1 Estructura del genoma humano y variación inter-individual. 2.2 El ADN repetitivo. 2.1 ESTRUCTURA DEL GENOMA HUMANO Y VARIACIÓN
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN
 INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987)
 EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
7. RESULTADOS. De las 18 cepas previamente congeladas (-70 C) se recuperaron 13 cepas. En los
 7. RESULTADOS De las 18 cepas previamente congeladas (-70 C) se recuperaron 13 cepas. En los pacientes infectados con estas cepas, 8 (61.5%) presentaron gastritis crónica, 2 (15.38%) gastritis folicular,
7. RESULTADOS De las 18 cepas previamente congeladas (-70 C) se recuperaron 13 cepas. En los pacientes infectados con estas cepas, 8 (61.5%) presentaron gastritis crónica, 2 (15.38%) gastritis folicular,
6.1 Células competentes y transformación. posterior transformación de éstas para la elaboración de un control positivo. La
 6. RESULTADOS 6.1 Células competentes y transformación La primera parte experimental del trabajo consistió en la obtención de células competentes y posterior transformación de éstas para la elaboración
6. RESULTADOS 6.1 Células competentes y transformación La primera parte experimental del trabajo consistió en la obtención de células competentes y posterior transformación de éstas para la elaboración
Transferencia de material genético II. Ensayos de restricción (digestión) del plásmido Electroforesis en geles de agarosa
 Transferencia de material genético II Ensayos de restricción (digestión) del plásmido Electroforesis en geles de agarosa ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar
Transferencia de material genético II Ensayos de restricción (digestión) del plásmido Electroforesis en geles de agarosa ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar
).- INTRODUCCIÓN: el sector vitivinícola en León
 Aislamiento, caracterización genética y conservación de levaduras vínicas autóctonas de la variedad de uva Prieto Picudo (Asociación Vinos de Calidad de Tierras de León: Castilla y León) RM-00006 IP: Juan
Aislamiento, caracterización genética y conservación de levaduras vínicas autóctonas de la variedad de uva Prieto Picudo (Asociación Vinos de Calidad de Tierras de León: Castilla y León) RM-00006 IP: Juan
Tipo Tm ( O C) Tamaño (pb) Segregación
 IV. RESULTADOS Se diseñaron iniciadores i-pcr para 12 marcadores AFLP, obteniendo la secuencia del producto de amplificación en 2 casos correspondientes a marcadores AFLP mayores a 190 pb con un contenido
IV. RESULTADOS Se diseñaron iniciadores i-pcr para 12 marcadores AFLP, obteniendo la secuencia del producto de amplificación en 2 casos correspondientes a marcadores AFLP mayores a 190 pb con un contenido
CAPÍTULO 3: RESULTADOS
 CAPÍTULO 3: RESULTADOS 3.1 Extracción y cuantificación de ADN La extracción de ADN realizada con el método modificado de Khanuja et al. (1999) de las muestras del grupo 4 (Pedernales Canoa Km 4, 65 individuos),
CAPÍTULO 3: RESULTADOS 3.1 Extracción y cuantificación de ADN La extracción de ADN realizada con el método modificado de Khanuja et al. (1999) de las muestras del grupo 4 (Pedernales Canoa Km 4, 65 individuos),
Ensayos de restricción
 Ensayos de restricción Vector con inserto= Vector recombinante 6500pb Objetivos de ensayos de restricción de plásmidos Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa.
Ensayos de restricción Vector con inserto= Vector recombinante 6500pb Objetivos de ensayos de restricción de plásmidos Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa.
Caracteristicas del ADN. Se rompen con las siguientes condiciones -Temperaturas >90 - ph >10.5
 PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
TECNICAS DE BIOLOGIA MOLECULAR
 Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006
 IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90. País: ESPAÑA
 XII OLIMPIADA IBEROAMERICANA DE BIOLOGÍA SETIEMBRE 2018 LOJA-ECUADOR EXAMEN PRÁCTICO BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90 TIEMPO PERMITIDO: 120 minutos País: ESPAÑA ESCRIBA EL CÓDIGO EN EL SIGUIENTE
XII OLIMPIADA IBEROAMERICANA DE BIOLOGÍA SETIEMBRE 2018 LOJA-ECUADOR EXAMEN PRÁCTICO BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90 TIEMPO PERMITIDO: 120 minutos País: ESPAÑA ESCRIBA EL CÓDIGO EN EL SIGUIENTE
BIOLOGIA MOLECULAR. Dra. Ma. Laura Tondo
 TÉCNICAS BASADAS EN HIBRIDACIÓN MOLECULAR BIOLOGIA MOLECULAR 2016 Dra. Ma. Laura Tondo DESNATURALIZACION Y RENATURALIZACION DEL DNA Cómo se mantienen unidas las dos hebras del DNA? - Puentes de hidrógeno
TÉCNICAS BASADAS EN HIBRIDACIÓN MOLECULAR BIOLOGIA MOLECULAR 2016 Dra. Ma. Laura Tondo DESNATURALIZACION Y RENATURALIZACION DEL DNA Cómo se mantienen unidas las dos hebras del DNA? - Puentes de hidrógeno
Técnicas del ADN recombinante Introducción
 Técnicas del ADN recombinante Introducción Estructura primaria del ADN Enzimas de restricción (ER) Son enzimas (endonucleasas) de origen bacteriano que reconocen y cortan secuencias específicas (4-8 pb)
Técnicas del ADN recombinante Introducción Estructura primaria del ADN Enzimas de restricción (ER) Son enzimas (endonucleasas) de origen bacteriano que reconocen y cortan secuencias específicas (4-8 pb)
BIOLOGIA CELULAR Y MOLECULAR. LABORATORIO No 5: ENZIMAS DE RESTRICCION
 BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 5: ENZIMAS DE RESTRICCION 1. INTRODUCION Las enzimas de restricción, también conocidas como endonucleasas, son enzimas que cortan los enlaces fosfodiéster del
BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 5: ENZIMAS DE RESTRICCION 1. INTRODUCION Las enzimas de restricción, también conocidas como endonucleasas, son enzimas que cortan los enlaces fosfodiéster del
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular. Biol 3051L
 Técnicas de Biología Molecular Biol 3051L Objetivos El cortar el DNA con enzimas de restricción y hacer corridas de geles usando electroforesis es muchas veces el primer paso para estudiar un gen. En este
Técnicas de Biología Molecular Biol 3051L Objetivos El cortar el DNA con enzimas de restricción y hacer corridas de geles usando electroforesis es muchas veces el primer paso para estudiar un gen. En este
Cromosoma Y. Microdeleciones de AZF. Correlación Infertilidad - Cáncer Testicular
 Cromosoma Y Microdeleciones de AZF Correlación Infertilidad - Cáncer Testicular Introducción Últimos 30-50 años: 50% en la concentración y motilidad espermática En la incidencia de los TGCT (tumores de
Cromosoma Y Microdeleciones de AZF Correlación Infertilidad - Cáncer Testicular Introducción Últimos 30-50 años: 50% en la concentración y motilidad espermática En la incidencia de los TGCT (tumores de
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
 Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar
 4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar - Agarosa de grado molecular - Agarosa de bajo punto de fusión
4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar - Agarosa de grado molecular - Agarosa de bajo punto de fusión
PLANIFICACIÓN DE ASIGNATURA
 PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
Código: KIT-005 Ver: 1 MTHFR C677T
 Sistema para detección de la alteración C677T en el gene que codifica para la Metilen-tetrahidrofolato reductasa humana 1 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598)
Sistema para detección de la alteración C677T en el gene que codifica para la Metilen-tetrahidrofolato reductasa humana 1 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598)
TAXONOMÍA MOLECULAR. Dra. Alicia Luque CEREMIC
 TAXONOMÍA MOLECULAR Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las condiciones ambientales. PLEOMORFISMO Debido al bajo poder
TAXONOMÍA MOLECULAR Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las condiciones ambientales. PLEOMORFISMO Debido al bajo poder
Estimación de las Frecuencias Alélicas del D16S539 en una Muestra Poblacional de ascendencia andina ancashina. Tito Tadeo, Raúl Yhossef.
 ANTECEDENTES Los estudios del genoma humano han revelado la existencia de secuencias repetitivas en tándem en muchos sitios a lo largo de todos los cromosomas, que varían en el número de veces que la unidad
ANTECEDENTES Los estudios del genoma humano han revelado la existencia de secuencias repetitivas en tándem en muchos sitios a lo largo de todos los cromosomas, que varían en el número de veces que la unidad
MATERIALES Y MÉTODOS. Muestras
 MATERIALES Y MÉTODOS Muestras Las muestras de sangre con el bacilo M. tuberculosis se obtuvieron de 2 pacientes con tuberculosis diagnosticada por clínica, laboratorio y con cultivo positivo como controles
MATERIALES Y MÉTODOS Muestras Las muestras de sangre con el bacilo M. tuberculosis se obtuvieron de 2 pacientes con tuberculosis diagnosticada por clínica, laboratorio y con cultivo positivo como controles
Protocolos de preparación de muestras
 Protocolos de preparación de muestras Contenido 1. TIPOS DE MUESTRA 2. PROTOCOLO DE PREPARACIÓN DE MUESTRA 2.1 Producto de PCR crudo (sin purificar). 2.2 Producto de PCR crudo en gel (sin purificar). 2.3
Protocolos de preparación de muestras Contenido 1. TIPOS DE MUESTRA 2. PROTOCOLO DE PREPARACIÓN DE MUESTRA 2.1 Producto de PCR crudo (sin purificar). 2.2 Producto de PCR crudo en gel (sin purificar). 2.3
PRÁCTICO Nº 6. ACIDOS NUCLEICOS II Espectrofotometría, Electroforesis, Mapas de Restricción
 PRÁCTICO Nº 6. ACIDOS NUCLEICOS II Espectrofotometría, Electroforesis, Mapas de Restricción Espectrofotometría de ácidos nucleicos Los dobles enlaces conjugados de las bases nitrogenadas hacen que los
PRÁCTICO Nº 6. ACIDOS NUCLEICOS II Espectrofotometría, Electroforesis, Mapas de Restricción Espectrofotometría de ácidos nucleicos Los dobles enlaces conjugados de las bases nitrogenadas hacen que los
Ensayos southwestern y northwestern
 Búsqueda de proteínas que se unen a DNA (secuencia conocida, promotor) Ensayo southwestern encontrar proteínas ligantes de DNA que reconozcan en éste una determinada secuencia de nucleótidos Ensayo northwestern
Búsqueda de proteínas que se unen a DNA (secuencia conocida, promotor) Ensayo southwestern encontrar proteínas ligantes de DNA que reconozcan en éste una determinada secuencia de nucleótidos Ensayo northwestern
o Y Cromosomas autosómicos: 22 pares Cromosoma sexual: 1 par (X o Y)
 Cromosomas autosómicos: 22 pares o Y Cromosoma sexual: 1 par (X o Y) Un alelo es dominante cuando su presencia se manifiesta siempre en el fenotipo, y el fenotipo es igual para el homocigótico que para
Cromosomas autosómicos: 22 pares o Y Cromosoma sexual: 1 par (X o Y) Un alelo es dominante cuando su presencia se manifiesta siempre en el fenotipo, y el fenotipo es igual para el homocigótico que para
Código: IDK-003 Ver: 1. Factor V Leiden
 Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
PCR. Técnica inventada por Kary Mullis :1987. Premio Nobel 1993. Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN
 PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
PRÁCTICA DE GENÉTICA
 PRÁCTICA DE GENÉTICA Diagnóstico molecular de una enfermedad genética, seguimiento en una genealogía y consejo genético. Profesores responsables: Paloma Martínez Rodríguez y José L. Bella Sombría NORMAS
PRÁCTICA DE GENÉTICA Diagnóstico molecular de una enfermedad genética, seguimiento en una genealogía y consejo genético. Profesores responsables: Paloma Martínez Rodríguez y José L. Bella Sombría NORMAS
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS. Es un ácido capaz de formar sales con iones cargados
 Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Figura Cariotipo con bandas G-C secuenciales de la especie Pan troglodytes. La ordenación del cariotipo se ha realizado siguiendo las
 116 Figura 4.20. Cariotipo con bandas G-C secuenciales de la especie Pan troglodytes. La ordenación del cariotipo se ha realizado siguiendo las homologías cromosómicas con la especie humana. Resultados
116 Figura 4.20. Cariotipo con bandas G-C secuenciales de la especie Pan troglodytes. La ordenación del cariotipo se ha realizado siguiendo las homologías cromosómicas con la especie humana. Resultados
Código: IDK-006 Ver: 1 MTHFR A1298C. Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa
 Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr. Criterios generales para el diseño de cebadores para RT qpcr
 Diseño de cebadores para RT qpcr ACTIVIDADES 1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr con SYBR Green 2 Mostración del diseño de un par de cebadores para cuantificar
Diseño de cebadores para RT qpcr ACTIVIDADES 1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr con SYBR Green 2 Mostración del diseño de un par de cebadores para cuantificar
Código: IDK-004 Ver: 1. Factor II 20210
 20210 Sistema para detección de la alteración G20210A en el gene que codifica para el de la coagulación humana PM 1 2 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2
20210 Sistema para detección de la alteración G20210A en el gene que codifica para el de la coagulación humana PM 1 2 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2
PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN DE LAS COMPETENCIAS PROFESIONALES CUESTIONARIO DE AUTOEVALUACIÓN PARA LAS TRABAJADORAS Y TRABAJADORES
 MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
Módulo 2 Biología Molecular Genética Molecular II Módulo 2. Clonación y Selección de moléculas recombinantes
 Módulo 2 Clonación y Selección de moléculas recombinantes El fragmento de ADN amplificado por PCR en el módulo anterior se introdujo en un vector pgem T Easy (Ver Anexo 2) utilizando una ligasa. En la
Módulo 2 Clonación y Selección de moléculas recombinantes El fragmento de ADN amplificado por PCR en el módulo anterior se introdujo en un vector pgem T Easy (Ver Anexo 2) utilizando una ligasa. En la
ELECTROFORESIS. Objetivo de la clase. *fundamentos *procedimientos *algunas aplicaciones. Conocer los métodos electroforéticos
 ELECTROFORESIS Objetivo de la clase Conocer los métodos electroforéticos *fundamentos *procedimientos *algunas aplicaciones Definición de electroforesis Proceso de separación que se basa en la diferente
ELECTROFORESIS Objetivo de la clase Conocer los métodos electroforéticos *fundamentos *procedimientos *algunas aplicaciones Definición de electroforesis Proceso de separación que se basa en la diferente
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
Seminario de Problemas
 Seminario de Problemas 1. Qué uniones relacionan: a) dos deoxirribosas entre si; b) 2 bases enfrentadas? 2. Un segmento de ADN de cadena simple tiene la siguiente composición de bases: A 31,5 % C 23,7
Seminario de Problemas 1. Qué uniones relacionan: a) dos deoxirribosas entre si; b) 2 bases enfrentadas? 2. Un segmento de ADN de cadena simple tiene la siguiente composición de bases: A 31,5 % C 23,7
Conclusiones y perspectivas...34 Bibliografía...36 Apéndices...41 Apéndice A: Lista de cepas...41 Apéndice B: Lista de plásmidos...42 Apéndice C: List
 Tabla de contenido Introducción...1 Sustratos y productos...2 Exposición en superficie celular...5 Ruta metabólica y exposición en superficie...6 Objetivos...8 Objetivo general...8 Objetivos específicos...8
Tabla de contenido Introducción...1 Sustratos y productos...2 Exposición en superficie celular...5 Ruta metabólica y exposición en superficie...6 Objetivos...8 Objetivo general...8 Objetivos específicos...8
Introducción General 1
 Índice General I Introducción General 1 1. Alimentos funcionales 1 1.1. Probióticos y prebióticos como componentes de alimentos funcionales 2 1.1.1. Probióticos 2 1.1.2. Prebióticos 3 2. El kefir: un ecosistema
Índice General I Introducción General 1 1. Alimentos funcionales 1 1.1. Probióticos y prebióticos como componentes de alimentos funcionales 2 1.1.1. Probióticos 2 1.1.2. Prebióticos 3 2. El kefir: un ecosistema
VII. RESULTADOS Y DISCUSIÓN
 7.1 Acopio de Baterías VII. RESULTADOS Y DISCUSIÓN Como primer paso en la realización de ésta tesis, se inició con el acopio de baterías de todo tipo. La recolección de baterías se hizo en los diferentes
7.1 Acopio de Baterías VII. RESULTADOS Y DISCUSIÓN Como primer paso en la realización de ésta tesis, se inició con el acopio de baterías de todo tipo. La recolección de baterías se hizo en los diferentes
Caracterización de la expresión del gen reca
 Figura 3.8. (A) Diagrama que representa los fragmentos amplificados a partir del promotor lexa de F. nucleatum usados en experimentos EMSA. Las posiciones numeradas se refieren al punto de inicio de traducción
Figura 3.8. (A) Diagrama que representa los fragmentos amplificados a partir del promotor lexa de F. nucleatum usados en experimentos EMSA. Las posiciones numeradas se refieren al punto de inicio de traducción
Repaso de PCR y qpcr
 Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola 2016 Dogma central de la biología
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola 2016 Dogma central de la biología
Transferencia de material genético II. 1) Aislamiento de plásmidos Lisis alcalina
 Transferencia de material genético II 1) Aislamiento de plásmidos Lisis alcalina ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar que hay células transformantes de acuerdo
Transferencia de material genético II 1) Aislamiento de plásmidos Lisis alcalina ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar que hay células transformantes de acuerdo
RESULTADOS Y DISCUSIÓN. Purificación de Tripsina
 RESULTADOS Y DISCUSIÓN Purificación de Tripsina Se purificó tripsina a partir de 86 gr de ciegos pilóricos, la enzima fue separada del resto de las moléculas del extracto crudo mediante cromatografía de
RESULTADOS Y DISCUSIÓN Purificación de Tripsina Se purificó tripsina a partir de 86 gr de ciegos pilóricos, la enzima fue separada del resto de las moléculas del extracto crudo mediante cromatografía de
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
Material complementario UD 5
 Material complementario UD 5 Componentes de la PCR ADN molde que es el que se quiere replicar. Oligonucleótidos iniciadores, primers o cebadores: - Son oligonucleótidos monocatenarios cuya secuencia es
Material complementario UD 5 Componentes de la PCR ADN molde que es el que se quiere replicar. Oligonucleótidos iniciadores, primers o cebadores: - Son oligonucleótidos monocatenarios cuya secuencia es
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ
 REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
Marcadores Bioquímicos: Isoenzimas y Proteínas de Reserva
 Marcadores morfológicos Influencia del ambiente Bajo número Caracteres de madurez Entrenamiento y subjetividad Marcadores moleculares Sin influencia ambiental Cantidad ilimitada Análisis en fases tempranas
Marcadores morfológicos Influencia del ambiente Bajo número Caracteres de madurez Entrenamiento y subjetividad Marcadores moleculares Sin influencia ambiental Cantidad ilimitada Análisis en fases tempranas
UNIVERSIDAD DE MAGALLANES FACULTAD DE CIENCIAS DEPARTAMENTO CIENCIAS Y RECURSOS NATURALES
 UNIVERSIDAD DE MAGALLANES FACULTAD DE CIENCIAS DEPARTAMENTO CIENCIAS Y RECURSOS NATURALES Diversidad Filogenética de Cianobacterias y Eubacterias en Estromatolitos Modernos de Laguna Amarga, Torres del
UNIVERSIDAD DE MAGALLANES FACULTAD DE CIENCIAS DEPARTAMENTO CIENCIAS Y RECURSOS NATURALES Diversidad Filogenética de Cianobacterias y Eubacterias en Estromatolitos Modernos de Laguna Amarga, Torres del
MATERIALES Y MÉTODOS. Cepas Utilizadas
 MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
Seminario Genética. Técnicas de Biología Molecular
 Seminario Genética Técnicas de Biología Molecular Contenidos Extracción de ADN Enzimas de restricción PCR Southern Blot Secuenciación método de Sanger y automática 1º Extracción de ADN Se pueden tomar
Seminario Genética Técnicas de Biología Molecular Contenidos Extracción de ADN Enzimas de restricción PCR Southern Blot Secuenciación método de Sanger y automática 1º Extracción de ADN Se pueden tomar
Área Virología Área Virología
 Área Virología Departamento de Microbiología Facultad de Medicina UBA 2009 1 Objetivos Generales: Conocer la metodología aplicada en el diagnóstico de laboratorio de infecciones virales humanas y la interpretación
Área Virología Departamento de Microbiología Facultad de Medicina UBA 2009 1 Objetivos Generales: Conocer la metodología aplicada en el diagnóstico de laboratorio de infecciones virales humanas y la interpretación
HERRAMIENTAS MOLECULARES APLICADAS A LA INVESTIGACIÓ PHYTOPHTHORA INFESTANS EN LA ESTACIÓ
 HERRAMIENTAS MOLECULARES APLICADAS A LA INVESTIGACIÓ INVESTIGACIÓN DE PHYTOPHTHORA INFESTANS EN LA ESTACIÓ ESTACIÓN CIPCIP-QUITO El tizón tardío de la papa causado por el hongo Phytophthora infestans continua
HERRAMIENTAS MOLECULARES APLICADAS A LA INVESTIGACIÓ INVESTIGACIÓN DE PHYTOPHTHORA INFESTANS EN LA ESTACIÓ ESTACIÓN CIPCIP-QUITO El tizón tardío de la papa causado por el hongo Phytophthora infestans continua
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos
 Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Temas actuales: Next Generation Sequencing (NGS) Bioinformática Elvira Mayordomo
 Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
ELECTROFORESIS Q U Í M I C A B I O L Ó G I C A. D E P A R T A M E N T O D E B Q C A. F C N Y C S. U N P S J B
 ELECTROFORESIS Q U Í M I C A B I O L Ó G I C A. D E P A R T A M E N T O D E B Q C A. F C N Y C S. U N P S J B 2 0 1 7 Que es la electroforesis? Qué permite analizar? Que factores influyen? Separar y analizar
ELECTROFORESIS Q U Í M I C A B I O L Ó G I C A. D E P A R T A M E N T O D E B Q C A. F C N Y C S. U N P S J B 2 0 1 7 Que es la electroforesis? Qué permite analizar? Que factores influyen? Separar y analizar
Métodos de secuenciación de ADN
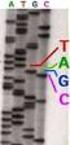 Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
POSTER P163 - UICC OSLO
 POSTER P163 - UICC OSLO 22 1 EFECTO DE LOS OLIGOELEMENTOS Se, Zn y Mn MAS LACHESIS MUTA EN EL CRECIMIENTO DE CELULAS PANC-1. ESTUDIOS IN VIVO E IN VITRO. EJV Crescenti 1, ES Rivera 2, M Croci 1, GP Cricco
POSTER P163 - UICC OSLO 22 1 EFECTO DE LOS OLIGOELEMENTOS Se, Zn y Mn MAS LACHESIS MUTA EN EL CRECIMIENTO DE CELULAS PANC-1. ESTUDIOS IN VIVO E IN VITRO. EJV Crescenti 1, ES Rivera 2, M Croci 1, GP Cricco
SECCIÓN II: Diversidad genética de aislamientos de F. graminearum empleando marcadores moleculares
 SECCIÓN II: Diversidad genética de aislamientos de F. graminearum empleando marcadores moleculares 39 SECCIÓN II: Diversidad genética de aislamientos de F. graminearum empleando marcadores moleculares
SECCIÓN II: Diversidad genética de aislamientos de F. graminearum empleando marcadores moleculares 39 SECCIÓN II: Diversidad genética de aislamientos de F. graminearum empleando marcadores moleculares
RESULTADOS Y DISCUSIONES. El análisis por medio de electroforesis a demostrado ser uno de los
 39 RESULTADOS Y DISCUSIONES El análisis por medio de electroforesis a demostrado ser uno de los mejores métodos de separación para complejos proteicos, además de necesitar cantidades muy pequeñas de muestra.
39 RESULTADOS Y DISCUSIONES El análisis por medio de electroforesis a demostrado ser uno de los mejores métodos de separación para complejos proteicos, además de necesitar cantidades muy pequeñas de muestra.
RESULTADOS Y DISCUSIÓN. Aislamiento de Inmunoglobulina de Conejo Anti-Ferritina
 RESULTADOS Y DISCUSIÓN Aislamiento de Inmunoglobulina de Conejo Anti-Ferritina El aislamiento de inmunoglobulinas provenientes del suero de conejo inmunizado con ferritina de bazo de caballo, se realizó
RESULTADOS Y DISCUSIÓN Aislamiento de Inmunoglobulina de Conejo Anti-Ferritina El aislamiento de inmunoglobulinas provenientes del suero de conejo inmunizado con ferritina de bazo de caballo, se realizó
Técnicas moleculares aplicadas en Bioquímica Clínica-2017 Dra. María Gabriela Lacoste
 Técnicas moleculares aplicadas en Bioquímica Clínica-2017 Dra. María Gabriela Lacoste TIPOS DE PCR SEGÚN LA MUTACION A DETECTAR GRANDES DELECIONES MUTACIONES PUNTUALES Y DELECIONES PEQUEÑAS PCR Multiplex
Técnicas moleculares aplicadas en Bioquímica Clínica-2017 Dra. María Gabriela Lacoste TIPOS DE PCR SEGÚN LA MUTACION A DETECTAR GRANDES DELECIONES MUTACIONES PUNTUALES Y DELECIONES PEQUEÑAS PCR Multiplex
Detección de mutaciones en el gen rpob de M. tuberculosis 8 MATERIALES Y MÉTODOS
 8 MATERIALES Y MÉTODOS 8.2 CEPAS DE M. TUBERCULOSIS. Se utilizó 1 cepa control y se analizaron un total de 13 extracciones diferentes de DNA de cepas de M. tuberculosis previamente aisladas de muestras
8 MATERIALES Y MÉTODOS 8.2 CEPAS DE M. TUBERCULOSIS. Se utilizó 1 cepa control y se analizaron un total de 13 extracciones diferentes de DNA de cepas de M. tuberculosis previamente aisladas de muestras
ENZIMAS USADAS EN LA MANIPULACION DE ACIDOS NUCLEICOS Adriana Krapp consulta: martes 16 hs
 ENZIMAS USADAS EN LA MANIPULACION DE ACIDOS NUCLEICOS 2016 Adriana Krapp consulta: martes 16 hs TECNOLOGIA DEL DNA RECOMBINANTE Insulina humana recombinante Cultivos con resistencia a herbicidas (glifosato):expresan
ENZIMAS USADAS EN LA MANIPULACION DE ACIDOS NUCLEICOS 2016 Adriana Krapp consulta: martes 16 hs TECNOLOGIA DEL DNA RECOMBINANTE Insulina humana recombinante Cultivos con resistencia a herbicidas (glifosato):expresan
Genética grupo B. Examen Final Primera Parte. 19 de Mayo de Apellidos Nombre Firma:
 Genética grupo B. Examen Final Primera Parte. 19 de Mayo de 2017. Apellidos Nombre Firma: 1/6- Los individuos marcados en negro en la genealogía presenta una anomalía muy infrecuente en la población. Establezca
Genética grupo B. Examen Final Primera Parte. 19 de Mayo de 2017. Apellidos Nombre Firma: 1/6- Los individuos marcados en negro en la genealogía presenta una anomalía muy infrecuente en la población. Establezca
Técnicas de Biología Molecular. Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Extracción de ácidos nucleicos o o DNA RNA Tipos de Muestras Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Extracción de ácidos nucleicos o o DNA RNA Tipos de Muestras Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
1) a) En la figura señale, englobando con un trazo continuo, lo siguiente:
 CÁTEDRA: BIOQUÍMICA Carreras: Farmacia Profesorado en Química Licenciatura en Química Lic. en C. y T. de los Alimentos ÁCIDOS NUCLEICOS 1) a) En la figura señale, englobando con un trazo continuo, lo siguiente:
CÁTEDRA: BIOQUÍMICA Carreras: Farmacia Profesorado en Química Licenciatura en Química Lic. en C. y T. de los Alimentos ÁCIDOS NUCLEICOS 1) a) En la figura señale, englobando con un trazo continuo, lo siguiente:
Validación de métodos, ensayos de aptitud y biometrología en OGM
 Validación de métodos, ensayos de aptitud y biometrología en OGM AMOUNT OF DNA 8 256 PCR Punto 9 Final 512 10 1,024 11 2,048 12 4,096 CYCLE No. de NUMBER ciclo 13 AMOUNT Copias OF de DNA ADN 8,192 140
Validación de métodos, ensayos de aptitud y biometrología en OGM AMOUNT OF DNA 8 256 PCR Punto 9 Final 512 10 1,024 11 2,048 12 4,096 CYCLE No. de NUMBER ciclo 13 AMOUNT Copias OF de DNA ADN 8,192 140
Fecha de elaboración: 25 de mayo de 2010 Fecha de última actualización: 27 de mayo de 2010
 PROGRAMA DE ESTUDIO TECNICAS AVANZADAS DE BIOLOGIA MOLECULAR Programa elaborado por: Programa Educativo: Licenciatura en Biología Área de Formación : Sustantiva Profesional Horas teóricas: 2 Horas prácticas:
PROGRAMA DE ESTUDIO TECNICAS AVANZADAS DE BIOLOGIA MOLECULAR Programa elaborado por: Programa Educativo: Licenciatura en Biología Área de Formación : Sustantiva Profesional Horas teóricas: 2 Horas prácticas:
Universidad de Los Andes Facultad de Medicina Instituto Inmunología Clínica Maestría en Inmunología
 TÉCNICAS EN BIOLOGÍA MOLECULAR RESPONSABLE: Dra. Lisbeth Berrueta. CREDITOS: 2 El curso de Técnicas de Biología Molecular está destinado a profesionales del área biomédica con conocimientos básicos relativos
TÉCNICAS EN BIOLOGÍA MOLECULAR RESPONSABLE: Dra. Lisbeth Berrueta. CREDITOS: 2 El curso de Técnicas de Biología Molecular está destinado a profesionales del área biomédica con conocimientos básicos relativos
CATÁLOGO DE SERVICIOS Y PRECIOS
 CATÁLOGO DE SERVICIOS Y PRECIOS ANÁLISIS DE SUELO. FERTILIDAD DETERMINACIONES POR MUESTRA/CONCEPTO Densidad aparente 35 ph 30 Conductividad eléctrica 30 Bases intercambiables (Calcio, 60 c/u Magnesio,
CATÁLOGO DE SERVICIOS Y PRECIOS ANÁLISIS DE SUELO. FERTILIDAD DETERMINACIONES POR MUESTRA/CONCEPTO Densidad aparente 35 ph 30 Conductividad eléctrica 30 Bases intercambiables (Calcio, 60 c/u Magnesio,
CYP 2C9 A1075C CYP2C9*3
 CYP 2C9 A1075C Sistema para la detección de la mutación A1075C en el gene del citocromo P450 2C9. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy
CYP 2C9 A1075C Sistema para la detección de la mutación A1075C en el gene del citocromo P450 2C9. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy
DESARROLLO DE TÉCNICAS MOLECULARES PARA LA IDENTIFICACIÓN DE BACTERIAS ACÉTICAS
 DESARROLLO DE TÉCNICAS MOLECULARES PARA LA IDENTIFICACIÓN DE BACTERIAS ACÉTICAS José M. Guillamón Departamento de Bioquimica i Biotecnologia Facultat d Enologia de Tarragona Universitat Rovira i Virgili
DESARROLLO DE TÉCNICAS MOLECULARES PARA LA IDENTIFICACIÓN DE BACTERIAS ACÉTICAS José M. Guillamón Departamento de Bioquimica i Biotecnologia Facultat d Enologia de Tarragona Universitat Rovira i Virgili
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR
 KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
TIPIFICACIÓN MOLECULAR
 TIPIFICACIÓN MOLECULAR TIPIFICACION MOLECULAR APLICACIONES GENERALES TIPIFICACIÓN MOLECULAR Son todas iguales? Para que diferenciarlas? APLICACION EN MICROBIOLOGIA CLINICA Y EPIDEMIOLOGIA Confirmación
TIPIFICACIÓN MOLECULAR TIPIFICACION MOLECULAR APLICACIONES GENERALES TIPIFICACIÓN MOLECULAR Son todas iguales? Para que diferenciarlas? APLICACION EN MICROBIOLOGIA CLINICA Y EPIDEMIOLOGIA Confirmación
PALINDROMOS: SECUENCIAS ESPECIALES DEL ADN
 PALINDROMOS: SECUENCIAS ESPECIALES DEL ADN En el genoma existen secuencias denominadas palindrómicas o simplemente palíndromos. Podrían considerarse como un tipo especial de secuencias capicúas. Lo propio
PALINDROMOS: SECUENCIAS ESPECIALES DEL ADN En el genoma existen secuencias denominadas palindrómicas o simplemente palíndromos. Podrían considerarse como un tipo especial de secuencias capicúas. Lo propio
Invest Clin 40(4): 257-266, 1999
 Invest Clin 40(4): 257-266, 1999 Análisis de la prevalencia de mutaciones puntuales en el codón 12 del oncogen K-ras en muestras no cancerosas de citología exfoliativa cervical positivas para VPH tipo
Invest Clin 40(4): 257-266, 1999 Análisis de la prevalencia de mutaciones puntuales en el codón 12 del oncogen K-ras en muestras no cancerosas de citología exfoliativa cervical positivas para VPH tipo
