CLONAJE DE GENES EN PLÁSMIDOS BACTERIANOS: Construcción del DNA recombinante
|
|
|
- María Montoya Ríos
- hace 8 años
- Vistas:
Transcripción
1 Construcción DNA recombinante 1 Práctica 1 CLONAJE DE GENES EN PLÁSMIDOS BACTERIANOS: Construcción del DNA recombinante Introducción Los plásmidos bacterianos son moléculas de DNA extracromosómico capaces de replicar autónomamente. La mayoría están constituidos por un DNA bicatenario circular cerrado de tamaño muy variable (1 a >200 kb), que se puede aislar de la bacteria en forma superenrollada. Se encuentran en una gran variedad de especies bacterianas, pero suelen tener un rango de hospedador estrecho. La replicación y transcripción de los plásmidos dependen, en mayor o menor extensión, de proteínas codificadas por genes de la bacteria. A su vez, los plásmidos contienen con frecuencia genes que codifican enzimas útiles para la bacteria hospedadora como genes de resistencia a o producción de antibióticos, de degradación de compuestos orgánicos, de toxinas, y de producción de enzimas de restricción y modificación, entre otros. Los plásmidos pueden utilizarse como vectores de clonaje y amplificación de fragmentos de DNA que se hayan insertado en los mismos mediante técnicas de Ingeniería Genética. Estos fragmentos replican y se transmiten a las células hijas conjuntamente con el resto del plásmido. Los primeros plásmidos usados como vectores eran de origen natural, pero en la actualidad se usan plásmidos modificados en los que se han introducido características útiles para el clonaje, a expensas de eliminar muchas secuencias innecesarias del plásmido original. Por ejemplo: 1. Marcadores de selección. Generalmente genes de resistencia a antibióticos como ampicilina, kanamicina, tetraciclina, etc. 2. Un sitio de policlonaje que es una secuencia sintética que contiene las secuencias reconocidas por varias enzimas de restricción. 3. Este sitio de policlonaje puede estar insertado dentro de un gen marcador lo que permite la selección directa de los recombinantes. Por ejemplo, existen plásmidos en los que el sitio de policlonaje se encuentra dentro de una secuencia de DNA que codifica un fragmento ( ) N-terminal de la -galactosidasa. En la Práctica 2 usaremos este gen marcador para la selección de transformantes. 4. El origen de replicación deriva normalmente del plásmido ColE1. Este origen permite conseguir un número de copias elevado del plásmido. Muchos plásmidos que se usan en Ingeniería Genética, como el que usamos en estas prácticas, contienen un segundo origen de replicación derivado de un fago con DNA monocatenario (M13, f1), que permite replicar una sola de las cadenas del plásmido originando copias monocatenarias del DNA clonado, útiles para secuenciación, mutagénesis, etc. 5. Promotores de fagos (T7, SP6) delante del sitio de policlonaje. Útiles para la transcripción in vitro del DNA clonado, o para su expresión regulada in vivo si el plámido recombinante se introduce en bacterias que expresan la RNA polimerasa viral. 6. En los vectores de expresión, se suelen introducir además de promotores regulables, para expresar el gen clonado en respuesta a cambios en las condiciones de cultivo, la secuencia Shine-Dalgarno ( ribosome binding site, RBS) que confiere alta eficiencia de traducción, y una secuencia de nucleótidos que codifica un pequeño polipéptido (una etiqueta ) que puede ser reconocido por anticuerpos específicos. El gen clonado y la etiqueta se expresan conjuntamente originando una proteína de
2 Construcción DNA recombinante 2 fusión, fácilmente detectable con los anticuerpos. También existen etiquetas (p.ej. 6 His) que permiten la purificación rápida de la proteína de fusión por cromatografía de afinidad. 7. Muchos plásmidos vectores contienen secuencias complementarias a primers universales que permiten secuenciar cualquier inserto introducido. Las enzimas de restricción del tipo II constituyen la herramienta enzimática esencial para la construcción de DNAs recombinantes. Se trata de endonucleasas que reconocen secuencias cortas y específicas en el DNA, cortándolo por dicha región siempre que determinadas bases de la secuencia no estén metiladas. Existen enzimas de restricción, denominadas isoesquizómeras, que reconocen total o parcialmente la misma secuencia. El corte puede producir extremos protuberantes (también denominados cohesivos) o romos. Se denominan con varias letras y un número romano. La primera letra se refiere al género, las dos siguientes a la especie y la última a la cepa de la bacteria de donde se aisló. Los números se usan cuando hay más de un sistema de restricción-modificación en la misma bacteria. Otras enzimas importantes en la construcción de DNAs recombinantes son las DNA ligasas, que catalizan la formación del enlace fosfodiéster entre los extremos 3 -OH y 5 -P adyacentes en un DNA, utilizando como cofactor ATP o NAD +. La más usada es la DNA ligasa del fago T4 que puede unir extremos cohesivos e incluso romos en presencia de ATP/Mg 2+. El fraccionamiento, identificación y purificación de moléculas de DNA se hace normalmente mediante electroforesis en geles de agarosa (o de poliacrilamida cuando se quieren resolver pequeños fragmentos de DNA de tamaños muy parecidos ya que tiene mayor nivel de resolución). La agarosa es un polímero lineal constituido por unidades alternantes de D- y L-galactosa unidas por enlaces glicosídicos -(1 3) y -(1 4). Se funde bien a temperaturas elevadas y al bajar la temperatura gelidifica formando un entramado tridimensional con poros de un diámetro entre 50 nm - >200 nm. En estos geles, las moléculas lineales de DNA migran hacia el polo positivo con una velocidad inversamente proporcional a su tamaño, dentro de ciertos límites. Estos geles también son útiles para distinguir diversas estructuras que puede formar una misma molécula de DNA bicatenaria, como DNA lineal, DNA circular cerrado superenrollado o DNA circular abierto. La movilidad del DNA lineal depende principalmente de su tamaño, de la presencia o no de agentes intercalantes, del voltaje aplicado, de la concentración iónica del tampón de electroforesis y de la concentración de agarosa, ya que a mayor concentración menor es el tamaño del poro formado. Por lo tanto, se deben usar concentraciones más altas a medida que se desea analizar moléculas más pequeñas de DNA. A los geles de agarosa se suele añadir bromuro de etidio (3,8 diamino-6-etil-5- fenilfenatridium bromuro) para visualizar el DNA. Este compuesto, que es un mutágeno y carcinógeno muy potente, se intercala entre las bases del DNA. Absorbe radiaciones a 302 y 366 nm (emitidas por las fuentes comerciales de luz ultravioleta), reemitiendo parte de la energía absorbida a 590 nm, que corresponde a la región rojo-naranja del espectro visible. Para la construcción de un plásmido recombinante que contenga el fragmento de DNA que deseamos clonar, el plásmido vector elegido se digiere con una o dos enzimas de restricción y, posteriormente, se incuba con el DNA que se desea clonar, conteniendo extremos compatibles con los extremos generados en el vector, y DNA ligasa.
3 Construcción DNA recombinante 3 Objetivos En esta primera práctica se pretende que el alumno obtenga una visión global de los pasos experimentales que hay que realizar para construir un plásmido recombinante constituido por el plásmido vector y un fragmento de DNA insertado en el mismo. En concreto, como vector elegimos inicialmente el plásmido de expresión prset-t7 ( por tener unas características muy interesantes que se discutirán en prácticas, y como fragmento de DNA a insertar un cdna que codifica el factor de iniciación de la traducción eif4e de Drosophila melanogaster. Este cdna fue aislado a partir de una genoteca de expresión de D. melanogaster construida en el fago lambda, y posteriormente clonado en el sitio EcoRI del plásmido pbluescript ( En consecuencia, el proceso de construcción del plásmido recombinante en esta Práctica consistiría en: 1) Digestión del plásmido recombinante pbluescript-cdna(eif4e) y del plásmido vector prset-t7 con EcoRI. Análisis cualitativo de los productos de la digestión mediante electroforesis en gel de agarosa. 2) Purificación del prset-t7 digerido mediante extracción con fenol/cloroformo y precipitación con etanol. 3) Aislamiento y purificación del cdna(eif4e) mediante electroforesis preparativa en gel de agarosa, elución de la banda correspondiente al cdna del gel y cromatografía. 4) Cuantificación de las concentraciones de ambos DNAs lineales purificados (prset-t7 y cdna) mediante electroforesis analítica en gel de agarosa. 5) Ligación de los dos DNAs lineales en presencia de DNA ligasa del fago T4. Sin embargo, dado el tiempo oficialmente permitido para el desarrollo del conjunto de prácticas (experimentales y de ordenador) de esta asignatura, se ha optado por hacer un protocolo experimental alternativo al anterior mucho más reducido, que se resume en los siguientes apartados: 1) Digestión del plásmido recombinante pbluescript-cdna(eif4e) con EcoRI. Análisis cualitativo de los productos de la digestión mediante electroforesis en gel de agarosa. 2) Purificación de los productos de la digestión (pbluescript y cdna) mediante extracción con fenol/cloroformo y precipitación con etanol. 3) Ligación de los dos DNAs lineales en presencia de DNA ligasa del fago T4. Es decir, se reconstruye el plásmido recombinante original. Es importante destacar que esta modificación del protocolo experimental tiene varias ventajas, adicionales al objetivo de reducción del tiempo, que se resumen en: a) La utilización del plásmido pbluescript nos permite hacer una doble selección (resistencia a ampicilina y color de las colonias) para distinguir los clones bacterianos que contienen el plásmido recombinante de los clones que contienen el plásmido recircularizado sin inserto según se verá en la Práctica 2. Por el contrario, el plásmido prset no permite esta doble selección. b) La ligación del DNA vector (pbluescript) con el cdna no se hace en una relación molar óptima lo que nos permite introducir al alumno aspectos de cuantificación de DNA, a los que está poco habituado, e interpretativos del resultado obtenido (% de bacterias con plásmido recombinante). Por otra parte, el protocolo reducido no ofrece ninguna desventaja significativa pues los alumnos siguen realizando todas las técnicas previstas en el protocolo inicial ya que una cromatografía similar a la suprimida en esta Práctica se realiza en la Práctica 3 y en las Prácticas 3 y 4 se hacen dos electroforesis adicionales en geles de agarosa.
4 Construcción DNA recombinante 4 Materiales Nota: Se utiliza material estéril. Material de vidrio y plástico se esteriliza en autoclave. Medios líquidos, a excepción de compuestos orgánicos (fenol, cloroformo, etanol), se esterilizan en autoclave o por filtración. Equipo y Materiales generales ubicados en las mesas y vitrinas laterales: Papel para secarse las manos; Jabón y escobillas; Papel de filtro; Pinzas; Cinta adhesiva de colores y para autoclave; Asas de siembra (6-12); Asas de extensión de plástico estériles (o de vidrio y vasos con etanol para esterilizarlas); Rotuladores para pizarra de varios colores y borrador; Probetas, vasos y matraces de vidrio/plástico de diversos tamaños; Pipetas estériles de 1 ml y 5 ml; Propipetas; Bolsas con tubos eppendorf de todos los tamaños y puntas de pipeta para reponer; Bolsas de plástico grandes para desechar material contaminado con Bromuro de Etidio (1) y placas de Petri usadas (1); Dos baños termostatizados con agitación a 37ºC: uno con gradilla para tubos de dilución y flotadores para tubos eppendorf; y el otro con abrazaderas para matraces de 100 ml y 25 ml; Frigorífico con congelador; Transiluminador luz UV y gafas protectoras (y/o pantalla protectora); Bloque térmico; Espectrofotómetro y cubetas de plástico; Pipetas Pasteur de plástico; Equipo PCR; Estufa a 30-37ºC; Balanza/granatario para equilibrar tubos con dos vasos; Centrifuga de mesa con rotor para tubos Falcon de 50 ml; Balanza de precisión; Papel de aluminio para pesar; Espátulas; Microondas y guantes aislantes(2); Maquina de hacer hielo y contenedores para hielo; Agarosa (Hispanaagar, low electroendosmosis). Equipo y Materiales generales ubicados en las mesas (3) de los alumnos: Papel de filtro (1/pareja); Guiones de prácticas (1/alumno); Gradillas plástico (1/pareja); Gradillas acero para tubos dilución (2/mesa); Gradillas para tubos eppendorf de 0,1 ml (2/mesa); Pipetas automáticas (1 juego/pareja); Puntas de pipeta estériles de tres tamaños (2 juegos/mesa); Contenedores para desecho de las puntas (2/mesa); Tubos eppendorf estériles en botes de vidrio de 1,5 ml, 0,5 ml y 0,1 ml (2 juegos/mesa); Rotuladores para tubos (2/mesa); Parafilm (1/mesa); Tijeras (1/mesa); Guantes tres tamaños (1 juego/mesa); H2O estéril (2 botellas de 50 ml /mesa); Equipos de electroforesis (1/mesa para la Practica 1; 2/mesa para las Prácticas 3+4); Fuentes de alimentación (1/mesa); Microfugas (1-2/mesa); Matraces de 250 ml (1/mesa) y probetas de 100 ml (1/mesa); Garrafa con H2O destilada (1/laboratorio); Mecheros de gas (2-4/mesa) y cerillas (2/mesa); Otros materiales necesarios para la Práctica 1: Plásmido recombinante pbluescript con el cdna(eif4e) ( ; search: nucleotide, for : U16139; referencia original: Hernández, G. & Sierra, J. M Biochim Biophys Acta 1261,427-31) clonado en el sitio EcoRI. Sin diluir y diluido 1/20. EcoRI y tampón H para la reacción de digestión. Consultar características en la información de la casa comercial que tiene el Profesor. Tampón TAE: 40 mm Tris-acetato, 1 mm EDTA. Bromuro de etidio (5 mg/ml). Marcadores de masa molecular de DNA del fago 29 digerido con HindIII.
5 Construcción DNA recombinante 5 Tampón de carga de electroforesis (6X) que contiene los colorantes bromophenol blue y xylene cyanol FF, así como sacarosa o glicerol en agua. Fenol (Sigma). Antes de la práctica preparar alícuotas de 0,5 ml en tubos eppendorf (2/mesa). Cloroformo (Merck). Antes de la práctica preparar alícuotas de 0,5 ml en tubos eppendorf (2/mesa). 3 M NaAc, ph 5,2. Antes de la práctica preparar alícuotas de 0,5 ml en tubos eppendorf (2/mesa). Etanol (Merck). Varias botellas conteniendo etanol absoluto y etanol al 70% en agua destilada estéril. Se guardan en el congelador del frigorífico. DNA ligasa del bacteriófago T4 y tampón de ligación. Consultar características en la información de la casa comercial que tiene el Profesor. Procedimiento A) Digestión del plásmido recombinante pbluescript-cdna(eif4e) con EcoRI. 1. Preparar el termoblock a 37ºC. 2. En un tubo eppendorf de 1,5 ml, rotulado con el número de la pareja, preparar la mezcla de incubación siguiente, añadiendo los componentes en el orden indicado (el alumno debe calcular previamente los volúmenes a añadir de DNA, tampón H y H2O estéril): Componente Concentración del stock Concentración o cantidad requerida en la mezcla de incubación Volumen a añadir H2O estéril l Tampón H 10X 1X l DNA (pblue-4e) 0,8 g/ l 4000 ng l EcoRI 10 unidades/ l 2 l* Volumen final 25 l * EcoRI se suministra en un tampón que contiene 50% glicerol. Las concentraciones superiores al 5% de glicerol en la mezcla de digestión pueden afectar a la actividad enzimática por lo que el volumen de EcoRI no debe superar 1/10 del volumen final de la mezcla de reacción. 3. Poner los tubos en el termoblock e incubar a 37ºC durante min. 4. Detener la reacción elevando la temperatura del termoblock a 65ºC e incubando las muestras durante 5 min. 5. Mantener las muestras en la nevera hasta su uso. B) Análisis de los productos de la digestión mediante electroforesis en gel de agarosa en presencia de bromuro de etidio. Notas de advertencia importantes sobre posibles peligros: El bromuro de etidio es un agente mutagénico y carcinogénico muy potente. Es obligatorio usar guantes para su manipulación y la de los geles, cubetas, puntas de
6 Construcción DNA recombinante 6 pipeta, etc., que hayan estado en contacto con este compuesto. Si hubiese que preparar una disolución stock habría que usar mascarilla para pesarlo. La electroforesis se realiza con corriente continua a 100 voltios por lo que una descarga eléctrica es peligrosa. Si fuese necesario manipular la cubeta durante la electroforesis hay que desenchufar previamente la fuente de alimentación. La visualización de las bandas de DNA en el gel de electroforesis se hace haciendo pasar luz ultravioleta a través del gel. Está absolutamente prohibido mirar el gel sin la debida protección con gafas y/o pantalla protectoras para evitar que la luz UV dañe la retina del ojo. Se preparará un único gel por mesa por lo que las operaciones que se indican a continuación se deben distribuir entre los alumnos de cada mesa como se considere más oportuno: 1. Preparar el soporte donde se colocará la agarosa sellándolo con cinta adhesiva en los dos laterales abiertos y colocar el peine adecuado en la posición indicada. Mantener encima de un papel de filtro fuera de la cubeta de electroforesis. 2. Una pareja de alumnos se encargará de preparar 2-3 litros de tampón TAE (1X) a partir de un stock TAE (50X) y de distribuir a todas las parejas y mesas. 3. Preparar el gel de agarosa al 1%: Pesar 0,7 g de agarosa y ponerla en un matraz de 250 ml. Añadir 70 ml de TAE (1X). Calentar en el microondas hasta que la agarosa quede fundida (unos 2 min). Sacar el matraz usando guantes aislantes. Dejar enfriar unos minutos y añadir 7 l de una disolución stock de bromuro de etidio de 5 mg/ml (concentración final en el gel: 0,5 g/ml). Verter inmediatamente en el soporte preparado según el apartado 1. Dejar unos 10 min en reposo para que el gel polimerice. Retirar con cuidado la cinta adhesiva y el peine e introducir el soporte con el gel en la cubeta de electroforesis ( dónde deben quedar situados los pocillos del gel con respecto a los electrodos de la cubeta?). Añadir suavemente tampón TAE (1X) hasta sobrepasar el nivel del gel. Nota: Normalmente el tampón TAE (1X) usado en la electroforesis se suplementa con bromuro de etidio (concentración final 0,5 g/ l) para mejorar la visualización de las bandas de DNA. Sin embargo, nosotros no lo añadimos para evitar la generación de grandes volúmenes de material de desecho que no pueden ser eliminados por la pila. En todo caso, la visualización de las bandas de DNA en nuestras condiciones es adecuada. 4. Aplicación de las muestras y electroforesis: Preparar ocho tubos eppendorf de 0,5 ml según se indica en la Tabla siguiente:
7 Construcción DNA recombinante l Mezcla de digestión Parejas 1(5, 9) Mezcla de digestión 3 l Parejas 2 (6, 10) pblue-4e sin digerir 6 l (dilución 1/20) Marcadores DNA 4 l ( 29) Mezcla de digestión 3 l Parejas 3 (7, 11) Mezcla de digestión 3 l Parejas 4 (8, 12) pblue-4e sin digerir (dilución 1/20) o Mezcla de digestión de una pareja Marcadores DNA ( 29) o Mezcla de digestión de una pareja H 2O (ajustar hasta 10 l) 7 l 7 l 4 l 6 l 7 l 7 l 4 l o 1 l 6 l o 9 l 3 l o 1 l 7 l o 9 l Tampón de carga 2 l 2 l 2 l 2 l 2 l 2 l 2 l 2 l (6X) Nota: Calcular los ng de DNA total presente en la mezcla de digestión y en la muestra del pblue-4e sin digerir que ha puesto en cada tubo. Aplicar suavemente los 12 l totales de cada muestra en los pocillos correspondientes del gel. Hay que evitar perforar el fondo del pocillo o que la muestra se salga por la parte superior del mismo. Cada alumno debe aplicar una muestra. Cerrar con cuidado la cubeta de electroforesis y conectarla a la fuente de alimentación. Correr la electroforesis a 100 V durante min (hasta que el colorante azul esté próximo al final del gel). El bromophenol blue migra aproximadamente como un DNA bicatenario lineal de 300 bp (prácticamente con el frente) y el xylene cyanol FF como un DNA bicatenario lineal de 4 kb. Parar la electroforesis y desconectar la cubeta de la fuente de alimentación. Sacar el soporte con el gel escurriéndolo bien y ponerlo encima de un papel de filtro doblado. Llevar el soporte con el gel al transiluminador y visualizar los DNAs haciendo pasar luz ultravioleta. NO mirar el gel sin gafas o pantalla protectora. Se pueden sacar fotografías con ayuda de un teléfono móvil que tenga cámara fotográfica. Analizar los resultados obtenidos y contestar a las preguntas que se formulan en el apartado de Resultados y cuestiones.
8 Construcción DNA recombinante 8 C) Purificación de los productos de la digestión (pbluescript y cdna) mediante extracción con fenol/cloroformo y precipitación con etanol. 1. Pasar la mezcla de digestión restante conteniendo los dos DNAs a un tubo eppendorf de 0,5 ml, estimando el volumen remanente. El tubo debe estar marcado con el número de pareja. 2. Eliminar la proteína contaminante añadiendo un volumen de fenol saturado y mezclar bien. 3. Centrifugar a rpm en microfuga durante 2 minutos a temperatura ambiente. NOTA: Como los tubos de 0,5 ml son mas pequeños que los huecos de la centrifuga hay que ponerlos DENTRO de un tubo eppendorf de 1,5 ml al que se ha cortado previamente la tapa. 4. Retirar la fase acuosa (la superior), con cuidado de no recoger la interfase que contiene la proteína desnaturalizada, y pasar a otro tubo eppendorf de 0,5 ml, marcado con el número de pareja, estimando el volumen recogido. 5. Si el volumen de esta fase acuosa es superior a 15 l (si es inferior NO es aconsejable hacer la extracción con cloroformo), añadir un volumen igual de cloroformo, mezclar bien y repetir la centrifugación y obtención de la fase acuosa como antes (pasos 3 y 4). El cloroformo elimina los restos de fenol que queden en la fase acuosa. 6. Precipitar el DNA añadiendo a la fase acuosa final (la obtenida después de la extracción con fenol o con cloroformo) 1/10 vol final de 3M NaAc, ph 5.2 y 2 volúmenes de etanol absoluto (enfriado a 20ºC). Mezclar suavemente y guardar en el congelador del frigorífico hasta el día siguiente. 7. Al día siguiente, sacar los tubos del congelador y centrifugar a rpm en microfuga durante 15 minutos. Marcar la posición del tubo en la microfuga. Decantar el sobrenadante sobre un papel de filtro. 8. Lavar el precipitado de DNA añadiendo 300 l de etanol al 70%. Agitar suavemente y centrifugar de nuevo en la microfuga durante 5 minutos. Decantar el sobrenadante sobre un papel de filtro y con golpecitos suaves para eliminar el resto de líquido. 9. Dejar secar a temperatura ambiente unos 5 minutos, manteniendo el tubo abierto entre los dedos. 10. Resuspender el DNA en 20 l de agua destilada estéril. Guardar en nevera hasta su uso. Los tubos deben seguir marcados con el número de la pareja. D) Ligación de los DNAs linearizados y desproteinizados (cdna del eif4e y plásmido pbluescript). 1. Preparar un tubo eppendorf de 0,1 ml marcado con el número de cada pareja conteniendo los componentes siguientes, añadidos en el orden que se indica: Componente Volumen H2O estéril 7 o 5 l Tampón para la ligasa (10X) 1 1 l Mezcla de DNAs resuspendidos 1 o 2 l en 20 l de H2O estéril (*) DNA ligasa de T4 (1 U/ l) 1 o 2 l Volumen final 10 l (*) Hay que tener presente que para favorecer la formación de moléculas recombinantes se debe usar, como mínimo, una razón molar inserto/vector de 3/1. Los alumnos deben
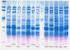
9 Construcción DNA recombinante 9 determinar la razón molar que usamos en este experimento y qué implicaciones tendrá en relación con los porcentajes de colonias bacterianas que se obtendrán conteniendo el plásmido recombinante y el plásmido recircularizado sin inserto. 2. Incubar a temperatura ambiente durante 60 minutos (normalmente la incubación se hace a 16ºC durante toda la noche). Resultados y cuestiones 1) a) Buscar la secuencia de nucleótidos del cdna del eif4e de Drosophila melanogaster usado en esta práctica en la base de datos del NCBI ( ; search: nucleotide, for : U16139). b) Fíjese en la secuencia de aa de la proteína codificada y deduzca los codones de iniciación y terminación de la traducción usados. c) Cómo buscaría vd el codón de iniciación de un mrna que codifica una proteína eucariótica?. Explique la respuesta. Podría aplicar el mismo criterio para un mrna procariótico?. Nota: El mapa de restricción de este cdna, con ayuda del programa informático accesible en la dirección: lo hará en las prácticas de Bioinformática de esta asignatura. Esperaría vd encontrar una diana para EcoRI dentro de este cdna?. Explique la respuesta. 2) Busque los mapas de los plásmidos pbluescript (Stratagene), usado en esta práctica, y prset-t7 (Invitrogen) y explique de forma concisa en su cuaderno de prácticas las características que le parecen más significativas y útiles de los mismos. 3) En la Figura se muestra un gel semejante al obtenido en esta Práctica: plásmido recombinante pbluescript-cdna(eif4e) digerido (canales 2, 3) o sin digerir (canales 4, 5) (160 ng, canales 2, 4 y 320 ng, canales 3, 5); marcadores de DNA de 29 digerido con HindIII (canales 1, 6) cuyos tamaños (en pb) y cantidades (en ng) se indican en la Tabla. En su cuaderno de prácticas dibuje un esquema del resultado real obtenido en su mesa y conteste a las preguntas siguientes: a) Hubo digestión del plásmido recombinante?, Fue una digestión parcial o completa?. b) Cuáles son los tamaños aproximados (en pb) de los DNAs obtenidos en la digestión?. Son los tamaños esperados?. c) Puede vd deducir alguna conclusión al comparar la movilidad del plásmido recombinante no digerido con respecto a la movilidad de los dos DNAs linearizados?. Explique su respuesta. d) Podría vd estimar, de forma aproximada, la cantidad de DNA presente en las bandas correspondientes al cdna(eif4e) y al pbluescript linearizado resultantes de la digestión?. Por qué disminuye la cantidad de DNA presente en las bandas al disminuir el tamaño del fragmento?. Fragmento Tamaño (pb) Cantidad (ng)
10 Construcción DNA recombinante 10 4) En el canal 5 de la Figura anterior, que corresponde a una muestra en la que se puso un exceso de plásmido recombinante sin digerir, se vislumbran bandas minoritarias con menor o mayor movilidad electroforética que la del plásmido recombinante. La mayoría de estas bandas parece que han desaparecido en la muestra digerida correspondiente (canal 3). a) Qué tipo de DNA puede estar presente en una banda minoritaria que migra más lentamente que el plásmido recombinante sin digerir?. b) Y en una banda que migra más rápidamente?. c) Las preguntas anteriores tienen varias contestaciones posibles, cómo podría vd distinguir experimentalmente cada una de estas posibilidades?. Razone con detalle sus respuestas. 5) a) Por qué hemos tenido que usar EcoRI para hacer la digestión y no otra enzima de restricción?. b) En el caso de que hubiésemos usado el prset como vector con qué enzima de restricción lo hubiésemos digerido?. Explique la respuesta. c) Escriba la secuencia diana de EcoRI. Qué tipo de extremos genera esta enzima?. Qué ventajas tienen este tipo de extremos para la clonación de fragmentos de DNA?. 6) En el laboratorio de prácticas hemos usado para la digestión 5 l de un stock 0,8 g/ l de pbluescript-cdna(eif4e) (4,2 kb). De los 25 l de la mezcla de digestión, se usaron 3 l para analizar los productos de la digestión y 22 l para la extracción con fenol/cloroformo seguida de precipitación con etanol. El DNA precipitado se resuspendió en 20 l de agua estéril. Suponga que durante todo el proceso anterior hemos recuperado solamente el 30% del DNA presente en los 22 l iniciales. Finalmente, en la reacción de ligación (volumen final, 10 l) hemos usado 1 o 2 l de los 20 l de disolución acuosa de DNA. Otros datos: la masa molar de 1 pb es 660 g/mol. a) Calcule los nanomoles del plásmido recombinante pbluescript-cdna(eif4e) añadidos a la mezcla de digestión. b) Cuántos nanomoles de pbluescript linearizado y de cdna(eif4e) hemos obtenido después de la digestión con EcoRI?. Y nanogramos de estos dos DNAs?. c) Cuántos nanogramos de pbluescript linearizado y de cdna(eif4e) estima vd que tenemos en los 20 l de disolución acuosa de DNA teniendo en cuenta el % de recuperación indicado?. Y nanomoles?. d) Qué cantidad (en nanogramos) de DNA total estima vd que tendremos en la mezcla de DNA ligado?. 7) a) Cuál es la proporción molar cdna(eif4e) / pbluescript que hemos usado en la reacción de ligación realizada en esta práctica?. b) Por qué se recomienda que la proporción molar inserto/vector sea al menos 3/1 para una reacción de ligación?. c) Qué otras dos estrategias se le ocurren para resolver los problemas que acaba de comentar?.
11 Transformación de bacterias 1 Práctica 2 CLONAJE DE GENES EN PLÁSMIDOS BACTERIANOS: Transformación de células competentes de Escherichia coli y selección de transformantes. Introducción Para el clonaje y amplificación del plásmido recombinante preparado en la práctica anterior es necesario introducirlo en una bacteria mediante un proceso que se denomina transformación. La mayoría de las bacterias, como por ejemplo E. coli, son impermeables a la entrada de ácidos nucleicos exógenos en condiciones normales de crecimiento. Sin embargo, se han desarrollado métodos químicos y físicos que facilitan este proceso al menos en E. coli. Todos los métodos químicos de transformación que se utilizan actualmente derivan de la observación de Mandel y Higa (1970, J. Mol. Biol. 53, ) según la cual las bacterias tratadas en frío con una disolución de CaCl2, y a continuación sometidas a un choque térmico a 42ºC, se hacen competentes y permiten la entrada del DNA del fago lambda. Este método se extrapoló posteriormente a DNA de plásmidos (Cohen et al., 1972, Proc. Natl. Acad. Sci. USA 69, ). Los métodos actualmente en uso son variaciones del original, encaminadas a optimizar la eficiencia de transformación de cada cepa bacteriana, que suele oscilar entre 10 6 a 10 9 transformantes por g de DNA plasmídico. Una posible explicación sobre el mecanismo molecular por el que las células se hacen competentes a la entrada de DNA exógeno lo puede ver en el siguiente enlace: Por otra parte, de entre los métodos físicos de transformación de bacterias podemos destacar la electroporación. Consiste en exponer las células de E. coli a una descarga eléctrica, que induce la formación transitoria de poros en las membranas por donde penetran las moléculas de DNA. Los plásmidos que estamos usando en estas prácticas de clonaje (pbluescript o prset-t7) tienen el gen de resistencia a ampicilina (bla). Este gen codifica una - lactamasa que hidroliza el anillo -lactámico de la ampicilina, inactivándola. La ampicilina es una aminopenicilina con actividad antimicrobiana de amplio espectro. Inhibe la síntesis del péptidoglicano, constituyente de la pared bacteriana, en células en fase activa de crecimiento. Las bacterias que contienen este tipo de plásmidos se pueden seleccionar creciéndolas en presencia de ampicilina. Obviamente, las bacterias que no han incorporado el plásmido no crecerán en este medio de cultivo. Para diferenciar las bacterias que han incorporado el plásmido recombinante de las que han incorporado el plásmido recircularizado sin inserto se pueden usar varios métodos. Uno de los más conocidos es el de la complementación del fragmento de la -galactosidasa. Este método requiere que el vector usado (por ejemplo, el plásmido pbluescript pero no así el plásmido prset-t7) contenga el sitio de policlonaje dentro de la región codificante del fragmento y la bacteria hospedadora exprese el otro fragmento de la -galactosidasa necesario para la complementación (lacz M15; presente normalmente en el profago 80 o en F ). En los plásmidos recombinantes, la incorporación del inserto inactiva el fragmento de forma que las bacterias que contienen estos plásmidos carecen de actividad -galactosidasa, originando colonias blancas en un medio de cultivo que contiene el sustrato incoloro X-GAL. Las bacterias
12 Transformación de bacterias 2 transformadas con plásmidos sin inserto aparecen como colonias azules en este medio pues son capaces de sintetizar -galactosidasa. Objetivos Preparación de bacterias competentes de E. coli, su transformación posterior mediante un choque térmico con los plásmidos obtenidos en la Práctica 1, y selección de los clones bacterianos conteniendo específicamente el plásmido recombinante pbluescript-cdna(eif4e) de Drosophila melanogaster. Uno de estos clones se utilizará en la Práctica 3. Materiales Equipo y Materiales generales: ver Práctica 1 Otros materiales necesarios para la Práctica 2: 2% (p/v) X-GAL (5-bromo-4-cloro-3-indolil- -D-galactósido) en dimetilformamida. 20% (p/v; 0,8 M) IPTG (isopropil-tio- -D-galactósido). Placas de Petri con medio LB (Luria-Bertani) sólido (10 g de triptona, 5 g de extracto de levadura, 10 g de NaCl, 15g de bacto agar, y agua destilada hasta 1000 ml) SIN ampicilina. Estas placas las utiliza el Profesor para crecer la bacteria usada en la transformación. Placas de Petri con medio LB (Luria-Bertani) sólido CON AMPICILINA (0,1 mg/ml). Para crecer bacterias transformadas. E. coli XL1-Blue (ver características en ) (1-2 placas de Petri con colonias/mesa). Medio LB líquido (igual composición que el LB sólido pero sin agar y SIN ampicilina): Tubos de dilución con 2 ml de medio y matraces de 100 ml con 25 ml de medio. Para crecer la bacteria antes de la transformación. Tubos Falcon de 50 ml (1/pareja). 0.1 M CaCl2 estéril (2 botellas/mesa). Mantener en frío (nevera; hielo). Mezcla de ligación realizada en la Práctica 1. Plásmido recombinante pbluescript-cdna(eif4e) sin digerir y diluido 1/80. Procedimiento A) Adición de IPTG y X-GAL a placas de Petri con medio LB + ampicilina 1. La tarde anterior al inicio de la práctica (día 1º) distribuir 3 placas de Petri conteniendo medio LB + ampicilina por pareja mas 2 placas por mesa. Cada pareja marcará en la base sus 3 placas con el número de pareja seguido de 1, 2, 3, respectivamente. Las dos placas sobrantes se marcaran con el número de la mesa. 2. Para cada mesa preparar en un tubo eppendorf de 1,5 ml, el premix que se indica en la última columna de la tabla siguiente: Componente l / placa l / 15 placas (1 mesa) X-GAL (2%) IPTG (20%) Volumen final
13 Transformación de bacterias 3 3. Poner 47 l del premix anterior en el centro de cada una de las placas de Petri y extender uniformemente con ayuda de un asa de plástico estéril. 4. Dejar secar las placas boca arriba y tapadas hasta el día siguiente. Si no se usaran este día se guardarán en la nevera. B) Crecimiento de Escherichia coli. 1. La tarde anterior al inicio de la práctica (día 1º) cada pareja picará una colonia de bacterias E. coli XL1-Blue de un placa de Petri conteniendo medio LB preparada recientemente y la transferirá a un tubo de dilución con tapón conteniendo 2 ml de medio LB (sin ampicilina). Sujetar el tapón del tubo con cinta aislante sin bloquear la entrada de aire al tubo, marcar con el número de pareja e incubar en baño a 37ºC con fuerte agitación durante toda la noche. 2. A la mañana siguiente (13:30-13:45 h), transferir 0,5-0,8 ml del cultivo crecido en cada tubo a sendos matraces de 100 ml conteniendo 25 ml de medio LB fresco precalentado a 37ºC. Incubar con fuerte agitación en baño a 37ºC. 3. A las 15:30-15:45 h, medir la A550 nm del cultivo. Parar el crecimiento cuando se alcance un valor 0,28-0,32, poniendo el matraz con el cultivo en hielo. Nota: En estas condiciones de crecimiento el número de bacterias duplica en unos 20 min. C) Preparación de células competentes. 1. Transferir el cultivo enfriado a un tubo Falcon de 50 ml estéril y mantener en hielo. 2. Equilibrar tubos de dos en dos con ayuda de una balanza/granatario. 3. Recuperar las bacterias por centrifugación a 5000 rpm durante 10 min. Tener cuidado de poner enfrentados los tubos que se han equilibrado previamente en parejas. 4. Decantar el sobrenadante rápidamente en la pila. Dejar el tubo invertido sobre el papel de filtro durante 1 min con objeto de eliminar los restos de medio. 5. Resuspender las bacterias en 5 ml de 0,1 M CaCl2 frío usando la propipeta P1000. Mezclar con suavidad con ayuda de la pipeta eliminando completamente todos los agregados de células. 6. Después de unos 20 min en hielo, recuperar las bacterias por centrifugación a 5000 rpm durante 10 min, y decantar de nuevo el sobrenadante como antes. 7. Resuspender las bacterias en 0,5 ml de 0.1 M CaCl2 frío y mantener en hielo. Notas: a) Dado que la centrifuga que disponemos en el laboratorio sólo tiene capacidad para 6 tubos Falcon de 50 ml, y el número de parejas por grupo es de 12, se puede reducir el número de centrifugaciones a la mitad mezclando los cultivos crecidos de dos parejas en un tubo Falcon. En este caso, las bacterias se resuspenderían inicialmente en 10 ml de de 0,1 M CaCl2 frío y después en 1,0 ml. Finalmente, cada pareja recuperaría 0,5 ml de esta última suspensión de bacterias. b) En el caso de que las bacterias competentes no se vayan a utilizar inmediatamente para la transformación se pueden dividir en alícuotas, congelar rápidamente y conservar a 70ºC siguiendo el protocolo siguiente: A 1 ml de bacterias añadir 35 l de DMSO (dimetilsulfóxido) y mezclar suavemente, manteniendo la suspensión en hielo durante 15 min. Añadir otros 35 l de DMSO, mezclar y volver a poner en hielo. Enfriar tubos eppendorf en hielo
14 Transformación de bacterias 4 seco/etanol (o nitrógeno líquido) y poner alícuotas de l de la suspensión de bacterias con DMSO en cada tubo. Las alícuotas se mantienen congeladas a -70ºC. Cuando se necesiten, descongelar el tubo con la mano y mantener en hielo durante 10 min. D) Transformación de las bacterias. 1. Preparar 3 tubos eppendorf de 1,5 ml por pareja en hielo. Marcarlos con el número de pareja y los números 1, 2 y 3, respectivamente. Añadir a cada uno de ellos los componentes que se indican en la tabla siguiente: Componente Tubo 1 Tubo 2 Tubo 3 Células competentes 100 l 100 l 100 l Mezcla ligación (práctica 1) 2 l pbluescript-eif4e sin digerir l (dilución 1/80) H2O estéril l 2. Mantener en hielo durante 30 min agitando suavemente la suspensión de vez en cuando. 3. Transferir los tubos a un bloque térmico a 42ºC y mantener 2 min. 4. Detener el choque térmico transfiriendo los tubos a contenedores con hielo. E) Crecimiento y selección de los transformantes. 1. Añadir a cada tubo 500 l de medio LB SIN ampicilina e incubar a 37ºC con agitación intensa durante 1 hora. Si la incubación se hace sin agitación, conviene agitar las células a intervalos de tiempo para resuspenderlas. 2. Plaquear alícuotas de 100 l de cada uno de los tubos en las placas de Petri correspondientes (es decir, tubo 1 a placa 1, etc.) conteniendo medio LB sólido CON ampicilina + IPTG + X-GAL preparadas el día anterior. Extender uniformemente la suspensión de bacterias sobre la superficie de la placa con ayuda de un asa de plástico estéril (usar un asa distinta para cada placa). Mantener las placas boca arriba y tapadas hasta que sequen bien. 3. Una vez secas, invertir las placas, comprobar que están debidamente marcadas e incubarlas en estufa a 37ºC durante unas horas. 4. Recoger las placas de la estufa y analizar los resultados: número y color de las colonias; uniformidad de las colonias en la placa; etc. Resultados y Cuestiones 1) a) Busque las características de la cepa bacteriana usada en esta práctica e interprételas brevemente en su cuaderno. b) Cuál le parece más imprescindible para la selección de clones recombinantes que hemos realizado en este experimento?. 2) Para qué se crecen las bacterias en medio LB líquido sin ampicilina durante una hora antes de pasarlas al medio sólido que contiene ampicilina (+IPTG + X-GAL)?. 3) a) Por qué se crecen las bacterias durante toda la noche en un medio sólido con ampicilina?. b) Qué resultado esperaría vd encontrar si nos hubiésemos olvidado de añadir ampicilina al medio de cultivo?.
15 Transformación de bacterias 5 4) a) Para qué usamos IPTG y X-GAL en el medio de cultivo?. b) Qué tipo de colonias (blancas o azules) aparecen mas frecuentemente en su placa 1?. Por qué?. c) De qué color son las colonias que aparecen en la placa 2?. Por qué?. d) Para qué nos sirve el análisis de la placa de Petri número 3 procedente del tubo 3?. Interprete los resultados que vd ha obtenido en dicha placa. 5) El método de selección de bacterias conteniendo plásmidos recombinantes basado en la complementación del fragmento falla en ocasiones, observándose resultados contrarios a lo esperado: colonias azules de bacterias transformadas con el plásmido recombinante y colonias blancas de bacterias transformadas con el plásmido sin inserto. A qué se pueden deber estos resultados?. 6) Determine la eficiencia de transformación (número de colonias/ g de DNA) utilizando la placa de Petri número 2 procedente de la transformación con el plásmido recombinante pbluescript-cdna(eif4e) no digerido y diluido 1/80 (tubo 2) suponiendo que en esta placa ha obtenido 100 colonias. 7) Podríamos hacer una estimación de la cantidad de DNA (plásmido recombinante + plásmido sin inserto) presente en la mezcla de ligación de la Práctica 1 usada en la transformación del tubo 1 observando los resultados de la placa 1?. Explique su respuesta y haga los cálculos en su cuaderno de prácticas suponiendo que en esta placa ha obtenido 70 colonias.
16 Práctica 3 CLONAJE DE GENES EN PLÁSMIDOS BACTERIANOS: Aislamiento de DNA plasmídico y análisis del mismo por digestión con enzimas de restricción. Introducción La aparición de colonias blancas en placas con ampicilina, si bien es un buen indicio, no garantiza que contengan el vector recombinante, ya que puede tratarse de falsos positivos, como sería el caso de dímeros de vectores religados o la presencia de mutaciones en la -galactosidasa. Por esa razón es imprescindible, después de la selección, hacer un análisis de los clones para comprobar la presencia del inserto digiriendo el DNA plasmídico con enzimas de restricción y analizando los fragmentos resultantes por electroforesis en gel de agarosa. Una vez comprobada la presencia del inserto, en algunas ocasiones hay que secuenciarlo para asegurarse que no contiene mutaciones. Los vectores de clonaje tienen secuencias flanqueantes al sitio de policlonaje complementarias a primers denominados universales, pues sirven para cualquier inserto. Objetivos Los clones recombinantes seleccionados en presencia de ampicilina y de color blanco debido a la inactivación de la -galactosidasa, se crecerán en medio LB en presencia de ampicilina y se aislará el DNA plasmídico para su posterior análisis. El análisis estructural de los plásmidos recombinantes tiene un doble objetivo: 1) la identificación y confirmación de la presencia del inserto en los clones que contengan el vector y 2) la determinación de la orientación del mismo. Todo ello se llevará a cabo mediante análisis de restricción del DNA plasmídico e identificación de los fragmentos resultantes por electroforesis en agarosa. Materiales Además del material general descrito en la Práctica 1, en esta práctica utilizaremos: - Erlenmeyer con 5 ml de medio LB conteniendo 100 µg/ml de ampicilina. - Kit Wizard Plus SV Minipreps (Promega). - Enzimas de restricción Eco RI, Bam HI y Kpn I, y sus correspondientes tampones. - Para la electroforesis necesitamos: Agarosa Tampón TAE: 40 mm Tris-acetato, 1 mm EDTA. GelRed DNA Molecular Weight Marker XVI, 250bp ladder Solución de carga de electroforesis conteniendo azul de bromofenol y xylene cyanol FF. Procedimiento A) Crecimiento de clones bacterianos. (dia 2) Coger una colonia blanca seleccionada de placas con ampicilina, IPTG y X-gal con un asa de cultivo e inocular un erlenmeyer con 5 ml de LB conteniendo 100 µg/ml de ampicilina. Crecer a 37 C con agitación durante la noche. B) Aislamiento del DNA plasmídico (Miniprep). (dia 3) Al día siguiente se aislará el DNA plasmídico a pequeña escala por el método de la lisis alcalina utilizando el Kit comercial Wizard Plus SV Minipreps (Promega). Información suministrada por el fabricante en: Para ello se seguirá el siguiente protocolo: - Poner 1.5 ml del cultivo crecido durante la noche en un tubo eppendorf. Recoger las células por centrifugación a máxima velocidad en minifuga 3 min. Tirar el sobrenadante, escurrir el tubo boca abajo sobre papel de filtro. - Resuspender en Vortex o con micropipeta el sedimento celular en 250 l de solución R (50 mm Tris-HCl ph 7.5, 10 mm EDTA, 100 g/ml RNasa A). QUE NO QUEDEN GRUMOS. - Lisar las células con 250 l de solución L (0.2 M NaOH, 1% SDS), mezclar suavemente por inversión. Observar el aumento de viscosidad. NO DEJARLO MÁS DE 5 min, pues el DNA desnaturalizado es mucho más frágil que el nativo.
17 - Degradar las proteínas del lisado con 10 l de solución P (proteasa alcalina), mezclar por inversión. NO DEJARLO MÁS DE 5 min. - Neutralizar con 350 l de solución N (4.09 M hidrocloruro de guanidina, M acetato potásico, 2.12 M acido acético), mezclar por inversión. Aparecerá un abundante precipitado blanco correspondiente al DNA bacteriano, que queremos eliminar. - Centrifugar 10 min. - En este paso hay que tener especial cuidado, pues es el más crítico. Transferir cuidadosamente por decantación o con micropipeta el sobrenadante claro conteniendo el DNA plasmídico al filtro suministrado en el Kit, adaptado a un tubo colector. Debemos coger CUANTO MAS SOBRENADANTE MEJOR PERO EVITANDO QUE NO CAIGA EL PRECIPITADO BLANCO. En caso de que esto ocurra, devolverlo al tubo y centrifugar otra vez. Repetir el proceso. - Centrifugar 1 min el material del filtro adaptado al tubo colector. Previamente dar dos o tres pulsos (centrifugación muy corta). El filtro no se tapa, pues la fuerza centrífuga impide que se salga el líquido. En estas condiciones el DNA plasmídico queda retenido en el filtro. - Vaciar el tubo colector, lavar el filtro con 750 l de solución W (8.3 mm Tris-HCl, 0.04 mm EDTA, 60 mm acetato potásico, 60% etanol), centrifugar 1 min. - Repetir el proceso lavando con 250 l de solución W, centrifugar 2 min. - Poner el filtro, asegurándose de que no tiene solución W, en un eppendorf nuevo al que previamente habremos cortado la tapa. La tapa la guardaremos para después de las centrifugaciones.. - Extraer el plásmido del filtro con 60 l de agua sin nucleasas (H). Dejar unos instantes y dar dos o tres pulsos antes de centrifugar 1 min. C) Análisis de restricción del plásmido: El análisis se realizará mediante digestión con tres enzimas de restricción: Bam HI, Eco RI y Kpn I. El DNA plasmídico se distribuirá en tres tubos marcados E, B y K para digerirlos con Eco RI, Bam HI y Kpn I, respectivamente, según el siguiente protocolo en el que cada enzima se incuba con su propio tampón recomendado por el fabricante para optimizar la digestión: tubo DNA plasmídico Tx10 enzima E 17 µl 2 µl TE 1 µl E B 17 µl 2 µl TB 1 µl B K 17 µl 2 µl TK 1 µl K Los tubos, con un volumen total de 20 µl, se incubarán a 37ºC al menos durante 1 h. D) Separación de fragmentos de restricción por electroforesis en geles de agarosa (día 4) Los fragmentos obtenidos en las digestiones enzimáticas se analizarán por electroforesis análogamente a como se describió en la práctica 1 Procedimiento B). - Preparar un gel de agarosa de 70 ml al 0.7% (0.49 gr) en TAE 1X, utilizando el peine de 15 pocillos. Añadir a la agarosa fundida 2 l de GelRed. - Añadir 4 µl de solución de carga a las muestras correspondientes a las digestiones enzimáticas, y cargar todas las muestras en el gel de electroforesis. Cargar en un extremo, 18 µl de marcadores de tamaño (Molecular Weight Marker XVI, 250bp ladder). - Correr el gel a 100V, hasta que el azul de bromofenol esté a 1/3 del final del gel, visualizarlo al ultravioleta. E) Análisis y discusión de los resultados. - Calcular, a partir de la posición de las bandas en el gel, el tamaño de los fragmentos obtenidos tras la digestión con las distintas enzimas de restricción. - Determinar la orientación del inserto.
18 Cuestiones 1) Deducir el número y tamaño de los fragmentos obtenidos tras la digestión con Eco RI, Bam HI y Kpn I (ver Apéndice, considerar despreciable el tamaño del sitio de policlonaje). 2) Con qué otros enzimas podríamos determinar la orientación del inserto? (ver Apéndice) 3) Buscar en el Apéndice la secuencia codificante del eif-4e (primer ATG). Teniendo en cuenta la fase de lectura y el código genético, determinar: a) el nº de aminoácidos de la proteína. b) la secuencia de los 5 aminoácidos N-terminales. c) la secuencia de los 5 aminoácidos C-terminales. 4) Con los datos obtenidos en la cuestión 3, diseña un primer de 10 nucleótidos para obtener por mutagénesis dirigida el cambio Arg 7 -Ser. 5) Determinar si en el recombinante con la orientación correcta el gen de la proteína eif-4e está en fase con el de la -galactosidasa (ver Apéndice). En caso negativo a) Cuántos aminoácidos tendría la proteína de fusión resultante?, b) Cómo se podrían poner en fase el gen de la -galactosidasa y el cdna del F-4E?. 6) Teóricamente el inserto se podría colocar en cualquier orientación, sin embargo en el análisis de los recombinantes mayoritariamente se obtiene una. Podrías proponer una hipótesis para explicar este resultado?. 7) Diseña los primers para insertar de forma unidireccional (empleando dos enzimas distintas) el cdna de eif-4e en el plásmido pbluescript IIKS, y detalla el procedimiento a seguir en el clonaje. Qué condiciones deben cumplir las enzimas seleccionados?.
19 PRÁCTICA 4 DETECCIÓN DE AGENTES INFECCIOSOS POR PCR (REACCIÓN EN CADENA DE LA POLIMERASA) Introducción Pocos avances en genética molecular han tenido tantos campos de aplicación como la PCR (reacción en cadena de la polimerasa, vulgarmente conocida como prueba del ADN), de ahí que, aunque existían precedentes, a quien la desarrolló en su forma actual, Kary Mullis, se le concediera el Premio Nobel en La idea es muy simple, la hibridación de unos primers flanqueantes a la secuencia a amplificar y su posterior elongación, permite la duplicación de su secuencia. Si la elongación se efectúa con una DNA polimerasa termoestable, la desnaturalización de los productos elongados y la repetición cíclica del proceso permite una enorme amplificación de la secuencia inicial, veces después de 40 ciclos. A pesar de su simplicidad hay ciertos parámetros que hay que tener muy en cuenta para una correcta amplificación. En primer lugar es esencial la elección de los primers, que han de hibridar con una secuencia única, lo que se logra con un tamaño suficientemente largo (20-25 nts), no han de presentar complementariedad inter o intra-molecular, Tms similares, y a ser posible con pares GC en sus extremos para una mayor estabilidad. Los mejores resultados se obtienen con primers distantes entre si, de 100 a 500 nucleótidos. En algunas ocasiones no existe una total complementariedad, como es el caso de la mutagénesis dirigida, donde el nucleótido cambiado, la deleción o inserción presente en el primer, debe colocarse en el centro de la secuencia, nuevamente para mayor estabilidad. Otro parámetro crucial es la temperatura de hibridación de los primers, unos 5-10 C por debajo de su temperatura de fusión (Tm). Una temperatura demasiado alta daría lugar a una pobre amplificación, por el contrario una temperatura demasiado baja podría dar lugar a hibridaciones inespecíficas con secuencias similares, obteniéndose múltiples productos.. Otro paso importante a tener en cuenta es el tiempo de extensión empleado, dado que la mayoría de las DNA polimerasas elongan en un minuto alrededor de 1 Kb de DNA, si se amplificasen fragmentos de mayor tamaño debe aumentarse dicho tiempo. Igualmente se puede amplificar una secuencia de RNA utilizando previamente la transcriptasa inversa para copiarla a DNA, esta técnica, RT-PCR (reverse transcripcion PCR) de amplia utilización, permite detectar cantidades minúsculas de RNA. La PCR no solo sirve para amplificar una secuencia, la PCR cuantitativa permite determinar la concentración inicial de un DNA (qpcr) ó un RNA (qrt-pcr) en una muestra. Utilizando agentes intercalantes fluorescentes podemos monitorizar la amplificación en tiempo real y, con DNAs de concentración conocida utilizados como estándar determinar la concentración inicial de nuestra muestra. Los campos de aplicación de la PCR son enormes, éstos son solo algunos ejemplos: en investigación es una herramienta imprescindible de uso cotidiano, la mutagénesis dirigida mencionada antes, la detección de agentes infecciosos, objetivo de esta práctica, diagnosis de enfermedades o riesgo potencial de padecerlas, pruebas de paternidad y aplicaciones forenses, tanto de identificación de restos biológicos como de pruebas criminalísticas. Objetivos El objetivo de esta práctica es el aprendizaje de la técnica de la PCR y su aplicación en la detección de agentes infecciosos. En este caso el agente infeccioso es un peligroso virus que contamina unas muestras de agua (en realidad esto es una simulación, pues el virus utilizado es el bacteriófago Ø29 que infecta a Bacilus subtillis y por tanto totalmente inocuo). Se suministrarán dos muestras, una infectada con el virus y otra no, marcadas una de color rojo y otra azul, y mediante esta técnica se determinará cuál de las muestras está infectada, si la roja o la azul. En esta práctica se explicarán las bases teóricas de la técnica y se indicarán los parámetros a tener en cuenta para el diseño de los primers de la reacción. La reacción en cadena de la polimerasa es la técnica estándar para la amplificación de un fragmento de DNA incluido en un DNA molde, utilizando oligonucleótidos de secuencia única (primers) complementaria a ambos lados del fragmento que se desea amplificar. Esta amplificación se lleva a cabo mediante una serie de ciclos, generalmente 30, cada uno de los cuales consta de tres etapas: 1) Desnaturalización del DNA; 2) Hibridación de los primers y 3)
ELECTROFORESIS BASICA
 Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
ELECTROFORESIS AVANZADA
 Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
39. Aislamiento y purificación del DNA de un plásmido recombinante
 39. Aislamiento y purificación del DNA de un plásmido recombinante Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento de Bioquímica
39. Aislamiento y purificación del DNA de un plásmido recombinante Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento de Bioquímica
3. COMPONENTES. Tampón de electroforesis concentrado 2 x 50 ml 10 X (2 envases 500ml)
 PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS
 Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico
 17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA. Grado en Medicina
 DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz porosa
 INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES
 PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL
 LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
Práctico: Genética bacteriana 2007.
 Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I
 UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I DRAS. MARÍA LUISA ORTIZ Y ANA PEDREGOSA 1 PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA
UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I DRAS. MARÍA LUISA ORTIZ Y ANA PEDREGOSA 1 PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA
Técnicas de ADN recombinante: la manipulación genética
 Técnicas de ADN recombinante: la manipulación genética A partir de los años 70 se desarrollaron las herramientas de la biología molecular o la ingeniería genética o lo que se ha llamado técnicas del ADN
Técnicas de ADN recombinante: la manipulación genética A partir de los años 70 se desarrollaron las herramientas de la biología molecular o la ingeniería genética o lo que se ha llamado técnicas del ADN
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
DANAGENE SALIVA KIT. Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones
 DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR
 PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
DETERMINACIÓN DEL FACTOR Rh por PCR
 Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR
 TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Andrés Ciocchini, Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN) por partición diferencial
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Andrés Ciocchini, Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN) por partición diferencial
3/22/2010. Iván Ferrer Rodríguez, PhD Catedrático. En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR.
 Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
AUTOR: JORGE CONTRERAS PINEDA 1. Titulo:
 Página 1 de 1 1. Titulo: Electroforesis de DNA 2. Objetivo Conocer los principios básicos de la electroforesis horizontal en geles de agarosa y aplicarlo para la separación de DNA humano, plasmídico, recombinante
Página 1 de 1 1. Titulo: Electroforesis de DNA 2. Objetivo Conocer los principios básicos de la electroforesis horizontal en geles de agarosa y aplicarlo para la separación de DNA humano, plasmídico, recombinante
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O. Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua
 C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
V MATERIALES Y MÉTODOS
 V MATERIALES Y MÉTODOS 5.1 Aislamiento de bacterias e identificación 5.1.1 Cepas tomadas del cepario de la Universidad de las Américas-Puebla En el cepario del Departamento de Química y Biología hay algunas
V MATERIALES Y MÉTODOS 5.1 Aislamiento de bacterias e identificación 5.1.1 Cepas tomadas del cepario de la Universidad de las Américas-Puebla En el cepario del Departamento de Química y Biología hay algunas
PRÁCTICA 5. CALORIMETRÍA
 PRÁCTICA 5. CALORIMETRÍA INTRODUCCIÓN Al mezclar dos cantidades de líquidos a distinta temperatura se genera una transferencia de energía en forma de calor desde el más caliente al más frío. Dicho tránsito
PRÁCTICA 5. CALORIMETRÍA INTRODUCCIÓN Al mezclar dos cantidades de líquidos a distinta temperatura se genera una transferencia de energía en forma de calor desde el más caliente al más frío. Dicho tránsito
DANAGENE RNA PURIFICATION KIT
 DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
BIOTECNOLOGÍA. 1.- Ingeniería Genética, ADN recombinante
 BIOTECNOLOGÍA 1.- Ingeniería Genética, ADN recombinante Técnica del ADN recombinante: es la más usada en ingeniería genética, esta basada en el uso de las enzimas de restricción o endonucleasas de restricción
BIOTECNOLOGÍA 1.- Ingeniería Genética, ADN recombinante Técnica del ADN recombinante: es la más usada en ingeniería genética, esta basada en el uso de las enzimas de restricción o endonucleasas de restricción
5. MATERIALES Y MÉTODOS
 5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN
 NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN 1. PROCEDIMIENTOS BÁSICOS: En este apartado pretendemos detallar algunos factores muy importantes a la hora de realizar
NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN 1. PROCEDIMIENTOS BÁSICOS: En este apartado pretendemos detallar algunos factores muy importantes a la hora de realizar
Práctica 5 CINÉTICA ENZIMÁTICA: DETERMINACIÓN ESPECTOFOTOMÉTRICA DE LA CONSTANTE DE MICHAELIS-MENTEN DE LA PAPAÍNA
 Práctica 5 CINÉTICA ENZIMÁTICA: DETERMINACIÓN ESPECTOFOTOMÉTRICA DE LA CONSTANTE DE MICHAELIS-MENTEN DE LA PAPAÍNA 1. Objetivo Se pretende calcular la constante de Michaelis-Menten (K M ), la constante
Práctica 5 CINÉTICA ENZIMÁTICA: DETERMINACIÓN ESPECTOFOTOMÉTRICA DE LA CONSTANTE DE MICHAELIS-MENTEN DE LA PAPAÍNA 1. Objetivo Se pretende calcular la constante de Michaelis-Menten (K M ), la constante
LABORATORIO DE QUÍMICA ANALÍTICA E INSTRUMENTAL 502503. GUÍA No 2.3- METODOS DE SEPARACIÓN POR DESTILACIÓN
 LABORATORIO DE QUÍMICA ANALÍTICA E INSTRUMENTAL 502503 GUÍA No 2.3- METODOS DE SEPARACIÓN POR DESTILACIÓN I. EL PROBLEMA Dos líquidos completamente miscibles se pueden separar por métodos físicos llamados
LABORATORIO DE QUÍMICA ANALÍTICA E INSTRUMENTAL 502503 GUÍA No 2.3- METODOS DE SEPARACIÓN POR DESTILACIÓN I. EL PROBLEMA Dos líquidos completamente miscibles se pueden separar por métodos físicos llamados
PRÁCTICA Nº 5 REACCIONES DE POLIMERIZACIÓN. Objetivos
 PRÁCTICA Nº 5 REACCINES DE PLIMERIZACIÓN bjetivos - Realizar un ejemplo práctico de reacción de polimerización por condensación: Preparación de un poliéster. Preparación de una poliamida. - Resaltar la
PRÁCTICA Nº 5 REACCINES DE PLIMERIZACIÓN bjetivos - Realizar un ejemplo práctico de reacción de polimerización por condensación: Preparación de un poliéster. Preparación de una poliamida. - Resaltar la
SÍNTESIS DEL ÁCIDO ACETIL SALICÍLICO
 PRÁCTICA 10: SÍNTESIS DEL ÁCIDO ACETIL SALICÍLICO 1. INTRODUCCIÓN En esta práctica llevaremos a cabo un proceso sencillo de síntesis de un fármaco: la síntesis del ácido acetilsalicílico. El extracto de
PRÁCTICA 10: SÍNTESIS DEL ÁCIDO ACETIL SALICÍLICO 1. INTRODUCCIÓN En esta práctica llevaremos a cabo un proceso sencillo de síntesis de un fármaco: la síntesis del ácido acetilsalicílico. El extracto de
PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO
 PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO I. OBJETIVO Determinar la presencia de bacterias Mesófilas Aerobias en una muestra de agua potable por la técnica de
PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO I. OBJETIVO Determinar la presencia de bacterias Mesófilas Aerobias en una muestra de agua potable por la técnica de
DETECCIÓN DE C. jejuni EN HAMBURGUESA DE POLLO POR PCR TRADICIONAL PRT-712.04-082
 Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA"
 P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
Centro de Capacitación en Informática
 Fórmulas y Funciones Las fórmulas constituyen el núcleo de cualquier hoja de cálculo, y por tanto de Excel. Mediante fórmulas, se llevan a cabo todos los cálculos que se necesitan en una hoja de cálculo.
Fórmulas y Funciones Las fórmulas constituyen el núcleo de cualquier hoja de cálculo, y por tanto de Excel. Mediante fórmulas, se llevan a cabo todos los cálculos que se necesitan en una hoja de cálculo.
OBTENCIÓN DE CULTIVOS PUROS
 Introducción Objetivo Fundamento Trabajo Práctico Nº 4 OBTENCIÓN DE CULTIVOS PUROS Procedimiento Resultados Instrucciones para el uso del Sistema Anaeróbico Gaspak Bibliografía INTRODUCCIÓN En los ambientes
Introducción Objetivo Fundamento Trabajo Práctico Nº 4 OBTENCIÓN DE CULTIVOS PUROS Procedimiento Resultados Instrucciones para el uso del Sistema Anaeróbico Gaspak Bibliografía INTRODUCCIÓN En los ambientes
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa
 38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS
 GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS Leidy Diana Ardila Leal Docente. INTRODUCCIÓN En esta práctica se va a realizar la extracción
GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS Leidy Diana Ardila Leal Docente. INTRODUCCIÓN En esta práctica se va a realizar la extracción
Genoma y Ensamblado Seminario de Modelos y Métodos cuantitativos (Bioinformática)
 Genoma y Ensamblado Seminario de Modelos y Métodos cuantitativos (Bioinformática) Rodrigo Fernández Cristián Maureira Gabriel Zamora Universidad Técnica Federico Santa María 18 de noviembre de 2010 Genoma
Genoma y Ensamblado Seminario de Modelos y Métodos cuantitativos (Bioinformática) Rodrigo Fernández Cristián Maureira Gabriel Zamora Universidad Técnica Federico Santa María 18 de noviembre de 2010 Genoma
SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO
 Actividad Experimental SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO Investigación previa 1.- Investigar las medidas de seguridad que hay que mantener al manipular KOH y H SO, incluyendo que acciones
Actividad Experimental SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO Investigación previa 1.- Investigar las medidas de seguridad que hay que mantener al manipular KOH y H SO, incluyendo que acciones
MEDIDA DEL CALOR ESPECÍFICO
 Laboratorio de Física General Primer Curso (Termodinámica) MEDIDA DEL CALOR ESPECÍFICO Fecha: 07/02/05 1. Objetivo de la práctica Familiarizarse con las medidas calorimétricas mediante la medida del calor
Laboratorio de Física General Primer Curso (Termodinámica) MEDIDA DEL CALOR ESPECÍFICO Fecha: 07/02/05 1. Objetivo de la práctica Familiarizarse con las medidas calorimétricas mediante la medida del calor
ELECTROFORESIS EN GELES DE AGAROSA. Agustín Garrido. agugarrido@hotmail.com
 1 ELECTROFORESIS EN GELES DE AGAROSA Agustín Garrido agugarrido@hotmail.com Introducción En este trabajo práctico se utilizó la técnica de electroforesis. Este proceso se basa en la migración de las moléculas
1 ELECTROFORESIS EN GELES DE AGAROSA Agustín Garrido agugarrido@hotmail.com Introducción En este trabajo práctico se utilizó la técnica de electroforesis. Este proceso se basa en la migración de las moléculas
Reacción en cadena de la polimerasa (PCR)
 Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Extracción sólido-líquido
 Extracción sólido-líquido Objetivos de la práctica! Determinar la concentración de saturación del soluto en el disolvente en un sistema ternario arena-azúcar-agua, estableciendo la zona operativa del diagrama
Extracción sólido-líquido Objetivos de la práctica! Determinar la concentración de saturación del soluto en el disolvente en un sistema ternario arena-azúcar-agua, estableciendo la zona operativa del diagrama
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Normas de seguridad Laboratorio de Química Física Universidad Pablo de Olavide NORMAS DE SEGURIDAD
 NORMAS DE SEGURIDAD El laboratorio debe ser un lugar seguro para trabajar donde no se deben permitir descuidos o bromas. Para ello se tendrán siempre presente los posibles peligros asociados al trabajo
NORMAS DE SEGURIDAD El laboratorio debe ser un lugar seguro para trabajar donde no se deben permitir descuidos o bromas. Para ello se tendrán siempre presente los posibles peligros asociados al trabajo
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
TRABAJO PRÁCTICO N 2: TÉCNICAS DE ESTERILIZACIÓN Y CULTIVO DE MICROORGANISMOS Objetivos:
 TRABAJO PRÁCTICO N 2: TÉCNICAS DE ESTERILIZACIÓN Y CULTIVO DE MICROORGANISMOS Objetivos: -Conocer las metodologías actuales de control y eliminación de microorganismos. -Obtener dominio de los métodos
TRABAJO PRÁCTICO N 2: TÉCNICAS DE ESTERILIZACIÓN Y CULTIVO DE MICROORGANISMOS Objetivos: -Conocer las metodologías actuales de control y eliminación de microorganismos. -Obtener dominio de los métodos
Temas de electricidad II
 Temas de electricidad II CAMBIANDO MATERIALES Ahora volvemos al circuito patrón ya usado. Tal como se indica en la figura, conecte un hilo de cobre y luego uno de níquel-cromo. Qué ocurre con el brillo
Temas de electricidad II CAMBIANDO MATERIALES Ahora volvemos al circuito patrón ya usado. Tal como se indica en la figura, conecte un hilo de cobre y luego uno de níquel-cromo. Qué ocurre con el brillo
Operación de Microsoft Excel. Guía del Usuario Página 79. Centro de Capacitación en Informática
 Manejo básico de base de datos Unas de las capacidades de Excel es la de trabajar con listas o tablas de información: nombres, direcciones, teléfonos, etc. Excel puede trabajar con tablas de información
Manejo básico de base de datos Unas de las capacidades de Excel es la de trabajar con listas o tablas de información: nombres, direcciones, teléfonos, etc. Excel puede trabajar con tablas de información
IES Menéndez Tolosa 3º ESO (Física y Química)
 IES Menéndez Tolosa 3º ESO (Física y Química) 1 De las siguientes mezclas, cuál no es heterogénea? a) azúcar y serrín. b) agua y aceite. c) agua y vino d) arena y grava. La c) es una mezcla homogénea.
IES Menéndez Tolosa 3º ESO (Física y Química) 1 De las siguientes mezclas, cuál no es heterogénea? a) azúcar y serrín. b) agua y aceite. c) agua y vino d) arena y grava. La c) es una mezcla homogénea.
Código: IDK-010 Ver: 1. Proteína G C825T. Sistema para la detección de la mutación C825T en el gene de la proteína G.
 C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
Procesado de muestras en el laboratorio de la clínica (I)
 12.prevención de la salud Procesado de muestras en el laboratorio de la clínica (I) A lo largo de esta primera parte veremos como realizar un manejo correcto de las muestras de sangre, orina y líquidos
12.prevención de la salud Procesado de muestras en el laboratorio de la clínica (I) A lo largo de esta primera parte veremos como realizar un manejo correcto de las muestras de sangre, orina y líquidos
ECOLOGÍA MICROBIANA Y COMPORTAMIENTO BACTERIANO COMUNITARIO
 5 TRABAJO PRÁCTICO ECOLOGÍA MICROBIANA Y COMPORTAMIENTO BACTERIANO COMUNITARIO OBJETIVOS: 1 Observar y comprender la diversidad bacteriana que coexiste en un mismo nicho en un dado ecosistema y las interrelaciones
5 TRABAJO PRÁCTICO ECOLOGÍA MICROBIANA Y COMPORTAMIENTO BACTERIANO COMUNITARIO OBJETIVOS: 1 Observar y comprender la diversidad bacteriana que coexiste en un mismo nicho en un dado ecosistema y las interrelaciones
INSTRUCCIONES DE INSTALACIÓN Y RECARGA CISS HP CARTUCHO SERIE 1
 Antes de empezar Es muy importante que no realice ninguna acción hasta leer completamente las instrucciones. Léalas de forma detenida y completa!!! Es muy importante que compruebe que su impresora se encuentra
Antes de empezar Es muy importante que no realice ninguna acción hasta leer completamente las instrucciones. Léalas de forma detenida y completa!!! Es muy importante que compruebe que su impresora se encuentra
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS
 EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS Objetivos: Mª Jesús González García Mª Amparo Mora Alcácer COLEGIO AVE Mª DE PENYA-ROJA Aprender a trabajar en el laboratorio y apreciar el orden,
EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS Objetivos: Mª Jesús González García Mª Amparo Mora Alcácer COLEGIO AVE Mª DE PENYA-ROJA Aprender a trabajar en el laboratorio y apreciar el orden,
En los programas de estudio para sus estudiantes los Aprendizajes incluidos en la actividad son:
 INTRODUCCIÓN Recordemos que lo que define a un ser vivo entre otras funciones, es su función reproductiva, que permite que una especie se perpetúe en el tiempo, manteniendo las características de sus progenitores,
INTRODUCCIÓN Recordemos que lo que define a un ser vivo entre otras funciones, es su función reproductiva, que permite que una especie se perpetúe en el tiempo, manteniendo las características de sus progenitores,
INGENIERÍA GENÉTICA 5 GAATTC 3 3 CTTAAG 5
 INGENIERÍA GENÉTICA 1. Fundamentos básicos de la ingeniería genética 2. Desnaturalización e hibridación del ADN 3. Reacción en cadena de la polimerasa (PCR) 4. Nuevas disciplinas surgidas de la ingeniería
INGENIERÍA GENÉTICA 1. Fundamentos básicos de la ingeniería genética 2. Desnaturalización e hibridación del ADN 3. Reacción en cadena de la polimerasa (PCR) 4. Nuevas disciplinas surgidas de la ingeniería
Laboratorio. Objetivos I N T R O D U C C I Ó N. Al finalizar este laboratorio el estudiante podrá:
 Laboratorio 12 Biología molecular Objetivos Al finalizar este laboratorio el estudiante podrá: 1. Conocer los principios básicos de la técnica de electroforesis y su aplicación al análisis del ADN. 2.
Laboratorio 12 Biología molecular Objetivos Al finalizar este laboratorio el estudiante podrá: 1. Conocer los principios básicos de la técnica de electroforesis y su aplicación al análisis del ADN. 2.
1. Generalidades del laboratorio
 1. Generalidades del laboratorio Isaac Túnez, María del Carmen Muñoz Departamento de Bioquímica y Biología Molecular, Facultad de Medicina, Universidad de Córdoba, Avda. Menéndez Pidal s/n, 14004-Córdoba
1. Generalidades del laboratorio Isaac Túnez, María del Carmen Muñoz Departamento de Bioquímica y Biología Molecular, Facultad de Medicina, Universidad de Córdoba, Avda. Menéndez Pidal s/n, 14004-Córdoba
TÉCNICAS GENÓMICAS. 2) Cuantificación del número de virus presentes en muestras de sangre o suero: carga vírica
 TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS.
 NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
EXTRACCIÓN DE CAFEÍNA DEL CAFÉ
 10-11-2010 EXTRACCIÓN DE CAFEÍNA DEL CAFÉ Colegio de San Francisco de Paula Enrique Jacobo Díaz Montaña José Antonio Vázquez de la Paz Enrique Gómez-Álvarez Hernández 1ºBACHILLERATO-B Índice: Objetivos
10-11-2010 EXTRACCIÓN DE CAFEÍNA DEL CAFÉ Colegio de San Francisco de Paula Enrique Jacobo Díaz Montaña José Antonio Vázquez de la Paz Enrique Gómez-Álvarez Hernández 1ºBACHILLERATO-B Índice: Objetivos
Procesos de Separación: Destilación Simple de un Vino para la Determinación de su Grado Alcohólico
 Destilación y Grado Alcohólico de un Vino EXPERIMENTACIÓN EN QUÍMICA EUITIG INGENIERO TÉCNICO EN QUÍMICA INDUSTRIAL PRÁCTICA Nº 14 Apellidos y Nombre: Grupo:. Apellidos y Nombre: Pareja:. Procesos de Separación:
Destilación y Grado Alcohólico de un Vino EXPERIMENTACIÓN EN QUÍMICA EUITIG INGENIERO TÉCNICO EN QUÍMICA INDUSTRIAL PRÁCTICA Nº 14 Apellidos y Nombre: Grupo:. Apellidos y Nombre: Pareja:. Procesos de Separación:
Curso de biología molecular CIMAT 2010
 Curso de biología molecular CIMAT 2010 INTRODUCCIÓN Este curso-taller tiene como propósito mostrar de manera teórica y práctica algunos principios básicos de biología molecular e ingeniería genética. Conocerás
Curso de biología molecular CIMAT 2010 INTRODUCCIÓN Este curso-taller tiene como propósito mostrar de manera teórica y práctica algunos principios básicos de biología molecular e ingeniería genética. Conocerás
PRÁCTICA 4 DETERMINACIÓN DE LA CONCENTRACIÓN IÓNICA TOTAL DEL AGUA POTABLE, USANDO LA CROMATOGRAFÍA DE INTERCAMBIO IÓNICO
 PRÁCTICA 4 DETERMINACIÓN DE LA CONCENTRACIÓN IÓNICA TOTAL DEL AGUA POTABLE, USANDO LA CROMATOGRAFÍA DE INTERCAMBIO IÓNICO 1.- FUNDAMENTO TEÓRICO. 1.1.- Materiales de intercambio iónico. El intercambio
PRÁCTICA 4 DETERMINACIÓN DE LA CONCENTRACIÓN IÓNICA TOTAL DEL AGUA POTABLE, USANDO LA CROMATOGRAFÍA DE INTERCAMBIO IÓNICO 1.- FUNDAMENTO TEÓRICO. 1.1.- Materiales de intercambio iónico. El intercambio
ENERGÍA ELÉCTRICA. Central Eólica
 ENERGÍA ELÉCTRICA. Central Eólica La energía eólica es la energía obtenida por el viento, es decir, la energía cinética obtenida por las corrientes de aire y transformada en energía eléctrica mediante
ENERGÍA ELÉCTRICA. Central Eólica La energía eólica es la energía obtenida por el viento, es decir, la energía cinética obtenida por las corrientes de aire y transformada en energía eléctrica mediante
TRABAJO EXPERIMENTAL
 TRABAJO EXPERIMENTAL Temas 1: PRESIÓN HIDRÁULICA DE LA MAREA Diariamente, la gravedad lunar provoca la subida y bajada de la marea. Estos cambios de altura del agua del mar pueden ser útiles para obtener
TRABAJO EXPERIMENTAL Temas 1: PRESIÓN HIDRÁULICA DE LA MAREA Diariamente, la gravedad lunar provoca la subida y bajada de la marea. Estos cambios de altura del agua del mar pueden ser útiles para obtener
CUESTIONES. 1. Por qué la PCR convencional no es cuantitativa?
 2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
Ejercicios de Programación Lineal
 Ejercicios de Programación Lineal Investigación Operativa Ingeniería Informática, UCM Curso 8/9 Una compañía de transporte dispone de camiones con capacidad de 4 libras y de 5 camiones con capacidad de
Ejercicios de Programación Lineal Investigación Operativa Ingeniería Informática, UCM Curso 8/9 Una compañía de transporte dispone de camiones con capacidad de 4 libras y de 5 camiones con capacidad de
Instrucciones técnicas Aparato de extracción y filtración en caliente para sistemas de microondas
 Instrucciones técnicas Aparato de extracción y filtración en caliente para sistemas de microondas Las condiciones experimentales de un experimento con microondas dependen de los datos técnicos del aparto
Instrucciones técnicas Aparato de extracción y filtración en caliente para sistemas de microondas Las condiciones experimentales de un experimento con microondas dependen de los datos técnicos del aparto
PRÁCTICA 7: EQUILIBRIO ÁCIDO-BASE
 PRÁCTICA 7: EQUILIBRIO ÁCIDO-BASE FUNDAMENTOS Concepto de ácido y base Los ácidos y las bases constituyen una clase de compuestos químicos de gran interés. El concepto de ácido y base ha evolucionado a
PRÁCTICA 7: EQUILIBRIO ÁCIDO-BASE FUNDAMENTOS Concepto de ácido y base Los ácidos y las bases constituyen una clase de compuestos químicos de gran interés. El concepto de ácido y base ha evolucionado a
2.3 SISTEMAS HOMOGÉNEOS.
 2.3 SISTEMAS HOMOGÉNEOS. 2.3.1 DISOLUCIONES. Vemos que muchos cuerpos y sistemas materiales son heterogéneos y podemos observar que están formados por varias sustancias. En otros no podemos ver que haya
2.3 SISTEMAS HOMOGÉNEOS. 2.3.1 DISOLUCIONES. Vemos que muchos cuerpos y sistemas materiales son heterogéneos y podemos observar que están formados por varias sustancias. En otros no podemos ver que haya
CUESTIONES TEMA 4: La revolución genética y la biotecnología.
 CUESTIONES TEMA 4: La revolución genética y la biotecnología. 1. El ADN no puede salir del núcleo: Cómo logra llevar a los ribosomas que están en el citoplasma la información que porta? 2. El individuo
CUESTIONES TEMA 4: La revolución genética y la biotecnología. 1. El ADN no puede salir del núcleo: Cómo logra llevar a los ribosomas que están en el citoplasma la información que porta? 2. El individuo
BLOQUE I. CUÁL ES LA COMPOSICIÓN DE LOS SERES VIVOS? LAS MOLÉCULAS DE LA VIDA
 I.E.S. Flavio Irnitano El Saucejo (Sevilla) Curso 2.015 2.016 Departamento de Biología y Geología NIVEL: 2º Bachillerato MATERIA: BIOLOGÍA 6.1. Concepto y estructura. BLOQUE I. CUÁL ES LA COMPOSICIÓN DE
I.E.S. Flavio Irnitano El Saucejo (Sevilla) Curso 2.015 2.016 Departamento de Biología y Geología NIVEL: 2º Bachillerato MATERIA: BIOLOGÍA 6.1. Concepto y estructura. BLOQUE I. CUÁL ES LA COMPOSICIÓN DE
Adaptación al NPGC. Introducción. NPGC.doc. Qué cambios hay en el NPGC? Telf.: 93.410.92.92 Fax.: 93.419.86.49 e-mail:atcliente@websie.
 Adaptación al NPGC Introducción Nexus 620, ya recoge el Nuevo Plan General Contable, que entrará en vigor el 1 de Enero de 2008. Este documento mostrará que debemos hacer a partir de esa fecha, según nuestra
Adaptación al NPGC Introducción Nexus 620, ya recoge el Nuevo Plan General Contable, que entrará en vigor el 1 de Enero de 2008. Este documento mostrará que debemos hacer a partir de esa fecha, según nuestra
ANÁLISIS DE AGUAS: Metodología
 FTTM06 Rev-2,21/11/2013 INSTITUTO DE TOXICOLOGÍA DE LA DEFENSA Hospital Central de la Defensa. Glorieta del Ejército s/n. 28047 MADRID. Tel.: 914222625. Fax: 914222624 E- mail : toxicologia@oc.mde.es Web
FTTM06 Rev-2,21/11/2013 INSTITUTO DE TOXICOLOGÍA DE LA DEFENSA Hospital Central de la Defensa. Glorieta del Ejército s/n. 28047 MADRID. Tel.: 914222625. Fax: 914222624 E- mail : toxicologia@oc.mde.es Web
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
VALORACIÓN ÁCIDO-BASE. Conocer y aplicar el método volumétrico para realizar una titulación ácido-base
 EXPERIMENTO 3 VALORACIÓN ÁCIDO-BASE Objetivo general Conocer y aplicar el método volumétrico para realizar una titulación ácido-base Objetivos específicos 1.- Determinar el punto de equivalencia de una
EXPERIMENTO 3 VALORACIÓN ÁCIDO-BASE Objetivo general Conocer y aplicar el método volumétrico para realizar una titulación ácido-base Objetivos específicos 1.- Determinar el punto de equivalencia de una
MANUAL DE PRÁCTICAS DE LABORATORIO
 MANUAL DE PRÁCTICAS DE LABORATORIO CARRERA Ingeniería en Biotecnología ASIGNATURA: Microbiología Gral. FICHA TECNICA Fecha: Nombre del catedrático: 13-SEPT-2012 FICHA TÉCNICA MICROBIOLOGÍA GENERAL Jesús
MANUAL DE PRÁCTICAS DE LABORATORIO CARRERA Ingeniería en Biotecnología ASIGNATURA: Microbiología Gral. FICHA TECNICA Fecha: Nombre del catedrático: 13-SEPT-2012 FICHA TÉCNICA MICROBIOLOGÍA GENERAL Jesús
ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN
 Universidad Centroccidental Lisandro Alvarado Decanato de Ciencias de la Salud ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN Octubre 2009 INTRODUCCION La molécula de ADN (que es la que se
Universidad Centroccidental Lisandro Alvarado Decanato de Ciencias de la Salud ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN Octubre 2009 INTRODUCCION La molécula de ADN (que es la que se
Facultad de Ciencias Experimentales Departamento de Ingeniería Química, Ambiental y de los Materiales. Cuaderno de prácticas de la asignatura
 UNIVERSIDAD DE JAÉN Facultad de Ciencias Experimentales Departamento de Ingeniería Química, Ambiental y de los Materiales Cuaderno de prácticas de la asignatura BASES DE LA INGENIERÍA AMBIENTAL Licenciatura
UNIVERSIDAD DE JAÉN Facultad de Ciencias Experimentales Departamento de Ingeniería Química, Ambiental y de los Materiales Cuaderno de prácticas de la asignatura BASES DE LA INGENIERÍA AMBIENTAL Licenciatura
I. Objetivos 1. Determinar el cambio de entalpía de una reacción de metal de magnesio con ácido clorhídrico.
 UNIVERSIDAD INTERAMERICANA Recinto de Bayamón Departamento de Ciencias Naturales y Matemáticas Fundamentos de Química: CHEM 1111 Experimento No. 9: Cambio de entalpía de una reacción I. Objetivos 1. Determinar
UNIVERSIDAD INTERAMERICANA Recinto de Bayamón Departamento de Ciencias Naturales y Matemáticas Fundamentos de Química: CHEM 1111 Experimento No. 9: Cambio de entalpía de una reacción I. Objetivos 1. Determinar
TEMA 11. MÉTODOS FÍSICOS DE SEPARACIÓN Y PURIFICACIÓN
 TEMA 11. MÉTODOS FÍSICOS DE SEPARACIÓN Y PURIFICACIÓN 1. Destilación 2. Extracción 3. Sublimación 4. Cristalización 5. Cromatografía 6. Fórmulas empíricas y moleculares 2 Tema 11 TEMA 11. Métodos físicos
TEMA 11. MÉTODOS FÍSICOS DE SEPARACIÓN Y PURIFICACIÓN 1. Destilación 2. Extracción 3. Sublimación 4. Cristalización 5. Cromatografía 6. Fórmulas empíricas y moleculares 2 Tema 11 TEMA 11. Métodos físicos
10B Reacciones de Esterificación de Ácidos Carboxílicos. Obtención de Acetato de Isoamilo (Aceite de Plátano).
 PRÁCTICA 10B Reacciones de Esterificación de Ácidos Carboxílicos. Obtención de Acetato de Isoamilo (Aceite de Plátano). I. OBJETIVOS. a) Preparar un éster a partir de un alcohol y un ácido carboxílico.
PRÁCTICA 10B Reacciones de Esterificación de Ácidos Carboxílicos. Obtención de Acetato de Isoamilo (Aceite de Plátano). I. OBJETIVOS. a) Preparar un éster a partir de un alcohol y un ácido carboxílico.
1. Fundamento teórico
 1 1. Fundamento teórico Los métodos espectroscópicos atómicos y moleculares figuran entre los métodos analíticos instrumentales más utilizados. La espectroscopia molecular basada en la radiación ultravioleta,
1 1. Fundamento teórico Los métodos espectroscópicos atómicos y moleculares figuran entre los métodos analíticos instrumentales más utilizados. La espectroscopia molecular basada en la radiación ultravioleta,
La separación de mezclas de las cuales existen dos tipos como son las homogéneas y heterogéneas
 Introducción En el tema operaciones fundamentales de laboratorio se dan una serie e pasos muy importantes para el desarrollo del programa de laboratorio por ejemplo podemos citar varios procedimientos
Introducción En el tema operaciones fundamentales de laboratorio se dan una serie e pasos muy importantes para el desarrollo del programa de laboratorio por ejemplo podemos citar varios procedimientos
PIP 4º ESO IES SÉNECA TRABAJO EXPERIMENTAL EN FÍSICA Y QUÍMICA
 MEZCLAS Las mezclas son agrupaciones de dos o más sustancias puras en proporciones variables. Si presentan un aspecto uniforme son homogéneas y también se denominan disoluciones, como la de azúcar en agua.
MEZCLAS Las mezclas son agrupaciones de dos o más sustancias puras en proporciones variables. Si presentan un aspecto uniforme son homogéneas y también se denominan disoluciones, como la de azúcar en agua.
GUÍA DE HIGIENE BUCAL
 GUÍA DE HIGIENE BUCAL La placa dental es una película de bacterias que se adhiere a la superficie de los dientes y que produce las caries y las enfermedades de las encías (gingivitis y periodontitis o
GUÍA DE HIGIENE BUCAL La placa dental es una película de bacterias que se adhiere a la superficie de los dientes y que produce las caries y las enfermedades de las encías (gingivitis y periodontitis o
Artículo 3 Solución isotónica de Cloruro de Sodio
 Banco de levaduras Esta es una serie traducida del original en https://eurekabrewing.wordpress.com/ y publicada en nuestro sitio web con permiso expreso del autor. Artículo 3 Solución isotónica de Cloruro
Banco de levaduras Esta es una serie traducida del original en https://eurekabrewing.wordpress.com/ y publicada en nuestro sitio web con permiso expreso del autor. Artículo 3 Solución isotónica de Cloruro
Tutorial de PowerPoint
 Tutorial de PowerPoint Este documento pretende explicar la elaboración de la presentación Los Planetas del Sistema Solar que se comenta en el apartado II de la Guía del curso. Las imágenes utilizadas están
Tutorial de PowerPoint Este documento pretende explicar la elaboración de la presentación Los Planetas del Sistema Solar que se comenta en el apartado II de la Guía del curso. Las imágenes utilizadas están
Guía para la migración de asignaturas de grado y másteres al nuevo espacio docente para el curso 2015/2016
 Guía para la migración de asignaturas de grado y másteres al nuevo espacio docente para el curso 2015/2016 El presente manual ha sido elaborado antes de la puesta en producción de la plataforma para el
Guía para la migración de asignaturas de grado y másteres al nuevo espacio docente para el curso 2015/2016 El presente manual ha sido elaborado antes de la puesta en producción de la plataforma para el
PRÁCTICA 2 DETERMINACIÓN ESPECTROFOTOMÉTRICA DE
 PRÁCTICA 2 DETERMINACIÓN ESPECTROFOTOMÉTRICA DE MnO 4-1.- FUNDAMENTO TEÓRICO. 1.1.- Introducción Un método espectrofotométrico está basado en la medida directa de la absorción de radiación electromagnética
PRÁCTICA 2 DETERMINACIÓN ESPECTROFOTOMÉTRICA DE MnO 4-1.- FUNDAMENTO TEÓRICO. 1.1.- Introducción Un método espectrofotométrico está basado en la medida directa de la absorción de radiación electromagnética
ANEJO 2: PROCESO DE ELABORACIÓN
 ANEJO 2: PROCESO DE ELABORACIÓN ANEJO 2: PROCESO DE ELABORACIÓN. 1.. 2. Descripción del proceso. 2.1. Fase 1: Elaboración de la mermelada. 2.1.1. Mezcla de ingredientes. 2.1.2. Cocido primera etapa. 2.1.3.
ANEJO 2: PROCESO DE ELABORACIÓN ANEJO 2: PROCESO DE ELABORACIÓN. 1.. 2. Descripción del proceso. 2.1. Fase 1: Elaboración de la mermelada. 2.1.1. Mezcla de ingredientes. 2.1.2. Cocido primera etapa. 2.1.3.
Grupo Nodrissa. Marta Bacardit Abellán EXTRACCIÓN, ALMACENAMIENTO Y CONSERVACIÓN DE LA LECHE MATERNA
 Grupo Nodrissa. Marta Bacardit Abellán EXTRACCIÓN, ALMACENAMIENTO Y CONSERVACIÓN DE LA LECHE MATERNA 1. Extracción de la leche materna Motivos de extracción: Incorporación al trabajo. Para estimular la
Grupo Nodrissa. Marta Bacardit Abellán EXTRACCIÓN, ALMACENAMIENTO Y CONSERVACIÓN DE LA LECHE MATERNA 1. Extracción de la leche materna Motivos de extracción: Incorporación al trabajo. Para estimular la
TRABAJO PRACTICO ESTERILIZACION
 TRABAJO PRACTICO ESTERILIZACION Introducción La esterilización es un proceso de suma importancia para la industria de las fermentaciones. Para comenzar la explicación de este tema es conveniente dejar
TRABAJO PRACTICO ESTERILIZACION Introducción La esterilización es un proceso de suma importancia para la industria de las fermentaciones. Para comenzar la explicación de este tema es conveniente dejar
2011-2012 RESOLUCIÓN DE ERRORES EN MOODLE CAMPUS VIRTUAL-BIRTUALA UPV-EHU
 2011-2012 RESOLUCIÓN DE ERRORES EN MOODLE CAMPUS VIRTUAL-BIRTUALA UPV-EHU Antecedentes:... 2 1. Introducción... 3 2. Imágenes que no se visualizan... 3 3. URLs de recursos o actividades que no son autocontenido...
2011-2012 RESOLUCIÓN DE ERRORES EN MOODLE CAMPUS VIRTUAL-BIRTUALA UPV-EHU Antecedentes:... 2 1. Introducción... 3 2. Imágenes que no se visualizan... 3 3. URLs de recursos o actividades que no son autocontenido...
Historia de los Indicadores Biológicos
 Historia de los Indicadores Biológicos E.U. Sandra Riveros C. El uso de un producto estéril, y el proceso que produce este producto estéril, es crítico para el programa del control de infecciones. Un producto
Historia de los Indicadores Biológicos E.U. Sandra Riveros C. El uso de un producto estéril, y el proceso que produce este producto estéril, es crítico para el programa del control de infecciones. Un producto
