CONTROLES DE CALIDAD. Taller 3
|
|
|
- Nieves Ponce Carrasco
- hace 9 años
- Vistas:
Transcripción
1 Taller 3 CONTROLES DE CALIDAD Coordina: Andres C. García Rosa Pinto (Banco Nacional de ADN, Salamanca) María Almeida Parra (Banco Nacional de ADN, Salamanca) María Pérez Caro (Banco Nacional de ADN, Salamanca) Raquel Paternain (Fundación para la Investigación Médica Aplicada, Navarra)
2 Taller 3: CONTROLES DE CALIDAD Cuantificación del ADN. Diferencias entre espectrofotometría UV y fluorimetría Dr. Andrés C. García Montero. Banco Nacional de ADN Carlos III- Universidad de Salamanca 12 de Noviembre de 2014
3 ABSORBANCIA UV VS FLUORESCENCIA
4 ESPECTROFOTOMETRÍA La Ley Lambert Beer describe cómo disminuye la cantidad de luz que atraviesa una solución (absorbancia) en función de: 1.- La concentración de la muestra (cantidad de material de absorción) 2.- La distancia de la trayectoria óptica 3.- El coeficiente de extinción (probabilidad de que el fotón a esa sea absorbido por el material) A = lc A = = l = c = Absorbancia Coeficiente de extinción Distancia en cm Concentración A 260 nm de, la media del Coeficiente de extinción ( ) : C = A / l dsdna ssdna ssrna Primers (μg/ml)-1 cm-1, (μg/ml)-1 cm-1, (μg/ml)-1 cm (μg/ml)-1 cm-1. Ac. nucleico Concentración (μg/ml) En cubeta 1 cm, 1 unidad OD ( optical density ) a 260nm dsdna 50 ssdna 37 ssrna 40
5 Espectrofotómetros de cubeta Espectrofotómetros en placa Espectrofotómetros de micromuestras
6 ABSORBANCIA A260 VENTAJAS.- barato.- rápido.- (poco volumen).- independiente de estándares de [DNA] conocidos.- detecta contaminación (proteínas, c.aromáticos, sales, ) Espectro típico de un ácido nucleótido Espectro típico de fenol
7 CONTROL DE CALIDAD DEL ADN PUREZA DEL ADN POSIBLES CONTAMINANTES: - ADN degradado, nucleótidos, cebadores, -proteínas - compuestos aromáticos (fenol, ) - alcoholes, - sales (Buffer TE),
8 ABSORBANCIA A260 LIMITACIONES.- detecta tanto DNA íntegro como nucleótidos libres.- límite inferior de detección >3-4 ng/ul.- cuantificación afectada por los contaminantes
9
10
11 CRITERIOS DE PUREZA DEL ADN DETECCIÓN DE CONTAMINANTES Relación de absorbancia 260/280 Se usa para establecer la pureza de una solución de ácidos nucleicos. El ratio 260/280 de cada nucleótido puro, si se mide independientemente, es:** Guanina: 1.15 Adenina: 4.50 Citosina: 1.51 Uracilo: 4.00 Timina: 1.47 (**Leninger, A. L. Biochemistry, 2nd ed., Worth Publishers, New York, 1975) Se acepta que un ratio de ~1.8 se correspondería a un ADN puro; mientras que para el ARN debería ser de ~2.0. Si el ratio es sensiblemente inferior indicaría la presencia de proteínas, fenoles u otros contaminantes que absorben próximos a 280 nm.
12 CRITERIOS DE PUREZA DEL ADN DETECCIÓN DE CONTAMINANTES RELACIÓN DE ABSORBANCIAS A260/A280 Contaminación c. aromáticos < 1,6 (proteínas, fenoles) 1,7 1,9 PUREZA ÓPTIMA > 2,1 Contaminación ARN Espectro típico de un ácido nucleótido Espectro típico de fenol
13 CRITERIO DE INTEGRIDAD DEL ADN ELECTROFORESIS EN GEL DE AGAROSA (0,7%): ADN íntegro ADN degradado
14 FLUORIMETRÍA Emisión de FLUORESCENCIA Picogreen
15 FLUORESCENCIA VENTAJAS.- únicamente ADN íntegro (doble cadena).- necesita cantidades mínimas de ADN (pg)
16 FLUORESCENCIA DESVENTAJAS.- técnica compleja (diluciones, volúmenes pequeños, ).- necesita datos previos de rango de concentración.- cuantificación relativa.- dependiente de estándares de [ADN] conocidos Picogreen Cuantificación relativa! Curvas de calibrado: patrones de concentración conocida!
17 EVALUACIÓN CUANTIFICACIÓN ADN Estudio multicéntrico: BBMRI 64 centros Absorbancia 260nm 37 centros Fluorescencia (Picogreen) 8 muestras (x3) concentraciones conocidas #1 270 ng/ul #2 220 ng/ul #3 140 ng/ul #4 180 ng/ul #5 60 ng/ul #6 40 ng/ul #7 10 ng/ul #8 100 ng/ul
18 Q2C_080 Q 2C_082 Q2C_083a Q2C_083b Q2C_084 Q2C_085a Q2C_086 Q2C_087a Q 2C_087b Q2C_088 Q2C_089 Q2C_091a Q 2C_092a Q2C_094 Q2C_078 Q2C_079a Q 2C_077b Q2C_075a Q 2C_077a Q2C_073b Q2C_074a Absorbance concentrations Absorbancia a 260nm Q2C_044 Q2C_045 Q2C_046 Q2C_050 Q2C_051 Q2C_052 Q2C_054 Q 2C_055a Q 2C_055b Q2C_057 Q2C_058a Q2C_058b Q2C_060 Q2C_061 Q2C_062a Q2C_062b Q 2C_064a Q 2C_064b Q2C_065 Q 2C_066 Q2C_068 Q 2C_069 Q2C_071A Q2C_072 Q2C_073a Q2C_043 Study Centre Fluorescence Concentrations Fluorescencia (Picogreen) A B C D E F G H Q2C_079b Q2C_081 Q2C_085b Q2C_087c Q2C_090a Q2C_090b Q2C_091b Q2C_075b Q2C_071B Q2C_074b Q2C_070 Q2C_067 Q2C_029a Q2C_029b Q2C_029c Q2C_029d ) Q2C_030 Q2C_031 Q2C_039 Q2C_042a Q2C_042b Q2C_047 Q2C_049 Q2C_059 Q2C_060 Q2C_062 Q2C_028 Q 2C_041 Q 2C_034 Q2C_038 Q2C_027b Q2C_027a Q 2C_033b Q2C_033c Q2C_023a Q2C_023c Q 2C_033a Q2C_017 Q2C_023b Q2C_020 Q2C_021 Q2C_016 Q2C_018 Q2C_014 Q2C_015 Q2C_011b Q2C_008 Q2C_011a Q2C_010 Q 2C_005 Q2C_002 Q2C_004 Q2C_004 Q2C_001a Valores esperados: Study Centre 1 A B C D E F G H Q2C_001b Concentrations ng/ul Concentration (ng/ul)
19 RECOMENDACIONES Utilizar de rutina espectrofotometría UV.- Lo que si es IMPORTANTE que tengáis en cuenta al medir el UV es: 1º.- Verificar la calibración del espectrofotómetro frecuentemente (p.ej. una vez al mes), para comprobar que mide bien..- En un espectrofotómetro UV de referencia (verificado/calibrado): medid varios ADNs de muy buena calidad (elevada pureza, buena concentración y ausencia de contaminantes proteínas, fenoles, sales -), conservadlos en condiciones óptimas (múltiples alícuotas congeladas), y utilizadlos como muestras de referencia (concentración conocida)..- Una vez al mes sacáis una alícuota de cada ADN y los medís en el Espect. UV que queréis verificar. Haced al menos 10 medidas de cada ADN. Si la precisión y exactitud no se van más allá de entre un 2%- 5%, entonces todo OK.
20
21 Exactitud: es la cualidad de un instrumento para proporcionar resultados (medidas) muy próximas al valor esperado. Precisión: es un término cualitativo que hace referencia a la habilidad de un instrumento para proporcionar resultados (medidas) muy similares entre ellos. También se suele referir como repetitividad o reproductibilidad del equipo. a) Exactitud y precisión buenas. b) Exactitud deficiente y buena precisión, c) Exactitud y precisión deficientes.
22 RECOMENDACIONES Utilizar de rutina espectrofotometría UV.- Lo que si es IMPORTANTE que tengáis en cuenta al medir el UV es: 1º.- Verificar la calibración del espectrofotómetro frecuentemente (p.ej. una vez al mes), para comprobar que mide bien. 2º.- En todas las muestras hacer un gel de agarosa para verificar la integridad del ADN (no está fragmentado). Es muy sencillo, rápido y barato..- Un gel agarosa al 0.8-1%es suficiente..- Migrar 20 minutos a 100V.- Revelar con EtBr (u otro menos tóxico de los que hay en el mercado). Resultado: Banda de gran tamaño y sin arrastre (o muy poco) es que el ADN está perfecto.
23 CONTROL DE CALIDAD DEL ADN INTEGRIDAD DEL ADN ELECTROFORESIS EN GEL DE AGAROSA (0,7%):
24 RECOMENDACIONES Utilizar de rutina espectrofotometría UV.- Lo que si es IMPORTANTE que tengáis en cuenta al medir el UV es: 1º.- Verificar la calibración del espectrofotómetro frecuentemente (p.ej. una vez al mes), para comprobar que mide bien. 2º.- En todas las muestras hacer un gel de agarosa para verificar la integridad del DNA (no está fragmentado). 3º.- Verificar periódicamente la eficiencia del protocolo de extracción de ADN, mediante la comparación de la cuantificación mediante PicoGreen vs Absorbancia 260nm. Con esto podremos conocer nuestra incertidumbre de medida y saber si ésta es aceptable o no
25 CONCENTRACIÓN (ng/ l): CONTROL DE CALIDAD DEL ADN CONCENTRACIÓN -Espectrofotometría: concentración y pureza Nanodrop ND1000 -Fluorimetría: concentración e integridad medida de absorbancia 260 nm todos los nucleótidos PicoGreen unión exclusiva al ADN de doble cadena RELACIÓN PicoGreen / Nanodrop ND1000 R < 1 muestra óptima de ADN: R > 0.8
26 GRACIAS!
27 Taller 3 CONTROLES DE CALIDAD Coordina: Andres C. García Rosa Pinto (Banco Nacional de ADN, Salamanca) María Almeida Parra (Banco Nacional de ADN, Salamanca) María Pérez Caro (Banco Nacional de ADN, Salamanca) Raquel Paternain (Fundación para la Investigación Médica Aplicada, Navarra)
28 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. Dra. María Pérez Caro. Área de Control de Calidad Banco Nacional de ADN Carlos III Universidad de Salamanca 12 de Noviembre de 2014
29 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. El término calidad engloba concentración, pureza e integridad. En el DNA y RNA el análisis de la calidad, tradicionalmente, comprende electroforesis y análisis del ratio 260/280 En el RNA concretamente, se determina la calidad del rrna (>80% ribosómico y 1 3% mrna) Cuando tratamos el control de calidad tenemos que distinguir entre: PUREZA: ausencia de contaminantes INTEGRIDAD: banda única de gran tamaño (DNA) /relación 28S:18S 2:1 (RNA) FUNCIONALIDAD: que la solución sea apta para realizar los ensayos que se propongan. CONCENTRACIÓN
30 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. CONCENTRACIÓN Y PUREZA: RELACIÓN ABSORBANCIAS 1.8 D.O A260/ Pureza aceptable PUREZA ÓPTIMA < 1.8 Contaminación comp. aromáticos D.O A260/230 ~2,0 PUREZA ÓPTIMA < 1.5 (contaminación sales, comp. orgánicos) INTEGRIDAD DNA RNA
31 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. FUNCIONALIDAD Long PCR Múltiple: DNA RT PCR oligos inter exónicos: RNA 5 parejas de oligonucleótidos (primers) simultáneamente amplificación en 6 cromosomas diferentes amplicones entre 17.5 kb y 2.0 kb M
32 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. Técnicas de microfluídos Agilent 2100 bioanalyzer combina técnicas de electroforesis capilar y fluorescencia. Evalúa calidad e integridad. Requiere de muy poca cantidad de muestra. Da valores numéricos a las imágenes que se ven habitualmente RIN (RNA Integrity Number) RIN < 4 RNA baja calidad RIN > 7 RNA alta calidad (arrays) RIN 4 7 RNA calidad media (qrt PCR)
33 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. Tapestation Agilent Uso de geles multi línea y microfluidos que permiten un ensayo semi automático. Simplifica el manejo de muestras y reduciendo los tiempos de ensayo.
34 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. OBJETIVO: Comprobar fiabilidad y repetitividad: RIN y concentración TÉCNICAS DE MICROFLUÍDOS Control de calidad del RNA 5 muestras RNA (diluciones) extraídas por métodos diferentes analizadas por duplicado yendías distintos
35 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas.
36 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. Well RIN e Conc. [ng/µl] Sample Description Replicas Datos Bioanalyzer A1 7,2 168 ladder [1]/[2] RIN [] B1 6, patol ng/ul C1 6, patol D1 6,3 61, patol 1/ ng/ul E1 6,4 55, patol 1/2 F1 6, patol 1/ ng/ul G1 6, patol 1/4 H1 1,8 75, patol ng/ul A2 2,1 79, patol B2 1,8 52, patol 1/ ng/ul C2 1,7 41, patol 1/2 D2 1,4 27, patol 1/ ng/ul E2 1,7 28, patol 1/4 F2 6, norm ng/ul G2 6,5 94, norm H2 6 56, norm 1/2 0.9 N/A 14ng/ul A3 6,3 61, norm 1/2 B3 6, norm 1/4 1 N/A 7ng/ul C3 6,5 26, norm 1/4 D3 7, neutro ng/ul E neutro F3 8, neutro 1/ ng/ul G3 8,1 97, neutro 1/2 H3 7,8 55, neutro 1/ ng/ul A4 7, neutro 1/4 B4 7, mono ng/ul C4 7, mono D4 8,2 61, mono 1/ ng/ul E4 8,2 58, mono 1/2 F4 7, mono 1/ ng/ul G4 8 33, mono 1/4
37 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. El análisis de las medidas de concentración aportada por bioanalizador y tapestation de las mismas muestras, por duplicado yendías distintos, determinó la baja reproducibilidad entre ambas técnicas, resultando ser la medida más estable la aportada por el nanodrop. DIA 1 DIA 2 DIA 1 DIA 2 Tape 1 Tape 2 Tape 1 Tape 2 Agilent Agilent muestras concentración concentración concentración concentración concentración concentración patol patol 1/2 61,4 55,7 46, patol 1/ ,7 38, patol 75,8 79,2 50,5 78, patol 1/2 52,4 41,4 48,2 50, patol 1/4 27,5 28,5 16,8 31, norm ,7 55,2 42, norm 1/2 56,3 61,4 62,6 31, norml 1/ ,9 16,9 32, neutro neutro 1/ , , neutro 1/4 55, ,1 29, mono , mono 1/2 61,5 58,2 34,6 28, mono 1/ ,9 19,3 12,
38 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. Well RIN e Conc. [ng/µl] Sample Description RIN e /RIN Observations A1 7,2 168 ladder RIN [] B1 6, patol 0, ng/ul C1 6, patol D1 6,3 61, patol 1/2 0, ng/ul E1 6,4 55, patol 1/2 F1 6, patol 1/4 0, ng/ul G1 6, patol 1/4 H1 1,8 75, patol ng/ul A2 2,1 79, patol B2 1,8 52, patol 1/ ng/ul C2 1,7 41, patol 1/2 D2 1,4 27, patol 1/ ng/ul E2 1,7 28, patol 1/4 F2 6, norm ng/ul G2 6,5 94, norm H2 6,0 56, norm 1/ N/A 14ng/ul A3 6,3 61, norm 1/2 B3 6, norm 1/ N/A 7ng/ul C3 6,5 26, norm 1/4 D3 7, neutro ng/ul E3 8, neutro F3 8, neutro 1/ ng/ul G3 8,1 97, neutro 1/2 H3 7,8 55, neutro 1/ ng/ul A4 7, neutro 1/4 B4 7, mono ng/ul C4 7, mono D4 8,2 61, mono 1/ ng/ul E4 8,2 58, mono 1/2 F4 7, mono 1/ ng/ul G4 8,0 33, mono 1/4
39 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas.
40 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas.
41 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. CONCLUSIONES: Resultados obtenidos en el BNADN muestran cómo las mismas muestras de RNA analizadas en días diferentes, en ambas plataformas, NO reproducen la concentración entre réplicas (a pesar del que cumplen con las especificaciones del aparato) pero SÍ reproduce los valores de RIN/ RIN e en el RNA, siendo la diferencia entre ambos ~1
42 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. TÉCNICAS DE MICROFLUÍDOS Control de calidad del DNA TAPESTATION AGILENT
43 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. Sample Description Ratio Picogreen/ TapeStation Ratio Nanodrop/ TapeStation Ratio Picogreen/ Nanodrop 1 1,1 1,2 0,91 2 1,2 1,2 0,94 3 1,3 1,3 0,97 4 1,2 1,2 0,98 5 1,1 1,1 0,96 6 1,2 1,4 0,87 7 1,3 1, ,3 1,4 0,93 9 1,2 1,4 0, ,5 1,7 0,89
44 Taller 3: CONTROLES DE CALIDAD Evaluación de los métodos de control de calidad de ácidos nucleicos mediante electroforesis capilar y electroferogramas. CONCLUSIÓN: Cada sistema de análisis aporta resultados diferentes, NO COMPARABLES, de ahí la importancia de utilizar siempre la misma plataforma, conociendo sus ventajas e inconvenientes.
45 DIRECCIÓN: Prof. Dr. Alberto Orfao de Matos COORDINACIÓN TÉCNICA: Dr. Andrés García Montero Dra. María Almeida Parra GESTIÓN DE CALIDAD: Ana Regalado Mayordomo CONTROL DE CALIDAD: Dra. Rosa Pinto Labajo Dra. María Pérez Caro SECRETARÍA y ADMINISTRACIÓN: Elena Chamorro Castro PERSONAL DE LABORATORIO: Javier García Palomo Teresa Malvar Ferreras Mª Teresa Márquez de Sousa Sheila Mateos Domínguez Mª Isabel Morante Arroyo
46 GRACIAS!
47
48 Objetivo inicial./,*0 $1221/(**)!"#$% &'()*+(,* ' -
49 Metodología. 31(*+1*4 1 / 5&. 6! 1*!$ 7. "$"8/1***!." 7' $$ 9*:/5 ( *8
50 Resultados iniciales %1(*1* Pureza* Rendimiento** N A2 60/280 < 0,1 0,1-1 > 1 Es ófago-estómago 7 1,86± 0, G anglio 5 1,92± 0, Hígado 6 1,93± 0, Intestino 27 1,93± 0, Mama 24 1,58± 0, O vario 7 1,81± 0, P.blandas y hueso 11 1,83± 0, Pulm ón 8 1,79± 0, Riñón-vejiga 20 1,91± 0, Útero 5 1,79± 0, TOTAL 120 * Media +/- Desviación estándar ** Nº de muestras cuyo rendimiento de RN A es (ug/mg tejido) : <0,1, >0,1 y >1, y >1 $;$
51 Resultados en tejido mamario % () )< MUESTRA EXTRACCIÓN NANODROP EXPERION # BIOPSIA AÑO CAJA HUECO TEJIDO CLASE FECHA EXTR. Peso (mg) RENDIMIENTO (ug/mg tejido) ng/ul ug RNA A260/280 CALIDAD 28S/18S RQI CLASE mama NT 08/02/ ,50 0, ,6000 7,9920 1,76 1 0,22 3, mama NT 16/02/ ,60 0,0078 9,8700 0,1974 1, mama NT 16/02/ ,20 0, ,7100 0,6342 1, mama NT 16/02/ ,50 0,0053 6,2500 0,1250 1, mama NT 16/02/ ,10 0, ,4400 0,2887 1, mama NT 16/02/ ,00 0, ,0000 1,6000 0, mama NT 16/02/ ,30 0,0057 9,0000 0,1799 0, mama NT 24/02/ ,60 0, , ,1692 1, mama NT 24/02/ ,50 0, ,8050 4,7761 1,89 1 0,62 6, mama NT 24/02/ ,50 0,0028 4,1400 0,0828 0, mama NT 04/03/ ,80 0, ,6650 0,7133 1, mama NT 05/05/ ,10 0, , ,9642 1,86 1 0,16 3, mama NT 11/05/ ,20 0, ,0300 3,2206 1, mama NT 23/05/ ,80 0, ,6950 2,3939 1,66 1 0,72 2, mama NT 23/05/ ,30 0, ,3650 2,9273 1,77 0 3,04 1, mama NT 23/05/ ,80 0, ,5400 0,4908 1, mama NT 18/05/ ,70 0, ,9750 2,2795 1, mama NT 18/05/ ,80 0, ,7750 1,2555 1, mama NT 18/05/ ,00 0, ,2050 2,5241 1, mama NT 23/05/ ,90 0, ,7550 3,8151 1, mama NT 23/05/ ,60 0, ,7500 0,5150 0, mama NT 18/05/ ,40 0, ,5150 0,9903 0, mama NT 18/05/ ,50 0, ,2300 1,0646 0, mama NT 18/05/ ,60 2, , ,9723 1,86 1 0,93 N/A Low con mama NT 31/05/ ,20 0, ,1700 3,4034 0, mama NT 31/05/ ,50 0, ,5200 2,3904 1, mama NT 31/05/ ,20 0, ,7600 5,6352 1,78 1 0,24 2, mama NT 31/05/ ,50 0, ,9300 1,6386 1, mama NT 31/05/ ,10 0, ,4100 2,1082 1, mama NT 31/05/ ,20 0, ,2600 7,7052 1,81 1 1,04 7, mama NT 31/05/ ,70 0, ,3200 2,2864 1, mama NT 31/05/ ,50 0, ,4400 3,9088 1, mama NT 31/05/ ,30 0, ,9100 2,4182 1, mama T 16/02/ ,10 0, ,6300 1,2100 1,60 0 1,28 8, mama T 16/02/ ,10 0, ,0000 2,3599 1, mama T 16/02/ ,30 0, , ,9071 2,01 1 1,44 9, mama T 24/02/ ,00 0, ,9300 5,7586 1,45 0 0,29 3, mama T 21/02/ ,90 0, , ,0138 1,95 1 1,25 9, mama T 21/02/ ,30 1, , ,9403 1,91 1 1,14 7, mama T 08/03/ ,40 1, , ,8774 1,98 1 1, mama T 03/05/ ,60 1, , ,2589 1,95 1 1,05 7, mama T 17/05/ ,50 1, , ,2163 1,94 1 0,19 2, mama T 11/05/ ,50 0, ,1400 1,4428 0, mama T 23/05/ ,30 2, , ,3105 1,96 1 1,36 8, mama T 18/05/ ,80 0, , ,9618 1,95 1 0,68 5, mama T 23/05/ ,10 0, ,8400 4,9168 1, mama T 23/05/ ,80 2, , ,2283 1,97 1 1, mama T 23/05/ ,70 1, , ,2466 1,96 1 1,72 9, mama T 23/05/ ,60 0, , ,7744 1,96 1 0,77 6, mama T 23/05/ ,70 0, , ,5366 1,98 1 0,85 2, mama T 23/05/ ,80 0, , ,5433 1,93 1 0,75 3, mama T 18/05/ ,30 0, , ,9886 1,73 1 1,08 8, mama T 18/05/ ,70 0, , ,8022 1,87 1 0,83 6, mama T 23/05/ ,90 1, , ,6751 1,98 1 1,00 7, mama T 31/05/ ,80 0, , ,6344 1,88 1 0,02 2, mama T 31/05/ ,60 1, , ,4972 1,97 1 0,80 6,6 2
52 Metodología. Comparar distribución frecuencias entre las muestras T y NT 1 / 5&. 6! 1*!$ 7. "$"8/1***!." 7' $$ 9*:/5 ( *8
53 Resultados en tejido mamario "# $% = <()!311>?:4 = <<)<!3?(>@:4 $%&'&" 70 Rendimiento RNA Cantidad RNA insuficiente para Experion Cantidad RNA suficiente para Experion 60 Número de muestras Total N=26 Insuficiente N=3 Total N=63 Insuficiente N=33 0 TUMOR NO TUMOR
54 Resultados en tejido mamario (3()*+(,*A1>9?4 )# *+ =?()!312>(:4 $%&'&" 70 Pureza RNA (A260/280) A260/280 < 1,74 A260/280 > 1,75 60 Número de muestras Total N=26 < 1,74 N=5 Total N=63 < 1,74 N=41 0 TUMOR NO TUMOR
55 Resultados en tejido mamario,# -!! -B93.$4 )3()>1:4 (139*:4 $%&'&" Integridad RNA (Análisis de RQI ) Mama TUMOR Mama NO TUMOR 23% 26% 12% 33% 13% 52% 17% 53% 19% 22% 46% 70% 8% 6% RQI > 7 4 < RQI > 7 RQI < 4 NO APTO RQI > 7 4 < RQI > 7 RQI < 4
56 Conclusiones = $$.!/$. "> = C $& # " 7$(!$ >.$(!!0!12$2 $1!34 5
57 Perspectivas G.!..#;$ F $ $D7!."!$.. 34 E$D7
58 GRACIAS! Biobanco Universidad de Navarra H; I& GI FF J
59 Taller 3 CONTROLES DE CALIDAD Coordina: Andres C. García Rosa Pinto (Banco Nacional de ADN, Salamanca) María Almeida Parra (Banco Nacional de ADN, Salamanca) María Pérez Caro (Banco Nacional de ADN, Salamanca) Raquel Paternain (Fundación para la Investigación Médica Aplicada, Navarra)
60 Taller 3. CONTROLES DE CALIDAD Controles de calidad en los sistemas de gestión de muestras para garantizar el seguimiento e identificación de muestras María Almeida Parra Banco Nacional de ADN Carlos III. Universidad de Salamanca Palma de Mallorca, 12 noviembre
61 MARCO JURÍDICO Ley 14/2007, de 3 de julio, de Investigación Biomédica Un biobanco tiene que estar organizado como una unidad técnica con criterios de CALIDAD, orden y destino Art. 66 ORGANIZACIÓN DEL BIOBANCO: Tiene que haber un responsable del fichero, un registro de actividades del mismo, y se debe garantizar la CALIDAD, la SEGURIDAD y la TRAZABILIDAD de los datos y muestras biológicas almacenadas y de los procedimientos asociados al funcionamiento del biobanco.
62 QUÉ ES LA TRAZABILIDAD? Ley 14/2007, de 3 de julio, de Investigación Biomédica (Art. 3. Definiciones): Capacidad de asociar un material biológico determinado con información registrada referida a cada paso en la cadena de su obtención, así como a lo largo de todo el proceso de investigación.
63 CALIDAD, SEGURIDAD y TRAZABILIDAD de muestras y datos SISTEMA INFORMÁTICO DE GESTIÓN DE MUESTRAS Y DATOS (LaboratoryIntegrated Management System) Características: Herramienta óptima, flexible y multifuncional: gestión muestras biológicas y datos, incremento eficiencia del biobanco y calidad de los productos. Cumplimiento requerimientos ético legales y técnicos. Ventajas: Incrementa la eficacia ylacalidad del trabajo en biobancos Reduce errores de tipo humano Aumenta el rendimiento de los procesos Control de la trazabilidad de las muestras y datos asociados
64 CALIDAD, SEGURIDAD y TRAZABILIDAD de muestras y datos ACCESO al LIMS ACCESO RESTRINGIDO (usuario y contraseña) Medidas de seguridad: Histórico registros de acceso. Control claves de entrada, autorizaciones y accesos no deseados. Bloqueo acceso con contraseña errónea. Tiempos de caducidad para contraseñas. Desconexión LIMS tiempo máximo de espera excedido.
65 REGISTRO de muestras y datos asociados LIMS: Identificación única e inequívoca de las muestras y de su información asociada como requisito básico de funcionamiento. Registro Manual AUTOMÁTICO NO RECOMENDADO Etiquetas/lectores código barras
66 Datos asociados
67 ADJUNTAR DOCUMENTOS
68
69 PROCESAMIENTO de muestras La correcta gestión de las muestras biológicas es un punto crítico y esencial El LIMS debe: Adaptarse a tipos de muestras y líneas de procesamiento, pero siempre manteniendo la trazabilidad de las muestras y de su información asociada. Proporcionar al usuario un seguimiento completo de la ejecución de todos los procesos que tiene que seguir una muestra de acuerdo al esquema previamente diseñado. Permitir su conexión al equipamiento del biobanco.
70 PROCESAMIENTO muestras: Sistemas AUTOMATIZACIÓN Etiquetas y lectores de código de barras Evitar rotular tubos a mano!!!
71 PROCESAMIENTO muestras: Sistemas AUTOMATIZACIÓN Automatización procesamiento (ROBOTS)
72 ALMACENAMIENTO de muestras y datos El LIMS debe: Tener perfectamente localizada cada alícuota almacenada en el sistema.
73 Laboratory Integrated Management System (LIMS)
74 ALMACENAMIENTO de muestras y datos Información disponible/alícuota almacenada Listado detallado de alícuotas que alberga cada almacén. Lugar exacto de almacenamiento. Código de la alícuota. Código de la muestra de procedencia. Información asociada (datos epidemiológicos, clínicos, etc. del donante de procedencia). Centro de procedencia. Fecha y hora de obtención de la muestra original. Tipo de muestra de procedencia. Tipo de subproducto que alberga la alícuota. Cantidad que contiene. Fecha y hora de almacenamiento. Proyectos de investigación que la han utilizado. etc.
75 ALMACENAMIENTO de muestras y datos El LIMS debe: Tener perfectamente localizada cada alícuota almacenada en el sistema. Controlar movimientos muestras (cambios de posición, proyectos de investigación que las han utilizado, etc.).
76 ALMACENAMIENTO de muestras y datos
77 ALMACENAMIENTO de muestras y datos
78 ALMACENAMIENTO de muestras y datos El LIMS debe: Tener perfectamente localizada cada alícuota almacenada en el sistema. Controlar movimientos muestras (cambios de posición, proyectos de investigación que las han utilizado, etc.). Gestionar sistemas de almacenamiento y espacios. Gestionar la información almacenada
79 ALMACENAMIENTO de muestras Sistemas de identificación de muestras almacenadas Tubos rotulados/etiquetados amano Tubos identificados con código alfanumérico de posición en caja/gradilla Tubos identificados mediante etiquetas con código de barras Tubos identificados con código bidimensional fijo y único
80 Almacenamiento muestras TUBOS código 2D
81 TRAZABILIDAD Etiquetas Lectores código barras LIMS Automatización Tubos código 2D
82 Gestión de SOLICITUDES de muestras y datos 1. Búsqueda de muestras
83 Gestión de SOLICITUDES de muestras y datos 1. Búsqueda de muestras
84 Gestión de SOLICITUDES de muestras y datos 2. Generación solicitud muestras y/o datos
85 Gestión de SOLICITUDES de muestras y datos 2. Generación solicitud muestras y/o datos Posiciones muestras
86 Gestión de SOLICITUDES de muestras y datos 2. Selección muestras a enviar
87 Gestión de SOLICITUDES de muestras y datos 3. VALIDACIÓN de muestras a enviar (Código bidimensional) Ideal para mantener trazabilidad (96)
88 Gestión de SOLICITUDES de muestras y datos 3. VALIDACIÓN de muestras a enviar Microtubos con código bidimensional
89 Gestión de SOLICITUDES de muestras y datos 3. VALIDACIÓN de muestras a enviar
90 Gestión de SOLICITUDES de muestras y datos 3. VALIDACIÓN de muestras a enviar
91 Gestión de SOLICITUDES de muestras y datos 3. VALIDACIÓN de muestras a enviar
92 Gestión de SOLICITUDES de muestras y datos 4. CONTROL DE CALIDAD de muestras a enviar A qué muestras hay que hacer el control de integridad?
93 Gestión de SOLICITUDES de muestras y datos 4. CONTROL DE CALIDAD de muestras a enviar Código 2D
94 Gestión de SOLICITUDES de muestras y datos 5. ALICUOTADO AUTOMÁTICO de muestras a enviar
95 CONCLUSIÓN Los sistemas informáticos de gestión de muestras y datos y de automatización constituyen una herramienta de trabajo imprescindible para el correcto funcionamiento de un biobanco, ya que permiten garantizar la TRAZABILIDAD de todos los procesos e incrementar la CALIDAD de los productos que éste ofrece a sus usuarios.
96 DIRECCIÓN: Prof. Dr. Alberto Orfao de Matos COORDINACIÓN TÉCNICA: Dr. Andrés García Montero Dra. María Almeida Parra GESTIÓN DE CALIDAD: Ana Regalado Mayordomo CONTROL DE CALIDAD: Dra. Rosa Pinto Labajo Dra. María Pérez Caro SECRETARÍA y ADMINISTRACIÓN: Elena Chamorro Castro PERSONAL DE LABORATORIO: Javier García Palomo Teresa Malvar Ferreras Mª Teresa Márquez de Sousa Sheila Mateos Domínguez Mª Isabel Morante Arroyo
97 MUCHAS GRACIAS!! /BancoNacionalADN
98 Taller 3 CONTROLES DE CALIDAD Coordina: Andres C. García - Rosa Pinto (Banco Nacional de ADN, Salamanca) María Almeida Parra (Banco Nacional de ADN, Salamanca) María Pérez Caro (Banco Nacional de ADN, Salamanca) Raquel Paternain (Fundación para la Investigación Médica Aplicada, Navarra)
99 Taller 3: Controles de Calidad Identificación y trazabilidad de las muestras mediante marcadores genéticos Dra. Rosa María Pinto Labajo. Área de Control de Calidad Banco Nacional de ADN Carlos III- Universidad de Salamanca
100 Laboratory Integrated Management System - Etiquetas y lectores de códigos de barras - Microtubos 2D-coded - Automatización (Robots)
101 IDENTIFICACIÓN DE INDIVIDUOS DE DIFERENTE SEXO PCR de Sexo: Utiliza una pareja de oligonucleótidos que amplifica en una región conservada en ambos cromosomas X e Y pero que da lugar a un fragmento de diferente tamaño en cada cromosoma Gen AME: AMEX y AMEY: diferencia de 6 pb por deleción en cromosoma X
102 IDENTIFICACIÓN DE SEXO: PCR de Sexo SEXO: amplificación gen ZF (Fredsten y Villesten, 2004) cromosoma X: inserción de un elemento Alu de 423 pb cromosoma Y: no existe elemento Alu PCR sexo l-hind III pb B pb hombre mujer
103 PERFIL GENÉTICO por MICROSATELITES o STR s STR s: Short Tandem Repeats -secuencias cortas de 2-7 pb que se repiten a lo largo de todo el genoma -el número de repeticiones es único en cada individuo -la amplificación de estas regiones constituye el perfil o huella genética del individuo kits comerciales con combinaciones de 15 y 18 STR s - medicina forense son caros - pruebas de paternidad requieren secuenciación
104 . - AmpFSTR Profiler Plus PCR Amplification Kit (Applied Biosystems) Amplificación de 9 loci STRs: D3S1358, D5S818, D7S820, D8S1179, D13S317, D18S51, D21S11, FGA, y vwa, y el locus del gen de la amelogenina para la identificación del sexo del individuo. -STR analysis using the Promega PowerPlex 16 System. Amplificación de 16 loci (15 loci STR y 1 para el gen de la amelogenina): Penta E, D18S51, D21S11, TH01, D3S1358, FGA, TPOX, D8S1179, vwa, Amelogenin, Penta D, CSF1PO, D16S539, D7S820, D13S317 y D5S818. La detección de la fluorescencia de los genotipos es compatible con los equipos ABI PRISM 310, 3100 y 3100-Avant Genetic Analyzers, Applied Biosystems 3130 and 3130xl Genetic Analyzers and ABI PRISM 377 DNA Sequencer.
105 EPICENTRE Forum 5 (3) Multiplex PCR Amplification of STRs from DNA Isolated with the MasterAmp Buccal Swab DNA Extraction Kit Haiying Grunenwald, Epicentre Technologies CHLC (Cooperative Human Linkage Center) markers Sheffield JC et al (1995), Gastier JM et al (1995) 5 pares de oligonucleótidos PCR MÚLTIPLE 5 parejas de oligonucleotidos: agarosa 3% marcadores: DXS7132 GATA31E08 DYS390 GATA175D03 DXS6789 tamaño producto PCR pb pb pb pb pb carriles 3, 5, 7, 100 bp DNA ladder 2: muestra mujer 4, 6, 8 : muestras hombres
106 VALIDACIÓN DEL PROTOCOLO AND molde 100 ng 50% Hi-Gel Matrix (Biotools) y 0,7% agarosa estándar 4 individuos en dos reacciones de PCR diferentes una con Tm a 55ºC y otra con Tm a 58ºC x174 HaeIII /
107 Tipo de Muestras Sangre EDTA Plasma ADN Sangre Procesamiento/ alicuotado automático Sangre ACD Leucocitos Inmortalización Líneas celulares Microtubos con código bidimiensional (Micronic ) -80ºC Papel FTA 15-30ºC N 2 líquido -196ºC
108 . Amplificación diferentes tipos de muestra: muestras obtenidas con diferentes protocolos
109 . muestras de distintos orígenes Muestras ADN de sangre (100 ng ADN molde) Muestras ADN papel FTA (2 discos de 1.2 mm ADN molde) A B C D A B C D A = D B = C
110 No permite diferenciar hermanos pero sí grado madre/ padre de hijos/as H1 H2 H3 M H M H1 H2 H3 P pb H1 H2 H3: tres hermanos M: madre H: hijo M: marcador tamaño molecular: φ X174 DNA-Hae III H1 H2 H3: tres hermanos P: padre
111 Pérdida de trazabilidad durante el procesamiento de muestras de sangre en EDTA 1- En un momento durante el procesamiento de la muestra de sangre en EDTA se sospecha un posible cruce entre la muestras 2708 y Se continua el procesamiento 3- Se compara el ADN de las muestras procesadas en EDTA con el ADN obtenido a partir de la línea celular (en ACD) 4- Se confirma el error en la codificación de la muestra 5- Se procede al cambio de codificación previamente al proceso de normalización
112 Muestras codificadas erróneamente en el centro de obtención de la muestra 1- Se reciben dos muestras rotuladas con el mismo código 0152 pero con 2 cuestionarios distintos: 0151 y En la aplicación se registran los dos códigos del cuestionario: 0151: A 0152: B 3- Se confirma que A y B son dos individuos distintos como indicaba el cuestionario 4- Se solicita al hospital que contacte M A B con uno de los dos donantes para obtener muestra de nuevo bien pb codificada A B M Se comparan los tres ADN: 0151: B 0152: A 6- Se cambia la relación de códigos en la aplicación
113 Muestras codificadas erróneamente en el centro de obtención de la muestra 1- Se reciben de un mismo centro dos muestras con el mismo código origen 269 C.O 269 = A C.O 270 = B 2- Vuelven a enviar desde el hospital la muestra C.O 269 M A B pb C.O 269= A C.O 269= B C.O 270= A 3- Se cambian los códigos en la aplicación
114 Muestras codificadas erróneamente en el centro de obtención de la muestra 1- Se recibe desde el hospital tres muestras de un proyecto correspondiente a un trío: padre, madre e hija. Estas muestras estas contenidas en tubos no identificados 2- Las muestras se procesan y se lleva a cabo una PCR de sexo para identificar al padre La muestra 2 es el padre 3- Se solicita al hospital una muestra de saliva de cada individuo identificado. 4- Se comparan los perfiles genéticos de las muestras procesadas de sangre (no identificadas) y de saliva (identificadas)
115 muestras saliva P: padre M: madre H: hija muestras sangre 2: padre 1 y 3: madre, hija? pb M H M P 1 = H 2 = P 3 = M 1 H 2 P 3 M
116 Confirmación de las placas alicuotadas previamente al envío de muestras Réplicas de dos cajas micronic A02801 y A01068 En dos placas de 96 pocillos S901CWNJ y S901CWNI
117 Identificación de cajas de envío tras el alicuotado A02801 S901CWNJ A01068 S901CWNI H1 H2 H1 H2 H1 H2 H1 H A02801 = S901CWNJ 1 = 3 2 = 4 * * A01068 = S901CWNI 5 = 7 6 = 8 * * H1 H2 1 = 3 5 = 7 2 = 4 6 = 8
118 Confirmación de las placas alicuotadas previamente al envío de muestras Réplicas en dos placas de muestras de tejido normal y tumoral 1 N 1T 2N 2T 3N 3T N: normal T: tumoral
119 Incidencia durante la inmortalización de líneas celulares (sangre ACD) ALÍCUOTAS LINFOBLASTOS EXTRACCIÓN ADN INFECCIÓN VIRUS EPSTEIN- BARR CULTIVO ALÍCUOTAS LINFOBLASTOS 1- Durante el proceso de inmortalización se sospecha de un cruce entre dos muestras (cada muestra se realiza por duplicado). Los códigos de las muestras son y Las muestras se denominan temporalmente como: a b a b2
120 Incidencia durante inmortalización de líneas celulares 3- Se realiza un perfil genético entre las 4 muestras problema y las muestras y procesadas a partir de sangre EDTA almacenadas en tubos micronic El técnico que realiza la inmortalización sugiere que las muestras a2 y b2 podrían corresponderse a la muestra almacenada en micronic Se cumple que las muestras a2 y b2 son el individuo y las muestras a1 y b1 son el individuo
121 Incidencia durante el proceso de normalización (1) Xxxx muestra de ADN en solución a 4ºC para ser normalizada Xxxx : muestra ya normalizada y alicuotada en micronic Xxxx = Xxxx?? M Xxxx = Xxxx La muestra Xxxx se normaliza y se alicuota en micronic
122 Incidencia durante el proceso de alicuotado Durante el proceso de alicuotado de muestras la aplicación informática detecta que uno de los códigos (11030) correspondiente a una muestra que va a ser alicuotada, y que hasta el momento se ha mantenido en la nevera a 4ºC, se corresponde a una muestra ya almacenada en caja micronic micronic ºC 1- La muestra fue procesada en dos tubos uno de ellos ha sido encontrado en la nevera pero su perfil genético no se corresponde con la muestra del mismo código alicuotada en micronic. 2- En la nevera también se ha encontrado un solo tubo de la muestra (código muy similar) mientras que el otro tubo no ha sido encontrado ni existe registro de que haya sido alicuotado. 3- Se sospecha que uno de los tubos de la muestra se ha mezclado con uno de los tubos de la muestra previamente al alicuotado de la muestra y que probablemente la mezcla de ambos sea la muestra alicuotada en caja micronic correspondiente al código
123 1º eliminamos la muestra del tubo micronic 2º inmortalizan las líneas celulares de y para obtener ADN de cada de una de ellas 3º se comparan los perfiles genéticos de las muestras a 4ºC y el ADN obtenido de la línea celular inmortalizada 4º para cada una de las muestras se unen el ADN almacenado a 4ºC y el ADN obtenido de la línea celular inmortalizada 5º ambas muestras (correspondientes a los códigos y 11130) se normalizan POR SEPARADO!! y se alicuotan en tubos micronic para su almacenamiento a -80ºC
124 Conclusión: Los cinco marcadores STR pertenecientes a Cooperative Human Linkage Center (CHLC) permiten diferenciar mediante PCR múltiple y electroforesis en gel de agarosa de alta resolución perfiles genéticos de diferentes individuos siempre y cuando dichos individuos no sean hermanos entre sí.
125 GRACIAS!
126 DIRECCIÓN: Prof. Dr. Alberto Orfao de Matos COORDINACIÓN TÉCNICA: Dr. Andrés García Montero Dra. María Almeida Parra GESTIÓN DE CALIDAD: Ana Regalado Mayordomo CONTROL DE CALIDAD: Dra. Rosa Pinto Labajo Dra. María Pérez Caro SECRETARÍA y ADMINISTRACIÓN: Elena Chamorro Castro PERSONAL DE LABORATORIO: Javier García Palomo Teresa Malvar Ferreras Mª Teresa Márquez de Sousa Sheila Mateos Domínguez Mª Isabel Morante Arroyo
El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN.
 PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
Taller: CONTROL DE CALIDAD DE MUESTRAS BIOLÓGICAS EN UN BIOBANCO. Situación del control de calidad en la Plataforma Nacional de Biobancos
 Taller: CONTROL DE CALIDAD DE MUESTRAS BIOLÓGICAS EN UN BIOBANCO Situación del control de calidad en la Plataforma Nacional de Biobancos Dr. Andrés C. García Montero. Banco Nacional de ADN Carlos III-
Taller: CONTROL DE CALIDAD DE MUESTRAS BIOLÓGICAS EN UN BIOBANCO Situación del control de calidad en la Plataforma Nacional de Biobancos Dr. Andrés C. García Montero. Banco Nacional de ADN Carlos III-
Taller 5. Controles de calidad para muestras biológicas
 Taller 5. Controles de calidad para muestras biológicas Coordina: Andrés C García Montero, Banco Nacional de ADN, Salamanca. Ponentes: Carmen García Macías, Servicio de Patología Molecular, CIC, Salamanca.
Taller 5. Controles de calidad para muestras biológicas Coordina: Andrés C García Montero, Banco Nacional de ADN, Salamanca. Ponentes: Carmen García Macías, Servicio de Patología Molecular, CIC, Salamanca.
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80
 AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS
 ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS Luis Sáenz Mateos R4 Análisis Clínicos- Hospital General Universitario de Ciudad Real Rotación Externa Hospital Morales Meseguer-Hospital
ESTUDIO DE QUIMERISMO POST TRASPLANTE DE MÉDULA ÓSEA POR ANÁLISIS DE FRAGMENTOS Luis Sáenz Mateos R4 Análisis Clínicos- Hospital General Universitario de Ciudad Real Rotación Externa Hospital Morales Meseguer-Hospital
Informe de Resultados de Análisis de Polimorfismos de ADN en Manchas de Sangre
 SOCIEDAD LATINOAMERICANA DE GENETICA FORENSE LABORATORIO GENETICA MOLECULAR CRUZ ROJA ECUATORIANA Ejercicio Control de Calidad Interlaboratorios Informe de Resultados de Análisis de Polimorfismos de ADN
SOCIEDAD LATINOAMERICANA DE GENETICA FORENSE LABORATORIO GENETICA MOLECULAR CRUZ ROJA ECUATORIANA Ejercicio Control de Calidad Interlaboratorios Informe de Resultados de Análisis de Polimorfismos de ADN
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP
 SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
Laboratorio de Ensayo Acreditado Nº LE-096. Referencia Laboratorio Clínico.
 Laboratorio de Ensayo Acreditado Nº LE-096 El Ente Costarricense de Acreditación, en virtud de la autoridad que le otorga la ley 8279, declara que Referencia Laboratorio Clínico. Ubicado en las instalaciones
Laboratorio de Ensayo Acreditado Nº LE-096 El Ente Costarricense de Acreditación, en virtud de la autoridad que le otorga la ley 8279, declara que Referencia Laboratorio Clínico. Ubicado en las instalaciones
Seminario: Técnicas de Biología Molecular aplicadas al control de calidad e identificación de líneas celulares
 Seminario: Técnicas de Biología Molecular aplicadas al control de calidad e identificación de líneas celulares Detección de Mycoplasma en cultivos celulares mediante PCR a tiempo real Autentificación de
Seminario: Técnicas de Biología Molecular aplicadas al control de calidad e identificación de líneas celulares Detección de Mycoplasma en cultivos celulares mediante PCR a tiempo real Autentificación de
TECNICAS DE BIOLOGIA MOLECULAR
 Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas [email protected] TIPO DE MUTACION DEFINE
Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas [email protected] TIPO DE MUTACION DEFINE
MARCADORES GENÉTICOS
 MARCADORES GENÉTICOS MARCADORES GENÉTICOS La diferencia entre individuos se denotaba señalando diferencias en su aspecto externo (fenotipo) Marcadores Morfológicos Carácter Informativo Variación POLIMORFISMO
MARCADORES GENÉTICOS MARCADORES GENÉTICOS La diferencia entre individuos se denotaba señalando diferencias en su aspecto externo (fenotipo) Marcadores Morfológicos Carácter Informativo Variación POLIMORFISMO
RESULTADOS Y DISCUSIÓN
 RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
INFORME DE RESULTADOS-PRUEBA DE PATERNIDAD
 INFORME DE RESULTADOS-PRUEBA DE PATERNIDAD T. 976 22 11 33 WWW.BIOSALUD.ORG RESIDENCIAL PARAÍSO, 9 50006 ZARAGOZA -SPAIN- INFORME DE RESULTADOS-PRUEBA DE PATERNIDAD Han sido remitidas al Centro de Análisis
INFORME DE RESULTADOS-PRUEBA DE PATERNIDAD T. 976 22 11 33 WWW.BIOSALUD.ORG RESIDENCIAL PARAÍSO, 9 50006 ZARAGOZA -SPAIN- INFORME DE RESULTADOS-PRUEBA DE PATERNIDAD Han sido remitidas al Centro de Análisis
FRAGMENTO OLIGO 5-3 Hebra SECUENCIA 5' - - > 3' TAMAÑO (pb) F1. hmtl 569 L AACCAAACCCCAAAGACACC hmth2982 H CTGATCCAACATCGAGGTCG
 ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
PERFILES GENÉTICOS. El ADN y la ciencia forense. El Perfil de ADN o CODIS (COmbined DNA Index System=Sistema indexado de combinaciones de ADN)
 PERFILES GENÉTICOS El ADN y la ciencia forense El Perfil de ADN o CODIS (COmbined DNA Index System=Sistema indexado de combinaciones de ADN) El CODIS es un conjunto de locus (genes) situados en diferentes
PERFILES GENÉTICOS El ADN y la ciencia forense El Perfil de ADN o CODIS (COmbined DNA Index System=Sistema indexado de combinaciones de ADN) El CODIS es un conjunto de locus (genes) situados en diferentes
Alberts Molecular Biology of the Cell 4ª, 5ª o 6ª Ed. (2002, 2008, 2014) Lehninger Principles of Biochemistry 4ª, 5ª o 6ª Ed. (2004, 2008, 2012)
 Bibliografía: Alberts Molecular Biology of the Cell 4ª, 5ª o 6ª Ed. (2002, 2008, 2014) Lehninger Principles of Biochemistry 4ª, 5ª o 6ª Ed. (2004, 2008, 2012) Lodish Molecular Cell Biology 5ª, 6ª o 7ª
Bibliografía: Alberts Molecular Biology of the Cell 4ª, 5ª o 6ª Ed. (2002, 2008, 2014) Lehninger Principles of Biochemistry 4ª, 5ª o 6ª Ed. (2004, 2008, 2012) Lodish Molecular Cell Biology 5ª, 6ª o 7ª
ESPECTROFOTOMETRO DE NANO VOLUMEN CON FLUOROMETRO Y CUBETA
 ESPECTROFOTOMETRO DE NANO VOLUMEN CON FLUOROMETRO Y CUBETA Aplicaciones: ADN, ARN, la cuantificación ssdna con las mediciones de pureza (260/280 y 260/230). aplicaciones preconfiguradas separados para
ESPECTROFOTOMETRO DE NANO VOLUMEN CON FLUOROMETRO Y CUBETA Aplicaciones: ADN, ARN, la cuantificación ssdna con las mediciones de pureza (260/280 y 260/230). aplicaciones preconfiguradas separados para
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO
 SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr. Criterios generales para el diseño de cebadores para RT qpcr
 Diseño de cebadores para RT qpcr ACTIVIDADES 1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr con SYBR Green 2 Mostración del diseño de un par de cebadores para cuantificar
Diseño de cebadores para RT qpcr ACTIVIDADES 1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr con SYBR Green 2 Mostración del diseño de un par de cebadores para cuantificar
Temas actuales: Next Generation Sequencing (NGS) Bioinformática Elvira Mayordomo
 Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Utilidad de las técnicas de biología molecular en el manejo de bacterias fastidiosas.
 Utilidad de las técnicas de biología molecular en el manejo de bacterias fastidiosas. Estudio rutinario de las bacterias en el laboratorio Examen microscópico (en fresco, Gram, Ziehl- Neelsen, otros) Cultivo
Utilidad de las técnicas de biología molecular en el manejo de bacterias fastidiosas. Estudio rutinario de las bacterias en el laboratorio Examen microscópico (en fresco, Gram, Ziehl- Neelsen, otros) Cultivo
REVISIÓN DE MÉTODOS ESPECTROSCÓPICOS
 REVISIÓN DE MÉTODOS ESPECTROSCÓPICOS OBJETIVOS Aplicar un método espectrofotométrico para medir la concentración de ADN. Conocer el manejo de micropipetas y espectrofotómetros. Espectrofotometría Es la
REVISIÓN DE MÉTODOS ESPECTROSCÓPICOS OBJETIVOS Aplicar un método espectrofotométrico para medir la concentración de ADN. Conocer el manejo de micropipetas y espectrofotómetros. Espectrofotometría Es la
Repaso de PCR y qpcr
 Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
Reacción en cadena de la polimerasa (PCR)
 Curso: Biología Molecular y Genómica Enero 2014. Conceptos elementales sobre la técnica de reacción en cadena de la polimerasa (PCR) Dr. Juan Venegas Hermosilla Programa de Biología Celular y Molecular
Curso: Biología Molecular y Genómica Enero 2014. Conceptos elementales sobre la técnica de reacción en cadena de la polimerasa (PCR) Dr. Juan Venegas Hermosilla Programa de Biología Celular y Molecular
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones
 TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones Dra. Mariana L. Ferramola Curso de Técnicas Moleculares Aplicadas a Bioquímica Clínica. Área de Qca. Biológica
TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones Dra. Mariana L. Ferramola Curso de Técnicas Moleculares Aplicadas a Bioquímica Clínica. Área de Qca. Biológica
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987)
 EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón
 RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
3. MATERIALES Y MÉTODOS
 3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
DNANÁLISE LABORATÓRIO ABRIL
 RESULTADOS Ejercicio de Control de Calidad entre laboratorios 20-2013 DNANÁLISE LABORATÓRIO ABRIL / 2013 (FLORIANÓPOLIS/BRASIL) Ejercicio de Control de Calidad entre laboratorios 20-2013 DETERMINACION
RESULTADOS Ejercicio de Control de Calidad entre laboratorios 20-2013 DNANÁLISE LABORATÓRIO ABRIL / 2013 (FLORIANÓPOLIS/BRASIL) Ejercicio de Control de Calidad entre laboratorios 20-2013 DETERMINACION
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
Código: KIT-005 Ver: 1 MTHFR C677T
 Sistema para detección de la alteración C677T en el gene que codifica para la Metilen-tetrahidrofolato reductasa humana 1 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598)
Sistema para detección de la alteración C677T en el gene que codifica para la Metilen-tetrahidrofolato reductasa humana 1 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598)
Pruebas de Paternidad
 Pruebas de Paternidad Por qué una prueba de paternidad? Confirmación de parentesco biológico Derechos de visita y custodia en procesos de separación conyugal Herencias en litigio Inmigrantes. Reagrupamiento
Pruebas de Paternidad Por qué una prueba de paternidad? Confirmación de parentesco biológico Derechos de visita y custodia en procesos de separación conyugal Herencias en litigio Inmigrantes. Reagrupamiento
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
NOTA DE APLICACIÓN Usando el NanoDrop One para cuantificar Proteínas y Péptidos a 205 nm
 NOTA DE APLICACIÓN Usando el NanoDrop One para cuantificar Proteínas y Péptidos a 205 nm Resumen Los investigadores en ciencias de la vida siempre han necesitado formas de cuantificar rápidamente diversas
NOTA DE APLICACIÓN Usando el NanoDrop One para cuantificar Proteínas y Péptidos a 205 nm Resumen Los investigadores en ciencias de la vida siempre han necesitado formas de cuantificar rápidamente diversas
El ADN como material genético Estructura de los ácidos nucleicos
 El ADN como material genético Estructura de los ácidos nucleicos Bibliografía: Lehninger Principles of Biochemistry 4ª, 5ª o 6ª Ed. (2004, 2008, 2012). Griffiths Introduction to Genetic Analysis 8a Ed.
El ADN como material genético Estructura de los ácidos nucleicos Bibliografía: Lehninger Principles of Biochemistry 4ª, 5ª o 6ª Ed. (2004, 2008, 2012). Griffiths Introduction to Genetic Analysis 8a Ed.
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. [email protected] www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. [email protected] www.atgen.com.uy
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
qprotein (BCA) Kit para cuantificar proteinas totales PB-L Productos Bio-Lógicos
 qprotein () Kit para cuantificar proteinas totales PB-L http://www.pb-l.com.ar qprotein () Cat. no. RA03 CONTENIDO Componentes RA0301 RA0302 Buffer A 250 ml 500 ml Buffer B 5 ml 10 ml Proteína Patrón 5
qprotein () Kit para cuantificar proteinas totales PB-L http://www.pb-l.com.ar qprotein () Cat. no. RA03 CONTENIDO Componentes RA0301 RA0302 Buffer A 250 ml 500 ml Buffer B 5 ml 10 ml Proteína Patrón 5
Métodos analíticos para el control de alergenos en alimentos. Dr. Luis Mata Responsable de I+D ZEULAB
 Métodos analíticos para el control de alergenos en alimentos Dr. Luis Mata Responsable de I+D ZEULAB Por qué analizar alergenos? Seguridad del consumidor Producto final Control de materias primas Asegurar
Métodos analíticos para el control de alergenos en alimentos Dr. Luis Mata Responsable de I+D ZEULAB Por qué analizar alergenos? Seguridad del consumidor Producto final Control de materias primas Asegurar
PLIEGO DE PRESCRIPCIONES TÉCNICAS CONTRATO DE SUMINISTROS. ADQUISICIÓN DE EQUIPOS Nº EXPEDIENTE: 2/2013
 PLIEGO DE PRESCRIPCIONES TÉCNICAS CONTRATO DE SUMINISTROS. ADQUISICIÓN DE EQUIPOS Nº EXPEDIENTE: 2/2013 REF/ PROYECTO: PROMIS 2012 NÚMERO DE LOTES: UNICO Este pliego contempla la adquisición de un sistema
PLIEGO DE PRESCRIPCIONES TÉCNICAS CONTRATO DE SUMINISTROS. ADQUISICIÓN DE EQUIPOS Nº EXPEDIENTE: 2/2013 REF/ PROYECTO: PROMIS 2012 NÚMERO DE LOTES: UNICO Este pliego contempla la adquisición de un sistema
TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR
 TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR Microbiología a General Área Micología Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las
TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR Microbiología a General Área Micología Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las
CYP 2C9 A1075C CYP2C9*3
 CYP 2C9 A1075C Sistema para la detección de la mutación A1075C en el gene del citocromo P450 2C9. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. [email protected]
CYP 2C9 A1075C Sistema para la detección de la mutación A1075C en el gene del citocromo P450 2C9. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. [email protected]
Calidad y eficacia se dan la mano. Espectrofotómetro UV-VIS DR6000
 Calidad y eficacia se dan la mano Espectrofotómetro UV-VIS DR6000 Combinación de calidad y rentabilidad El nuevo espectrofotómetro UV-VIS DR6000 proporciona un rendimiento máximo para la rutina de laboratorio
Calidad y eficacia se dan la mano Espectrofotómetro UV-VIS DR6000 Combinación de calidad y rentabilidad El nuevo espectrofotómetro UV-VIS DR6000 proporciona un rendimiento máximo para la rutina de laboratorio
Técnicas moleculares.
 Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR
 KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
GenomeLab GeXP Sistema de Análisis Genético
 GenomeLab GeXP Sistema de Análisis Genético PLATAFORMA CON MULTIPLES APLICACIONES DETECCIÓN DE VIRUS, BACTERIAS, LEVADURAS, HONGOS, APLICACIONES ESPECIALES, ETC. GenomeLab GeXP GenomeLab GeXP Qué es? Es
GenomeLab GeXP Sistema de Análisis Genético PLATAFORMA CON MULTIPLES APLICACIONES DETECCIÓN DE VIRUS, BACTERIAS, LEVADURAS, HONGOS, APLICACIONES ESPECIALES, ETC. GenomeLab GeXP GenomeLab GeXP Qué es? Es
Código: IDK-003 Ver: 1. Factor V Leiden
 Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
DETECCIÓN DE UN FRAGMENTO DEL GEN ENV DEL VIH EN PLASMA Y GLÓBULOS BLANCOS EN PACIENTES CON VIH/sida. Presenta: Fernando I. Puerto
 DETECCIÓN DE UN FRAGMENTO DEL GEN ENV DEL VIH EN PLASMA Y GLÓBULOS BLANCOS EN PACIENTES CON VIH/sida Presenta: Fernando I. Puerto ANTECEDENTES En los 80 s, esta pandemia se identificó como peligro para
DETECCIÓN DE UN FRAGMENTO DEL GEN ENV DEL VIH EN PLASMA Y GLÓBULOS BLANCOS EN PACIENTES CON VIH/sida Presenta: Fernando I. Puerto ANTECEDENTES En los 80 s, esta pandemia se identificó como peligro para
IES Santa Clara. PAU BIOLOGÍA. 2º BACHILLER.
 ACIDOS NUCLEICOS 1. Representa mediante un dibujo la estructura del ADN, indicando las regiones de la misma donde se encuentran situados los grupos fosfato, las desoxirribosas y las bases nitrogenadas,
ACIDOS NUCLEICOS 1. Representa mediante un dibujo la estructura del ADN, indicando las regiones de la misma donde se encuentran situados los grupos fosfato, las desoxirribosas y las bases nitrogenadas,
Análisis y Identificación de evidencias biológicas en la escena del crimen Jorge Hau Camoretti, MSc Cmdte. Bio. PNP Biólogo Forense
 Análisis y Identificación de evidencias biológicas en la escena del crimen Jorge Hau Camoretti, MSc Cmdte. Bio. PNP Biólogo Forense GAMETOGENESIS ESPERMATOGONIO 44 + XY. OVOGONIO 44+XX ESPERMATOCITO
Análisis y Identificación de evidencias biológicas en la escena del crimen Jorge Hau Camoretti, MSc Cmdte. Bio. PNP Biólogo Forense GAMETOGENESIS ESPERMATOGONIO 44 + XY. OVOGONIO 44+XX ESPERMATOCITO
Análisis de paternidad y parentesco en alpacas (Vicugna pacos) mediante marcadores moleculares
 Análisis de paternidad y parentesco en alpacas (Vicugna pacos) mediante marcadores moleculares MSc. Juan Agapito Panta Laboratorio de Genómica y Biología Molecular Instituto Peruano de Energía Nuclear
Análisis de paternidad y parentesco en alpacas (Vicugna pacos) mediante marcadores moleculares MSc. Juan Agapito Panta Laboratorio de Genómica y Biología Molecular Instituto Peruano de Energía Nuclear
INFORME DE PATERNIDAD BIOLÓGICA
 INFORME DE PATERNIDAD BIOLÓGICA Comparecieron en el centro de extracciones de los laboratorios LABCO, para ser debidamente identificados: Nombre de la hija: XXXXXXXXX DNI de la hija: Número de petición
INFORME DE PATERNIDAD BIOLÓGICA Comparecieron en el centro de extracciones de los laboratorios LABCO, para ser debidamente identificados: Nombre de la hija: XXXXXXXXX DNI de la hija: Número de petición
Biología Forense. Dra. Ana María López Parra. Laboratorio de Genética Forense
 Dra. Ana María López Parra Laboratorio de Genética Forense Departamento de Toxicología y Legislación Sanitaria. Facultad de Medicina. Universidad Complutense de Madrid (UCM) Ventajas del análisis de ADN
Dra. Ana María López Parra Laboratorio de Genética Forense Departamento de Toxicología y Legislación Sanitaria. Facultad de Medicina. Universidad Complutense de Madrid (UCM) Ventajas del análisis de ADN
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Introducción a la Secuenciación Masiva y a la Bioinformática
 Introducción a la Secuenciación Masiva y a la Bioinformática Dietmar Fernández Orth, PhD 24 de Abril de 2014 1 El DNA (Ácido desoxirribonucleico) contiene la información genética usada en el desarrollo
Introducción a la Secuenciación Masiva y a la Bioinformática Dietmar Fernández Orth, PhD 24 de Abril de 2014 1 El DNA (Ácido desoxirribonucleico) contiene la información genética usada en el desarrollo
Muestra humanas de origen único Marcadores autosómicos
 XVIII Jornadas del GHEP-ISFG Sevilla, 18-20 septiembre 2013 GRUPO DE HABLA ESPAÑOL A Y PORTUGUESA DE LA ISFG GRUPO DE LÍNGUAS ESPANHOLA E PORTUGUESA DA ISFG Muestra humanas de origen único Marcadores autosómicos
XVIII Jornadas del GHEP-ISFG Sevilla, 18-20 septiembre 2013 GRUPO DE HABLA ESPAÑOL A Y PORTUGUESA DE LA ISFG GRUPO DE LÍNGUAS ESPANHOLA E PORTUGUESA DA ISFG Muestra humanas de origen único Marcadores autosómicos
Código: IDK-006 Ver: 1 MTHFR A1298C. Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa
 Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
Métodos de secuenciación de ADN
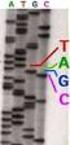 Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA
 UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
La Patología a Molecular y el Control de Calidad
 UNIDAD DIDÁCTICA 7: LOS PROCESOS EN ANATOMÍA PATOLÓGICA La Patología a Molecular y el Control de Calidad Asunción n Olmo Sevilla Master Diagnóstica S. L. Patología Molecular: Patología del futuro X Personal:
UNIDAD DIDÁCTICA 7: LOS PROCESOS EN ANATOMÍA PATOLÓGICA La Patología a Molecular y el Control de Calidad Asunción n Olmo Sevilla Master Diagnóstica S. L. Patología Molecular: Patología del futuro X Personal:
Simposio El papel del patólogo en el tratamiento de los tumores cerebrales
 CUESTIONARIO DE AUTOEVALUACIÓN Simposio El papel del patólogo en el tratamiento de los tumores cerebrales Madrid, 4 de febrero de 2010 1. El análisis del 1p y 19q está indicado actualmente para el manejo
CUESTIONARIO DE AUTOEVALUACIÓN Simposio El papel del patólogo en el tratamiento de los tumores cerebrales Madrid, 4 de febrero de 2010 1. El análisis del 1p y 19q está indicado actualmente para el manejo
NTE AS Centro Producción de Componentes Sanguíneos NORMAS TECNICAS ESPECÍFICAS AUTORIZACION SANITARIA UNIDADES DE PRODUCCION COMPONENTES SANGUÍNEOS
 NORMAS TECNICAS ESPECÍFICAS AUTORIZACION SANITARIA UNIDADES DE PRODUCCION COMPONENTES SANGUÍNEOS I. INTRODUCCIÓN Definición: La Unidad de Procesamiento de Componentes Sanguíneos está constituida por las
NORMAS TECNICAS ESPECÍFICAS AUTORIZACION SANITARIA UNIDADES DE PRODUCCION COMPONENTES SANGUÍNEOS I. INTRODUCCIÓN Definición: La Unidad de Procesamiento de Componentes Sanguíneos está constituida por las
Espectrofotómetros V-10 PLUS, UV-20 y UV-30 SCAN
 Espectrofotómetros V-10 PLUS, UV-20 y UV-30 SCAN GARANTÍA AÑOS Características Componentes pre-alineados: Emisor de Luz y la lámpara de deuterio Monocromador : Rejilla con 1200 líneas/mm Dos parámetros
Espectrofotómetros V-10 PLUS, UV-20 y UV-30 SCAN GARANTÍA AÑOS Características Componentes pre-alineados: Emisor de Luz y la lámpara de deuterio Monocromador : Rejilla con 1200 líneas/mm Dos parámetros
CONTENIDO DE HIERRO EN LAS LECHES DE FORMULA EMPLEADAS EN LA ALIMENTACIÓN INFANTIL: DISTRIBUCION EN EL SUERO LACTEO Y EN LA GRASA
 CONTENIDO DE HIERRO EN LAS LECHES DE FORMULA EMPLEADAS EN LA ALIMENTACIÓN INFANTIL: DISTRIBUCION EN EL SUERO LACTEO Y EN LA GRASA R. Domínguez 3,1, J.M. Fraga 1,2, J.A. Cocho 1,2, P. Bermejo 3, A. Bermejo
CONTENIDO DE HIERRO EN LAS LECHES DE FORMULA EMPLEADAS EN LA ALIMENTACIÓN INFANTIL: DISTRIBUCION EN EL SUERO LACTEO Y EN LA GRASA R. Domínguez 3,1, J.M. Fraga 1,2, J.A. Cocho 1,2, P. Bermejo 3, A. Bermejo
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR)
 METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
ANEXO TÉCNICO ACREDITACIÓN Nº 665/LE1398. Entidad: ERTZAINTZA - POLICÍA AUTÓNOMA DEL PAÍS VASCO. UNIDAD DE POLICÍA CIENTÍFICA
 Hoja 1 de 3 ANEXO TÉCNICO ACREDITACIÓN Nº 665/LE1398 Entidad: ERTZAINTZA - POLICÍA AUTÓNOMA DEL PAÍS VASCO. UNIDAD DE POLICÍA CIENTÍFICA Dirección: C/ Larrauri Mendotxe Bidea, 18; 48950 Erandio (VIZCAYA)
Hoja 1 de 3 ANEXO TÉCNICO ACREDITACIÓN Nº 665/LE1398 Entidad: ERTZAINTZA - POLICÍA AUTÓNOMA DEL PAÍS VASCO. UNIDAD DE POLICÍA CIENTÍFICA Dirección: C/ Larrauri Mendotxe Bidea, 18; 48950 Erandio (VIZCAYA)
BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA
 BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA PROFESORADO CURSO 1994-1995: Dr. J. Ariño (Unidad de Bioquímica) Dra. F. Bosch (Unidad de Bioquímica) Dr. A. Sánchez (Unidad de Genética y Mejora)
BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA PROFESORADO CURSO 1994-1995: Dr. J. Ariño (Unidad de Bioquímica) Dra. F. Bosch (Unidad de Bioquímica) Dr. A. Sánchez (Unidad de Genética y Mejora)
NAT: Experiencia en el Centro Regional de Hemoterapia Garrahan
 NAT: Experiencia en el Centro Regional de Hemoterapia Garrahan Jornadas SAV-AAHI. Actualización en la detección de ácidos nucleicos virales, su alcance en el tamizaje de infecciones en gran escala. 6 de
NAT: Experiencia en el Centro Regional de Hemoterapia Garrahan Jornadas SAV-AAHI. Actualización en la detección de ácidos nucleicos virales, su alcance en el tamizaje de infecciones en gran escala. 6 de
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006
 IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
I SIMPOSIO Biopsia líquida
 I SIMPOSIO Biopsia líquida Detección de mutaciones en biopsia líquida con tecnología CLART SANTIAGO DE COMPOSTELA, 30/01/16 Biopsia líquida: ADNtc Biopsia líquida comprende tres biomarcadores diferentes:
I SIMPOSIO Biopsia líquida Detección de mutaciones en biopsia líquida con tecnología CLART SANTIAGO DE COMPOSTELA, 30/01/16 Biopsia líquida: ADNtc Biopsia líquida comprende tres biomarcadores diferentes:
2. NIVEL MEDIO 1. NIVEL BASICO BIOLOGIA MOLECULAR 2.1 DNA SALIVA
 BIOLOGIA MOLECULAR Hemos elaborado un programa en 3 niveles, en función del tipo de estudiante y las posibilidades del centro educativo: 1. NIVEL BASICO 2. NIVEL MEDIO DANAEXTRACTOR KIT Práctica que constituye
BIOLOGIA MOLECULAR Hemos elaborado un programa en 3 niveles, en función del tipo de estudiante y las posibilidades del centro educativo: 1. NIVEL BASICO 2. NIVEL MEDIO DANAEXTRACTOR KIT Práctica que constituye
TAXONOMÍA MOLECULAR. Dra. Alicia Luque CEREMIC
 TAXONOMÍA MOLECULAR Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las condiciones ambientales. PLEOMORFISMO Debido al bajo poder
TAXONOMÍA MOLECULAR Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las condiciones ambientales. PLEOMORFISMO Debido al bajo poder
Laboratorio Clínico Gendiag.exe, S.L. GENDIAG.LAB. Cartera Prestación Servicios
 Laboratorio Clínico Gendiag.exe, S.L. GENDIAG.LAB Cartera Prestación Servicios 10/10/2014 RESUMEN CARTERA SERVICIOS PRUEBA DESCRIPCIÓN TECNOLOGÍA (MÉTODO) Análisis (genotipado) de 113 marcadores genéticos
Laboratorio Clínico Gendiag.exe, S.L. GENDIAG.LAB Cartera Prestación Servicios 10/10/2014 RESUMEN CARTERA SERVICIOS PRUEBA DESCRIPCIÓN TECNOLOGÍA (MÉTODO) Análisis (genotipado) de 113 marcadores genéticos
Desarrollo y Validación de Técnicas Analíticas
 Desarrollo y Validación de Técnicas Analíticas Nerea Leal Eguiluz DynaKin, S.L. Farmacoterapia Personalizada en Oncología 3 de julio de 2009 Definiciones Método Bioanálitico: conjunto de todos los procedimientos
Desarrollo y Validación de Técnicas Analíticas Nerea Leal Eguiluz DynaKin, S.L. Farmacoterapia Personalizada en Oncología 3 de julio de 2009 Definiciones Método Bioanálitico: conjunto de todos los procedimientos
Actividades del CNA en la detección de alérgenos alimentarios: Detección de Avellana mediante PCR
 Actividades del CNA en la detección de alérgenos alimentarios: Detección de Avellana mediante PCR Mª del Camino Martín-Forero Servicio de Biotecnología Reglamento (UE) Nº 1169/2011 del Parlamento Europeo
Actividades del CNA en la detección de alérgenos alimentarios: Detección de Avellana mediante PCR Mª del Camino Martín-Forero Servicio de Biotecnología Reglamento (UE) Nº 1169/2011 del Parlamento Europeo
PCR gen 18S ARNr humano
 PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
Kit artus EBV QS-RGQ. Características de rendimiento. Mayo Sample & Assay Technologies. Sensibilidad analítica (plasma)
 Kit artus EBV QS-RGQ Características de rendimiento Kit artus EBV QS-RGQ, versión 1, 4501363 Compruebe la disponibilidad de nuevas revisiones electrónicas de las especificaciones en www.qiagen.com/products/artusebvpcrkitce.aspx
Kit artus EBV QS-RGQ Características de rendimiento Kit artus EBV QS-RGQ, versión 1, 4501363 Compruebe la disponibilidad de nuevas revisiones electrónicas de las especificaciones en www.qiagen.com/products/artusebvpcrkitce.aspx
PRÁCTICA No 7: PRUEBAS DE RELACION FILIAL E IDENTIFICACION
 PRÁCTICA No 7: PRUEBAS DE RELACION FILIAL E IDENTIFICACION 1. INTRODUCCION Las pruebas de relación filial son un instrumento muy útil para determinar la relación familiar ya sea una paternidad, identificación
PRÁCTICA No 7: PRUEBAS DE RELACION FILIAL E IDENTIFICACION 1. INTRODUCCION Las pruebas de relación filial son un instrumento muy útil para determinar la relación familiar ya sea una paternidad, identificación
Aplicación de la biología molecular en la detección del quimerismo hematopoyético post-transplante alogénico de médula ósea.
 Aplicación de la biología molecular en la detección del quimerismo hematopoyético post-transplante alogénico de médula ósea. Dra. Karina Casanueva Calero. Especialista de 1er grado en Bioquímica Clínica
Aplicación de la biología molecular en la detección del quimerismo hematopoyético post-transplante alogénico de médula ósea. Dra. Karina Casanueva Calero. Especialista de 1er grado en Bioquímica Clínica
Septiembre 2015 artus CMV QS-RGQ Kit: Características de rendimiento
 Septiembre 2015 artus CMV QS-RGQ Kit: Características de rendimiento artus CMV QS-RGQ Kit, versión 1 4503363 Compruebe la disponibilidad de nuevas versiones de la documentación electrónica en www.qiagen.com/products/artuscmvpcrkitce.aspx
Septiembre 2015 artus CMV QS-RGQ Kit: Características de rendimiento artus CMV QS-RGQ Kit, versión 1 4503363 Compruebe la disponibilidad de nuevas versiones de la documentación electrónica en www.qiagen.com/products/artuscmvpcrkitce.aspx
TEST de PATERNIDAD. Ref.PCR paternidad (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO
 Ref.PCR paternidad (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO TEST de PATERNIDAD Este experimento introduce a los alumnos en el uso del ADN y la PCR para simular una determinación de paternidad. Los estudiantes
Ref.PCR paternidad (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO TEST de PATERNIDAD Este experimento introduce a los alumnos en el uso del ADN y la PCR para simular una determinación de paternidad. Los estudiantes
