BF GBP1 Hs AU INDO Hs.840 dioxygenase down
|
|
|
- Pablo Francisco Álvarez Martin
- hace 5 años
- Vistas:
Transcripción
1 Supplementary Table 2. Post- RASP SEB specific genes which passed the four different comparative tests and 1.5 #Fold- Change (FC) cut- off : 1.5 #Pairing option : Pairs of conditions S- 2: post- RASP- SEB exposed #[S- 2] vs [C- 1] leukocytes C- 1: untreated pre- RASP leukocytes (control) S- 1: pre- RASP SEB- exposed leukocytes #[S- 2] vs [C- 2] (SEB) C- 2: untreated post- RASP leukocytes (stress) #[S- 2] vs [S- 1] GeneBankAcceSymbol UniGene Description FC ([S- 2] vfc ([S- 2] vsfc ([S- 2] vs [S- 1]) BF WARS Hs Tryptophanyl- trna synthetase down NM_ CXCL10 Chemokine (C- X- C motif) ligand Hs down Chemokine (C- X- C motif) ligand NM_ CXCL9 Hs down BF GBP1 Hs Guanylate binding protein 1, interferon- inducible, 67kDa down AV CCL8 Hs Chemokine (C- C motif) ligand down Indoleamine- pyrrole 2,3 AU INDO Hs.840 dioxygenase down Colony stimulating factor 2 BE CSF2 Hs.1349 (granulocyte- macrophage) down AL XCL1 CHEMOKINE (C MOTIF) LIGAND down Interleukin 1 receptor BG IL1RN Hs antagonist down NM_ IL1A Hs.1722 Interleukin 1, alpha down Suppressor of cytokine signaling AB SOCS3 Hs down AV HLA- DPA1 Major histocompatibility Hs complex, class II, DP alpha down Solute carrier organic anion transporter family, member AF SLCO1A2 Hs A down Early growth response 2 (Krox- AF EGR2 Hs homolog, Drosophila) down PROSTAGLANDIN- ENDOPEROXIDE SYNTHASE 2 (PROSTAGLANDIN G/H SYNTHASE AND AL PTGS2 CYCLOOXYGENASE) down L26165 CDKN1A Hs Cyclin- dependent kinase inhibitor 1A (p21, Cip1) down NM_ SELL Hs Selectin L (lymphocyte adhesion molecule 1) down
2 BG SOD2 Superoxide dismutase 2, Hs mitochondrial down Receptor- interacting serine- BG RIPK2 Hs threonine kinase down CD74 molecule, major histocompatibility complex, BG CD74 Hs class II invariant chain ####### down Leukocyte immunoglobulin- like receptor, subfamily B (with TM AF LILRB3 and ITIM domains), member down AV GPR171 Hs G protein- coupled receptor down NM_ GK Hs.1466 Glycerol kinase down Leukocyte immunoglobulin- like receptor, subfamily B (with TM NM_ LILRB3 Hs and ITIM domains), member down XM_ SCYB5 small inducible cytokine subfamily B (Cys- X- Cys), down BF HLA- DRA Major histocompatibility Hs complex, class II, DR alpha down Proteoglycan 1, secretory AV PRG1 Hs.1908 granule ####### down MAJOR HISTOCOMPATIBILITY X87344 HLA- DMA COMPLEX, CLASS II, DM ALPHA down Major histocompatibility M20503 HLA- DRB1 Hs complex, class II, DR beta down AV IL8 Hs.624 Interleukin down Protein tyrosine phosphatase, NM_ PTPRC Hs receptor type, C down tumor necrosis factor, alpha- XM_ TNFAIP6 induced protein down NM_ BTG2 Hs BTG family, member down Chemokine (C- X- C motif) ligand 1 (melanoma growth BG CXCL1 Hs.789 stimulating activity, alpha) down BE TRIM21 Hs Tripartite motif- containing down Acyl- CoA synthetase long- chain BE ACSL1 Hs family member down Integrin, alpha M (complement component 3 receptor 3 NM_ ITGAM Hs subunit) down BE HLA- B Major histocompatibility Hs complex, class I, B down BF HLA- DRB1 Major histocompatibility Hs complex, class II, DR beta down BG AKAP13 A kinase (PRKA) anchor protein Hs down XM_ LOC similar to phosphodiesterase 4D interacting protein down
3 W38319 IL1B Hs Interleukin 1, beta down Similar to Ig heavy chain V- II AL LOC Hs region ARH- 77 precursor down AV B2M Hs Beta- 2- microglobulin down BG LYN Hs V- yes- 1 Yamaguchi sarcoma viral related oncogene homolog down Suppression of tumorigenicity AL ST14 Hs (colon carcinoma) down XM_ PPBP pro- platelet basic protein (includes platelet basic down AL SERPINB2 Serpin peptidase inhibitor, Hs clade B (ovalbumin), member ####### down N32077 IER3 Hs Immediate early response down BG SLC7A11 Solute carrier family 7, (cationic amino acid transporter, y+ Hs system) member down Nuclear factor of kappa light polypeptide gene enhancer in B- NM_ NFKB1 Hs cells 1 (p105) down AU KLF6 Hs.4055 Kruppel- like factor down CD48 ANTIGEN (B- CELL AL CD48 MEMBRANE PROTEIN) down BG HLA- B Hs Major histocompatibility complex, class I, B down XM_ CCR7 chemokine (C- C motif) receptor down Gardner- Rasheed feline sarcoma viral (v- fgr) oncogene AI FGR Hs.1422 homolog down Special AT- rich sequence binding protein 1 (binds to nuclear matrix/scaffold- BE SATB1 Hs associating DNA's) down Major histocompatibility AW HLA- C Hs complex, class I, C down Hematopoietic cell- specific Lyn AL HCLS1 Hs substrate down M12824 CD8A Hs CD8a molecule down AI IGHG1 Immunoglobulin heavy constant Hs gamma 1 (G1m marker) down NM_ AQP9 Hs Aquaporin down V- ets erythroblastosis virus E26 AL ETS2 Hs oncogene homolog 2 (avian) down Major histocompatibility BF HLA- DRB1 Hs complex, class II, DR beta down XM_ PRKCH protein kinase C, eta down
4 NM_ DUSP6 Hs Dual specificity phosphatase down Fc fragment of IgG, low affinity J04162 FCGR3A Hs IIIa, receptor (CD16a) down NM_ IRF1 Hs Interferon regulatory factor down AW LTB Lymphotoxin beta (TNF Hs superfamily, member 3) down Fc fragment of IgE, high affinity I, receptor for; gamma AV FCER1G Hs polypeptide down XM_ HLX1 H2.0 (Drosophila)- like homeo box down AA CDKN1A Hs Cyclin- dependent kinase inhibitor 1A (p21, Cip1) down Leukocyte immunoglobulin- like receptor, subfamily B (with TM NM_ LILRB1 Hs and ITIM domains), member down M83664 HLA- DPB1 Major histocompatibility Hs complex, class II, DP beta down Platelet factor 4 (chemokine (C- AI PF4 Hs X- C motif) ligand 4) down BG NINJ1 Hs Ninjurin down AL FAS Fas (TNF receptor superfamily, Hs member 6) down BE PLAUR Plasminogen activator, Hs urokinase receptor down AF CCR- 5 mutant protein containing 215 amino acids (versus wild- type 352 amino acids) in which the C- terminal 31 amino acids are translated out of frame.; Homo sapiens CC- chemokine receptor (CCR- 5) gene, delta- 32 allele, complete cds down Leukocyte immunoglobulin- like receptor, subfamily B (with TM NM_ LILRB1 Hs and ITIM domains), member down AA SLC31A2 Hs Solute carrier family 31 (copper transporters), member down NM_ CD40 Hs CD40 molecule, TNF receptor superfamily member down NM_ IRF7 Hs Interferon regulatory factor down M57888 CTLA1 Human (clone lambda B34) cytotoxic T- lymphocyte- associated serine esterase 1 (CTLA1) gene, complete cds down Colony stimulating factor 2 receptor, beta, low- affinity AW CSF2RB Hs (granulocyte- macrophage) down
5 AL CTSL Hs Cathepsin L down U83582 HLA- DQB1 Major histocompatibility Hs complex, class II, DQ beta down NM_ LCP2 Lymphocyte cytosolic protein 2 (SH2 domain containing Hs leukocyte protein of 76kDa) down BG S100A9 Hs S100 calcium binding protein A down BF CASP4 Caspase 4, apoptosis- related Hs cysteine peptidase down NM_ PLEK Hs Pleckstrin down Colony stimulating factor 1 BE CSF1 Hs (macrophage) down BG TRIM22 Hs Tripartite motif- containing down BF IGHA1 Immunoglobulin heavy constant Hs alpha down U72882 IFI35 Hs Interferon- induced protein down NM_ IKZF1 IKAROS family zinc finger 1 (Ikaros) down NM_ CASP5 Caspase 5, apoptosis- related Hs cysteine peptidase down BG UBD Hs Ubiquitin D down TCR gamma alternate reading AI TARP Hs frame protein down NM_ PLK3 Hs Polo- like kinase 3 (Drosophila) down NM_ TLR2 Hs Toll- like receptor down AC HBGF heparin- binding EGF- like growth factor precursor; Homo sapiens chromosome 5, BAC clone 189n23 (LBNL H168), complete sequence down NM_ IL2RB Hs Interleukin 2 receptor, beta down XM_ LOC similar to chemokine (C- C motif) receptor 1 (H down BF NAPA N- ethylmaleimide- sensitive factor attachment protein, Hs alpha down NM_ MICA MHC class I polypeptide- related Hs sequence A down NM_ KIAA0247 Hs KIAA down NM_ PF4V1 Hs Platelet factor 4 variant down BF BCL2A1 Hs BCL2- related protein A down AL CREM camp RESPONSIVE ELEMENT MODULATOR down AF CCRL2 Chemokine (C- C motif) receptor- Hs like down
6 AF TLR4 synonym: Lps; encodes most common amino acid sequence in humans; membrane spanning component of the human LPS receptor; human homolog of the mouse Lps gene product; Homo sapiens toll- like receptor 4 (TLR4) gene, TLR4A allele, complete cds down AI PPP1R16B Hs Protein phosphatase 1, regulatory (inhibitor) subunit 16B down BF SAT1 Hs Spermidine/spermine N1- acetyltransferase down AA STAT1 Hs Signal transducer and activator of transcription 1, 91kDa down AL CTSW Hs Cathepsin W down XM_ HKR3 GLI- Kruppel family member HKR3 (HKR3), mrna down AL LRRC8C Leucine rich repeat containing 8 Hs family, member C down NM_ IL4R Hs Interleukin 4 receptor down PLATELET- ACTIVATING FACTOR BG PTAFR Hs RECEPTOR down XM_ NBS1 Nijmegen breakage syndrome 1 (nibrin) down BG HLA- DOB Hs.1802 Major histocompatibility complex, class II, DO beta down XM_ CD226 CD226 antigen; DNAX accessory molecule- 1; T lineage- specific activation antigen 1 antigen; adhesion glycoprotein; platelet and T cell activation antigen down AI PSCDBP Hs.270 Pleckstrin homology, Sec7 and coiled- coil domains, binding protein down Major histocompatibility AI HLA- DMB Hs complex, class II, DM beta down AL LAIR1 Hs Leukocyte- associated immunoglobulin- like receptor down Chemokine (C- C motif) ligand 18 (pulmonary and activation- NM_ CCL18 Hs regulated) down Interferon stimulated NM_ ISG20 Hs exonuclease gene 20kDa down XM_ CTSH cathepsin H down
7 NM_ ELL2 Elongation factor, RNA Hs polymerase II, down AI PSMB9 Proteasome (prosome, macropain) subunit, beta type, 9 (large multifunctional Hs peptidase 2) down Potassium channel tetramerisation domain AA KCTD12 Hs containing down N39944 ATF3 Hs.460 Activating transcription factor down X63741 EGR3 Hs Early growth response down Leukocyte immunoglobulin- like receptor, subfamily A (without NM_ LILRA3 Hs TM domain), member down AA ATF1 Hs Activating transcription factor down Purinergic receptor P2Y, G- BF P2RY10 Hs protein coupled, down AI IFIT2 Interferon- induced protein with Hs tetratricopeptide repeats down BF MX1 Myxovirus (influenza virus) resistance 1, interferon- Hs inducible protein p78 (mouse) down NM_ BATF Basic leucine zipper Hs transcription factor, ATF- like down BG CTNS Hs Cystinosis, nephropathic down NM_ STK4 Hs Serine/threonine kinase down Caspase 1, apoptosis- related cysteine peptidase (interleukin NM_ CASP1 Hs , beta, convertase) down NM_ PTGER4 Hs Prostaglandin E receptor 4 (subtype EP4) down Interleukin 6 (interferon, beta M54894 IL6 Hs ) down AW EMP1 Hs Epithelial membrane protein down NM_ MAPKAPK3 Mitogen- activated protein kinase- activated protein kinase Hs down B- cell CLL/lymphoma 6 (zinc NM_ BCL6 Hs finger protein 51) down AV GCH1 Hs GTP cyclohydrolase 1 (dopa- responsive dystonia) down S82692 IL2 Hs Interleukin down NM_ CD83 Hs CD83 molecule down AB KIAA0323 Hs KIAA down Plasminogen activator, NM_ PLAU Hs urokinase down
8 NM_ MTF1 Hs Metal- regulatory transcription factor down NM_ PIM1 Hs Pim- 1 oncogene down NM_ SLC16A6 Hs Solute carrier family 16, member 6 (monocarboxylic acid transporter 7) down AP KIAA0136 Homo sapiens genomic DNA, chromosome 21q22.2, BAC clone:kb739c11, CBR1- HLCS region down T- cell activation NFKB- like AI TA- NFKBH Hs protein down NM_ TNF Tumor necrosis factor (TNF Hs superfamily, member 2) down NM_ SERPINB8 Serpin peptidase inhibitor, Hs clade B (ovalbumin), member down AY APOL3 Hs Apolipoprotein L, down XM_ PTPN1 protein tyrosine phosphatase, non- receptor type down Aldehyde dehydrogenase 3 AU ALDH3B2 Hs family, member B down Adaptor- related protein AI AP1G2 Hs complex 1, gamma 2 subunit down AF LST1 Hs Leukocyte specific transcript down AB FNBP1 Hs Formin binding protein down Fucosyltransferase 4 (alpha (1,3) fucosyltransferase, AA FUT4 Hs myeloid- specific) down XM_ ELK3 ELK3, ETS- domain protein (SRF accessory protein 2) down Hypothetical protein AK MGC48972 Hs MGC down AA CTBS Hs Chitobiase, di- N- acetyl down S59049 BL34 B cell activation gene; BL34=B cell activation gene [human, mrna, 1398 nt] down XM_ LOC similar to PHORBOLIN down Tumor necrosis factor (ligand) NM_ TNFSF13 Hs superfamily, member down AA PSME1 Hs Proteasome (prosome, macropain) activator subunit 1 (PA28 alpha) down BE RALB Hs V- ral simian leukemia viral oncogene homolog B (ras related; GTP binding protein) down BF SLC2A3P1 Hs Solute carrier family 2 (facilitated glucose transporter), member 3 pseudogene down
9 AA BCL2L11 BCL2- like 11 (apoptosis Hs facilitator) down Ubiquitin- conjugating enzyme AL UBE2A Hs E2A (RAD6 homolog) down AF USP15 Hs Ubiquitin specific peptidase down AI FCER2 Fc fragment of IgE, low affinity Hs II, receptor for (CD23) down ER degradation enhancer, BG EDEM1 Hs mannosidase alpha- like down Major histocompatibility NM_ HLA- DMB Hs complex, class II, DM beta down Chemokine (C- C motif) ligand 4- AV CCL4L2 Hs like down AA ZCCHC10 Hs Zinc finger, CCHC domain containing down U90912 KIAA1128 Hs KIAA down AI FOSL2 Hs FOS- like antigen down U25029 NR3C1 Nuclear receptor subfamily 3, group C, member 1 Hs (glucocorticoid receptor) down AU TRIB1 Hs Tribbles homolog 1 (Drosophila) down M24364 HLA- DQB1 Major histocompatibility Hs complex, class II, DQ beta down AI SCLY Hs Selenocysteine lyase down TRAF- type zinc finger domain AU TRAFD1 Hs.5148 containing down ATPase, H+ transporting, lysosomal 56/58kDa, V1 subunit BE ATP6V1B2 Hs B down U16996 DUSP5 Hs.2128 Dual specificity phosphatase down Phosphodiesterase 4B, camp- specific (phosphodiesterase E4 L12686 PDE4B Hs dunce homolog, Drosophila) down BF HLA- DOA Major histocompatibility Hs complex, class II, DO alpha down CDC42 effector protein (Rho AF CDC42EP2 Hs GTPase binding) down NM_ IL18R1 Hs Interleukin 18 receptor down Mitogen- activated protein NM_ MAPK12 Hs kinase down AI IL32 Hs.943 Interleukin down AF NRP2 Hs Neuropilin down NM_ LIMK2 Hs LIM domain kinase down Granzyme A (granzyme 1, cytotoxic T- lymphocyte- AA GZMA Hs associated serine esterase 3) down NM_ IL15RA Hs Interleukin 15 receptor, alpha down XM_ TXK TXK tyrosine kinase down
10 N20599 CTSO Hs Cathepsin O down XM_ GGTLA1 gamma- glutamyltransferase- like activity down NM_ KIAA0125 KIAA down AB CTSL2 Hs Cathepsin L down XM_ LOC similar to nuclear receptor subfamily 4, group A, down B- cell CLL/lymphoma 6 (zinc AI BCL6 Hs finger protein 51) down NM_ CIITA Hs Class II, major histocompatibility complex, transactivator down CD40 ligand (TNF superfamily, member 5, hyper- IgM NM_ CD40LG Hs syndrome) down BE TAF4B TAF4b RNA polymerase II, TATA box binding protein (TBP)- Hs associated factor, 105kDa down AI FREQ Frequenin homolog Hs (Drosophila) down AK LRRN3 Hs.3781 Leucine rich repeat neuronal down BC KIAA0258 Hs KIAA down NM_ IRF2 Hs Interferon regulatory factor down Myeloid differentiation primary AW MYD88 Hs response gene (88) down Ribosomal protein S6 kinase, AF RPS6KA5 Hs kDa, polypeptide down Killer cell immunoglobulin- like receptor, three domains, long NM_ KIR3DL1 Hs cytoplasmic tail, down SLIT and NTRK- like family, N30155 SLITRK2 Hs member down AI BACH1 BTB and CNC homology 1, basic leucine zipper transcription Hs factor down NM_ PLSCR1 Hs Phospholipid scramblase down NM_ MNDA Myeloid cell nuclear Hs differentiation antigen down AV AKAP13 A kinase (PRKA) anchor protein Hs down AL CUL1 Hs Cullin down BF YWHAH Tyrosine 3- monooxygenase/tryptophan 5- monooxygenase activation Hs protein, eta polypeptide down AW ATP13A3 Hs ATPase type 13A down AA ZC3H12A Zinc finger CCCH- type Hs containing 12A down
11 AK DERL1 Der1- like domain family, Hs member down D26121 SF1 Hs Splicing factor down Signal- induced proliferation- AB SIPA1L1 Hs associated 1 like down Hypothetical protein AA LOC Hs LOC down BG IFITM1 Interferon induced transmembrane protein 1 (9- Hs ) down AA KCNJ2 Hs.1547 Potassium inwardly- rectifying channel, subfamily J, member down V- abl Abelson murine leukemia viral oncogene homolog 2 (arg, NM_ ABL2 Hs Abelson- related gene) down Caspase 7, apoptosis- related U37449 CASP7 Hs.9216 cysteine peptidase down BG PCYT1A Hs Phosphate cytidylyltransferase 1, choline, alpha down Chromosome 6 open reading AB C6orf32 Hs frame up BF TCF3 Transcription factor 3 (E2A immunoglobulin enhancer Hs binding factors E12/E47) up U10991 C11orf41 Chromosome 11 open reading Hs frame up BG SH3BP5 SH3- domain binding protein 5 Hs (BTK- associated) up XM_ MLL2 myeloid/lymphoid or mixed- lineage leukemia up AW FDFT1 Farnesyl- diphosphate Hs farnesyltransferase up AJ C17orf85 Chromosome 17 open reading Hs frame up BE SMARCE1 SWI/SNF related, matrix associated, actin dependent regulator of chromatin, Hs subfamily e, member up XM_ ZNF6 zinc finger protein 6 (CMPX1) up BE PTTG1IP Pituitary tumor- transforming 1 Hs interacting protein up AL GLB1 Hs Galactosidase, beta up AI TYMS Hs Thymidylate synthetase up Complement component 5a AL C5AR1 Hs.2161 receptor up Carnitine palmitoyltransferase BC CPT1A Hs A (liver) up
12 BF PRKCSH Hs Protein kinase C substrate 80K- H up Keratin 5 (epidermolysis bullosa simplex, Dowling- Meara/Kobner/Weber- L42611 KRT5 Hs Cockayne types) up Myeloid/lymphoid or mixed- lineage leukemia (trithorax homolog, Drosophila); AA MLLT10 Hs translocated to, up AL COPA Hs Coatomer protein complex, subunit alpha up UDP- N- acetyl- alpha- D- galactosamine:polypeptide N- acetylgalactosaminyltransferase X85019 GALNT2 Hs (GalNAc- T2) up NM_ CYP19A1 Cytochrome P450, family 19, subfamily A, polypeptide up AI ZNF297B Hs Zinc finger protein 297B up AW RIMS1 Regulating synaptic membrane Hs exocytosis up MRE11 meiotic recombination NM_ MRE11A Hs homolog A (S. cerevisiae) up Dexamethasone- induced AW DEXI Hs transcript up BG RAB9 RAB9, member RAS oncogene Hs family up Hepatoma- derived growth factor (high- mobility group BF HDGF Hs protein 1- like) up DNA2 DNA replication helicase BE DNA2L Hs like (yeast) up BE HMGB1 Hs High- mobility group box up D44640 ADD1 Hs Adducin 1 (alpha) up AA AA Hs Transcribed locus up AA ARPC5L Actin related protein 2/3 Hs complex, subunit 5- like up AV RSRC1 Arginine/serine- rich coiled- coil Hs up AA SLC3A1 Solute carrier family 3 (cystine, dibasic and neutral amino acid transporters, activator of cystine, dibasic and neutral amino acid transport), member Hs up NM_ TFAP2C Transcription factor AP- 2 gamma (activating enhancer Hs binding protein 2 gamma) up AA AA Hs Transcribed locus up
13 NM_ DNM1L Hs Dynamin 1- like up BF POLR2A Polymerase (RNA) II (DNA Hs directed) polypeptide A, 220kDa up Serpin peptidase inhibitor, clade F (alpha- 2 antiplasmin, pigment epithelium derived AL SERPINF1 Hs factor), member up Chromosome 14 open reading N48003 C14orf126 Hs frame up NM_ MAGEA9 Hs Melanoma antigen family A, up AW SP4 Hs Sp4 transcription factor up Chromosome 22 open reading AA C22orf23 Hs frame up BE PTTG1IP Pituitary tumor- transforming 1 Hs interacting protein up BF RAGE Hs Renal tumor antigen up Endoplasmic reticulum protein BE ERP29 Hs up AW JOSD3 Hs Josephin domain containing up AA EIF3S9 Eukaryotic translation initiation Hs factor 3, subunit 9 eta, 116kDa up Coagulation factor C homolog, NM_ COCH Hs cochlin (Limulus polyphemus) up Endothelial differentiation- AI EDF1 Hs related factor up Complement component 1, r M14058 C1R Hs subcomponent up Discs, large homolog 3 (neuroendocrine- dlg, AA DLG3 Hs Drosophila) up AW GAPVD1 GTPase activating protein and Hs VPS9 domains up AA SMA3 SMA up AW CST7 Hs Cystatin F (leukocystatin) up AA BPTF Bromodomain PHD finger Hs transcription factor up AK SUDS3 Suppressor of defective silencing 3 homolog (S. Hs cerevisiae) up N64757 TIGD3 Tigger transposable element Hs derived up Polymerase (RNA) I polypeptide BC POLR1B Hs B, 128kDa up AI ULK4 Hs Unc- 51- like kinase 4 (C. elegans) up Ornithine aminotransferase AL OAT Hs (gyrate atrophy) up
14 BE CHCHD7 Coiled- coil- helix- coiled- coil- Hs helix domain containing up AI LTA4H Hs Leukotriene A4 hydrolase up BC TARS Hs Threonyl- trna synthetase up NM_ GLUD1 Hs Glutamate dehydrogenase up XM_ CAPN5 calpain up Complement component 1, s NM_ C1S Hs subcomponent up NM_ KRT2 Hs.707 Keratin 2 (epidermal ichthyosis bullosa of Siemens) up BCL2- like 13 (apoptosis AI BCL2L13 Hs facilitator) up BE H2AFY Hs H2A histone family, member Y up Leucine- rich PPR- motif AI LRPPRC Hs containing up NM_ KAL1 Hs Kallmann syndrome 1 sequence up AF HSD17B12 Hydroxysteroid (17- beta) Hs dehydrogenase up AU GAL3ST4 GALACTOSE- 3- O- SULFOTRANSFERASE up AI SLC26A8 Solute carrier family 26, Hs member up NM_ PAH Hs Phenylalanine hydroxylase up Discs, large homolog 7 NM_ DLG7 Hs (Drosophila) up AL CBX1 Hs Chromobox homolog 1 (HP1 beta homolog Drosophila ) up AI METAP2 Hs Methionyl aminopeptidase up V- fos FBJ murine osteosarcoma AV FOS Hs viral oncogene homolog up BG GANAB Hs Glucosidase, alpha; neutral AB up Mitogen- activated protein BC MAPK3 Hs.861 kinase up AJ MMP25 Hs Matrix metallopeptidase up NM_ SUV39H1 Suppressor of variegation 3-9 Hs homolog 1 (Drosophila) up Nucleolar complex associated 3 NM_ NOC3L Hs homolog (S. cerevisiae) up AW MAP3K9 Hs Mitogen- activated protein kinase kinase kinase up AW C9orf140 Hs Chromosome 9 open reading frame up AF FLJ43806 Hs Hypothetical protein FLJ up Nucleosome assembly protein 1- BG NAP1L4 Hs like up XM_ LOC similar to Selenide,water dikinase 1 (Selenophosphate up NM_ MCC Mutated in colorectal cancers up
15 AB AOF2 Amine oxidase (flavin Hs containing) domain up L25275 SULT1A3 Sulfotransferase family, cytosolic, 1A, phenol- preferring, Hs member up AW MTFR1 Mitochondrial fission regulator Hs up Zinc finger and BTB domain AA ZBTB4 Hs containing up MutS homolog 2, colon cancer, AW MSH2 Hs nonpolyposis type 1 (E. coli) up AA DCN Hs Decorin up Myeloid/lymphoid or mixed- lineage leukemia (trithorax homolog, Drosophila); AA MLLT1 Hs translocated to, up AW KIAA0913 Hs KIAA up NM_ GTF3C1 Hs General transcription factor IIIC, polypeptide 1, alpha 220kDa up XM_ UNG2 uracil- DNA glycosylase up Mannosidase, alpha, class 1B, AL MAN1B1 Hs member up AL PTPRU PROTEIN TYROSINE PHOSPHATASE, RECEPTOR TYPE, U up AA HMGCR 3- hydroxy- 3- methylglutaryl- Hs Coenzyme A reductase up AA LILRB3 Leukocyte immunoglobulin- like receptor, subfamily B (with TM Hs and ITIM domains), member up AA BLMH Hs Bleomycin hydrolase up Nuclear factor (erythroid- NM_ NFE2 Hs derived 2), 45kDa up BE CENTG3 Hs Centaurin, gamma up BG VDAC2 Voltage- dependent anion Hs channel up BF TMPO Thymopoietin up AI AI Hs Transcribed locus up AI TNFRSF12A Tumor necrosis factor receptor Hs superfamily, member 12A up NM_ EPHB3 Hs.2913 EPH receptor B up BC GATA3 Hs GATA binding protein up AK HDAC7A Hs Histone deacetylase 7A up BG MCFD2 Multiple coagulation factor Hs deficiency up AA EIF3S8 Eukaryotic translation initiation Hs factor 3, subunit 8, 110kDa up
16 BE GBAS Glioblastoma amplified Hs sequence up AV ATP5C1 ATP synthase, H+ transporting, mitochondrial F1 complex, Hs gamma polypeptide up BF MGP Hs Matrix Gla protein up Aldo- keto reductase family 1, BF AKR1B1 Hs member B1 (aldose reductase) up NM_ THBS2 Hs Thrombospondin up NM_ ICA1 Hs Islet cell autoantigen 1, 69kDa up XM_ XPNPEP2 X- prolyl aminopeptidase (aminopeptidase P) 2, up AI MGC3207 Hs Hypothetical protein MGC up M23234 ABCB4 ATP- binding cassette, sub- Hs family B (MDR/TAP), member up AI FGB Hs Fibrinogen beta chain up Malate dehydrogenase 1, NAD BF MDH1 Hs (soluble) up AL RCN2 Hs Reticulocalbin 2, EF- hand calcium binding domain up AL SCARB1 Hs Scavenger receptor class B, member up BE PRKDC Hs Protein kinase, DNA- activated, catalytic polypeptide up Poly (ADP- ribose) polymerase BE PARP1 Hs family, member up AI LOXL1 Hs Lysyl oxidase- like up AL DKK1 Hs Dickkopf homolog 1 (Xenopus laevis) up AW KIAA1804 Hs Mixed lineage kinase up AV UCRC Ubiquinol- cytochrome c Hs reductase complex (7.2 kd) up AL CTGF Hs Connective tissue growth factor up Protein phosphatase BE PPME1 Hs methylesterase up AI XYLB Hs Xylulokinase homolog (H. influenzae) up Component of oligomeric golgi AF COG7 Hs complex up AV YY1 Hs YY1 transcription factor up NM_ PLOD2 Procollagen- lysine, 2- Hs oxoglutarate 5- dioxygenase up BE RNF157 Hs Ring finger protein up ATP synthase, H+ transporting, mitochondrial F0 complex, BE ATP5G1 Hs subunit C1 (subunit 9) up Chromosome 1 open reading AW C1orf187 Hs frame up
17 AU XYLB Hs Xylulokinase homolog (H. influenzae) up XM_ COX15 COX15 (yeast) homolog, cytochrome c oxidase assembly up NM_ RUFY3 Hs.7972 RUN and FYVE domain containing up BG STRAP Hs Serine/threonine kinase receptor associated protein up BE ARFGEF2 Hs ADP- ribosylation factor guanine nucleotide- exchange factor 2 (brefeldin A- inhibited) up Peroxidasin homolog AF PXDN Hs (Drosophila) up XM_ ORM1 orosomucoid up BG MT1A Hs Metallothionein 1A (functional) up AL BIRC5 Baculoviral IAP repeat- Hs containing 5 (survivin) up Adhesion molecule with Ig- like AA AMIGO1 Hs domain up BE RRM1 Hs Ribonucleotide reductase M1 polypeptide up U2 small nuclear RNA auxiliary AW U2AF1 Hs factor up BE DHCR7 Hs dehydrocholesterol reductase up AI AKR7A2 Aldo- keto reductase family 7, member A2 (aflatoxin aldehyde Hs reductase) up AL KDELR1 KDEL (Lys- Asp- Glu- Leu) endoplasmic reticulum protein Hs retention receptor up AA FARP1 FERM, RhoGEF (ARHGEF) and pleckstrin domain protein 1 Hs (chondrocyte- derived) up BE UQCRH Ubiquinol- cytochrome c Hs reductase hinge protein up Milk fat globule- EGF factor 8 AI MFGE8 Hs.3745 protein up AL DPAGT1 Hs Dolichyl- phosphate (UDP- N- acetylglucosamine) N- acetylglucosaminephosphotrans ferase 1 (GlcNAc- 1- P transferase) up NM_ SERPINA5 Hs Serpin peptidase inhibitor, clade A (alpha- 1 antiproteinase, antitrypsin), member up
18 BE DHCR dehydrocholesterol Hs reductase up Epidermal growth factor NM_ EPS8 Hs receptor pathway substrate up NM_ FBN1 Hs Fibrillin up AI LZTS2 Leucine zipper, putative tumor Hs suppressor up N56946 BBS2 Hs Bardet- Biedl syndrome up Aldolase C, fructose- T03444 ALDOC Hs bisphosphate up M95585 HLF Hs Hepatic leukemia factor up 2,4- dienoyl CoA reductase 2, AV DECR2 Hs.9235 peroxisomal up BG HMGB2 Hs High- mobility group box up NM_ LGMN Hs Legumain up NM_ MAGEA1 Hs Melanoma antigen family A, 1 (directs expression of antigen MZ2- E) up BE KPNA2 Hs Karyopherin alpha 2 (RAG cohort 1, importin alpha 1) up AL GGH Hs Gamma- glutamyl hydrolase (conjugase, folylpolygammaglutamyl hydrolase) up AU LUM Hs Lumican up AW COL1A1 Hs Collagen, type I, alpha up BE TK1 Hs Thymidine kinase 1, soluble up AL ID1 INHIBITOR OF DNA BINDING 1, DOMINANT NEGATIVE HELIX- LOOP- HELIX PROTEIN up AU LAMB1 Hs Laminin, beta up AI PRAME Hs Preferentially expressed antigen in melanoma up AL OPRS1 Hs Opioid receptor, sigma up XM_ MMP2 matrix metalloproteinase 2 (gelatinase A, 72kD up NM_ DHRS2 Hs Dehydrogenase/reductase (SDR family) member up NM_ AURKA Hs Aurora kinase A up BG IGJ Immunoglobulin J polypeptide, linker protein for immunoglobulin alpha and mu Hs polypeptides up AL ITGB7 Hs Integrin, beta up XM_ NID1 nidogen1 (enactin) (NID) up Isoleucine- trna synthetase 2, AU IARS2 Hs mitochondrial up AL COL6A1 Hs Collagen, type VI, alpha up
19 NM_ PSMB10 Hs Proteasome (prosome, macropain) subunit, beta type, up XM_ NCKAP1 NCK- associated protein up AW LOC91316 Hs Similar to bk246h3.1 (immunoglobulin lambda- like polypeptide 1, pre- B- cell specific) up Insulin- like growth factor 2 AW IGF2 Hs (somatomedin A) up NM_ DSP Hs Desmoplakin up BF MAGEA6 Hs Melanoma antigen family A, up AA KRT7 Hs Keratin up Secreted frizzled- related NM_ SFRP1 Hs protein up XM_ HBG2 hemoglobin, gamma G up AU A2M Hs Alpha- 2- macroglobulin up BG HP Hs Haptoglobin up AL IGFBP1 Insulin- like growth factor Hs binding protein up NM_ CENPF synonyms: CENF, PRO1779; centromere protein F (400kD); centromere protein F (350/400kD, mitosin); CENP- F kinetochore protein; AH antigen; cell- cycle- dependent 350K nuclear protein; Homo sapiens centromere protein F, 350/400ka (mitosin) (CENPF), mrna up BG KRT18 Hs Keratin up NM_ BAMBI Hs BMP and activin membrane- bound inhibitor homolog (Xenopus laevis) up AI PAICS Phosphoribosylaminoimidazole carboxylase, phosphoribosylaminoimidazole Hs succinocarboxamide synthetase up AV CSNK1G1 Hs Casein kinase 1, gamma up BE PODXL Hs Podocalyxin- like up AL ALB Hs Albumin up AA AFP Hs Alpha- fetoprotein up Non- metastatic cells 1, protein BF NME1 Hs (NM23A) expressed in up AW MDK Hs Midkine (neurite growth- promoting factor 2) up AW MLXIP Hs MLX interacting protein up BF SILV Hs Silver homolog (mouse) up
20 D89729 XPO1 Hs Exportin 1 (CRM1 homolog, yeast) up N72014 PLA2G12A Hs Phospholipase A2, group XIIA up M62403 IGFBP4 Insulin- like growth factor Hs binding protein up X56160 TNC Hs Tenascin C (hexabrachion) up BG SHC1 SHC (Src homology 2 domain containing) transforming Hs protein up NM_ GAGE2 Hs G antigen up
21 )
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
Gene Symbol Assay ID Gene Name. ACTB l Hs _m1 actin, beta. AKT1 b Hs _m1 v-akt murine thymoma viral oncogene homolog 1
 Electronic Supplementary Material (ESI) for Metallomics. This journal is The Royal Society of Chemistry 2015 Gene Symbol Assay ID Gene Name ACTB l Hs99999903_m1 actin, beta AKT1 b Hs00178289_m1 v-akt murine
Electronic Supplementary Material (ESI) for Metallomics. This journal is The Royal Society of Chemistry 2015 Gene Symbol Assay ID Gene Name ACTB l Hs99999903_m1 actin, beta AKT1 b Hs00178289_m1 v-akt murine
Table S1. Gene expression levels from Affymetrix microarray data
 Table S1. Gene expression levels from Affymetrix microarray data Gene UniGene GeneBank hesc hesc-mec FH-Ep-P3 FH-Ep-PP OCT4 Hs.249184 NM_002701 10.8 6.8 5.4 5.5 NANOG Hs.661360 NM_024865 9.0 5.8 5.2 5.4
Table S1. Gene expression levels from Affymetrix microarray data Gene UniGene GeneBank hesc hesc-mec FH-Ep-P3 FH-Ep-PP OCT4 Hs.249184 NM_002701 10.8 6.8 5.4 5.5 NANOG Hs.661360 NM_024865 9.0 5.8 5.2 5.4
Supplemental Table 1. Significant genes and their values necessary for computing multi-factorial L value in the prediction model.
 Supplemental Table 1. Significant genes and their values necessary for computing multi-factorial L value in the prediction model. UG cluster Symbol Description t-value Midpoint p-value Unique id Hs.36566
Supplemental Table 1. Significant genes and their values necessary for computing multi-factorial L value in the prediction model. UG cluster Symbol Description t-value Midpoint p-value Unique id Hs.36566
Impacto de la Hiperestimulación Ovárica Controlada sobre el la Expresión Génica del Endometrio durante la Ventana de Implantación
 Impacto de la Hiperestimulación Ovárica Controlada sobre el la Expresión Génica del Endometrio durante la Ventana de Implantación J.A. Horcajadas, A. Riesewijk, A. Cervero, A. Pellicer, S. Mosselman and
Impacto de la Hiperestimulación Ovárica Controlada sobre el la Expresión Génica del Endometrio durante la Ventana de Implantación J.A. Horcajadas, A. Riesewijk, A. Cervero, A. Pellicer, S. Mosselman and
Secuenciación*Genómica*y*Medicina*Personalizada*del*Cáncer
 Secuenciación*Genómica*y*Medicina*Personalizada*del*Cáncer Ricardo Armisén Y. Centro de Investigación y Tratamiento del Cáncer U-CANCER: Red de Medicina Traslacional en Cáncer Programa de Fisiopatología!Instituto
Secuenciación*Genómica*y*Medicina*Personalizada*del*Cáncer Ricardo Armisén Y. Centro de Investigación y Tratamiento del Cáncer U-CANCER: Red de Medicina Traslacional en Cáncer Programa de Fisiopatología!Instituto
similar to ADAM DEC1 precursor (A disintegrin and metalloproteinase domain-like protein decysin 1) (ADAM-like LOC ADAMDEC
 Table 2. Top list of the genes (n = 100) displaying the highest differential expression in the AI vs. SCNT profiles in bovine endometrial caruncles and intercaruncular areas Gene accession n Unigene ID
Table 2. Top list of the genes (n = 100) displaying the highest differential expression in the AI vs. SCNT profiles in bovine endometrial caruncles and intercaruncular areas Gene accession n Unigene ID
Mejora en la Composición de los Alimentos. Guillermo Reglero Director IMDEA Alimentación
 Mejora en la Composición de los Alimentos Guillermo Reglero Director IMDEA Alimentación 1 2 3 Enfermos hospitalizados por ECV 4 Alimentación para la salud 5 6 7 8 13 de marzo de 2014 9 10 11 12 13 8,00%
Mejora en la Composición de los Alimentos Guillermo Reglero Director IMDEA Alimentación 1 2 3 Enfermos hospitalizados por ECV 4 Alimentación para la salud 5 6 7 8 13 de marzo de 2014 9 10 11 12 13 8,00%
Síntesis de ácidos grasos
 Síntesis de ácidos grasos Las vías de síntesis y degradación son distintas Es similar a lo que ocurre entre glicolisis y gluneogénesis, síntesis y degradación de glicógeno Existen 4 diferencias esenciales
Síntesis de ácidos grasos Las vías de síntesis y degradación son distintas Es similar a lo que ocurre entre glicolisis y gluneogénesis, síntesis y degradación de glicógeno Existen 4 diferencias esenciales
Zinc en metaloproteinas y enzimas - 2
 Zinc en metaloproteinas y enzimas - 2 » Se les denomina dedos porque contienen dominios estructurales capaces de anclarse en los surcos de la doble hélice del ADN como si fueran dedos.» La mayoría de estas
Zinc en metaloproteinas y enzimas - 2 » Se les denomina dedos porque contienen dominios estructurales capaces de anclarse en los surcos de la doble hélice del ADN como si fueran dedos.» La mayoría de estas
Cortex-specific genes ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs AA Unknown sc_id
![Cortex-specific genes ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs AA Unknown sc_id Cortex-specific genes ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs AA Unknown sc_id](/thumbs/85/92751791.jpg) Cortex-specific genes 184016 ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs.291676 AA767401 Unknown sc_id6474 187003 KIAA0007 KIAA0007 protein Hs.90315 AA251235 Similar to KIAA0007
Cortex-specific genes 184016 ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs.291676 AA767401 Unknown sc_id6474 187003 KIAA0007 KIAA0007 protein Hs.90315 AA251235 Similar to KIAA0007
Biología General: Macromoléculas biológicas. Prof. Bernal Gerardo Garro Mora
 Biología General: Macromoléculas biológicas Prof. Bernal Gerardo Garro Mora Objetivos: Al finalizar la clase el estudiante será capaz de: Definir a las moléculas orgánicas y los principales grupos funcionales
Biología General: Macromoléculas biológicas Prof. Bernal Gerardo Garro Mora Objetivos: Al finalizar la clase el estudiante será capaz de: Definir a las moléculas orgánicas y los principales grupos funcionales
ESCUELA INTERNACIONAL DE IDIOMAS Avenida Pedro de Heredia, Calle 49a #31-45, barrio el Libano 6600671
 Página: Pág: 1 HORARIOS DE CLASES IDIOMAS Jornada: M Sem:01 Curso:01 A.1.1 AA A.1.1 AA A.1.1 AA 11:00AM-12:00PM VIONIS VIONIS Jornada: M Sem:01 Curso:02 A.1.1 AB A.1.1 AB A.1.1 AB VIONIS VIONIS Jornada:
Página: Pág: 1 HORARIOS DE CLASES IDIOMAS Jornada: M Sem:01 Curso:01 A.1.1 AA A.1.1 AA A.1.1 AA 11:00AM-12:00PM VIONIS VIONIS Jornada: M Sem:01 Curso:02 A.1.1 AB A.1.1 AB A.1.1 AB VIONIS VIONIS Jornada:
As was previously described, gene expression profiles of the 70 CCRCCs (performed on cdna
 RSNA, 2015 10.1148/radiol.2015150800 Appendix E1 Gene Expression Profiling Training Set As was previously described, gene expression profiles of the 70 CCRCCs (performed on cdna microarrays containing
RSNA, 2015 10.1148/radiol.2015150800 Appendix E1 Gene Expression Profiling Training Set As was previously described, gene expression profiles of the 70 CCRCCs (performed on cdna microarrays containing
ESTUDIO DE GMI. ESTUDIO PROTEÓMICO COMPARATIVO ENTRE LOS IMPLANTES DENTALES DE GMI Y DE STRAUMANN
 ESTUDIO DE GMI. ESTUDIO PROTEÓMICO COMPARATIVO ENTRE LOS IMPLANTES DENTALES DE GMI Y DE STRAUMANN 2 Hipótesis. Estudio proteómico de la primera capa de proteínas depositada Después de la implantación:
ESTUDIO DE GMI. ESTUDIO PROTEÓMICO COMPARATIVO ENTRE LOS IMPLANTES DENTALES DE GMI Y DE STRAUMANN 2 Hipótesis. Estudio proteómico de la primera capa de proteínas depositada Después de la implantación:
DEPARTAMENTO DE BIOLOGÍA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA
 DEPARTAMENTO DE BIOLOGÍA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA DINÁMICA DE LA ACTINA Y TRÁFICO DE MEMBRANAS ASOCIADO AL COMPLEJO DE GOLGI: PAPEL REGULADOR DE RHOA, RAC1 Y CDC42 Tesis presentada
DEPARTAMENTO DE BIOLOGÍA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA DINÁMICA DE LA ACTINA Y TRÁFICO DE MEMBRANAS ASOCIADO AL COMPLEJO DE GOLGI: PAPEL REGULADOR DE RHOA, RAC1 Y CDC42 Tesis presentada
Supplemental Data. Hu et al. (2012). Plant Cell /tpc
 1 ATGGCGCGCCGCGCCGCTTCCCGCGCTGTTGGCGCCCTTCGCTCGGACGGCTCGATCCAA M A R R A A S R A V G A L R S D G S I Q 61 GGGCGAGGAGGCCGCGCGGGGGGCAGTGGCGCCGAGGACGCACGCCACGTGTTCGACGAA G R G G R A G G S G A E D A R H V
1 ATGGCGCGCCGCGCCGCTTCCCGCGCTGTTGGCGCCCTTCGCTCGGACGGCTCGATCCAA M A R R A A S R A V G A L R S D G S I Q 61 GGGCGAGGAGGCCGCGCGGGGGGCAGTGGCGCCGAGGACGCACGCCACGTGTTCGACGAA G R G G R A G G S G A E D A R H V
y su relación con cáncer
 Cir Cir 2012;80:389-398. y su relación con cáncer Alejandra Berenice Ochoa-Hernández,* Clara Ibet Juárez-Vázquez,** Mónica Alejandra Rosales-Reynoso,** Patricio Barros-Núñez* Resumen los procesos de regulación,
Cir Cir 2012;80:389-398. y su relación con cáncer Alejandra Berenice Ochoa-Hernández,* Clara Ibet Juárez-Vázquez,** Mónica Alejandra Rosales-Reynoso,** Patricio Barros-Núñez* Resumen los procesos de regulación,
USO DE NUEVAS HERRAMIENTAS DE SECUENCIACIÓN ( RNA-SEQ ) APLICADAS EN SALMÓN DEL ATLÁNTICO
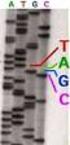 USO DE NUEVAS HERRAMIENTAS DE SECUENCIACIÓN ( RNA-SEQ ) APLICADAS EN SALMÓN DEL ATLÁNTICO Ingeniero en Acuicultura Mg. Sc. Dra. Natalia Lam P. Licenciado en Biología Mg. Sc. Dr. Cristian Araneda T. Licenciada
USO DE NUEVAS HERRAMIENTAS DE SECUENCIACIÓN ( RNA-SEQ ) APLICADAS EN SALMÓN DEL ATLÁNTICO Ingeniero en Acuicultura Mg. Sc. Dra. Natalia Lam P. Licenciado en Biología Mg. Sc. Dr. Cristian Araneda T. Licenciada
Dr. Norberto W. Zwirner
 Dr. Norberto W. Zwirner nzwirner@hospitaldeclinicas.uba.ar Respuesta inmune innata vs. Respuesta inmune adaptativa Propiedad Inmunidad Innata Inmunidad Adaptativa Receptores Codificados en el genoma Reordenamiento
Dr. Norberto W. Zwirner nzwirner@hospitaldeclinicas.uba.ar Respuesta inmune innata vs. Respuesta inmune adaptativa Propiedad Inmunidad Innata Inmunidad Adaptativa Receptores Codificados en el genoma Reordenamiento
Las Citocinas (II) PATRON DE TIPO TH1
 Las Citocinas (II) Dr. Juan Rodríguez-Tafur D. Profesor Asociado de Farmacología e Inmunología Laboratorio de Farmacología de la Respuesta Inmune Coordinador del Comité de Residentado Médico de Inmunología
Las Citocinas (II) Dr. Juan Rodríguez-Tafur D. Profesor Asociado de Farmacología e Inmunología Laboratorio de Farmacología de la Respuesta Inmune Coordinador del Comité de Residentado Médico de Inmunología
Tema 6 Expresión y Regulación de Genes Cap. 10, pág
 Tema 6 Expresión y Regulación de Genes Cap. 10, pág. 255-317 Qué es un gen? Qué es un gen? DNA transcripción mrna no se traducen trna rrna snrna traducción remoción de intrones en eucariotas Proteína construcción
Tema 6 Expresión y Regulación de Genes Cap. 10, pág. 255-317 Qué es un gen? Qué es un gen? DNA transcripción mrna no se traducen trna rrna snrna traducción remoción de intrones en eucariotas Proteína construcción
 Zoonosis parasitaria del hombre, mamíferos y aves producida por un protozoo coccidio el Toxoplasma gondii. Parásito intracelular obligado. En el hombre esta infección habitualmente es asintomática, las
Zoonosis parasitaria del hombre, mamíferos y aves producida por un protozoo coccidio el Toxoplasma gondii. Parásito intracelular obligado. En el hombre esta infección habitualmente es asintomática, las
ÁNGEL MIGUEL GARCÍA LORA
 Ficha de investigador ÁNGEL MIGUEL GARCÍA LORA Grupo de Investigación: INMUNOLOGIA. HOSPITAL UNIVERSITARIO VIRGEN DE LAS NIEVES (Cod.: CTS143) Departamento: Sistema Sanitario Público de Andalucía. Hospital
Ficha de investigador ÁNGEL MIGUEL GARCÍA LORA Grupo de Investigación: INMUNOLOGIA. HOSPITAL UNIVERSITARIO VIRGEN DE LAS NIEVES (Cod.: CTS143) Departamento: Sistema Sanitario Público de Andalucía. Hospital
Comunicaciòn Celular. Copyright 2005 Pearson Education, Inc. publishing as Benjamin Cummings
 Comunicaciòn Celular La comunicaciòn intercelular es esencial para los organismos multicelulares Existen algunos mecanismos de regulaciòn celular universales Las señales extracelulares son convertidas
Comunicaciòn Celular La comunicaciòn intercelular es esencial para los organismos multicelulares Existen algunos mecanismos de regulaciòn celular universales Las señales extracelulares son convertidas
Supplementary Table 1: List of the 316 genes regulated during hyperglycemic euinsulinemic clamp in skeletal muscle.
 Supplementary Table 1: List of the 316 genes regulated during hyperglycemic euinsulinemic clamp in skeletal muscle. UGCluster Name Symbol Fold Change Cytoband Response to stress Hs.517581 Heme oxygenase
Supplementary Table 1: List of the 316 genes regulated during hyperglycemic euinsulinemic clamp in skeletal muscle. UGCluster Name Symbol Fold Change Cytoband Response to stress Hs.517581 Heme oxygenase
Las citoquinas son proteínas secretadas por las células de la inmunidad innata y adaptativa que median muchas funciones de estas células.
 CITOQUINAS Las citoquinas son proteínas secretadas por las células de la inmunidad innata y adaptativa que median muchas funciones de estas células. Propiedades generales La secreción de citoquinas es
CITOQUINAS Las citoquinas son proteínas secretadas por las células de la inmunidad innata y adaptativa que median muchas funciones de estas células. Propiedades generales La secreción de citoquinas es
Células de la piel. Capa córnea Capa granulosa. Estrato basal. Células del sistema inmunitario. Células de Langerhans. Queratinocito.
 Células de la piel Capa córnea Capa granulosa Células de Langerhans Queratinocito Estrato basal Melanocito Fibroblasto Células del sistema inmunitario 1 Células de la piel Células del sistema inmunitario
Células de la piel Capa córnea Capa granulosa Células de Langerhans Queratinocito Estrato basal Melanocito Fibroblasto Células del sistema inmunitario 1 Células de la piel Células del sistema inmunitario
P é ptidos Gonadales. Regulación Intraovárica. Dr. Alfredo Celis L.
 P é ptidos Gonadales Regulación Intraovárica Dr. Alfredo Celis L. Foliculogenesis Folículo es unidad funcional Foliculos : Primordiales (90%) Reserva folicular Folículo primario Folículo preantral Folículo
P é ptidos Gonadales Regulación Intraovárica Dr. Alfredo Celis L. Foliculogenesis Folículo es unidad funcional Foliculos : Primordiales (90%) Reserva folicular Folículo primario Folículo preantral Folículo
Enzyme 2. D Reaction 1. Reaction 3 Starting. Reaction 2. Product molecule
 Figure 8-01 LE 8-UN141 Enzyme 1 Enzyme 2 Enzyme 3 A B C D Reaction 1 Reaction 2 Reaction 3 Starting Product molecule LE 8-2 On the platform, the diver has more potential energy. Diving converts potential
Figure 8-01 LE 8-UN141 Enzyme 1 Enzyme 2 Enzyme 3 A B C D Reaction 1 Reaction 2 Reaction 3 Starting Product molecule LE 8-2 On the platform, the diver has more potential energy. Diving converts potential
FACTORS AFFECTING GROWTH IN CHILDREN WITH CHRONIC KIDNEY DISEASE. THE GH-IGF1 Axis.
 FACTORS AFFECTING GROWTH IN CHILDREN WITH CHRONIC KIDNEY DISEASE. THE GH-IGF1 Axis. Francisco Cano MD Titular Professor Division of Pediatric Nephrology Luis Calvo Mackenna Children s Hospital Department
FACTORS AFFECTING GROWTH IN CHILDREN WITH CHRONIC KIDNEY DISEASE. THE GH-IGF1 Axis. Francisco Cano MD Titular Professor Division of Pediatric Nephrology Luis Calvo Mackenna Children s Hospital Department
DEL GEN A LA PROTEINA
 DEL GEN A LA PROTEINA Parte I: Transcripción Capítulo 12 Expresión de Genes fenotipo Nivel molecular afecta la estructura y función de las células lo que determina que caracteres son expresados (fenotipo)
DEL GEN A LA PROTEINA Parte I: Transcripción Capítulo 12 Expresión de Genes fenotipo Nivel molecular afecta la estructura y función de las células lo que determina que caracteres son expresados (fenotipo)
Degradación de Proteínas
 Degradación de Proteínas La degradación de las proteínas desempeña importantes funciones, ya sea por la modulación de los niveles intracelulares de proteínas específicas (proteólisis limitada), como por
Degradación de Proteínas La degradación de las proteínas desempeña importantes funciones, ya sea por la modulación de los niveles intracelulares de proteínas específicas (proteólisis limitada), como por
Cáncer. Dr. Monterrubio. Capitulo 14
 Cáncer Capitulo 14 Cáncer 1. Caracterizado por la división celular sin control 2. ~1.5 millón/año de americanos diagnosticados (0.5 millón/año mueren por la enfermedad) 3. 10% de canceres con predisposición
Cáncer Capitulo 14 Cáncer 1. Caracterizado por la división celular sin control 2. ~1.5 millón/año de americanos diagnosticados (0.5 millón/año mueren por la enfermedad) 3. 10% de canceres con predisposición
GENOMAS DEL CLOROPLASTO Y LA MITOCONDRIA. prof. Jorge Hernández T., Ph.D.
 GENOMAS DEL CLOROPLASTO Y LA MITOCONDRIA prof. Jorge Hernández T., Ph.D. La Teoría Endosimbiótica prof. Jorge Hernández T., Ph.D. Evidence for Endosymbiotic Theory Mitochondria have very similar characteristics
GENOMAS DEL CLOROPLASTO Y LA MITOCONDRIA prof. Jorge Hernández T., Ph.D. La Teoría Endosimbiótica prof. Jorge Hernández T., Ph.D. Evidence for Endosymbiotic Theory Mitochondria have very similar characteristics
HISTOSONDA DR. JESÚS ALBA LOSADA
 HISTOSONDA DR. JESÚS ALBA LOSADA HISTOSONDA Sondas de DNA monocatenario para el estudio de la expresión génica en tejidos fijados en formol e incluidos en parafina mediante la hibridación in situ HIBRIDACIÓN
HISTOSONDA DR. JESÚS ALBA LOSADA HISTOSONDA Sondas de DNA monocatenario para el estudio de la expresión génica en tejidos fijados en formol e incluidos en parafina mediante la hibridación in situ HIBRIDACIÓN
Mecanismos de aparición de genes
 Mecanismos de aparición de genes - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera - Duplicación génica - Duplicación cromosómica - Duplicación genómica Introducción
Mecanismos de aparición de genes - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera - Duplicación génica - Duplicación cromosómica - Duplicación genómica Introducción
MINIMAL GENOME En busca del GENOMA MÍNIMO. Dra. Rosario Gil RVGP, octubre 2004
 MINIMAL GENOME En busca del GENOMA MÍNIMO Dra. Rosario Gil RVGP, octubre 2004 El concepto de genoma mínimo Genoma mínimo (Koonin, 2000): grupo más pequeño de genes que debe ser suficiente para mantener
MINIMAL GENOME En busca del GENOMA MÍNIMO Dra. Rosario Gil RVGP, octubre 2004 El concepto de genoma mínimo Genoma mínimo (Koonin, 2000): grupo más pequeño de genes que debe ser suficiente para mantener
Intro Redes Biológicas 1
 Intro Redes Biológicas 1 Cuestión de escala Sistemas biológicos: complejidad en diferentes escalas de organización Ecosistema Comunidad Población Organismo Sistema de órganos Tejido Célula Organelas Moléculas
Intro Redes Biológicas 1 Cuestión de escala Sistemas biológicos: complejidad en diferentes escalas de organización Ecosistema Comunidad Población Organismo Sistema de órganos Tejido Célula Organelas Moléculas
Desarrollo de recursos para estudios genómicos en cítricos: creación de una colección de ESTs y una micromatriz de cdna
 Desarrollo de recursos para estudios genómicos en cítricos: creación de una colección de ETs y una micromatriz de cdna Proyecto de Genómica Funcional de Cítricos Instituto de Agroquímica y Tecnología de
Desarrollo de recursos para estudios genómicos en cítricos: creación de una colección de ETs y una micromatriz de cdna Proyecto de Genómica Funcional de Cítricos Instituto de Agroquímica y Tecnología de
Regulación de la expresión genética
 Regulación de la expresión genética E. coli en presencia de: Glucosa---crecimiento Lactosa---crecimiento mas lento Glucosa mas Lactosa---solo usa glucosa Depleción de glucosa---cambia a Lactosa Por qué
Regulación de la expresión genética E. coli en presencia de: Glucosa---crecimiento Lactosa---crecimiento mas lento Glucosa mas Lactosa---solo usa glucosa Depleción de glucosa---cambia a Lactosa Por qué
SISTEMA MAYOR DE HISTOCOMPATIBILIDAD II
 Departamento de Especialidades Médicas Facultad de Medicina Universidad de Concepción Cómo Reconocemos lo propio de lo ajeno? SISTEMA MAYOR DE HISTOCOMPATIBILIDAD II Por que rechazamos un órgano no compatible?
Departamento de Especialidades Médicas Facultad de Medicina Universidad de Concepción Cómo Reconocemos lo propio de lo ajeno? SISTEMA MAYOR DE HISTOCOMPATIBILIDAD II Por que rechazamos un órgano no compatible?
Transcripcion: Proceso selectivo Reiterativo Conservador No requiere cebador
 Transcripcion: Proceso selectivo Reiterativo Conservador No requiere cebador Procariotas 1 ARN pol Eucariotas 3 ARN pol separada de la traducción ARN r y ARNt grandes moleculas precursoras ARNm policistronicos
Transcripcion: Proceso selectivo Reiterativo Conservador No requiere cebador Procariotas 1 ARN pol Eucariotas 3 ARN pol separada de la traducción ARN r y ARNt grandes moleculas precursoras ARNm policistronicos
Respuesta inmune adaptativa
 CURSO DE FORMACIÓN CBMSO-UAM 2010 Respuesta inmune adaptativa Balbino Alarcón CBMSO 5 de julio de 2010 La respuesta inmune adaptativa tiene dos características distintivas: -Existencia de un repertorio
CURSO DE FORMACIÓN CBMSO-UAM 2010 Respuesta inmune adaptativa Balbino Alarcón CBMSO 5 de julio de 2010 La respuesta inmune adaptativa tiene dos características distintivas: -Existencia de un repertorio
Acido ribonucleico RNA En RNA U aparea con A
 Metabolismo del RNA Transcripcion, enzimas, localizacion, inhibicion En procariotas, factor sigma En eucariotas, sequencias cis-regulatorios, TATA, inr, enhancer In eucariotas, Pol II, TF, iniciacion,
Metabolismo del RNA Transcripcion, enzimas, localizacion, inhibicion En procariotas, factor sigma En eucariotas, sequencias cis-regulatorios, TATA, inr, enhancer In eucariotas, Pol II, TF, iniciacion,
5. Respuesta inmune celular. Presentación de antígeno. El complejo mayor de histocompatibilidad.
 Bioquímica inmunológica 5. Respuesta inmune celular. Presentación de antígeno. El complejo mayor de histocompatibilidad. Los linfocitos T están implicados en lo que tradicionalmente se conoce como respuesta
Bioquímica inmunológica 5. Respuesta inmune celular. Presentación de antígeno. El complejo mayor de histocompatibilidad. Los linfocitos T están implicados en lo que tradicionalmente se conoce como respuesta
TEÒRICO 4 INMUNOLOGÌA 2010
 TEÒRICO 4 INMUNOLOGÌA 2010 Indique el enunciado incorrecto: a) Los queratinocitos expresan receptores de reconocimiento de patrones (RRP). b) La proteína C reactiva es una proteína de fase aguda y un RRP.
TEÒRICO 4 INMUNOLOGÌA 2010 Indique el enunciado incorrecto: a) Los queratinocitos expresan receptores de reconocimiento de patrones (RRP). b) La proteína C reactiva es una proteína de fase aguda y un RRP.
Respuesta inmune. Bioq. Liliana B. Rateni
 Bioq. Liliana B. Rateni Respuesta inmune Los microorganismos desafían el sistema inmune del individuo El sistema inmune responde de dos formas diferente Innata Adaptativa Péptidos antimicrobianos defensinas
Bioq. Liliana B. Rateni Respuesta inmune Los microorganismos desafían el sistema inmune del individuo El sistema inmune responde de dos formas diferente Innata Adaptativa Péptidos antimicrobianos defensinas
Eje de la Hormona de Crecimiento en Enfermedad Renal Crónica pediátrica. Implicancias terapéuticas
 Eje de la Hormona de Crecimiento en Enfermedad Renal Crónica pediátrica. Implicancias terapéuticas FRANCISCO CANO M.D. UNIDAD DE NEFROLOGÍA HOSPITAL DE NIÑOS LUIS CALVO MACKENNA FACULTAD DE MEDICINA UNIVERSIDAD
Eje de la Hormona de Crecimiento en Enfermedad Renal Crónica pediátrica. Implicancias terapéuticas FRANCISCO CANO M.D. UNIDAD DE NEFROLOGÍA HOSPITAL DE NIÑOS LUIS CALVO MACKENNA FACULTAD DE MEDICINA UNIVERSIDAD
Regulación transcripcional en semillas de cebada cv. Bomi y su mutante alto en lisina Riso-1508
 IX Reunión de Biología Molecular de Plantas Regulación transcripcional en semillas de cebada cv. Bomi y su mutante alto en lisina Riso-58 Centro de Biotecnología y Genómica de Plantas Universidad Politécnica
IX Reunión de Biología Molecular de Plantas Regulación transcripcional en semillas de cebada cv. Bomi y su mutante alto en lisina Riso-58 Centro de Biotecnología y Genómica de Plantas Universidad Politécnica
Regulación de la expresión en Eucariotas
 Regulación de la expresión en Eucariotas Amplificación Génica Proteínas o RNAs que se requieren en abundancia están codificados por genes múltiples Duplicación génica Control genómico La estructura de
Regulación de la expresión en Eucariotas Amplificación Génica Proteínas o RNAs que se requieren en abundancia están codificados por genes múltiples Duplicación génica Control genómico La estructura de
Dr. Mario Galindo Programa de Biología Celular y Molecular ICBM, Facultad de Medicina Universidad de Chile
 DIFERENCIACIÓN CELULAR ASPECTOS GENERALES MECANISMOS CELULARES Y MOLECULARES Dr. Mario Galindo Programa de Biología Celular y Molecular ICBM, Facultad de Medicina Universidad de Chile EL PROCESO DE FORMACION
DIFERENCIACIÓN CELULAR ASPECTOS GENERALES MECANISMOS CELULARES Y MOLECULARES Dr. Mario Galindo Programa de Biología Celular y Molecular ICBM, Facultad de Medicina Universidad de Chile EL PROCESO DE FORMACION
Métodos y técnicas clásicas aplicadas al estudio de la regulación de la expresión de genes
 BIOQUIMICA Biología molecular 2018 Bases Moleculares de la regulación de la Expresión génica en eucariontes Métodos y técnicas clásicas aplicadas al estudio de la regulación de la expresión de genes Bibliografía:
BIOQUIMICA Biología molecular 2018 Bases Moleculares de la regulación de la Expresión génica en eucariontes Métodos y técnicas clásicas aplicadas al estudio de la regulación de la expresión de genes Bibliografía:
Secuenciación Masiva en Cáncer
 Secuenciación Masiva en Cáncer Javier Freire Salinas Biólogo Molecular HUMV Coordinación científica: Dr. Fernando Rivera Hospital Universitario Marqués de Valdecilla, Santander Organizado por: Fundación
Secuenciación Masiva en Cáncer Javier Freire Salinas Biólogo Molecular HUMV Coordinación científica: Dr. Fernando Rivera Hospital Universitario Marqués de Valdecilla, Santander Organizado por: Fundación
Ficha de grupo de investigación
 Ficha de grupo de n EPIGENÉTICA EN CÉLULAS MADRE Y CÁNCER Código: BIO342 Responsable del grupo: DAVID LANDEIRA FRIAS Miembros DAVID LANDEIRA FRIAS Profesor contratado doctor Tiempo completo AURORA SERRANO
Ficha de grupo de n EPIGENÉTICA EN CÉLULAS MADRE Y CÁNCER Código: BIO342 Responsable del grupo: DAVID LANDEIRA FRIAS Miembros DAVID LANDEIRA FRIAS Profesor contratado doctor Tiempo completo AURORA SERRANO
Oncogenes DR. JOSE MORDOH. Maestría Biología Molecular Médica UBA 2016
 Oncogenes DR. JOSE MORDOH Maestría Biología Molecular Médica UBA 2016 What is the molecular basis of cancer? Cancers are formed from repeated rounds of DNA mutation, competition, and natural selection
Oncogenes DR. JOSE MORDOH Maestría Biología Molecular Médica UBA 2016 What is the molecular basis of cancer? Cancers are formed from repeated rounds of DNA mutation, competition, and natural selection
10. TRAFICO INTRACELULAR DE PROTEINAS. Verónica González Núñez Universidad de Salamanca
 10. TRAFICO INTRACELULAR DE PROTEINAS Verónica González Núñez Universidad de Salamanca ESQUEMA. Tráfico intracelular de proteínas 1. Introducción al tráfico intracelular de proteínas 2. Traslocación al
10. TRAFICO INTRACELULAR DE PROTEINAS Verónica González Núñez Universidad de Salamanca ESQUEMA. Tráfico intracelular de proteínas 1. Introducción al tráfico intracelular de proteínas 2. Traslocación al
Transcripción. - Generalidades - DNA RNA. - Mecanismo. - Modelos Protein. - Moléculas. - Métodos de detección de RNA. Chromosome RNA DNA
 TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
Mycobacterias. Dra. en C. Monserrat Alvarez Zavala
 Mycobacterias Dra. en C. Monserrat Alvarez Zavala Infección del Epitelio alveolar por M.tb La mycobacteria puede invadir y replicarse en las células alveolares epiteliales, esta invasión en benéfica para
Mycobacterias Dra. en C. Monserrat Alvarez Zavala Infección del Epitelio alveolar por M.tb La mycobacteria puede invadir y replicarse en las células alveolares epiteliales, esta invasión en benéfica para
Dogma central de la biología molecular
 GENÉTICA MOLECULAR Dogma central de la biología molecular replicación de DNA virus a DNA DNA reparación recombinación transcripción procesamiento de RNA splicing, edición, modificación de nucleótidos y
GENÉTICA MOLECULAR Dogma central de la biología molecular replicación de DNA virus a DNA DNA reparación recombinación transcripción procesamiento de RNA splicing, edición, modificación de nucleótidos y
Moléculas Adhesión. T.M. MsC Juan Luis Castillo N. CAM. Cellular Adhesion Molecules. Glicoproteínas: Dominio citoplasmático (extremo carboxilo)
 Departamento de Especialidades Médicas Facultad de Medicina Universidad de Concepción Moléculas Adhesión T.M. MsC Juan Luis Castillo N. Miércoles 30 de Marzo de 2006 Glicoproteínas: Dominio citoplasmático
Departamento de Especialidades Médicas Facultad de Medicina Universidad de Concepción Moléculas Adhesión T.M. MsC Juan Luis Castillo N. Miércoles 30 de Marzo de 2006 Glicoproteínas: Dominio citoplasmático
regulación de la transcripción Dr. Víctor Romanowski, 2009
 regulación de la transcripción Dr. Víctor Romanowski, 2009 1 regulación de la transcripción en procariotas Promotores Secuencias de DNA que posicionan a la RNA polimerasa (RNAP) en el sitio de iniciación
regulación de la transcripción Dr. Víctor Romanowski, 2009 1 regulación de la transcripción en procariotas Promotores Secuencias de DNA que posicionan a la RNA polimerasa (RNAP) en el sitio de iniciación
Fisiología General. Tema 7. Receptores específicos de patógenos en la R.I.I. Las células de alarma en la Respuesta Inmune
 El fagocito se adhiere al microorganismo para poder fagocitarlo Complemento Inmunoglobulinas Patógenos Durante su evolución, los organismos eucariotas han desarrollado receptores para estructuras moleculares
El fagocito se adhiere al microorganismo para poder fagocitarlo Complemento Inmunoglobulinas Patógenos Durante su evolución, los organismos eucariotas han desarrollado receptores para estructuras moleculares
NOMENCLATURA BASE + AZUCAR + FOSFATO: Base Azúcar Fosfatos Nucleótido Ac. nucleico
 NUCLEOTIDOS Los nucleótidos están formados por una base orgánica unida a una azúcar la que tiene 1 a 3 grupos fosfatos. Las bases pueden ser Púricas: adenina y guanina; Pirimídicas: citocina, uracilo y
NUCLEOTIDOS Los nucleótidos están formados por una base orgánica unida a una azúcar la que tiene 1 a 3 grupos fosfatos. Las bases pueden ser Púricas: adenina y guanina; Pirimídicas: citocina, uracilo y
Specific Detection of Inflammation Markers with Tissue Context Using RNAscope
 Application Review Inflammation Advanced Cell Diagnostics Specific Detection of Inflammation Markers with Tissue Context Using RNAscope Technology Detect your target of interest with ease High Sensitivity
Application Review Inflammation Advanced Cell Diagnostics Specific Detection of Inflammation Markers with Tissue Context Using RNAscope Technology Detect your target of interest with ease High Sensitivity
Unificando las hipótesis de los daños crónicos por Hiperglicemia
 Unificando las hipótesis de los daños crónicos por Hiperglicemia Mecanismos moleculares del daño vascular por diabetes: Incremento de la vía del polioles Acumulación de Productos Finales de la Glicosilación
Unificando las hipótesis de los daños crónicos por Hiperglicemia Mecanismos moleculares del daño vascular por diabetes: Incremento de la vía del polioles Acumulación de Productos Finales de la Glicosilación
Información proporcionada de conformidad con el Convenio sobre el registro de objetos lanzados al espacio ultraterrestre
 Naciones Unidas Secretaría Distr. general 30 de junio de 2004 Español Original: inglés Comisión sobre la Utilización del Espacio Ultraterrestre con Fines Pacíficos Información proporcionada de conformidad
Naciones Unidas Secretaría Distr. general 30 de junio de 2004 Español Original: inglés Comisión sobre la Utilización del Espacio Ultraterrestre con Fines Pacíficos Información proporcionada de conformidad
Bacteriófagos. 0.5 m. Capitulo 5 Microbial Genetics Capitulo 3 y 7 Molecular genetics of Bacteria
 Bacteriófagos 0.5 m Capitulo 5 Microbial Genetics Capitulo 3 y 7 Molecular genetics of Bacteria Bacteriofagos (fagos) Definición Parásito intracelular obligado que se multiplica dentro de una bacteria
Bacteriófagos 0.5 m Capitulo 5 Microbial Genetics Capitulo 3 y 7 Molecular genetics of Bacteria Bacteriofagos (fagos) Definición Parásito intracelular obligado que se multiplica dentro de una bacteria
BIOLOGIA TUMORAL MATERIA ONCOLOGIA MOLECULAR. Dr. José Mordoh. Oncología Molecular, 2013
 BIOLOGIA TUMORAL MATERIA ONCOLOGIA MOLECULAR Dr. José Mordoh. Oncología Molecular, 2013 Orquesta. Bruno Brudovic LOCURA, de Francis Hammond ENSAYO DE ORQUESTA. FEDERICO FELLINI BIOLOGIA DE LA CELULA TUMORAL
BIOLOGIA TUMORAL MATERIA ONCOLOGIA MOLECULAR Dr. José Mordoh. Oncología Molecular, 2013 Orquesta. Bruno Brudovic LOCURA, de Francis Hammond ENSAYO DE ORQUESTA. FEDERICO FELLINI BIOLOGIA DE LA CELULA TUMORAL
Mario Chiong. U Chile. 2007
 Receptores de membrana con actividad enzimática intrínseca Mario Chiong. U Chile. 2007 1 Tipo de receptores tirosina kinasa Factor de crecimiento nervioso (NGF) Factor de crecimiento derivado de plaquetas
Receptores de membrana con actividad enzimática intrínseca Mario Chiong. U Chile. 2007 1 Tipo de receptores tirosina kinasa Factor de crecimiento nervioso (NGF) Factor de crecimiento derivado de plaquetas
MECANISMOS DE SEÑALIZACIÓN INTRACELULAR DE SUPERVIVENCIA Y MUERTE NEURONAL REGULADOS POR FACTORES TRÓFICOS
 DEPARTAMENT DE BIOLOGIA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA UNIVERSITAT DE BARCELONA MECANISMOS DE SEÑALIZACIÓN INTRACELULAR DE SUPERVIVENCIA Y MUERTE NEURONAL REGULADOS POR FACTORES TRÓFICOS
DEPARTAMENT DE BIOLOGIA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA UNIVERSITAT DE BARCELONA MECANISMOS DE SEÑALIZACIÓN INTRACELULAR DE SUPERVIVENCIA Y MUERTE NEURONAL REGULADOS POR FACTORES TRÓFICOS
DIFERENCIACION SUPERVIVENCIA / MUERTE
 CICLO CELULAR EXPRESIÓN de GENES Para dividirse, la célula recibe señales, las transmite y las interpreta como EXPRESIÓN de GENES ESPECÍFICOS para poder pasar por cada una de las fases del CICLO CELULAR
CICLO CELULAR EXPRESIÓN de GENES Para dividirse, la célula recibe señales, las transmite y las interpreta como EXPRESIÓN de GENES ESPECÍFICOS para poder pasar por cada una de las fases del CICLO CELULAR
COMPLEJO PRINCIPAL DE HISTOCOMPATIBILIDAD (MHC)
 DRA. LUISA BARBOZA Abbas, A. Inmunología Celular y Molecular. 6ª ed. 2008. Kuby. Immunology 5ªed 2004 HISTORIA COMPLEJO PRINCIPAL DE HISTOCOMPATIBILIDAD (MHC) Abbas, A. Inmunología Celular y Molecular.
DRA. LUISA BARBOZA Abbas, A. Inmunología Celular y Molecular. 6ª ed. 2008. Kuby. Immunology 5ªed 2004 HISTORIA COMPLEJO PRINCIPAL DE HISTOCOMPATIBILIDAD (MHC) Abbas, A. Inmunología Celular y Molecular.
Regulación de la expresión en Eucariotas
 Regulación de la expresión en Eucariotas Amplificación Génica Proteínas o RNAs que se requieren en abundancia están codificados por genes múltiples Duplicación génica Control genómico La estructura de
Regulación de la expresión en Eucariotas Amplificación Génica Proteínas o RNAs que se requieren en abundancia están codificados por genes múltiples Duplicación génica Control genómico La estructura de
Expresión de la información genética en eucariotas. transcripción
 Expresión de la información genética en eucariotas transcripción regulación de la transcripción ver cap. 15 Alberts: receptores y señales Alberts Molecular Biology Of The Cell. 5th.Ed. pdf Víctor Romanowski,
Expresión de la información genética en eucariotas transcripción regulación de la transcripción ver cap. 15 Alberts: receptores y señales Alberts Molecular Biology Of The Cell. 5th.Ed. pdf Víctor Romanowski,
Bases para el control de la reproducción y conocimiento del sistema de defensas naturales en el lenguado senegalés (Solea senegalensis)
 PLAN NACIONAL PARA EL CULTIVO DEL LENGUADO Bases para el control de la reproducción y conocimiento del sistema de defensas naturales en el lenguado senegalés (Solea senegalensis) 2009-2011 Objetivos Generales
PLAN NACIONAL PARA EL CULTIVO DEL LENGUADO Bases para el control de la reproducción y conocimiento del sistema de defensas naturales en el lenguado senegalés (Solea senegalensis) 2009-2011 Objetivos Generales
Tema V: Modelado computacional de rutas señalizadoras
 Tema V: Modelado computacional de rutas señalizadoras V 1.Una ruta del factor de crecimiento epidérmico El factor de crecimiento epidérmico (EGFR) es quizás el sistema receptor mejor conocido. En ello
Tema V: Modelado computacional de rutas señalizadoras V 1.Una ruta del factor de crecimiento epidérmico El factor de crecimiento epidérmico (EGFR) es quizás el sistema receptor mejor conocido. En ello
Fisiología de la Respuesta Innata
 Jornadas Nacionales Conjuntas de Alergia e Inmunología en Pediatría Buenos Aires, 13 al 15 de mayo de 2010 Mesa Redonda NUEVAS INMUNODEFICIENCIAS CON COMPROMISO DE LA INMUNIDAD INNATA Fisiología de la
Jornadas Nacionales Conjuntas de Alergia e Inmunología en Pediatría Buenos Aires, 13 al 15 de mayo de 2010 Mesa Redonda NUEVAS INMUNODEFICIENCIAS CON COMPROMISO DE LA INMUNIDAD INNATA Fisiología de la
El complejo mayor de histocompatibilidad. Dr. Norberto W. Zwirner
 El complejo mayor de histocompatibilidad Dr. Norberto W. Zwirner Descubrimiento como genes que determinan la histocompatibilidad Moléculas clásicas Restricción (Dogherty & Zinkernagel) Polimorfismo Poligenismo
El complejo mayor de histocompatibilidad Dr. Norberto W. Zwirner Descubrimiento como genes que determinan la histocompatibilidad Moléculas clásicas Restricción (Dogherty & Zinkernagel) Polimorfismo Poligenismo
Regulación de la expresión génica génica en en eucariontes Transcripción en en eucariotes eucariotes
 Regulación de la expresión génica en eucariontes Factores transcripcionales Mario Chiong Transcripción en eucariotes Las 3 RNAs polimerasas (I, II y III) interactúan con sus promotores a través de los
Regulación de la expresión génica en eucariontes Factores transcripcionales Mario Chiong Transcripción en eucariotes Las 3 RNAs polimerasas (I, II y III) interactúan con sus promotores a través de los
ANTIGENOS Y PRESENTACION DE ANTIGENOS
 ANTIGENOS Y PRESENTACION DE ANTIGENOS AGENTE EXTRAÑO RECEPTOR CPA PRESENTACION CELULA EFECTORA http://webdelprofesor.ula.ve/medicina/jacova Dr. José Angel Cova Instituto de Inmunología Clínica Universidad
ANTIGENOS Y PRESENTACION DE ANTIGENOS AGENTE EXTRAÑO RECEPTOR CPA PRESENTACION CELULA EFECTORA http://webdelprofesor.ula.ve/medicina/jacova Dr. José Angel Cova Instituto de Inmunología Clínica Universidad
Sistema HLA Nomenclatura actual Rol biológico Antígenos Clase I y II.
 Sistema HLA Nomenclatura actual Rol biológico Antígenos Clase I y II. BQ. Alejandra Arenas C. Sección Histocompatibilidad Subdepto. Enfermedades no Transmisibles Complejo Mayor de Histocompatibilidad(MHC)
Sistema HLA Nomenclatura actual Rol biológico Antígenos Clase I y II. BQ. Alejandra Arenas C. Sección Histocompatibilidad Subdepto. Enfermedades no Transmisibles Complejo Mayor de Histocompatibilidad(MHC)
Diferentes mecanismos de transporte de proteínas
 Diferentes mecanismos de transporte de proteínas Todos los mecanismos involucran señales de tránsito presentes en las proteínas y receptores sobre la organela en cuestión Existen dos clases de señales
Diferentes mecanismos de transporte de proteínas Todos los mecanismos involucran señales de tránsito presentes en las proteínas y receptores sobre la organela en cuestión Existen dos clases de señales
CURSO MEDICINA GENOMICA. Karol Carrillo Sánchez INMEGEN
 CURSO MEDICINA GENOMICA Karol Carrillo Sánchez INMEGEN kcarrillo@inmegen.gob.mx 27 de enero 2009 ÍNDICE DE TEMAS Estructura Química: DNA, RNA y Proteínas (1a Clase) Organelos Celulares (2da Clase) Ciclo
CURSO MEDICINA GENOMICA Karol Carrillo Sánchez INMEGEN kcarrillo@inmegen.gob.mx 27 de enero 2009 ÍNDICE DE TEMAS Estructura Química: DNA, RNA y Proteínas (1a Clase) Organelos Celulares (2da Clase) Ciclo
Respuesta inmune adaptativa
 CURSO DE FORMACIÓN CBMSO-UAM 2012 Respuesta inmune adaptativa Balbino Alarcón CBMSO 19 de enero de 2012 La respuesta inmune adaptativa tiene dos características distintivas: -Existencia de un repertorio
CURSO DE FORMACIÓN CBMSO-UAM 2012 Respuesta inmune adaptativa Balbino Alarcón CBMSO 19 de enero de 2012 La respuesta inmune adaptativa tiene dos características distintivas: -Existencia de un repertorio
CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO
 Alianza para el Aprendizaje de Ciencias y Matemáticas CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO GUÍA DEL MAESTRO ESTÁNDARES ATENDIDOS: 1. LA NATURALEZA
Alianza para el Aprendizaje de Ciencias y Matemáticas CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO GUÍA DEL MAESTRO ESTÁNDARES ATENDIDOS: 1. LA NATURALEZA
TRANSDUCCIÓN DE SEÑALES: TIROSINA QUINASAS
 TRANSDUCCIÓN DE SEÑALES: TIROSINA QUINASAS Dra Claudia Tomes REACCIÓN de FOSFORILACIÓN CATALIZADA POR PROTEINA QUINASAS SUSTRATO-aa-OH + APPP (ATP) SUSTRATO-aa-OP + APP (ADP) Ser/Tre quinasas fosforilan
TRANSDUCCIÓN DE SEÑALES: TIROSINA QUINASAS Dra Claudia Tomes REACCIÓN de FOSFORILACIÓN CATALIZADA POR PROTEINA QUINASAS SUSTRATO-aa-OH + APPP (ATP) SUSTRATO-aa-OP + APP (ADP) Ser/Tre quinasas fosforilan
EXPRESIÓN de GENES. o TRANSCRIPCIONAL. o TRADUCCIONAL REGULACIÓN. o MODIFICACIÓN DE PROTEÍNAS (Fosforilación)
 EL CICLO CELULAR EUCARIONTE Y SU REGULACIÓN G2 M S INTERFASE G1 EXPRESIÓN de GENES o TRANSCRIPCIONAL REGULACIÓN o TRADUCCIONAL o MODIFICACIÓN DE PROTEÍNAS (Fosforilación) La célula percibe señales para:
EL CICLO CELULAR EUCARIONTE Y SU REGULACIÓN G2 M S INTERFASE G1 EXPRESIÓN de GENES o TRANSCRIPCIONAL REGULACIÓN o TRADUCCIONAL o MODIFICACIÓN DE PROTEÍNAS (Fosforilación) La célula percibe señales para:
Transcripción del DNA y Regulación de la expresión genética
 Universidad de Chile, Facultad de Medicina, ICBM Programa de Biología Celular y Molecular Transcripción del DNA y Regulación de la expresión genética Curso de Biología Molecular y Genómica 2018 Dr. Héctor
Universidad de Chile, Facultad de Medicina, ICBM Programa de Biología Celular y Molecular Transcripción del DNA y Regulación de la expresión genética Curso de Biología Molecular y Genómica 2018 Dr. Héctor
CANCER UNKNOWN PRIMARY
 CANCER UNKNOWN PRIMARY 3-5 % of diagnosed cancers Variability depending of centers 50% adenocarcinomas Immunohistochemistry helps in 30-85% Gene expression assays help in 75-90% Both methods solve > 95%
CANCER UNKNOWN PRIMARY 3-5 % of diagnosed cancers Variability depending of centers 50% adenocarcinomas Immunohistochemistry helps in 30-85% Gene expression assays help in 75-90% Both methods solve > 95%
CÉLULAS NATURAL KILLER (NK)
 CÉLULAS NATURAL KILLER (NK) Linfocitos granulocitos de 8-10 micras que representan 2-18% de los linfocitos totales, no requieren de sensibilidad previa. Eliminan células tumorales e infectadas, liberación
CÉLULAS NATURAL KILLER (NK) Linfocitos granulocitos de 8-10 micras que representan 2-18% de los linfocitos totales, no requieren de sensibilidad previa. Eliminan células tumorales e infectadas, liberación
Mecanismos de evolución de los genomas. - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera
 Mecanismos de evolución de los genomas - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera Evolución del tamaño de los genes Contenido Introducción: mecanismos de amplificación
Mecanismos de evolución de los genomas - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera Evolución del tamaño de los genes Contenido Introducción: mecanismos de amplificación
Clases de GPCRs. C. Tipo metabotrópico glutamato/ feromona. E. Receptores camp (Dictiostelium) -- Subfamilia Frizzled/Smoothened
 Clases de GPCRs A. Tipo rodopsina B. Tipo secretina C. Tipo metabotrópico glutamato/ feromona D. Feromona hongos E. Receptores camp (Dictiostelium) -- Subfamilia Frizzled/Smoothened Cada GPCR tiene requerimientos
Clases de GPCRs A. Tipo rodopsina B. Tipo secretina C. Tipo metabotrópico glutamato/ feromona D. Feromona hongos E. Receptores camp (Dictiostelium) -- Subfamilia Frizzled/Smoothened Cada GPCR tiene requerimientos
Señalización Celular
 Señalización Celular Importancia de la Señalización Celular Metabolismo Movimiento Proliferación Supervivencia Muerte Diferenciación Señalización Celular Principios de Señalización Celular Moléculas de
Señalización Celular Importancia de la Señalización Celular Metabolismo Movimiento Proliferación Supervivencia Muerte Diferenciación Señalización Celular Principios de Señalización Celular Moléculas de
Regulación Mitocondrial y. Micro-Inmunoterapia
 A S O C I A C I Ó N Micro-Inmunoterapia Regulación Mitocondrial y Micro-Inmunoterapia Prevenir el desarrollo o limitar la progresión de enfermedades crónicas, regulando la función mitocondrial a diferentes
A S O C I A C I Ó N Micro-Inmunoterapia Regulación Mitocondrial y Micro-Inmunoterapia Prevenir el desarrollo o limitar la progresión de enfermedades crónicas, regulando la función mitocondrial a diferentes
Fagocitosis FAGOCITOSIS. Marcelo Castillo Navarrete, TM MsC (c) Facultad de Medicina Dpto. Especialidades Médicas Carrera Tecnología Médica
 Fagocitosis Marcelo Castillo Navarrete, TM MsC (c) Facultad de Medicina Dpto. Especialidades Médicas Carrera Tecnología Médica 09 de Marzo de 2006 SISTEMA INMUNE INMUNIDAD INNATA INFLAMACION INMUNIDAD
Fagocitosis Marcelo Castillo Navarrete, TM MsC (c) Facultad de Medicina Dpto. Especialidades Médicas Carrera Tecnología Médica 09 de Marzo de 2006 SISTEMA INMUNE INMUNIDAD INNATA INFLAMACION INMUNIDAD
CURSO TEÓRICO-PRÁCTICO DE MÉTODOS METAGENÓMICOS PARA EL ESTUDIO DE COMUNIDADES MICROBIANAS COMPLEJAS
 CURSO TEÓRICO-PRÁCTICO DE MÉTODOS METAGENÓMICOS PARA EL ESTUDIO DE COMUNIDADES MICROBIANAS COMPLEJAS Universidad Pablo de Olavide, Sevilla 2-6 Febrero 2009 1 Introducción Vivimos en un mundo rodeado de
CURSO TEÓRICO-PRÁCTICO DE MÉTODOS METAGENÓMICOS PARA EL ESTUDIO DE COMUNIDADES MICROBIANAS COMPLEJAS Universidad Pablo de Olavide, Sevilla 2-6 Febrero 2009 1 Introducción Vivimos en un mundo rodeado de
