Supplementary Table 1: List of the 316 genes regulated during hyperglycemic euinsulinemic clamp in skeletal muscle.
|
|
|
- María Elena Valenzuela Maldonado
- hace 5 años
- Vistas:
Transcripción
1 Supplementary Table 1: List of the 316 genes regulated during hyperglycemic euinsulinemic clamp in skeletal muscle. UGCluster Name Symbol Fold Change Cytoband Response to stress Hs Heme oxygenase (decycling) 1 HMOX q12 Hs Metallothionein 1X MT1X q13 Hs Metallothionein 1B (functional) MT1B q13 Hs Oxidation resistance 1 OXR q23 Hs Metallothionein 1F (functional) MT1F q13 Hs Metallothionein 2A MT2A q13 Hs Metallothionein 1H MT1H q13 Hs Glutathione S-transferase pi GSTP q13 Hs Stannin SNN p13 Immune response, cytokines & related Hs TNF (ligand) superfamily, member 10 (TRAIL) TNFSF q26 Hs CD59 antigen p18-20 (protectin) CD p13 Hs Complement component 4B, telomeric C4A p21.3 Hs Immunoglobulin lambda variable 6-57 IGLV q11.2 Hs Calcium modulating ligand CAMLG q23 Hs B-cell CLL/lymphoma 10 BCL p22 Hs.840 Indoleamine-pyrrole 2,3 dioxygenase INDO p12-p11 Hs Mal, T-cell differentiation protein 2 MAL Hs CD99 antigen-like 2 CD99L Xq28 Hs Chemokine (C-C motif) ligand 19 CCL p13 Hs Interferon regulatory factor 2 binding protein 2 IRF2BP q42.3 Hs Contactin 1 CNTN q11-q12 Hs MHC class I mrna fragment p21.3 Hs Carcinoembryonic antigen-related cell adhesion molecule 19 CEACAM q13.31 Hs Selectin E (endothelial adhesion molecule 1) SELE q22-q25 Hs Family with sequence similarity 19 (chemokine (C-C motif)-like), member A5 FAM19A q13.32 Hs Fc fragment of IgG, low affinity IIIb, receptor (CD16b) FCGR3B q23 Hs Junctional adhesion molecule 2 JAM q21.2 Hs S100 calcium binding protein A12 (calgranulin C) S100A q21 Hs Nuclear factor of activated T-cells NFATC q23 Hs Deleted in colorectal carcinoma DCC q21.3 Hs Sema domain, immunoglobulin domain (Ig), transmembrane domain (TM) and short cytoplasmic domain, (semaphorin) 4D SEMA4D q22-q31 Circulating active peptides Hs.2561 Nerve growth factor, beta polypeptide NGFB p13.1 Hs Transforming growth factor, beta 2 TGFB q41 Hs Stanniocalcin 1 STC p21-p11.2 Hs Vascular endothelial growth factor C VEGFC q34.1-q34.3 Hs Coagulation factor II (thrombin) F p11-q12 Receptors Hs Patched homolog (Drosophila) PTCH q22.3 Hs Tumor-associated calcium signal transducer 2 TACSTD p32-p31 Hs Smoothened homolog (Drosophila) SMO q32.3 Hs Low density lipoprotein receptor (familial hypercholesterolemia) LDLR p13.3 Hs Integrin, beta 7 ITGB q13.13 Hs Adrenergic, alpha-1a-, receptor ADRA1A p21-p11.2 Hs Taste receptor, type 2, member 49 TAS2R p13.2 Hs Endothelial differentiation, lysophosphatidic acid G-proteincoupled receptor, 2 EDG q31.3 Hs.1041 Proto-oncogene tyrosine-protein kinase ROS precursor ROS q22 Hs Transmembrane protein 71 TMEM q24.22 Hs Poliovirus receptor-related 3 PVRL q13 Hs Prostaglandin E receptor 3 (subtype EP3) PTGER p31.2 Hs Receptor tyrosine kinase-like orphan receptor 2 ROR q22 Signaling pathways Protein phosphatases Hs Protein phosphatase 1, catalytic subunit, gamma isoform PPP1CC q24.1-q24.2 Hs Protein tyrosine phosphatase-like A domain containing 1 PTPLAD q22.2 Hs Protein tyrosine phosphatase, receptor type, O PTPRO p13.3-p13.2 Hs Protein tyrosine phosphatase, non-receptor type 14 PTPN q32.2 Hs Protein phosphatase 2 (formerly 2A), regulatory subunit B (PR 52), alpha isoform PPP2R2A p21.2 Hs Protein phosphatase 1, regulatory subunit 10 PPP1R p21.3 Protein kinases Hs Mitogen-activated protein kinase 1 MAPK q11.2 Hs Protein kinase D2 PRKD q13.3 Hs Cyclin-dependent kinase-like 3 CDKL q31 Hs TAO kinase 2 TAOK p11.2 Hs Tyrosine-protein kinase HCK DDEF q24.1-q24.2 Hs Cyclin-dependent kinase 6 CDK q21-q22
2 Hs LIM domain kinase 1 LIMK q11.23 Hs AP2 associated kinase 1 AAK p24.3-p14 Others signaling pathways Hs Homer homolog 1 (Drosophila) HOMER q14.2 Hs Calcyphosine CAPS p13.3 Hs FYVE, RhoGEF and PH domain containing 1 (faciogenital dysplasia) FGD Xp11.21 Hs Calneuron 1 CALN q11 Hs GRB2-associated binding protein 2 GAB q14.1 Hs Secreted frizzled-related protein 1 SFRP p12-p11.1 Hs Pleckstrin homology domain containing, family A member 5 PLEKHA p12 Hs SH3 domain and tetratricopeptide repeats 2 SH3TC q32 Hs Ras-like without CAAX 2 RIT q12.3 Hs Centaurin, alpha 1 CENTA p22.3 Hs Arrestin, beta 2 ARRB p13 Hs Insulin receptor substrate 1 IRS q36 Hs Nuclear factor of kappa light polypeptide gene enhancer in B-cells inhibitor, alpha NFKBIA q13 Hs Ectonucleotide pyrophosphatase/phosphodiesterase 2 (autotaxin) ENPP q24.1 Hs SH3-domain GRB2-like (endophilin) interacting protein 1 SGIP p31.3 Hs.2722 Inositol 1,4,5-trisphosphate 3-kinase A ITPKA q14-q21 Hs Rap guanine nucleotide exchange factor (GEF) 2 RAPGEF q32.1 Hs GTP-binding protein PTD004 PTD q31.1 Hs RalA binding protein 1 RALBP p11.3 Hs RAB27A, member RAS oncogene family RAB27A q15-q21.1 Hs Regulator of G-protein signalling 4 RGS q23.3 Hs Phosphodiesterase 6A, cgmp-specific, rod, alpha PDE6A q31.2-q34 Hs Inhibitor of kappa light polypeptide gene enhancer in B-cells, kinase gamma IKBKG Xq28 Hs Phosphoprotein enriched in astrocytes 15 PEA q21.1 Hs Rho GTPase activating protein 29 ARHGAP p22.1 Enzymes Carbohydrate metabolism Hs Aldolase A, fructose-bisphosphate ALDOA q22-q24 Hs Phosphorylase, glycogen; muscle (McArdle syndrome, glycogen storage disease type V) PYGM q12-q13.2 Hs Phosphoglucomutase 3 PGM q14.1-q15 Hs Phosphorylase kinase, alpha 2 (liver) PHKA Xp22.2-p22.1 Hs Fructosamine-3-kinase-related protein FN3KRP q25.3 Hs.8364 Pyruvate dehydrogenase kinase, isoenzyme 4 PDK q21.3-q22.1 Lipid metabolism Hs Acetyl-Coenzyme A carboxylase beta ACACB q24.1 Hs Monoacylglycerol O-acyltransferase 3 MOGAT q22.1 Hs Sterol-C4-methyl oxidase-like SC4MOL q32-q34 Hs Arachidonate 15-lipoxygenase, second type ALOX15B p13.1 Hs Lipase, member H LIPH q27 ATPase & related Hs ATPase, Na+/K+ transporting, beta 1 polypeptide ATP1B q24 Hs ATPase, Class I, type 8B, member 1 ATP8B q21-q22 Others enzymes Hs Creatine kinase, muscle CKM q13.2-q13.3 Hs Alanine-glyoxylate aminotransferase (oxalosis I; hyperoxaluria I; glycolicaciduria; serine-pyruvate aminotransferase) AGXT q36-q37 Hs Guanosine monophosphate reductase GMPR p23 Hs N-acetylgalactosaminidase, alpha- NAGA q13-qter Hs ,10-methylenetetrahydrofolate reductase (NADPH) MTHFR p36.3 Hs Collapsin response mediator protein 1 CRMP p16.1-p15 Hs Dehydrogenase/reductase (SDR family) member 2 DHRS q11.2 Structural proteins & cytoskeleton Hs Actin, gamma 2, smooth muscle, enteric ACTG p13.1 Hs Kinesin 2 KNS q32.3 Hs Nexilin (F actin binding protein) NEXN p31.1 Hs Myosin binding protein C, slow type MYBPC q23.3 Hs Titin TTN q31 Hs EF hand domain family, member D1 EFHD q37.1 Hs Collagen, type IX, alpha 1 COL9A q12-q14 Hs Collagen, type IV, alpha 5 (Alport syndrome) COL4A Xq22 Hs Plakophilin 4 PKP q23-q31 Hs Keratin 5b K5B q13.13 Hs Leucine-rich repeats and calponin homology (CH) domain containing 3 LRCH q29 Hs Microtubule-associated protein, RP/EB family, member 2 MAPRE q12.1 Hs Nidogen 2 (osteonidogen) NID q21-q22 Hs Myosin VA (heavy polypeptide 12, myoxin) MYO5A q21
3 Hs Tubulin tyrosine ligase-like family, member 2 TTLL q27 Hs Spectrin, beta, non-erythrocytic 1 SPTBN p21 Hs Tubulin, alpha 6 TUBA q12-q14 Hs Growth arrest-specific 8 GAS q24.3 Hs Doublecortex; lissencephaly, X-linked (doublecortin) DCX Xq22.3-q23 Hs Claudin 3 CLDN q11.23 Hs Sarcoglycan, delta (35kDa dystrophin-associated glycoprotein) SGCD q33-q34 Hs Neurofibromin 2 (bilateral acoustic neuroma) NF q12.2 Hs Erythrocyte membrane protein band 4.1 EPB p33-p32 Hs Ectodermal-neural cortex (with BTB-like domain) ENC q12-q13.3 Hs Myosin VB MYO5B q21 Hs Laminin, gamma 1 (formerly LAMB2) LAMC q31 Hs Collagen, type VI, alpha 2 COL6A q22.3 Transporters & carriers Hs Fatty acid binding protein 3, muscle and heart (mammary-derived growth inhibitor) FABP p33-p32 Hs Solute carrier family 43, member 2 SLC43A p13.3 Hs Neuroglobin NGB q24 Hs Solute carrier family 2 (facilitated glucose transporter), member 12 SLC2A q23.2 Hs Synaptosomal-associated protein, 25kDa SNAP p12-p11.2 Hs Trans-golgi network protein 2 TGOLN p11.2 Hs Sodium channel, nonvoltage-gated 1, delta SCNN1D p36.3-p36.2 Hs Solute carrier family 2 (facilitated glucose transporter), member 2 SLC2A q26.1-q26.2 Hs Stomatin (EPB72)-like 1 STOML q24-q25 Hs Solute carrier family 19 (thiamine transporter), member 2 SLC19A q23.3 Hs Solute carrier family 1 (neuronal/epithelial high affinity glutamate transporter, system Xag), member 1 SLC1A p24 Hs Syntaxin binding protein 6 (amisyn) STXBP q12 Hs Potassium large conductance calcium-activated channel, subfamily M, beta member 1 KCNMB q34 Hs Hyperpolarization activated cyclic nucleotide-gated potassium channel 4 HCN q24-q25 Hs Lipocalin 2 (oncogene 24p3) LCN q34 Transcription and DNA related proteins Transcription factors and transcription regulation Hs TSC22 domain family, member 1 TSC22D q14 Hs SET and MYND domain containing 1 SMYD p11.2 Hs Forkhead box O1A (rhabdomyosarcoma) FOXO1A q14.1 Hs Zinc finger protein 281 ZNF q32.1 Hs Retinoid X receptor, alpha RXRA q34.3 Hs Four and a half LIM domains 3 FHL p34 Hs Four and a half LIM domains 1 FHL Xq26 Hs SMAD, mothers against DPP homolog 5 SMAD q31 Hs General transcription factor IIB GTF2B p22-p21 Hs Yip1 interacting factor homolog B YIF1B q13.2 Hs Myeloid/lymphoid or mixed-lineage leukemia MLL q23 Hs Zinc finger protein 187 ZNF p21.31 Hs Orthopedia homolog (Drosophila) OTP q13.3 Hs Basic leucine zipper transcription factor, ATF-like BATF q24.3 Hs NK2 transcription factor related, locus 2 (Drosophila) NKX pter-q11.23 Hs AF4/FMR2 family, member 3 AFF q11.2-q12 Hs Zinc finger protein 81 (HFZ20) ZNF Xp11.23 Hs Nuclear factor of activated T-cells, cytoplasmic, calcineurindependent 3 NFATC q22.2 Hs Iroquois homeobox protein 5 IRX q11.2-q13 Hs Retinoblastoma binding protein 8 RBBP q11.2 Hs Nuclear factor I/X (CCAAT-binding transcription factor) NFIX p13.3 Hs.1545 Caudal type homeo box transcription factor 1 CDX q31-q33 Hs Period homolog 1 (Drosophila) PER p p12 Hs T-cell leukemia, homeobox 2 TLX p13.1-p12 Hs Transcription elongation factor B (SIII), polypeptide 3 (110kDa, elongin A) TCEB p36.1 Hs Zinc finger protein 138 (clone phz-32) ZNF q11.21-q11.23 Hs AT rich interactive domain 5B (MRF1-like) ARID5B q21.2 Hs Immediate early response 5 IER q25.3 Hs Zinc finger protein 549 ZNF q13.43 Hs V-myc myelocytomatosis viral related oncogene, neuroblastoma derived (avian) MYCN p24.1 Hs Paired box gene 5 (B-cell lineage specific activator) PAX p13 Hs V-myb myeloblastosis viral oncogene homolog (avian) MYB q22-q23 DNA associated proteins Hs Topoisomerase (DNA) II beta 180kDa TOP2B p24 Hs Proliferation-inducing protein 38 RP11-301I q14.11 Hs MRE11 meiotic recombination 11 homolog A (S. cerevisiae) MRE11A q21 Hs Additional sex combs like 1 (Drosophila) ASXL q11.1 Hs Postmeiotic segregation increased 2-like 4 PMS2L q11-q22
4 Hs ZXD family zinc finger C ZXDC q21.2 Hs Topoisomerase (DNA) III beta TOP3B q11.22 Hs Histone 1, H2al HIST1H2AL p22-p21.3 Translation RNA transport and processing Hs Muscleblind-like (Drosophila) MBNL q25 Hs Heterogeneous nuclear ribonucleoprotein A0 HNRPA q31 Hs Poly(A) polymerase gamma PAPOLG p16.1 Hs Nuclear RNA export factor 1 NXF q12-q13 Hs TIA1 cytotoxic granule-associated RNA binding protein TIA p13 Hs Nucleoporin-like protein RIP HRB q36.3 Hs Apobec-1 complementation factor ACF q11.23 Hs LSM6 homolog, U6 small nuclear RNA associated (S. cerevisiae) LSM q31.22 Translation and post-translational modifications Hs Transmembrane and tetratricopeptide repeat containing 1 TMTC p11.22 Hs Glycosyltransferase 8 domain containing 1 GLT8D p21.1 Hs Ribosomal protein L30 RPL q22 Hs Ribosomal protein S15a RPS15A p Hs Arginyltransferase 1 ATE q26.13 Hs Lysyl oxidase-like 2 LOXL p21.3-p21.2 Hs UDP-glucose ceramide glucosyltransferase-like 1 UGCGL q14.3 Hs Dolichyl-phosphate mannosyltransferase polypeptide 1, catalytic subunit DPM q13.13 Hs Threonyl-tRNA synthetase TARS p13.2 Hs Eukaryotic elongation factor, selenocysteine-trna-specific EEFSEC q21.3 Hs Mannose-P-dolichol utilization defect 1 MPDU p13.1-p12 Hs UDP-glucose ceramide glucosyltransferase-like 2 UGCGL q32.1 Hs Mannosyl (beta-1,4-)-glycoprotein beta-1,4-nacetylglucosaminyltransferase MGAT q13.1 Hs Phosphatidylinositol N-acetylglucosaminyltransferase GPI1 subunit PIGQ p13.3 Hs Eukaryotic translation initiation factor 2-alpha kinase 2 EIF2AK p22-p21 Protein catabolism Ubiquitin-proteasome pathway Hs Ubiquitin specific protease 13 (isopeptidase T-3) USP q26.2-q26.3 Hs Ring finger protein 43 RNF q22 Hs Cullin-associated and neddylation-dissociated 1 CAND q14 Proteases and protease inhibitors Hs Proprotein convertase subtilisin/kexin type 1 PCSK q15-q21 Hs Matrix metalloproteinase 19 MMP q14 Hs Prolyl endopeptidase PREP q22 Hs Inter-alpha (globulin) inhibitor H2 ITIH p15 Hs Reelin RELN q22 Hs Crm, cramped-like (Drosophila) CRAMP1L p13.3 Hs Cathepsin O CTSO q31-q32 Hs Protease, serine, 12 (neurotrypsin, motopsin) PRSS q28.1 Others and proteins with unknown function Hs Putative lymphocyte G0/G1 switch gene G0S q32.2-q41 Hs LIM and cysteine-rich domains 1 LMCD p26-p24 Hs Arsenate resistance protein ARS2 ARS q21 Hs Transmembrane protein 125 TMEM p34.2 Hs Downregulated in ovarian cancer 1 DOC q12.1 Hs Ankyrin repeat domain 36 ANKRD q11.2 Hs Vitamin K epoxide reductase complex, subunit 1-like 1 VKORC1L q11.21 Hs Transmembrane protein 80 TMEM p15.5 Hs Mast cell-expressed membrane protein 1 MCEMP p13.2 Hs Endomucin EMCN q24 Hs Kin of IRRE like (Drosophila) KIRREL q21-q25 Hs Butyrophilin, subfamily 3, member A3 BTN3A p21.3 Hs Male sterility domain containing 1 MLSTD p11.22 Hs Fasciculation and elongation protein zeta 1 (zygin I) FEZ q24.2 Hs Family with sequence similarity 38, member B FAM38B p11.22 Hs WD repeat domain 73 WDR q25.2 Hs Myelin oligodendrocyte glycoprotein MOG p22.1 Hs Family with sequence similarity 32, member A FAM32A pter-p13.3 Hs Growth hormone regulated TBC protein 1 GRTP q34 Hs Neuron navigator 1 NAV Hs Transmembrane protein 115 TMEM p21.3 Hs Torsin A interacting protein 1 TOR1AIP q24.2 Hs Acrosomal vesicle protein 1 ACRV p12-q13 Hs WD repeat domain 22 WDR q23-q24.1 Hs Translocation associated membrane protein 1-like 1 TRAM1L q26 Hs Zinc finger, DHHC domain containing 14 ZDHHC q25.3 Hs Major facilitator superfamily domain containing 7 MFSD p16.3 Hs Huntingtin interacting protein E HYPE q24.1
5 Hypothetical proteins and EST Hs Hypothetical protein LOC LOC q13.1 Hs Transcribed locus 1.93 Hs Transcribed locus q35.3 Hs Hypothetical protein LOC LOC q24.22 Hs Transcribed locus 1.40 Hs Chromosome 20 open reading frame 117 C20orf q11.23 Hs Clone mrna sequence Hs Transcribed locus Hs hypothetical protein LOC q13.11 Hs FLJ16124 protein FLJ p14 Hs Hypothetical protein MGC26694 MGC p13.11 Hs Chromosome 5 open reading frame 3 C5orf q31-q33 Hs Chromosome 6 open reading frame 165 C6orf q15 Hs.4988 Hypothetical protein LOC LOC q21.3 Hs Chromosome 15 open reading frame 40 C15orf q25.2 Hs Transcribed locus Hs Transcribed locus Hs Hypothetical protein MGC10646 MGC q21.21 Hs hypothetical protein H q24.2 Hs Transcribed locus Hs Transcribed locus Hs Transcribed locus Hs Transcribed locus Hs Transcribed locus Hs.8429 Transcribed locus Hs Transcribed locus Hs Transcribed locus Hs Hypothetical gene supported by AK LOC q27 Hs chromosome 17 open reading frame p11.2 Hs Transcribed locus Hs Transcribed locus Hs Hypothetical BC331191_1 LOC q13.2 Hs Transcribed locus] Hs.6224 KIAA0895 protein KIAA p14.2 Hs Hypothetical protein FLJ12700 FLJ q36.1 Hs.9003 Chromosome 16 open reading frame 58 C16orf p11.2 Hs Full-length cdna clone CS0DC009YH Hs Transcribed locus Hs Transcribed locus Hs KIAA0922 protein KIAA q31.3 Hs Transcribed locus Hs Transcribed locus Hs Hypothetical protein FLJ40243 FLJ p13.1 Hs Hypothetical gene supported by AK q34.11 Hs Transcribed locus Hs Hypothetical protein LOC LOC p23.3 Hs Hypothetical protein FLJ13231 FLJ p13.2 Hs Transcribed locus Hs Transcribed locus Hs Hypothetical protein LOC LOC q36.1 Hs Full-length cdna clone CS0DK005YO Hs Transcribed locus Hs Hypothetical protein MGC35048 MGC p12.3 Hs Transcribed locus Hs Transcribed locus Hs KIAA1217 KIAA p12.1 Hs Transcribed locus Hs Transcribed locus Hs Hypothetical protein MGC24381 MGC p13.3 Hs hypothetical protein FLJ32949 FLJ q14.2 Hs Chromosome Y open reading frame 15B CYorf15B Yq Hs Transcribed locus Hs Hypothetical protein FLJ20366 FLJ q23.2 Hs Transcribed locus, -2.34
Gene Symbol Assay ID Gene Name. ACTB l Hs _m1 actin, beta. AKT1 b Hs _m1 v-akt murine thymoma viral oncogene homolog 1
 Electronic Supplementary Material (ESI) for Metallomics. This journal is The Royal Society of Chemistry 2015 Gene Symbol Assay ID Gene Name ACTB l Hs99999903_m1 actin, beta AKT1 b Hs00178289_m1 v-akt murine
Electronic Supplementary Material (ESI) for Metallomics. This journal is The Royal Society of Chemistry 2015 Gene Symbol Assay ID Gene Name ACTB l Hs99999903_m1 actin, beta AKT1 b Hs00178289_m1 v-akt murine
Supplemental Table 1. Significant genes and their values necessary for computing multi-factorial L value in the prediction model.
 Supplemental Table 1. Significant genes and their values necessary for computing multi-factorial L value in the prediction model. UG cluster Symbol Description t-value Midpoint p-value Unique id Hs.36566
Supplemental Table 1. Significant genes and their values necessary for computing multi-factorial L value in the prediction model. UG cluster Symbol Description t-value Midpoint p-value Unique id Hs.36566
As was previously described, gene expression profiles of the 70 CCRCCs (performed on cdna
 RSNA, 2015 10.1148/radiol.2015150800 Appendix E1 Gene Expression Profiling Training Set As was previously described, gene expression profiles of the 70 CCRCCs (performed on cdna microarrays containing
RSNA, 2015 10.1148/radiol.2015150800 Appendix E1 Gene Expression Profiling Training Set As was previously described, gene expression profiles of the 70 CCRCCs (performed on cdna microarrays containing
Impacto de la Hiperestimulación Ovárica Controlada sobre el la Expresión Génica del Endometrio durante la Ventana de Implantación
 Impacto de la Hiperestimulación Ovárica Controlada sobre el la Expresión Génica del Endometrio durante la Ventana de Implantación J.A. Horcajadas, A. Riesewijk, A. Cervero, A. Pellicer, S. Mosselman and
Impacto de la Hiperestimulación Ovárica Controlada sobre el la Expresión Génica del Endometrio durante la Ventana de Implantación J.A. Horcajadas, A. Riesewijk, A. Cervero, A. Pellicer, S. Mosselman and
BF GBP1 Hs AU INDO Hs.840 dioxygenase down
 Supplementary Table 2. Post- RASP SEB specific genes which passed the four different comparative tests and 1.5 #Fold- Change (FC) cut- off : 1.5 #Pairing option : Pairs of conditions S- 2: post- RASP-
Supplementary Table 2. Post- RASP SEB specific genes which passed the four different comparative tests and 1.5 #Fold- Change (FC) cut- off : 1.5 #Pairing option : Pairs of conditions S- 2: post- RASP-
Table S1. Gene expression levels from Affymetrix microarray data
 Table S1. Gene expression levels from Affymetrix microarray data Gene UniGene GeneBank hesc hesc-mec FH-Ep-P3 FH-Ep-PP OCT4 Hs.249184 NM_002701 10.8 6.8 5.4 5.5 NANOG Hs.661360 NM_024865 9.0 5.8 5.2 5.4
Table S1. Gene expression levels from Affymetrix microarray data Gene UniGene GeneBank hesc hesc-mec FH-Ep-P3 FH-Ep-PP OCT4 Hs.249184 NM_002701 10.8 6.8 5.4 5.5 NANOG Hs.661360 NM_024865 9.0 5.8 5.2 5.4
Zinc en metaloproteinas y enzimas - 2
 Zinc en metaloproteinas y enzimas - 2 » Se les denomina dedos porque contienen dominios estructurales capaces de anclarse en los surcos de la doble hélice del ADN como si fueran dedos.» La mayoría de estas
Zinc en metaloproteinas y enzimas - 2 » Se les denomina dedos porque contienen dominios estructurales capaces de anclarse en los surcos de la doble hélice del ADN como si fueran dedos.» La mayoría de estas
Mejora en la Composición de los Alimentos. Guillermo Reglero Director IMDEA Alimentación
 Mejora en la Composición de los Alimentos Guillermo Reglero Director IMDEA Alimentación 1 2 3 Enfermos hospitalizados por ECV 4 Alimentación para la salud 5 6 7 8 13 de marzo de 2014 9 10 11 12 13 8,00%
Mejora en la Composición de los Alimentos Guillermo Reglero Director IMDEA Alimentación 1 2 3 Enfermos hospitalizados por ECV 4 Alimentación para la salud 5 6 7 8 13 de marzo de 2014 9 10 11 12 13 8,00%
Cortex-specific genes ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs AA Unknown sc_id
![Cortex-specific genes ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs AA Unknown sc_id Cortex-specific genes ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs AA Unknown sc_id](/thumbs/85/92751791.jpg) Cortex-specific genes 184016 ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs.291676 AA767401 Unknown sc_id6474 187003 KIAA0007 KIAA0007 protein Hs.90315 AA251235 Similar to KIAA0007
Cortex-specific genes 184016 ESTs, Weakly similar to A46010 X-linked retinopathy protein [H.sapiens] Hs.291676 AA767401 Unknown sc_id6474 187003 KIAA0007 KIAA0007 protein Hs.90315 AA251235 Similar to KIAA0007
BIOLOGIA TUMORAL MATERIA ONCOLOGIA MOLECULAR. Dr. José Mordoh. Oncología Molecular, 2013
 BIOLOGIA TUMORAL MATERIA ONCOLOGIA MOLECULAR Dr. José Mordoh. Oncología Molecular, 2013 Orquesta. Bruno Brudovic LOCURA, de Francis Hammond ENSAYO DE ORQUESTA. FEDERICO FELLINI BIOLOGIA DE LA CELULA TUMORAL
BIOLOGIA TUMORAL MATERIA ONCOLOGIA MOLECULAR Dr. José Mordoh. Oncología Molecular, 2013 Orquesta. Bruno Brudovic LOCURA, de Francis Hammond ENSAYO DE ORQUESTA. FEDERICO FELLINI BIOLOGIA DE LA CELULA TUMORAL
similar to ADAM DEC1 precursor (A disintegrin and metalloproteinase domain-like protein decysin 1) (ADAM-like LOC ADAMDEC
 Table 2. Top list of the genes (n = 100) displaying the highest differential expression in the AI vs. SCNT profiles in bovine endometrial caruncles and intercaruncular areas Gene accession n Unigene ID
Table 2. Top list of the genes (n = 100) displaying the highest differential expression in the AI vs. SCNT profiles in bovine endometrial caruncles and intercaruncular areas Gene accession n Unigene ID
Secuenciación*Genómica*y*Medicina*Personalizada*del*Cáncer
 Secuenciación*Genómica*y*Medicina*Personalizada*del*Cáncer Ricardo Armisén Y. Centro de Investigación y Tratamiento del Cáncer U-CANCER: Red de Medicina Traslacional en Cáncer Programa de Fisiopatología!Instituto
Secuenciación*Genómica*y*Medicina*Personalizada*del*Cáncer Ricardo Armisén Y. Centro de Investigación y Tratamiento del Cáncer U-CANCER: Red de Medicina Traslacional en Cáncer Programa de Fisiopatología!Instituto
Comunicaciòn Celular. Copyright 2005 Pearson Education, Inc. publishing as Benjamin Cummings
 Comunicaciòn Celular La comunicaciòn intercelular es esencial para los organismos multicelulares Existen algunos mecanismos de regulaciòn celular universales Las señales extracelulares son convertidas
Comunicaciòn Celular La comunicaciòn intercelular es esencial para los organismos multicelulares Existen algunos mecanismos de regulaciòn celular universales Las señales extracelulares son convertidas
USO DE NUEVAS HERRAMIENTAS DE SECUENCIACIÓN ( RNA-SEQ ) APLICADAS EN SALMÓN DEL ATLÁNTICO
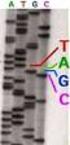 USO DE NUEVAS HERRAMIENTAS DE SECUENCIACIÓN ( RNA-SEQ ) APLICADAS EN SALMÓN DEL ATLÁNTICO Ingeniero en Acuicultura Mg. Sc. Dra. Natalia Lam P. Licenciado en Biología Mg. Sc. Dr. Cristian Araneda T. Licenciada
USO DE NUEVAS HERRAMIENTAS DE SECUENCIACIÓN ( RNA-SEQ ) APLICADAS EN SALMÓN DEL ATLÁNTICO Ingeniero en Acuicultura Mg. Sc. Dra. Natalia Lam P. Licenciado en Biología Mg. Sc. Dr. Cristian Araneda T. Licenciada
Síntesis de ácidos grasos
 Síntesis de ácidos grasos Las vías de síntesis y degradación son distintas Es similar a lo que ocurre entre glicolisis y gluneogénesis, síntesis y degradación de glicógeno Existen 4 diferencias esenciales
Síntesis de ácidos grasos Las vías de síntesis y degradación son distintas Es similar a lo que ocurre entre glicolisis y gluneogénesis, síntesis y degradación de glicógeno Existen 4 diferencias esenciales
DEPARTAMENTO DE BIOLOGÍA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA
 DEPARTAMENTO DE BIOLOGÍA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA DINÁMICA DE LA ACTINA Y TRÁFICO DE MEMBRANAS ASOCIADO AL COMPLEJO DE GOLGI: PAPEL REGULADOR DE RHOA, RAC1 Y CDC42 Tesis presentada
DEPARTAMENTO DE BIOLOGÍA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA DINÁMICA DE LA ACTINA Y TRÁFICO DE MEMBRANAS ASOCIADO AL COMPLEJO DE GOLGI: PAPEL REGULADOR DE RHOA, RAC1 Y CDC42 Tesis presentada
RECEPTORES ACOPLADOS A ENZIMAS. FISIOLOGÍA MOLECULAR UAM CUAJIMALPA Prof. M en C MANUEL GUTIÉRREZ VILLÁN
 RECEPTORES ACOPLADOS A ENZIMAS FISIOLOGÍA MOLECULAR UAM CUAJIMALPA Prof. M en C MANUEL GUTIÉRREZ VILLÁN RECEPTORES ACOPLADOS A ENZIMAS PROTEÍNAS TRANSMEMBRANALES CON UN DOMINIO EXTRACELULAR DE UNIÓN A
RECEPTORES ACOPLADOS A ENZIMAS FISIOLOGÍA MOLECULAR UAM CUAJIMALPA Prof. M en C MANUEL GUTIÉRREZ VILLÁN RECEPTORES ACOPLADOS A ENZIMAS PROTEÍNAS TRANSMEMBRANALES CON UN DOMINIO EXTRACELULAR DE UNIÓN A
ESTUDIO PARALELO CON MICROARRAYS GENÓMICOS Y DE EXPRESIÓN IDENTIFICA A LOS R-TRAIL, DR4 Y DR5, COMO GENES SUPRESORES CANDIDATOS EN LINFOMA B
 ESTUDIO PARALELO CON MICROARRAYS GENÓMICOS Y DE EXPRESIÓN IDENTIFICA A LOS R-TRAIL, DR4 Y DR5, COMO GENES SUPRESORES CANDIDATOS EN LINFOMA B F. Rubio-Moscardo, D.Blesa, C.Mestre R.Siebert, A.Rosenwald,
ESTUDIO PARALELO CON MICROARRAYS GENÓMICOS Y DE EXPRESIÓN IDENTIFICA A LOS R-TRAIL, DR4 Y DR5, COMO GENES SUPRESORES CANDIDATOS EN LINFOMA B F. Rubio-Moscardo, D.Blesa, C.Mestre R.Siebert, A.Rosenwald,
Tema 6 Expresión y Regulación de Genes Cap. 10, pág
 Tema 6 Expresión y Regulación de Genes Cap. 10, pág. 255-317 Qué es un gen? Qué es un gen? DNA transcripción mrna no se traducen trna rrna snrna traducción remoción de intrones en eucariotas Proteína construcción
Tema 6 Expresión y Regulación de Genes Cap. 10, pág. 255-317 Qué es un gen? Qué es un gen? DNA transcripción mrna no se traducen trna rrna snrna traducción remoción de intrones en eucariotas Proteína construcción
ESTUDIO DE GMI. ESTUDIO PROTEÓMICO COMPARATIVO ENTRE LOS IMPLANTES DENTALES DE GMI Y DE STRAUMANN
 ESTUDIO DE GMI. ESTUDIO PROTEÓMICO COMPARATIVO ENTRE LOS IMPLANTES DENTALES DE GMI Y DE STRAUMANN 2 Hipótesis. Estudio proteómico de la primera capa de proteínas depositada Después de la implantación:
ESTUDIO DE GMI. ESTUDIO PROTEÓMICO COMPARATIVO ENTRE LOS IMPLANTES DENTALES DE GMI Y DE STRAUMANN 2 Hipótesis. Estudio proteómico de la primera capa de proteínas depositada Después de la implantación:
Secuenciación Masiva en Cáncer
 Secuenciación Masiva en Cáncer Javier Freire Salinas Biólogo Molecular HUMV Coordinación científica: Dr. Fernando Rivera Hospital Universitario Marqués de Valdecilla, Santander Organizado por: Fundación
Secuenciación Masiva en Cáncer Javier Freire Salinas Biólogo Molecular HUMV Coordinación científica: Dr. Fernando Rivera Hospital Universitario Marqués de Valdecilla, Santander Organizado por: Fundación
Respuesta inmune adaptativa
 CURSO DE FORMACIÓN CBMSO-UAM 2010 Respuesta inmune adaptativa Balbino Alarcón CBMSO 5 de julio de 2010 La respuesta inmune adaptativa tiene dos características distintivas: -Existencia de un repertorio
CURSO DE FORMACIÓN CBMSO-UAM 2010 Respuesta inmune adaptativa Balbino Alarcón CBMSO 5 de julio de 2010 La respuesta inmune adaptativa tiene dos características distintivas: -Existencia de un repertorio
y su relación con cáncer
 Cir Cir 2012;80:389-398. y su relación con cáncer Alejandra Berenice Ochoa-Hernández,* Clara Ibet Juárez-Vázquez,** Mónica Alejandra Rosales-Reynoso,** Patricio Barros-Núñez* Resumen los procesos de regulación,
Cir Cir 2012;80:389-398. y su relación con cáncer Alejandra Berenice Ochoa-Hernández,* Clara Ibet Juárez-Vázquez,** Mónica Alejandra Rosales-Reynoso,** Patricio Barros-Núñez* Resumen los procesos de regulación,
Señalización Celular
 Señalización Celular Importancia de la Señalización Celular Metabolismo Movimiento Proliferación Supervivencia Muerte Diferenciación Señalización Celular Principios de Señalización Celular Moléculas de
Señalización Celular Importancia de la Señalización Celular Metabolismo Movimiento Proliferación Supervivencia Muerte Diferenciación Señalización Celular Principios de Señalización Celular Moléculas de
DEL GEN A LA PROTEINA
 DEL GEN A LA PROTEINA Parte I: Transcripción Capítulo 12 Expresión de Genes fenotipo Nivel molecular afecta la estructura y función de las células lo que determina que caracteres son expresados (fenotipo)
DEL GEN A LA PROTEINA Parte I: Transcripción Capítulo 12 Expresión de Genes fenotipo Nivel molecular afecta la estructura y función de las células lo que determina que caracteres son expresados (fenotipo)
DIFERENCIACION SUPERVIVENCIA / MUERTE
 CICLO CELULAR EXPRESIÓN de GENES Para dividirse, la célula recibe señales, las transmite y las interpreta como EXPRESIÓN de GENES ESPECÍFICOS para poder pasar por cada una de las fases del CICLO CELULAR
CICLO CELULAR EXPRESIÓN de GENES Para dividirse, la célula recibe señales, las transmite y las interpreta como EXPRESIÓN de GENES ESPECÍFICOS para poder pasar por cada una de las fases del CICLO CELULAR
Rendimiento Potencial de Materiales para un Ambiente Específico
 Rendimiento Potencial de Materiales para un Ambiente Específico Hernán Mauricio Romero, Ph.D. Coordinador del Programa de Biología y Mejoramiento Cenipalma Profesor Asociado Departamento de Biología Universidad
Rendimiento Potencial de Materiales para un Ambiente Específico Hernán Mauricio Romero, Ph.D. Coordinador del Programa de Biología y Mejoramiento Cenipalma Profesor Asociado Departamento de Biología Universidad
 Zoonosis parasitaria del hombre, mamíferos y aves producida por un protozoo coccidio el Toxoplasma gondii. Parásito intracelular obligado. En el hombre esta infección habitualmente es asintomática, las
Zoonosis parasitaria del hombre, mamíferos y aves producida por un protozoo coccidio el Toxoplasma gondii. Parásito intracelular obligado. En el hombre esta infección habitualmente es asintomática, las
Desarrollo de recursos para estudios genómicos en cítricos: creación de una colección de ESTs y una micromatriz de cdna
 Desarrollo de recursos para estudios genómicos en cítricos: creación de una colección de ETs y una micromatriz de cdna Proyecto de Genómica Funcional de Cítricos Instituto de Agroquímica y Tecnología de
Desarrollo de recursos para estudios genómicos en cítricos: creación de una colección de ETs y una micromatriz de cdna Proyecto de Genómica Funcional de Cítricos Instituto de Agroquímica y Tecnología de
IDENTIFICANDO GENES QUE FAVORECEN LA ADAPTACIÓN LOCAL EN Apis mellifera iberiensis
 IDENTIFICANDO GENES QUE FAVORECEN LA ADAPTACIÓN LOCAL EN Apis mellifera iberiensis Julio Chávez-Galarza, Dora Henriques, J. Spencer Johnston, J. Rufino, M. Alice Pinto School of Sciences University of
IDENTIFICANDO GENES QUE FAVORECEN LA ADAPTACIÓN LOCAL EN Apis mellifera iberiensis Julio Chávez-Galarza, Dora Henriques, J. Spencer Johnston, J. Rufino, M. Alice Pinto School of Sciences University of
MINIMAL GENOME En busca del GENOMA MÍNIMO. Dra. Rosario Gil RVGP, octubre 2004
 MINIMAL GENOME En busca del GENOMA MÍNIMO Dra. Rosario Gil RVGP, octubre 2004 El concepto de genoma mínimo Genoma mínimo (Koonin, 2000): grupo más pequeño de genes que debe ser suficiente para mantener
MINIMAL GENOME En busca del GENOMA MÍNIMO Dra. Rosario Gil RVGP, octubre 2004 El concepto de genoma mínimo Genoma mínimo (Koonin, 2000): grupo más pequeño de genes que debe ser suficiente para mantener
MOLECULAS DE ADHESION
 MOLECULAS DE ADHESION Marcelo Castillo Navarrete, TM MsC (c) Facultad de Medicina Dpto. Especialidades Médicas Carrera Tecnología Médica 11 de Abril de 2005 Moléculas de Adhesión Familia de las Integrinas
MOLECULAS DE ADHESION Marcelo Castillo Navarrete, TM MsC (c) Facultad de Medicina Dpto. Especialidades Médicas Carrera Tecnología Médica 11 de Abril de 2005 Moléculas de Adhesión Familia de las Integrinas
Mecanismos de invasión del cáncer de mama: Diferencias génicas CDIS vs CDI
 Mecanismos de invasión del cáncer de mama: Diferencias génicas CDIS vs CDI Siglo XX Enrique Lerma Hospital de la Santa Creu i Sant Pau Barcelona. Siglo XXI Índice Progresión del cáncer de mama Carcinoma
Mecanismos de invasión del cáncer de mama: Diferencias génicas CDIS vs CDI Siglo XX Enrique Lerma Hospital de la Santa Creu i Sant Pau Barcelona. Siglo XXI Índice Progresión del cáncer de mama Carcinoma
Mario Chiong. U Chile. 2007
 Receptores de membrana con actividad enzimática intrínseca Mario Chiong. U Chile. 2007 1 Tipo de receptores tirosina kinasa Factor de crecimiento nervioso (NGF) Factor de crecimiento derivado de plaquetas
Receptores de membrana con actividad enzimática intrínseca Mario Chiong. U Chile. 2007 1 Tipo de receptores tirosina kinasa Factor de crecimiento nervioso (NGF) Factor de crecimiento derivado de plaquetas
Supplemental Data. Hu et al. (2012). Plant Cell /tpc
 1 ATGGCGCGCCGCGCCGCTTCCCGCGCTGTTGGCGCCCTTCGCTCGGACGGCTCGATCCAA M A R R A A S R A V G A L R S D G S I Q 61 GGGCGAGGAGGCCGCGCGGGGGGCAGTGGCGCCGAGGACGCACGCCACGTGTTCGACGAA G R G G R A G G S G A E D A R H V
1 ATGGCGCGCCGCGCCGCTTCCCGCGCTGTTGGCGCCCTTCGCTCGGACGGCTCGATCCAA M A R R A A S R A V G A L R S D G S I Q 61 GGGCGAGGAGGCCGCGCGGGGGGCAGTGGCGCCGAGGACGCACGCCACGTGTTCGACGAA G R G G R A G G S G A E D A R H V
MECANISMOS INTRACELULARES DE SUPERVIVENCIA Y MUERTE NEURONAL EN MODELOS EXCITOTÓXICOS Y TRANSGÉNICOS DE LA ENFERMEDAD DE HUNTINGTON
 DEPARTAMENT DE BIOLOGIA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA MECANISMOS INTRACELULARES DE SUPERVIVENCIA Y MUERTE NEURONAL EN MODELOS EXCITOTÓXICOS Y TRANSGÉNICOS DE LA ENFERMEDAD DE HUNTINGTON
DEPARTAMENT DE BIOLOGIA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA MECANISMOS INTRACELULARES DE SUPERVIVENCIA Y MUERTE NEURONAL EN MODELOS EXCITOTÓXICOS Y TRANSGÉNICOS DE LA ENFERMEDAD DE HUNTINGTON
CICLO CELULAR Y SU REGULACIÓN G1
 G2 M S CICLO CELULAR Y SU REGULACIÓN G1 EXPRESIÓN de GENES Para dividirse, la célula recibe señales, las transmite y las interpreta como EXPRESIÓN de GENES ESPECÍFICOS para poder pasar por cada una de
G2 M S CICLO CELULAR Y SU REGULACIÓN G1 EXPRESIÓN de GENES Para dividirse, la célula recibe señales, las transmite y las interpreta como EXPRESIÓN de GENES ESPECÍFICOS para poder pasar por cada una de
EXPRESIÓN de GENES. o TRANSCRIPCIONAL. o TRADUCCIONAL REGULACIÓN. o MODIFICACIÓN DE PROTEÍNAS (Fosforilación)
 EL CICLO CELULAR EUCARIONTE Y SU REGULACIÓN G2 M S INTERFASE G1 EXPRESIÓN de GENES o TRANSCRIPCIONAL REGULACIÓN o TRADUCCIONAL o MODIFICACIÓN DE PROTEÍNAS (Fosforilación) La célula percibe señales para:
EL CICLO CELULAR EUCARIONTE Y SU REGULACIÓN G2 M S INTERFASE G1 EXPRESIÓN de GENES o TRANSCRIPCIONAL REGULACIÓN o TRADUCCIONAL o MODIFICACIÓN DE PROTEÍNAS (Fosforilación) La célula percibe señales para:
Diferentes mecanismos de transporte de proteínas
 Diferentes mecanismos de transporte de proteínas Todos los mecanismos involucran señales de tránsito presentes en las proteínas y receptores sobre la organela en cuestión Existen dos clases de señales
Diferentes mecanismos de transporte de proteínas Todos los mecanismos involucran señales de tránsito presentes en las proteínas y receptores sobre la organela en cuestión Existen dos clases de señales
Intro Redes Biológicas 1
 Intro Redes Biológicas 1 Cuestión de escala Sistemas biológicos: complejidad en diferentes escalas de organización Ecosistema Comunidad Población Organismo Sistema de órganos Tejido Célula Organelas Moléculas
Intro Redes Biológicas 1 Cuestión de escala Sistemas biológicos: complejidad en diferentes escalas de organización Ecosistema Comunidad Población Organismo Sistema de órganos Tejido Célula Organelas Moléculas
Angiogénesis, invasividad y metástasis
 Angiogénesis, invasividad y metástasis Papel del factor transcripcional Snail1 y otros represores transcripcionales en la regulación de la transición epitelio-mesénquima (EMT) Antonio García de Herreros
Angiogénesis, invasividad y metástasis Papel del factor transcripcional Snail1 y otros represores transcripcionales en la regulación de la transición epitelio-mesénquima (EMT) Antonio García de Herreros
Respuesta transcripcional global de C. albicans al estrés producido por la eliminación de la pared celular (protoplastos) o de una de sus proteínas
 Respuesta transcripcional global de C. albicans al estrés producido por la eliminación de la pared celular (protoplastos) o de una de sus proteínas (Ssr1). Pared Celular Protección física y química a la
Respuesta transcripcional global de C. albicans al estrés producido por la eliminación de la pared celular (protoplastos) o de una de sus proteínas (Ssr1). Pared Celular Protección física y química a la
Investigación traslacional en vías de transducción. Miguel Quintela-Fandino, MD, PhD, M.Sc. 26 de octubre de 2012 Simposio SEOM
 Investigación traslacional en vías de transducción Miguel Quintela-Fandino, MD, PhD, M.Sc. 26 de octubre de 2012 Simposio SEOM Guión Porqué éinvestigar en señalización ñli ió en clínica Retos de muestreo
Investigación traslacional en vías de transducción Miguel Quintela-Fandino, MD, PhD, M.Sc. 26 de octubre de 2012 Simposio SEOM Guión Porqué éinvestigar en señalización ñli ió en clínica Retos de muestreo
METABOLISMO DEL GLUCOGENO
 METABLISM DEL GLUCGEN QUIMICA BILGICA II Dra. Ma. Victoria Aguirre Profesora Titular bjetivos: Describir las etapas de la glucogenólisis y la glucogenogénesis Distinguir las regulaciones del metabolismo
METABLISM DEL GLUCGEN QUIMICA BILGICA II Dra. Ma. Victoria Aguirre Profesora Titular bjetivos: Describir las etapas de la glucogenólisis y la glucogenogénesis Distinguir las regulaciones del metabolismo
Moléculas Adhesión. T.M. MsC Juan Luis Castillo N. CAM. Cellular Adhesion Molecules. Glicoproteínas: Dominio citoplasmático (extremo carboxilo)
 Departamento de Especialidades Médicas Facultad de Medicina Universidad de Concepción Moléculas Adhesión T.M. MsC Juan Luis Castillo N. Miércoles 30 de Marzo de 2006 Glicoproteínas: Dominio citoplasmático
Departamento de Especialidades Médicas Facultad de Medicina Universidad de Concepción Moléculas Adhesión T.M. MsC Juan Luis Castillo N. Miércoles 30 de Marzo de 2006 Glicoproteínas: Dominio citoplasmático
Biología General: Macromoléculas biológicas. Prof. Bernal Gerardo Garro Mora
 Biología General: Macromoléculas biológicas Prof. Bernal Gerardo Garro Mora Objetivos: Al finalizar la clase el estudiante será capaz de: Definir a las moléculas orgánicas y los principales grupos funcionales
Biología General: Macromoléculas biológicas Prof. Bernal Gerardo Garro Mora Objetivos: Al finalizar la clase el estudiante será capaz de: Definir a las moléculas orgánicas y los principales grupos funcionales
Oncogenes DR. JOSE MORDOH. Maestría Biología Molecular Médica UBA 2016
 Oncogenes DR. JOSE MORDOH Maestría Biología Molecular Médica UBA 2016 What is the molecular basis of cancer? Cancers are formed from repeated rounds of DNA mutation, competition, and natural selection
Oncogenes DR. JOSE MORDOH Maestría Biología Molecular Médica UBA 2016 What is the molecular basis of cancer? Cancers are formed from repeated rounds of DNA mutation, competition, and natural selection
GENOMAS DEL CLOROPLASTO Y LA MITOCONDRIA. prof. Jorge Hernández T., Ph.D.
 GENOMAS DEL CLOROPLASTO Y LA MITOCONDRIA prof. Jorge Hernández T., Ph.D. La Teoría Endosimbiótica prof. Jorge Hernández T., Ph.D. Evidence for Endosymbiotic Theory Mitochondria have very similar characteristics
GENOMAS DEL CLOROPLASTO Y LA MITOCONDRIA prof. Jorge Hernández T., Ph.D. La Teoría Endosimbiótica prof. Jorge Hernández T., Ph.D. Evidence for Endosymbiotic Theory Mitochondria have very similar characteristics
UNIVERSIDAD CENTRAL DE VENEZUELA FACULTAD DE MEDICINA ESCUELA DE MEDICINA LUIS RAZETTI CÁTEDRA DE FISIOLOGIA. Emilia Díaz 6 de marzo de 2012
 UNIVERSIDAD CENTRAL DE VENEZUELA FACULTAD DE MEDICINA ESCUELA DE MEDICINA LUIS RAZETTI CÁTEDRA DE FISIOLOGIA Emilia Díaz 6 de marzo de 2012 CONTENIDO PROGRAMÁTICO El eje hipotálamo-hipófisis-tiroides:
UNIVERSIDAD CENTRAL DE VENEZUELA FACULTAD DE MEDICINA ESCUELA DE MEDICINA LUIS RAZETTI CÁTEDRA DE FISIOLOGIA Emilia Díaz 6 de marzo de 2012 CONTENIDO PROGRAMÁTICO El eje hipotálamo-hipófisis-tiroides:
Cáncer. Dr. Monterrubio. Capitulo 14
 Cáncer Capitulo 14 Cáncer 1. Caracterizado por la división celular sin control 2. ~1.5 millón/año de americanos diagnosticados (0.5 millón/año mueren por la enfermedad) 3. 10% de canceres con predisposición
Cáncer Capitulo 14 Cáncer 1. Caracterizado por la división celular sin control 2. ~1.5 millón/año de americanos diagnosticados (0.5 millón/año mueren por la enfermedad) 3. 10% de canceres con predisposición
Las células reciben señales
 Las células reciben señales Ubicación espacial Temporalidad del efecto Mensaje Bifurcación y convergencia de las señales La química de la transducción de señales Tyr, Ser, Thr: aminoácidos fosforilables
Las células reciben señales Ubicación espacial Temporalidad del efecto Mensaje Bifurcación y convergencia de las señales La química de la transducción de señales Tyr, Ser, Thr: aminoácidos fosforilables
Biología general de la membrana plasmática. Prof. Bernal Gerardo Garro Mora UCR
 Biología general de la membrana plasmática Prof. Bernal Gerardo Garro Mora UCR Las membranas celulares son estructuras muy complejas, cuyos elementos estructurales difieren según el tipo de organismo,
Biología general de la membrana plasmática Prof. Bernal Gerardo Garro Mora UCR Las membranas celulares son estructuras muy complejas, cuyos elementos estructurales difieren según el tipo de organismo,
HISTOSONDA DR. JESÚS ALBA LOSADA
 HISTOSONDA DR. JESÚS ALBA LOSADA HISTOSONDA Sondas de DNA monocatenario para el estudio de la expresión génica en tejidos fijados en formol e incluidos en parafina mediante la hibridación in situ HIBRIDACIÓN
HISTOSONDA DR. JESÚS ALBA LOSADA HISTOSONDA Sondas de DNA monocatenario para el estudio de la expresión génica en tejidos fijados en formol e incluidos en parafina mediante la hibridación in situ HIBRIDACIÓN
Enzyme 2. D Reaction 1. Reaction 3 Starting. Reaction 2. Product molecule
 Figure 8-01 LE 8-UN141 Enzyme 1 Enzyme 2 Enzyme 3 A B C D Reaction 1 Reaction 2 Reaction 3 Starting Product molecule LE 8-2 On the platform, the diver has more potential energy. Diving converts potential
Figure 8-01 LE 8-UN141 Enzyme 1 Enzyme 2 Enzyme 3 A B C D Reaction 1 Reaction 2 Reaction 3 Starting Product molecule LE 8-2 On the platform, the diver has more potential energy. Diving converts potential
ADENDUM TEMA 1 RUTA FOSFATIDIL INOSITOL 3 QUINASA. 1.6.2.2 Ruta de la Fisfatidil inositol 3 quinasa (PI3K)
 ADENDUM TEMA 1 RUTA FOSFATIDIL INOSITOL 3 QUINASA 1.6.2.2 Ruta de la Fisfatidil inositol 3 quinasa (PI3K) Además de la ruta de las MAPK, descrita anteriormente, otra de las rutas que activan los receptores
ADENDUM TEMA 1 RUTA FOSFATIDIL INOSITOL 3 QUINASA 1.6.2.2 Ruta de la Fisfatidil inositol 3 quinasa (PI3K) Además de la ruta de las MAPK, descrita anteriormente, otra de las rutas que activan los receptores
Regulación transcripcional en semillas de cebada cv. Bomi y su mutante alto en lisina Riso-1508
 IX Reunión de Biología Molecular de Plantas Regulación transcripcional en semillas de cebada cv. Bomi y su mutante alto en lisina Riso-58 Centro de Biotecnología y Genómica de Plantas Universidad Politécnica
IX Reunión de Biología Molecular de Plantas Regulación transcripcional en semillas de cebada cv. Bomi y su mutante alto en lisina Riso-58 Centro de Biotecnología y Genómica de Plantas Universidad Politécnica
FACTORS AFFECTING GROWTH IN CHILDREN WITH CHRONIC KIDNEY DISEASE. THE GH-IGF1 Axis.
 FACTORS AFFECTING GROWTH IN CHILDREN WITH CHRONIC KIDNEY DISEASE. THE GH-IGF1 Axis. Francisco Cano MD Titular Professor Division of Pediatric Nephrology Luis Calvo Mackenna Children s Hospital Department
FACTORS AFFECTING GROWTH IN CHILDREN WITH CHRONIC KIDNEY DISEASE. THE GH-IGF1 Axis. Francisco Cano MD Titular Professor Division of Pediatric Nephrology Luis Calvo Mackenna Children s Hospital Department
ONCOLOGIA MOLECULAR. Maestría en Biología Molecular Médica 2008
 ONCOLOGIA MOLECULAR Maestría en Biología Molecular Médica 2008 QUE ES EL CANCER? 1) Proliferación n ilimitada 2) Capacidad de dar metástasis Normal cell division: enough to replace MBMM 2008 Excessive
ONCOLOGIA MOLECULAR Maestría en Biología Molecular Médica 2008 QUE ES EL CANCER? 1) Proliferación n ilimitada 2) Capacidad de dar metástasis Normal cell division: enough to replace MBMM 2008 Excessive
Respuesta inmune adaptativa
 CURSO DE FORMACIÓN CBMSO-UAM 2012 Respuesta inmune adaptativa Balbino Alarcón CBMSO 19 de enero de 2012 La respuesta inmune adaptativa tiene dos características distintivas: -Existencia de un repertorio
CURSO DE FORMACIÓN CBMSO-UAM 2012 Respuesta inmune adaptativa Balbino Alarcón CBMSO 19 de enero de 2012 La respuesta inmune adaptativa tiene dos características distintivas: -Existencia de un repertorio
Regulación de la expresión en Eucariotas
 Regulación de la expresión en Eucariotas Amplificación Génica Proteínas o RNAs que se requieren en abundancia están codificados por genes múltiples Duplicación génica Control genómico La estructura de
Regulación de la expresión en Eucariotas Amplificación Génica Proteínas o RNAs que se requieren en abundancia están codificados por genes múltiples Duplicación génica Control genómico La estructura de
Transcripción y Traducción
 Transcripción y Traducción Traducido Prof. Wanda Ortiz Carrión UIPR-Fajardo Uso exclusivo estudiantes BIOL 1102 Puede estar sujeto a leyes Derecho de Autor 2016-10 Figure 17.1 Transcripción y Traducción
Transcripción y Traducción Traducido Prof. Wanda Ortiz Carrión UIPR-Fajardo Uso exclusivo estudiantes BIOL 1102 Puede estar sujeto a leyes Derecho de Autor 2016-10 Figure 17.1 Transcripción y Traducción
Células de la piel. Capa córnea Capa granulosa. Estrato basal. Células del sistema inmunitario. Células de Langerhans. Queratinocito.
 Células de la piel Capa córnea Capa granulosa Células de Langerhans Queratinocito Estrato basal Melanocito Fibroblasto Células del sistema inmunitario 1 Células de la piel Células del sistema inmunitario
Células de la piel Capa córnea Capa granulosa Células de Langerhans Queratinocito Estrato basal Melanocito Fibroblasto Células del sistema inmunitario 1 Células de la piel Células del sistema inmunitario
Las Citocinas (II) PATRON DE TIPO TH1
 Las Citocinas (II) Dr. Juan Rodríguez-Tafur D. Profesor Asociado de Farmacología e Inmunología Laboratorio de Farmacología de la Respuesta Inmune Coordinador del Comité de Residentado Médico de Inmunología
Las Citocinas (II) Dr. Juan Rodríguez-Tafur D. Profesor Asociado de Farmacología e Inmunología Laboratorio de Farmacología de la Respuesta Inmune Coordinador del Comité de Residentado Médico de Inmunología
Diagnóstico Molecular en Linfomas. Mauricio Lema Medina MD Clínica SOMA / Clínica Astorga, Medellín
 Diagnóstico Molecular en Linfomas Mauricio Lema Medina MD Clínica SOMA / Clínica Astorga, Medellín Curso de Biología Molecular, Diagnóstico e Investigación para Hematólogos y Oncólogos, Villa de Leyva,
Diagnóstico Molecular en Linfomas Mauricio Lema Medina MD Clínica SOMA / Clínica Astorga, Medellín Curso de Biología Molecular, Diagnóstico e Investigación para Hematólogos y Oncólogos, Villa de Leyva,
INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR. Señalización Celular
 INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR Señalización Celular 12-6-2017 Señal/estímulo (Químico, mecánico, lumínico, etc.) Transducción de la señal (suele haber amplificación de la misma) Respuesta
INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR Señalización Celular 12-6-2017 Señal/estímulo (Químico, mecánico, lumínico, etc.) Transducción de la señal (suele haber amplificación de la misma) Respuesta
Selectinas e Inflamación. Dr. Iván Martínez Duncker R.
 Selectinas e Inflamación Dr. Iván Martínez Duncker R. Qué es la inflamación? Estimulos nocivos causan una reacción vascular protectora llamada inflamación que puede ser aguda o crónica Diluir Destruir
Selectinas e Inflamación Dr. Iván Martínez Duncker R. Qué es la inflamación? Estimulos nocivos causan una reacción vascular protectora llamada inflamación que puede ser aguda o crónica Diluir Destruir
Prevalencia e interpretación de las variantes de significado incierto
 Prevalencia e interpretación de las variantes de significado incierto Orland Díez i Gibert Àrea de Genètica Clínica i Molecular Hospital Universitari Vall d Hebron Grup d Oncogenètica Vall d Hebron Institut
Prevalencia e interpretación de las variantes de significado incierto Orland Díez i Gibert Àrea de Genètica Clínica i Molecular Hospital Universitari Vall d Hebron Grup d Oncogenètica Vall d Hebron Institut
Enzimas: conceptos básicos y cinéticos
 Enzimas: conceptos básicos y cinéticos ENZIMAS CATALIZADOR BIOLÓGICO Poder catalítico Especificidad S + E ES E + P S= Sustrato P= Producto ES= Complejo enzima-sustrato Enzyme Nonenzymatic half-life Uncatalyzed
Enzimas: conceptos básicos y cinéticos ENZIMAS CATALIZADOR BIOLÓGICO Poder catalítico Especificidad S + E ES E + P S= Sustrato P= Producto ES= Complejo enzima-sustrato Enzyme Nonenzymatic half-life Uncatalyzed
Transcripción del DNA y Regulación de la expresión genética
 Universidad de Chile, Facultad de Medicina, ICBM Programa de Biología Celular y Molecular Transcripción del DNA y Regulación de la expresión genética Curso de Biología Molecular y Genómica 2018 Dr. Héctor
Universidad de Chile, Facultad de Medicina, ICBM Programa de Biología Celular y Molecular Transcripción del DNA y Regulación de la expresión genética Curso de Biología Molecular y Genómica 2018 Dr. Héctor
Un viaje histórico por la biología molecular. que nos permitirá conocer algunos conceptos
 Biología Molecular Un viaje histórico por la biología molecular que nos permitirá conocer algunos conceptos La herencia de fenotipos sigue reglas predecibles El inicio de la genética (G. Mendel) Cruces
Biología Molecular Un viaje histórico por la biología molecular que nos permitirá conocer algunos conceptos La herencia de fenotipos sigue reglas predecibles El inicio de la genética (G. Mendel) Cruces
SECCIÓN 1: INVENTORES
 SECCIÓN 1: INVENTORES INVENTORES Nombre: Cargo: Laboratorio: Teléfonos: Dirección: Email: Ciudadanía: Nombre: Cargo: Laboratorio: Teléfonos: Dirección: Email: Ciudadanía: Nombre: Cargo: Laboratorio: Teléfonos:
SECCIÓN 1: INVENTORES INVENTORES Nombre: Cargo: Laboratorio: Teléfonos: Dirección: Email: Ciudadanía: Nombre: Cargo: Laboratorio: Teléfonos: Dirección: Email: Ciudadanía: Nombre: Cargo: Laboratorio: Teléfonos:
Los genes se expresan con diferente eficiencia
 De ADN a proteína Los genes se expresan con diferente eficiencia El mecanismo de transcripción Diferentes clases de ARN s Transcripción en bacterias La cadena codificante tiene la misma secuencia que el
De ADN a proteína Los genes se expresan con diferente eficiencia El mecanismo de transcripción Diferentes clases de ARN s Transcripción en bacterias La cadena codificante tiene la misma secuencia que el
Mycobacterias. Dra. en C. Monserrat Alvarez Zavala
 Mycobacterias Dra. en C. Monserrat Alvarez Zavala Infección del Epitelio alveolar por M.tb La mycobacteria puede invadir y replicarse en las células alveolares epiteliales, esta invasión en benéfica para
Mycobacterias Dra. en C. Monserrat Alvarez Zavala Infección del Epitelio alveolar por M.tb La mycobacteria puede invadir y replicarse en las células alveolares epiteliales, esta invasión en benéfica para
Clases de GPCRs. C. Tipo metabotrópico glutamato/ feromona. E. Receptores camp (Dictiostelium) -- Subfamilia Frizzled/Smoothened
 Clases de GPCRs A. Tipo rodopsina B. Tipo secretina C. Tipo metabotrópico glutamato/ feromona D. Feromona hongos E. Receptores camp (Dictiostelium) -- Subfamilia Frizzled/Smoothened Cada GPCR tiene requerimientos
Clases de GPCRs A. Tipo rodopsina B. Tipo secretina C. Tipo metabotrópico glutamato/ feromona D. Feromona hongos E. Receptores camp (Dictiostelium) -- Subfamilia Frizzled/Smoothened Cada GPCR tiene requerimientos
Mecanismos de aparición de genes
 Mecanismos de aparición de genes - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera - Duplicación génica - Duplicación cromosómica - Duplicación genómica Introducción
Mecanismos de aparición de genes - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera - Duplicación génica - Duplicación cromosómica - Duplicación genómica Introducción
Receptores de membrana y transduccion de la señal
 Receptores de membrana y transduccion de la señal Elementos efectores Proteínas reguladoras Transducción de la señal HORMONAS Mensajeros bioquímicos que actúan integrando las respuestas de las diferentes
Receptores de membrana y transduccion de la señal Elementos efectores Proteínas reguladoras Transducción de la señal HORMONAS Mensajeros bioquímicos que actúan integrando las respuestas de las diferentes
Bacteriófagos. 0.5 m. Capitulo 5 Microbial Genetics Capitulo 3 y 7 Molecular genetics of Bacteria
 Bacteriófagos 0.5 m Capitulo 5 Microbial Genetics Capitulo 3 y 7 Molecular genetics of Bacteria Bacteriofagos (fagos) Definición Parásito intracelular obligado que se multiplica dentro de una bacteria
Bacteriófagos 0.5 m Capitulo 5 Microbial Genetics Capitulo 3 y 7 Molecular genetics of Bacteria Bacteriofagos (fagos) Definición Parásito intracelular obligado que se multiplica dentro de una bacteria
Hay regulación a nivel actividad GTPasa de la beta-tubulina?
 Hay regulación a nivel actividad GTPasa de la beta-tubulina? GPCRs y proteínas G triméricas Schappi et al., BBA 2014 Citoesqueleto (CSK) y morfología celular Microfilamentos (8 nm): polímeros bicatenarios
Hay regulación a nivel actividad GTPasa de la beta-tubulina? GPCRs y proteínas G triméricas Schappi et al., BBA 2014 Citoesqueleto (CSK) y morfología celular Microfilamentos (8 nm): polímeros bicatenarios
Seminario de Fisiología Renal MECANISMOS DE TRANSPORTE TUBULAR. Dr. Ernesto Castro Aguilar Julio 2014
 ! Seminario de Fisiología Renal MECANISMOS DE TRANSPORTE TUBULAR Dr. Ernesto Castro Aguilar Julio 2014 TRANSPORTE EPITELIAL Filtrado glomerular sufre una serie de modificaciones antes de convertirse en
! Seminario de Fisiología Renal MECANISMOS DE TRANSPORTE TUBULAR Dr. Ernesto Castro Aguilar Julio 2014 TRANSPORTE EPITELIAL Filtrado glomerular sufre una serie de modificaciones antes de convertirse en
Las células reciben señales. Transmiten la señal. Asimilan la señal. Responden
 Las células reciben señales Transmiten la señal Asimilan la señal Responden Ubicación espacial de la señal Temporalidad del efecto Modificación posttraduccional de proteínas Expresión genética Mensaje
Las células reciben señales Transmiten la señal Asimilan la señal Responden Ubicación espacial de la señal Temporalidad del efecto Modificación posttraduccional de proteínas Expresión genética Mensaje
Catenina Beta: características estructurales y funcionales
 Catenina Beta: características estructurales y funcionales Alfredo Trejo Córdova *, Maria del Carmen Navarro Maldonado, Adolfo Rosado García **. Recibido: 19 de noviembre de 2009. Aceptado: 08 de febrero
Catenina Beta: características estructurales y funcionales Alfredo Trejo Córdova *, Maria del Carmen Navarro Maldonado, Adolfo Rosado García **. Recibido: 19 de noviembre de 2009. Aceptado: 08 de febrero
INTERACCIONES Y COMUNICACIÓN ENTRE CELULAS
 INTERACCIONES Y COMUNICACIÓN ENTRE CELULAS 1 CITOESQUELETO Las células presentan muy variadas formas neuronas eritrocitos epitelio 2 CITOESQUELETO -Resistencia Mecánica -Forma - Polarización - Organización
INTERACCIONES Y COMUNICACIÓN ENTRE CELULAS 1 CITOESQUELETO Las células presentan muy variadas formas neuronas eritrocitos epitelio 2 CITOESQUELETO -Resistencia Mecánica -Forma - Polarización - Organización
TRANSDUCCIÓN DE SEÑALES EN EUCARIOTAS TRANSDUCCIÓN DE LA SEÑAL INTRACELULAR PROTEÍNAS G
 TRANSDUCCIÓN DE SEÑALES EN EUCARIOTAS TRANSDUCCIÓN DE LA SEÑAL INTRACELULAR PROTEÍNAS G Esta diapositiva la pusimos para introducir el tipo de modificaciones que se realizan en las moléculas que van a
TRANSDUCCIÓN DE SEÑALES EN EUCARIOTAS TRANSDUCCIÓN DE LA SEÑAL INTRACELULAR PROTEÍNAS G Esta diapositiva la pusimos para introducir el tipo de modificaciones que se realizan en las moléculas que van a
MECANISMOS DE AMPLIFICACIÓN DE LA RESPUESTA INMUNOLÓGICA. José Angel Cova, MD
 MECANISMOS DE AMPLIFICACIÓN DE LA RESPUESTA INMUNOLÓGICA José Angel Cova, MD jacova@ula.ve INMUNIDAD INNATA MECANISMOS DE DEFENSA ACTIVOS PREVIO AL ENCUENTRO CON EL ANTÍGENO SON MECANISMOS NO MODIFICADOS
MECANISMOS DE AMPLIFICACIÓN DE LA RESPUESTA INMUNOLÓGICA José Angel Cova, MD jacova@ula.ve INMUNIDAD INNATA MECANISMOS DE DEFENSA ACTIVOS PREVIO AL ENCUENTRO CON EL ANTÍGENO SON MECANISMOS NO MODIFICADOS
NUEVAS PERSPECTIVAS: ESTATINAS Y CANCER
 NUEVAS PERSPECTIVAS: ESTATINAS Y CANCER NEW PERSPECTIVE: STATINS AND CANCER Guzmán Velásquez Luis Jesús Junior 1 RESUMEN El presente artículo analiza la participación de los inhibidores de la enzima 3-hidroxi-3-
NUEVAS PERSPECTIVAS: ESTATINAS Y CANCER NEW PERSPECTIVE: STATINS AND CANCER Guzmán Velásquez Luis Jesús Junior 1 RESUMEN El presente artículo analiza la participación de los inhibidores de la enzima 3-hidroxi-3-
de metabolitos secundarios
 Elicitación de la producción de metabolitos secundarios Los vegetales no poseen células ni tejidos especializados en la defensa TODAS sus células poseen la potencialidad de responder por si mismas para
Elicitación de la producción de metabolitos secundarios Los vegetales no poseen células ni tejidos especializados en la defensa TODAS sus células poseen la potencialidad de responder por si mismas para
MECANISMOS DE SEÑALIZACIÓN INTRACELULAR DE SUPERVIVENCIA Y MUERTE NEURONAL REGULADOS POR FACTORES TRÓFICOS
 DEPARTAMENT DE BIOLOGIA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA UNIVERSITAT DE BARCELONA MECANISMOS DE SEÑALIZACIÓN INTRACELULAR DE SUPERVIVENCIA Y MUERTE NEURONAL REGULADOS POR FACTORES TRÓFICOS
DEPARTAMENT DE BIOLOGIA CEL LULAR I ANATOMIA PATOLÒGICA FACULTAT DE MEDICINA UNIVERSITAT DE BARCELONA MECANISMOS DE SEÑALIZACIÓN INTRACELULAR DE SUPERVIVENCIA Y MUERTE NEURONAL REGULADOS POR FACTORES TRÓFICOS
Microbial Genetics. Capítulo 13 07/11/2014. Central Dogma Regulation of Gene Expression Genetic Exchange. DNA (genotype) Transcription.
 Capítulo 13 Microbial Genetics Central Dogma Regulation of Gene Expression Genetic Exchange R E P L I C A C I Ó N Y E X P R E S I Ó N D E L G E N O M A B A C T E R I A N O Dogma Central de la Genética
Capítulo 13 Microbial Genetics Central Dogma Regulation of Gene Expression Genetic Exchange R E P L I C A C I Ó N Y E X P R E S I Ó N D E L G E N O M A B A C T E R I A N O Dogma Central de la Genética
UD. III. BIOLOGIA CEL LULAR. Ll. III. 1. Organització cel lular
 UD. III. BIOLOGIA CEL LULAR. Ll. III. 1. Organització cel lular UD. III. BIOLOGIA CEL LULAR. Ll. III. 1. Organització cel lular 1. Cèl lula eucariota vegetal UD. III. BIOLOGIA CEL LULAR. Ll. III. 1. Organització
UD. III. BIOLOGIA CEL LULAR. Ll. III. 1. Organització cel lular UD. III. BIOLOGIA CEL LULAR. Ll. III. 1. Organització cel lular 1. Cèl lula eucariota vegetal UD. III. BIOLOGIA CEL LULAR. Ll. III. 1. Organització
ETAPAS PRINCIPALES DEL CICLO CELULAR EN EUCARIONTES. Mitosis
 ETAPAS PRINCIPALES DEL CICLO CELULAR EN EUCARIONTES Mitosis REPASO SOBRE LAS DIFERENCIAS ENTRE MITOSIS Y MEIOSIS Mitosis Meiosis Sinapsis (apareamiento de cromosomas homologos) 1a. División 2da. división
ETAPAS PRINCIPALES DEL CICLO CELULAR EN EUCARIONTES Mitosis REPASO SOBRE LAS DIFERENCIAS ENTRE MITOSIS Y MEIOSIS Mitosis Meiosis Sinapsis (apareamiento de cromosomas homologos) 1a. División 2da. división
Segunda Parte: El camino de vuelta
 Segunda Parte: El camino de vuelta Del núcleo a la sinapsis Cómo llegan las macromoléculas a las sinapsis específicas? Hipótesis para explicar la especificidad sináptica Synaptic Tagging New gene products
Segunda Parte: El camino de vuelta Del núcleo a la sinapsis Cómo llegan las macromoléculas a las sinapsis específicas? Hipótesis para explicar la especificidad sináptica Synaptic Tagging New gene products
p53: estructura y actividad biológica. La ruta p53-mdm2-arf Ignacio Palmero Master de Oncología Molecular
 : estructura y actividad biológica. La ruta --ARF Ignacio Palmero Master de Oncología Molecular alteraciones oncogénicas (estímulos mitogénicos inapropiados, daño a DNA) Mecanismos de defensa supresión
: estructura y actividad biológica. La ruta --ARF Ignacio Palmero Master de Oncología Molecular alteraciones oncogénicas (estímulos mitogénicos inapropiados, daño a DNA) Mecanismos de defensa supresión
Mecanismos de evolución de los genomas. - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera
 Mecanismos de evolución de los genomas - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera Evolución del tamaño de los genes Contenido Introducción: mecanismos de amplificación
Mecanismos de evolución de los genomas - Duplicación de dominios y alargamientos de genes. - Exon-shuffling - Genes quimera Evolución del tamaño de los genes Contenido Introducción: mecanismos de amplificación
GENOMA, ENVEJECIMIENTO Y VIH/SIDA
 GENOMA, ENVEJECIMIENTO Y VIH/SIDA Felipe García Vallejo. PhD Profesor Titular y Director Científico Laboratorio de Biología Molecular y Patogénesis. Escuela de Ciencias Básicas. Facultad de Salud. Universidad
GENOMA, ENVEJECIMIENTO Y VIH/SIDA Felipe García Vallejo. PhD Profesor Titular y Director Científico Laboratorio de Biología Molecular y Patogénesis. Escuela de Ciencias Básicas. Facultad de Salud. Universidad
RELACIÓN DE PRODUCTOS DE LAS LÍNEAS DE GENERACIÓN Y APLICACIÓN DEL CONOCIMIENTO DIAGNÓSTICO, PREVENCIÓN Y RIESGOS PARA LA SALUD EN LA REGIÓN NAYARITA
 RELACIÓN DE PRODUCTOS DE LAS LÍNEAS DE GENERACIÓN APLICACIÓN DEL CONOCIMIENTO NOMBRE DEL ARTÍCULO THE RELEASE KINETICS OF ß- CAROTENE NANOCAPSULES/XANTHAN GUM COATING AND QUALIT CHANGES IN FRESH-CUT MELON
RELACIÓN DE PRODUCTOS DE LAS LÍNEAS DE GENERACIÓN APLICACIÓN DEL CONOCIMIENTO NOMBRE DEL ARTÍCULO THE RELEASE KINETICS OF ß- CAROTENE NANOCAPSULES/XANTHAN GUM COATING AND QUALIT CHANGES IN FRESH-CUT MELON
CURSO TEÓRICO-PRÁCTICO DE MÉTODOS METAGENÓMICOS PARA EL ESTUDIO DE COMUNIDADES MICROBIANAS COMPLEJAS
 CURSO TEÓRICO-PRÁCTICO DE MÉTODOS METAGENÓMICOS PARA EL ESTUDIO DE COMUNIDADES MICROBIANAS COMPLEJAS Universidad Pablo de Olavide, Sevilla 2-6 Febrero 2009 1 Introducción Vivimos en un mundo rodeado de
CURSO TEÓRICO-PRÁCTICO DE MÉTODOS METAGENÓMICOS PARA EL ESTUDIO DE COMUNIDADES MICROBIANAS COMPLEJAS Universidad Pablo de Olavide, Sevilla 2-6 Febrero 2009 1 Introducción Vivimos en un mundo rodeado de
Unificando las hipótesis de los daños crónicos por Hiperglicemia
 Unificando las hipótesis de los daños crónicos por Hiperglicemia Mecanismos moleculares del daño vascular por diabetes: Incremento de la vía del polioles Acumulación de Productos Finales de la Glicosilación
Unificando las hipótesis de los daños crónicos por Hiperglicemia Mecanismos moleculares del daño vascular por diabetes: Incremento de la vía del polioles Acumulación de Productos Finales de la Glicosilación
