UNIVERSIDAD DE LA LAGUNA OPEN WARE COURSE HERRAMIENTAS DE GENÉTICA MICROBIANA TÉCNICAS BÁSICAS DE BIOLOGÍA MOLECULAR
|
|
|
- Purificación García Montes
- hace 7 años
- Vistas:
Transcripción
1 UNIVERSIDAD DE LA LAGUNA OPEN WARE COURSE HERRAMIENTAS DE GENÉTICA MICROBIANA TÉCNICAS BÁSICAS DE BIOLOGÍA MOLECULAR María Antonieta Quispe Ricalde.
2 INDICE Páginas Marcaje de ADN. Electroforesis de ADN en gel de agarosa. Aislamiento y purificación de fragmentos de ADN a partir de geles de agarosa. Cuantificación de la sonda. Inmovilización de ácidos nucleicos y Southern-blot Electroforesis de proteínas en geles de poliacrilamida. Tinción de proteínas en geles de poliacrilamida. Transferencia de proteínas a membranas de nitrocelulosa desde SDS- PAGE. Western-blot
3 MARCAJE DE LA MOLÉCULA DE ADN. En la actualidad existen diversos compuestos descritos que se tienen la capacidad de unirse al ADN, los mismos que son utilizados para la visualización del ADN y los podemos dividir en: Marcaje enzimático. Marcaje fluorescente. Marcaje radiactivo. En esta asignatura vamos a desarrollar el marcaje enzimático, en el cual la molécula de ADN se marca mediante la incorporación de deoxiuridintrifosfato (dutp) unido a digoxigenina. El dutp se une a la digoxigenina con el hapteno eteroide (DIG-11) por medio de un espaciador. Una vez que el ADN objetivo incorpora la molécula dutp- DIG11, es posible detectarla mediante ensayos inmuno-enzimáticos usando un anticuerpo conjugado (anti-digoxigenina conjugada con fosfatasa alcalina (anti-dig- AP) y acoplado a una reacción colorimétrica utilizando 5 bromo-4 clor-3 indolil fosfato (BCIP) y sal azul de nitrotetrazolio (NBT). Tenemos dos métodos de marcaje utilizando la molécula dutp-dig11, y son seguidamente descritos a detalle. Método de marcaje al azar.- El método de marcaje al azar del ADN se realiza con la enzima Klenow de E. coli. Esta enzima tiene actividad polimerasa 5-3 y actividad endonucleasa 3-5. En un sistema de reacción en presencia de dutp-dig11, la cadena molde de ADN es hidrolizada por la actividad endonucleasa 3-5 y simultáneamente la enzima sintetiza una nueva cadena dada su actividad polimerasa 5-3 permitiendo la incorporación de los nucléotidos marcados. La reacción se puede realizar con cantidades variables de ADN, que oscila entre 10 ng y 3 ug por prueba. La reacción de marcaje depende de la actividad enzimática de la enzima Klenow, sin embargo la reacción suele ser rápida (1 hora) y se traduce en la incorporación de digoxigenina cada nucleótidos en el ADN recién sintetizado. Esta densidad de haptenos en el ADN determina una sensibilidad máxima en la reacción de detección. Según el molde de ADN, el tamaño del fragmento marcado variará entre 200 a 2000 pares de bases. Método de marcaje por PCR.- Un segundo método que tiene la misma eficiencia y es más sencillo para quieres suelen utilizarlo es la Reacción en Cadena de la Polimerasa 3
4 (PCR). En la reacción de PCR se combinan 5 nucleótidos, uno de ellos es el nucleótido Dig-dUTP a una concentración 0.35X veces que los otros cuatro nucleótidos. Esta modificación del método se puede utilizar cuando se tienen los cebadores que flanqueen la región que queremos utilizar como sonda y por otro lado cuando estamos seguros que el producto de amplificación corresponde a la región de ADN que deseamos marcar. Soluciones: a) Mezcla deoxinucleótidos (dntp) de marcaje (10X): dctp 1mM, dgtp 1mM, dttp 0,65mM, Dig-dUTP 0,35mM. b) Tampón de PCR 5X. c) Solución de MgCl2 25 mm. d) Solución de cebadores de extensión a 100pmol/ul. e) Agua pura grado miliq o bidestilada. f) Enzima Taq Polimerasa 5U/ul. Procedimiento: a) Para evitar cualquier problema de contaminación, el laboratorio debe de tener un área exclusiva para realizar la reacción de PCR, donde se mezclen todas las soluciones excepto el ADN, esta zona se denomina pre-pcr. El ADN se añade en una segunda zona, que puede ser la misma mesa de trabajo. b) Realizar la mezcla de reacción de la PCR con la mezcla de nucleótidos de marcaje, de acuerdo a la siguiente tabla: Reactivo Concentración stock Concentración final Volumen por 1 reacción Volumen por X reacciones Agua bd - - Tampón PCR 5X 1X MgCl 2 25 mm 1,5 mm dntps DIG 1 mm 100 um Cebador 1 10 pmol/ul 15 pmol Cebador 2 10 pmol/ul 15 pmol ADN 2-10 ng/ul 4-20 ng Volumen total 20 ul 4
5 c) Una vez realizada mezcla y añadido el ADN, la reacción se lleva a cabo en el termociclador. El tiempo de reacción puede variar desde 1 hora hasta 3 horas, el tiempo de reacción depende de la enzima utilizada y del tamaño del ADN que se quiere amplificar. 5
6 ELECTROFORESIS DE ADN EN GELES DE AGAROSA. La electroforesis en geles de agarosa u otro polímero son procedimientos básicos analíticos para caracterizar macromoléculas, tienen alta resolución, son fáciles de realizar y el costo es relativamente accesible. La agarosa es un polímero lineal se forma por la repetición de unidades de beta-1-galactosil-alfa-1-4 3,6 anhidrogalactosa, de gran longitud, dando lugar a una estructura rígida que posee unos poros, cuyo tamaño depende de la concentración del azúcar. Los ácidos nucleicos se pueden separar en estos medios semisólidos, la agarosa en particular permite una mayor resolución para las macromoléculas de gran tamaño, y por lo tanto se utilizan principalmente para ácidos nucleicos. La capacidad de resolución de un gel de agarosa va desde los 50 pb hasta varios Kb. Desde el punto de vista instrumental también la elaboración de los geles de agarosa son accesibles porque actualmente en el mercado están disponibles diversos modelos de cámaras electroforéticas de tamaños variados. La electroforesis de ácidos nucleicos en geles de agarosa, es el método rutinario empleado para separar y purificar fragmentos de ADN y ARN. En una electroforesis en gel de agarosa, los ácidos nucleicos debido a su carga neta negativa, se moverán hacia el ánodo y su movilidad depende fundamentalmente de los siguientes aspectos: a) Tamaño de ácido nucleico. b) Porosidad del gel (% de agarosa o % de poliacrilamida). c) Conformación estructural del ácido nucleico. d) Corriente eléctrica aplicada a la electroforesis. e) Composición de bases y temperatura. El peso molecular de ácidos nucleicos se puede determinar, debido a la linealidad de la relación empírica: logaritmo decimal del peso molecular Vs movilidad, siempre que las macromoléculas tengan una relación carga/masa constante. Así, se pueden medir los pesos moleculares de las macromoléculas usando patrones de peso molecular conocido. Existe una relación lineal entre el logaritmo decimal de la movilidad electroforética del ADN y la porosidad del gel. Como referencia para una buena separación de las moléculas de ADN, se puede seguir la siguiente tabla: 6
7 Concentración de agarosa Intervalo de separación 0.3% kb 0.7% kb 1.2% kb 1.5% kb 2.0% kb El ADN de doble cadena migra a una velocidad inversamente proporcional a su peso molecular, pero si tienen diferente estructura secundaria migran a diferentes velocidades, por ejemplo, el ADN de plásmido superenrollado avanza más rápido que el ADN lineal y éste más que el ADN circular relajado. La migración se puede seguir por la adición de colorantes: azul de bromofenol (BPB) y/o xilenocial (XC). Preparación de geles de agarosa. Soluciones a) Tampón TAE 50X (ph: 8.1): Reactivo cantidad Reactivo cantidad Tris base 242 gr. EDTA 0,5M 100 ml Ácido acético 57,1 ml Agua bidestilada c.s.p 1000 ml b) Loading buffer (tampón de carga): Reactivo cantidad Xilenocianol 0.25% Azul de bromofenol 0.25% Glicerol 30% O ficol en lugar de glicerol 5 a 15% c) Bromuro de etidio (BrEt): Disolver 10 mg de BrEt en 1ml de agua. El BrEt es un agente que forma planos intercalantes entre las bases de los ácidos nucleicos confiriendo fluorescencia al ser iluminados con luz ultravioleta. Es mutagénico y por tanto cancerígeno, debe manejarse con guantes. 7
8 El bromuro de etidio es un componente altamente tóxico y su uso está siendo reemplazado por otros agentes que de igual modo se unen al ADN pero que son menos tóxicos. Algunos ejemplos son: realsafe de la marca Realsafe. Procedimiento: 1. Pesar la cantidad adecuada de agarosa de acuerdo con la reticulación deseada y resuspenderla en el volumen de tampón TAE 1X o TBE 1X. Para disolverla se puede usar varios sistemas: a) Mediante horno microondas. b) Mediante autoclave de laboratorio. c) Utilizar una placa calefactora. d) Calentar a la llama, agitando de vez en cuando. 2. Mientras se disuelve la agarosa, preparar el molde, y poner un peine adecuado. 3. Enfriar la agarosa hasta unos C, al aire, y añadir bromuro de etidio a una concentración final de 1ug/ml (a partir de una solución de 10mg/ml). 4. Verter la agarosa en el molde con cuidado. Si aparecen burbujas, se eliminan con una pipeta pasteur o con una punta de pipeta. Dejar solidificar la agarosa durante al menos 30 minutos. Retirar el peine y colocar el gel en la cubeta de electroforesis. Adicionar suficiente tampón (TBE o TAE), para cubrir el gel (aproximadamente 1mm). 5. Añadir a las muestras de ADN a separar el tampón de electroforesis (6X). La electroforesis se realiza con los geles totalmente sumergidos, lo cual es útil para electroforesis rápidas y un paso homogéneo de electricidad a través del gel. 6. La visualización de los ácidos nucleicos se realiza con luz utravioleta a una longitud de onda de 360 ηm. 8
9 AISLAMIENTO Y PURIFICACIÓN DE FRAGMENTOS DE ADN A PARTIR DE GELES DE AGAROSA. Para recuperar y purificar ADN de geles de agarosa se utilizan columnas de silica, este material y sus modificaciones son ampliamente utilizados. Se suelen purificar fragmentos pequeños desde 40 pb hasta fragmentos de gran tamaño como 50 kb. Uno de los sistemas que utiliza columnas de silica gel, o silica gel en suspensión es el Kit Qiaex II. El procedimiento se basa en la afinidad del ADN por la resina cuando el medio que le rodea tiene altas condiciones de sal, de igual modo cuando las concentraciones de sal disminuyen ocurre el desprendimiento o separación de la molécula de ADN al medio soluble. Soluciones: a) QIAEX II suspensión. b) Tampón QX1 (con idicador de ph) c) Tampón PE (concentrado) Procedimiento: 1. Cortar la banda del gel de agarosa. Determinar el volumen aproximado del gel separado mediante pesada (1g = 1ml aprox.) y transferirlo a un tubo de centrífuga. 2. Añadir 3 volúmenes de tampón QX1 a 1 volumen de gel de ADN para fragmentos de 100 pb a 4 kb (si el fragmento de ADN es menor de 100 pb añadir 6 volúmenes QX1 y si es mayor a 4 kb añadir 3 volúmenes de tampón QX1 y 2 volúmenes de agua). 3. Resuspender el QIAEX II por vortex durante 30 segundos. Añadir 10 ul de esta para más o menos 2 ug de ADN (si hay entre 2 y 10 ug de ADN añadir 30 ul), e incubar a 50 C durante 10 minutos agitando cada 2 minutos hasta que se disuelva la agarosa. (Si el color de la mezcla es naranja o morado, añadir 10 ul de acetato sódico 3M ph 5.0 y mezclar. El color cambiará a amarillo. La incubación se aumentará unos 5 minutos más). La absorción de las partículas de ADN al QIAEX II es eficiente solamente a ph 7.5. El tampón QX1 tiene un indicador de ph cn el amarillo a ph 7.5, y el color naranja o morado tienen un ph más alcalino. 4. Centrifugar durante 30 segundos. Eliminar el sobrenadante. 9
10 5. Lavar el pellet con 500 ul de tampón QX1. Resuspender el pellet con vortex (para fragmentos mayores a 10 kb resuspender por inversión del tubo. El vortex podría producir rotura de fragmentos grandes de ADN). Centrifugar durante 30 segundos y eliminar todos los restos del sobrenadante con la pipeta para evitar contaminación. 6. Lavar el pellet 2 veces con 500 ul de tampón PE. Resuspender el pellet con vortex (para fragmentos mayores a 10 kb resuspender por inversión del tubo. El vortex podría producir rotura de fragmentos grandes de ADN). Centrifugar durante 30 segundos y eliminar todos los restos del sobrenadante con la pipeta para evitar contaminación. 7. Dejar secar durante 10 a 15 minutos hasta que el pellet se este poniendo blanco (el exceso de secado del pellet del QIAEX II podría disminuir la eficiencia de la elusión). 8. Eluir el ADN añadiendo 20 ul de 10mM Tris-HCl ph 8.0 y resuspender el pellet por vortex. Incubar el tubo a 50 C durante 5 minutos. Centrifugar unos 30 segundos. La eficiencia de la elusión es dependiente del ph. Si utilizamos agua para la elusión, debe de tener ph entre 7 y 8.5. La elusión también se puede hacer con TE pero el EDTA puede inhibir la reacción de las enzimas. 9. Centrifugar unos 30 segundos. Cuidadosamente (sin tocar la resina) recogemos el sobrenadante. Guardar el ADN a -20 C para evitar la degradación del ADN. 10. Se puede repetir los pasos 8 y 9 una vez más. WIZARD PCR Clean-up System (Promega). Este es otro método de purificación de fragmentos de amplificación por PCR de la casa Promega. Utiliza el mismo fundamente que el protocolo anterior, solo que la resina está unida a una pequeña columna, lo cual puede facilitar la extracción. Procedimiento: 1. Cortar la banda del gel de agarosa, poner en un eppendorf de 1.5 ml. 2. Añadir 10 ul de Membrana Binding Solution por cada 10 mg de gel. Mezclar por vortex e incubar a C hasta que el gel esté completamente disuelto (unos 10 minutos agitando suavemente cada 2 minutos). 3. Poner el SVMiniculum en un eppendorf. Añadir la mezcla del gel disuelto. Incubar 1 minuto a temperatura ambiente. 4. Centrifugar a rpm durante 1 minuto. Decantar la solución y reinsertar la columna en el eppendorf. 10
11 5. Añadir 700 ul de solución de lavado (diluida previamente con etanol al 95%). Centrifugar a rpm durante 1 minuto. Decantar la solución y reinsertar la columna en el eppendorf. 6. Añadir 500ul de solución de lavado (diluida previamente con etanol al 95%). Centrifugar a rpm durante 1 minuto. Decantar la solución y reinsertar la columna en el eppendorf. 7. Centrifugar 1 minuto para eliminar el etanol. 8. Transferir la columna a un eppendorf nuevo. 9. Añadir 20 ul de agua libre de nucleasas (añadir en el centro de la columna).incubar 1 minuto a temperatura ambiente. Centrifugar a rpm durante 1 minuto. 10. Decantar la minicolumna y guardar el ADN a 4 C ó a -20 C. 11
12 CUANTIFICACIÓN DE LA SONDA. La cantidad de sonda recomendada para hibridaciones es de 20 ng/ml. Por lo tanto es conveniente cuantificar el marcaje antes de proceder a la hibridación con la sonda. Para realizar este procedimiento, utilizaremos el kit de marcaje del ADN con dioxigenina (Roche Diagnostic). Soluciones: a) Control de ADN marcado. b) Control de ADN sin marcar. c) Tampón de disolución del ADN. d) Solución de pre-hibridación: SSC 5X. Agente de bloqueo 1% (p/v). N-laurilsarcosina, sal sódica 0.1% (p/v) SDS 0,02% (p/v) El agente de bloqueo no se disuelve con rapidez, de modo que hay que preparar la solución 1 hora antes, disolviendo a C en tampón 1. Es conveniente autoclavar y tratar de mantener su esterilidad. e) Solución de ácido maleico (Tampón 1): Ácido maleico 0.1 M NaCl 0,15 M Ajustar el ph a 7.5 con NaOH sólido. f) Solución bloqueadora 10X: Agente bloqueante 10%(p/v) en tampón 1. Disolver el agente bloqueante en agitación constante a 65 C o calentar en microondas, autoclavar y guardar a 4 C. La solución permanece opaca. g) Solución bloqueadora 1X (Tampón 2): Es la solución bloqueadora diluida a 1X en tampón 1. h) Solución 3: Solución de detección: Tris-HCl 0,1 M NaCl 0,1 M MgCl2 50 mm ph 9.5 (20 C). i) Solución 4: Solución de substrato: 12
13 Preparar siempre antes de utilizar: añadir 200 ul de solución de NBT/BCIP (Roche) a 10 ml de solución 3. Procedimiento: a) Realizar diluciones seriadas del control de ADN marcado (de 10 fg/µl a 10 ρg/µl). b) Realizar diluciones de la sonda elaborada. Las diluciones pueden ser 1/5, 1/10 y 1/100. c) Colocar 2µL de ADN tanto del control como de la muestra problema, sobre una membrana de nylon cargada positivamente (Roche Diagnostic). d) Fijar el ADN a 80 C durante 20 minutos, o fijar con luz UV únicamente 3 minutos. DETECCIÓN DE LAS SONDAS MARCADAS CON DIGOXIGENINA. La detección de la presencia de la sonda que lleva unida la digoxigenina, se realiza mediante reconocimiento inmunológico. Procedimiento: a) La membrana de nylon se equilibra por 3 minutos en tampón 1 [ácido maléico 0.1M, NaCl 0.15M, ph 7.5] y Tween-20 al 0.3% (v/v). b) Bloquear con el tampón 2 (Agente bloqueante 1% en tampón 1) durante 30 minutos. c) Incubar durante 1 hora a temperatura ambiente con el anticuerpo anti-dig-fab (Roche Diagnostic) a una dilución 1/5000 en el tampón 2. d) Lavar por 3 veces con el tampón 1 durante 15 minutos por vez. e) Equilibrar con el tampón de reacción de la enzima fosfatasa alcalina [Tris HCl 0.1M ph 8.0, NaCl 0.1 MgCl M, ph 9.5] por 5 minutos. f) Revelar la membrana utilizando el sustrato para la enzima Nitro Blue Tetrazolium Salt/X-fosfato (5-bromo-cloro-3-indolil-fosfato). NBT/BCIP (Roche Diagnostic) en el mismo tampón de reacción de la fosfatasa alcalina. 13
14 INMOVILIZACIÓN DE ÁCIDOS NUCLEICOS. La transferencia de fragmentos de ADN separados en geles de electroforesis a una matriz inmovilizante se ha convertido en un proceso rutinario que permite una detección sensible y específica de macromoléculas de interés biológico. Después de la transferencia, se obtienen réplicas inmovilizadas exactas de las macromoléculas separadas, y éstas pueden ser detectadas por hibridación con sondas específicas. La técnica básica de transferencia de ADN, fue descrita por Southern en 1975 y consiste en la transferencia por capilaridad del ADN, desde geles de agarosa a membranas de nitrocelulosa, nylon o PVDF. Las moléculas de la matriz de un gel son relativamente inaccesibles a sondas como ADN complementario o antígenos, ya que son demasiado grandes para difundir a través del gel y unirse específicamente con su homólogo. Así la primera ventaja de la transferencia es la accesibilidad; la transferencia a la superficie de una membrana hace posible análisis que de otro modo serían difíciles de ralizar. Una segunda ventaja importante es que las moléculas transferidas se retienen en la membrana de manera que no hay pérdida de resolución y además, en muchos casos la actividad biológica permanece incluso almacenando las membranas transferidas durante cierto tiempo. Otra ventaja es que las membranas son relativamente fuertes y fáciles de manejar, en comparación con los geles que son más débiles. TRANSFERENCIA E INMOVILIZACIÓN DE ADN A SOPORTES SÓLIDOS: SOUTHERN BLOT. Una vez concluida la electroforesis, se recomienda que se degrade parcialmente el ADN para aumentar la eficiencia de la transferencia. Se puede irradiar con luz UV en presencia de bromuro de etidio (procedimiento habitual empleado para ver el estado del ADN separado por electroforesis y para obtención de una foto del gel). Alternativamente se trata con ácido clorhidrico diluido para depurinarlo. El enlace fosfodiéster de sitios donde se ha perdido la base es muy sensible al álcali, por lo que el ADN se degrada al desnaturalizarlo. Soluciones: a) Solución de transferencia de ADN: NaOH 0,4 N 14
15 b) Tampón SSC 20X: NaCl 3 M Citrato de sodio 0,3 M. ph: 7,2 c) Solución de bloqueo 10X: Agente bloqueante (Roche) al 10 % en agua tampón 1. d) Tampón 1: Ácido maléico 0,1 M NaCl 0,15 M ph: 7,.5 e) Tampón 2: Solución de bloqueo diluida 1/10 en tampón 1. f) Tampón de pre-hibridación: SSC 5X Solución de bloqueo 1 % (p/v) N-lauril-sarcosina 0,1 % (p/v) SDS 0,2 % (p/v) g) Tampón de hibridación: Tampón de pre-hibridación Sonda marcada ηg. h) Tampón de reacción de la Fosfatasa Alcalina: Tris HCl 0,1 M ph 8,0 NaCl 0,1M MgCl2 0,05M ph 9,5 Procedimiento: a) Depurinar parcialmente al ADN con HCl 0.2M por 10 minutos. b) Enjuagar con agua miliq. c) Sumergir el gel en una solución de NaCl 1.5M y NaOH 0.5M por 15 minutos en agitación constante, en este paso se hidroliza el enlace fosfodiester de la hebra de ADN en los sitios depurinados. d) Equilibrar con la solución de transferencia (NaOH 0.4 M) por minutos. e) Mientras se hacen las incubaciones tener preparado 2 tiras de papel whatman 3MM, un trozo de membrana de nylon del mismo tamaño que el gel y 5 trozos de papel Whatman con un tamaño exacto del gel. f) Realizar la transferencia alcalina de forma descendente con NaOH 0.4M por dos horas. 15
16 g) Finalizada la transferencia, lavar la membrana con el tampón SSC 2X durante 5 minutos. h) Fijar el ADN a la membrana mediante exposición a luz UV en el transiluminador High Performance Ultraviolet Transilluminator (Ultra-violet products) durante 3 minutos, asegurándose que, la cara de la membrana donde ha sido transferido al ADN esté en contacto con la luz UV. i) Las membranas de nylon que llevan el ADN transferido, pre-hidridarlas en tampón de pre-hidridación por 1 hora a 68º C con agitación suave, en un horno de hibridación. j) Disolver la sonda marcada en tampón de pre-hidridación e incubar durante 10 minutos a 95º C para su desnaturalización, con esta solución se reemplazará el tampón de prehibridación e hibridar la membrana de 8 a 12 horas a 68 C. k) Realizada la hibridación, lavar la membrana 2 veces con la solución SSC 2X/SDS 0,1% durante 5 minutos a temperatura ambiente cada una. l) Realizar dos lavados de mayor astringencia, con la solución SSC 0,1X/SDS 0.1% dos veces durante 5 minutos a 68º C. DETECCIÓN INMUNOLÓGICA. La detección de la hibridación de la sonda, se realizó por el reconocimiento inmunológico de la digoxigenina. Todos los pasos del protocolo se realizan a temperatura ambiente con agitación suave. Procedimiento: a) Lavar las membranas de nylon durante 2 minutos con 20ml de tampón 1. b) Incubar durante 30 minutos con el tampón 2. c) Incubar por 1 hora con el anticuerpo anti-dig conjugada con fosfatasa alcalina en el tampón 2 a una dilución 1: d) Lavar los filtros durante 15 minutos con tampón 1 por 3 veces. e) Equilibrar las membranas con el tampón de reacción de la enzima por 5 minutos. f) Finalmente se incuba la membrana con la solución de substratos cromogénicos NBT/BCIP en el tampón de reacción de fosfatasa alcalina, hasta la aparición de un precipitado de color violeta. 16
17 ELECTROFORESIS DE PROTEÍNAS EN GELES DE POLIACRILAMIDA. Introducción y técnica básica. La electroforesis de proteínas en geles con una matriz de poliacrilamida, comúnmente denominada electroforesis en poliacrilamida o PAGE (polyacrilamide gel electroforesis) es una de las técnicas más usadas para caracterizar mezclas complejas de proteínas y separar estas por tamaño. La electroforesis en poliacrilamida es un método conveniente, rápido y económico a nivel de muestra pues se requieren sólo cantidades del orden de microgramos de proteína. Las proteínas presentan una carga eléctrica neta si se encuentran en un medio que tenga un ph diferente al de su punto isoeléctrico y por eso tienen la propiedad de desplazarse cuando se someten a un campo eléctrico. La velocidad de migración es proporcional a la relación entre las cargas de la proteína y su masa. Cuanto mayor carga por unidad de masa más rápida será la migración. El gel de poliacrilamida es un enrejado tridimensional de fibras lineales de acrilamida, unidas por puntos de cruce consistentes en moléculas de bisacrilamida. El tamaño del poro del gel depende de la concentración de acrilamida y bisacrilamida utilizada. Una ventaja importante de los geles de poliacrilamida es que son químicamente inertes, transparentes y estables en un amplio rango de phs, temperatura y fuerza iónica. La velocidad de polimerización viene determinada por la concentración de persulfato de amonio (catalizador) y TEMED (iniciador). La porosidad del gel determina las proporciones relativas de acrilamida y bisacrilamida, siendo menores poro cuanta más bisacrilamida y arcrilamida se use. El porcentaje total de acrilamida/bisacrilamida determina el rango de separación del gel. Habitualmente los geles se denominan en función del % de arcrilamida/bisacrilamida que contiene. Así, la mayoría de las proteínas se separan bien en el rango de 5 a 10%. Un menor porcentaje (mayor tamaño de poro) es mejor para separar proteínas de gran tamaño. La electroforesis en geles de arcrilamida se puede realizar empleando sistemas de uno o más tampones, en estos casos se habla de sistemas tampón continuos o discontinuos. En los sistemas discontinuos el primer tampón asegura la migración de todas las proteínas en el frente de migración, provocándose la acumulación de todas las que se han cargado en el pocillo. La separación realmente comienza a partir del momento en que el frente de migración alcanza la frontera del segundo tampón. El primer gel (compactación 17
18 stacking ) es de mayor poro (menor porcentaje de arcrilamida + bisacrilamida) y tiene un ph más ácido que el segundo gel que es el que realmente separa las proteínas, este es denominado gel de resolución. Este sistema es especialmente adecuado para analizar muestras diluidas sin perder resolución. SDS-PAGE, significa que la electroforesis en geles de poliacrilamida se realiza en presencia de SDS. Fue descrito por Laemmli en Se trata de un tipo de electroforesis desnaturalizante en la que las muestras se desnaturalizan por calor en presencia de agentes desnaturalizantes (beta-mercaptoetanol, que destruye los puentes disulfuro, SDS que desnaturaliza y recubre a la proteína), y se separan como cadenas polipeptídicas aisladas. En general se emplean sistemas de dos tampones (discontinuos). Este sistema permite la separación de volúmenes relativamente grandes de muestra sin pérdida de resolución. SDS es un detergente de acción desnaturalizante que se une a las cadenas polipeptídicas desnaturalizadas con una relación de 1.4 g de SDS por g de proteína, uniéndose aproximadamente una molécula de SDS por cada dos aminoácidos de la cadena. Esta unión masiva de moléculas de SDS bloquea la carga propia d la molécula de proteína y le confiere al complejo una carga neta negativa proporcional a su masa, haciendo que todas las proteínas complejas con SDS viajen hacia el ánodo. La separación de los complejos SDS-proteína es proporcional sólo a la masa de la proteína pues todas tienen la misma carga por unidad de masa. Se puede entonces determinar el peso molecular aparente de cualquier proteína por comparación con un patrón de proteínas de pesos moleculares conocidos. Las movilidades de las proteínas en los geles de SDS-PAGE son funciones lineales de logaritmo de su peso molecular. 18
19 Preparación de los geles: Soluciones: Tampón de muestra (Laemly): Reactivo Volumen /peso Concentración final Tris 0,5M ph 6.8 2,5 ml 50 mm SDS al 10% 10 ml 2% Glicerol 5 ml 10% Azul de bromofenol 0.05 g 0,1% Mezclar los componentes Añadir DTT ( g/ml de laemly Tampón de muestra (Laemly) 2X: Reactivo Volumen /peso Concentración final Tris 0,5M ph mm SDS al 10% 2 % Glicerol 20 % Azul de bromofenol 0,004 % β-mercaptoetanol 10 % Mezclar los componentes Tampón de electroforesis 10X Reactivo Volumen /peso Concentración final Tris base g 0.25 M Glicina g 2 M SDS 1 gr 1% Completar con 100 ml de agua bidestilada Acrilamida/bisacrilamida 30%: Reactivo Volumen /peso Concentración final Acrilamida 29.2 g 29.2 % Bisacrilamida 0.8 g 0.8 % Mezcla total 30 gr 30 % Disolver con 100 ml de agua bidestilada. Filtrar empleando filtros de 0.45 um y cubrir 19
20 el recipiente con papel aluminio. Tampón Lower-Tris: Reactivo Volumen /peso Concentración final Tris- HCl 18.1 g SDS 0.4 gr Disolver con 100 ml de agua bidestilada. Ajustar el ph a 8.8 Stacking gel: Reactivo Volumen /peso Concentración final Acrilamida /Bisacrilamida 2 ml Tris-HCl 0.5M ph ml SDS 10% 200 ul Agua bd 12,4 ml Completar con 100 ml de agua bidestilada Persulfato de amonio (APS 10%): Pesar 0.01 g de persulfato de monónio y diluir en un volumen final de 1 ml con agua bidestilada. Este reactivo en polvo es altamente hidrofílico, por lo tanto, es importante mantener el envase herméticamente cerrado. Esta preparación debe ser de 1 semana de antigüedad como máximo. Preparación de los geles: Lower Gel o gel de resolución: 8% 10% 12% 14% 16% 20% Lower-Tris 1.25 ml 1.25 ml 1 ml 1 ml 1.25 ml 1 ml Acril/Bisacril 30% 1.33 ml 1.66 ml 1.66 ml 1.86 ml 2.66 ml 2.66 ml APS 10% 17 ul 17 ul 17 ul 17 ul 17 ul 17 ul Temed 3 ul 3 ul 3 ul 3 ul 3 ul 3 ul Agua 1.7 ml 1.75 ml 1.4 ml 1.14 ml 1.09 ml 0.33 ml 20
21 Stacking Gel o gel de apilamiento: 8% 10% 12% 14% 16% 20% Stacking 2 ml 2 ml 2 ml 2 ml 2 ml 2 ml APS 10% 40 ul 40 ul 40 ul 40 ul 40 ul 40 ul Temed 3 ul 3 ul 3 ul 3 ul 3 ul 3 ul Preparación de la muestra biológica. La preparación de la proteína consiste en un proceso en el cual se rompen enlaces peptídicos y puetes disulfuro gracias a la acción de detergentes iónicos, reactivos reductores y altas temperaturas, los que en conjunto consiguen desnaturalizar la proteína hasta su estructura primaria. Procedimiento. a) Mezclar la proteína con el tampón de muestra concentrada cuatro veces (4X) en proporción 1:1. b) Hervir la muestra a 100 C durante 5 minutos. c) Finalizado el procedimiento, centrifugar las muestras durante 30 segundos a rpm. Mantenerlas en hielo hasta el proceso de electroforesis. Preparación del gel de resolución: a) Realizar la preparación del gel de resolución a una concentración de acuerdo a las necesidades, mezclando los componentes en el orden que se indica en la tabla (Staking gel o gel de resolución). b) Aproximadamente 5 ml de solución deberán ser preparados en caso de trabajar con geles pequeños de 0.75 mm de grosor. c) Una vez finalizada la preparación de la solución, añadir los catalizadores APS y TEMED uno a uno, tratando de mezclar cada reactivo de manera uniforme en todo el volumen. d) Agregar un volumen de la solución de poliacrilamida entre las placas de vidrio. e) Posteriormente, añadir una capa de isopropanol sobre las solución de poliacrilamida para evitar el contacto de moléculas de oxígeno que retarden la polimerización. f) Dejar polimerizar a temperatura ambiente durante 15 a 30 minutos, usando como control de polimerización la solución de poliacrilamida sobrante. 21
22 g) Una vez formado el gel, descartar la capa de isopropanol y lavar la parte superior del gel varias veces con agua destilada hasta eliminar completamente los restos de arcrilamida no polimerizada y de isopropanol. Preparación del de apilamiento: a) Mezclar los componentes del gel tal como lo indica la tabla: Gel de stacking o gel de apilamiento, y agitar moderadamente con el fin de que el TEMED y el APS se distribuyan de manera uniforme. b) Agregar la poliacrilamida entre los vidrios de la cámara y sobre el gel de resolución ya polimerizado. Inmediatamente después, colocar el pine entre ambos vidrios. c) Dejar polimerizar la poliacrilamida a temperatura ambiente durante 15 a 20 minutos usando como control de polimerización la solución de poliacrilamida que sobró en el tubo. d) Una vez formado el gel, retirar el pine cuidadosamente y lavar todos los pocillos ya formados, con agua destilada. Electroforesis: Protocolo: a) Sumergir el sistema conteniendo los geles polimerizados en la cubeta de la cámara de electroforesis que contiene el tampón de electroforesis 1X. Dicha cubeta lleva dos electrodos: uno positivo y otro negativo que serán conectados a una fuente de poder. Hay que asegurarse que los pocillos del gel estén totalmente cubiertos de tampón. b) Cargar la muestra en los pocillos evitando la contaminación del pocillo contiguo. c) Cargar el marcador de peso molecular de proteínas. Dicho marcador debe ser sometido a los mismos tratamientos que el de la proteína, excepto cuando se trate de un marcador previamente teñido en el que se obviará el uso del tampón de muestra. d) Una vez finalizada la colocación de las proteínas en cada pocillo del gel, tapar el tanque y conectar los cables correspondientes de los electrodos en la fuente de poder. e) Encender la fuente y seleccionar el voltaje y amperaje correspondientes. Se puede poner desde 120 V a 180 V dependiendo de cómo avance la electroforesis. Si el frente se deforma debe de bajarse el voltaje para que se iguale. f) El valor del amperaje dependerá de la fuerza iónica del tampón. En el caso de trabajar con tampón Tris-glicina 1X el valor recomendado es de 25 a 30 ma. También se puede realizar la electroforesis con ma cuando se trata de el sistema Mini-Protean. 22
23 g) Realizar al electroforsis hasta que el frente (visualizado como una línea azul por el colorante del tampón) haya llegado a los límites de la parte inferior del gel. h) Finalizada la electroforesis proceder al desmontaje del equipo. i) Separar el gel de empacamiento del gel de resolución realizando un corte transversal y hacer una pequeña muesca en una de las esquinas del gel, a fin de que sirva de orientación para ubicar el orden en que fueron cargadas las muestras. j) Para desprender el gel del vidrio remojarlo nuevamente con tampón de electroforesis o agua destilada y usar uno de los espaciadores a manera de espátula para elevarlo por las puntas. Coger cuidadosamente el gel y luego sumergirlo en el colorante o seguir el tratamiento para realizar la transferencia de proteínas a soporte sólido. k) El tampón de electroforesis puede ser reciclado varias veces. Como marcadores de peso molecular más utilizados: - High Molecular weight Standard Mixture for SDS gel electroforesis SDS 6H: 205, 116, 97.4, 66, 45, 29 kda. (Sigma) - Dalton Mark VII-L for SDS Electroforesis SDS-7: 66, 45, 36, 29, 24, 20, 14.2 kda (Sigma). - Kaleidoscope Prestained Standars: 218, 126, 90, 43,5, 33,90 17,40, 7,6 kda (Bio- Rad). 23
24 TINCIÓN DE PROTEÍNAS EN GELES DE POLIACRILAMIDA. Una vez realizada la electroforesis, los geles pueden ser teñidos con colorante azul brillante de Coomassie R-250 al 0.2% (p/v), en solución de metanol al 40% (v/v) y ácido acético al 10% (v/v), según el método descrito por Fairbanks y col (1971). El proceso de desteñido se llevo a cabo con lavados en solución decolorante [metanol 40% (v/v) y ácido acético10% (v/v)] hasta visualizar las bandas. Los geles se pueden conservar en ácido acético 5% (v/v) y/o se secar en papel celofán. Existen otros métodos para visualizar proteínas, sobretodo las que se encuentre en menor cantidad o visualizar la purificación de las mismas, uno de ellos es la coloración con nitrato de plata, con el sistema Silver stain plus (Bio-Rad) el cual utiliza el método descrito por Gottleib and Chavko (1997) modificado. Se recomienda seguir el procedimiento utilizado por el fabricante. 24
25 TRANSFERENCIA DE PROTEÍNAS A MEMBRANAS DE NITROCELULSA DESDE SDS-PAGE. Soluciones: Tampón de transferencia. Reactivo Concentración final Volumen /peso Tris base 25 mm 3.03 g Glicina 192 mm 14.4 g Metanol 15 % 150 ml SDS 10% 1 g Completar con 1000 ml de agua bidestilada Procedimiento: a) Una vez realizada la electroforesis, separar los vidrios, despegar el gel y humedecer en tampón de transferencia e introducirlo en una cubeta con abundante tampón para equilibrarlo durante 20 minutos. b) Cortar una pieza de membrana de nitrocelulosa, nylon o Inmobilon-P del tamaño del gel. Debe guardarse mucho cuidado de no tocar la membrana con los dedos (debe cogerse entre sus dos papeles de protección y utiizar guantes y pinzas). Cortamos también cuatro papeles 3MM del tamaño de el casete de transferencia. c) Equilibrar la membrana en 5 a 10 ml en tampón de electrotransferencia. d) Montamos el casete de transferencia de la siguiente manera: Sobre una superficie plana, como puede ser la mesa de trabajo, colocar dos papeles 3MM, uno sobre otro y sumergir en buffer de transferencia sin SDS. Para eliminar las posibles burbujas que impidan el paso de corriente, pasar sobre los papeles un rodillo (puede ser una pipeta pasteur). Sobre los papeles mojados colocar el gel a transferir, pasar el rodillo para eliminar burbujas, colocar sobre el gel la membrana equilibrada en buffer y sobre el pasar de nuevo el rodillo. Colocar sobre la membrana dos papeles 3MM que previamente se hayan sumergido en buffer y de nuevo pasar el rodillo. Colocar el sándwich entre las esponjillas mojadas en el buffer y todo el sistema dentro del casete de manera que el gel quede del mismo lado que la cara gris oscura del casete. e) Colocar el casete en la cubeta, poniendo la parte negra junto a la parte negra y ésta en la cubeta de transferencia poniendo a parte negra cerca del electrodo negativo (negro), 25
26 así queda la membrana más cerca del electrodo posito (rojo) y el gel cerca del negativo. Las proteínas cargadas negativamente debido al SDS migran hacia el polo positivo y quedan retenidas en la membrana). Añadir el tampón de transferencia. f) Aplicar corriente eléctrica a 70 V durante 2 horas. Se puede emplear otros voltajes y tiempos por ejemplo 100 V por 1 hora, 20 V durante toda la noche. Otra posibilidad es a 150 ma durante 2 horas o a 30 ma toda la noche. g) Desmontar la transferencia y colocar la membrana en una cubeta o recipiente de plástico para realizar el western-blot. Evitar que la membrana se seque pues se alteran sus propiedades. 26
27 WESTERN-BLOT Soluciones: Solución de lavado (TBS) Reactivo Concentración final Volumen /peso Tris base 20 mm NaCl 136 mm ClK 0.2 g Tween % BSA o leche desnatada 0.1 % Completar con 1000 ml de agua bidestilada Solución de bloqueo. Reactivo Concentración final Volumen /peso BSA o leche desnatada 5 % Preparar la solución de bloqueo en TBS. También se puede añadir 1% de suero de cabra. Solución de incubación de anticuerpos. Reactivo Concentración final Volumen /peso BSA o leche desnatada 1 % Tween % Preparar la solución de incubación de anticuerpos en TBS. Tampón de reacción de fosfatasa alcalina. Reactivo Concentración final Volumen /peso Tris- HCl ph mm MgCl2 5 mm NaCl 100 mm Solución de substrato: Disolver 200ul de solución NBT/BCIP en 10 ml de tampón de reacción de fosfatasa alcalina. 27
28 Procedimiento: a) Incubar la membrana en solución de bloqueo de 1 a 5 horas con agitación suave a temperatura ambiente, o toda la noche a 4 C. b) Lavar la membrana 3 veces durante 10 minutos con solución de lavado y con agitación suave. c) Incubar la membrana con el primer anticuerpo en solución de incubación de anticuerpos. En caso de anticuerpos producidos en conejo, usar dilución 1:200. Preparar 0.1 ml de solución por centímetro cuadrado de membrana. Se incuba durante 2 horas a temperatura ambiente y con agitación suave o, alternativamente, toda la noche a 4 C. d) Lavar la membrana 3 veces durante 10 minutos con solución de lavado y con agitación suave. e) Incubar la membrana con el segundo anticuerpo en solución de incubación de anticuerpos. El segundo anticuerpo es comercial u es un anti IgG conjugado con la enzima fosfatasa alcalina. Se emplea a una dilución 1:1000 o según las indicaciones del fabricante. Se prepara 0.1 ml por centímetro cuadrado de membrana e incuba durante 2 horas (mínimo 1 h) con agitación suave a temperatura ambiente. f) Lavar la membrana 3 veces durante 10 minutos con solución de lavado y con agitación suave. g) Equilibrar la membrana durante 5 minutos en el tampón de reacción de la fosfatasa alcalina. h) Incubar la membrana con solución de revelado en oscuridad y sin agitación de 1-5 minutos o hasta que el color alcance la coloración deseada. Puede tardar en revelarse hasta 24 horas. i) Parar la reacción bañando la membrana en agua o se sumerge la membrana en una solución de EDTA 1mM. 28
Electroforesis de proteínas en geles de poliacrilamida
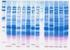 Electroforesis de proteínas en geles de poliacrilamida Electroforesis de proteínas en geles de poliacrilamida: Introducción y técnica básica La electroforesis de proteínas en geles con una matriz de poliacrilamida,
Electroforesis de proteínas en geles de poliacrilamida Electroforesis de proteínas en geles de poliacrilamida: Introducción y técnica básica La electroforesis de proteínas en geles con una matriz de poliacrilamida,
ELECTROFORESIS EN GEL DE POLIACRILAMIDA CON SDS SDS-PAGE
 Departamento de Biología de Sistemas Unidad Docente de Bioquímica y Biología Molecular ELECTROFORESIS EN GEL DE POLIACRILAMIDA CON SDS SDS-PAGE 1. FUNDAMENTO TEÓRICO La electroforesis es una técnica de
Departamento de Biología de Sistemas Unidad Docente de Bioquímica y Biología Molecular ELECTROFORESIS EN GEL DE POLIACRILAMIDA CON SDS SDS-PAGE 1. FUNDAMENTO TEÓRICO La electroforesis es una técnica de
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987)
 EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
ELECTROFORESIS EN GEL DE POLIACRILAMIDA CON SDS SDS-PAGE
 1. FUNDAMENTO TEÓRICO ELECTROFORESIS EN GEL DE POLIACRILAMIDA CON SDS SDS-PAGE Departamento de Bioquímica La electroforesis es una técnica de separación de moléculas cargadas por migración en un campo
1. FUNDAMENTO TEÓRICO ELECTROFORESIS EN GEL DE POLIACRILAMIDA CON SDS SDS-PAGE Departamento de Bioquímica La electroforesis es una técnica de separación de moléculas cargadas por migración en un campo
1. OBTENCIÓN DE ADN DE BACTERIAS PATÓGENAS
 1. OBTENCIÓN DE ADN DE BACTERIAS PATÓGENAS Existen diferentes métodos para la obtención de ADN bacteriano que varían en duración y complejidad del procedimiento en función de la calidad del ADN que queramos
1. OBTENCIÓN DE ADN DE BACTERIAS PATÓGENAS Existen diferentes métodos para la obtención de ADN bacteriano que varían en duración y complejidad del procedimiento en función de la calidad del ADN que queramos
4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar
 4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar - Agarosa de grado molecular - Agarosa de bajo punto de fusión
4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar - Agarosa de grado molecular - Agarosa de bajo punto de fusión
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 1 Virología y Micología Veterinaria Trabajo Práctico No. 4 ELECTROFORESIS La electroforesis es una técnica utilizada para separar grandes moléculas por tamaño a través de la aplicación de un campo eléctrico
1 Virología y Micología Veterinaria Trabajo Práctico No. 4 ELECTROFORESIS La electroforesis es una técnica utilizada para separar grandes moléculas por tamaño a través de la aplicación de un campo eléctrico
ELECTROFORESIS Q U Í M I C A B I O L Ó G I C A. D E P A R T A M E N T O D E B Q C A. F C N Y C S. U N P S J B
 ELECTROFORESIS Q U Í M I C A B I O L Ó G I C A. D E P A R T A M E N T O D E B Q C A. F C N Y C S. U N P S J B 2 0 1 7 Que es la electroforesis? Qué permite analizar? Que factores influyen? Separar y analizar
ELECTROFORESIS Q U Í M I C A B I O L Ó G I C A. D E P A R T A M E N T O D E B Q C A. F C N Y C S. U N P S J B 2 0 1 7 Que es la electroforesis? Qué permite analizar? Que factores influyen? Separar y analizar
DEPARTAMENTO DE GENÉTICA UNIVERSIDAD DE GRANADA FUNDAMENTOS DE BIOLOGÍA APLICADA I 4º CURSO DE BIOLOGÍA
 DEPARTAMENTO DE GENÉTICA UNIVERSIDAD DE GRANADA FUNDAMENTOS DE BIOLOGÍA APLICADA I 4º CURSO DE BIOLOGÍA MÓDULO DE GENÉTICA TÉCNICAS MOLECULARES DE ANÁLISIS GENÉTICO AISLAMIENTO Y CLONACIÓN DE ADN SATÉLITE
DEPARTAMENTO DE GENÉTICA UNIVERSIDAD DE GRANADA FUNDAMENTOS DE BIOLOGÍA APLICADA I 4º CURSO DE BIOLOGÍA MÓDULO DE GENÉTICA TÉCNICAS MOLECULARES DE ANÁLISIS GENÉTICO AISLAMIENTO Y CLONACIÓN DE ADN SATÉLITE
PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS. Es un ácido capaz de formar sales con iones cargados
 Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Módulo 2 Biología Molecular Genética Molecular II Módulo 2. Clonación y Selección de moléculas recombinantes
 Módulo 2 Clonación y Selección de moléculas recombinantes El fragmento de ADN amplificado por PCR en el módulo anterior se introdujo en un vector pgem T Easy (Ver Anexo 2) utilizando una ligasa. En la
Módulo 2 Clonación y Selección de moléculas recombinantes El fragmento de ADN amplificado por PCR en el módulo anterior se introdujo en un vector pgem T Easy (Ver Anexo 2) utilizando una ligasa. En la
PRÁCTICA 9 ELECTROFORESIS EN GEL DE AGAROSA
 INTRODUCCIÓN PRÁCTICA 9 ELECTROFORESIS EN GEL DE AGAROSA La electroforesis es una técnica que permite separar moléculas cargadas eléctricamente, entre las cuales se encuentran los ácidos nucleicos (DNA
INTRODUCCIÓN PRÁCTICA 9 ELECTROFORESIS EN GEL DE AGAROSA La electroforesis es una técnica que permite separar moléculas cargadas eléctricamente, entre las cuales se encuentran los ácidos nucleicos (DNA
BIOLOGIA CELULAR Y MOLECULAR. LABORATORIO No 9: ELECTROFORESIS EN GELES DE POLIACRILAMIDA (SDS-PAGE)
 BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 9: ELECTROFORESIS EN GELES DE POLIACRILAMIDA (SDS-PAGE) 1. INTRODUCCIÓN Uno de los métodos que permite la purificación y separación de proteínas de un organismo
BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 9: ELECTROFORESIS EN GELES DE POLIACRILAMIDA (SDS-PAGE) 1. INTRODUCCIÓN Uno de los métodos que permite la purificación y separación de proteínas de un organismo
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
MONITOREO DE UN PROCESO DE PURIFICACIÓN DE PROTEÍNAS. Corrimiento electroforético de las muestras de la purificación de la lactato deshidrogenasa.
 MONITOREO DE UN PROCESO DE PURIFICACIÓN DE PROTEÍNAS Corrimiento electroforético de las muestras de la purificación de la lactato deshidrogenasa. Qué es la electroforesis? Es la técnica que permite la
MONITOREO DE UN PROCESO DE PURIFICACIÓN DE PROTEÍNAS Corrimiento electroforético de las muestras de la purificación de la lactato deshidrogenasa. Qué es la electroforesis? Es la técnica que permite la
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
PCR SIMULADA. Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO
 Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ensayos de restricción
 Ensayos de restricción Vector con inserto= Vector recombinante 6500pb Objetivos de ensayos de restricción de plásmidos Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa.
Ensayos de restricción Vector con inserto= Vector recombinante 6500pb Objetivos de ensayos de restricción de plásmidos Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa.
DANAGENE PLASMID MIDI/MAXI KIT
 REF.0702.4 25 Midis REF.0702.5 10 Maxis DANAGENE PLASMID MIDI/MAXI KIT 1. INTRODUCCION DANAGENE PLASMID Midi/Maxiprep Kit proporciona un método simple para purificar ADN plasmídico a partir de cultivos
REF.0702.4 25 Midis REF.0702.5 10 Maxis DANAGENE PLASMID MIDI/MAXI KIT 1. INTRODUCCION DANAGENE PLASMID Midi/Maxiprep Kit proporciona un método simple para purificar ADN plasmídico a partir de cultivos
Transferencia de material genético II. 1) Aislamiento de plásmidos Lisis alcalina
 Transferencia de material genético II 1) Aislamiento de plásmidos Lisis alcalina ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar que hay células transformantes de acuerdo
Transferencia de material genético II 1) Aislamiento de plásmidos Lisis alcalina ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar que hay células transformantes de acuerdo
PRÁCTICAS AMPLIACIÓN DE MICROBIOLOGÍA
 PRÁCTICAS AMPLIACIÓN DE MICROBIOLOGÍA Día 1º.- EXPLICACIÓN GENERAL. TRANSFORMACIONES: E. coli, Streptomyces Día 2º.- INOCULAR MINIPREPS (8:45 de la mañana). OBTENCIÓN DE PLÁSMIDO ( MINIPREPS, Lisis alcalina)
PRÁCTICAS AMPLIACIÓN DE MICROBIOLOGÍA Día 1º.- EXPLICACIÓN GENERAL. TRANSFORMACIONES: E. coli, Streptomyces Día 2º.- INOCULAR MINIPREPS (8:45 de la mañana). OBTENCIÓN DE PLÁSMIDO ( MINIPREPS, Lisis alcalina)
K ADN PuriPrep-GP kit. Extracción y purificación rápida de ADN a partir de: Gel de agarosa y Productos de PCR en solución
 K 1206 ADN PuriPrep-GP kit Extracción y purificación rápida de ADN a partir de: Gel de agarosa y Productos de PCR en solución Indice Presentación... 6 Importante... 6 Componentes del kit... 6 Condiciones
K 1206 ADN PuriPrep-GP kit Extracción y purificación rápida de ADN a partir de: Gel de agarosa y Productos de PCR en solución Indice Presentación... 6 Importante... 6 Componentes del kit... 6 Condiciones
Monitoreo de un proceso de purificación de proteínas. Corrimiento electroforético de las muestras de la purificación de la lactato deshidrogenasa
 Monitoreo de un proceso de purificación de proteínas Corrimiento electroforético de las muestras de la purificación de la lactato deshidrogenasa Qué es la electroforesis? Es la técnica que permite la separación
Monitoreo de un proceso de purificación de proteínas Corrimiento electroforético de las muestras de la purificación de la lactato deshidrogenasa Qué es la electroforesis? Es la técnica que permite la separación
Diagnóstico Avanzado en Enfermedades Tropicales
 Diagnóstico Avanzado en Enfermedades Tropicales Máster universitario en Enfermedades Tropicales Código 302731 (3 créditos ECTS) Área de Parasitología Departamento de Biología Animal, Parasitología, Ecología,
Diagnóstico Avanzado en Enfermedades Tropicales Máster universitario en Enfermedades Tropicales Código 302731 (3 créditos ECTS) Área de Parasitología Departamento de Biología Animal, Parasitología, Ecología,
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón
 RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS
 Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS DICIEMBRE 2006 Tratamiento de Imágenes por Computadora Proyecto
Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS DICIEMBRE 2006 Tratamiento de Imágenes por Computadora Proyecto
ELECTROFORESIS AVANZADA
 Ref. ELECAVANZADA (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref. ELECAVANZADA (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80
 AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
ANEXOS. Anexo 1. Solución Reguladora de Acetato 0.1 M, ph 4.0 y metanol al 2% (v/v).
 ANEXOS Anexo 1 Solución Reguladora de Acetato 0.1 M, ph 4.0 y metanol al 2% (v/v). 1. Tomar 6.011mL de ácido acético y aforar a 1L de agua HPLC. 2. Pesar 13.609 g de acetato de sodio y aforar a 1L de agua
ANEXOS Anexo 1 Solución Reguladora de Acetato 0.1 M, ph 4.0 y metanol al 2% (v/v). 1. Tomar 6.011mL de ácido acético y aforar a 1L de agua HPLC. 2. Pesar 13.609 g de acetato de sodio y aforar a 1L de agua
Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato
 Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de
 Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de ASEBIR). 1 2 3 4 Anexo2: Ejemplo de protocolo para translocaciones
Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de ASEBIR). 1 2 3 4 Anexo2: Ejemplo de protocolo para translocaciones
INMUNOBLOTTING (Electroforesis, transferencia a papel, detección de antígenos)
 INMUNOBLOTTING (Electroforesis, transferencia a papel, detección de antígenos) Mª Dolores García González y Leonila Laiz Trabajo Introducción La utilización cada vez más frecuente de técnicas inmunológicas
INMUNOBLOTTING (Electroforesis, transferencia a papel, detección de antígenos) Mª Dolores García González y Leonila Laiz Trabajo Introducción La utilización cada vez más frecuente de técnicas inmunológicas
GENERALIDADES Y APLICACIONES DE LA ELECTROFORESIS. Influencia de campo cargado eléctricamente Movimiento al cátodo (-) o al anodo (+)
 GENERALIDADES Y APLICACIONES DE LA ELECTROFORESIS Electroforésis Separación de especies cargadas (anión (-) y catión (+) ) Influencia de campo cargado eléctricamente Movimiento al cátodo (-) o al anodo
GENERALIDADES Y APLICACIONES DE LA ELECTROFORESIS Electroforésis Separación de especies cargadas (anión (-) y catión (+) ) Influencia de campo cargado eléctricamente Movimiento al cátodo (-) o al anodo
Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497.
 Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497. Extracción de DNA genómico El DNA de A. brasilense CBG 497 se extrajo siguiendo el método descrito por la casa comercial distribuidora
Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497. Extracción de DNA genómico El DNA de A. brasilense CBG 497 se extrajo siguiendo el método descrito por la casa comercial distribuidora
Electroforesis. Dr. Héctor L. Ayala del Río AMGEN Bruce Wallace Program Departamento de Biología UPR-Humacao VER VIDEO CARGADO GEL
 Electroforesis Dr. Héctor L. Ayala del Río AMGEN Bruce Wallace Program Departamento de Biología UPR-Humacao VER VIDEO CARGADO GEL 1 Electroforesis l Método utilizado para separar y purificar macromoléculas:
Electroforesis Dr. Héctor L. Ayala del Río AMGEN Bruce Wallace Program Departamento de Biología UPR-Humacao VER VIDEO CARGADO GEL 1 Electroforesis l Método utilizado para separar y purificar macromoléculas:
Código: IDK-003 Ver: 1. Factor V Leiden
 Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
Bqca.. Esp. Marcela Alejandra Guastavino Dra. María a Mercedes Tiscornia
 Bqca.. Esp. Marcela Alejandra Guastavino Dra. María a Mercedes Tiscornia Cátedra de Biología Molecular y Genética - 2015 OBJETIVOS Comprender el fundamento teórico de las técnicas t básicas de aislamiento
Bqca.. Esp. Marcela Alejandra Guastavino Dra. María a Mercedes Tiscornia Cátedra de Biología Molecular y Genética - 2015 OBJETIVOS Comprender el fundamento teórico de las técnicas t básicas de aislamiento
Trabajo Práctico 5: Electroforesis de proteínas Electroforesis de proteínas en Acetato de celulosa
 Trabajo Práctico 5: Electroforesis de proteínas Electroforesis de proteínas en Acetato de celulosa OBJETIVO: -Realizar electroforesis de las proteínas presentes en suero utilizando acetato de celulosa
Trabajo Práctico 5: Electroforesis de proteínas Electroforesis de proteínas en Acetato de celulosa OBJETIVO: -Realizar electroforesis de las proteínas presentes en suero utilizando acetato de celulosa
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Técnicas de Estudio de las células
 Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Código: KIT-005 Ver: 1 MTHFR C677T
 Sistema para detección de la alteración C677T en el gene que codifica para la Metilen-tetrahidrofolato reductasa humana 1 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598)
Sistema para detección de la alteración C677T en el gene que codifica para la Metilen-tetrahidrofolato reductasa humana 1 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598)
ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDS-PAGE. Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE)
 ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDSPAGE Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE) Método rápido, reproducible y de bajo costo Utilizado para cuantificar, comparar
ELECTROFORESIS EN GELES DE POLIACRILAMIDA SDSPAGE Presencia de Sodio Dodecil Sulfato bajo condiciones reductoras (SDSPAGE) Método rápido, reproducible y de bajo costo Utilizado para cuantificar, comparar
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
qprotein (BCA) Kit para cuantificar proteinas totales PB-L Productos Bio-Lógicos
 qprotein () Kit para cuantificar proteinas totales PB-L http://www.pb-l.com.ar qprotein () Cat. no. RA03 CONTENIDO Componentes RA0301 RA0302 Buffer A 250 ml 500 ml Buffer B 5 ml 10 ml Proteína Patrón 5
qprotein () Kit para cuantificar proteinas totales PB-L http://www.pb-l.com.ar qprotein () Cat. no. RA03 CONTENIDO Componentes RA0301 RA0302 Buffer A 250 ml 500 ml Buffer B 5 ml 10 ml Proteína Patrón 5
Anexo Conservar a 4 o C.
 Anexo 1 Purificación de ADN viral 1. Partir de un sobrenadante de un cultivo celular infectado y clarificado. 2. Concentrar las formas brotantes de los baculovirus por centrifugación (centrifugrar 1,5
Anexo 1 Purificación de ADN viral 1. Partir de un sobrenadante de un cultivo celular infectado y clarificado. 2. Concentrar las formas brotantes de los baculovirus por centrifugación (centrifugrar 1,5
ANEXOS. Anexo 1 Transformación mediante electroporación
 Anexo 1 Transformación mediante electroporación ANEXOS En este método de transformación de E. coli se requieren células electrocompetentes. La preparación de estas células consta de los siguientes pasos:
Anexo 1 Transformación mediante electroporación ANEXOS En este método de transformación de E. coli se requieren células electrocompetentes. La preparación de estas células consta de los siguientes pasos:
Código: IDK-006 Ver: 1 MTHFR A1298C. Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa
 Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
TP2: Extracción y cuantificación de proteínas
 Objetivo TP2: Extracción y cuantificación de proteínas Comparar métodos de extracción de proteínas a partir de distintos organismos. Cuantificar las proteínas resultantes de cada extracto. Introducción
Objetivo TP2: Extracción y cuantificación de proteínas Comparar métodos de extracción de proteínas a partir de distintos organismos. Cuantificar las proteínas resultantes de cada extracto. Introducción
EXTRACCIÓN Y PURIFICACIÓN DE ÁCIDOS NUCLEICOS
 EXTRACCIÓN Y PURIFICACIÓN DE ÁCIDOS NUCLEICOS Consideraciones iniciales - Condiciones de limpieza y esterilidad - Peligrosidad de algunos reactivos usados en la purificación Fases - Rotura-homogeneización:
EXTRACCIÓN Y PURIFICACIÓN DE ÁCIDOS NUCLEICOS Consideraciones iniciales - Condiciones de limpieza y esterilidad - Peligrosidad de algunos reactivos usados en la purificación Fases - Rotura-homogeneización:
DANAGENE BLOOD DNA KIT
 Ref. 0601 100 ml Ref. 0602 200 ml DANAGENE BLOOD DNA KIT 1.INTRODUCCION DANAGENE BLOOD DNA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de sangre total o médula ósea.
Ref. 0601 100 ml Ref. 0602 200 ml DANAGENE BLOOD DNA KIT 1.INTRODUCCION DANAGENE BLOOD DNA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de sangre total o médula ósea.
11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS. Actualmente existen diversos métodos en el mercado que pueden utilizarse
 11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS Actualmente existen diversos métodos en el mercado que pueden utilizarse para el diagnóstico de rotavirus, las cuales pueden realizarse directamente a partir
11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS Actualmente existen diversos métodos en el mercado que pueden utilizarse para el diagnóstico de rotavirus, las cuales pueden realizarse directamente a partir
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
SOUTHERN BLOT. A Fragmentos estándar de ADN. E ADN padre 2 cortado con enzimas de restricción. C ADN Hijo cortado con enzimas de restricción
 SOUTHERN BLOT 5 grupos de estudiantes 1. OBJETIVO DEL EXPERIMENTO El objetivo de esta práctica es introducir a los estudiantes en el conocimiento de una de las técnicas de hibridación utilizadas en laboratorio,
SOUTHERN BLOT 5 grupos de estudiantes 1. OBJETIVO DEL EXPERIMENTO El objetivo de esta práctica es introducir a los estudiantes en el conocimiento de una de las técnicas de hibridación utilizadas en laboratorio,
Electroforesis de proteínas
 Electroforesis de proteínas Introducción general La electroforesis es un método de separación basado en la tasa diferencial de migración de especies cargadas al estar en un campo eléctrico de DC La electroforesis
Electroforesis de proteínas Introducción general La electroforesis es un método de separación basado en la tasa diferencial de migración de especies cargadas al estar en un campo eléctrico de DC La electroforesis
DANAGENE SALIVA KIT. Ref Extracciones / Ref Extracciones
 DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION Este kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras de saliva
DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION Este kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras de saliva
41. Comprobación de colonias transformantes mediante PCR y electroforesis
 41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
CYP 2C9 A1075C CYP2C9*3
 CYP 2C9 A1075C Sistema para la detección de la mutación A1075C en el gene del citocromo P450 2C9. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy
CYP 2C9 A1075C Sistema para la detección de la mutación A1075C en el gene del citocromo P450 2C9. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy
TP3: Electroforesis en gel de poliacrilamida
 Objetivos TP3: Electroforesis en gel de poliacrilamida Comparar los perfiles proteicos obtenidos mediante SDS-PAGE. Calcular la movilidad electroforética de diferentes bandas proteicas. Introducción La
Objetivos TP3: Electroforesis en gel de poliacrilamida Comparar los perfiles proteicos obtenidos mediante SDS-PAGE. Calcular la movilidad electroforética de diferentes bandas proteicas. Introducción La
TRABAJO PRÁCTICO No. 2 EXTRACION DE ACIDO NUCLEICO VIRAL
 Unidad Curricular: Virología y Micología Veterinaria TRABAJO PRÁCTICO No. 2 EXTRACION DE ACIDO NUCLEICO VIRAL Las pruebas para el diagnostico específico de una infección viral se clasifican en dos tipos:
Unidad Curricular: Virología y Micología Veterinaria TRABAJO PRÁCTICO No. 2 EXTRACION DE ACIDO NUCLEICO VIRAL Las pruebas para el diagnostico específico de una infección viral se clasifican en dos tipos:
MATERIALES Y MÉTODOS. Cepa de Mycobacterium tuberculosis
 MATERIALES Y MÉTODOS Cepa de Mycobacterium tuberculosis Se utilizó una cepa de referencia de Mycobacterium tuberculosis H37Rv tipificada en el Instituto Nacional de Diagnóstico y Referencia Epidemiológica
MATERIALES Y MÉTODOS Cepa de Mycobacterium tuberculosis Se utilizó una cepa de referencia de Mycobacterium tuberculosis H37Rv tipificada en el Instituto Nacional de Diagnóstico y Referencia Epidemiológica
EXTRACCIÓN DE ADN. BIOLOGÍA GENERAL (Evolución y Genética)
 EXTRACCIÓN DE ADN BIOLOGÍA GENERAL (Evolución y Genética) Curso Académico 2015-2016 Abril 2016 INTRODUCCIÓN Y CONCEPTOS Extracción de ADN La extracción de ADN requiere una serie de etapas básicas. En primer
EXTRACCIÓN DE ADN BIOLOGÍA GENERAL (Evolución y Genética) Curso Académico 2015-2016 Abril 2016 INTRODUCCIÓN Y CONCEPTOS Extracción de ADN La extracción de ADN requiere una serie de etapas básicas. En primer
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
Tema 6 Cuantificación de proteínas
 Tema 6 Cuantificación de proteínas Procedimiento de estudio de proteínas Selección de fuente Fraccionamiento de células Centrifugación Cromatografía Cromatografía en capa fina Cromatografía en columna
Tema 6 Cuantificación de proteínas Procedimiento de estudio de proteínas Selección de fuente Fraccionamiento de células Centrifugación Cromatografía Cromatografía en capa fina Cromatografía en columna
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR
 KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
Proyecto de Fin de Cursos
 Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Purificación de ADN plasmídico, utilizando soluciones homeopatizadas
 Purificación de ADN plasmídico, utilizando soluciones homeopatizadas Dra. Niurka Meneses Moreno Universidad de Berna, Suiza niurka.meneses@unibe.dcb.ch, niurkam@gmail.com En los laboratorios de investigación
Purificación de ADN plasmídico, utilizando soluciones homeopatizadas Dra. Niurka Meneses Moreno Universidad de Berna, Suiza niurka.meneses@unibe.dcb.ch, niurkam@gmail.com En los laboratorios de investigación
PRÁCTICA III. AISLAMIENTO DE MITOCONDRIAS: ACTIVIDAD MALATO DESHIDROGENASA
 PRÁCTICA III. AISLAMIENTO DE MITOCONDRIAS: ACTIVIDAD MALATO DESHIDROGENASA 1. Fundamento Teórico La mayoría de los procesos bioquímicos que producen energía química se realizan en orgánulos intracelulares
PRÁCTICA III. AISLAMIENTO DE MITOCONDRIAS: ACTIVIDAD MALATO DESHIDROGENASA 1. Fundamento Teórico La mayoría de los procesos bioquímicos que producen energía química se realizan en orgánulos intracelulares
Biología Celular y Molecular. Guía de TP Nro. 5. Actividad proteolítica celular.
 Biología Celular y Molecular. Guía de TP Nro. 5 Actividad proteolítica celular. Introducción La actividad proteolítica secretada por las células es un elemento indispensable para la proliferación y migración
Biología Celular y Molecular. Guía de TP Nro. 5 Actividad proteolítica celular. Introducción La actividad proteolítica secretada por las células es un elemento indispensable para la proliferación y migración
Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo
 Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo ESQUEMA GENERAL Sesión 1. Extracción y precipitación con sulfato de amonio. Sesión 2. Purificación por cromatografía Sesión
Purificación de la enzima lactato deshidrogenasa de músculo esquelético de pollo ESQUEMA GENERAL Sesión 1. Extracción y precipitación con sulfato de amonio. Sesión 2. Purificación por cromatografía Sesión
DETERMINACIÓN DEL PESO MOLECULAR DE PROTEÍNAS
 DETERMINACIÓN DEL PESO MOLECULAR DE PROTEÍNAS 6 grupos de estudiantes 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es desarrollar el conocimiento de la estructura de las proteínas y determinar
DETERMINACIÓN DEL PESO MOLECULAR DE PROTEÍNAS 6 grupos de estudiantes 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es desarrollar el conocimiento de la estructura de las proteínas y determinar
MATERIALES Y MÉTODOS. Tipo de Estudio. Muestra
 MATERIALES Y MÉTODOS Tipo de Estudio Experimental. Este tipo de estudio tiene como característica primordial el poder manipular y controlar las variables, siendo en este caso la fuerza iónica y el ph.
MATERIALES Y MÉTODOS Tipo de Estudio Experimental. Este tipo de estudio tiene como característica primordial el poder manipular y controlar las variables, siendo en este caso la fuerza iónica y el ph.
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
DESARROLLO COGNOSCITIVO Y APRENDIZAJE
 Prof. Jhonis Valbuena Cátedra: Análisis Instrumental La Electrofóresis es un método de la Química Analítica que utiliza corriente eléctrica controlada con la finalidad de separar biomolèculas según su
Prof. Jhonis Valbuena Cátedra: Análisis Instrumental La Electrofóresis es un método de la Química Analítica que utiliza corriente eléctrica controlada con la finalidad de separar biomolèculas según su
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
Conceptos y técnicas en ecología fluvial
 Conceptos y técnicas en ecología fluvial Edición a cargo de: ARTURO ELOSEGI Profesor titular de Ecología en la Universidad del País Vasco SERGI SABATER Catedrático de Ecología en la Universidad de Girona
Conceptos y técnicas en ecología fluvial Edición a cargo de: ARTURO ELOSEGI Profesor titular de Ecología en la Universidad del País Vasco SERGI SABATER Catedrático de Ecología en la Universidad de Girona
TP3: Electroforesis en gel de poliacrilamida
 TP3: Electroforesis en gel de poliacrilamida Objetivos Comparar las fracciones de proteínas obtenidas en el TP Nº2 mediante SDS-PAGE. Calcular la movilidad electroforética de diferentes bandas proteicas.
TP3: Electroforesis en gel de poliacrilamida Objetivos Comparar las fracciones de proteínas obtenidas en el TP Nº2 mediante SDS-PAGE. Calcular la movilidad electroforética de diferentes bandas proteicas.
Manual de Uso (Ref /2)
 Fabricado por: BIOTOOLS B&M Labs, S.A. Valle de Tobalina - 52 - Nave 39 28021 Madrid España Tel. (34) 91 710 00 74 Fax (34) 93 843 78 84 e-mail: info@biotools.eu www.biotools.eu Diseñado para la purificación
Fabricado por: BIOTOOLS B&M Labs, S.A. Valle de Tobalina - 52 - Nave 39 28021 Madrid España Tel. (34) 91 710 00 74 Fax (34) 93 843 78 84 e-mail: info@biotools.eu www.biotools.eu Diseñado para la purificación
DETERMINACIONES ESPECTROFOTOMETRICAS EN ALIMENTOS. Carotenoides totales y nitritos.
 1 PRACTICA Nº 5 DETERMINACIONES ESPECTROFOTOMETRICAS EN ALIMENTOS. Carotenoides totales y nitritos. I. INTRODUCCIÓN: En espectrofotometría se usa como fuente luminosa la luz blanca natural o artificial
1 PRACTICA Nº 5 DETERMINACIONES ESPECTROFOTOMETRICAS EN ALIMENTOS. Carotenoides totales y nitritos. I. INTRODUCCIÓN: En espectrofotometría se usa como fuente luminosa la luz blanca natural o artificial
Ensayos southwestern y northwestern
 Búsqueda de proteínas que se unen a DNA (secuencia conocida, promotor) Ensayo southwestern encontrar proteínas ligantes de DNA que reconozcan en éste una determinada secuencia de nucleótidos Ensayo northwestern
Búsqueda de proteínas que se unen a DNA (secuencia conocida, promotor) Ensayo southwestern encontrar proteínas ligantes de DNA que reconozcan en éste una determinada secuencia de nucleótidos Ensayo northwestern
UNIVERSIDAD TECNOLÓGICA EQUINOCCIAL Laboratorio de Química. MEDICIÓN DEL ph DE ÁCIDOS, BASES Y SALES 2. INTRODUCCIÓN
 UNIVERSIDAD TECNOLÓGICA EQUINOCCIAL Laboratorio de Química 1 MEDICIÓN DEL ph DE ÁCIDOS, BASES Y SALES 1. OBJETIVOS. Reconocer la acidez o basicidad de una sustancia mediante el uso de indicadores. Relacionar
UNIVERSIDAD TECNOLÓGICA EQUINOCCIAL Laboratorio de Química 1 MEDICIÓN DEL ph DE ÁCIDOS, BASES Y SALES 1. OBJETIVOS. Reconocer la acidez o basicidad de una sustancia mediante el uso de indicadores. Relacionar
Purificación de Proteínas Parte II
 Purificación de Proteínas Parte II Fase I. Obtención del extracto crudo Lisis celular. Válido para células sin pared celular como las células de tejidos animales, pero no es suficiente para células vegetales
Purificación de Proteínas Parte II Fase I. Obtención del extracto crudo Lisis celular. Válido para células sin pared celular como las células de tejidos animales, pero no es suficiente para células vegetales
Proceso que cuenta con varias etapas cuyo objetivo es separar una proteína de interés de una mezcla de proteínas.
 Proceso que cuenta con varias etapas cuyo objetivo es separar una proteína de interés de una mezcla de proteínas. La selección de técnicas de purificación depende de las propiedades de las proteínas. Características
Proceso que cuenta con varias etapas cuyo objetivo es separar una proteína de interés de una mezcla de proteínas. La selección de técnicas de purificación depende de las propiedades de las proteínas. Características
Tópicos en Biofísica Molecular
 Tópicos en Biofísica Molecular 2do Cuatrimestre de 2013 Docentes: Lía Pietrasanta y Catalina von Bilderling Práctica de laboratorio nº 1: Soluciones, ph OBJETIVOS Familiarizarse con el material de uso
Tópicos en Biofísica Molecular 2do Cuatrimestre de 2013 Docentes: Lía Pietrasanta y Catalina von Bilderling Práctica de laboratorio nº 1: Soluciones, ph OBJETIVOS Familiarizarse con el material de uso
Dice Debe decir Justificación*
 COMENTARIOS Con fundamento en el numeral 4.11.1 de la Norma Oficial Mexicana NOM-001-SSA1-2010, se publica el presente proyecto a efecto de que los interesados, a partir del 1º de febrero y hasta el 31
COMENTARIOS Con fundamento en el numeral 4.11.1 de la Norma Oficial Mexicana NOM-001-SSA1-2010, se publica el presente proyecto a efecto de que los interesados, a partir del 1º de febrero y hasta el 31
Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a
 Introducción Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a los investigadores verificar la expresión de una proteína, determinar la cantidad relativa
Introducción Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a los investigadores verificar la expresión de una proteína, determinar la cantidad relativa
DETERMINACIÓN DEL MAPA DE UN PLÁSMIDO CON ENZIMAS DE RESTRICCIÓN
 DETERMINACIÓN DEL MAPA DE UN PLÁSMIDO CON ENZIMAS DE RESTRICCIÓN 6 grupos de estudiantes 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es desarrollar un conocimiento del mapaje de ADN determinando
DETERMINACIÓN DEL MAPA DE UN PLÁSMIDO CON ENZIMAS DE RESTRICCIÓN 6 grupos de estudiantes 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es desarrollar un conocimiento del mapaje de ADN determinando
CROMATOGRAFÍA. A.- INTERCAMBIO IONICO: A.1. ANIONICA A.2. CATIONCA B.- FILTRACIÓN EN GEL
 CROMATOGRAFÍA. A.- INTERCAMBIO IONICO: A.1. ANIONICA A.2. CATIONCA B.- FILTRACIÓN EN GEL C.- AFINIDAD B. CROMATOGRAFÍA DE FILTRACIÓN EN GEL. 1. Principios. No requiere la unión de la proteína, lo cual
CROMATOGRAFÍA. A.- INTERCAMBIO IONICO: A.1. ANIONICA A.2. CATIONCA B.- FILTRACIÓN EN GEL C.- AFINIDAD B. CROMATOGRAFÍA DE FILTRACIÓN EN GEL. 1. Principios. No requiere la unión de la proteína, lo cual
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
 EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
Código: IDK-004 Ver: 1. Factor II 20210
 20210 Sistema para detección de la alteración G20210A en el gene que codifica para el de la coagulación humana PM 1 2 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2
20210 Sistema para detección de la alteración G20210A en el gene que codifica para el de la coagulación humana PM 1 2 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2
RESULTADOS Y DISCUSIÓN
 RESULTADOS Y DISCUSIÓN Estimación de la Concentración Proteica del Filtrado de Cultivo de Mycobacterium tuberculosis H37Rv La curva estándar con la que se estimó la concentración proteica del filtrado
RESULTADOS Y DISCUSIÓN Estimación de la Concentración Proteica del Filtrado de Cultivo de Mycobacterium tuberculosis H37Rv La curva estándar con la que se estimó la concentración proteica del filtrado
Preparación y Caracterización de Ácidos Nucleicos
 Práctico Nº 5. Ácidos Nucleicos I Preparación y Caracterización de Ácidos Nucleicos Facultad de Ciencias Bioquímica (Licenciatura en Ciencias Biológicas) Objetivos Laboratorio Ácidos Nucleicos I: Extracción
Práctico Nº 5. Ácidos Nucleicos I Preparación y Caracterización de Ácidos Nucleicos Facultad de Ciencias Bioquímica (Licenciatura en Ciencias Biológicas) Objetivos Laboratorio Ácidos Nucleicos I: Extracción
PROGRAMA DE CURSO DE FORMACION PROFESIONAL OCUPACIONAL Técnicas De Análisis Cromosómico Y Ácidos Nucleicos
 MINISTERIO DE TRABAJO Y ASUNTOS SOCIALES INSTITUTO NACIONAL DE EMPLEO PROGRAMA DE CURSO DE FORMACION PROFESIONAL OCUPACIONAL Técnicas De Análisis Cromosómico Y Ácidos Nucleicos NIPO: DATOS GENERALES DEL
MINISTERIO DE TRABAJO Y ASUNTOS SOCIALES INSTITUTO NACIONAL DE EMPLEO PROGRAMA DE CURSO DE FORMACION PROFESIONAL OCUPACIONAL Técnicas De Análisis Cromosómico Y Ácidos Nucleicos NIPO: DATOS GENERALES DEL
PROGRAMA DE CURSO DE FORMACION PROFESIONAL OCUPACIONAL
 PROGRAMA DE CURSO DE FORMACION PROFESIONAL OCUPACIONAL TÉCNICAS DE ANÁLISIS CROMOSÓMICO Y ÁCIDOS NUCLEICOS 1 DATOS GENERALES DEL CURSO 1. FAMILIA PROFESIONAL: SANIDAD ÁREA PROFESIONAL: ESPECIALIDADES SIN
PROGRAMA DE CURSO DE FORMACION PROFESIONAL OCUPACIONAL TÉCNICAS DE ANÁLISIS CROMOSÓMICO Y ÁCIDOS NUCLEICOS 1 DATOS GENERALES DEL CURSO 1. FAMILIA PROFESIONAL: SANIDAD ÁREA PROFESIONAL: ESPECIALIDADES SIN
SECRETARIA DE COMERCIO FOMENTO INDUSTRIAL NORMA MEXICANA NMX-K DETERMINACION DE ORTOFOSFATOS EN AGUAS PARA CALDERAS
 SECRETARIA DE COMERCIO Y FOMENTO INDUSTRIAL NORMA MEXICANA NMX-K-450-1978 DETERMINACION DE ORTOFOSFATOS EN AGUAS PARA CALDERAS WATER FOR BOILERS DETERMINATION OF ARTHO PHOSPHATES DIRECCION GENERAL DE NORMAS
SECRETARIA DE COMERCIO Y FOMENTO INDUSTRIAL NORMA MEXICANA NMX-K-450-1978 DETERMINACION DE ORTOFOSFATOS EN AGUAS PARA CALDERAS WATER FOR BOILERS DETERMINATION OF ARTHO PHOSPHATES DIRECCION GENERAL DE NORMAS
PROGRAMA FORMATIVO Técnicas De Análisis Cromosómico Y Ácidos Nucleicos
 PROGRAMA FORMATIVO Técnicas De Análisis Cromosómico Y Ácidos Nucleicos DATOS GENERALES DEL CURSO 1. Familia Profesional: SANIDAD Área Profesional: TÉCNICAS DE LABORATORIO ANATOMÍA PATOLÓGICA 2. Denominación
PROGRAMA FORMATIVO Técnicas De Análisis Cromosómico Y Ácidos Nucleicos DATOS GENERALES DEL CURSO 1. Familia Profesional: SANIDAD Área Profesional: TÉCNICAS DE LABORATORIO ANATOMÍA PATOLÓGICA 2. Denominación
MATERIALES Y MÉTODOS. Muestras
 MATERIALES Y MÉTODOS Muestras Las muestras de sangre con el bacilo M. tuberculosis se obtuvieron de 2 pacientes con tuberculosis diagnosticada por clínica, laboratorio y con cultivo positivo como controles
MATERIALES Y MÉTODOS Muestras Las muestras de sangre con el bacilo M. tuberculosis se obtuvieron de 2 pacientes con tuberculosis diagnosticada por clínica, laboratorio y con cultivo positivo como controles
