Centro de Investigación en Alimentación y Desarrollo, A.C.
|
|
|
- María Pilar Moya Ojeda
- hace 6 años
- Vistas:
Transcripción
1 Centro de Investigación en Alimentación y Desarrollo, A.C. CARACTERIZACIÓN BIOQUÍMICA Y BIOFÍSICA DE NUCLEÓSIDO DIFOSFATO CINASA DE CAMARÓN BLANCO (Litopenaeus vannamei) POR: IDANIA EMEDITH QUINTERO REYES TESIS APROBADA POR: COORDINACIÓN DE TECNOLOGÍA DE ALIMENTOS DE ORIGEN ANIMAL Como requisito parcial para obtener el grado de DOCTOR EN CIENCIAS Hermosillo, Sonora Diciembre 2012
2 ii
3 iii
4 FINANCIAMIENTO Este trabajo fue realizado en el Laboratorio de Biología Molecular de Organismos Acuáticos, de la Coordinación de Tecnología de Alimentos de Origen Animal del Centro de Investigación en Alimentación y Desarrollo, A.C. El apoyo financiero para su realización fue proporcionado por el Consejo Nacional de Ciencia y Tecnología (CONACYT), a través del proyecto CB "Inhibición del metabolismo de nucleótidos como estrategia contra el virus de la mancha blanca del camarón" y de la beca de estudios de doctorado otorgada por CONACYT. iv
5 AGRADECIMIENTOS Agradezco al Consejo Nacional de Ciencia y Tecnología (CONACYT) por el apoyo brindado durante ésta etapa fundamental de mi desarrollo académico. De igual manera se agradece al Centro de Investigación en Alimentación y Desarrollo, A.C. (CIAD) por la oportunidad de continuar preparándome como profesionista, además de todas las facilidades y apoyos otorgados para que este trabajo se pudiera llevar a cabo. Además al apoyo financiero para la realización de esta tesis que fue proporcionado a través del proyecto CB "Inhibición del metabolismo de nucleótidos como estrategia contra el virus de la mancha blanca del camarón" el cual fue proporcionado por el Consejo Nacional de Ciencia y Tecnología (CONACYT). Quiero agradecer infinitamente al Dr. Rogerio R. Sotelo Mundo por su guía, consejos y formación académica. A las observaciones, asesorías y sugerencias tan atinadas del Dr. Jesús Hernández López y la Dra. Adriana Muhlia Almazán. A la maestra Karina García Orozco por su asesoría y paciencia. A la Dra. María Auxiliadora Islas por su amistad y contribuciones en mi formación académica. v
6 Dra. Gloria Yepiz Plascencia agradezco de todo corazón esos momentos que me regaló de su valioso tiempo, lo cual me hizo crecer personal y académicamente. Al Dr. Enrique Velázquez Contreras por la asesoría brindada en la parte de calorimetría de titulación isotérmica de este trabajo, la cual lo enriqueció infinitamente. A la Dra. Rocío Sugich Miranda por su asesoría y apoyo en los análisis de calorimetría de titulación isotérmica, además por las facilidades otorgadas en el Departamento de Polímeros de la Universidad de Sonora, por su disponibilidad, consejos brindados y sobre todo por su amistad. Al Dr. Enrique Rudiño Pinera por la disponibilidad y asesoría brindada en la parte de Cristalografía. amistad. A la maestra Alma B. Peregrino por su disponibilidad, confianza y A las maestras Gisela Robles, María Elena Lugo, Guillermina García, Dra. Tere Gollas, por sus finas atenciones. Gerard. A Gerardo Reyna, por las facilidades otorgadas en Biblioteca, thanks Al Dr. Ramón Pacheco por su asesoría y consejos en esta etapa. A toda la Coordinación de Programas Académicos, Laura, Verónica, Argelia, Héctor, Aurora, Norma, Alejandra y Alfonso. vi
7 A mis compañeros de laboratorio Lalo, Max, Alonso, Monsterrat, Laura, Cindy, José Alfredo. A Salvador Carrasco y a las gusanitas Magda Pacheco, Citla Colín y Claudia Berlanga, por todo su apoyo y amistad, mil gracias. A mi tía Any (Dra. Ana María Calderón de la Barca) por sus finas atenciones, asesoría y facilidades otorgadas en su lab. Finalmente quiero agradecer al Dr. Francisco J. Castillo Yáñez por su guía, confianza, consejos y paciencia que me ha regalado desde la licenciatura, mil gracias. vii
8 DEDICATORIA A Dios por permitirme cumplir con una etapa más como profesionista. A mis padres Yosy y Juan, por ser la fuente de inspiración a mi superación como profesionista y como humano. Por su amor, paciencia y confianza. A mis hermanos, Oliver y Joan, por compartir este sendero conmigo. A mis tíos Lupita y José Luis, por su apoyo y presencia. A mi Tatito, al fin te cumplí. viii
9 ÍNDICE Páginas APROBACIÓN ii DECLARACIÓN INSTITUCIONAL iii FINANCIAMIENTO iv AGRADECIMIENTOS v DEDICATORIA viii INDICE ix LISTA DE FIGURAS xii LISTA DE TABLAS xiv RESUMEN xv ABSTRACT xvii INTRODUCCIÓN 1 ANTECEDENTES 3 Metabolismo de Nucleótidos 3 Biosíntesis de novo: Nucelótidos de Purinas 7 Biosíntesis de novo: Nucleótidos de Pirimidinas 12 Biosíntesis de Ribonucleótidos de Timina: vía de novo y de 14 Rescate Biosíntesis de Desoxirribonucleótidos: Reducción de 16 Ribonucleótidos a Desoxirribonucleótidos Análogos Nucleosídicos 18 Antivirales 18 Análogos Nucleosídicos no Fosforilados 20 Análogos Nucleosídicos Fosforilados 22 Enzimas Cinasas 24 Cinasas que Participan en la Fosforilación de Análogos 24 Nucleosídicos ix
10 Nucleósido Difosfato Cinasa (NDK) 26 Nucleósido Difosfato Cinasas Humanas: Grupo I 31 Nucleósido Difosfato Cinasas Humanas: Grupo II 32 Nucleósido Difosfato Cinasa en Virus 33 Virus de la Mancha Blanca (WSSV) 34 Estrategias de Control 37 Tratamiento a base de productos naturales 37 Tratamiento a base de vacunas de ADN/ARN 38 JUSTIFICACIÓN 40 HIPÓTESIS 41 OBJETIVO GENERAL 42 OBJETIVOS PARTICULARES 42 MATERIALES Y MÉTODOS 43 Reactivos 43 Producción de la Proteína Recombinante NDK de L. vannamei 43 Metales (IMAC) 44 Ensayo de Actividad Específica de NDK de L. vannamei Frente 45 a Desoxinucleótidos Difosfato Mediante Ensayo Enzimático Acoplado Determinación de la Estructura Cuaternaria de NDK de L. 45 vannamei Mediante Cromatografía de Exclusión Molecular Determinación de Parámetros Termodinámicos de NDK de L. 46 vannamei Frente a Desoxinucleótidos Difosfato Mediante Calorimetría de Titulación Isotérmica Modelamiento in silico y Anclaje de NDK de L. vannamei 47 Determinación de Condiciones de Cristalización del Complejo 48 NDK-dGDP RESULTADOS Y DISCUSIÓN 49 CONCLUSIONES 67 REFERENCIAS BIBLIOGRÁFICAS 69 ANEXO I.- 74 x
11 Idania E. Quintero-Reyes, Karina D. García-Orozco, Rocío Sugich- Miranda, Aldo A. Arivizu-Flores, Enrique F. Velázquez-Contreras, Francisco J. Castillo-Yáñez, Rogerio S. Sotelo-Mundo (2012). Shrimp oncoprotein nm23 is a functional nucleoside diphosphate kinase. Journal of Bioenergetics and Biomembranes, 44: ANEXO II.- Idania E. Quintero-Reyes, Alonso A. López-Zavala, Jesús S. Carrasco-Miranda, Karina D. García-Orozco, Enrique Rudiño- Piñera, Rogerio R. Sotelo-Mundo. Crystallization studies for a nucleoside diphosphate kinase from Pacific whiteleg Shrimp (Litopenaeus vannamei). Enviado a Acta Crystallographica F. 82 xi
12 LISTA DE FIGURAS Figura Página 1 Estructura química de bases nitrogenadas 4 2 Esquema general del metabolismo de nucleótidos 6 3 Reacciones necesarias para la activación de la ribos-5-8 fosfato para generación de PRPP 4 Vía de novo para la biosíntesis de IMP a partir de PRPP 10 5 Biosíntesis de novo para la obtención de purinas a partir 11 de IMP 6 Síntesis de novo de nucleótidos de pirimidina a partir de 13 carbamoil fosfato 7 Ruta de novo y del rescate para la biosíntesis de 15 núcleotidos de timina 8 Biosíntesis de desoxirribonucleótidos trifosfato a partir de 17 ribonucleótido reductasa 9 Estructura química de análogos nucleosídicos (a,c) y 19 desoxinucleótidos 10 Esquema representativo del mecanismo de acción de 21 aciclovir (ACV) 11 Esquema representativo del mecanismo de acción de 23 cidofovir 12 Representación esquemática de la fosforilación de 25 nucleósidos naturales que participan en la vía del rescate en humanos 13 Modelo tridimensional del posicionamiento del sustrato en la cavidad catalítica de NDK 30 xii
13 14 Número de granjas afectadas por WSSV durante el 34 periodo Representación esquemática de la morfología de WSSV Análisis electroforético de la cinética de sobreexpresión 50 de NDK de L. vannamei. 17 Análisis electroforético de fracciones provenientes de la 51 purificación de NDK recombinante de L. vannamei mediante IMAC 18 Cromatograma obtenido a partir de cromatografía de 55 exclusión molecular 19 Modelo estructural de NDK de L. vannamei obtenido 57 mediante el paquete computacional MOE versión Modelo teórico del anclaje (docking) entre NDK de L. 61 vannamei y deoxinucleótidos difosfato 21 Cristales del complejo de NDK-dGDP obtenidos en la solución de 0.2 M de acetato de magnesio tetrahidratado, ph 6.5 y PEG 8000 al 20%, 66 xiii
14 LISTA DE TABLAS Tabla 1 Evaluación de la actividad específica de NDK recombinante de L. vannamei frente a desoxinucleótidos bifosfato. 2 Estructura cuaternaria de NDK en diferentes organismos modificado de (Lascu et al., 2000) 3 Parámetros termodinámicos y constantes de unión de NDK de L. vannamei frente a desoxinucleótidos bifosfato obtenidos mediante calorimetría de titulación isotérmica Página xiv
15 RESUMEN La nucleósido difosfato cinasa (NDK, E.C ) es una enzima que fosforila nucleósidos-difosfatos para generar nucleósidos-trifosfatos (NTPs), dicha función es de gran importancia para la replicación de ácidos nucleicos como el ácido desoxirribonucléico (ADN). La del camarón blanco (Litopenaeus vannamei) se expresa durante la infección con el virus de la mancha blanca (WSSV), donde los NTPs generados por NDK pudieran ser utilizados para la replicación viral. En base a lo anterior, es conveniente investigar sobre las rutas metabólicas que producen los NTPs necesarios para la replicación viral, además de estudiar las características bioquímicas y biofísicas de la proteína NDK del camarón L. vannamei con la finalidad de bloquear la replicación del WSSV. En este estudio se sobreexpresó la enzima NDK de forma recombinante y se determinó su actividad enzimática con los deoxi-nucleótidos dgdp, dadp, dtdp y dcdp. La mayor actividad se presentó con dtdp (19,344 U/mg de proteína) y la menor actividad con dgdp (5,505 U/mg de proteína). La afinidad de la enzima por los deoxi-nucleósidos difosfatos se determinó mediante calorimetría de titulación isotérmica (ITC) y se obtuvieron resultados en el rango micromolar (Kd 10-6 ) a excepción de dcdp, ya que este sustrato no mostró interacción detectable con la NDK mediante esta técnica. La estructura cuaternaria obtenida mediante cromatografía de exclusión molecular muestra que la NDK de L. vannamei es un trímero. Se obtuvieron cristales de la enzima por el método microbatch en complejo binario con dgdp, lo cual permitirá obtener la estructura tridimensional en estudios posteriores. Los datos obtenidos en este trabajo son de gran importancia ya que la enzima pudiera utilizar análogos nucleosídicos con la finalidad de bloquear la replicación WSSV xv
16 y el conocimiento de la estructura de la proteína NDK permitirá investigar a largo plazo la interacción de fármacos trifosfosforilados como terminadores de cadena de la replicación. Palabras clave: Deoxinucleótidos, fosforilación, replicación viral, WSSV, Litopenaeus vannamei, cinasas. xvi
17 ABSTRACT Nucleoside diphosphate kinase (NDK) is an enzyme that has a key role in DNA replication as catalyzes the phosphorylation of nucleotides diphosphates to generate nucleotides triphosphates (NTP s). NDK has been described in the white leg pacific shrimp as a housekeeping enzyme expressed during the infection with the white spot syndrome virus (WSSV). In this study, NDK was obtained as a recombinant protein and its specific activity was measured towards deoxynucleotides diphosphates dgdp, dadp, dtdp and dcdp. NDK activity was 19, 344 U/mg of protein using dtdp as substrate while the lower activity was detected with dgdp. The affinity toward each substrate was determined by isothermal titration calorimetry (ITC) where the data were in the micro-molar range, except for dctp which had no detectable interaction by this method. The quaternary structure, obtained by size-exclusion chromatography, of the L. vannamei NDK corresponds to a trimer. Crystals of NDK as a binary complex with dgdp were obtained using the microbatch method, and these crystals are essential for the determination of the three dimensional structure of the protein in further studies. Further work may be directed to test if NDK could phosphorylate nucleosides analogs with the aim of blocking the WSSV. Keywords: Deoxynucleotides, phosphorylation, viral replication, WSSV, L. vannamei, deoxinucleoside kinases. xvii
18 INTRODUCCIÓN La industria camaronícola es una de las principales actividades económicas en el país, donde Sonora ocupa el primer lugar ya que ha generado derramas económicas alrededor de 4000 m.d.p. durante los últimos 4 años. Sin embargo, se ve amenazada por la presencia de patógenos como el virus de la mancha blanca (WSSV, White Spot Syndrome Virus, por sus siglas en inglés), el cual ataca a crustáceos como el camarón blanco (Litopenaeus vannamei) provocando la pérdida del cultivo dentro de los primeros días de infección. De acuerdo a la literatura, durante la infección viral éste se apodera de la maquinaria del hospedero utilizando moléculas que participan en la biosíntesis de nucleótidos trifosfato (NTPs) los cuales son fundamentales para la replicación del ADN viral. Estudios realizados en el transcriptoma de L. vannamei identificaron el ADNc de una enzima que participan en la biosíntesis de NTP s, identificada como nucleósido difosfato cinasa (NDK) de acuerdo a su identidad con su homóloga en humano (Clavero-Salas et al., 2007). NDK cataliza la tercera fosforilación de la cascada de fosforilaciones de desoxinucleósidos difosfato dependiente de ATP, con la finalidad de mantener la poza de NTPs lo cual, como ya se mencionó, es clave para la replicación del ADN (Leu et al., 2009, Deville-Bonne et al., 2010). Lo anterior la vuelve un blanco para la generación de fármacos antivirales ya que pudiera llevar a cabo el proceso de activación de análogos nucleosídicos (AN). En el mercado existen AN como el aciclovir y ganciclovir que son prodrogas dirigidas a cinasas virales, que requieren ser fosforiladas primero por enzimas como la timidina cinasa y posteriormente por otras cinasas como NDK. Sin embargo, hasta el momento no existen estrategias antivirales dirigidas a WSSV, siendo el objetivo de este 1
19 estudio obtener la NDK recombinante de L. vannamei para caracterizar su actividad bioquímica y unión a desoxinucleótidos difosfato, lo cual permitirá a largo plazo establecer estrategias para la generación de un fármaco dirigido a WSSV. 2
20 ANTECEDENTES Metabolismo de Nucleótidos Las bases nitrogenadas (Figura 1), tanto purinas como pirimidinas, son bases que forman la columna vertebral del ácido desoxirribonucleico (ADN) y el ácido ribonucleico (ARN), de acuerdo a la combinación de cada base con una pentosa forma nucleósidos y la adición de un grupo fosfato genera nucleótidos. Los nucleótidos son moléculas que participan en diferentes procesos celulares como el metabolismo energético, llevan a cabo actividades como reguladores metabólicos así como precursores del ADN. La mayoría de los organismos pueden sintetizar nucleótidos a partir de precursores de bajo peso molecular en cantidades necesarias de acuerdo a las demandas celulares, y a esta ruta se le conoce como vía de novo. La ruta por la cual los organismos pueden sintetizar nucleótidos a partir de nucleósidos procedentes de la dieta o degradación enzimática de ácidos nucleicos se le conoce como vía del rescate o del salvamento (Figura 2). Se le conoce así, ya que involucra la utilización de purinas y pirimidinas que de otra forma se pudieran perder en la biodegradación (Mathews, 2002). 3
21 Figura 1.- Estructura química de las bases nitrogenadas (Mathews, 2002). 4
22 La degradación intracelular de ácidos nucleicos debido a vías de reparación del mismo o ingesta de los mismos en la dieta tiene como resultado que la vía del salvamento represente la ruta principal por la cual las bases nitrogenadas y los nucleósidos se vuelvan disponibles en el organismo. El proceso de degradación se inicia atacando los enlaces fosfodiester por endonucleasas tales como la ribonucleasa pancreática o la desoxirribonucleasa. La acción de las enzimas mencionadas produce oligonucleótidos los cuales a su vez son modificados por exonucleasas como la fosfodiesterasa para generar mononucleótidos. Estos últimos son degradados hidrolíticamente por fosfomonoesterasas llamadas nucleotidasas, proceso en el cual, se generan ortofosfatos más el nucleósido correspondiente (Mathews et al., 2002, Lehninger Albert, 1981). 5
23 Figura 2.- Esquema general del metabolismo de nucleótidos (Mathews, 2002). 6
24 En la Figura 2 se muestran los precursores que participan en la formación de nucleótidos, con actividades específicas para la síntesis de ADN o ARN, actividades metabólicas o con función de coenzimas. Ambas vías tienen una función importante ya que se han convertido en blanco de la industria farmacéutica para generar fármacos dirigidos a enfermedades microbianas y parasitarias. Además, por ensayos de mutagénesis se ha logrado manipular algunas enzimas para la generación de anticuerpos monoclonales. Las enzimas llamadas fosforribosiltransferasas y el azúcar activado llamado 5-fosfo-α-Dribosil-1-pirofosfato ó PRPP son centrales en ambas vías (Mathews et al., 2002). Biosíntesis de novo: Nucleótidos de Purina El PRPP es un intermediario clave en la biosíntesis de novo de nucleótidos de purinas y pirimidinas. El cual se forma por la acción de la PRPP sintetasa, ya que provoca la transferencia del pirofosfato al carbón 1 de la ribosa-5-fosfato, además esta reacción es reversible por la acción de la fosforribosiltransferasa con la finalidad de generar monofosfatos y pirofosfato (Figura 3). 7
25 Figura 3.- a) Activación de la ribosa-5-fosfato para la generación de PRPP, molécula clave en la vía de novo y del salvamento. b) Generación de monofosfatos a partir de fosforribosiltransferasa (Mathews, 2002). 8
26 El PRPP es el primer intermediario para la generación del primer anillo purinico, la inosina-5-monofosfato (IMP) o ácido inosínico (Mathews et al., 2002). De manera general, los pasos para la generación de IMP involucran la adición de un grupo amino provocando un cambio en la posición del carbono α de PRPP para generar 5-fosforribosilamina o PARA, los grupos aminos son donados por amidotransferasas. El metabolito 10-formil-tetrahidrofolato participa en la adición de unidades de carbono y las moléculas utilizadas para construir la estructura del anillo son glicina y fumarato, que intervienen en los últimos pasos para generar 5-aminoimidazol-4-ribonucleotido de carboxamida o AICAR y N- formilaminoimidazol-4-ribonucleótido de carboxamida o FAICAR. Posterior a FAICAR se lleva cabo una reacción de condensación la cual produce el primer compuesto púrico IMP (Figura 4). 9
27 PRPP H C Gln N N Ribosa P Glu, PPi O CH 2 O NH 2 O HN N P OH HO Gly, ATP H C N N Ribosa ADP, Pi O C NH P NH 2 H 2C NH 2 OHC O C NH THF Ribosa P H 2C NH 10-Formil- THF THF CHO HC COO - CH + 10-Formil- THF O C N H C N NH 2 Ribos a P O C NH COO - NH 2 Ribosa P Gln, ATP COO - H C Glu, ADP, Pi CH 2 H C H N C N N NH 2 Ribosa P H 2 C NH CHO COO - O HN HC NH Ribosa P ATP N H C N Ribosa CO 2 N H C N Ribosa ADP, Pi ADP, Pi NH 2 P - OOC NH 2 P Asp, ATP Figura 4.- Vía de novo para la biosíntesis de purina (IMP) a partir de PRPP. Se representa cada una de las reacciones involucradas en el proceso (Mathews, 2002). 10
28 La generación de IMP es un punto de divergencia para la producción de AMP y GMP, iniciando la vía para la generación de nucleótidos de guanina con la hidroxilación del anillo de IMP dependiente de NAD+ dando como resultado hipoxantina o XMP la cual es transformada a GMP por la intervención de amidotransferasa dependiente de glutamina. La generación de AMP involucra la transferencia de un nitrógeno del aspartato a IMP generando el intermediario adenilsuccinato; que seguida de una reacción de eliminación α, β produce AMP más fumarato (Mathews et al., 2002) (Figura 5). O NAD +,H 2 O HN N Asp, GTP NADH+H + N N RIBOSA--P GDP, Pi HN O N - OOC H 2 C H C COO - NH O Gln ATP N N RIBOSA--P N N N N RIBOSA--P Glu ADP, Pi - OOC CH 2 O H 2 C COO - HN N NH 2 H 2 N N N RIBOSA--P N N GMP AMP N N RIBOSA--P Figura 5.- Vía de novo para la obtención de AMP y GMP a partir de IMP (Mathews, 2002). 11
29 Durante el metabolismo, los nucleótidos son activos como trifosfatos, por medio de una cascada de fosforilaciones. Enzimas como la guanilato cinasa y la adenilato cinasa generan GDP y ADP, respectivamente. El ATP es el donador del grupo fosfato para generar los trifosfatos mediante la intervención de la enzima nucleósido difosfato cinasa o NDK (Mathews et al., 2002). adenilato cinasa AMP + ATP ADP + 2ATP guanilato cinasa GMP + ATP GDP + ATP Es importante mencionar que GMP y AMP controlan su propia formación por inhibición de la síntesis de adenilsuccinato. La acumulación de GTP tiende a promover el camino para la producción de nucleótidos de adenina. La conversión de XMP a GMP es dependiente de ATP, de esta manera la acumulación de ATP puede promover la síntesis de nucleótidos de guanina (Mathews et al., 2002). Biosíntesis de novo: Nucleótidos de Pirimidinas La generación de nucleótidos de pirimidina es mucho más sencilla e involucra dos características distintivas con respecto a la generación de purinas. La primera es que el anillo pirimídinico se ensambla como una base libre para posteriormente convertirse en un nucleótido, además que esta vía no se divide o ramifica en otras vías. Esta vía inicia con la formación de carbamoil fosfato mediante la enzima carbamoil fosfato sintetasa generando carbamoil aspartato, reacción catalizada por la enzima aspartato transcarbamoilasa. Posteriormente se produce orotidina monofosfato la cual es procesada a UMP o uridina monofosfato por la acción de la enzima orotidilato descarboxilasa. La enzima 12
30 UMP cinasa y la NDK provocan la formación de UTP al cual se transfiere un grupo amino por la CTP sintetasa para dar como producto final CTP o citidin trifosfato (Figura 6). La enzima aspartato transcarbamoilasa puede ser inhibida por el producto final de la vía CTP, y puede ser activada por ATP para mantener el balance de la biosíntesis de pirimidinas (Mathews et al., 2002). Figura 6.- Síntesis de novo de nucleótidos de pirimidinas, ruta que no se ramifica además de iniciar con una base libre llamada ácido orótico (Mathews, 2002). 13
31 Biosíntesis de Desoxirribonucleótidos de Timina: Vía de novo y de Rescate La biosíntesis de desoxitimidina o dttp empieza con desoxiuridina difosfato o dudp (cuya formación se tratará más adelante). dudp es fosforilado a dutp el cual es transformado por una difosfohidrolasa (dutpasa) a dump. La formación de dump a partir de dcdp, se da por la defosforilación de dcdp para formar dcmp, el cual sufre una desaminación por la dcmp desaminasa para generar dump. En este punto se genera una ramificación donde el dump generado es utilizado como sustrato para la formación de dtmp o desoxitimidina monofosfato. La reacción anterior es catalizada por la timidilato sintasa o TS. La TS adiciona un grupo carbono el cual es donado por la 5,10- metilentetrahidrofolato, además de un par de electrones para reducirlo (Mathews et al., 2002). El dtmp formado es convertido a dttp por dos fosforilaciones consecutivas (Figura 7). 14
32 Timidilato Sintasa (TS) Timidilato Cinasa (TMK) Nucleósido Difosfato Cinasa (NDK) Figura 7.- Vía de novo y de rescate para la biosíntesis de nucleótidos de timina (Mathews, 2002). 15
33 Biosíntesis de Desoxirribonucleótidos: Reducción de Ribonucleótidos a Desoxirribonucleótidos Los ribonucleótidos tienen varias funciones en el metabolismo de la célula, mientras que los desoxirribonucleótidos son utilizados solo como constituyentes del ADN. De acuerdo a lo anterior, su producción es regulada en relación a la síntesis de ADN (Mathews, 2002). La biosíntes de los desoxirribonucleótidos inicia con la reducción de la ribosa a desoxirribosa, es decir, ocurre un reemplazo del hidroxilo del C2 por un ion hidronio. La enzima que reduce los cuatro desoxirribonucleótidos es la enzima ribonucleótido reductasa, la cual utiliza como sustratos a los ribonucleósidos difosfato. La ribonucleótido reductasa es un tetrámero α 2 β 2, el cual se divide en R1 (sitio catalítico) que corresponde a las α 2 y posee tres cisteínas y se llevan a cabo reacciones redox y R2 corresponde a β 2. R2 posee una tirosina que actúa en conjunto con R1 además de poseer un átomo de oxígeno el cual forma un puente con un centro dinuclear de fierro (Mathews, 2002). En la Figura 8 se muestra la vía en la que interviene la ribonucleótido reductasa para generar los desoxinucleótidos a partir de nucleótidos difosfato. 16
34 Figura 8.- Biosíntesis de desoxirribonucleótidos trifosfato a partir de ribonucleótido reductasa (Mathews, 2002). 17
35 Análogos Nucleosídicos Antivirales Desde hace cuatro décadas el uso de antivirales ha sido crucial para el manejo de distintas infecciones virales, incluyendo el virus del herpes simple (HSV, por sus siglas en inglés), virus de la hepatitis B (HBV), el virus de la inmunodeficiencia humana (VIH) y el citomegalovirus (CMV). Dentro de los antivirales se encuentran los derivados de nucleósidos o análogos nucleosídicos (AN). La actividad antiviral radica en que actúan inhibiendo a la ADN polimerasa viral así como a la transcriptasa reversa, enzimas que desempeñan un papel importante en el ciclo de replicación viral (De Clercq, 2009). De manera inicial, alrededor de 1960, los AN se identificaron como moléculas que actuaban en conjunto con el metabolismo de una batería de fármacos utilizados para el tratamiento de cáncer, ya que actuaban como antimetabolitos interfiriendo con el crecimiento de las células cancerígenas. El efecto terapéutico de los análogos nucleosídicos de manera invariable, provocaba daño a las células del hospedero, efecto secundario, que se creía que podía evitarse durante la eliminación del fármaco del cuerpo de los pacientes. Con la finalidad de disminuir los efectos secundarios se llevaron a cabo experimentos que incluían la modificación de la base heterocíclica así como la cavidad del azúcar por lo que se clasifican en dos grupos: análogos nucleosídicos fosforilados y análogos nucleosídicos no fosforilados (Deville- Bonne et al., 2010). En la Figura 9 se muestran 2 análogos nucleosídicos y sus análogos naturales. 18
36 Figura 9.- Estructura química de análogos nucleosídicos (a, c) y desoxinucleótidos (b, d) (Deville-Bonne et al., 2010). 19
37 Análogos nucleosídicos no fosforilados (AN).- la generación de este tipo de fármacos se inició con el descubrimiento de (S)-9-(2,3-dihidroxipropilo) adenina ó DHPA. DHPA fue utilizado para tratar las ámpulas generadas durante el resfriado y fue considerado como un antiviral de amplio espectro; pero en realidad poseía una actividad muy débil frente varios virus, independientemente de su composición química. El mecanismo de acción se basa en su composición análoga a la adenosina, ya que éste ocupa el sitio de unión a adenosina en el sitio catalítico de la S-adenosil-L-homocisteína hidrolasa, enzima que participa en la regulación de metilaciones de la S-adenosil-Lmetionina (De Clercq, 2002). Este antiviral duró poco en el mercado como pomada para tratamiento tópico, sin embargo, fue una gran herramienta para investigaciones subsecuentes. Este tipo de fármacos tienen como blanco la ADN polimerasa viral y actúan como terminadores de la replicación. Para lograr lo anterior, primero tiene que ser fosforilado por varias enzimas cinasas de manera consecutiva para generar un metabolito activo trifosforado (De Clercq, 2009). La primera fosforilación generalmente la realizan enzimas virales, acción que permite que este tipo de fármacos sea dirigido a las células infectadas. Las fosforilaciones consecutivas se llevan a cabo por enzimas celulares como GMP (guanilato cinasa) y NDK (nucleósido difosfato cinasa). El aciclovir (ACV) es el estándar de oro dentro de los análogos nucleosídicos no fosforilados, y sirve para tratar el virus del herpes simple tipo 1 y 2 (HSV-1 y HSV-2, respectivamente). El mecanismo de acción descrito por Gertrude Elion se representa en la Figura 10 (De Clercq, 2001, 2002, 2009, De Clercq & Holy, 2005, Elion, 1983). 20
38 Figura 10.- Esquema representativo del mecanismo de acción de aciclovir (ACV), análogo nucleosídico de la guanina. Se muestran los pasos de fosforilación consecutiva que genera un metabolito trifosforado el cual termina con la replicación viral (De Clercq & Holy, 2005). 21
39 Análogos nucleosídicos fosforilados (ANP).- El mecanismo de acción de los análogos nucleosídicos fosforilados, a diferencia de los análogos nucleosídicos clásicos como zidovudine, aciclovir o penciclovir, no requiere de la primera fosforilación para su activación, la cual es catalizada por una nucleósido cinasa. En las células donde esta enzima es menos activa o que no la poseen, los análogos nucleosídicos no fosforilados son inactivos, mientras que los análogos nucleosídicos fosforilados son activos por la participación de nucleótido monofosfato cinasas (GMP cinasa o AMP cinasa) y la nucleósido difosfato cinasa (NDK), enzimas que los convierten a la forma di- y trifosfato, respectivamente (Figura 11) (De Clercq, 2001, 2002, 2009, De Clercq & Holy, 2005). 22
40 Figura 11.- Esquema representativo del mecanismo de acción de cidofovir (S-1- (3-hidroxi-2-fosfonilmetoxipropil) citosina o HPMPC, por sus siglas en inglés. Se observa que el análogo de manera inicial, posee en sus estructura un grupo fosfato, lo cual permite las 2 fosforilaciones consecutivas para la generación del metabolito trifosfato que termina con la replicación viral (De Clercq & Holy, 2005). 23
41 Enzimas Cinasas Cinasas Que Participan en la Activación de Análogos Nucleosídicos Las enzimas son en general proteínas con una capacidad de catálisis asombrosa. Estas moléculas llevan a cabo reacciones biológicas a altas velocidades, pueden actuar bajo condiciones suaves de reacción, es decir, temperaturas por debajo de los 100 C y actúan a phs neutros. También poseen un alto grado de especificidad por su sustrato y su tipo de catálisis varía en respuesta a la concentración de otras sustancias diferentes al sustrato, es decir tienen capacidad de regulación catalítica (Alberts et al., 2010). Los fármacos antivirales, principalmente análogos nucleosídicos como el 2 -,3 -dideoxi-3- azidotimidina (AZT), aciclovir, cidofovir, etc., tienen como blanco la ADN polimerasa o la transcriptasa reversa viral. Estos fármacos deben de ser procesados a su forma trifosfato, la cual compite con los sustratos naturales para la incorporación a la cadena de ADN en síntesis. Los trifosfatos, no pueden ser utilizados como fármacos debido a su polaridad, ya que no pueden atravesar la membrana celular, por tanto las fosforilaciones ocurren de forma intracelular. La activación de los fármacos, es decir, el procesamiento de nucleósidos a nucleótidos trifosfatos involucra fosforilaciones consecutivas (Figura 12). Este proceso generalmente se lleva a cabo por enzimas intracelulares llamadas cinasas, las cuales pertenecen a la vía del salvamento; aunque este proceso también lo pueden llevar a cabo cinasas virales (De Clercq & Holy, 2005, Deville-Bonne et al., 2010). 24
42 dn BASE NTP dn cinasas dgk, dck TK1, TK2 NDP O dnmp BASE HO O - O P O O dn H H OH OH O - H H OH H H OH NMP cinasas hamok 1-6, hgmpk htmpk, hump-cmpk NTP MEMBRANA CELULAR O O dndp NDP BASE - O P O P O O O - O - H H - O O P O - O O P O - O dntp O P O O - H H OH O BASE H H OH NDP NTP H OH NDP cinasa PG cinasa Creatinina cinasa H OH Figura 12.- Representación esquemática de la fosforilación de nucleósidos naturales que participan en la vía del rescate en humanos. Paso 1 desoxinucleósido cinasas llevan a cabo la primera fosforilación, paso 2, nucleótido monofosfato cinasas llevan acabo la segunda fosforilación, paso 3 nucleótido difosfato cinasas llevan acabo la tercera fosforilación (Deville-Bonne et al., 2010). 25
43 Las cinasas de la vía del rescate y aquellas que son codificadas por genomas virales son proteínas pequeñas de alrededor de kda. La mayoría son monoméricas, aunque también existen dímeros, tetrámeros y hexámeros. No presentan un comportamiento alostérico, a excepción de desoxicitocina cinasa (dck), timidina cinasa 1 y 2 de humano (htk1 y htk2, respectivamente) las cuales presentan una cooperatividad negativa frente a sus sustratos (Munch-Petersen et al., 1991). En el caso de las desoxinucleósido cinasas, que son las que llevan a cabo la adición del primer fosfato, éstas son inhibidas por el producto trifosforado (dttp) el cual se une como un bisustrato. Las cinasas catalizan la transferencia de un grupo fosfato de un donador que normalmente es el fosfato γ del ATP a un aceptor ya sea el hidroxilo 5 de un nucleósido o el fosfato α o β de un nucleótido. La molécula donadora o aceptora se une a dos sitios distintos normalmente, en una forma no ordenada, aunque la unión del primer sustrato facilita o inhibe la unión del segundo. Existen dos excepciones a lo descrito anteriormente, uno por la nucleósido difosfato cinasa la cual tiene un solo sitio de unión para ambos sustratos y la timidina cinasa del virus del herpes simple tipo 1, el cual une primero a la timidina antes que el ATP (Perozzo et al., 2000). Nucleósido Difosfato Cinasa La nucleósido difosfato cinasa (NDK o NDP) fue descubierta en levaduras (Berg & Joklik, 1953) y en músculo de pichones (Krebs & Hems, 1953). La NDK cataliza la transferencia del fosfato γ de un nucleótido trifosfato a un nucleósido difosfato aceptor, la reacción incluye la formación de un intermediario, siendo la histidina de la enzima la que se fosforila de manera inicial. En base a lo anterior, la transferencia del grupo fosfato se lleva a cabo en dos pasos mediante el mecanismo conocido como ping-pong (Garces & Cleland, 1969). En este mecanismo los productos son liberados paso a paso. 26
44 Dos sustratos son involucrados en la reacción, el primer sustrato se une provocando la liberación de un producto y dejando a la enzima químicamente modificada. El segundo sustrato se une a la enzima modificada y es procesada para generar un segundo producto, provocando que la enzima regrese a su forma nativa. Lo anterior se puede representar de la siguiente forma: donde E* denota la enzima modificada y P y Q representan los productos (Deville-Bonne et al., 2010). Cada paso es totalmente reversible, la fosfohistidina es estable hasta por una hora en ausencia del sustrato aceptor (Deville-Bonne et al., 2010). La reacción se puede seguir utilizando ensayos espectofotométricos en combinación con enzimas acopladas o por ensayos fluorométricos. Diferentes estudios hacen referencia a que ésta enzima es no específica y los niveles de su actividad varían de acuerdo a su localización celular. La actividad de esta enzima se ha detectado en el citosol (Cheng et al., 1973), en el núcleo (Veer Reddy & Pardee, 1980), en la mitocondria (Herbert et al., 1955, Jacobus & Evans, 1977) y asociada a la membrana plasmática (Kimura & Shimada, 1988). NDK es una enzima altamente conservada ya que estudios en organismos de diferentes especies muestran que tiene un sito catalítico muy conservado con pequeñas variaciones en su estructura. Prácticamente una de las diferencias que presentan las NDKs es la asociación de su estructura cuaternaria (Lacombe et al., 2000). Datos cristalográficos de NDK de humano, específicamente la isoforma B (Webb et al., 1995) y D (Milon et al., 2000), demuestran que se encuentra como hexámeros, similar a la NDK de Drosophila melanogaster (Chiadmi et al., 1993) y de Dictyostelium discoideum (Morera et al., 1994), mientras que la NDK de Myxococcus es tetramérica (Williams et al., 1993). La estabilidad de las subunidades ya sean hexámeros o tetrámeros 27
45 radica en la forma en que se asocian las subunidades entre sí. Los tetrámeros se forman por la asociación de dos dímeros mientras que los hexámeros se asocian ya sea por un dímero de trímeros o un trímero de dímeros (Webb et al., 1995). Las interfases de los dímeros están compuestas principalmente por hojas β. El rearreglo trimérico de los dímeros involucra la participación de residuos del lazo (loop) llamado Kpn y la extensión de la cadena polipeptídica en el carboxilo terminal (Deville-Bonne et al., 2010). En el caso de las enzimas que se ensamblan como hexámeros, los aminoácidos del extremo carboxilo terminal interaccionan con el dímero vecino contribuyendo a la estabilidad del hexámero. En Myxococcus la NDK se ensambla como tetrámero, el segmento del carboxilo terminal es más pequeño e interactúa con la subunidad del mismo dímero y tiene características cinéticas particulares como su alta k cat que por lo general excede a los 1000 s -1. Esta enzima es considerada como la principal fuente en la generación de los cuatro nucleótidos trifosfato que son sustratos de la ADN polimerasa y en el caso de la generación de CTP y UTP necesarios para la ARN polimerasa. La biosíntesis de estos componentes es una función esencial en todos los organismos, lo cual la define como una enzima constitutiva. En algunos organismos como Mycoplasma, carecen del gen que codifica para NDK y la deleción en E. coli o S. cerevisiae no previene su crecimiento; lo cual implica que otras enzimas cumplen con la producción de nucleótidos y desoxinucleótidos trifosfato necesarios para la célula (Bilitou et al., 2009). En mamíferos incluyendo humanos, la NDK es el producto del gen nm23 (non-metastatic, por sus siglas en inglés) el cual incluye hasta 10 miembros. El nombre de nm23 deriva por su función de supresor de metástasis. En experimentos de líneas celulares tumorales en ratas, se detectaron niveles de ARN de nm23 mas altos en células o tumores con bajo potencial a desarrollar metástasis (Steeg et al., 1988). El gen nm23-h1 muestra similitud al gen awd de Drosophila cuyo producto es NDK A, en el caso de nm23-h2 es similar a NDK B. NDK A y B poseen 88% de identidad y características enzimáticas similares. Datos recientes reportan una interacción de NDK con caderinas, alfacateninas y plakoglobinas, que son proteínas de adhesión intracelular, 28
46 provocando la expresión elevada de NDK, lo cual sugiere que esto promueve la actividad supresora de tumores (Aktary et al., 2010). En estructuras cristalográficas disponibles en el banco de proteínas se incluyen complejos de la NDK con sustratos naturales o análogos, donde la histidina reactiva (H118 en las isoformas humanas) se encuentra en la parte inferior de la cavidad de unión, lo que permite que el nucleótido se posicione en la hendidura mediante los grupos fosfato (Figura 13). El anillo aromático de la base rodea la región hidrofóbica de las cadenas laterales pero los grupos polares no interaccionan con la proteína a excepción de la guanina, siendo el hidrógeno del grupo amino el que forma un puente con el carboxilo terminal de glutamato de la subunidad adyacente del hexámero. Lo anterior explica o define el hecho de que la NDK A y B de humano tienen una amplia gamma de sustratos (Moréra et al., 1995). 29
47 Figura 13.- Modelo tridimensional del posicionamiento de sustrato en la cavidad catalítica de NDK (Matte et al., 1998). 30
48 De manera general, la NDK es una enzima cuya actividad varía dependiendo de su localización en la célula así como en el tejido en que se encuentre, además ya se ha mencionado que participa en diferentes procesos celulares (Bilitou et al., 2009). A partir de una gran variedad de organismos hasta el momento, se han obtenido alrededor de 70 secuencias de ADNc de NDK mostrando que son enzimas conservadas. En humano se han documentado 8 genes denominados nm23-h1-nm23-h8 (Lacombe et al., 2000). Análisis filogenéticos definen que la familia de NDK se divide en dos grupos, los genes del primer grupo codifican para proteínas que generalmente tienen homólogos en vertebrados y presentan la actividad clásica de NDK. Este grupo incluye nm23-h1 a nm23-h4 y comparten entre 58 y 88% de identidad. Los genes del segundo grupo abarcan nm23-h5 a nm23-h8 que son más divergentes y presentan del 35 al 45% de identidad con el primer grupo y a excepción de nm23-h6, se encuentran predominantemente en testículos. Solo el producto del gen nm23-h6 cataliza la reacción de transferencia del fosfato-γ a partir de un donador (Lacombe et al., 2000). Cinasas humanas del grupo I.- Estudios cristalográficos las definen como hexámeros y poseen el motivo (motif) NXXHG/ASD y tienen parámetros cinéticos similares. Todas poseen nueve residuos los cuales son importantes para que se lleve a cabo la catálisis y para la estabilidad, lo cual fue definido en base a estudios de mutagénesis dirigida. nm23-h1 y nm23-h2 pueden formar homo y hetero hexámeros lo cual pudiera definir su función celular. En condiciones fisiológicas normales, participan en proliferación celular, desarrollo y diferenciación. Sin embargo, el mecanismo no ha sido del todo definido (Gilles et al., 1991). Nm23-H3 fue descrita en leucemia crónica mielogénica como la proteína que posee una extensión en el amino terminal de 17 aminoácidos a diferencia de nm23-h1 y nm23-h2 y su coexpresión está inversamente relacionada con la diferenciación. Esta característica de su amino terminal define que es una enzima citosólica aunque no se precisa exactamente el lugar. La sobreexpresión de nm23-h3 de médula ósea de ratón inhibe la diferenciación de granulocitos y provoca apoptosis, aunque varía dependiendo 31
49 de la línea celular en estudio. En condiciones normales se encuentra principalmente en cerebro, cerebelo y glándula pituitaria (Lacombe et al., 2000). Nm23-H4 contiene una extensión de 33 aminoácidos en el amino terminal, señal que le permite su localización a mitocondria (Milon et al., 1997). También una serina en la posición 129 equivalente a la mutación en el lazo (loop) Kpn en Drosophila, cuya mutación provoca la pérdida del color ciruela en los ojos de la mosca. Se sugiere que nm23-h4 se encuentra asociada como una porina, que permite el contacto entre la membrana interna y externa de la mitocondria. Cinasas humanas del grupo II.- La NDK generada de estos genes son muy diferentes entre ellas y comparadas con las del grupo I, poseen un motivo (motif) en el lazo Kpn que es X-A-R/K, aunque no es estrictamente conservado a excepción de nm23-h6 y nm23-h8. Nm23-H5 es abundante en testículos y se encuentra en bajas concentraciones en cerebro y riñón; se sugiere que está involucrada en etapas tempranas de espermatogénesis (Yelick et al., 1989). Secuencias homólogas se han encontrado en ratón y pez zebra entre otros. nm23-h6 tiene 7 aminoácidos en el amino terminal y 22 en el carboxilo terminal a diferencia de nm23-h1 y nm23-h2, y se encuentra en la mayoría de los tejidos humanos aunque en niveles bajos a diferencia de las enzimas del grupo I. El gen de nm23-h6 se ha visto implicado en una variedad de tumores malignos (Mehus et al., 1999, Tsuiki et al., 2000). Nm23-H7 posee una extensión de 85 aminoácidos en el amino terminal y carece de 13 residuos en el carboxilo terminal, se ha encontrado en hígado, corazón, cerebro, ovario, entre otros. El gen nm23-h7 se localiza dentro o cerca del locus susceptible a cáncer de próstata. La histidina catalítica es sustituida por una tirosina en nm23-h8 y en el amino terminal es homóloga a tiorredoxina y es expresada en testículos principalmente (Lacombe et al., 2000). 32
50 Nucleósido Difosfato Cinasa en Virus En el 2003 se descubrió un virus de cadena doble de ADN que infecta amibas, llamado Mimivirus; hasta el momento es el virus más grande que se conoce. Es una partícula icosahédrica de 600 nm de diámetro y posee 911 genes. Contiene genes que no han sido encontrados en otros virus. Ejemplos de lo anterior son genes que codifican para la aminoacil ARNt sintetasa, componentes del aparato de traducción, enzimas que participan en la reparación del ADN entre otras. Lo anterior sugiriere que el ciclo de vida de Mimivirus necesita componentes específicos que la célula hospedera no le provee. La identificación de la primera NDK homologa viral de Mimivirus de Acanthamoeba polyphaga (NDK apm ) fue de gran importancia ya que permitió demostrar su actividad a nivel molecular para conocer su función en el ciclo del virus y sus variaciones a nivel de secuencia con respecto a la de otros organismos (Janin et al., 2000, La Scola et al., 2003). La NDK apm es un hexámero que posee una prolina conservada en la posición 95 así como una glicina en la posición 96 en el lazo Kpn que está involucrado en la estabilidad del oligómero. Presenta mayor actividad frente a desoxipirimidinas lo cual pudiera estar relacionado con el alto contenido de A+T en el genoma del virus (72%). Durante la replicación dttp es el nucleótido limitante para ser incorporado a la cadena naciente de ADN, lo que sugiere que la NDK apm pudiera haber evolucionado con el fin de tomar ventaja de la baja concentración de dtdp en la amiba (célula hospedera). Además, el genoma del Mimivirus codifica para enzimas involucradas en la biosíntesis de dttp tales como la timidina cinasa (TK), como se mencionó previamente, la TK es responsable de la transformación de dt/du a dtmp/dump (Mathews et al., 2002, Jeudy et al., 2006) y la timidilato sintasa (TS), la cual produce dtmp a partir de dump. Además posee su propia desoxirribunocleótido monofosfato cinasa para generar cualquier nucleótido difosfato y por lo tanto poder producir dtdp a partir de dtmp. Así también produce su propia ribonucleósido reductasa, 33
51 enzima responsable de la generación de ribonucleótido difosfato a desoxirribonucleósido difosfato como dgdp y dcdp. Virus de la Mancha Blanca (WSSV) El virus de la mancha blanca afecta a los camarones peneidos y es de los más devastadores ya que provoca hasta el 100% de mortalidad del cultivo dentro de los primeros días de infección (Lightner, 1996). Desde su primer reporte en Taiwan, en 1992, se ha expandido en todo el mundo provocando pérdidas económicas en la industria camaronícola. En Sonora, de acuerdo a la COSAES (Figura 14), para el 2011 se contaba con 75 granjas infectadas a diferencia del 2010 que se tenían 106. A pesar de que disminuyó el número de granjas infectadas, la infección por el WSSV sigue siendo un problema para la camaronicultura. Figura 14.- Número de granjas afectadas por WSSV durante el periodo , información del informe final ciclo 2011, COSAES. 34
52 El WSSV es un virus con un genoma de doble cadena de ADN, de alrededor de 300 kpb, el cual codifica para 181 marcos de lectura (ORFs, por sus siglas en inglés, open reading frame) (Escobedo-Bonilla et al., 2008, van Hulten et al., 2001). Los viriones de WSSV presentan una morfología ovoide a baciliforme, con un apéndice cuya función no se conoce. El tamaño de los viriones varía entre 210 a 420 nm de largo y de diámetro de 70 a 167 nm. La envoltura viral es de 6-7 nm de grueso y presenta una estructura que asemeja a una membrana con bicapa lipídica, que posee un área entre la envoltura y la nucleocápside (Figura 15). Las dimensiones de la nucleocápside son de nm de largo y nm de diámetro indicando que se encuentra estrechamente empaquetada dentro del virión (Durand et al., 1997, Leu et al., 2009). De acuerdo a la información molecular y análisis filogenéticos de la ADN polimerasa del WSSV, se asignó a una nueva familia de virus llamada Nimaviridae, del género Whispovirus (Mayo, 2002a, 2002b, van Hulten et al., 2001). Figura 15.- Representación esquemática de la morfología de WSSV (Sánchez- Paz, 2010). 35
53 La envoltura del WSSV tiene al menos 35 proteínas diferentes, de las cuales VP28 y VP26 son las proteínas más estudiadas hasta el momento. El ORF 421 codifica para la proteína VP28 de la envoltura; varios estudios indican que esta proteína juega un papel importante en los pasos iniciales de la infección sistémica en camarón (van Hulten et al., 2001). Además, existe evidencia sobre su papel como proteína de unión a la células de camarón lo cual provoca la internalización del virus al citoplasma (Yi et al., 2004). VP28 a la vez contribuye al reconocimiento de receptores de la superficie de la célula de camarón debido a que posee varios sitios de glicosilación, aunque todavía no se ha demostrado (Tsai et al., 2004). El gen wsv 311 codifica para la proteína VP26, proteína asociada de manera inicial a la nucleocápside, donde se mostró mediante un estudio comparativo de análisis de secuencias que VP26 era idéntica a p22. La proteína p22 está localizada en el espacio entre la envoltura y la nucleocápside, que actúa como unidor, linker o proteína de unión entre éstos dos compartimentos. Es decir, el N-terminal de la VP26 (que es una región hidrofóbica) ancla la envoltura, mientras que el C-terminal (contiene un secuencia hidrofílica) está unida a la nucleocápside. VP26 es capaz de unir actina o proteínas asociadas a actina por lo que se menciona que pudiera ayudar al WSSV a desplazarse al núcleo interactuando con actina o proteínas de unión a actina celulares, con la finalidad de que el virus esté cerca del sitio de transcripción y replicación. Gracias al conocimiento generado sobre algunas proteínas en el WSSV se han generado alternativas o estrategias para el control de la infección. Sin embargo, es importante mencionar que en la actualidad no existe ningún tratamiento disponible para controlar la dispersión y la viremia ocasionada por el WSSV. 36
54 Estrategias de Control Productos naturales.- Durante los últimos años se ha incrementado el interés de utilizar productos naturales como tratamiento para el WSSV, ya que se cree, que estimulan el sistema inmune para prevenir infecciones virales. Algunos compuestos como el metanol extraído de hierbas como Cynodon dactylon, Aegle marmelos, etc., ha sido utilizado como tratamiento de camarones infectados. En camarones tratados con extractos metabólicos de C. dactylon, incrementaron su supervivencia, presentando la actividad antiviral más fuerte. Otro ejemplo importante a mencionar es Pongamia pinnata, cuyos extractos de la planta originaria de la India, fueron exitosos para lograr una supervivencia de los camarones de hasta un 80% posterior a 15 días de infección (Rameshthangam & Ramasamy, 2007). Tratamiento a base de vacunas virales.- La literatura reporta que los crustáceos al igual que otros invertebrados carecen de un sistema inmune adaptativo verdadero; por lo que su defensa se basa en mecanismos montados por la respuesta inmune innata. Por lo anterior, el desarrollo de vacunas en contra del WSSV no ha sido fácil, es decir, la falta de un sistema inmune de memoria así como el poco conocimiento sobre la etiología de la enfermedad han sido determinantes. Sin embargo, hay un caso que rompe con el esquema anterior donde se encontraron camarones (P. japonicus) sobrevivientes a un brote por WSSV 4 meses después del evento. En este caso se reportó una supervivencia del 94% sugiriendo que la supervivencia de los organismos previamente infectado desarrollaron una respuesta inmune (Venegas et al., 2000). Estudios posteriores asocian semejante resistencia a un factor de neutralización presente en el plasma del camarón, aunque no caracterizado aún, sugiriendo que la respuesta inmune adaptativa en el camarón pudiera existir sin que se haya demostrado aún su mecanismo (Wu et al., 2002). Varios estudios utilizan proteínas recombinantes de la envoltura de WSSV con la finalidad de inducir una resistencia en el camarón. La inoculación 37
55 de P. japonicus con rvp26 mostró mayor resistencia a WSSV, mientras que en los organismos inoculados con rvp28 se observó 4% de disminución en la mortalidad (Namikoshi et al., 2004). En la literatura se les llama vacunas a estas proteínas recombinantes, haciendo analogía a los antígenos que desencadenan la respuesta inmune en animales superiores, aun cuando estrictamente no lo son. Es importante mencionar que el tratamiento a base de vacunas es prometedor como una estrategia de control; sin embargo, se requiere demostrar los mecanismos de dicha protección. Tratamiento a base de vacunas con ADN/ARN.- Este tipo de estrategia es de las más novedosas para el tratamiento de infecciones virales, donde puede contribuir en la activación del sistema inmune de los organismos inclusive; ya han sido probadas en una variedad de organismos. El ARN de doble cadena (ARNdc) actúa como una molécula que dispara la respuesta celular del hospedero frente a una infección viral. En crustáceos, específicamente en P. vannamei, hay evidencia sobre la inducción de respuesta frente al virus de Taura y WSSV con vacunas de ARNdc. De acuerdo a Robalino et al. (2004), camarones infectados con WSSV que fueron tratados con ARNdc (VP19) mostraron mayor protección a diferencia de camarones tratados con ARNdc distintos, lo cual indica que los camarones pueden utilizar protección patógeno-específica utilizando sistemas de ARNdc. Resultados similares se obtuvieron con ARNdc correspondientes a VP28 y VP281 en P. chinensis (Sánchez-Paz, 2010). Estudios recientes demuestran que camarones infectados con WSSV presentan protección de hasta 50 días gracias a la utilización de vacunas con plásmidos de ADN recombinante que codifican para la proteínas VP28 o VP281. Aquellos organismos vacunados con ADN recombinante correspondiente a VP28 mostraron niveles altos en la producción de superóxido dismutasa así como profenol oxidasa lo cual pudiera estar involucrado en el desarrollo de la resistencia contra el WSSV (Rajesh Kumar et al., 2008). Plásmidos que contienen información de proteínas correspondientes a la nucleocápside (VP15 y VP35) no generaron ningún tipo de respuesta, dato 38
56 que se puede relacionar con el hecho de que la nucleocápside confiere protección al virus (van Hulten et al., 2001). De acuerdo a lo mencionado se puede decir que existen diferentes alternativas para el tratamiento de WSSV; sin embargo, hasta el momento no existe ninguna que sea definitiva. Además la cantidad disponible de ribo- y desoxinucleósido cinasas varía en las células y en todos los organismos. Los virus son capaces de redirigir el metabolismo de la célula hospedera de acuerdo a lo observado en bacterias infectadas por fagos; donde la nucleósido difosfato cinasa (NDK) es una de las principales enzimas del hospedero que provee una fuente de precursores necesarios para la síntesis de ADN viral (Mathews et al., 2002). 39
57 JUSTIFICACIÓN El camarón blanco (Litopenaeus vannamei) posee un gen cuya secuencia nucleotídica codifica para la enzima nucleósido difosfato cinasa (NDK), la cual no había sido caracterizada con técnicas bioquímicas y biofísicas. Esta enzima pudiera ser blanco del virus de la mancha blanca (WSSV) para poder llevar a cabo su replicación, debido a la función clave de NDK en la síntesis de nucleótidos. Por lo que es de gran importancia la investigación basada en el conocimiento molecular y catalítico de NDK de camarón blanco (L. vannamei) frente a sustratos específicos. 40
58 HIPÓTESIS La enzima nucleósido difosfato cinasa (NDK) del camarón blanco (L. vannamei) es capaz de fosforilar los cuatro desoxinucleótidos difosfatos (dadp, dtdp, dcdp, dgdp) con la misma afinidad, lo cual es consistente con los parámetros termodinámicos y el anclaje (docking) de los mismos en el sitio activo de la enzima. 41
59 OBJETIVO GENERAL Obtener la nucleósido difosfato cinasa (NDK) recombinante del camarón blanco (L. vannamei) y evaluar su actividad enzimática y unión a desoxinucleótidos difosfato. OBJETIVOS PARTICULARES 1.- Obtener la nucleósido difosfato cinasa (NDK) recombinante de camarón blanco (L. vannamei) y en base a su contenido de 6 histidinas purificarla mediante cromatografía de afinidad a metales. 2.- Determinar la actividad enzimática de la nucleósido difosfato cinasa (NDK) frente a sustratos específicos (dadp, dcdp, dgdp, dtdp) utilizando un ensayo enzimático acoplado. 3.- Determinar la estructura cuaternaria de la nucleósido difosfato cinasa (NDK) por cromatografía de exclusión molecular. 4.- Determinar parámetros termodinámicos, Kasoc., ΔH, ΔS, ΔG, de la interacción de nucleósido difosfato cinasa (NDK) con dadp, dcdp, dgdp, dtdp mediante calorimetría de titulación isotérmica (ITC). 5.- Elaborar un modelo tridimensional por métodos computacionales (in silico) de nucleósido difosfato cinasa (NDK) unido a dadp, dcdp, dgdp, dtdp. 6.- Determinar condiciones experimentales para la generación de cristales de nucleósido difosfato cinasa (NDK) en complejo binario con dgdp. 42
60 MATERIALES Y MÉTODOS Reactivos Los reactivos utilizados fueron de grado analítico, bioquímico o de biología molecular según se requirió y fueron adquiridos de las firmas Sigma- Aldrich, Merck, J.T. Baker y USB. Producción de la Proteína Recombinante NDK de L. vannamei Se partió de la secuencia de aminoácidos depositada en el banco de genes con la clave de acceso DQ (Clavero-Salas et al., 2007). La secuencia codificante fue optimizada y producida sintéticamente en el vector pjexpress414 (DNA 2.0, U.S.A) con la finalidad de obtener una expresión óptima en Escherichia coli. Se agregó una etiqueta de histidinas en el constructo y un sitio susceptible a corte por proteasas (PreScission Protease). El constructo fue transformado en células químicamente competentes (E. coli BL21-SI, NOVAGEN, USA) las cuales fueron previamente transformadas con un plásmido bacteriano que contenía las chaperonas dnak, dnaj, grpe y el complejo groes/el (pgjke8, Takara, USA). El vector pjexpress 414 contiene un marcador de resistencia a ampicilina y un promotor T7 inducible por β-d-1- tiogalactopiranosido (IPTG). Las bacterias transformadas se crecieron en medio LB sin NaCl a 37 C en presencia de ampicilina y cloranfenicol a 50 µg/ml y 20 µg/ml respectivamente. Al alcanzar la densidad óptica de 0.6 a 600 nm, el 43
61 cultivo fue inducido con IPTG 1 mm para inducir la expresión de NDK y la T7 ARN polimerasa con cloruro de sodio 0.3 M y 5 mg/ml de L-arabinosa con la finalidad de inducir la expresión de las chaperonas. La inducción se llevó a cabo durante 5 horas y la biomasa bacteriana fue colectada por centrifugación a 5,000 x g durante 15 minutos a 4 C y el lavado de la misma fue con cloruro de sodio al 0.9% bajo las mismas condiciones. La biomasa obtenida a partir de 1L de cultivo se almacenó congelada a - 80 C hasta su uso. La biomasa fue resuspendida en buffer lisis (5 ml por gramo de biomasa), el cual contenía Na 2 HPO 4 20 mm ph 7.5, NaCl 0.5 M, DTT 1 mm, PMSF 0.5 mm y lisozima de clara de huevo 0.1 mg/ml concentración final. El lisado bacteriano se colocó en un baño con agua y hielo, para mantener la temperatura a 0 C, y ahí mismo se sonicó con 4 pulsos de 15 segundos con pausas de 1 min. El sonicado se clarificó mediante centrifugación a 35, 000 x g durante 30 minutos a 4 C. La proteína de interés se encontró en la fracción soluble, y el precipitado se desechó. La cinética de expresión se evaluó en un gel de poliacrilamida al 15% en condiciones desnaturalizantes y reductoras comparando la fracción soluble de las bacterias antes y después de inducir el cultivo. Purificación de NDK de L. vannamei Mediante Cromatografía de Afinidad a Metales, (IMAC) El clarificado obtenido por centrifugación se trató con sulfato de estreptomicina al 10% (peso/volumen), se mezcló con agitación magnética suave en un baño de agua con hielo y se centrifugó a 35,000 x g durante 30 minutos a 4 C. El sobrenadante contenía la proteína de interés, el cual se cargó a una columna His-Trap FF de 5 ml (GE Healthcare, USA) previamente equilibrada con (10 volúmenes de columna) búfer de fosfato de sodio 20 mm ph 7.4, cloruro de sodio 500 mm. La cromatografía se realizó en un equipo 44
62 automático (AKTA, GE LifeSciences Pharmacia). La NDK de camarón fue recuperada en el búfer de equilibrio adicionando imidazol 500 mm. Las fracciones obtenidas fueron de 3 ml y se analizaron en un gel de poliacrilamida al 15% en condiciones desnaturalizantes y reductoras. El gel fue teñido con nitrato de plata (Laemmli,1970). Ensayo de Actividad Específica de NDK de L. vannamei Frente a Desoxinucleótidos Difosfato Mediante Ensayo Enzimático Acoplado La actividad de NDK de L. vannamei se determinó siguiendo la oxidación de NADH a NAD + a 340 nm utilizando el ensayo enzimático acoplado previamente descrito (Kandeel & Kitade, 2010a). El volumen total de reacción fue de 1.5 ml, la mezcla contenía Tris-HCl 50 mm ph 7.5, ATP 2 mm, 5 µg de NDK de L. vannamei, MgCl 2 5 mm, KCl 10 mm, NADH 0.14 mm, 2 unidades de fosfoenolpiruvato (PEP), 1.3 unidades de piruvato cinasa (PK) y 2.3 unidades de lactato deshidrogenasa (LDH). La reacción se inició al adicionar 0.5 mm del deoxinucleótido difosfato (dtdp, dadp, dcdp o dgdp). Una unidad se define como la conversión de 1 µmol de sustrato por segundo. Determinación de la Estructura Cuaternaria de NDK de L. vannamei Mediante Cromatografía de Exclusión Molecular Se determinó el estado oligomérico de NDK de L. vannamei utilizando una columna Superdex 200 (GE Healthcare), la cual fue previamente equilibrada con estándares moleculares. Los marcadores se prepararon en búfer de fosfatos 20 mm ph 7.4, NaCl 200 mm y 5% de glicerol. Los marcadores utilizados fueron albúmina (66 kda), ovoalbúmina (44 kda) además 45
63 de inhibidor de tripsina de soya (21.5 kda). La cromatografía se realizó en un equipo automático (AKTA, GE LifeSciences Pharmacia). Determinación de Parámetros Termodinámicos de NDK de L. vannamei Frente a Desoxinucléotidos Difosfatos Mediante Calorimetría de Titulación Isotérmica Para determinar los parámetros de unión (binding) de NDK de L. vannamei frente a desoxinuclótidos difosfatos, se utilizó un microcalorímetro VP-ITC (Microcal, Inc., USA). 1.4 ml de NDK de L. vannamei con una concentración de 30 µm fue titulada con soluciones 1 mm de cada uno de los desoxinucleótidos difosfatos (dgdp, dcdp, dadp y dtdp). Todas las soluciones de NDK de L. vannamei fueron preparadas utilizando búfer diálisis (Tris-HCl 50 mm ph 7.5, MgCl mm). Cada uno de los experimentos se llevó a cabo a 30 C, la velocidad de agitación fue de 249 RPM. Se llevaron a cabo inyecciones de 10 µl con un espaciamiento de 300 s con la finalidad de asegurar el equilibrio entre cada inyección. El calor de dilución se determinó en base a dos experimentos, primero; el búfer diálisis de NDK fue titulado con cada nucleótido difosfato, segundo; la NDK fue titulada con búfer diálisis. Los datos fueron ajustados al modelo de unión a un solo sitio, donde se arrojan de forma directa y se obtiene la entalpía de unión ( H), la constante de asociación (Ka) y la estequiometría de unión (n). De manera indirecta mediante sustitución en la fórmula G = RT lnk se obtiene la energía libre de unión ( G) y los cambios en la entropía ( S) se obtuvieron de la fórmula G = H-T S. 46
64 Modelamiento in silico y Anclaje de NDK de L. vannamei El modelo teórico de la estructura molecular se realizó en base a la identidad que presenta a nivel de secuencia aminoacídica con la NDK de humano, dicha estructura se encuentra depositada en el PDB con la clave de acceso: 2HDV (Giraud et al., 2006). El modelamiento y anclaje se llevó a cabo utilizando el programa MOE versión (Chemical Computing Group, Canada). El modelamiento molecular se inició con alineamientos múltiples de la secuencia de NDK con la finalidad de mejorar el modelo. Un total de 25 intermediarios se generaron bajo el campo de fuerza CHARMM27, el cual incluyó el ligando de ATP de la estructura templado. El modelo final fue refinado con los parámetros estándar del programa MOE. El anclaje de los desoxinucleótidos al sitio activo de la enzima fue utilizado para predecir la forma de unión y así poder correlacionarlo con la afinidad de la enzima hacia los mismos en base a su estructura. Para el anclaje, de manera inicial se eligió la conformación cerrada del sitio de unión de los nucelótidos de la NDK de L. vannamei sin seleccionar los residuos de Mg ++ en el sitio de unión. Con la finalidad de obtener convergencia de las mejores posiciones de unión de ligando-ndk de L. vannamei se llevaron a cabo tres corridas estocásticas, independientes al anclaje. El método de triángulo-alfa se utilizó para determinar la orientación de los nucleótidos hacia el sitio activo de la enzima. Partiendo de 60,000 posiciones colectadas, solo 30 fueron retenidas de acuerdo a las funciones estándares aplicadas por MOE. Estas posiciones fueron sujetas a un paso de refinamiento con la finalidad de relajar las posiciones en el sitio activo, las cuales fueron seleccionadas de acuerdo a la función dg-scoring, la cual estima la contribución entálpica a la energía libre de la unión de las interacciones predichas en el posicionamiento del anclaje. 47
65 Determinación de Condiciones de Cristalización del Complejo NDK-dGDP Se utilizó el kit comercial de cristalización de Hampton Research (Laguna Niguel, USA) Crystal Screen 1 y se utilizó la técnica de Microbatch. Se utilizaron distintas concentraciones de proteína (7, 10 y 13 mg), mezclando un microlitro de la solución de proteína más un microlitro de la solución de precipitación. Las placas se incubaron a 16 C y se hicieron observaciones cada semana con la finalidad de encontrar un cristal definido. Los cristales formados fueron recolectados en una micro-asa especial la cual se introdujo en la solución crioprotectante e inmediatamente fueron superenfriados en nitrógeno líquido para ser difractados posteriormente. 48
66 RESULTADOS Y DISCUSIÓN En este trabajo la co-expresión de NDK de L. vannamei con chaperonas permitió obtener la proteína recombinante de manera soluble (Figura 16), la cual fue purificada en un solo paso utilizando una columna de afinidad a metales o IMAC (por sus siglas en inglés) His-trap FF (Figura 17). Reportes en la literatura mencionan que las NDKs son enzimas oligoméricas donde su actividad es independiente de su estructura cuaternaria. Varias estructuras cristalográficas y estudios bioquímicos indican que en el caso de eucariotes como Drosophila melanoganster, Homo sapiens y Mus musculus las NDKs se encuentra formando hexámeros y en el caso de procariotas como Myxococcus xhantus se encuentra como trímero. En el caso de Salmonella typhimurium o E. coli se encuentra formando tetrámeros (Lascu et al., 2000, Williams et al., 1993). 49
67 Figura Análisis electroforético de la cinética de sobreexpresión de NDK de L. vannamei. MPM: marcador de peso molecular de amplio rango, A.I. sobrenadante antes de inducción, 5 h D.I. : sobrenadante obtenido 5 horas después de la inducción con IPTG. Las bandas de alrededor de 14 kda corresponden a lisozima de huevo utilizado para lisar bacterias. El gel fue teñido con azul de Coomasie R
68 kda MPM E.C. Fr lavado Fracciones obtenidas durante elusión 21.5 NDK Figura 17.- Análisis electroforético de fracciones provenientes de la purificación de NDK recombinante de L. vannamei mediante IMAC. MPM: marcador de peso molecular de amplio rango; E.C.: extracto crudo del clarificado de E. coli, Fr lavado: fracción que no se unió a la columna; fracciones obtenidas durante la elusión: proteína recuperada en un gradiente de imidazol de 50 a 150 mm.. 51
69 La actividad específica de la enzima se determinó mediante un ensayo enzimático acoplado y se utilizaron como sustratos cuatro desoxinucleótidos difosfato, dadp, dgdp, dtdp y dcdp. Se encontró que la NDK de L. vannamei es activa frente a todos los dndps, donde la mayor actividad la mostró frente a dtdp, seguida de dadp, dcdp y dgdp (Tabla 1). Pocos datos sobre la actividad específica de NDKs frente a los cuatro dndps han sido publicados. En el caso de la NDK mitocondrial recombinante de chícharo (Pisum sativum), ésta mostró una actividad específica de 2,700 U/mg frente a dcdp siendo 3.6 veces más grande el valor de NDK de L. vannamei frente a éste sustrato (9,648 U/mg) (Quintero-Reyes et al., 2012) (Johansson et al., 2004). La actividad específica de NDK mitocondrial de chícharo frente a dtdp fue de 5,100 U/mg mientras que la actividad de NDK de L. vannamei frente al mismo nucleótido difosfato es 3.8 veces mayor (19,344 U/mg). Valores frente al dtdp de NDK s como Dictyostelium discoideum y de humano (Nm23-H2) presentan valores menores que los de chícharo (2200 U/mg y 731 U/mg respectivamente) (Mesnildrey et al., 1998, Postel et al., 2002). La actividad de NDK de L. vannamei frente a dadp y dgdp fue de 14, 626 U/mg y 5,505 U/mg, respectivamente. 52
70 Tabla 1.- Evaluación de la actividad específica de NDK recombinante de L. vannamei frente a desoxinucleótidos difosfato. Actividad de Nucleósido Difosfato Cinasa después de su purificación por IMAC SUSTRATO U/mg de proteína dtdp 19, 344 dadp 14, 626 dcdp 9, 648 dgdp 5,505 53
71 La actividad de las enzimas está relacionada con la estabilidad de su estructura cuaternaria lo cual ha sido demostrado mediante estudios de mutagénesis, las NDKs comparten similitud en su estructura primaria, secundaria y terciaria pero difieren en su estructura cuaternaria. En este trabajo se determinó la asociación multimérica de NDK de L. vannamei utilizando cromatografía de exclusión molecular (Superdex 200) donde la proteína eluyó en una sola fracción (Figura 18); es decir no muestra la presencia de otras especies, revelando que se encuentra como un trímero de 66 kda. En la Tabla 2 se presentan algunas diferencias en la estructura cuaternaria de NDK de diferentes organismos, básicamente se reflejan los hexámeros en eucariotas aunque a la vez se presentan como tetrámeros como el caso de NDK de eritrocitos de humano de acuerdo a (Stoeckler et al., 1978). 54
72 Figura 18.- Cromatograma obtenido a partir de cromatografía de exclusión molecular. El peso molecular nativo de la NDK de L. vannamei fue de 66 kda de acuerdo a la calibración con estándares moleculares, indicando que es un trímero. 55
73 La estructura cuaternaria está relacionada con la estabilidad de las enzimas y una de las primeras evidencias se obtuvo al analizar las mutantes en el lazo (loop) Kpn de Drosophila. La mutación puntual de P97S en NDK generó un fenotipo letal (Timmons & Shearn, 1997). Se observó que la estabilidad de la enzima disminuyó frente a diferentes temperaturas y concentraciones de urea en comparación con la NDK nativa. Al momento de llevar a cabo el replegamiento, la NDK solo generaba monómeros, lo cual indica que la mutación afecta el ensamblado de las subunidades (Lascu et al., 1992). La misma mutación en Dictyostelium discoideum mostró una sutil modificación en la interacción entre las subunidades al igual que la NDK de mitocondria de humano, provocando que frente a la desnaturalización con urea, la ausencia de la prolina, que es un residuo conservado, incrementa la estabilidad de la enzima (Milon et al., 2000). Análisis de la mutación P105G en la NDK de Dictyostelium discoideum mostró que la estructura cuaternaria era idéntica a la que muestra la estructura nativa con la excepción de la ausencia de la prolina en la cadena lateral, lo cual provocó la disminución del área de interacción entre las áreas no polares de la cadena. Aunque de manera inicial este cambio es pequeño, afecta en gran magnitud a la estabilidad de la enzima (Lascu et al., 1993). La Figura 19 muestra el modelo molecular de la NDK de L. vannamei el cual predice que es un trímero de acuerdo a la alta identidad que muestra en alineamiento con su homóloga en humano nm23-h1. 56
74 Figura 19.- Modelo estructural de NDK de L. vannamei obtenido mediante el paquete computacional MOE versión Se utilizó como templado nm23-h1 PDB 3L7U. 57
75 Cada monómero de NDK de L. vannamei posee un dominio α/β con dos hélices α a un costado de la hoja β, la cual es estabilizada por interacciones hidrofóbicas. En la Tabla 2 se muestran algunas diferencias de la estructura cuaternaria de NDK de distintos organismos. En organismos como H. sapiens, D. melanogaster, M. musculus por mencionar algunos se encuentra en forma de hexámero (Webb et al., 1995). Pocos organismos presentan una estructura tetramérica como el caso de E. coli, y trimérica como X. laevis, donde el ensamblado de los monómeros en los diferentes tipos de estructura cuaternaria es idéntico. La asociación de cada monómero influye en la formación de la estructura cuaternaria, es decir, tres dímeros se asocian para generar un hexámero y dos dímeros se asocian por interacción en una región específica de la superficie del dímero contiguo para generar un tetrámero. En el caso de los residuos que forman parte de la interfase (dímero-dímero) involucrados en la formación del hexámero han sido bien conservados durante la evolución. 58
76 Tabla 2.- Estructura cuaternaria de NDK en diferentes organismos. Organismo Tejido o Recombinante Estructura Cuaternaria Humano (MI) Eritrocitos Hexámero Xenopus laevis Trímero-Tetrámero Myxococcus xanthus Recombinante Trímero Dictyostelium discoideum Recombinante Hexámero Drosophila melanogaster Hexámero Modificado de (Lascu et al., 2000). 59
77 Diferentes residuos juegan un papel importante en el ensamblado de las subunidades, en el caso de NDK de L. vannamei no se han definido por la estructura cristalográfica como los ejemplos mencionados anteriormente, sin embargo se llevó a cabo una predicción in silico de la interacción de los cuatro desoxinucleótidos en el sitio activo de NDK o anclaje con los desoxinucleótidos difosfato (docking). De acuerdo al anclaje, la H117 que es el residuo encargado de la transferencia del grupo fosfato es conservado así como S119 y E128 (Figura 20). La presencia de otros residuos como K11 y N114 son importantes en la unión con el OH 3 de la ribosa, en el caso de F59 participa en el posicionamiento del anillo de la base, así como otros aminoácidos básicos que alinean la entrada de los fosfatos. En el modelo se observa que tanto desoxirribonucleótidos de purina y pirimidinas embonan en el sitio activo de la enzima (Janin et al., 2000), pero se ha reportado que la desoxicitidina muestra una pobre afinidad (Kandeel & Kitade, 2010b). 60
78 Figura 20.- Modelo teórico del anclaje (docking) entre NDK de L. vannamei y desoxinucleótidos difosfato. a) Interacción de dgdp y el sitio activo, se observa la interacción favorable entre el anillo de la purina y V111, F59 y L63. b) La interacción con dadp inclusive dtdp (c) se observa la misma interacción aunque L63 no participa en la interacción. d) Se encontraron varios confórmeros durante el experimento lo cual es consistente con la falta de interacción en ITC. 61
79 Las constantes de afinidad de NDK frente a los nucleótidos, así como los parámetros termodinámicos ΔG, ΔH y ΔS, se determinaron mediante calorimetría de titulación isotérmica (Tabla 3). Dentro de los nucleótidos estudiados dgdp mostró la mayor constante de afinidad (K d = 7.6 µm), mientras que dadp y dtdp muestran valores menores de constante de afinidad, muy similares entre ambos, K d = 14.6 µm y 13.7 µm, respectivamente. dcdp no generó calor de interacción, a pesar de haber presentado una actividad enzimática de 9,648 U/mg proteína. Lo anterior puede explicarse si se considera que en este caso la formación del complejo enzima-sustrato es debida exclusivamente a la entropía de la interacción, presentando una entalpía muy cercana a cero, con lo cual el calor liberado o absorbido durante la reacción de complejación no es detectado por el equipo. Por otra parte, para todos los nucleótidos estudiados a excepción de dcdp la formación de los complejos con NDK presentan entalpías y entropías favorables. En el caso de dgdp la principal contribución a la estabilidad del complejo es del factor entálpico, con un ΔH = -4.4 kcal/mol que a su vez es el mayor valor de entalpía para los nucleótidos estudiados; de estos datos se infiere que dgdp presenta un mayor número de enlaces de hidrógeno en el sitio activo. Los valores del cambio de entalpía para dadp (ΔH = -2.7 kcal/mol) y dtdp (ΔH = -2.2 kcal/mol) son menores en comparación con dgdp, lo cual indica que, en los respectivos complejos, hay un menor número de enlaces de hidrógeno formados entre el sitio activo de la enzima y el sustrato. Por otra parte, conforme en la serie de nucleótidos estudiados la entalpía se torna menos favorable, la entropía se vuelve mas favorable: dgdp(tδs=2.7kcal/mol)<dadp(tδs=4.0kcal/mol)<dtdp(tδs=4.6kcal/mol), fenómeno conocido como compensación entalpía-entropía, en cual conforme uno de los parámetros termodinámicos se vuelve mas favorable, el otro tiende a ser menos favorable. Esto es posible, ya que a mayor número de enlaces formados (entalpía mas favorable) el complejo debe ser mas rígido, con el consecuente costo entrópico; por otra parte, menor número de enlaces implica menos rigidez (o mayor flexibilidad) y por lo tanto menor costo entrópico. En 62
80 nuestro caso de estudio, la formación de un mayor número de enlaces de hidrógeno proporciona una mayor estabilidad del complejo enzima sustrato, a pesar del costo entrópico que ello ocasiona, por lo cual NDK presenta una mayor afinidad hacia dgdp. 63
81 Tabla 3.- Parámetros termodinámicos y constantes de unión de NDK de L. vannamei frente a desoxinucleótidos difosfato obtenidos mediante calorimetría de titulación isotérmica. Ligando ΔH TΔS ΔG K d (µm) kcal/mol kcal/mol kcal/mol dgdp dadp dtdp dcdp sin calor de interacción detectable 64
82 De acuerdo a los datos anteriores, la estabilidad del complejo de NDK con dgdp permitió la generación del análisis de condiciones de cristalización a partir de un total de 190 soluciones de un kit comercial de Hampton Research, Laguna Niguel, USA. Se obtuvieron cristales del complejo binario (NDK-dGDP) en la solución 19 del kit 1 (Crystal screen 1) el cual contiene 0.2 M de acetato de magnesio tetrahidratado, ph 6.5 y PEG 8000 al 20% (Figura 21). Los resultados de la difracción de dichos cristales generarán información básica e importante sobre las bases moleculares de la interacción NDK-dGDP, lo cual será clave para la generación de un fármaco dirigido a WSSV. 65
83 Figura 21.- a) Cristales obtenidos a los 30 días en 0.2 M de acetato de magnesio tetrahidratado, ph 6.5 y PEG 8000 al 20%, (NDK-dGDP). b) Cristal roto utilizando un cabello de cola de caballo, lo cual indica que el cristal es de proteína y no de sal. Ambos corresponden a el complejo binario NDK dgdp. 66
84 CONCLUSIONES Se obtuvo la enzima nucleósido difosfato cinasa (NDK) de L. vannamei de forma soluble a partir de un gen sintético, el cual fue construido a partir de la secuencia depositada de la misma en el banco de genes con la clave de acceso DQ Lo anterior fue corroborado de acuerdo al análisis electroforético de la cinética de sobreexpresión al comparar la muestra antes y posterior a la inducción. Se purificó la enzima nucleósido difosfato cinasa (NDK) de L. vannamei mediante cromatografía de afinidad a metales (IMAC). Se obtuvo un rendimiento protéico de 10 mg/ml a partir de un litro de cultivo. En base a los resultados arrojadas del análisis in silico, donde se utilizó como templado nm23-h1 (PDB 3L7U), se identificó a la enzima nucleósido difosfato cinasa (NDK) de L. vannamei como un trímero; lo cual fue consistente con los resultados obtenidos mediante cromatografía de filtración en gel. Los resultados del anclaje (docking) permitieron hacer la simulación de la interacción de los desoxinucleótidos difosfato (dadp, dcdp, dtdp y dgdp) con el sitio activo de nucleósido difosfato cinasa (NDK) de L. vannamei y se definieron los residuos que intervienen en la catálisis. Además se observó que dcdp, de acuerdo al refinamiento, el comportamiento del anclaje no fue consistente con los desoxinucleótidos difosfato restantes. Los parámetros termodinámicos de nucleósido difosfato cinasa (NDK) de L. vannamei frente a los desoxinucleótidos difosfato (dadp, dcdp, dtdp y 67
85 dgdp) definen que la interacción de NDK con dgdp presenta la mayor constante de afinidad con respecto a los desoxinucleótidos difosfato restantes. dadp muestra una constante de disociación comparable con dgdp aunque la unión del complejo es guiado por la entropía. dcdp no muestra calor de interacción por lo que pudiera deberse a un valor bajo en la entalpía, que no es detectado, definiendo que la interacción es guiada por la entropía. Lo anterior es consistente con los resultados de anclaje (docking). 68
86 REFERENCIAS Aktary, Z., Chapman, K., Lam, L., Lo, A., Ji, C., Graham, K., Cook, L., Li, L., Mackey, J. & Pasdar, M. (2010). Oncogene 29, Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., Walter, P., BRAY, D. & WATSON, J. (2010). Classic textbook now in its 5th Edition. Berg, P. & Joklik, W. (1953). Nature 172, Bilitou, A., Watson, J., Gartner, A. & Ohnuma, S. (2009). Molecular and cellular biochemistry 329, Cheng, Y. C., Robison, B. & Parks Jr, R. (1973). Biochemistry 12, Chiadmi, M., Morera, S., Lascu, I., Dumas, C., Bras, G. L., Veron, M. & Janin, J. (1993). Structure 1, Clavero-Salas, A., Sotelo-Mundo, R. R., Gollas-Galván, T., Hernández-López, J., Peregrino-Uriarte, A. B., Muhlia-Almazán, A. & Yepiz-Plascencia, G. (2007). Fish & shellfish immunology 23, De Clercq, E. (2001). Journal of Pharmacology and experimental Therapeutics 297, De Clercq, E. (2002). Nature Reviews Drug Discovery 1, De Clercq, E. (2009). Med Res Rev 29, De Clercq, E. & Holy, A. (2005). Nature Reviews Drug Discovery 4, Deville-Bonne, D., El Amri, C., Meyer, P., YUXING, C., Agrofoglio, L. A. & Janin, J. (2010). Antiviral research 86, Durand, S., Lightner, D., Redman, R. & Bonami, J. R. (1997). Diseases of Aquatic Organisms 29, Elion, G. B. (1983). Journal of Antimicrobial Chemotherapy 12, 9. Escobedo-Bonilla, C. M., Alday-Sanz, V., Wille, M., Sorgeloos, P., Pensaert, M. B. & Nauwynck, H. J. (2008). Journal of Fish Diseases 31, Garces, E. & Cleland, W. W. (1969). Biochemistry 8,
87 Gilles, A. M., Presecan, E., Vonica, A. & Lascu, I. (1991). Journal of Biological Chemistry 266, Giraud, M. F., Georgescauld, F., Lascu, I. & Dautant, A. (2006). Journal of bioenergetics and biomembranes 38, Herbert, E., Potter, V. R. & Takagi, Y. (1955). Journal of Biological Chemistry 213, Jacobus, W. & Evans, J. (1977). Journal of Biological Chemistry 252, Janin, J., Dumas, C., Moreira, S., Xu, Y., Meyer, P., Chiadmi, M. & Cherfils, J. (2000). Journal of bioenergetics and biomembranes 32, Jeudy, S., Claverie, J. M. & Abergel, C. (2006). Journal of bioenergetics and biomembranes 38, Johansson, M., MacKenzie-Hose, A., Andersson, I. & Knorpp, C. (2004). Plant physiology 136, Kandeel, M. & Kitade, Y. (2010a). Journal of bioenergetics and biomembranes 1-9. Kandeel, M. & Kitade, Y. (2010b). Journal of bioenergetics and biomembranes 42, Kimura, N. & Shimada, N. (1988). Journal of Biological Chemistry 263, Krebs, H. & Hems, R. (1953). Biochimica et biophysica acta 12, 172. La Scola, B., Audic, S., Robert, C., Jungang, L., de Lamballerie, X., Drancourt, M., Birtles, R., Claverie, J. M. & Raoult, D. (2003). Science 299, Lacombe, M. L. L., Munier, A., Mehus, J. G. & Lambeth, D. O. (2000). Journal of bioenergetics and biomembranes 32, Lascu, I., Chaffotte, A., Limbourg-Bouchon, B. & Veron, M. (1992). Journal of Biological Chemistry 267, Lascu, I., Deville-Bonne, D., Glaser, P. & Veron, M. (1993). Journal of Biological Chemistry 268,
88 Lascu, I., Giartosio, A., Ransac, S. & Erent, M. (2000). Journal of bioenergetics and biomembranes 32, Lehninger Albert, L. B. (1981). Replicación y transcripción del DNA. Segunda edición. Ed. Revolucionaria Leu, J. H., Yang, F., Zhang, X., Xu, X., Kou, G. H. & Lo, C. F. (2009). Lesser Known Large dsdna Viruses Lightner, D. V. (1996). A handbook of shrimp pathology and diagnostic procedures for diseases of cultured penaeid shrimp. World Aquaculture Society. Mathews, C. K., Ahern, K. G. & Van Holde, K. E. (2002). Biochemistry, Cuarta edición ed. Pearson Education Limited. Matte, A., Tari, L. W. & Delbaere, L. T. J. (1998). Structure 6, Mayo, M. (2002a). Archives of virology 147, Mayo, M. (2002b). Archives of virology 147, Mehus, J. G., Deloukas, P. & Lambeth, D. O. (1999). Human genetics 104, Mesnildrey, S., Agou, F., Karlsson, A., Bonne, D. D. & Veron, M. (1998). Journal of Biological Chemistry 273, Milon, L., Meyer, P., Chiadmi, M., Munier, A., Johansson, M., Karlsson, A., Lascu, I., Capeau, J., Janin, J. & Lacombe, M. L. (2000). Journal of Biological Chemistry 275, Milon, L., Rousseau-Merck, M. F., Munier, A., Erent, M., Lascu, I., Capeau, J. & Lacombe, M. L. (1997). Human genetics 99, Moréra, S., Lacombe, M. L., Yingwu, X. & Joel Janin, G. L. B. (1995). Structure 3, Morera, S., LeBras, G., Lascu, I., Lacornbe, M., Veron, M. & Janin, J. (1994). Journal of molecular biology 243, Munch-Petersen, B., Cloos, L., Tyrsted, G. & Eriksson, S. (1991). Journal of Biological Chemistry 266, Namikoshi, A., Wu, J. L., Yamashita, T., Nishizawa, T., Nishioka, T., Arimoto, M. & Muroga, K. (2004). Aquaculture 229,
89 Perozzo, R., Jelesarov, I., Bosshard, H. R., Folkers, G. & Scapozza, L. (2000). Journal of Biological Chemistry 275, Postel, E., Berberich, S., Flint, S. & Ferrone, C. (1993). Science (New York, NY) 261, 478. Postel, E. H., Abramczyk, B. A., Gursky, S. K. & Xu, Y. (2002). Biochemistry 41, Quintero-Reyes, I. E., Garcia-Orozco, K. D., Sugich-Miranda, R., Arvizu-Flores, A. A., Velazquez-Contreras, E. F., Castillo-Yañez, F. J. & Sotelo-Mundo, R. R. (2012). Journal of bioenergetics and biomembranes 1-7. Rajesh Kumar, S., Ishaq Ahamed, V., Sarathi, M., Nazeer Basha, A. & Sahul Hameed, A. (2008). Fish & shellfish immunology 24, Rameshthangam, P. & Ramasamy, P. (2007). Virus research 126, Sánchez-Paz, A. (2010). Veterinary research 41. Steeg, P. S., Bevilacqua, G., Kopper, L., Thorgeirsson, U. P., Talmadge, J. E., Liotta, L. A. & Sobel, M. E. (1988). Journal of the National Cancer Institute 80, Stoeckler, J. D., Agarwal, R. P., Agarwal, K. C., Schmid, K. & Parks Jr, R. E. (1978). Biochemistry 17, Timmons, L. & Shearn, A. (1997). Advances in Genetics 35, Tsai, J. M., Wang, H. C., Leu, J. H., Hsiao, H. H., Wang, A. H. J., Kou, G. H. & Lo, C. F. (2004). Journal of virology 78, Tsuiki, H., Nitta, M., Furuya, A., Hanai, N., Fujiwara, T., Inagaki, M., Kochi, M., Ushio, Y., Saya, H. & Nakamura, H. (2000). Journal of cellular biochemistry 76, van Hulten, M. C. W., Witteveldt, J., Peters, S., Kloosterboer, N., Tarchini, R., Fiers, M., Sandbrink, H., Lankhorst, R. K. & Vlak, J. M. (2001). Virology 286, Veer Reddy, G. P. & Pardee, A. B. (1980). Proceedings of the National Academy of Sciences 77, Venegas, C., Nonaka, L., Mushiake, K., Nishizawa, T. & Muroga, K. (2000). Diseases of Aquatic Organisms 42,
90 Webb, P. A., Perisic, O., Mendola, C. E., Backer, J. M. & Williams, R. L. (1995). Journal of molecular biology 251, Williams, R. L., Oren, D. A., Muñoz-Dorado, J., Inouye, S., Inouye, M. & Arnold, E. (1993). Journal of molecular biology 234, Wu, J., Nishioka, T., Mori, K., Nishizawa, T. & Muroga, K. (2002). Fish & shellfish immunology 13, Yelick, P. C., Kwon, Y. K., Flynn, J. F., Borzorgzadeh, A., Kleene, K. C. & Hecht, N. B. (1989). Molecular reproduction and development 1, Yi, G., Wang, Z., Qi, Y., Yao, L., Qian, J. & Hu, L. (2004). Journal of biochemistry and molecular biology 37,
91 74
Los nucleótidos están formados por una base nitrogenada, ribosa o desoxirribosa y un grupo fosfato.
 Introducción Los nucleótidos están formados por una base nitrogenada, ribosa o desoxirribosa y un grupo fosfato. Origen de los átomos que componen: Las bases púricas Base Nucleósido Nucleótido monofosfato
Introducción Los nucleótidos están formados por una base nitrogenada, ribosa o desoxirribosa y un grupo fosfato. Origen de los átomos que componen: Las bases púricas Base Nucleósido Nucleótido monofosfato
Bioquímica ACIDOS NUCLEICOS. Tema:5. Dra. Silvia Varas.
 Bioquímica ACIDOS NUCLEICOS 2013 Tema:5 Dra. Silvia Varas bioquimica.enfermeria.unsl@gmail.com Descubrimiento El descubrimiento de los ácidos nucleicos se realiza en el año 1869. A partir de núcleos de
Bioquímica ACIDOS NUCLEICOS 2013 Tema:5 Dra. Silvia Varas bioquimica.enfermeria.unsl@gmail.com Descubrimiento El descubrimiento de los ácidos nucleicos se realiza en el año 1869. A partir de núcleos de
UNIDAD ÁCIDOS NUCLEICOS
 UNIDAD ÁCIDOS NUCLEICOS DIRECTRICES Y ORIENTACIONES GENERALES PARA LAS PRUEBAS DE ACCESO A LA UNIVERSIDAD BLOQUE I. CUÁL ES LA COMPOSICIÓN DE LOS SERES VIVOS? LAS MOLÉCULAS DE LA VIDA I. PRINCIPALES TEMAS
UNIDAD ÁCIDOS NUCLEICOS DIRECTRICES Y ORIENTACIONES GENERALES PARA LAS PRUEBAS DE ACCESO A LA UNIVERSIDAD BLOQUE I. CUÁL ES LA COMPOSICIÓN DE LOS SERES VIVOS? LAS MOLÉCULAS DE LA VIDA I. PRINCIPALES TEMAS
Acidos Nucleicos. Adenina + Ribosa Adenosina
 Dr. Luis Rebolledo Acidos Nucleicos Prof. Iván Rebolledo Introducción Los ácidos nucleicos son macromoléculas orgánicas excepcionales por la cantidad considerable de fósforo que contienen y, sobre todo,
Dr. Luis Rebolledo Acidos Nucleicos Prof. Iván Rebolledo Introducción Los ácidos nucleicos son macromoléculas orgánicas excepcionales por la cantidad considerable de fósforo que contienen y, sobre todo,
Ácidos nucléicos. Los ácidos nucleicos fueron descubiertos por Freidrich Miescher en Mirel Nervenis
 Ácidos nucléicos Los ácidos nucleicos fueron descubiertos por Freidrich Miescher en 1869 La información genética o genoma, está contenida en unas moléculas llamadas ácidos nucleicos. Existen dos tipos
Ácidos nucléicos Los ácidos nucleicos fueron descubiertos por Freidrich Miescher en 1869 La información genética o genoma, está contenida en unas moléculas llamadas ácidos nucleicos. Existen dos tipos
BIOSINTESIS DE AMINOÁCIDOS INTEGRANTES: ZACHARY FERNANDA CUELLAR CARDOZO PAOLA ANDREA CASTAÑO PAYA
 BIOSINTESIS DE AMINOÁCIDOS INTEGRANTES: ZACHARY FERNANDA CUELLAR CARDOZO PAOLA ANDREA CASTAÑO PAYA Aminoácidos Es una molécula orgánica con un grupo amino (-NH 2 ) y un grupo carboxilo (- COOH). Los aminoácidos
BIOSINTESIS DE AMINOÁCIDOS INTEGRANTES: ZACHARY FERNANDA CUELLAR CARDOZO PAOLA ANDREA CASTAÑO PAYA Aminoácidos Es una molécula orgánica con un grupo amino (-NH 2 ) y un grupo carboxilo (- COOH). Los aminoácidos
TEMA 5 ACIDOS NUCLEICOS
 Bioquímica-Lic. En Enfermería TEMA 5 ACIDOS NUCLEICOS Dra. María Gabriela Lacoste Área de Química Biológica FQByF-UNSL 2016 DESCUBRIMIENTO El descubrimiento de los ácidos nucleicos se debe a Friedrich
Bioquímica-Lic. En Enfermería TEMA 5 ACIDOS NUCLEICOS Dra. María Gabriela Lacoste Área de Química Biológica FQByF-UNSL 2016 DESCUBRIMIENTO El descubrimiento de los ácidos nucleicos se debe a Friedrich
Acidos Nucleicos. Cap.3 Dra. Millie L. González
 Acidos Nucleicos Cap.3 Dra. Millie L. González Acidos Nucleicos Los ácidos nucleicos son de suma importancia para las células, ya que almacenan, transmiten y expresan la información genética Son polímeros
Acidos Nucleicos Cap.3 Dra. Millie L. González Acidos Nucleicos Los ácidos nucleicos son de suma importancia para las células, ya que almacenan, transmiten y expresan la información genética Son polímeros
INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR
 INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR - Componentes químicos de la célula - Un poco de química 1 Un poco de química El 99% del peso de una célula corresponde a C, H, N y O. La sustancia más abundante
INTRODUCCION A LA BIOLOGIA CELULAR Y MOLECULAR - Componentes químicos de la célula - Un poco de química 1 Un poco de química El 99% del peso de una célula corresponde a C, H, N y O. La sustancia más abundante
SUMARIO Ácidos nucleicos. Estructura general. Tipos principales: ADN y ARN. ADN. Estructura primaria. Estructura secundaria o modelo de Watson y Crick
 COFERENCIA 5 TÍTULO: COMPONENTES MOLECULARES: MACROMOLÉCULAS. ÁCIDOS NUCLEICOS Autor: Dr. Daniel Sánchez Serrano SUMARIO Ácidos nucleicos. Estructura general. Tipos principales: ADN y ARN. ADN. Estructura
COFERENCIA 5 TÍTULO: COMPONENTES MOLECULARES: MACROMOLÉCULAS. ÁCIDOS NUCLEICOS Autor: Dr. Daniel Sánchez Serrano SUMARIO Ácidos nucleicos. Estructura general. Tipos principales: ADN y ARN. ADN. Estructura
Proteínas y Ácidos Nucleicos
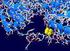 Proteínas y Ácidos Nucleicos Mapa conceptual Biomoléculas. Biomoléculas inorgánicas: Moléculas que no presentan carbono en su estructura. Biomoléculas orgánicas: Moléculas que presentan carbono en su estructura.
Proteínas y Ácidos Nucleicos Mapa conceptual Biomoléculas. Biomoléculas inorgánicas: Moléculas que no presentan carbono en su estructura. Biomoléculas orgánicas: Moléculas que presentan carbono en su estructura.
BIOSÍNTESIS DE NUCLEÓTIDOS
 Importancia: BIOSÍNTESIS DE NUCLEÓTIDOS - Componentes de ác. nucleicos -Moléculas de alto poder energético (ATP, GTP) -Biosíntesis orgánica (UDP-glucosa, CDP-etanolamina, CDP-diacilglicerol) -Transducción
Importancia: BIOSÍNTESIS DE NUCLEÓTIDOS - Componentes de ác. nucleicos -Moléculas de alto poder energético (ATP, GTP) -Biosíntesis orgánica (UDP-glucosa, CDP-etanolamina, CDP-diacilglicerol) -Transducción
Biomoléculas orgánicas: Proteínas y Ácidos nucleicos. Propiedad Intelectual Cpech
 Biología Biomoléculas orgánicas: Proteínas y Ácidos nucleicos Repaso Moléculas orgánicas Carbohidratos Lípidos 1. Monosacáridos: glucosa 2. Disacáridos: maltosa 3. Polisacáridos: glucógeno 1. Ácidos grasos.
Biología Biomoléculas orgánicas: Proteínas y Ácidos nucleicos Repaso Moléculas orgánicas Carbohidratos Lípidos 1. Monosacáridos: glucosa 2. Disacáridos: maltosa 3. Polisacáridos: glucógeno 1. Ácidos grasos.
Del ADN a las Proteínas
 Del ADN a las Proteínas ADN MATERIAL GENETICO CAPACIDAD PARA ALMACENAR INFORMACION ESTABILIDAD FISICA Y QUIMICA PARA NO PERDER INFORMACION CAPACIDAD PARA TRANSFERIR INFORMACION A CELULAS HIJAS Y HACERLO
Del ADN a las Proteínas ADN MATERIAL GENETICO CAPACIDAD PARA ALMACENAR INFORMACION ESTABILIDAD FISICA Y QUIMICA PARA NO PERDER INFORMACION CAPACIDAD PARA TRANSFERIR INFORMACION A CELULAS HIJAS Y HACERLO
EL CONTROL DE LAS ACTIVIDADES CELULARES
 EL CONTROL DE LAS ACTIVIDADES CELULARES LAS REACCIONES CELULARES BÁSICAS Todas las células llevan a cabo funciones vitales: Ingestión de nutrientes Eliminación de desperdicios Crecimiento Reproducción
EL CONTROL DE LAS ACTIVIDADES CELULARES LAS REACCIONES CELULARES BÁSICAS Todas las células llevan a cabo funciones vitales: Ingestión de nutrientes Eliminación de desperdicios Crecimiento Reproducción
ACIDOS NUCLEICOS. Dra. Elena Alvarado León Área de Genética y Biología Celular Depto. De Morfología Humana Fac. de Medicina UNT
 ACIDOS NUCLEICOS Dra. Elena Alvarado León Área de Genética y Biología Celular Depto. De Morfología Humana Fac. de Medicina UNT ÁCIDOS NUCLEICOS Son las biomoléculas esenciales de un organismo Monómero:
ACIDOS NUCLEICOS Dra. Elena Alvarado León Área de Genética y Biología Celular Depto. De Morfología Humana Fac. de Medicina UNT ÁCIDOS NUCLEICOS Son las biomoléculas esenciales de un organismo Monómero:
-Biomoléculas orgánicas con carácter ácido que se encuentran en el núcleo celular.
 ÁCIDOS NUCLÉICOS Definición: -Biomoléculas orgánicas con carácter ácido que se encuentran en el núcleo celular. - También en el citoplasma de células procariotas, en algunos orgánulos (ribosomas, mitocondrias
ÁCIDOS NUCLÉICOS Definición: -Biomoléculas orgánicas con carácter ácido que se encuentran en el núcleo celular. - También en el citoplasma de células procariotas, en algunos orgánulos (ribosomas, mitocondrias
Los nucleótidos están formados de: Una base nitrogenada Un azúcar de cinco carbonos Uno o más grupos fosfato
 ACIDOS NUCLEICOS Los monómeros de los ácidos nucleicos son los nucleótidos Los nucleótidos están formados de: Una base nitrogenada Un azúcar de cinco carbonos Uno o más grupos fosfato Las bases nitrogenadas
ACIDOS NUCLEICOS Los monómeros de los ácidos nucleicos son los nucleótidos Los nucleótidos están formados de: Una base nitrogenada Un azúcar de cinco carbonos Uno o más grupos fosfato Las bases nitrogenadas
CAPÍTULO 2 FLUJO DE LA INFORMACIÓN BIOLÓGICA FACULTAD DE AGRONOMÍA CURSO DE BIOQUÍMICA
 CAPÍTULO 2 FLUJO DE LA INFORMACIÓN BIOLÓGICA FACULTAD DE AGRONOMÍA CURSO DE BIOQUÍMICA Contenido 2.1 Información biológica e interacciones no covalentes. 2.2 Almacenamiento de la información biológica
CAPÍTULO 2 FLUJO DE LA INFORMACIÓN BIOLÓGICA FACULTAD DE AGRONOMÍA CURSO DE BIOQUÍMICA Contenido 2.1 Información biológica e interacciones no covalentes. 2.2 Almacenamiento de la información biológica
RESPIRACIÓN CELULAR. C 6 H 12 O 6 + O 2 + 6H 2 O CO H 2 O + Energía
 RESPIRACIÓN CELULAR Es el proceso por el cual la energía química de las moléculas de "alimento" es liberada y parcialmente capturada en forma de ATP. Los carbohidratos, grasas y proteínas pueden ser usados
RESPIRACIÓN CELULAR Es el proceso por el cual la energía química de las moléculas de "alimento" es liberada y parcialmente capturada en forma de ATP. Los carbohidratos, grasas y proteínas pueden ser usados
TEMA 9: NUCLEÓTIDOS Y ÁCIDOS NUCLEICOS.
 TEMA 9: NUCLEÓTIDOS Y ÁCIDOS NUCLEICOS. 1 1.-INTRODUCCIÓN. Entre las biomoléculas más importantes, por su papel en el almacenamiento y transmisión de la información genética, se encuentran los ácidos nucleicos.
TEMA 9: NUCLEÓTIDOS Y ÁCIDOS NUCLEICOS. 1 1.-INTRODUCCIÓN. Entre las biomoléculas más importantes, por su papel en el almacenamiento y transmisión de la información genética, se encuentran los ácidos nucleicos.
ACIDO FOSFÓRICO (ENLACE ESTER-FOSFATO) MONOSACÁRIDO (ENLACE N GLICOSÍDICO) BASE NITROGENADA RIBONUCLEÓTIDOS: DESOXINUCLEÓTIDOS:
 NUCLEÓTIDOS ACIDO FOSFÓRICO (ENLACE ESTER-FOSFATO) MONOSACÁRIDO (ENLACE N GLICOSÍDICO) BASE NITROGENADA NUCLEÓTIDOS NUCLEICOS RIBONUCLEÓTIDOS: AC. FOSFÓRICO RIBOSA ADENINA GUANINA CITOSINA URACILO DESOXINUCLEÓTIDOS:
NUCLEÓTIDOS ACIDO FOSFÓRICO (ENLACE ESTER-FOSFATO) MONOSACÁRIDO (ENLACE N GLICOSÍDICO) BASE NITROGENADA NUCLEÓTIDOS NUCLEICOS RIBONUCLEÓTIDOS: AC. FOSFÓRICO RIBOSA ADENINA GUANINA CITOSINA URACILO DESOXINUCLEÓTIDOS:
UNIDAD 5. AMINOÁCIDOS Y PROTEÍNAS
 UNIDAD 5. AMINOÁCIDOS Y PROTEÍNAS Dr en C. MPA MVZ Carlos Gutiérrez Olvera AMINOÁCIDOS: Como su nombre lo implica, los aminoácidos son moléculas orgánicas que contienen un grupo amino (NH2) en uno de los
UNIDAD 5. AMINOÁCIDOS Y PROTEÍNAS Dr en C. MPA MVZ Carlos Gutiérrez Olvera AMINOÁCIDOS: Como su nombre lo implica, los aminoácidos son moléculas orgánicas que contienen un grupo amino (NH2) en uno de los
UD 5: LOS ÁCIDOS NUCLEICOS
 UD 5: LOS ÁCIDOS NUCLEICOS ÍNDICE: 1. COMPOSICIÓN QUÍMICA Y CLASIFICACIÓN 2. NUCLEÓTIDOS 3. ADN. COMPOSICIÓN QUÍMICA Y ESTRUCTURA Y PROPIEDADES. 4. ARN 1. ARN MENSAJERO 2. ARN TRANSFERENTE 3. OTROS ARNs
UD 5: LOS ÁCIDOS NUCLEICOS ÍNDICE: 1. COMPOSICIÓN QUÍMICA Y CLASIFICACIÓN 2. NUCLEÓTIDOS 3. ADN. COMPOSICIÓN QUÍMICA Y ESTRUCTURA Y PROPIEDADES. 4. ARN 1. ARN MENSAJERO 2. ARN TRANSFERENTE 3. OTROS ARNs
Temas 14. Estructura y función de los ácidos nucleicos
 Temas 14. Estructura y función de los ácidos nucleicos Composición química: azúcares, fosfato y bases nitrogenadas Estructura y propiedades de las bases púricas y pirimidínicas ucleósidos y nucleótidos
Temas 14. Estructura y función de los ácidos nucleicos Composición química: azúcares, fosfato y bases nitrogenadas Estructura y propiedades de las bases púricas y pirimidínicas ucleósidos y nucleótidos
Es la capacidad de realizar un trabajo. A pesar que existen varias formas de energía: química, luminosa, mecánica, etc., solo hay dos tipos básicos:
 Es la capacidad de realizar un trabajo. A pesar que existen varias formas de energía: química, luminosa, mecánica, etc., solo hay dos tipos básicos: Potencial: es la capacidad de realizar trabajo como
Es la capacidad de realizar un trabajo. A pesar que existen varias formas de energía: química, luminosa, mecánica, etc., solo hay dos tipos básicos: Potencial: es la capacidad de realizar trabajo como
TEMA 5: LOS ÁCIDOS NUCLEICOS
 TEMA 5: LOS ÁCIDOS NUCLEICOS 1. Características químicas 2. Nucleósidos y Nucleótidos 3. Estructura del ADN 4. Estructura y tipos de ARN 5. Importancia biológica de estos compuestos 1. CARACTERÍSTICAS
TEMA 5: LOS ÁCIDOS NUCLEICOS 1. Características químicas 2. Nucleósidos y Nucleótidos 3. Estructura del ADN 4. Estructura y tipos de ARN 5. Importancia biológica de estos compuestos 1. CARACTERÍSTICAS
Metabolismo de nucleótidos, purinas y pirimidinas
 Metabolismo de nucleótidos, purinas y pirimidinas Repaso Un nucleótido consta de: 1. una base nitrogenada 2. una pentosa 3. un fosfato Nucleósido Adenosina Un nucleósido consta de una ribosa y una base
Metabolismo de nucleótidos, purinas y pirimidinas Repaso Un nucleótido consta de: 1. una base nitrogenada 2. una pentosa 3. un fosfato Nucleósido Adenosina Un nucleósido consta de una ribosa y una base
BIOLOGÍA GENERAL Ing.MSc. Sigfredo Ramos Cortez
 BIOLOGÍA GENERAL Ing.MSc. Sigfredo Ramos Cortez UNIDAD 2: ENERGÍA Y METABOLISMO TEMA: LA FUENTE DE ENERGÍA PARA LAS CELULAS SUBTEMAS: La glucosa El trifosfato de adenosina (ATP) OBJETIVOS DE LA CLASE:
BIOLOGÍA GENERAL Ing.MSc. Sigfredo Ramos Cortez UNIDAD 2: ENERGÍA Y METABOLISMO TEMA: LA FUENTE DE ENERGÍA PARA LAS CELULAS SUBTEMAS: La glucosa El trifosfato de adenosina (ATP) OBJETIVOS DE LA CLASE:
ÁCIDOS NUCLEICOS. Son biopolímeros, de elevado peso molecular, formados por otras subunidades estructurales o monómeros, denominados nucleótidos.
 ÁCIDOS NUCLEICOS Son biopolímeros, de elevado peso molecular, formados por otras subunidades estructurales o monómeros, denominados nucleótidos. El descubrimiento de los ácidos nucleicos se debe a Meischer
ÁCIDOS NUCLEICOS Son biopolímeros, de elevado peso molecular, formados por otras subunidades estructurales o monómeros, denominados nucleótidos. El descubrimiento de los ácidos nucleicos se debe a Meischer
Componentes químicos de
 Componentes químicos de las célulasc Componentes químicos Las células están compuestas por una enorme cantidad y variedad de moléculas que pueden clasificarse en: Componentes inorgánicos Componentes orgánicos
Componentes químicos de las célulasc Componentes químicos Las células están compuestas por una enorme cantidad y variedad de moléculas que pueden clasificarse en: Componentes inorgánicos Componentes orgánicos
ACIDOS NUCLEICOS. Andrea Arreola Guerrero Carlos A. Córdova Mendoza José A. Román Velásquez
 ACIDOS NUCLEICOS Andrea Arreola Guerrero Carlos A. Córdova Mendoza José A. Román Velásquez Ácidos Nucleicos. ADN Ácidos desoxirribonucleicos. ARN Ácidos ribonucleicos. Las unidades basicazo monómeros de
ACIDOS NUCLEICOS Andrea Arreola Guerrero Carlos A. Córdova Mendoza José A. Román Velásquez Ácidos Nucleicos. ADN Ácidos desoxirribonucleicos. ARN Ácidos ribonucleicos. Las unidades basicazo monómeros de
Glucólisis. OpenStax College. Based on Glycolysis by. 1 El ATP en los seres vivos
 OpenStax-CNX module: m53380 1 Glucólisis OpenStax College Based on Glycolysis by OpenStax College This work is produced by OpenStax-CNX and licensed under the Creative Commons Attribution License 4.0 Al
OpenStax-CNX module: m53380 1 Glucólisis OpenStax College Based on Glycolysis by OpenStax College This work is produced by OpenStax-CNX and licensed under the Creative Commons Attribution License 4.0 Al
EL A.D.N. Existen 2 tipos de Acidos Nucleicos : ADN (Acido Desoxirribonucleico) y ARN (Acido Ribonucleico) Diferencias entre ADN y ARN
 EL A.D.N Existen 2 tipos de Acidos Nucleicos : ADN (Acido Desoxirribonucleico) y ARN (Acido Ribonucleico) Diferencias entre ADN y ARN Hay tres tipos netamente diferenciados de ARN, tanto en su estructura
EL A.D.N Existen 2 tipos de Acidos Nucleicos : ADN (Acido Desoxirribonucleico) y ARN (Acido Ribonucleico) Diferencias entre ADN y ARN Hay tres tipos netamente diferenciados de ARN, tanto en su estructura
Lectura para aprender más
 Lectura para aprender más Estructura del ADN. Al ácido desoxirribonucleico (ADN) se le considera la base molecular de la vida, ya que constituye las unidades hereditarias que llamamos genes. El color de
Lectura para aprender más Estructura del ADN. Al ácido desoxirribonucleico (ADN) se le considera la base molecular de la vida, ya que constituye las unidades hereditarias que llamamos genes. El color de
Tema XI: Ácidos nucléicos (Introducción)
 República Bolivariana de Venezuela Ministerio del Poder Popular para la Educación U.E. Colegio Santo Tomás de Villanueva Departamento de Ciencias Cátedra: Ciencias Biológicas 3 Año Tema XI: Ácidos nucléicos
República Bolivariana de Venezuela Ministerio del Poder Popular para la Educación U.E. Colegio Santo Tomás de Villanueva Departamento de Ciencias Cátedra: Ciencias Biológicas 3 Año Tema XI: Ácidos nucléicos
El nitrógeno es un elemento sumamente importante para los organismo ya que
 METABOLISMO DE LOS COMPUESTOS NITROGENADOS MVZ Cuauhtémoc Nava Cuéllar Introducción El nitrógeno es un elemento sumamente importante para los organismo ya que forma parte de los aminoácidos y los ácidos
METABOLISMO DE LOS COMPUESTOS NITROGENADOS MVZ Cuauhtémoc Nava Cuéllar Introducción El nitrógeno es un elemento sumamente importante para los organismo ya que forma parte de los aminoácidos y los ácidos
Unidad 3: Organización molecular de la célula Proteínas
 Proteínas Las proteínas son las biomoléculas más abundantes de las células, constituyendo el 50% de su peso seco aproximadamente. Estructuralmente, son polímeros de aminoácidos. Existe una enorme variedad
Proteínas Las proteínas son las biomoléculas más abundantes de las células, constituyendo el 50% de su peso seco aproximadamente. Estructuralmente, son polímeros de aminoácidos. Existe una enorme variedad
Es la capacidad de realizar un trabajo. En términos bioquímicos: representa la capacidad de cambio, ya que la vida depende de que la energía pueda
 Es la capacidad de realizar un trabajo. En términos bioquímicos: representa la capacidad de cambio, ya que la vida depende de que la energía pueda ser transformada de una forma a otra, cuyo estudio es
Es la capacidad de realizar un trabajo. En términos bioquímicos: representa la capacidad de cambio, ya que la vida depende de que la energía pueda ser transformada de una forma a otra, cuyo estudio es
J. L. Sánchez Guillén
 J. L. Sánchez Guillén 1 LOS ÁCIDOS NUCLEICOS CONCEPTO: Químicamente, los ácidos nucleicos son polímeros constituidos por la unión mediante enlaces químicos de unidades menores llamadas nucleótidos. Los
J. L. Sánchez Guillén 1 LOS ÁCIDOS NUCLEICOS CONCEPTO: Químicamente, los ácidos nucleicos son polímeros constituidos por la unión mediante enlaces químicos de unidades menores llamadas nucleótidos. Los
Introducción a la Biología Molecular
 Introducción a la Biología Molecular Gonzalo Gómez ggomez@cnio.es Unidad de Bioinformática (CNIO) http://bioinfo.cnio.es/ Facultad de Informática, Ourense Mayo 2009 1 Niveles de organización DNA El ácido
Introducción a la Biología Molecular Gonzalo Gómez ggomez@cnio.es Unidad de Bioinformática (CNIO) http://bioinfo.cnio.es/ Facultad de Informática, Ourense Mayo 2009 1 Niveles de organización DNA El ácido
CARACTERÍSTICAS QUÍMICAS
 ÁCIDOS NUCLEICOS INTRODUCCIÓN Son polímeros formados por la unión de monómeros, los nucleótidos. Contienen toda la información genética del individuo y las instrucciones precisas para su lectura. Presentan
ÁCIDOS NUCLEICOS INTRODUCCIÓN Son polímeros formados por la unión de monómeros, los nucleótidos. Contienen toda la información genética del individuo y las instrucciones precisas para su lectura. Presentan
GENÉTICA MOLECULAR. El ADN, LA MOLÉCULA DE LA HERENCIA
 GENÉTICA MOLECULAR El ADN, LA MOLÉCULA DE LA HERENCIA INTRODUCCIÓN: COMENCEMOS PREGUNTÁNDONOS Qué significan estas dos palabras? GENÉTICA MOLECULAR Estudio de la genética (genes) desde el punto de vista
GENÉTICA MOLECULAR El ADN, LA MOLÉCULA DE LA HERENCIA INTRODUCCIÓN: COMENCEMOS PREGUNTÁNDONOS Qué significan estas dos palabras? GENÉTICA MOLECULAR Estudio de la genética (genes) desde el punto de vista
4. El esquema adjunto representa la estructura de una molécula denominada lisozima.
 BLOQUE I : La base físico-química de la materia 1. a. Nombre y formule un monosacárido que contenga seis átomos de carbono. b. Indica esquemáticamente una diferencia estructural existente entre el almidón
BLOQUE I : La base físico-química de la materia 1. a. Nombre y formule un monosacárido que contenga seis átomos de carbono. b. Indica esquemáticamente una diferencia estructural existente entre el almidón
Estrategias de control del Virus del Síndrome de la Mancha Blanca. Dr. Arturo Sánchez Paz
 Estrategias de control del Virus del Síndrome de la Mancha Blanca Dr. Arturo Sánchez Paz WSSV: un patógeno letal para camarones Datos: http://www.cosaes.com/ WSSV: un patógeno letal para camarones Extremadamente
Estrategias de control del Virus del Síndrome de la Mancha Blanca Dr. Arturo Sánchez Paz WSSV: un patógeno letal para camarones Datos: http://www.cosaes.com/ WSSV: un patógeno letal para camarones Extremadamente
Unidad 7: Respiración Celular
 1 La energía lumínica es capturada por las plantas verdes y otros organismos fotosintéticos, que la transforman en energía química fijada en moléculas como la glucosa. Estas moléculas son luego degradadas
1 La energía lumínica es capturada por las plantas verdes y otros organismos fotosintéticos, que la transforman en energía química fijada en moléculas como la glucosa. Estas moléculas son luego degradadas
Bases moleculares de la herencia. Código genético y Traducción.
 UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Código genético y Traducción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira
UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Código genético y Traducción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira
TEMA 5: LOS ÁCIDOS NUCLEICOS
 TEMA 5: LOS ÁCIDOS NUCLEICOS 1. LOS NUCLEÓTIDOS Los ácidos nucleicos son biomoléculas orgánicas formadas por C, H, O, N y P. Nunca llevan S. Se forman por la polimerización de nucleótidos, que se unen
TEMA 5: LOS ÁCIDOS NUCLEICOS 1. LOS NUCLEÓTIDOS Los ácidos nucleicos son biomoléculas orgánicas formadas por C, H, O, N y P. Nunca llevan S. Se forman por la polimerización de nucleótidos, que se unen
METABOLISMO DE AMINOACIDOS. Kinesiología 2011
 METABOLISMO DE AMINOACIDOS Kinesiología 2011 BALANCE NITROGENADO BN=Nitrógeno total ingerido Nitrógeno total excretado BN=0 existe equilibrio nitrogenado BN es negativo, cuando hay condiciones de síntesis
METABOLISMO DE AMINOACIDOS Kinesiología 2011 BALANCE NITROGENADO BN=Nitrógeno total ingerido Nitrógeno total excretado BN=0 existe equilibrio nitrogenado BN es negativo, cuando hay condiciones de síntesis
ÁCIDOS NUCLEICOS. Aguanten Gregor Mendel y los guisantes!
 ÁCIDOS NUCLEICOS Aguanten Gregor Mendel y los guisantes! ÁCIDOS NUCLEICOS caracts. grales. ADN y ARN son los mensajeros químicos de la información genética de los seres vivos. Son polímeros de nucleótidos,
ÁCIDOS NUCLEICOS Aguanten Gregor Mendel y los guisantes! ÁCIDOS NUCLEICOS caracts. grales. ADN y ARN son los mensajeros químicos de la información genética de los seres vivos. Son polímeros de nucleótidos,
CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO
 Alianza para el Aprendizaje de Ciencias y Matemáticas CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO GUÍA DEL MAESTRO ESTÁNDARES ATENDIDOS: 1. LA NATURALEZA
Alianza para el Aprendizaje de Ciencias y Matemáticas CÓMO SE PUEDE DETERMINAR LA SECUENCIA DEL DNA A PARTIR DE UNA PROTEÍNA? DR. MANUEL E. AQUINO GUÍA DEL MAESTRO ESTÁNDARES ATENDIDOS: 1. LA NATURALEZA
RESULTADO DE APRENDIZAJE:
 Explicar las reacciones químicas del ciclo de Krebs y su regulación RESULTADO DE APRENDIZAJE: Relacionar el metabolismo de las distintas macromoléculas alrededor del Ciclo de Krebs Las reacciones se llevan
Explicar las reacciones químicas del ciclo de Krebs y su regulación RESULTADO DE APRENDIZAJE: Relacionar el metabolismo de las distintas macromoléculas alrededor del Ciclo de Krebs Las reacciones se llevan
SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS. Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida
 SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida La secuencia de aminoácidos de un polipéptido está programada en una unidad heredable denominada gen.
SESIÓN 5 ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS Los Ácidos Nucleicos. Moléculas Esenciales Para La Vida La secuencia de aminoácidos de un polipéptido está programada en una unidad heredable denominada gen.
Enzimas Departamento de Bioquímica Noviembre de 2005
 U.T.I. Biología Celular Enzimas Departamento de Bioquímica Noviembre de 2005 Enzimas A. Propiedades generales de las enzimas B. Principios fundamentales de su acción catalítica C. Introducción a la cinética
U.T.I. Biología Celular Enzimas Departamento de Bioquímica Noviembre de 2005 Enzimas A. Propiedades generales de las enzimas B. Principios fundamentales de su acción catalítica C. Introducción a la cinética
TEMA 6. LOS ÁCIDOS NUCLEICOS.
 TEMA 6. LOS ÁCIDOS NUCLEICOS. 1.-Importancia de los ácidos nucleicos. 2.-Componentes de los ácidos nucleicos. 2.1. Los nucleótidos. 2.2. Funciones de los nucleótidos. 3.- El enlace nucleotídico. 4.- El
TEMA 6. LOS ÁCIDOS NUCLEICOS. 1.-Importancia de los ácidos nucleicos. 2.-Componentes de los ácidos nucleicos. 2.1. Los nucleótidos. 2.2. Funciones de los nucleótidos. 3.- El enlace nucleotídico. 4.- El
Fisiología de Guyton Capitulo 3
 Fisiología de Guyton Capitulo 3 Introducción En general se conoce que los genes son el medio principal para la herencia de genes de padres a hijos, que se encuentra en el núcleo de las células de todo
Fisiología de Guyton Capitulo 3 Introducción En general se conoce que los genes son el medio principal para la herencia de genes de padres a hijos, que se encuentra en el núcleo de las células de todo
Metabolismo. Forma de obtención de carbono. Corresponde a la actividad. participan sistemas multienzimáticos (rutas metabólicas) RUTAS METABÓLICAS
 Facultad de Ciencias de la Salud BIO160 Bioquímica i General Metabolismo METABOLISMO Corresponde a la actividad coordinada que ocurre dentro de una célula, en la cual participan sistemas multienzimáticos
Facultad de Ciencias de la Salud BIO160 Bioquímica i General Metabolismo METABOLISMO Corresponde a la actividad coordinada que ocurre dentro de una célula, en la cual participan sistemas multienzimáticos
BIOQUIMICA Y FISIOLOGÍA MICROBIANA. Dra. Yenizey Merit Alvarez Cisneros
 BIOQUIMICA Y FISIOLOGÍA MICROBIANA Dra. Yenizey Merit Alvarez Cisneros Moléculas importantes para la vida Compuestos Orgánicos (contienen carbono e hidrógeno) y forma enlaces covalentes Azucares Aminoácidos
BIOQUIMICA Y FISIOLOGÍA MICROBIANA Dra. Yenizey Merit Alvarez Cisneros Moléculas importantes para la vida Compuestos Orgánicos (contienen carbono e hidrógeno) y forma enlaces covalentes Azucares Aminoácidos
CONVERGENCIA METABÓLICA. En el catabolismo aerobio existen tres fases principales:
 CONVERGENCIA METABÓLICA Rutas catabólicas En el catabolismo aerobio existen tres fases principales: - Fase I.- Transformación de Macromoléculas en sus Monómeros correspondientes. 1) Polisacáridos Monosacáridos
CONVERGENCIA METABÓLICA Rutas catabólicas En el catabolismo aerobio existen tres fases principales: - Fase I.- Transformación de Macromoléculas en sus Monómeros correspondientes. 1) Polisacáridos Monosacáridos
PROGRAMA DE ESTUDIO. 2. CICLO O AREA: División de Ciencias e Ingeniería/Ingeniería Ambiental.
 PROGRAMA DE ESTUDIO 1. NOMBRE DE LA ASIGNATURA: BIOQUÍMICA. 2. CICLO O AREA: División de Ciencias e Ingeniería/Ingeniería Ambiental. 3. CLAVE: 4. SERIACION: Química Orgánica. 5. H.T.S. H.P.S. T.H.S. C.
PROGRAMA DE ESTUDIO 1. NOMBRE DE LA ASIGNATURA: BIOQUÍMICA. 2. CICLO O AREA: División de Ciencias e Ingeniería/Ingeniería Ambiental. 3. CLAVE: 4. SERIACION: Química Orgánica. 5. H.T.S. H.P.S. T.H.S. C.
Metabolismo de Hidratos de. Parte II
 Metabolismo de Hidratos de Carbono Parte II GLUCONEOGENESIS: Cómo se la define?: Es la síntesis de glucosa a partir de compuestos no glucídicos, como: aminoácidos, piruvato proveniente de la transaminación
Metabolismo de Hidratos de Carbono Parte II GLUCONEOGENESIS: Cómo se la define?: Es la síntesis de glucosa a partir de compuestos no glucídicos, como: aminoácidos, piruvato proveniente de la transaminación
Revisión- Opción Múltiple Procesamiento de energía
 Revisión- Opción Múltiple Procesamiento de energía 1. El mmetabolismo es considerado como las "reacciones químicas totales que ocurren dentro de un organismo". Estas reacciones químicas pueden estar vinculados
Revisión- Opción Múltiple Procesamiento de energía 1. El mmetabolismo es considerado como las "reacciones químicas totales que ocurren dentro de un organismo". Estas reacciones químicas pueden estar vinculados
BIOLOGIA COMÚN BC-04 U N I D A D I : C É L U L A ÁCIDOS NUCLEICOS
 BIOLOGIA COMÚN UNIDAD I: CÉLULA ÁCIDOS NUCLEICOS BC-04 COMPUESTOS ORGÁNICOS: Ácidos Nucleicos Son las únicas moléculas biológicas que poseen la capacidad de autoduplicación y están formados por C, H, O,
BIOLOGIA COMÚN UNIDAD I: CÉLULA ÁCIDOS NUCLEICOS BC-04 COMPUESTOS ORGÁNICOS: Ácidos Nucleicos Son las únicas moléculas biológicas que poseen la capacidad de autoduplicación y están formados por C, H, O,
DUPLICACION DEL ADN. Dra Carmen Aída Martínez
 DUPLICACION DEL ADN Dra Carmen Aída Martínez Definción El proceso de replicación de ADN es el mecanismo que permite al ADN duplicarse (es decir, sintetizar una copia idéntica) Replicación La reproducción
DUPLICACION DEL ADN Dra Carmen Aída Martínez Definción El proceso de replicación de ADN es el mecanismo que permite al ADN duplicarse (es decir, sintetizar una copia idéntica) Replicación La reproducción
Metabolismo de AMINOÁCIDOS
 UNIVERSIDAD DE SAN CARLOS DE GUATEMALA FACULTAD DE CIENCIAS MÉDICAS FASE I, Unidad Didáctica: BIOQUÍMICA MÉDICA 2º AÑO CICLO ACADÉMICO 2,011 Metabolismo de AMINOÁCIDOS Dr. Mynor A. Leiva Enríquez Fuente:
UNIVERSIDAD DE SAN CARLOS DE GUATEMALA FACULTAD DE CIENCIAS MÉDICAS FASE I, Unidad Didáctica: BIOQUÍMICA MÉDICA 2º AÑO CICLO ACADÉMICO 2,011 Metabolismo de AMINOÁCIDOS Dr. Mynor A. Leiva Enríquez Fuente:
En el estudio de los mecanismos de acción farmacológica hay que distinguir.
 TEMA : ASPECTOS MOLECULARES DEL MECANISMO DE ACCIÓN DE LOS FÁRMACOS En el estudio de los mecanismos de acción farmacológica hay que distinguir. Fármacos de Acción Específica: son aquellos que han de interaccionar
TEMA : ASPECTOS MOLECULARES DEL MECANISMO DE ACCIÓN DE LOS FÁRMACOS En el estudio de los mecanismos de acción farmacológica hay que distinguir. Fármacos de Acción Específica: son aquellos que han de interaccionar
Energía y metabolismo
 Energía y metabolismo Sesión 17 Introducción a la Biología Prof. Nelson A. Lagos Los sistemas vivos son abiertos y requieren energía para mantenerse La energía es la capacidad de hacer trabajo. Cinético
Energía y metabolismo Sesión 17 Introducción a la Biología Prof. Nelson A. Lagos Los sistemas vivos son abiertos y requieren energía para mantenerse La energía es la capacidad de hacer trabajo. Cinético
ADN, ARN Y SÍNTESIS DE PROTEÍNAS
 UNIVERSIDAD DEL VALLE DE GUATEMALA CU125 - CIENCIAS NATURALES FLOR DE MARÍA PELÁEZ ADN, ARN Y SÍNTESIS DE PROTEÍNAS DIEGO FERNANDO GODOY RECKHOLDER #09602 OSCAR ALESSANDRO REYES LICO #09565 10 de marzo
UNIVERSIDAD DEL VALLE DE GUATEMALA CU125 - CIENCIAS NATURALES FLOR DE MARÍA PELÁEZ ADN, ARN Y SÍNTESIS DE PROTEÍNAS DIEGO FERNANDO GODOY RECKHOLDER #09602 OSCAR ALESSANDRO REYES LICO #09565 10 de marzo
Plataforma para la monitorización de la Gripe en España
 Plataforma para la monitorización de la Gripe en España La gripe es una de las enfermedades más contagiosas que existen y cada año es responsable de la muerte de cientos de miles de personas en todo el
Plataforma para la monitorización de la Gripe en España La gripe es una de las enfermedades más contagiosas que existen y cada año es responsable de la muerte de cientos de miles de personas en todo el
ESTUDIOS CINÉTICOS SE MIDE LA VELOCIDAD DE LA REACCIÓN CATALIZADA. La velocidad de reacciones químicas: Para una reacción:
 ESTUDIOS CINÉTICOS SE MIDE LA VELOCIDAD DE LA REACCIÓN CATALIZADA La velocidad de reacciones químicas: Para una reacción: A B La velocidad depende de la concentración del reactante: v = k[a] k = constante
ESTUDIOS CINÉTICOS SE MIDE LA VELOCIDAD DE LA REACCIÓN CATALIZADA La velocidad de reacciones químicas: Para una reacción: A B La velocidad depende de la concentración del reactante: v = k[a] k = constante
CADENA RESPIRATORIA O CADENA DE TRANSPORTE DE ELECTRONES
 CADENA RESPIRATORIA O CADENA DE TRANSPORTE DE ELECTRONES El NADH y FADH2 obtenidos contienen un par de electrones que se transfieren al O2 con liberación de energía. La cadena respiratoria transporta los
CADENA RESPIRATORIA O CADENA DE TRANSPORTE DE ELECTRONES El NADH y FADH2 obtenidos contienen un par de electrones que se transfieren al O2 con liberación de energía. La cadena respiratoria transporta los
Bases moleculares de la herencia. Transcripción.
 UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Transcripción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira 2. Eduardo
UNIVERSIDAD AUTÓNOMA DE SINALOA Unidad Académica de Ciencias de la Nutrición y Gastronomía. Bases moleculares de la herencia. Transcripción. Dr. Javier Magaña 1. Anna Islas 2. Diego Moreira 2. Eduardo
NUCLEOSIDOS Y NUCLEOTIDOS Semana 31. Licda. Lilian Judith Guzmán Melgar
 NUCLEOSIDOS Y NUCLEOTIDOS Semana 31 Licda. Lilian Judith Guzmán Melgar PURINAS Y PIRIMIDINAS + RIBOSA O DESOXIRIBOSA NUCLEÓSIDOS + ACIDO FOSFORICO NUCLEÓTIDOS ACIDO NUCLEICO 2 BASES PIRIMIDICAS Las bases
NUCLEOSIDOS Y NUCLEOTIDOS Semana 31 Licda. Lilian Judith Guzmán Melgar PURINAS Y PIRIMIDINAS + RIBOSA O DESOXIRIBOSA NUCLEÓSIDOS + ACIDO FOSFORICO NUCLEÓTIDOS ACIDO NUCLEICO 2 BASES PIRIMIDICAS Las bases
Tema 10. Regulación de la actividad enzimática
 Tema 10. Regulación de la actividad enzimática Control de la actividad enzimática Regulación por cambios en la concentración de enzima Regulación alostérica Modificaciones covalentes reversibles Activación
Tema 10. Regulación de la actividad enzimática Control de la actividad enzimática Regulación por cambios en la concentración de enzima Regulación alostérica Modificaciones covalentes reversibles Activación
TEORIA CELULAR. En el mundo vivo, la unidad fundamental es la célula. DECUBRIMIENTO DE LAS CELULAS
 TEORIA CELULAR En el mundo vivo, la unidad fundamental es la célula. DECUBRIMIENTO DE LAS CELULAS El nombre de célula significa celda, así las llamo Robert Hooke. En 1839 el zoólogo alemán Theodore Schwann
TEORIA CELULAR En el mundo vivo, la unidad fundamental es la célula. DECUBRIMIENTO DE LAS CELULAS El nombre de célula significa celda, así las llamo Robert Hooke. En 1839 el zoólogo alemán Theodore Schwann
Trascripción. Traducción
 Trascripción y Traducción 1 2 Tipos de ARN 3 Molécula lineal que contiene información genética copiada del ADN. Tiene regiones codificadoras y regiones no codificadoras como la cabeza o líder y la cola.
Trascripción y Traducción 1 2 Tipos de ARN 3 Molécula lineal que contiene información genética copiada del ADN. Tiene regiones codificadoras y regiones no codificadoras como la cabeza o líder y la cola.
RESPIRACIÓN AEROBIA Y ANAEROBIA
 RESPIRACIÓN AEROBIA Y ANAEROBIA Las células llevan a cabo diversos procesos para mantener su funcionamiento normal, muchos de los cuales requieren energía. La respiración celular es una serie de reacciones
RESPIRACIÓN AEROBIA Y ANAEROBIA Las células llevan a cabo diversos procesos para mantener su funcionamiento normal, muchos de los cuales requieren energía. La respiración celular es una serie de reacciones
TEMA 2 LA INFORMACIÓN GENÉTICA COLEGIO LEONARDO DA VINCI BIOLOGÍA Y GEOLOGÍA 4º ESO CURSO 2014/15
 TEMA 2 LA INFORMACIÓN GENÉTICA COLEGIO LEONARDO DA VINCI BIOLOGÍA Y GEOLOGÍA 4º ESO CURSO 2014/15 OBJETIVOS DEL TEMA * Ácidos nucleicos. Composición. Estructura. Tipos. Funciones. * Procesos del dogma
TEMA 2 LA INFORMACIÓN GENÉTICA COLEGIO LEONARDO DA VINCI BIOLOGÍA Y GEOLOGÍA 4º ESO CURSO 2014/15 OBJETIVOS DEL TEMA * Ácidos nucleicos. Composición. Estructura. Tipos. Funciones. * Procesos del dogma
Introducción. Expresión génica. Regulación de la expresión génica en procariotas
 Introducción La secuencia genética siempre es la misma en todas las células y siempre está presente en organismos unicelulares, pero no se expresa igual en cada célula y en cada momento. Expresión génica
Introducción La secuencia genética siempre es la misma en todas las células y siempre está presente en organismos unicelulares, pero no se expresa igual en cada célula y en cada momento. Expresión génica
Ciclo de Krebs Destino del piruvato. Descarboxilación oxidativa. Aspectos estructurales y mecanismos de la piruvato deshidrogenasa.
 Ciclo de Krebs Destino del piruvato. Descarboxilación oxidativa. Aspectos estructurales y mecanismos de la piruvato deshidrogenasa. Relevancia del ciclo de Krebs dentro del metabolismo en general. Descripción
Ciclo de Krebs Destino del piruvato. Descarboxilación oxidativa. Aspectos estructurales y mecanismos de la piruvato deshidrogenasa. Relevancia del ciclo de Krebs dentro del metabolismo en general. Descripción
Conceptos básicos de biología molecular, I
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 21 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Conceptos básicos de biología molecular 21 de mayo del 2013 1 / 28 1 Introducción Tarea Dr.
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 21 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Conceptos básicos de biología molecular 21 de mayo del 2013 1 / 28 1 Introducción Tarea Dr.
CICLO CELULAR Y SU REGULACIÓN G1
 G2 M S CICLO CELULAR Y SU REGULACIÓN G1 EXPRESIÓN de GENES Para dividirse, la célula recibe señales, las transmite y las interpreta como EXPRESIÓN de GENES ESPECÍFICOS para poder pasar por cada una de
G2 M S CICLO CELULAR Y SU REGULACIÓN G1 EXPRESIÓN de GENES Para dividirse, la célula recibe señales, las transmite y las interpreta como EXPRESIÓN de GENES ESPECÍFICOS para poder pasar por cada una de
LA NUEVA BIOTECNOLOGÍA
 LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
Click para ir al sitio web:
 Slide 1 / 46 New Jersey Center for Teaching and Learning Iniciativa de Ciencia Progresiva Este material está disponible gratuitamente en www.njctl.org y está pensado para el uso no comercial de estudiantes
Slide 1 / 46 New Jersey Center for Teaching and Learning Iniciativa de Ciencia Progresiva Este material está disponible gratuitamente en www.njctl.org y está pensado para el uso no comercial de estudiantes
Transcripción. - Generalidades - DNA RNA. - Mecanismo. - Modelos Protein. - Moléculas. - Métodos de detección de RNA. Chromosome RNA DNA
 TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
TRANSCRIPCIÓN Transcripción - Generalidades Chromosome - DNA RNA - Mecanismo - Modelos Protein RNA - Moléculas - Métodos de detección de RNA DNA Transcripción - Primer proceso de la expresión genética
Conversión del glucoso 6 fosfato en otras hexosas
 BIOSINTESIS DE LOS CARBOHIDRATOS TABLA DE CONTENIDO Introducción Justificación Objetivos Síntesis del glucoso fosfato a partir del ácido pirúvico Regulación de la ruta que va desde el piruvato al glucoso
BIOSINTESIS DE LOS CARBOHIDRATOS TABLA DE CONTENIDO Introducción Justificación Objetivos Síntesis del glucoso fosfato a partir del ácido pirúvico Regulación de la ruta que va desde el piruvato al glucoso
Degradación de aminoácidos
 Degradación de aminoácidos Familias de aminoácidos Degradación de aminoácidos Cuándo es necesario degradar los aminoácidos? Son una buena fuente de energía? Qué pasa con el nitrógeno? Y los átomos de carbono?
Degradación de aminoácidos Familias de aminoácidos Degradación de aminoácidos Cuándo es necesario degradar los aminoácidos? Son una buena fuente de energía? Qué pasa con el nitrógeno? Y los átomos de carbono?
NUCLEOTIDOS. Metabolismo
 NUCLEOTIDOS -Composición -Bases nitrogenadas 1.-bases púricas 2.-bases pirimidínicas -Otras bases 1.-bases modificadas a) mutilaciones b) isopentenil derivados c) tioderivados 2.-productos naturales 3.-análogos
NUCLEOTIDOS -Composición -Bases nitrogenadas 1.-bases púricas 2.-bases pirimidínicas -Otras bases 1.-bases modificadas a) mutilaciones b) isopentenil derivados c) tioderivados 2.-productos naturales 3.-análogos
Ácidos Nucleicos. Compuestos orgánicos formados por: Su estructura básica (monómero) es el nucleotido Existen principalmente 2 tipos DNA y RNA
 Ácidos Nucleicos Características genérales Ácidos Nucleicos Compuestos orgánicos formados por: Su estructura básica (monómero) es el nucleotido Existen principalmente 2 tipos DNA y RNA C, H, O, N, P Nucleotidos:
Ácidos Nucleicos Características genérales Ácidos Nucleicos Compuestos orgánicos formados por: Su estructura básica (monómero) es el nucleotido Existen principalmente 2 tipos DNA y RNA C, H, O, N, P Nucleotidos:
Metabolismo de hidratos de carbono
 Introducción a la Botánica 2016 10ma. clase teórica: 13/4/2016 Metabolismo de hidratos de carbono La fotosíntesis y la respiración 1 La fotosíntesis y la respiración Azúcares + ATP + nutrientes del suelo
Introducción a la Botánica 2016 10ma. clase teórica: 13/4/2016 Metabolismo de hidratos de carbono La fotosíntesis y la respiración 1 La fotosíntesis y la respiración Azúcares + ATP + nutrientes del suelo
ADN ---- Proteínas. ADN ---? --- Proteínas
 El dogma central de la biología molecular es un concepto que explica los mecanismos de transmisión y expresión de la herencia genética tras el descubrimiento de la molécula del ADN en doble hélice. Propone
El dogma central de la biología molecular es un concepto que explica los mecanismos de transmisión y expresión de la herencia genética tras el descubrimiento de la molécula del ADN en doble hélice. Propone
Ciclo del ácido cítrico (Krebs o Ciclo de los ácidos tricarboxílicos
 Ciclo del ácido cítrico (Krebs o Ciclo de los ácidos tricarboxílicos Oxidación de los derivados de la glucosa para dar CO 2 Dentro de la mitocondria (eucariotas) Eje central del metabolismo celular Vía
Ciclo del ácido cítrico (Krebs o Ciclo de los ácidos tricarboxílicos Oxidación de los derivados de la glucosa para dar CO 2 Dentro de la mitocondria (eucariotas) Eje central del metabolismo celular Vía
Dogma Central de la Biología Molecular
 Replicación Dogma Central de la Biología Molecular Ciclo celular Modelo semiconservador Comprobación Polimerización Procariontes (Modelo Theta) Procariontes y Eucariontes Mitocondrias Origen de la replicación
Replicación Dogma Central de la Biología Molecular Ciclo celular Modelo semiconservador Comprobación Polimerización Procariontes (Modelo Theta) Procariontes y Eucariontes Mitocondrias Origen de la replicación
TRABAJO SOBRE INGENIERÍA GENÉTICA 4º P.D.C.- A.C.T. CURSO:2012/2013. Ingeniería genética
 Ingeniería genética Es la tecnología del control y transferencia de ADN de un organismo a otro, lo que posibilita la creación de nuevas especies, la corrección de defectos genéticos y la fabricación de
Ingeniería genética Es la tecnología del control y transferencia de ADN de un organismo a otro, lo que posibilita la creación de nuevas especies, la corrección de defectos genéticos y la fabricación de
CLASIFICACION POR LA CADENA LATERAL POR SU REQUERIMIENTO POR SU POLARIDAD. Esenciales No esenciales CADENA LATERAL CON GRUPO
 AMINOACIDOS Y PEPTIDOS Licda. Lilian Judith Guzmán Melgar 2016 29 AMINOACIDOS Compuestos que contienen un grupo carboxílico y un grupo amino. Los más importantes en el mundo biológico son los α-aminoácidos,
AMINOACIDOS Y PEPTIDOS Licda. Lilian Judith Guzmán Melgar 2016 29 AMINOACIDOS Compuestos que contienen un grupo carboxílico y un grupo amino. Los más importantes en el mundo biológico son los α-aminoácidos,
Lehninger, cap. 15, ps ; Mathews.- cap. 13, ps y cap.16. ps ; Stryer.- cap. 21, ps ; Voet.-cap. 15, ps
 BBM II. 2012/13 (R-T8-1) Tema 8.- Metabolismo del glucógeno: Metabolismo de polisacáridos de reserva. Degradación y Síntesis del glucógeno. Regulación metabólica y hormonal de la glucogenolisis y glucogénesis.
BBM II. 2012/13 (R-T8-1) Tema 8.- Metabolismo del glucógeno: Metabolismo de polisacáridos de reserva. Degradación y Síntesis del glucógeno. Regulación metabólica y hormonal de la glucogenolisis y glucogénesis.
BIOMOLÉCULAS. Son moléculas fundamentales para la constitución y funcionamiento de todo ser vivo. Se clasifican en dos grupos:
 FICHA N 2 UNIDAD III: CÉLULAS A ORGANISMOS Profesora Verónica Abasto Córdova Ciencias Naturales 8 Básico Nombre : Curso : Fecha : Revisión BIOMOLÉCULAS Son moléculas fundamentales para la constitución
FICHA N 2 UNIDAD III: CÉLULAS A ORGANISMOS Profesora Verónica Abasto Córdova Ciencias Naturales 8 Básico Nombre : Curso : Fecha : Revisión BIOMOLÉCULAS Son moléculas fundamentales para la constitución
Niveles de organización biológica. La composición de los seres vivos
 Niveles de organización biológica. La composición de los seres vivos Imagen de Fondo bajo licencia Creative Commons, autor: René Ehrtardt. Animación ADN de dominio público, fuente:wikipedia Con este tema
Niveles de organización biológica. La composición de los seres vivos Imagen de Fondo bajo licencia Creative Commons, autor: René Ehrtardt. Animación ADN de dominio público, fuente:wikipedia Con este tema
RESPIRACIÓN CELULAR (I): CICLO DE KREBS
 Mediante la respiración celular, el acido pirúvico formado durante la glucólisis se oxida completamente a CO 2 y H 2 O en presencia de oxígeno, Este proceso de respiración se desarrolla en dos etapas sucesivas:
Mediante la respiración celular, el acido pirúvico formado durante la glucólisis se oxida completamente a CO 2 y H 2 O en presencia de oxígeno, Este proceso de respiración se desarrolla en dos etapas sucesivas:
Clave Genética y Síntesis de Proteínas
 UNIVERSIDAD DE SAN CARLOS DE GUATEMALA FACULTAD DE CIENCIAS MÉDICAS FASE I, Unidad Didáctica: BIOQUÍMICA MÉDICA 2º AÑO CICLO ACADÉMICO 2,011 Clave Genética y Síntesis de Proteínas Dr. Mynor A. Leiva Enríquez
UNIVERSIDAD DE SAN CARLOS DE GUATEMALA FACULTAD DE CIENCIAS MÉDICAS FASE I, Unidad Didáctica: BIOQUÍMICA MÉDICA 2º AÑO CICLO ACADÉMICO 2,011 Clave Genética y Síntesis de Proteínas Dr. Mynor A. Leiva Enríquez
