Predicción de Estructura Tridimensional de Proteínas
|
|
|
- María Victoria Rey Castillo
- hace 8 años
- Vistas:
Transcripción
1 Estructura de Proteínas Predicción de Estructura Tridimensional de Proteínas Florencio Pazos (CNB-CSIC) Florencio Pazos Cabaleiro Computational Systems Biology Group (CNB-CSIC) CNB Marzo 2008
2 Genómica Estructural Vitkup, D., Melamud, E., Moult, J. and Sander, C. (2001) Completeness in structural genomics. Nat Struct Biol, 8,
3 Genómica estructural y predicción de estructura
4 Genómica estructural y predicción de estructura
5 Predicción de estructura de proteínas Clasificación de los métodos de predicción Nivel estructura proteínas Representación de la proteína Uso de información extra Secundaria terciaria cuaternaria 1D 2D 3D AAVLYFGREDHTLLVY AAVLYFGREDHTLLVY AAVLYFGREDHTLLVY 4D Ab Initio pred. str. secundaria mutaciones correlacionadas - dinámica molecular - minimización de energía docking No Ab-Initio pred. str. secundaria - modelado por homología - threading docking con filtros
6 Predicción de Estructura de Proteínas Métodos 3D - Ab initio - Diseño por homología/modelado comparativo. - Reconocimiento de plegamiento/ diseño por homología remota/threading
7 Ab Initio Basados en principios físico/químicos básicos (potenciales de interacción,...). Solo usan la secuencia primaria como entrada. Interesantes pues son los que más conocimiento aportan sobre el proceso de plegamiento. No usables aún (en general) para predicción 3D por - Potenciales empíricos y semiempíricos con pequeñas inexactitudes que se acumulan para proteínas grandes y/o simulaciones largas. - Requieren muchísima potencia de calculo.
8 Modelado por Homología vs. Threading Espacio de secuencias Espacio de estructuras Diseño por homología threading
9 Modelado por homología Relación entre parecido estructural y parecido en secuencia Chothia, C. & Lesk, A.M. (1986) The relation between the divergence of sequence and structure in proteins. EMBO J., 5, Sander, C. & Schneider, R. (1993) The HSSP data base of protein structure-sequence alignments. Nucleic Acids Res., 21,
10 Modelado por Homología. Estrategia general Localizar molde Generar alineamiento entre secuencia problema y molde Para átomos del esqueleto tomar las coordenadas del molde. Para aa. conservados tomar mismas coordenadas para las cadenas laterales Cadenas laterales de otros aa. - usar librerías de rotámeros - usar la mayor cantidad de átomos posibles (Cβ -> C γ,...) Modelar loops (inserciones y delecciones) Optimizar empaquetamiento (MD,...) Evaluar calidad del modelo
11 Modelado por Homología. Alineamiento molde-proteína problema? target template model
12 Modelado por Homología. Alineamiento molde-proteína problema
13 Modelado por Homología. Cadenas laterales
14 Modelado por Homología. Nucleo Estructural
15 Modelado por Homología. Loops
16 Modelado por Homología Evaluación de Modelos
17 Modelado por Homología Servidores Públicos y Repositorios de Modelos SWISS-MODEL - An automated comparative modelling server (ExPASy, CH) CPHmodels - Server using homology modelling (BioCentrum, Denmark) SDSC1 - cl.sdsc.edu/hm.html Protein structure homology modeling server (San Diego, USA) 3D-JIGSAW - Automated system for 3D models for proteins (Cancer Research UK)
18 Modelado por Homología vs. Threading Diseño por homología threading Espacio de secuencias Espacio de estructuras 20% id
19 Secuencia problema Threading. Estrategia general
20 Algoritmos de threading. General. 1. Library of protein structures (fold library) all known structures representative subset (seq. similarity filters) structural cores with loops removed ALMVWTGH Binary alignment algorithm with Scoring function contact potential environments Instead of aligning a sequence to a sequence, align strings of descriptors that represent 3D structual features. Usual Dynamic Programming: score matrix relates two amino acids Threading Dynamic Programming: relates amino acids to environments in 3D structure 3. Method for generating models via alignments (same as homology modl.)...
21 Algoritmos de threading Función de Puntuación - Aminoácido en ambiente similar a como suele estar en estructuras conocidas. - Potenciales de solvatación. - Potenciales de contacto. - Coincidencia de estructuras secundarias (real y predicha) y accesibilidades. - Matrices de homología remota extraídas de alineamientos estructurales Búsqueda con Modelos de Markov (HMMs).... ALMVWTGH...
22 Algoritmos de threading Potenciales de contacto (potenciales estadísticos) counts A V Energy of interaction = -KT ln (frequency of interactions) Boltzmann principle d Count pairs of each residue type at different separations E d Jones, D., Taylor, W. and Thornton, J. (1992) A new approach to protein fold recognition. Nature, 358, Sippl, M.J. (1995) Knowledge-based potentials for proteins. Curr Opin Struct Biol, 5,
23 Algoritmos de threading Coincidencia de estructura secundaria y accesibilidad Secondary structure prediction Rost, B. (1995) TOPITS: Threading one dimensional predictions into three dimensional structures. In Rawlings, C. and als, e. (eds.), Third International Conference on Intelligent Systems for Molecular Biology. AAAI Press. Menlo Park, CA. USA., England, pp
24 Algoritmos de threading Perfiles de secuencia + estructura secundaria Kelley, L.A., MacCallum, R.M. and Sternberg, M.J.E. (2000) Enhanced genome annotation using structural profiles in the program 3D-PSSM. J Mol Biol, 299,
25 Threading Post-procesamiento de resultados Combinación con información adicional
26 Threading Post-procesamiento de resultados Meta-servidores (Metaserver + 3D-jury ) Ginalski K, Rychlewski L. (2003). Detection of reliable and unexpected protein fold predictions using 3D-Jury.Nucleic Acids Res. 31(13):
27 Predicción de Estructura Mini-threading + ab-initio Rosetta I-sites PWGCVV AALWLV Ab-initio simulación MonteCarlo Simons, K.T., Kooperberg, C., Huang, E. & Baker, D. (1997) Assembly of protein tertiary structures from fragments with similar local sequences using simulated annealing and bayesian scoring functions. J Mol Biol, 268,
28 Modelado por Homología vs. Threading % id seq. con alguna estructura threading modelado por homología aplicación calidad modelos cualquier secuencia fold alineamiento - diferencia en loops y gaps - movimientos de dominios - cambios en el backbone >= 30% con algun PDB nivel atómico - cadenas laterales - loops 50 70
29 R. Russell EMBL
30 Evaluación de métodos de predicción I) CASP (bianual 94-06) MAKEFGIPAAVAGTVLNVVEAGGWVTTIVSILTAVGSG GLSLLAAAGRESIKAYLKKEI KKGKRAVIAW Databases Algorithm Computer evaluation MODEL(S) EVALUATION Proteins biannual issue on CASP/CAFASP
31 Evaluación de métodos de predicción II) CAFASP (bianual 98-06) MAKEFGIPAAVAGTVLNVVEAGGWVTTIVSILTAVGSG GLSLLAAAGRESIKAYLKKEI KKGKRAVIAW EVALUATION MODEL(S) Proteins biannual issue on CASP/CAFASP
32 Evaluación de métodos III) EVA/LiveBench Koh, I.Y.Y., Eyrich, V.A., Marti-Renom, M.A., Przybylski, D., Madhusudhan, M.S., Eswar, N., Grana, O., Pazos, F., Valencia, A., Sali, A., et al. (2003) EVA: evaluation of protein structure prediction servers. Nucl. Acids. Res., 31, Eyrich, V.A., Marti-Renom, M.A., Przybylski, D., Madhusudhan, M.S., Fiser, A., Pazos, F., Valencia, A., Sali, A. and Rost, B. (2001) EVA: continuous automatic evaluation of protein structure prediction servers. Bioinformatics, 17,
33 4 weeks running EVA => mora targets than CASP
34 Predicción de Estructura de Proteínas Futuro? - Combinación de métodos de predicción con datos estructurales a baja resolución - Integración en los proyectos de structural genomics. - Predicción de función Qu, Y., Guo, J.T., Olman, V. and Xu, Y. (2004) Protein structure prediction using sparse dipolar coupling data. Nucleic Acids Res, 32, Print Arakaki, A.K., Zhang, Y. and Skolnick, J. (2004) Large-scale assessment of the utility of low-resolution protein structures for biochemical function assignment. Bioinformatics, 20, Meiler, J. and Baker, D. (2003) Rapid protein fold determination using unassigned NMR data. Proc Natl Acad Sci U S A, 100, Li, W., Zhang, Y., Kihara, D., Huang, Y.J., Zheng, D., Montelione, G.T., Kolinski, A. and Skolnick, J. (2003) TOUCHSTONEX: Protein structure prediction with sparse NMR data. Proteins, 53, Zheng, W. and Doniach, S. (2002) Protein structure prediction constrainded by solution x-ray scattering data and structural homology identification. J Mol Biol, 316,
35
Modelado 3D. Introducción
 Modelado 3D. Introducción Centro de Investigacion y Tecnologia Agroalimentaria. CITA - DGA. Paulino Gómez Puertas. Centro de Biología Molecular "Severo Ochoa" CSIC-UAM, Madrid http://www.cbm.uam.es/bioweb
Modelado 3D. Introducción Centro de Investigacion y Tecnologia Agroalimentaria. CITA - DGA. Paulino Gómez Puertas. Centro de Biología Molecular "Severo Ochoa" CSIC-UAM, Madrid http://www.cbm.uam.es/bioweb
Programas para Alineamientos Múltiples
 Programas para Alineamientos Múltiples http://en.wikipedia.org/wiki/sequence_alignment_software Alindamientos múltiples de proteínas y comparaciones DB Los análisis filogenéticos basados en alineamientos
Programas para Alineamientos Múltiples http://en.wikipedia.org/wiki/sequence_alignment_software Alindamientos múltiples de proteínas y comparaciones DB Los análisis filogenéticos basados en alineamientos
Modelado Molecular 3D de Sistemas de Interés Biológico
 Curso de postgrado de la Carrera de Doctorado en Ciencias Biológicas Modelado Molecular 3D de Sistemas de Interés Biológico Docentes a cargo: Drs. Daniel E. Rodrigues y Silvano J. Sferco (teoría), y Drs.
Curso de postgrado de la Carrera de Doctorado en Ciencias Biológicas Modelado Molecular 3D de Sistemas de Interés Biológico Docentes a cargo: Drs. Daniel E. Rodrigues y Silvano J. Sferco (teoría), y Drs.
Estructura 3D de proteínas (2)
 Estructura 3D de proteínas (2) Aportaciones de la Bioinformática Structural Bioinformatics Laboratory Research Group on Biomedical Informatics (GRIB) Universitat Pompeu Fabra Sumario Conceptos básicos
Estructura 3D de proteínas (2) Aportaciones de la Bioinformática Structural Bioinformatics Laboratory Research Group on Biomedical Informatics (GRIB) Universitat Pompeu Fabra Sumario Conceptos básicos
Identificación de sitios en proteínas usando máquinas con vectores de soporte
 NOVA - PUBLICACIÓN CIENTÍFICA ISSN:1794-2370 VOL.1 No. 1 ENERO - DICIEMBRE DE 2003:1-116 ARTÍCULO ORIGINAL 65 Identificación de sitios en proteínas usando máquinas con vectores de soporte Jaime Leonardo
NOVA - PUBLICACIÓN CIENTÍFICA ISSN:1794-2370 VOL.1 No. 1 ENERO - DICIEMBRE DE 2003:1-116 ARTÍCULO ORIGINAL 65 Identificación de sitios en proteínas usando máquinas con vectores de soporte Jaime Leonardo
Modelado Molecular 3D de Sistemas de Interés Biológico
 Curso de posgrado de la Carrera de Doctorado en Ciencias Biológicas Modelado Molecular 3D de Sistemas de Interés Biológico Docentes a cargo: Dres. Alberto S. Garay, Daniel E. Rodrigues y Silvano J. Sferco
Curso de posgrado de la Carrera de Doctorado en Ciencias Biológicas Modelado Molecular 3D de Sistemas de Interés Biológico Docentes a cargo: Dres. Alberto S. Garay, Daniel E. Rodrigues y Silvano J. Sferco
Modelado Molecular 3D de Sistemas de Interés Biológico
 FACULTAD DE BIOQUÍMICA Y CIENCIAS BIOLÓGICAS UNIVERSIDAD NACIONAL DEL LITORAL SANTA FE, ARGENTINA Modelado Molecular 3D de Sistemas de Interés Biológico Docentes a cargo: Drs. Daniel E. Rodrigues y Silvano
FACULTAD DE BIOQUÍMICA Y CIENCIAS BIOLÓGICAS UNIVERSIDAD NACIONAL DEL LITORAL SANTA FE, ARGENTINA Modelado Molecular 3D de Sistemas de Interés Biológico Docentes a cargo: Drs. Daniel E. Rodrigues y Silvano
Definición de Umbral Mínimo para la Predicción de Estructura Secundaria de Proteínas
 Definición de Umbral Mínimo para la Predicción de Estructura Secundaria de Proteínas Alfonso E. Márquez Chamorro 1, Jesús S. Aguilar Ruiz 1 y Eloy Anguiano Rey 2 1 Departamento de lenguajes y sistemas,
Definición de Umbral Mínimo para la Predicción de Estructura Secundaria de Proteínas Alfonso E. Márquez Chamorro 1, Jesús S. Aguilar Ruiz 1 y Eloy Anguiano Rey 2 1 Departamento de lenguajes y sistemas,
Usando potenciales nativo-céntricos para simular el plegamiento de proteínas.
 DEPARTAMENTO DE BIOLOGIA FACULTAD DE CIENCIAS UNIVERSIDAD DE CHILE Usando potenciales nativo-céntricos para simular el plegamiento de proteínas. Cesar A. Ramirez-Sarmiento. Laboratorio de Bioquimica y
DEPARTAMENTO DE BIOLOGIA FACULTAD DE CIENCIAS UNIVERSIDAD DE CHILE Usando potenciales nativo-céntricos para simular el plegamiento de proteínas. Cesar A. Ramirez-Sarmiento. Laboratorio de Bioquimica y
Bioinformática Clásica
 Tema 4: Alineamiento Múltiple y Filogenias (1) Motivos y Perfiles Dr. Oswaldo Trelles Universidad de Málaga Los motivos o perfiles son pequeños fragmentos, trozos o zonas conservadas presentes en un grupo
Tema 4: Alineamiento Múltiple y Filogenias (1) Motivos y Perfiles Dr. Oswaldo Trelles Universidad de Málaga Los motivos o perfiles son pequeños fragmentos, trozos o zonas conservadas presentes en un grupo
Elucidación Estructural de Biomoléculas utilizando Resonancia Magnética Nuclear
 Universidad de Los Andes Facultad de Ciencias Postgrado Interdisciplinario en Química Aplicada Mención Estudio de Materiales Elucidación Estructural de Biomoléculas utilizando Resonancia Magnética Nuclear
Universidad de Los Andes Facultad de Ciencias Postgrado Interdisciplinario en Química Aplicada Mención Estudio de Materiales Elucidación Estructural de Biomoléculas utilizando Resonancia Magnética Nuclear
Alineamientos de Secuencias. CeCalCULA - C.P.T.M. Mérida. Venezuela.
 Alineamientos de Secuencias Análisis comparativo El alineamiento de secuencias es similar a otros tipos de análisis comparativo. En ambos es necesario cuantificar las similitudes y diferencias (scoring)
Alineamientos de Secuencias Análisis comparativo El alineamiento de secuencias es similar a otros tipos de análisis comparativo. En ambos es necesario cuantificar las similitudes y diferencias (scoring)
Estructura y estabilidad de un dominio proteico BRCT. Obregón Mansilla, Alexandra J. I. CAPÍTULO II ANTECEDENTES
 CAPÍTULO II ANTECEDENTES Frecuentemente se observa alteraciones en la abundancia de proteínas reguladoras críticas en células cancerosas (20). La pérdida de proteínas supresoras de tumores o el incremento
CAPÍTULO II ANTECEDENTES Frecuentemente se observa alteraciones en la abundancia de proteínas reguladoras críticas en células cancerosas (20). La pérdida de proteínas supresoras de tumores o el incremento
UNIVERSIDAD NACIONAL MAYOR DE SAN MARCOS (Universidad del Perú, Decana de América) SYLLABUS DE MODELIZACIÓN DE BIOMOLÉCULAS
 UNIVERSIDAD NACIONAL MAYOR DE SAN MARCOS (Universidad del Perú, Decana de América) FACULTAD DE CIENCIAS BIOLÓGICAS ESCUELA ACADÉMICO PROFESIONAL DE GENÉTICA Y BIOTECNOLOGÍA DEPARTAMENTO ACADÉMICO DE BIOLOGÍA
UNIVERSIDAD NACIONAL MAYOR DE SAN MARCOS (Universidad del Perú, Decana de América) FACULTAD DE CIENCIAS BIOLÓGICAS ESCUELA ACADÉMICO PROFESIONAL DE GENÉTICA Y BIOTECNOLOGÍA DEPARTAMENTO ACADÉMICO DE BIOLOGÍA
Identificación de Familias de Proteínas Cursos de Doctorado 2007
 Identificación de Familias de Proteínas Cursos de Doctorado 2007 Luis Sánchez Pulido Centro Nacional de Biotecnología Madrid Por qué analizamos secuencias? Proteínas de Secuencia conocida 3D Función Ambas?????
Identificación de Familias de Proteínas Cursos de Doctorado 2007 Luis Sánchez Pulido Centro Nacional de Biotecnología Madrid Por qué analizamos secuencias? Proteínas de Secuencia conocida 3D Función Ambas?????
Búsqueda de secuencias en Bases de Datos.
 Búsqueda de secuencias en Bases de Datos. Existe una amplia red de bases de datos en diferentes servidores científicos que permiten acceder a una gran cantidad de información científica. Y entre ella,
Búsqueda de secuencias en Bases de Datos. Existe una amplia red de bases de datos en diferentes servidores científicos que permiten acceder a una gran cantidad de información científica. Y entre ella,
CARACTERIZACIÓN DE LA INTERACCIÓN ENTRE CLUSTER DE Aun Y AMINOÁCIDOS AZUFRADOS WALDO ANDRÉS ACEVEDO CASTILLO INGENIERO EN BIOINFORMÁTICA RESUMEN
 CARACTERIZACIÓN DE LA INTERACCIÓN ENTRE CLUSTER DE Aun Y AMINOÁCIDOS AZUFRADOS WALDO ANDRÉS ACEVEDO CASTILLO INGENIERO EN BIOINFORMÁTICA RESUMEN En el ámbito biológico es posible caracterizar ciertas reacciones
CARACTERIZACIÓN DE LA INTERACCIÓN ENTRE CLUSTER DE Aun Y AMINOÁCIDOS AZUFRADOS WALDO ANDRÉS ACEVEDO CASTILLO INGENIERO EN BIOINFORMÁTICA RESUMEN En el ámbito biológico es posible caracterizar ciertas reacciones
Práctica modelado por homología
 Práctica modelado por homología La estructura de las proteínas está más conservada que su secuencia. Esto implica que si una proteína con estructura desconocida comparte más de un 30% de porcentaje de
Práctica modelado por homología La estructura de las proteínas está más conservada que su secuencia. Esto implica que si una proteína con estructura desconocida comparte más de un 30% de porcentaje de
200630 - FBIO - Fundamentos de Bioinformática
 Unidad responsable: Unidad que imparte: Curso: Titulación: Créditos ECTS: 2016 200 - FME - Facultad de Matemáticas y Estadística 1004 - UB - Universitat de Barcelona MÁSTER UNIVERSITARIO EN ESTADÍSTICA
Unidad responsable: Unidad que imparte: Curso: Titulación: Créditos ECTS: 2016 200 - FME - Facultad de Matemáticas y Estadística 1004 - UB - Universitat de Barcelona MÁSTER UNIVERSITARIO EN ESTADÍSTICA
Análisis de Secuencias, Familias de Proteínas. Masters en Bioinformática Madrid 2005. Michael Tress Protein Design Group
 Análisis de Secuencias, Familias de Proteínas Masters en Bioinformática Madrid 2005 Michael Tress Protein Design Group Lo que encontramos en las bases de datos Observación: las proteínas homólogas pueden
Análisis de Secuencias, Familias de Proteínas Masters en Bioinformática Madrid 2005 Michael Tress Protein Design Group Lo que encontramos en las bases de datos Observación: las proteínas homólogas pueden
UPR RECINTO DE RÍO PIEDRAS FACULTAD DE CIENCIAS NATURALES DEPARTAMENTO DE BIOLOGÍA LABORATORIO DE BIOTECNOLOGÍA (BIOL. 3365)
 UPR RECINTO DE RÍO PIEDRAS FACULTAD DE CIENCIAS NATURALES DEPARTAMENTO DE BIOLOGÍA LABORATORIO DE BIOTECNOLOGÍA (BIOL. 3365) Análisis del gen AmyE mediante el uso de herramientas de bioinformática A. Objetivos
UPR RECINTO DE RÍO PIEDRAS FACULTAD DE CIENCIAS NATURALES DEPARTAMENTO DE BIOLOGÍA LABORATORIO DE BIOTECNOLOGÍA (BIOL. 3365) Análisis del gen AmyE mediante el uso de herramientas de bioinformática A. Objetivos
TRANSCRIPCIÓN Y REPLICACIÓN DEL RNA DEL VIRUS DE LA GRIPE
 TRANSCRIPCIÓN Y REPLICACIÓN DEL RNA DEL VIRUS DE LA GRIPE Juan Ortín Resumen Los virus de la gripe tienen gran incidencia en la salud humana, ya que disponen de un amplio reservorio animal y reaparecen
TRANSCRIPCIÓN Y REPLICACIÓN DEL RNA DEL VIRUS DE LA GRIPE Juan Ortín Resumen Los virus de la gripe tienen gran incidencia en la salud humana, ya que disponen de un amplio reservorio animal y reaparecen
Predicción de la estructura terciara de las proteínas
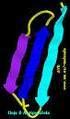 Predicción de la estructura terciara de las proteínas Cuando la estructura terciaria de una proteína no se ha determinado experimentalmente, se puede intentar construir un modelo tridimensional a partir
Predicción de la estructura terciara de las proteínas Cuando la estructura terciaria de una proteína no se ha determinado experimentalmente, se puede intentar construir un modelo tridimensional a partir
IDENTIFICACIÓN GENERAL. Bioinformática aplicada. Maestría en Ciencias Básicas Biomédicas. IDENTIFICACION ESPECÍFICA (Ver nota 1)
 Fecha: 07-04-2015 Pág. 1 de 5 IDENTIFICACIÓN GENERAL Nombre del curso Programa académico Bioinformática aplicada Maestría en Ciencias Básicas Biomédicas IDENTIFICACION ESPECÍFICA (Ver nota 1) Código 8501-279
Fecha: 07-04-2015 Pág. 1 de 5 IDENTIFICACIÓN GENERAL Nombre del curso Programa académico Bioinformática aplicada Maestría en Ciencias Básicas Biomédicas IDENTIFICACION ESPECÍFICA (Ver nota 1) Código 8501-279
Análisis de secuencias. Patrones, perfiles y dominios.
 Análisis de secuencias. Patrones, perfiles y dominios. Curso de verano de Bioinformática y Biología Computacional de la UCM, Madrid 2004 Federico Abascal Museo Nacional de Ciencias Naturales Recordatorio
Análisis de secuencias. Patrones, perfiles y dominios. Curso de verano de Bioinformática y Biología Computacional de la UCM, Madrid 2004 Federico Abascal Museo Nacional de Ciencias Naturales Recordatorio
Análisis in silico: Modelado de Proteínas.
 Tlamati Sabiduría Volumen 8 Número Especial 2, Octubre 2017 Introducción 4 r. Encuentro de Jóvenes en la Investigación de Bachillerato-CONACYT Acapulco, Guerrero 4, 5 y 6 de octubre 2017 Memorias Análisis
Tlamati Sabiduría Volumen 8 Número Especial 2, Octubre 2017 Introducción 4 r. Encuentro de Jóvenes en la Investigación de Bachillerato-CONACYT Acapulco, Guerrero 4, 5 y 6 de octubre 2017 Memorias Análisis
DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS
 DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS COMPARACION DE DOS ATPASAS DE PECES El DOT PLOT permite una visualización rápida de la similitud entre dos secuencias Inconvenientes: No identifica
DOT PLOT: VISUALIZACIÓN DE LA SIMILITUD ENTRE DOS SECUENCIAS COMPARACION DE DOS ATPASAS DE PECES El DOT PLOT permite una visualización rápida de la similitud entre dos secuencias Inconvenientes: No identifica
ANÁLISIS IN SILICO DE ESTRUCTURAS TRIDIMENSIONALES DE LA PROTEÍNA RPN11 DEL PROTEOSOMA 26S PRESENTES EN DIFERENTES ESPECIES
 INFORME FINAL DE INVESTIGACIÓN ANÁLISIS IN SILICO DE ESTRUCTURAS TRIDIMENSIONALES DE LA PROTEÍNA RPN11 DEL PROTEOSOMA 26S PRESENTES EN DIFERENTES ESPECIES Responsable del Estudio Dr. Gustavo Adolfo Sandoval
INFORME FINAL DE INVESTIGACIÓN ANÁLISIS IN SILICO DE ESTRUCTURAS TRIDIMENSIONALES DE LA PROTEÍNA RPN11 DEL PROTEOSOMA 26S PRESENTES EN DIFERENTES ESPECIES Responsable del Estudio Dr. Gustavo Adolfo Sandoval
Bancos de datos en Biología Molecular, un desarrollo vertiginoso.
 Bancos de datos en Biología Molecular, un desarrollo vertiginoso. Resumen: la introducción del computador como pieza clave en el trabajo en biologia molecular trajo consigo tambien un gran cambio en la
Bancos de datos en Biología Molecular, un desarrollo vertiginoso. Resumen: la introducción del computador como pieza clave en el trabajo en biologia molecular trajo consigo tambien un gran cambio en la
FACULTAD DE CIENCIAS EXACTAS Y NATURALES
 FACULTAD DE CIENCIAS EXACTAS Y NATURALES Información para el cumplimiento de la Resolución CS Nº 2210/03 Asignatura (6.1) Fundamentos y Objetivos. (6.2) Puntos de articulación con respecto al plan de la
FACULTAD DE CIENCIAS EXACTAS Y NATURALES Información para el cumplimiento de la Resolución CS Nº 2210/03 Asignatura (6.1) Fundamentos y Objetivos. (6.2) Puntos de articulación con respecto al plan de la
Algoritmos Genéticos Y
 Algoritmos Genéticos Y Optimización n Heurística Dr. Adrian Will Grupo de Aplicaciones de Inteligencia Artificial Universidad Nacional de Tucumán awill@herrera.unt.edu.ar Optimización n Tradicional Problemas
Algoritmos Genéticos Y Optimización n Heurística Dr. Adrian Will Grupo de Aplicaciones de Inteligencia Artificial Universidad Nacional de Tucumán awill@herrera.unt.edu.ar Optimización n Tradicional Problemas
2 Congreso Colombiano de Bioinformática y biología computacional.
 2 Congreso Colombiano de Bioinformática y biología computacional. Presentación y evaluación de ABMS (Automatic Blast for Massive Annotation) Nelson Perez nelsonp@correo.udistrital.edu.co Cristian Rojas
2 Congreso Colombiano de Bioinformática y biología computacional. Presentación y evaluación de ABMS (Automatic Blast for Massive Annotation) Nelson Perez nelsonp@correo.udistrital.edu.co Cristian Rojas
DES: Programa(s) Educativo(s): Tipo de materia: Clave de la materia: Semestre:
 : : lemas propios de la. lemas propios de la. lemas propios de la. lemas propios de la. lemas propios de la. lemas propios de la. lemas propios de la. 12 6 lemas propios de la. 12 6 lemas propios de la.
: : lemas propios de la. lemas propios de la. lemas propios de la. lemas propios de la. lemas propios de la. lemas propios de la. lemas propios de la. 12 6 lemas propios de la. 12 6 lemas propios de la.
Práctica X: Genómica comparativa
 Práctica X: Genómica comparativa Objetivo: Ø Conocer y emplear diversos programas bioinformáticos para realizar estudios de genómica comparativa. Objetivos particulares: Analizar las secuencias de genomas
Práctica X: Genómica comparativa Objetivo: Ø Conocer y emplear diversos programas bioinformáticos para realizar estudios de genómica comparativa. Objetivos particulares: Analizar las secuencias de genomas
Real Time Systems. Part 2: Cyclic schedulers. Real Time Systems. Francisco Martín Rico. URJC. 2011
 Real Time Systems Part 2: Cyclic schedulers Scheduling To organise the use resources to guarantee the temporal requirements A scheduling method is composed by: An scheduling algorithm that calculates the
Real Time Systems Part 2: Cyclic schedulers Scheduling To organise the use resources to guarantee the temporal requirements A scheduling method is composed by: An scheduling algorithm that calculates the
Relatos de Viaje: Últimas tendencias en modelos predictivos y su aplicación en y fuera del salón de clases
 Relatos de Viaje: Últimas tendencias en modelos predictivos y su aplicación en y fuera del salón de clases Marta Álvarez, Ph.D. Catedrática Instituto de Estadística y Sistemas Computadorizados de Información
Relatos de Viaje: Últimas tendencias en modelos predictivos y su aplicación en y fuera del salón de clases Marta Álvarez, Ph.D. Catedrática Instituto de Estadística y Sistemas Computadorizados de Información
NOMBRE DE LA ASIGNATURA: Métodos Computacionales en Bioinformática
 NOMBRE DE LA ASIGNATURA: Métodos Computacionales en Bioinformática CLAVE DE LA ASIGNATURA: 9310 RESPONSABLE DE LA ASIGNATURA: Dr. Ricardo Vázquez Juárez Suplente: Dr. Joaquín Gutiérrez Jagüey PARTICIPANTES:
NOMBRE DE LA ASIGNATURA: Métodos Computacionales en Bioinformática CLAVE DE LA ASIGNATURA: 9310 RESPONSABLE DE LA ASIGNATURA: Dr. Ricardo Vázquez Juárez Suplente: Dr. Joaquín Gutiérrez Jagüey PARTICIPANTES:
Introducción a la Bioinformática Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia
 Introducción a la Bioinformática Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Andrés M. Pinzón cphd - Universidad de los Andes 7'000.000 de habitantes Capital Mundial
Introducción a la Bioinformática Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Andrés M. Pinzón cphd - Universidad de los Andes 7'000.000 de habitantes Capital Mundial
La Bioinformática: una perspectiva de la estructura de proteínas
 La bioinformática: una perspectiva... La Bioinformática: una perspectiva de la estructura de proteínas Darrell Conklin* Resumen El Proyecto Genoma Humano está produciendo una inmensa cantidad de secuencias
La bioinformática: una perspectiva... La Bioinformática: una perspectiva de la estructura de proteínas Darrell Conklin* Resumen El Proyecto Genoma Humano está produciendo una inmensa cantidad de secuencias
Introducción al NCBI
 Introducción al NCBI National Center for Biotechnology Information Andrés M. Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Qué es el NCBI? http://www.ncbi.nlm.nih.gov/
Introducción al NCBI National Center for Biotechnology Information Andrés M. Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Qué es el NCBI? http://www.ncbi.nlm.nih.gov/
Métodos Markov Chain Monte Carlo
 Métodos Markov Chain Monte Carlo David J. Rios Optimización Combinatoria 19 de mayo del 2008 MCMC Introducción Que son Cadenas de Markov? Que es Monte Carlo? Que es Markov Chain Monte Carlo? Algoritmo
Métodos Markov Chain Monte Carlo David J. Rios Optimización Combinatoria 19 de mayo del 2008 MCMC Introducción Que son Cadenas de Markov? Que es Monte Carlo? Que es Markov Chain Monte Carlo? Algoritmo
BASES DE DATOS DE INTERÉS EN BIOQUÍMICA
 BASES DE DATOS DE INTERÉS EN BIOQUÍMICA Las técnicas de alto rendimiento desarrolladas en las últimas décadas han permitido la adquisición masiva de información de biología molecular que se depositan en
BASES DE DATOS DE INTERÉS EN BIOQUÍMICA Las técnicas de alto rendimiento desarrolladas en las últimas décadas han permitido la adquisición masiva de información de biología molecular que se depositan en
PRACTICA XV: PREDICCION DE ESTRUCTURA SECUNDARIA DE PROTEINAS Y PROPIEDADES 1D
 PRACTICA XV: PREDICCION DE ESTRUCTURA SECUNDARIA DE PROTEINAS Y PROPIEDADES 1D Objetivo General: Ø Que el alumno realice la predicción de la estructura secundaria de proteínas utilizando distintas herramientas
PRACTICA XV: PREDICCION DE ESTRUCTURA SECUNDARIA DE PROTEINAS Y PROPIEDADES 1D Objetivo General: Ø Que el alumno realice la predicción de la estructura secundaria de proteínas utilizando distintas herramientas
Enfermedad Mendeliana. Búsqueda en bases de datos con proteínas homólogas. Clonaje in silico del gen. Gen candidato
 Enfermedad Mendeliana Defecto determinado por métodos bioquímicos Sin pistas bioquímicas Identificación proteína Clonaje funcional del gen Búsqueda en bases de datos con proteínas homólogas Clonaje in
Enfermedad Mendeliana Defecto determinado por métodos bioquímicos Sin pistas bioquímicas Identificación proteína Clonaje funcional del gen Búsqueda en bases de datos con proteínas homólogas Clonaje in
Analisis de Secuencia Orientado a Dominios Cursos de Doctorado 2006
 Analisis de Secuencia Orientado a Dominios Cursos de Doctorado 2006 UAM Luis Sánchez Pulido Centro Nacional de Biotecnología CSIC Por qué analizamos secuencias? Proteínas de Secuencia conocida 3D Función
Analisis de Secuencia Orientado a Dominios Cursos de Doctorado 2006 UAM Luis Sánchez Pulido Centro Nacional de Biotecnología CSIC Por qué analizamos secuencias? Proteínas de Secuencia conocida 3D Función
Bioinformática estructural
 Bioinformática estructural Predicción de estructuras de proteínas y ARN Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 25 de julio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Bioinformática estructural
Bioinformática estructural Predicción de estructuras de proteínas y ARN Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 25 de julio del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) Bioinformática estructural
APLICACIONES BIOLÓGICAS DE LA ESPECTROSCOPIA VIBRACIONAL. P. Carmona Instituto de Estructura de la Materia (CSIC)
 APLICACIONES BIOLÓGICAS DE LA ESPECTROSCOPIA VIBRACIONAL P. Carmona Instituto de Estructura de la Materia (CSIC) GRUPO DE BIOESPECTROSCOPIA Aplicaciones Biológicas de la Espectroscopía Vibracional Estudios
APLICACIONES BIOLÓGICAS DE LA ESPECTROSCOPIA VIBRACIONAL P. Carmona Instituto de Estructura de la Materia (CSIC) GRUPO DE BIOESPECTROSCOPIA Aplicaciones Biológicas de la Espectroscopía Vibracional Estudios
TÓPICO: MODELADO MOLECULAR DE PROTEÍNAS
 TÓPICO: MODELADO MOLECULAR DE PROTEÍNAS DOCENTES A CARGO: Dr. Ramón Garduño Juárez (ICF) y Dr. Sergio Mares Samano (Catedrático CONACyT) INTRODUCCIÓN Los métodos de modelado molecular se han convertido
TÓPICO: MODELADO MOLECULAR DE PROTEÍNAS DOCENTES A CARGO: Dr. Ramón Garduño Juárez (ICF) y Dr. Sergio Mares Samano (Catedrático CONACyT) INTRODUCCIÓN Los métodos de modelado molecular se han convertido
Cloud computing y diseño de fármacos: Docking Virtual sobre Amazon EC2 Diego Alonso López Ingeniero Informático Director: Luis M. A.
 Cloud computing y diseño de fármacos: Docking Virtual sobre Amazon EC2 Diego Alonso López Ingeniero Informático Director: Luis M. A. Quintales Facultad & Ciencias Universidad & Salamanca Objetivos Conceptos
Cloud computing y diseño de fármacos: Docking Virtual sobre Amazon EC2 Diego Alonso López Ingeniero Informático Director: Luis M. A. Quintales Facultad & Ciencias Universidad & Salamanca Objetivos Conceptos
UTILIZACIÓN DE MODELOS DE INTERPOLACIÓN DE MARKOV PARA LA
 UTILIZACIÓN DE MODELOS DE INTERPOLACIÓN DE MARKOV PARA LA IDENTIFICACIÓN DE SECUENCIAS DE GENES Marco Gerardo Torres Andrade Resumen Uno de los principales problemas a resolver en la bioinformática es
UTILIZACIÓN DE MODELOS DE INTERPOLACIÓN DE MARKOV PARA LA IDENTIFICACIÓN DE SECUENCIAS DE GENES Marco Gerardo Torres Andrade Resumen Uno de los principales problemas a resolver en la bioinformática es
Nociones básicas para el análisis bioinformático de proteínas
 Nociones básicas para el análisis bioinformático de proteínas Esther Menéndez Gutiérrez, Raúl Rivas González {esthermenendez, raulrg}@usal.es Departamento de Microbiología y Genética, Universidad de Salamanca
Nociones básicas para el análisis bioinformático de proteínas Esther Menéndez Gutiérrez, Raúl Rivas González {esthermenendez, raulrg}@usal.es Departamento de Microbiología y Genética, Universidad de Salamanca
OPTATIVA I: MINERIA DE DATOS
 UNIVERSIDAD AUTÓNOMA DE CHIHUAHUA Clave: 08MSU007H Clave: 08USU4053W FACULTAD DE INGENIERÍA PROGRAMA DEL CURSO: OPTATIVA I: MINERIA DE DATOS DES: Programa(s) Educativo(s): Tipo de materia: Clave de la
UNIVERSIDAD AUTÓNOMA DE CHIHUAHUA Clave: 08MSU007H Clave: 08USU4053W FACULTAD DE INGENIERÍA PROGRAMA DEL CURSO: OPTATIVA I: MINERIA DE DATOS DES: Programa(s) Educativo(s): Tipo de materia: Clave de la
Pymol - Práctica guiada
 Pymol - Práctica guiada Pymol es un potente visualizor de moleculas muy práctico para trabajar con proteínas. Su interfaz puede parecer compleja en un primer momento, pero vereis que con un poco de práctica
Pymol - Práctica guiada Pymol es un potente visualizor de moleculas muy práctico para trabajar con proteínas. Su interfaz puede parecer compleja en un primer momento, pero vereis que con un poco de práctica
Uso de herramientas para alineación
 Uso de herramientas para alineación de secuencias y creación de árboles filogenéticos para la determinación de especies Using tools for sequence alignment and outline of phylogenetic trees to determine
Uso de herramientas para alineación de secuencias y creación de árboles filogenéticos para la determinación de especies Using tools for sequence alignment and outline of phylogenetic trees to determine
Dinámica Molecular de Proteínas Modelado y Simulación Computacional
 Dinámica Molecular de Proteínas Modelado y Simulación Computacional Profesores: Eliana K. Asciutto & Ignacio J. General 2do cuatrimestre 2017 Escuela de Ciencia y Tecnología UNSAM Dinámica Molecular de
Dinámica Molecular de Proteínas Modelado y Simulación Computacional Profesores: Eliana K. Asciutto & Ignacio J. General 2do cuatrimestre 2017 Escuela de Ciencia y Tecnología UNSAM Dinámica Molecular de
DATOS DE CONTACTO. Otros: Community Management. Universidad Nacional de Educación a Distancia (UNED). 2013.
 DATOS DE CONTACTO Apellidos y Nombre: Valdés González Haydée Correo electrónico: hvaldesg10@gmail.com Página Web: www.hvaldesg.com Linked in: Haydée Valdés Research ID: D-8612-2013 D DATOS PERSONALES TOS
DATOS DE CONTACTO Apellidos y Nombre: Valdés González Haydée Correo electrónico: hvaldesg10@gmail.com Página Web: www.hvaldesg.com Linked in: Haydée Valdés Research ID: D-8612-2013 D DATOS PERSONALES TOS
Bases de datos biológicas
 Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 28 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) 28 de mayo del 2013 1 / 50 1 Introducción Desventajas de las bases de datos biológicas Recuperación
Dr. Eduardo A. RODRÍGUEZ TELLO CINVESTAV-Tamaulipas 28 de mayo del 2013 Dr. Eduardo RODRÍGUEZ T. (CINVESTAV) 28 de mayo del 2013 1 / 50 1 Introducción Desventajas de las bases de datos biológicas Recuperación
Construcción de matrices de distancia para comparar proteínas. Alexis Gómez, Thalía Rebón, Claudia Rendón, Pablo Soberón Colegio Marymount
 Construcción de matrices de distancia para comparar proteínas Alexis Gómez, Thalía Rebón, Claudia Rendón, Pablo Soberón Colegio Marymount Construcción de matrices de distancia para comparar proteínas Alexis
Construcción de matrices de distancia para comparar proteínas Alexis Gómez, Thalía Rebón, Claudia Rendón, Pablo Soberón Colegio Marymount Construcción de matrices de distancia para comparar proteínas Alexis
Darío Canales Ignacio Salas Oscar Yañez
 Estructura de RNA Darío Canales Ignacio Salas Oscar Yañez RNA Está presente tanto en las células procariotas como en las eucariotas. Pares de bases canónicas A/U G/C G/U (par inestable) G/U (par inestable)
Estructura de RNA Darío Canales Ignacio Salas Oscar Yañez RNA Está presente tanto en las células procariotas como en las eucariotas. Pares de bases canónicas A/U G/C G/U (par inestable) G/U (par inestable)
Departamento Biología Molecular Universidad Autónoma de Madrid. Redes Biológicas y Biología de Sistemas Abril 2008
 Departamento Biología Molecular Universidad Autónoma de Madrid Redes Biológicas y Biología de Sistemas 21-29 Abril 2008 Coordinación: Florencio Pazos (CNB-CSIC) Redes Metabólicas lorencio Pazos Cabaleiro
Departamento Biología Molecular Universidad Autónoma de Madrid Redes Biológicas y Biología de Sistemas 21-29 Abril 2008 Coordinación: Florencio Pazos (CNB-CSIC) Redes Metabólicas lorencio Pazos Cabaleiro
PRACTICA VI: BUSQUEDA DE SIMILITUDES EN BASES DE DATOS
 Objetivo: PRACTICA VI: BUSQUEDA DE SIMILITUDES EN BASES DE DATOS Ø Conocer el fundamento de los programas BLAST y FASTA y utilizarlos eficientemente para la búsqueda de similitudes de secuencias de DNA
Objetivo: PRACTICA VI: BUSQUEDA DE SIMILITUDES EN BASES DE DATOS Ø Conocer el fundamento de los programas BLAST y FASTA y utilizarlos eficientemente para la búsqueda de similitudes de secuencias de DNA
Grupo de investigación en Minería de Datos http://mida.usal.es
 Departamento de Informática y Automática Postgrado en Informática y Automática MÁSTER EN SISTEMAS INTELIGENTES ASIGNATURAS Introducción a la Minería de Datos Minería Web María N. Moreno García http://avellano.usal.es/~mmoreno
Departamento de Informática y Automática Postgrado en Informática y Automática MÁSTER EN SISTEMAS INTELIGENTES ASIGNATURAS Introducción a la Minería de Datos Minería Web María N. Moreno García http://avellano.usal.es/~mmoreno
Optimización global en espacios restringidos mediante el sistema inmune artificial.
 Optimización global en espacios restringidos mediante el sistema inmune artificial. Tesista: Daniel Trejo Pérez dtrejo@computacion.cs.cinvestav.mx Director de tesis: Dr. Carlos Artemio Coello Coello CINVESTAV-IPN
Optimización global en espacios restringidos mediante el sistema inmune artificial. Tesista: Daniel Trejo Pérez dtrejo@computacion.cs.cinvestav.mx Director de tesis: Dr. Carlos Artemio Coello Coello CINVESTAV-IPN
Bases de datos Biológicas. Andrés Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia
 Bases de datos Biológicas Andrés Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Por qué las bases de datos? Crecimiento exponencial de los datos biológicos
Bases de datos Biológicas Andrés Pinzón Centro de Bioinformática Instituto de Biotecnología Universidad Nacional de Colombia Por qué las bases de datos? Crecimiento exponencial de los datos biológicos
Martes 28. Alineamientos múltiples. Cedric Notredame (CRG) Lunes 17 Dominios de proteínas y homología remota. Viernes 7 Miércoles 12.
 Hoy Lunes 27 Martes 28 Jueves 30 Bases de datos Federico Abascal Alineamiento y búsqueda de homólogos Federico Abascal Alineamientos múltiples Cedric Notredame (CRG) Búsquedas con perfiles Tirso Pons,
Hoy Lunes 27 Martes 28 Jueves 30 Bases de datos Federico Abascal Alineamiento y búsqueda de homólogos Federico Abascal Alineamientos múltiples Cedric Notredame (CRG) Búsquedas con perfiles Tirso Pons,
Zinc en metaloproteinas y enzimas - 2
 Zinc en metaloproteinas y enzimas - 2 » Se les denomina dedos porque contienen dominios estructurales capaces de anclarse en los surcos de la doble hélice del ADN como si fueran dedos.» La mayoría de estas
Zinc en metaloproteinas y enzimas - 2 » Se les denomina dedos porque contienen dominios estructurales capaces de anclarse en los surcos de la doble hélice del ADN como si fueran dedos.» La mayoría de estas
CONTROL E INTEGRACIÓN EN RED DE SISTEMAS DE GENERACIÓN DISTRIBUÍDA
 eman ta zabal zazu EUSKAL HERRIKO UNIBERTSITATEA UNIVERSIDAD DEL PAÍS VASCO Encuentro Sectorial de Energías Renovables 2006 CONTROL E INTEGRACIÓN EN RED DE SISTEMAS DE GENERACIÓN DISTRIBUÍDA J. Xabier
eman ta zabal zazu EUSKAL HERRIKO UNIBERTSITATEA UNIVERSIDAD DEL PAÍS VASCO Encuentro Sectorial de Energías Renovables 2006 CONTROL E INTEGRACIÓN EN RED DE SISTEMAS DE GENERACIÓN DISTRIBUÍDA J. Xabier
51 Int. CI.: G06F 11/34 (2006.01) 71 Solicitantes: 72 Inventor/es: 74 Agente/Representante:
 19 OFICINA ESPAÑOLA DE PATENTES Y MARCAS ESPAÑA 11 21 Número de publicación: 2 427 645 Número de solicitud: 201131833 51 Int. CI.: G06F 11/34 (2006.01) 12 INFORME SOBRE EL ESTADO DE LA TÉCNICA R1 22 Fecha
19 OFICINA ESPAÑOLA DE PATENTES Y MARCAS ESPAÑA 11 21 Número de publicación: 2 427 645 Número de solicitud: 201131833 51 Int. CI.: G06F 11/34 (2006.01) 12 INFORME SOBRE EL ESTADO DE LA TÉCNICA R1 22 Fecha
Búsqueda de conformaciones estables de interacción proteínaproteína utilizando métodos de inteligencia computacional. Juan Guillermo Carvajal Patiño
 Búsqueda de conformaciones estables de interacción proteínaproteína utilizando métodos de inteligencia computacional Juan Guillermo Carvajal Patiño Universidad Nacional de Colombia Facultad de Ingeniería,
Búsqueda de conformaciones estables de interacción proteínaproteína utilizando métodos de inteligencia computacional Juan Guillermo Carvajal Patiño Universidad Nacional de Colombia Facultad de Ingeniería,
Agrupación de secuencias genómicas bajo la distancia de edición
 Agrupación de secuencias genómicas bajo la distancia de edición Dr. Abdiel Emilio Cáceres González Resumen La distancia de edición, también conocida como distancia de Levenshtein ha sido utilizada frecuentemente
Agrupación de secuencias genómicas bajo la distancia de edición Dr. Abdiel Emilio Cáceres González Resumen La distancia de edición, también conocida como distancia de Levenshtein ha sido utilizada frecuentemente
Diseño e implementación de un sistema informático para la evaluación rápida de impactos ambientales
 Diseño e implementación de un sistema informático para la evaluación rápida de impactos ambientales Kiko Alexi Delgado Villanueva To cite this version: Kiko Alexi Delgado Villanueva. Diseño e implementación
Diseño e implementación de un sistema informático para la evaluación rápida de impactos ambientales Kiko Alexi Delgado Villanueva To cite this version: Kiko Alexi Delgado Villanueva. Diseño e implementación
ANX-PR/CL/001-02 GUÍA DE APRENDIZAJE ASIGNATURA CURSO ACADÉMICO - SEMESTRE FECHA DE PUBLICACION
 ANX-PR/CL/001-02 GUÍA DE APRENDIZAJE ASIGNATURA Modelling and control of dc dc converters CURSO ACADÉMICO - SEMESTRE 2014/2015 Primer semestre FECHA DE PUBLICACION Fecha Datos Descriptivos Nombre de la
ANX-PR/CL/001-02 GUÍA DE APRENDIZAJE ASIGNATURA Modelling and control of dc dc converters CURSO ACADÉMICO - SEMESTRE 2014/2015 Primer semestre FECHA DE PUBLICACION Fecha Datos Descriptivos Nombre de la
PREREQUISITOS: Biología Molecular de la Célula, Biomoléculas: estructura, función y propiedades, Fisiología y Metabolismo Microbiano.
 80111 LABORATORIO DE TÉCNICAS EXPERIMENTALES Pág 1 de 5 ASIGNATURA: LABORATORIO DE TÉCNICAS EXPERIMENTALES ESTUDIOS: MÁSTER EN BIOINGENIERÍA CÓDIGO: 80135 TIPO: O SEMESTRE: 1º CRÉDITOS: 9 (15 horas/semana)
80111 LABORATORIO DE TÉCNICAS EXPERIMENTALES Pág 1 de 5 ASIGNATURA: LABORATORIO DE TÉCNICAS EXPERIMENTALES ESTUDIOS: MÁSTER EN BIOINGENIERÍA CÓDIGO: 80135 TIPO: O SEMESTRE: 1º CRÉDITOS: 9 (15 horas/semana)
a. Viva a más de 3 millas del centro de la ciudad dado que la persona cambiaria a transportación publica.
 1) Entre 150 personas que fueron encuestadas como parte de un estudio de transporte urbano masivo, algunos vivían a más de 3 millas del centro de la ciudad (A), algunos de ellos van en sus propios carros
1) Entre 150 personas que fueron encuestadas como parte de un estudio de transporte urbano masivo, algunos vivían a más de 3 millas del centro de la ciudad (A), algunos de ellos van en sus propios carros
Dra. T. I. Grokhlina Institute of Mathematical Problems of Biology, Pushchino 14290, Russia
 1. Nombre del Proyecto, participantes y afiliaciones. Cálculos de estructura electrónica y de mecánica molecular para biomoléculas con ligandos en fase gaseosa y en solución Integrantes del Cuerpo Académico
1. Nombre del Proyecto, participantes y afiliaciones. Cálculos de estructura electrónica y de mecánica molecular para biomoléculas con ligandos en fase gaseosa y en solución Integrantes del Cuerpo Académico
Transferencia horizontal en eucariotas
 Transferencia horizontal en eucariotas Núria Rius 3 junio 2010 Tranferencia horizontal en eucariotas Qué es la transferencia horizontal y como se detecta? Casos en eucariotas Mitocondrias y Cloroplastos
Transferencia horizontal en eucariotas Núria Rius 3 junio 2010 Tranferencia horizontal en eucariotas Qué es la transferencia horizontal y como se detecta? Casos en eucariotas Mitocondrias y Cloroplastos
Planificaciones. 6665 - Control Digital. Docente responsable: ZANINI ANIBAL JOSE ANTONIO. 1 de 5
 Planificaciones 6665 - Control Digital Docente responsable: ZANINI ANIBAL JOSE ANTONIO 1 de 5 OBJETIVOS El alumno deberá finalizar la asignatura manejando fluidamente las diferentes técnicas de modelización
Planificaciones 6665 - Control Digital Docente responsable: ZANINI ANIBAL JOSE ANTONIO 1 de 5 OBJETIVOS El alumno deberá finalizar la asignatura manejando fluidamente las diferentes técnicas de modelización
Predicción de Estructura Secundaria y otras Características 1D
 Estructura de Proteínas Predicción de Estructura Secundaria y otras Características 1D Florencio Pazos (CNB-CSIC) Florencio Pazos Cabaleiro Computational Systems Biology Group (CNB-CSIC) pazos@cnb.csic.es
Estructura de Proteínas Predicción de Estructura Secundaria y otras Características 1D Florencio Pazos (CNB-CSIC) Florencio Pazos Cabaleiro Computational Systems Biology Group (CNB-CSIC) pazos@cnb.csic.es
Asignatura: Horas: Total (horas): Obligatoria Teóricas 3.0 Semana 5.0 Optativa X Prácticas 2.0 16 Semanas 80.0 de elección
 UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE INGENIERÍA PROGRAMA DE ESTUDIO Aprobado por el Consejo Técnico de la Facultad de Ingeniería en su sesión ordinaria del 15 de octubre de 2008 CONTROL
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE INGENIERÍA PROGRAMA DE ESTUDIO Aprobado por el Consejo Técnico de la Facultad de Ingeniería en su sesión ordinaria del 15 de octubre de 2008 CONTROL
Seminario Bioinformática: The ten most wanted solutions in protein bioinformatics. Problem 1
 Seminario Bioinformática: The ten most wanted solutions in protein bioinformatics. Problem 1 Gloria Inés Alvarez V. Departamento de Ciencias e Ingeniería de la Computación Pontificia Universidad Javeriana
Seminario Bioinformática: The ten most wanted solutions in protein bioinformatics. Problem 1 Gloria Inés Alvarez V. Departamento de Ciencias e Ingeniería de la Computación Pontificia Universidad Javeriana
Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA
 Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA Martin A. Lozano, Jaime Velasco-Medina Grupo de Bio-nanoelectrónica EIEE, Universidad del Valle, A.A. 25360, Cali, Colombia E-mail:
Diseño de un Procesador para el Alineamiento Global de Secuencias de DNA Martin A. Lozano, Jaime Velasco-Medina Grupo de Bio-nanoelectrónica EIEE, Universidad del Valle, A.A. 25360, Cali, Colombia E-mail:
Proteoma mitocondrial de Saccharomyces cerevisiae
 Proteoma mitocondrial de Saccharomyces cerevisiae PNAS, 23: 13207-13212 (2003) Mitocondria Orgánulos celulares presentes en casi todas las células eucariotas. La mitocondria desempeña un papel principal
Proteoma mitocondrial de Saccharomyces cerevisiae PNAS, 23: 13207-13212 (2003) Mitocondria Orgánulos celulares presentes en casi todas las células eucariotas. La mitocondria desempeña un papel principal
Benemérita Universidad Autónoma de Puebla Doctorado en Ciencias Químicas BIOINFORMATICA
 Benemérita Universidad Autónoma de Puebla Doctorado en Ciencias Químicas BIOINFORMATICA Programa semestral: 96 h. Objetivos del curso: Que los estudiantes adquieran las bases teóricos y aplicaciones en
Benemérita Universidad Autónoma de Puebla Doctorado en Ciencias Químicas BIOINFORMATICA Programa semestral: 96 h. Objetivos del curso: Que los estudiantes adquieran las bases teóricos y aplicaciones en
CURRICULUM VITAE. Diego Moitre
 CURRICULUM VITAE Diego Moitre A. ANTECEDENTES PERSONALES Nombre : Diego Carlos Alberto Moitre Fecha de Nacimiento : 13 de Noviembre de 1953 Nacionalidad : Argentina Domicilio Profesional: Universidad Nacional
CURRICULUM VITAE Diego Moitre A. ANTECEDENTES PERSONALES Nombre : Diego Carlos Alberto Moitre Fecha de Nacimiento : 13 de Noviembre de 1953 Nacionalidad : Argentina Domicilio Profesional: Universidad Nacional
Análisis bioinformático de la proteína SAG29 de Arabidopsis thaliana involucrada en la respuesta al estrés salino.
 / Ágora Rev. Cient.2015; 02(02):158-163 Análisis bioinformático de la proteína SAG29 de Arabidopsis thaliana involucrada en la respuesta al estrés salino. Bioinformatics analysis of SAG29 protein from
/ Ágora Rev. Cient.2015; 02(02):158-163 Análisis bioinformático de la proteína SAG29 de Arabidopsis thaliana involucrada en la respuesta al estrés salino. Bioinformatics analysis of SAG29 protein from
ANX-PR/CL/ GUÍA DE APRENDIZAJE. ASIGNATURA Estructura e ingenieria de proteinas. CURSO ACADÉMICO - SEMESTRE Primer semestre
 ANX-PR/CL/001-01 GUÍA DE APRENDIZAJE ASIGNATURA Estructura e ingenieria de proteinas CURSO ACADÉMICO - SEMESTRE 2016-17 - Primer semestre GA_02BT_25004411_1S_2016-17 Datos Descriptivos Nombre de la Asignatura
ANX-PR/CL/001-01 GUÍA DE APRENDIZAJE ASIGNATURA Estructura e ingenieria de proteinas CURSO ACADÉMICO - SEMESTRE 2016-17 - Primer semestre GA_02BT_25004411_1S_2016-17 Datos Descriptivos Nombre de la Asignatura
Sistema de Reconocimiento de Iris Implementado en un Procesador Digital de Señales
 Sistema de Reconocimiento de Iris Implementado en un Procesador Digital de Señales Arles Felipe García Maya Juan Camilo Moreno Ruiz Director MSc. Edwin Andrés Quintero Salazar Ing. Electrónica Orden del
Sistema de Reconocimiento de Iris Implementado en un Procesador Digital de Señales Arles Felipe García Maya Juan Camilo Moreno Ruiz Director MSc. Edwin Andrés Quintero Salazar Ing. Electrónica Orden del
Mas de 20 años especializado en la Planificación y Optimización Avanzada de Inventario.
 Mas de 20 años especializado en la Planificación y Optimización Avanzada de Inventario. Que es Valogix? Herramienta de planificación y pronóstico de última generación permite pronosticar, planificar, y
Mas de 20 años especializado en la Planificación y Optimización Avanzada de Inventario. Que es Valogix? Herramienta de planificación y pronóstico de última generación permite pronosticar, planificar, y
Análisis genético usando datos de secuenciación masiva. Michael Hackenberg
 Análisis genético usando datos de secuenciación masiva Michael Hackenberg hackenberg@ugr.es Motivación Existen diferentes situaciones en las que interesa detectar el genotipo de un individuo Identificación
Análisis genético usando datos de secuenciación masiva Michael Hackenberg hackenberg@ugr.es Motivación Existen diferentes situaciones en las que interesa detectar el genotipo de un individuo Identificación
AMINOÁCIDOS SE PUEDEN UNIR POR ENLACES PEPTÍDICOS
 Tema 4. Proteínas: funciones biológicas y estructura primaria. Enlace peptídico. Péptidos y proteínas. Diversidad de funciones biológicas. Niveles de organización estructural de las proteínas. Separación
Tema 4. Proteínas: funciones biológicas y estructura primaria. Enlace peptídico. Péptidos y proteínas. Diversidad de funciones biológicas. Niveles de organización estructural de las proteínas. Separación
Comisión Interministerial de Ciencia y Tecnología Curriculum vitae
 Comisión Interministerial de Ciencia y Tecnología Curriculum vitae Nombre: ELENA ROIBÁS MILLÁN Fecha: 13/02/2015 Apellidos: ROIBÁS MILLÁN Nombre: ELENA Situación profesional actual Organismo: Escuela o
Comisión Interministerial de Ciencia y Tecnología Curriculum vitae Nombre: ELENA ROIBÁS MILLÁN Fecha: 13/02/2015 Apellidos: ROIBÁS MILLÁN Nombre: ELENA Situación profesional actual Organismo: Escuela o
ASIGNATURA: ADVANCED BIOINFORMATICS
 Página 1 de 5 CARACTERÍSTICAS GENERALES * Tipo: Formación básica, Obligatoria, Optativa Trabajo de fin de grado, Prácticas externas Duración: Cuatrimestral Semestre / s: 1 Número de créditos ECTS: 4 Idioma
Página 1 de 5 CARACTERÍSTICAS GENERALES * Tipo: Formación básica, Obligatoria, Optativa Trabajo de fin de grado, Prácticas externas Duración: Cuatrimestral Semestre / s: 1 Número de créditos ECTS: 4 Idioma
Grupo de Genómica Computacional & Bioinformática Universidad de Granada
 Grupo de Genómica Computacional & Bioinformática Universidad de Granada INTRODUCCIÓN Metilación del ADN Proceso bioquímico de la metilación de citosinas ARN polimerasa Splicing alternativo Unión de factores
Grupo de Genómica Computacional & Bioinformática Universidad de Granada INTRODUCCIÓN Metilación del ADN Proceso bioquímico de la metilación de citosinas ARN polimerasa Splicing alternativo Unión de factores
Redes de regulación génica. David Ochoa García
 Redes de regulación génica David Ochoa García 1RUN 1RUN 1LBG 1LBG http://phet.colorado.edu/sims/gene-network/genemachine-lac-operon_en.jnlp Redes de regulación génica Represor Activador RegulonDB
Redes de regulación génica David Ochoa García 1RUN 1RUN 1LBG 1LBG http://phet.colorado.edu/sims/gene-network/genemachine-lac-operon_en.jnlp Redes de regulación génica Represor Activador RegulonDB
GANETEC SOLUTIONS HPC Farmacéuticas
 GANETEC SOLUTIONS HPC Farmacéuticas La integración de tecnologías HPC en el sector Farmacéutico y de la Bioinformática ha permitido grandes avances en diversos campos. NUESTRA VISIÓN Estas nuevas posibilidades
GANETEC SOLUTIONS HPC Farmacéuticas La integración de tecnologías HPC en el sector Farmacéutico y de la Bioinformática ha permitido grandes avances en diversos campos. NUESTRA VISIÓN Estas nuevas posibilidades
Estructura y propiedades de las proteínas
 Estructura y propiedades de las proteínas Matilde Julián Seguí Introducción Las proteínas son las macromoléculas biológicas más importantes. Hay gran variedad de proteínas y cumplen gran variedad de funciones
Estructura y propiedades de las proteínas Matilde Julián Seguí Introducción Las proteínas son las macromoléculas biológicas más importantes. Hay gran variedad de proteínas y cumplen gran variedad de funciones
ANX-PR/CL/ GUÍA DE APRENDIZAJE. ASIGNATURA Estructura e ingenieria de proteinas. CURSO ACADÉMICO - SEMESTRE Primer semestre
 ANX-PR/CL/001-02 GUÍA DE APRENDIZAJE ASIGNATURA Estructura e ingenieria de proteinas CURSO ACADÉMICO - SEMESTRE 2015-16 - Primer semestre GA_02BT_25004411_1S_2015-16 Datos Descriptivos Nombre de la Asignatura
ANX-PR/CL/001-02 GUÍA DE APRENDIZAJE ASIGNATURA Estructura e ingenieria de proteinas CURSO ACADÉMICO - SEMESTRE 2015-16 - Primer semestre GA_02BT_25004411_1S_2015-16 Datos Descriptivos Nombre de la Asignatura
Revista Colombiana de Química ISSN: 0120-2804 orodriguez@unal.edu.co Universidad Nacional de Colombia Colombia
 Revista Colombiana de Química ISSN: 0120-2804 orodriguez@unal.edu.co Universidad Nacional de Colombia Colombia Vivas-Reyes, Ricardo; Ahumedo, Maicol; Cabezas, José MODELAMIENTO POR HOMOLOGÍA DE LA ESTRUCTURA
Revista Colombiana de Química ISSN: 0120-2804 orodriguez@unal.edu.co Universidad Nacional de Colombia Colombia Vivas-Reyes, Ricardo; Ahumedo, Maicol; Cabezas, José MODELAMIENTO POR HOMOLOGÍA DE LA ESTRUCTURA
INSTITUTO UNIVERSITARIO DE SISTEMAS INTELIGENTES Y APLICACIONES NUMÉRICAS EN INGENIERÍA TRABAJO FINAL DE MÁSTER:
 INSTITUTO UNIVERSITARIO DE SISTEMAS INTELIGENTES Y APLICACIONES NUMÉRICAS EN INGENIERÍA TRABAJO FINAL DE MÁSTER: Sistema Biométrico de Detección Facial sobre Alumno: Marcos del Pozo Baños Tutor: Dr. Modesto
INSTITUTO UNIVERSITARIO DE SISTEMAS INTELIGENTES Y APLICACIONES NUMÉRICAS EN INGENIERÍA TRABAJO FINAL DE MÁSTER: Sistema Biométrico de Detección Facial sobre Alumno: Marcos del Pozo Baños Tutor: Dr. Modesto
