EXTRACCIÓN DE DNA Y AMPLIFICACIÓN DE SECUENCIAS DEL ITS DEL DNAr DE Ganoderma (FUNGI, BASIDIOMYCETES) PARA SU USO EN EL ANÁLISIS FILOGENÉTICO.
|
|
|
- Enrique Luis Miguel Miranda Correa
- hace 7 años
- Vistas:
Transcripción
1 2005 Avances en la Investigación Científica en el CUCBA 450 ISBN: EXTRACCIÓN DE DNA Y AMPLIFICACIÓN DE SECUENCIAS DEL ITS DEL DNAr DE Ganoderma (FUNGI, BASIDIOMYCETES) PARA SU USO EN EL ANÁLISIS FILOGENÉTICO. Laura Izascum Pérez Valencia, Anne Santerre, Alma Rosa Villalobos Arámbula. Laboratorio de Genética, Departamento de Biología Celular y Molecular. Adrián Galván Corona, Mabel Gisela Torres-Torres, Aarón Rodríguez Contreras, Laura Guzmán Dávalos. Departamento de Botánica y Zoología. Introducción El estudio de la biodiversidad es de gran importancia para entender la estructura y función de la vida en nuestro planeta; lamentablemente en las últimas décadas la diversidad biológica se ha visto afectada profundamente, principalmente a través de la pérdida acelerada de las especies, que se traduce en el deterioro de los ciclos biológicos. En cuanto a hongos se refiere la pérdida de diversidad también es muy grande, pero no evidente, debido al bajo número de especies descritas en relación con las que se calcula que existen (Hawksworth, 1991; Rossman, 1994). Esta pérdida tan grande, esta estimulando a muchos científicos de diferentes áreas a investigar más sobre las especies. Ciencias como la sistemática y la genética aportan técnicas que son útiles para la obtención de los datos necesarios para el conocimiento de la biodiversidad. El género Ganoderma P. Karst. pertenece a la familia Ganodermataceae (Basidiomycetes, Fungi), que esta constituida por un pequeño numero de géneros que presentan distribución cosmopolita y que se desarrollan sobre el suelo o madera muerta (Ryvarden y Johansen, 1980). Ryvarden y Moncalvo (1997) reconocen para la familia un total de 220 especies. Ganoderma se caracteriza por tener basidiomas bi o perianuales, pileados, sésiles o estipitados central o lateralmente, con la superficie del píleo lisa, opaca o brillante con una cutícula distintiva de color café claro a púrpura oscuro. Presenta poros pequeños a medianos, los tubos generalmente son estratificados, ocráceos a veces blanquecinos, y el contexto es blanco a color café. Su sistema hifal es di o trimítico, generalmente no presenta cistidios, y sus basidiosporas son ovoides a elipsoides, truncadas, de grandes a muy grandes (7-25 m de longitud), amarillentas o de color café, con doble pared muy distintiva, la externa hialina con un exosporio cubierto de ornamentaciones y un endosporio de color café (Furtado, 1965; Bazzalo y Wright, 1982; Corner, 1983; Nuñez y Ryvarden, 2000). Son hongos saprobios o parásitos facultativos de diversas plantas, incluyendo cultivos perennes, como el caucho, té, palmas aceiteras, café y árboles de madera dura, en donde causan pudrición blanca. Tienen una distribución amplia, en climas templados y tropicales (Bazzalo y Wright, 1982; Miller et al., 1995). De acuerdo con la estructura de la superficie
2 2005 Avances en la Investigación Científica en el CUCBA 451 del píleo, Steyaert (1980) estableció cuatro subgéneros (Ganoderma, Elfvingia, Plecoderma, Anamixoderma) y dividió el subgénero Ganoderma en dos secciones (Ganoderma y Characoderma). Otros autores, como Corner (1983) y Ryvarden (1991), han referido a las especies laqueadas como del complejo G. lucidum y las especies opacas como del complejo G. applanatum; los cuales han sido denominados como los subgéneros Ganoderma y Elfvingia, respectivamente. Aunque el género ha sido estudiado por diversos autores (e.g. Furtado, 1965; Steyaert, 1980; Bazzalo y Wright, 1982; Gilbertson y Ryvarden, 1986; Adaskaveg y Gilbertson, 1988; Ryvarden, 1991, 2000), incluso con datos moleculares y de otro tipo (Adaskaveg y Gilbertson, 1986; Moncalvo et al.; 1995; Gottlieb et al., 1998, 2000; Moncalvo, 2000; Smith y Sivasithamparam, 2000; Hong y Jung, 2004), su taxonomía sigue siendo controversial. Hasta hace algunos años, en los estudios en sistemática se utilizaban casi siempre caracteres morfológicos. Sin embargo, existen caracteres que pueden estar sometidos a la subjetividad de quien los analiza o bien no todos los caracteres relevantes son apreciables a simple vista, o no tenemos la capacidad para definir inequívocamente las homologías. En la actualidad las herramientas que se utilizan en la sistemática se han extendido; ahora no sólo se toman en cuenta los caracteres morfológicos, sino también los caracteres moleculares. La región espaciadora interna transcrita (ITS) del DNA ribosomal (DNAr) nuclear, contiene dos regiones variables no codificadoras, que se encuentran dentro de las repeticiones altamente conservadas del DNAr a, entre la subunidad pequeña, la región 5.8S y los genes que codifican para RNAr de la subunidad grande (Gardes y Bruns, 1993). La funcionalidad del ITS esta relacionada con la división específica dentro del transcripto primario ITS-1 e ITS-2 durante la maduración de las subunidades pequeñas (SSU), 5.8S, y la subunidad ribosomal grande (LSU). Entre las características convenientes para el análisis de secuencias ITS en hongos, se encuentran: la región ITS completa de 600 a 800 pb puede amplificarse fácilmente con iniciadores universales; la naturaleza multicopia de las repeticiones del DNAr hace de esta región una secuencia fácil de amplificar, aun cuando se utilicen muestras de DNA pequeñas o muy diluidas, o altamente degradadas (DNA obtenido de material viejo y especímenes de
3 2005 Avances en la Investigación Científica en el CUCBA 452 herbarios). Finalmente, diversos estudios han demostrado su utilidad en el análisis filogenético (Gardes y Bruns, 1993; Álvarez y Wendel, 2003). Este trabajo es parte de un proyecto más amplio, que tiene como objetivo el de establecer las relaciones filogenéticas (historia evolutiva y parentesco entre las especies), en la mayoría de los casos a nivel infragenérico de un grupo selecto de macromicetos, empleando caracteres morfológicos, moleculares y químicos. En ese proyecto para el género Ganoderma en particular, se pretende: comprobar su monofilia, estudiar con caracteres morfológicos, moleculares y químicos las relaciones filogenéticas de las especies, y definir si la presencia o ausencia de laca en el píleo de los basidiomas de Ganoderma es la sinapomorfia a nivel infragenérico. Así, en este trabajo se presentan los resultados obtenidos de una de las fases iniciales del proyecto, referente a la obtención de datos moleculares. En particular se presentan los resultados sobre la extracción de DNA y amplificación de ITS de especímenes de Ganoderma. Material y Métodos Material Hasta ahora se han seleccionado 93 especímenes de 19 especies del género Ganoderma de acuerdo a la clasificación tradicional del grupo. Los especímenes se recolectaron entre 1904 y 2004; la mayoría de las muestras se obtuvieron a partir de ejemplares de herbario, y muy pocas de materiales recién recolectados. De los 93 especímenes se realizaron 170 extracciones de distintos tejidos del basidioma: 73 corresponden a contexto, mientras que 84 corresponden a tubos. En trece muestras no se indica el tipo de tejido. Extracción de DNA Experimentos de optimización para el género Ganoderma Para la extracción de DNA a partir de muestras de herbario o frescas se ensayaron dos protocolos, uno con SDS y proteinasa K para la eliminación de proteínas (Aljanabi et al. 1997), ya utilizado en el Laboratorio de Genética para la extracción de DNA de insectos y plantas (Palomera, 2005); y otro con CTAB y cloroformo-isoamílico para la eliminación de proteínas (Doyle y Doyle, 1987), optimizado para extraer DNA de diversos tejidos de pinos (Palomera, 2002). En ambos protocolos se incluyó en el buffer de lisis PVP al 1%, además se adaptaron los protocolos a la cantidad de tejido de las muestras de herbario de Ganoderma. Se llevaron a cabo dos experimentos, cada uno con ocho muestras ( mg) que se trabajaron con los dos métodos de extracción de DNA ). En el primer experimento se utilizaron 8 ml de buffer de extracción en el protocolo de Doyle & Doyle y 4 ml de buffer de extracción para el protocolo con Proteinasa K. En el segundo experimento se utilizaron 4 ml de buffer de extracción para los dos protocolos. La calidad del DNA extraido se determinó por electroforesis en geles de agarosa al 0.8% (Sambrook et al., 1989).
4 2005 Avances en la Investigación Científica en el CUCBA 453 Extracción de DNA Para la extracción de DNA con el protocolo basado en proteinasa K el tejido se maceró con nitrógeno líquido y se transfirió a un tubo de 15 ml. Se agregaron 4 ml de buffer salino (NaCl 0.4M, Tris-HCl 10mM, EDTA 2mM) con PVP al 1%. Se mezcló suavemente por inversión y se añadieron 400 µl de SDS al 20% y 20µl de Proteinasa K (10 mg/ml). Se mezcló por inversión y se incubó por una hora a 65 C. Se añadieron 3 ml de NaCl 6M y se mezcló lentamente por 30 seg. Se centrifugó a 4000 rpm por 45 min a 24 C. Se transfirió el sobrenadante a un tubo nuevo y se agregó la misma cantidad de isopropanol frío. Se mezcló suavemente por inversión, se incubó toda la noche a -20 C, se centrifugó a 4 C por 30 min a 4000 rpm y se transfirió el sobrenadante a un tubo limpio. Se centrifugó nuevamente a 4000 rpm por 20 min y se agregaron 2.5 ml de etanol al 70% frío. Se mezcló por inversión cada 5 min al menos 3 veces. Se centrifugó a 4000 rpm por 10 min a 4 C y se decantó el etanol. La pastilla de DNA se transfirió a un tubo nuevo de 1.5 ml con 1 ml de etanol al 70% frío, repitiendo el lavado dos veces más. Se centrifugó a 4000 rpm por 10 min a 4 C y se decantó el etanol. La pastilla se diluyó en 100µl-500µl de TE (Tris 10mM ph 7.4, EDTA 0.2mM), disolviendo a temperatura ambiente toda la noche. Se almacenó a - 20 C hasta su uso. Con el método de extracción de Doyle & Doyle el tejido se maceró con nitrógeno líquido y se transfirió la muestra a un tubo de 15 ml con 4 ml de buffer CTAB 2X (CTAB 2%, Tris 100mM, EDTA 20 mm, NaCl 1.4M) con PVP al 1 % previamente calentado a 65 C. Se incubo por 60 min. a 65 C. Una vez fría la muestra se añadió 4 ml de cloroformo (alcohol isoamílico 24:1). Se centrifugo a 4000 rpm. por 45 min. Se transfirió el sobrenadante a un nuevo tubo. Se agrego el mismo volumen de isopropanol frío. Se incubó toda la noche a - 20 C. Se centrifugo a 4000 rpm. por 20 min. Se decanto el sobrenadante. Se añadió 4ml de buffer de lavado(etanol absoluto, acetato de amonio 7.5M). Se centrifugo a 2000 rpm. por 2 min. Se decantó y seco. Se añadió 800µl de buffer TE (Tris 1M ph 7.4, EDTA 0.5 M) y 280µl de agua destilada para disolver la pastilla. Se añadió 520µl de acetato de amonio 7.5M. Se agrego 3.5ml. de etanol absoluto frío. Se incubó por una hora a -20 C. Se centrifugó a 4000 rpm por 10 min. Se decanto y seco la pastilla. Se añadió 3 ml. de etanol al 70 %. Se centrifugó a 2000rpm por 5. Se decantó. Se añadió 1 ml de etanol al 70% frio. Se transfirió la pastilla de DNA a un tubo eppendorf de 1.5 ml. Se repitió el lavado con 1ml de etanol al 70% frío. Se centrifugó a 2000 rpm por 5 min. Se decanto. Se disolvió la pastilla de DNA en µl de TE. Se dejó a temperatura ambiente toda la noche. Amplificación de ITS Para la amplificación por PCR de la secuencia completa del ITS se utilizó el par de cebadores (ITS1F e ITS4) de acuerdo con las condiciones descritas en Guzmán-Dávalos et al. (2003) con algunas modificaciones. El volumen total de la reacción para la PCR fue de 25 µl por muestra. Con 23 µl de mezcla PCR (buffer PCR 10X, MgCl 2 50 mm, dntps 5 mm, BSA 1 mg/ml), 1 µl de mezcla primer-taq (primer 5 y 3 10 µm y 0.2 µl de TaqPol 5U/µl) y 1 µl de DNA (que se diluye 1:1 en caso de presentarse una alta concentración de pigmentos). Las muestras se amplificaronn con las siguientes condiciones: desnaturalización inicial a 95 C por 3 min,
5 2005 Avances en la Investigación Científica en el CUCBA ciclos de desnaturalización a 95 C por 1 min, alineación 50 C por 45 seg y elongación a 72 C por 2 min; 14 ciclos de desnaturalización a 95 C por 1 min, alineación 50 C por 45 seg, (aumentar 5 seg por ciclo) y elongación a 72 C por 2 min. Elongación prolongada final a 72 C por 10 min. El fragmento amplificado se separó en geles de agarosa (2 %) con amortiguador TBE 1X, las muestras se corrieron en promedio a 80 voltios por dos horas y los geles se tiñeron con bromuro de etidio para visualizar la banda de aproximadamente 500 pb en un transiluminador de luz UV (UVP-TS20E). Se tomaron fotografías de los geles con una cámara polaroid y con el sistema de fotodocumentación EDAS (Kodak Digital Science 1D v.3.0.2), posteriormente se procedió a su análisis. Resultados y Discusión Optimización de los protocolos de Extracción de DNA Con base en los resultados de la electroforesis del primer experimento para las muestras de Ganoderma con los dos protocolos de extracción, se encontró que de los dos métodos probados, el más adecuado para la extracción de DNA de especímenes de Ganoderma es el de proteinasa K, ya que el producto final contiene menos pigmentos que aquellas muestras procesadas con el método de Doyle & Doyle, además de que el protocolo es más sencillo. Sin embargo, el segundo experimento, nos revela que en el caso de Ganoderma no siempre se eliminan los pigmentos, aún con el método de proteinasa K. Así, la cantidad de pigmentos en la muestra dependerá del tejido utilizado. Es importante señalar que con estos dos experimentos se logró establecer tanto la cantidad de reactivos óptima, como la cantidad de muestra en mg a utilizar. Extracción de DNA En cuanto a los resultados obtenidos de las 130 extracciones de DNA de las muestras de Ganodermas, 115 se efectuaron con el protocolo con Proteinasa K, mientras que sólo 15 fueron procesadas con el método de Doyle & Doyle. El análisis del DNA obtenido en el gel de agarosa al 2% mostró que la mayoría de las muestras (67%) obtenidas con el método con proteinasa K, no presentan ninguna banda de DNA de alto peso molecular o degradado, el 9% de las muestras procesadas mostraron un barrido que indica DNA altamente degradado, y solo el 24% mostró una banda de DNA de alto peso molecular. Como puede observarse en el Cuadro I, el análisis de los resultados obtenidos en la extracción de DNA a partir de los dos tipos de tejidos: contexto y tubo, no muestran diferencias. Con respecto a las muestras obtenidas con el protocolo Doyle & Doyle, el análisis mostró que en el 53% de las muestras no se observó ninguna banda en el gel, mientras que en el 47% de las muestras se registró una banda de DNA de alto peso molecular. La mayoría de los especimenes de Ganodermas, fueron colectados hace siete a cincuenta años atrás, lo que explica la ausencia de banda en el gel. De cualquier manera, sabemos que ambos protocolos de extracción de DNA son adecuados para muestras frescas o colectados en los últimos cinco años ya que en muestras con estas características se observan bandas de DNA de alto peso molecular en el gel de agarosa. Es
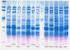
6 2005 Avances en la Investigación Científica en el CUCBA 455 importante señalar que en el gel también se observaron pigmentos co-extraídos con el DNA, como un barrido intenso en el fondo del gel, lo que impide la cuantificación del DNA por espectrofotometría. Cuadro I. Resultados del análisis del DNA obtenido de muestras de Ganodermas. Proteinasa K Doyle & Doyle Análisis de la Electroforesis Tubo Contexto Tubo Contexto Sin banda en el gel Banda de DNA de alto peso molecular Banda de DNA altamente degradado Amplificación con PCR de secuencias ITS Los productos de PCR obtenidos con el par de cebadores ITS1-F e ITS4 se observaron en la mayoría de los casos como una banda en los geles de agarosa teñidos con bromuro de etidio. El tamaño de los fragmentos de PCR, para el ITS completo es las muestras de Ganoderma fue de 700 pb. Algunas muestras de DNA también amplificaron una banda inespecíficas de 500 pb (Fig. 1A). Hasta ahora se cuenta con los resultados de 253 reacciones de PCR, en donde sólo el 30% mostró amplificación en la banda de interés. Finalmente es importe señalar que una vez que se prueba que las muestras de DNA no amplifican para el ITS completo, se probará una reamplificación con 1µl del producto de PCR y aumentando en el programa de alineación 1 C. En donde como se puede observar en la Fig. 1B, ya se ha logrado la amplificación de algunas muestras de Ganoderma. (A) (B) Figura 1. Amplificación por PCR de la secuencia ITS completa (A). Reamplificación de la región ITS2 (B).
7 2005 Avances en la Investigación Científica en el CUCBA 456 Agradecimientos Proyecto CONACYT con LGD como investigador responsable. Proyecto U de G P3E (2005) con ARVA como investigador participante. Beca CONACYT para Laura Izascum Pérez Valencia. PAY-SNI para Adrián Galván Corona. Bibliografía Adaskaveg, J.E. y R.L. Gilbertson Cultural studies and genetics of sexuality of Ganoderma lucidum and Ganoderma tsugae in relation with taxonomy of the G. lucidum complex. Mycologia 80: Adaskaveg, J.E. y R.L. Gilbertson Basidiospores, pylocistidia and other basidiocarp characters in several species of Ganoderma lucidum complex. Mycologia 80: Aljanabi, S.M. y I. Martinez Universal and rapid salt-extraction of high quality genomic DNA for PCR- based techniques. Nucleic Acid research 25: Alvarez, I. y J.F. Wendel Ribosomal ITS sequences and plant phylogenetic inference. Molecular phylogenetics and evolution. 29: Bailey, C.D., T.G Carr, S.A. Harris y C.E. Hughes Molecular Phylogenetics and evolution 29: Básalo, M. E. y J.E. Weight Survey of the argentine species of the Ganoderma lucidum complex. Mycotaxon 16: Corner, E. J. H Ad Polyporaceas I, Amauroderma and Ganoderma. Beih. Nova Hedw. 75, Cramer, Vaduz. Doyle, J.J., J.L. Doyle. (1987) A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytocheml Bull, 19, Furtado, J. S Relation of microstructure of the taxonomy of the Ganodermoideae (Polyporaceae) with special reference to the structure of the cover of the pilear surface. Miciologia Gilbertson, R. L. y L. Ryvarden North American Polypores. Vol. 1 Fungiflora, Oslo. Hawksworth, D.L The fungal dimension of biodiversity: magnitude, significance and conservation. Mycological Research 95: Hawksworth, D.L. P. M. Kirk, B.C. Sutton & D. N. Pegler Ainsworth & Bisby s dictionary of the fungi. CAB. International, Wallingford. 8 ed.
8 2005 Avances en la Investigación Científica en el CUCBA 457 Hong, S.G. y H.S. Jung, Phylogenetic analysis of Ganoderma based on nearly complete mitochondrial small-subunit ribosomal DNA sequences. Mycology 96: Miller, R.N., G. M. Holderness, P.D. Bridge, R.R.M. Paterson, M. Sariah, M.Z. Hussin y E. J. Hilsley A multidisciplinary approach to the characterization of Ganoderma in oil-palm cropping systems. In : Buchanan, P.K., R. S. Hseu y J.M. Moncalvo (eds). Proceedings of contributed symposium 59A, B5th International Mycological Congres, pp Taiper. Moncalvo, J.M., R. Vilgalys, S.A. Redhead, et al. (2002). One hundred and seventeen clades of euagarics. Molecular Phylogenetics and Evolution, 23, Palomera, V Comparación de métodos de extracción de DNA de diferentes tejidos de tres especies de pinos blancos (Pinus spp.). Tesis de licenciatura. Universidad de Guadalajara, México. Palomera, V Interacción entre Spiroplasma kunkelii (Mycoplasmastales, Spiroplasmataceae), su huésped Dalbulus maidis (Hemiptera, Cicadellidae) y enemigos naturales. Tesis de maestría. Universidad de Guadalajara, México. Ryvarden, L Genera of Polypores Nomenclature and taxonomy synopsis Fungorum 5. Fungiflora. Oslo. Ryvarden, L Studies in neotropical polypores 2. A preliminary key to neotropical species of Ganoderma with laccate pileus. Mycology 92: Rossman, A.Y A strategy for an all-taxa inventory of fungal biodiversity. In: Peng. C. I. & C.H. Chou (eds.). Biodiversity and terrestrial ecosystems Academia Sinica Monographs, series 14, Taiper. Steyaert, R. L Study of some Ganoderma species. Bull. Jard. Bot. Nat. Belg. 50: Zervakis, G. I., J. Moncalvo y R. Vilgalys Molecular phylogeny, biogeography and speciation of the mushroom species Pleurotus cystidiosus and allied taxa. Mycrobiology. 150:
Investigación, Biodiversidad y Desarrollo 2012; 31 (1): 61-7
 Investigación, Biodiversidad y Desarrollo 2012; 31 (1): 61-7 Extracción de ADN y amplificación de secuencias del ITS del ADNr de Ganoderma (Fungi, Basidiomycetes) para su uso en el análisis filogenético
Investigación, Biodiversidad y Desarrollo 2012; 31 (1): 61-7 Extracción de ADN y amplificación de secuencias del ITS del ADNr de Ganoderma (Fungi, Basidiomycetes) para su uso en el análisis filogenético
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987)
 EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
MATERIALES Y MÉTODOS. Muestras
 MATERIALES Y MÉTODOS Muestras Las muestras de sangre con el bacilo M. tuberculosis se obtuvieron de 2 pacientes con tuberculosis diagnosticada por clínica, laboratorio y con cultivo positivo como controles
MATERIALES Y MÉTODOS Muestras Las muestras de sangre con el bacilo M. tuberculosis se obtuvieron de 2 pacientes con tuberculosis diagnosticada por clínica, laboratorio y con cultivo positivo como controles
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón
 RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
 EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS. Es un ácido capaz de formar sales con iones cargados
 Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
1. OBTENCIÓN DE ADN DE BACTERIAS PATÓGENAS
 1. OBTENCIÓN DE ADN DE BACTERIAS PATÓGENAS Existen diferentes métodos para la obtención de ADN bacteriano que varían en duración y complejidad del procedimiento en función de la calidad del ADN que queramos
1. OBTENCIÓN DE ADN DE BACTERIAS PATÓGENAS Existen diferentes métodos para la obtención de ADN bacteriano que varían en duración y complejidad del procedimiento en función de la calidad del ADN que queramos
4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar
 4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar - Agarosa de grado molecular - Agarosa de bajo punto de fusión
4. TIPADO GENÉTICO DE BACTERIAS PATÓGENAS MEDIANTE ELECTROFORESIS EN CAMPOS PULSADOS (PFGE) Materiales - Bacterias crecidas en placas de agar - Agarosa de grado molecular - Agarosa de bajo punto de fusión
Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497.
 Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497. Extracción de DNA genómico El DNA de A. brasilense CBG 497 se extrajo siguiendo el método descrito por la casa comercial distribuidora
Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497. Extracción de DNA genómico El DNA de A. brasilense CBG 497 se extrajo siguiendo el método descrito por la casa comercial distribuidora
CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL
 LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
Obtención de ADN de cactáceas: comparación de dos métodos de extracción en Ferocactus histrix
 Obtención de ADN de cactáceas: comparación de dos métodos de extracción en Ferocactus histrix Brenda Díaz Cárdenas 1, Liliana Gómez Flores Ramos 1, Verónica Carolina Rosas-Espinoza 2, Laura Izascum Pérez
Obtención de ADN de cactáceas: comparación de dos métodos de extracción en Ferocactus histrix Brenda Díaz Cárdenas 1, Liliana Gómez Flores Ramos 1, Verónica Carolina Rosas-Espinoza 2, Laura Izascum Pérez
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
RESULTADOS Y DISCUSIÓN
 RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
EXTRACCIÓN Y PURIFICACIÓN DE ÁCIDOS NUCLEICOS
 EXTRACCIÓN Y PURIFICACIÓN DE ÁCIDOS NUCLEICOS Consideraciones iniciales - Condiciones de limpieza y esterilidad - Peligrosidad de algunos reactivos usados en la purificación Fases - Rotura-homogeneización:
EXTRACCIÓN Y PURIFICACIÓN DE ÁCIDOS NUCLEICOS Consideraciones iniciales - Condiciones de limpieza y esterilidad - Peligrosidad de algunos reactivos usados en la purificación Fases - Rotura-homogeneización:
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80
 AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
3. MATERIALES Y MÉTODOS
 3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006
 IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
PCR Múltiplex. Listeria monocytogenes
 PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR
 KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
Ensayos de restricción
 Ensayos de restricción Vector con inserto= Vector recombinante 6500pb Objetivos de ensayos de restricción de plásmidos Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa.
Ensayos de restricción Vector con inserto= Vector recombinante 6500pb Objetivos de ensayos de restricción de plásmidos Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa.
Palabras clave: Encalyptaceae, Musgos, Península Ibérica, Amplificación ADN, ITS
 ISSN: 0214-4565 Notas preliminares sobre las técnicas de amplificación y variación de la región ITS (Internal Transcribed Spacer) en el género Encalypta (Encalyptaceae, Bryophyta) María Paz MARTÍN* & Isabel
ISSN: 0214-4565 Notas preliminares sobre las técnicas de amplificación y variación de la región ITS (Internal Transcribed Spacer) en el género Encalypta (Encalyptaceae, Bryophyta) María Paz MARTÍN* & Isabel
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO
 SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
Reacción en cadena de la polimerasa (PCR)
 Curso: Biología Molecular y Genómica Enero 2014. Conceptos elementales sobre la técnica de reacción en cadena de la polimerasa (PCR) Dr. Juan Venegas Hermosilla Programa de Biología Celular y Molecular
Curso: Biología Molecular y Genómica Enero 2014. Conceptos elementales sobre la técnica de reacción en cadena de la polimerasa (PCR) Dr. Juan Venegas Hermosilla Programa de Biología Celular y Molecular
Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato
 Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
DANAGENE BLOOD DNA KIT
 Ref. 0601 100 ml Ref. 0602 200 ml DANAGENE BLOOD DNA KIT 1.INTRODUCCION DANAGENE BLOOD DNA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de sangre total o médula ósea.
Ref. 0601 100 ml Ref. 0602 200 ml DANAGENE BLOOD DNA KIT 1.INTRODUCCION DANAGENE BLOOD DNA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de sangre total o médula ósea.
Conceptos y técnicas en ecología fluvial
 Conceptos y técnicas en ecología fluvial Edición a cargo de: ARTURO ELOSEGI Profesor titular de Ecología en la Universidad del País Vasco SERGI SABATER Catedrático de Ecología en la Universidad de Girona
Conceptos y técnicas en ecología fluvial Edición a cargo de: ARTURO ELOSEGI Profesor titular de Ecología en la Universidad del País Vasco SERGI SABATER Catedrático de Ecología en la Universidad de Girona
TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR
 TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR Microbiología a General Área Micología Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las
TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR Microbiología a General Área Micología Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las
5. MATERIALES Y MÉTODOS
 5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
Instrucciones de uso. BAG Cycler Check. Kit de para la validación de la uniformidad de la temperatura en los termocicladores
 Instrucciones de uso BAG Cycler Check Kit de para la validación de la uniformidad de la temperatura en los termocicladores listo para usar, prealicuotado REF 7104 (10 tests) REF 71044 (4 tests) Contenidos
Instrucciones de uso BAG Cycler Check Kit de para la validación de la uniformidad de la temperatura en los termocicladores listo para usar, prealicuotado REF 7104 (10 tests) REF 71044 (4 tests) Contenidos
Documentos de Referencia para Diagnóstico de Semilla de Papa de Canadá
 SECRETARIA DE AGRICULTURA, GANADERÍA. DESARROLLO RURAL, PESCA Y ALIMENTACIÓN Dirección General de Sanidad Vegetal Centro Nacional de Referencia Fitosanitaria Subdirección de Diagnóstico Fitosanitario Documentos
SECRETARIA DE AGRICULTURA, GANADERÍA. DESARROLLO RURAL, PESCA Y ALIMENTACIÓN Dirección General de Sanidad Vegetal Centro Nacional de Referencia Fitosanitaria Subdirección de Diagnóstico Fitosanitario Documentos
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos
 Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Transferencia de material genético II. 1) Aislamiento de plásmidos Lisis alcalina
 Transferencia de material genético II 1) Aislamiento de plásmidos Lisis alcalina ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar que hay células transformantes de acuerdo
Transferencia de material genético II 1) Aislamiento de plásmidos Lisis alcalina ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar que hay células transformantes de acuerdo
Fig. 1. Modelo de activación e inactivación de las proteínas G heterotriméricas.
 ANÁLISIS COMPARATIVO DEL DNA GENÓMICO Y mrna S DE LOS GENES GPA DE Mucor circinelloides Ayala Lugo A.; Martínez Cadena Ma. G.; Sánchez Patlán A.A. División de Ciencias Naturales y Exactas Universidad de
ANÁLISIS COMPARATIVO DEL DNA GENÓMICO Y mrna S DE LOS GENES GPA DE Mucor circinelloides Ayala Lugo A.; Martínez Cadena Ma. G.; Sánchez Patlán A.A. División de Ciencias Naturales y Exactas Universidad de
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
PLANIFICACIÓN DE ASIGNATURA
 PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
DANAGENE SALIVA KIT. Ref Extracciones / Ref Extracciones
 DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION Este kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras de saliva
DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION Este kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras de saliva
Guía Práctica 9. Producción de micelio en medio líquido para extracción de ADN. Micelio de hongos. Contenido
 Guía Práctica 9 Micelio de hongos Producción de micelio en medio líquido para extracción de ADN Guillermo Castellanos, Experto en Investigación 2 Carlos Jara, Ing. Agr. M.Sc., Asociado en Investigación
Guía Práctica 9 Micelio de hongos Producción de micelio en medio líquido para extracción de ADN Guillermo Castellanos, Experto en Investigación 2 Carlos Jara, Ing. Agr. M.Sc., Asociado en Investigación
Caracteristicas del ADN. Se rompen con las siguientes condiciones -Temperaturas >90 - ph >10.5
 PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
Optimización de la extracción de ADN de Passiflora ligularis para el análisis por medio de marcadores moleculares
 UNIVERSITAS SCIENTIARUM Disponible en línea en: www.javeriana.edu.co/universitas_scientiarum 2009, Vol. 14 N x, xx-xx Comunicación corta Optimización de la extracción de ADN de Passiflora ligularis para
UNIVERSITAS SCIENTIARUM Disponible en línea en: www.javeriana.edu.co/universitas_scientiarum 2009, Vol. 14 N x, xx-xx Comunicación corta Optimización de la extracción de ADN de Passiflora ligularis para
Código de barras de ADN en Dactylopius coccus Costa cochinilla del carmín (Hemíptera: Dactylopiidae): Estudio preliminar
 IPEN Informe Científico Tecnológico. Volumen 13 (2013). p. 25-30. ISSN 1684-1662 Código de barras de ADN en Dactylopius coccus Costa cochinilla del carmín (Hemíptera: Dactylopiidae): Estudio preliminar
IPEN Informe Científico Tecnológico. Volumen 13 (2013). p. 25-30. ISSN 1684-1662 Código de barras de ADN en Dactylopius coccus Costa cochinilla del carmín (Hemíptera: Dactylopiidae): Estudio preliminar
TAXONOMÍA MOLECULAR. Dra. Alicia Luque CEREMIC
 TAXONOMÍA MOLECULAR Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las condiciones ambientales. PLEOMORFISMO Debido al bajo poder
TAXONOMÍA MOLECULAR Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las condiciones ambientales. PLEOMORFISMO Debido al bajo poder
DANAGENE PLASMID MIDI/MAXI KIT
 REF.0702.4 25 Midis REF.0702.5 10 Maxis DANAGENE PLASMID MIDI/MAXI KIT 1. INTRODUCCION DANAGENE PLASMID Midi/Maxiprep Kit proporciona un método simple para purificar ADN plasmídico a partir de cultivos
REF.0702.4 25 Midis REF.0702.5 10 Maxis DANAGENE PLASMID MIDI/MAXI KIT 1. INTRODUCCION DANAGENE PLASMID Midi/Maxiprep Kit proporciona un método simple para purificar ADN plasmídico a partir de cultivos
Caracterización morfológica y molecular de una cepa silvestre mexicana perteneciente al género Ganoderma
 Caracterización morfológica y molecular de una cepa silvestre mexicana perteneciente al género Ganoderma Jorge Suárez-Medellín, Mauricio Luna-Rodríguez, Guillermo Mendoza, Alejandro Salinas 2 1 Mirna Leonor
Caracterización morfológica y molecular de una cepa silvestre mexicana perteneciente al género Ganoderma Jorge Suárez-Medellín, Mauricio Luna-Rodríguez, Guillermo Mendoza, Alejandro Salinas 2 1 Mirna Leonor
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN
 INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
BIOLOGIA CELULAR Y MOLECULAR. LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR)
 BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR) 1. INTRODUCCIÓN La Reacción en Cadena de la Polimerasa (PCR) es una técnica in Vitro que imita la habilidad natural
BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR) 1. INTRODUCCIÓN La Reacción en Cadena de la Polimerasa (PCR) es una técnica in Vitro que imita la habilidad natural
PCR SIMULADA. Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO
 Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Código: IDK-006 Ver: 1 MTHFR A1298C. Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa
 Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
Obtención de Secuencias de DNAr ITS y LSU para la Identificación de un Aislado de Nematodo Entomopatógeno Nativo (Rhabditida: Steinernematidae).
 Obtención de Secuencias de DNAr ITS y LSU para la Identificación de un Aislado de Nematodo Entomopatógeno Nativo (Rhabditida: Steinernematidae). Mariana del Carmen Orozco Uribe 1, Eva Judith Hueso Guerrero
Obtención de Secuencias de DNAr ITS y LSU para la Identificación de un Aislado de Nematodo Entomopatógeno Nativo (Rhabditida: Steinernematidae). Mariana del Carmen Orozco Uribe 1, Eva Judith Hueso Guerrero
Invest Clin 40(4): 257-266, 1999
 Invest Clin 40(4): 257-266, 1999 Análisis de la prevalencia de mutaciones puntuales en el codón 12 del oncogen K-ras en muestras no cancerosas de citología exfoliativa cervical positivas para VPH tipo
Invest Clin 40(4): 257-266, 1999 Análisis de la prevalencia de mutaciones puntuales en el codón 12 del oncogen K-ras en muestras no cancerosas de citología exfoliativa cervical positivas para VPH tipo
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
41. Comprobación de colonias transformantes mediante PCR y electroforesis
 41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
Laboratorio de Marcadores Moleculares del Programa de Cereales y Granos Nativos
 Laboratorio de Marcadores Moleculares del Programa de Cereales y Granos Nativos GUÍA PRÁCTICA: INTRODUCCIÓN AL USO DE MARCADORES MOLECULARES EN EL ANÁLISIS DE DIVERSIDAD GENÉTICA Bloque Práctico (Chirinos
Laboratorio de Marcadores Moleculares del Programa de Cereales y Granos Nativos GUÍA PRÁCTICA: INTRODUCCIÓN AL USO DE MARCADORES MOLECULARES EN EL ANÁLISIS DE DIVERSIDAD GENÉTICA Bloque Práctico (Chirinos
Transferencia de material genético II. Ensayos de restricción (digestión) del plásmido Electroforesis en geles de agarosa
 Transferencia de material genético II Ensayos de restricción (digestión) del plásmido Electroforesis en geles de agarosa ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar
Transferencia de material genético II Ensayos de restricción (digestión) del plásmido Electroforesis en geles de agarosa ESQUEMA GENERAL SESIÓN I: TRANSFORMAR cepas de E. coli con un plásmido. Verificar
UNIVERSIDAD AUTÓNOMA CHAPINGO PROGRAMA DE ASIGNATURA: BIO-613 MÉTODOS DE BIOLOGÍA MOLECULAR
 UNIVERSIDAD AUTÓNOMA CHAPINGO PROGRAMA DE ASIGNATURA: BIO-613 MÉTODOS DE BIOLOGÍA MOLECULAR I. DATOS GENERALES Departamento Programa educativo Nivel educativo Área del conocimiento Asignatura Carácter
UNIVERSIDAD AUTÓNOMA CHAPINGO PROGRAMA DE ASIGNATURA: BIO-613 MÉTODOS DE BIOLOGÍA MOLECULAR I. DATOS GENERALES Departamento Programa educativo Nivel educativo Área del conocimiento Asignatura Carácter
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR).
 Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
TRABAJO PRÁCTICO No. 2 EXTRACION DE ACIDO NUCLEICO VIRAL
 Unidad Curricular: Virología y Micología Veterinaria TRABAJO PRÁCTICO No. 2 EXTRACION DE ACIDO NUCLEICO VIRAL Las pruebas para el diagnostico específico de una infección viral se clasifican en dos tipos:
Unidad Curricular: Virología y Micología Veterinaria TRABAJO PRÁCTICO No. 2 EXTRACION DE ACIDO NUCLEICO VIRAL Las pruebas para el diagnostico específico de una infección viral se clasifican en dos tipos:
Código: IDK-003 Ver: 1. Factor V Leiden
 Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de
 Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de ASEBIR). 1 2 3 4 Anexo2: Ejemplo de protocolo para translocaciones
Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de ASEBIR). 1 2 3 4 Anexo2: Ejemplo de protocolo para translocaciones
Extracción de almidón de plátano cuadrado (Musa balbisiana Colla)
 Extracción de almidón de plátano cuadrado (Musa balbisiana Colla) Resumen M. en C. Lázaro De la Torre Gutiérrez Para la extracción del almidón se utilizó y se modifico la metodología indicada por Bello-Pérez
Extracción de almidón de plátano cuadrado (Musa balbisiana Colla) Resumen M. en C. Lázaro De la Torre Gutiérrez Para la extracción del almidón se utilizó y se modifico la metodología indicada por Bello-Pérez
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Preparación y Caracterización de Ácidos Nucleicos
 Práctico Nº 5. Ácidos Nucleicos I Preparación y Caracterización de Ácidos Nucleicos Facultad de Ciencias Bioquímica (Licenciatura en Ciencias Biológicas) Objetivos Laboratorio Ácidos Nucleicos I: Extracción
Práctico Nº 5. Ácidos Nucleicos I Preparación y Caracterización de Ácidos Nucleicos Facultad de Ciencias Bioquímica (Licenciatura en Ciencias Biológicas) Objetivos Laboratorio Ácidos Nucleicos I: Extracción
NUEVOS REGISTROS DE HONGOS POLIPOROIDES (BASIDIOMYCETES) PARA COSTA RICA
 NUEVOS REGISTROS DE HONGOS POLIPOROIDES (BASIDIOMYCETES) PARA COSTA RICA por M. MATA 1, A. RUIZ-BOYER 2, J. CARRANZA 3 & L. RYVARDEN 4 1 Instituto Nacional de Biodiversidad (INBio), Apdo. Postal 22-3100,
NUEVOS REGISTROS DE HONGOS POLIPOROIDES (BASIDIOMYCETES) PARA COSTA RICA por M. MATA 1, A. RUIZ-BOYER 2, J. CARRANZA 3 & L. RYVARDEN 4 1 Instituto Nacional de Biodiversidad (INBio), Apdo. Postal 22-3100,
Trabajo práctico 1: Ecología microbiana
 Trabajo práctico 1: Ecología microbiana Las bacterias y las arqueas presentan una increíble diversidad metabólica que excede ampliamente la encontrada en organismos superiores. Los procariotas mantienen
Trabajo práctico 1: Ecología microbiana Las bacterias y las arqueas presentan una increíble diversidad metabólica que excede ampliamente la encontrada en organismos superiores. Los procariotas mantienen
CAPÍTULO 3: RESULTADOS
 CAPÍTULO 3: RESULTADOS 3.1 Extracción y cuantificación de ADN La extracción de ADN realizada con el método modificado de Khanuja et al. (1999) de las muestras del grupo 4 (Pedernales Canoa Km 4, 65 individuos),
CAPÍTULO 3: RESULTADOS 3.1 Extracción y cuantificación de ADN La extracción de ADN realizada con el método modificado de Khanuja et al. (1999) de las muestras del grupo 4 (Pedernales Canoa Km 4, 65 individuos),
TÉCNICAS DE EXTRACCIÓN Y ANÁLISIS DE ÁCIDOS NUCLEICOS Dr. GERARDO RODRÍGUEZ ALVARADO
 TÉCNICAS DE EXTRACCIÓN Y ANÁLISIS DE ÁCIDOS NUCLEICOS Dr. GERARDO RODRÍGUEZ ALVARADO NUMERO DE CREDITOS: 8 SEMESTRE FEBRERO 2017 JULIO 2017 PRERREQUISITOS: Bioquímica, Microbiología, Micología NUMERO MAXIMO
TÉCNICAS DE EXTRACCIÓN Y ANÁLISIS DE ÁCIDOS NUCLEICOS Dr. GERARDO RODRÍGUEZ ALVARADO NUMERO DE CREDITOS: 8 SEMESTRE FEBRERO 2017 JULIO 2017 PRERREQUISITOS: Bioquímica, Microbiología, Micología NUMERO MAXIMO
Código: KIT-005 Ver: 1 MTHFR C677T
 Sistema para detección de la alteración C677T en el gene que codifica para la Metilen-tetrahidrofolato reductasa humana 1 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598)
Sistema para detección de la alteración C677T en el gene que codifica para la Metilen-tetrahidrofolato reductasa humana 1 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598)
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Funga Veracruzana. Laetiporus sulphureus. Núm Laetiporus sulphureus (Bull.: Fr.) Murrill = Polyporus sulphureus Bull.: Fr.
 Núm. 142 Enero 2015 Basidiomycota: Coriolaceae Laetiporus sulphureus Funga Veracruzana http://fungavera.blogspot.mx Por: Armando López R.*, Juventino García A.* y Alberto S. González M. e-mail: armlopez@uv.mx
Núm. 142 Enero 2015 Basidiomycota: Coriolaceae Laetiporus sulphureus Funga Veracruzana http://fungavera.blogspot.mx Por: Armando López R.*, Juventino García A.* y Alberto S. González M. e-mail: armlopez@uv.mx
TECNICAS DE BIOLOGIA MOLECULAR
 Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Desarrollo de Diagnósticos basados en Biología Molecular de las Principales Enfermedades Infecciosas de Importancia en Salud Pública
 Título: Detección por PCR del gen meca A través del cofinanciamiento con la SENESCYT del proyecto Desarrollo de los diagnósticos basados en biología molecular de las principales enfermedades infecciosas
Título: Detección por PCR del gen meca A través del cofinanciamiento con la SENESCYT del proyecto Desarrollo de los diagnósticos basados en biología molecular de las principales enfermedades infecciosas
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
Determinación de proteínas, carbohidratos y lípidos en semillas
 Determinación de proteínas, carbohidratos y lípidos en semillas de ajonjolí germinadas y sin germinar Objetivo: encontrar la variación de los compuestos de reserva de la semilla de ajonjolí en su germinación
Determinación de proteínas, carbohidratos y lípidos en semillas de ajonjolí germinadas y sin germinar Objetivo: encontrar la variación de los compuestos de reserva de la semilla de ajonjolí en su germinación
FRAGMENTO OLIGO 5-3 Hebra SECUENCIA 5' - - > 3' TAMAÑO (pb) F1. hmtl 569 L AACCAAACCCCAAAGACACC hmth2982 H CTGATCCAACATCGAGGTCG
 ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
Incluye Certificado de aprovechamiento, Refrigerio y almuerzo Acreditación de Colegio Microbiólogos Declaratoria de Interés institucional.
 CURSO BASICO Curso Básico Teórico-Práctico de Biología Molecular. Reacción en Cadena de Polimerasa (PCR) sus principios y fundamentos. Instructores Ysea. Fecha 2 y 3 noviembre 2016 Horario 8am a 6 pm Aula
CURSO BASICO Curso Básico Teórico-Práctico de Biología Molecular. Reacción en Cadena de Polimerasa (PCR) sus principios y fundamentos. Instructores Ysea. Fecha 2 y 3 noviembre 2016 Horario 8am a 6 pm Aula
7. RESULTADOS. De las 18 cepas previamente congeladas (-70 C) se recuperaron 13 cepas. En los
 7. RESULTADOS De las 18 cepas previamente congeladas (-70 C) se recuperaron 13 cepas. En los pacientes infectados con estas cepas, 8 (61.5%) presentaron gastritis crónica, 2 (15.38%) gastritis folicular,
7. RESULTADOS De las 18 cepas previamente congeladas (-70 C) se recuperaron 13 cepas. En los pacientes infectados con estas cepas, 8 (61.5%) presentaron gastritis crónica, 2 (15.38%) gastritis folicular,
Evaluación de daño del ADN en células bucales
 Evaluación de daño del ADN en células bucales Técnica del cometa alcalino A.D. Vanesa Verónica Miana Dr. Elio Prieto Universidad Abierta Interamericana, CAECIHS 04 de diciembre de 2013 Cometa alcalino:
Evaluación de daño del ADN en células bucales Técnica del cometa alcalino A.D. Vanesa Verónica Miana Dr. Elio Prieto Universidad Abierta Interamericana, CAECIHS 04 de diciembre de 2013 Cometa alcalino:
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa
 38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
Anexo Conservar a 4 o C.
 Anexo 1 Purificación de ADN viral 1. Partir de un sobrenadante de un cultivo celular infectado y clarificado. 2. Concentrar las formas brotantes de los baculovirus por centrifugación (centrifugrar 1,5
Anexo 1 Purificación de ADN viral 1. Partir de un sobrenadante de un cultivo celular infectado y clarificado. 2. Concentrar las formas brotantes de los baculovirus por centrifugación (centrifugrar 1,5
RESULTADOS Y DISCUSIÓN
 RESULTADOS Y DISCUSIÓN Estimación de la Concentración Proteica del Filtrado de Cultivo de Mycobacterium tuberculosis H37Rv La curva estándar con la que se estimó la concentración proteica del filtrado
RESULTADOS Y DISCUSIÓN Estimación de la Concentración Proteica del Filtrado de Cultivo de Mycobacterium tuberculosis H37Rv La curva estándar con la que se estimó la concentración proteica del filtrado
Bioquímica III- 2009
 Facultad de Ciencias Exactas, Universidad Nacional de La Plata Bioquímica III- 2009 Trabajo Práctico Nro 4 Extracción de RNA y DNA bacteriano INTRODUCCIÓN El RNA es el ácido nucleico más abundante en la
Facultad de Ciencias Exactas, Universidad Nacional de La Plata Bioquímica III- 2009 Trabajo Práctico Nro 4 Extracción de RNA y DNA bacteriano INTRODUCCIÓN El RNA es el ácido nucleico más abundante en la
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PROGRAMA ANALÍTICO DE LA ASIGNATURA ENTOMOLOGIA SISTEMATICA
 Datos generales. PROGRAMA ANALÍTICO DE LA ASIGNATURA ENTOMOLOGIA SISTEMATICA Programa: Entomología y Acarología. Programa educativo: Maestría y Doctorado en Ciencias. Nivel Educativo: Maestría y Doctorado.
Datos generales. PROGRAMA ANALÍTICO DE LA ASIGNATURA ENTOMOLOGIA SISTEMATICA Programa: Entomología y Acarología. Programa educativo: Maestría y Doctorado en Ciencias. Nivel Educativo: Maestría y Doctorado.
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico
 17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
MATERIA: BIOLOGÍA MOLECULAR
 UNIVERSIDAD DE GUADALAJARA CENTRO UNIVERSITARIO DE CIENCIAS EXACTAS E INGENIERIAS DEPARTAMENTO DE FARMACOBIOLOGÍA MATERIA: BIOLOGÍA MOLECULAR Nivel: Licenciatura Clave: FB224 Horas por semana: 5 Tipo:
UNIVERSIDAD DE GUADALAJARA CENTRO UNIVERSITARIO DE CIENCIAS EXACTAS E INGENIERIAS DEPARTAMENTO DE FARMACOBIOLOGÍA MATERIA: BIOLOGÍA MOLECULAR Nivel: Licenciatura Clave: FB224 Horas por semana: 5 Tipo:
Biología Molecular y Filogenia en Micología
 Biología Molecular y Filogenia en Micología Biól. Nicolás Pastor Micología 2014 Por qué usar herramientas moleculares? Sistemática - Delimitación de especies Concepto filogenético de especie Diagnosis
Biología Molecular y Filogenia en Micología Biól. Nicolás Pastor Micología 2014 Por qué usar herramientas moleculares? Sistemática - Delimitación de especies Concepto filogenético de especie Diagnosis
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP
 SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
 Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS. Aislamiento de plásmido
 ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS Aislamiento de plásmido Transferencia de material genético intercelular Transformación Conjugación Transformación Transducción Existe algún mecanismo
ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS Aislamiento de plásmido Transferencia de material genético intercelular Transformación Conjugación Transformación Transducción Existe algún mecanismo
