INFORME FINAL EVALUAR LA EFECTIVIDAD GENÉTICA DE CINCO GENES DE RESISTENCIA A BEGOMOVIRUS EN EL TOMATE. PROYECTO FODECYT No.
|
|
|
- Vicente Camacho Rodríguez
- hace 5 años
- Vistas:
Transcripción
1 CONSEJO NACIONAL DE CIENCIA Y TECNOLOGIA CONCYT- SECRETARIA NACIONAL DE CIENCIA Y TECNOLOGIA SENACYT- FONDO NACIONAL DE CIENCIA Y TECNOLOGIA FONACYT- FACULTAD DE AGRONOMIA FAUSAC UNIVERSIDAD DE SAN CARLOS DE GUATEMALA INFORME FINAL EVALUAR LA EFECTIVIDAD GENÉTICA DE CINCO GENES DE RESISTENCIA A BEGOMOVIRUS EN EL TOMATE PROYECTO FODECYT No Dr. Luis Mejía Investigador Principal Guatemala, agosto
2 AGRADECIMIENTOS: La realización de este trabajo, ha sido posible gracias al apoyo financiero dentro del Fondo Nacional de Ciencia y Tecnología, -FONACYT-, otorgado por la Secretaría Nacional de Ciencia y Tecnología -SENACYT- y el Consejo Nacional de Ciencia y Tecnología -CONCYT-. 2
3 RESUMEN Se determinó la efectividad genética de cinco genes asociados con la resistencia a los begomovirus presentes en Guatemala. Se han desarrollado marcadores moleculares para la detección de todos estos genes de resistencia a los begomovirus. Por medio de la utilización de estos marcadores es posible construir líneas portadoras de diferentes combinaciones de genes y luego poder evaluar la efectividad asociada con cada uno de los genes. Se evaluó el nivel de resistencia de líneas portadoras de diferentes combinaciones de estos genes en comparación con líneas portadoras de uno solo de los genes y líneas sin ninguno de los genes. Los resultados obtenidos indican que los genes Ty3a y Ty3 son los que confieren un mayor grado de resistencia, mientras que los genes Ty4 y Ty2 por si mismos no confieren ningún nivel significativo de resistencia. Sin embargo, en forma combinada con los genes Ty3a y Ty3, los genes Ty4 y Ty2 contribuyen con efecto aditivo a la resistencia. Estos resultados pueden ser utilizados para dirigir la investigación sobre cuáles genes utilizar en sobreposición para aumentar los niveles de resistencia y para producir híbridos con mayor durabilidad. 3
4 SUMMARY (ABSTRACT) The genetic effectiveness of five genes associated with resistance to begomoviruses present in Guatemala was determined. Molecular markers for the detection of these genes were developed, their utilization allowed the construction of lines carrying different gene combinations and subsequently the evaluation of the contribution of these genes to resistance to begomoviruses. The level of resistance of lines carrying different gene combinations was determined in comparison with lines carrying only one or none of these genes. The results indicate that genes Ty3 and Ty3a confer a greater level of resistance, while genes Ty4 and Ty2 alone do not confer any significant level of resistance. However, in combination with genes Ty3 and Ty3a, genes Ty4 and Ty2 contribute with an additive effect to resistance. Gen Ty4 appears to be associated with lethal or sub lethal factors which produce a deficit of the gametes that carry it. The data collectively supports the presence of additional yet unknown genes of resistance. These results can be used in research related to the genes to be used to increase the levels of resistance and the production of hybrids with more durable resistance. 4
5 EQUIPO DE INVESTIGACIÓN: Luis Mejía De León, Rudy Estuardo Teni Cacao, Brenda Esperanza García Contreras, Ana Cristina Barillas Palma y Luis Méndez, Facultad de Agronomía, Universidad de San Carlos; Douglas Paul Maxwell, Departamento de Patología Vegetal, Universidad de Wisconsin-Madison. 5
6 TABLA DE CONTENIDOS RESUMEN... 3 SUMMARY (ABSTRACT)... 4 PARTE I... 7 I.1 INTRODUCCIÓN... 7 I.2 Planteamiento del problema... 9 I.2.1 Antecedentes en Guatemala... 9 I.2.2 Justificación del trabajo de investigación... 9 I.3 OBJETIVOS E HIPOTESIS I.3.1 Objetivos I General I Específicos I.3.2 Hipótesis I.4 METODOLOGÍA I.4.1 Localización I.4.2 Materiales I.4.3 Métodos PARTE II II. MARCO TEÓRICO PARTE III III.1 RESULTADOS III.2 DISCUSIÓN DE RESULTADOS: PARTE IV IV.1 CONCLUSIONES IV.2 RECOMENDACIONES: IV.3 REFERENCIAS BIBLIOGRÁFICAS IV.4 Anexos Parte V V.1 Informe financiero
7 PARTE I I.1 INTRODUCCIÓN I.1.1 Geminivirus y Begomovirus Los Geminivirus son virus de ADN que infectan un amplio rango de plantas hospederas. Sus genomas consisten de una o dos moléculas de ADN (ADN-A y ADN-B) de cadena sencilla covalentemente cerradas (circular). Los geminivirus miembros del género Begomovirus (nombre derivado de la especie tipo Bean Golden Mosaic Virus) son transmitidos por la mosca blanca y naturalmente infectan a varios cultivos de las Solanaceas tales como tomate y chile. Las infecciones mixtas, con dos o más geminivirus infectando una misma planta, son comunes en áreas tropicales y subtropicales del mundo (Polston y Anderson, 1997). Los Begomovirus han sido la principal causa de importantes pérdidas en la producción de tomate en Centro América y otras regiones productoras de tomate del mundo durante los últimos 20 años. El manejo de esta enfermedad ha sido difícil debido a la alta diversidad de los begomovirus (Ala-Poikela et al., 2006; Garcia-Andres et al. 2009; Fernandes et al., 2008; Nakhla et al., 2005; Seal et al., 2006) y al aumento en las poblaciones del vector, la mosca blanca, Bemisia tabaci, que se han producido debido principalmente a cambios en las practicas de cultivo (Morales y Anderson, 2001). En algunas regiones, el tomate se cultiva durante todo el año, lo cual constituye una constante fuente de inoculo viral y la disponibilidad de hospederos para la mosca blanca. I.1.2 Control de los begomovirus Los begomovirus infecciosos del tomate han permanecido un obstáculo principal a la producción de tomate en muchas regiones tropicales y sub-tropicales del planeta. Se han intentado estrategias de manejo que incluyen 1) el uso extensivo de insecticidas para el control del vector, la mosca blanca, Bemisia tabaci, 2) producción de transplantes libres de virus, 3) períodos libres de hospederos, 4) uso de coberturas a prueba de insectos en micro o macrotuneles durante 30 días o más y 5) utilización de híbridos resistentes. Durante la última década la disponibilidad de híbridos con resistencia moderada ha aumentado y la mayoría de las compañías de semillas han invertido recursos para la incorporación de genes de resistencia a begomovirus a sus últimas generaciones de híbridos. I.1.3 Genes de resistencia a los begomovirus Se han identificado varios genes de resistencia a los begomovirus en las últimas décadas (Ji et al., 2007). Zamir et al. (1994) asdcribieron el primer gen de resistencia, el gen Ty1, proveniente de la especie silvestre Solanum chilense, accesión LA1969, a una region comprendida entre 4cM y 10cM en el brazo corto del cromosoma 6. El gen Ty2 7
8 fue incorporado al genoma del tomate a partir de la especie silvestre Solanum habrochaites por Hanson et al. (2000), la introgresión fue localizada en una región entre 84 y 91cM en el cromosoma 11. Más recientemente, Ji et al. (2007b) reportaron una introgresión, Ty3, proveniente de S. chilense accesión LA2779, localizad entre 19 a 25cM en el cromosoma 6. Otra introgresión en esta misma región proveniente de S. chilense accesión LA1932 fue designada como Ty3a (Ji et al., 2007a). Las líneas resistentes derivadas de la accesión LA1932 contenían otro gen, Ty4, recientemente localizado en la mitad superior del cromosoma 3 (Ji et al., 2008) cerca de los 82cM. Un QTL, denominado Ty5, fue recientemente localizado en una línea, TY172, con introgresiones de S. peruvianum a 16 a 46cM en el cromosoma 4 (Anbinder et al., 2009). I.1.4 Marcadores moleculares Los marcadores moleculares son secuencias de nucleótidos ligadas a genes, en este caso a los genes de resistencia a begomovirus Ty1, Ty2, Ty3, Ty3a y Ty4, que son utilizados para identificar la presencia del gen. Su utilización en el mejoramiento de plantas consiste en la capacidad de seleccionar plantas resistentes mediante el proceso conocido como selección asistida por marcadores. 8
9 I.2 Planteamiento del problema I.2.1 Antecedentes en Guatemala En Guatemala, se han introducido muchas prácticas para el control de la virosis transmitida por la mosca blanca; estas incluyen, la utilización de plántulas libres de virus, la cobertura con tela en el campo de las plantas transplantadas y el uso de nuevos insecticidas. Pero, aun con estas estrategias de manejo, estos patógenos continúan ocasionado grandes pérdidas a la agricultura nacional. Una práctica de manejo que podría ser efectiva en la reducción de las pérdidas es el desarrollo de híbridos de tomate resistentes a los begomovirus y con una calidad hortícola aceptable. Hasta hace poco, no existía ningún híbrido comercial de este tipo, en el 2007, Semillas Tropicales, S. A., una nueva compañía de semillas localizada en Antigua, ha introducido varios híbridos con niveles moderados a altos de resistencia a begomovirus. Estos híbridos fueron desarrollados utilizando líneas desarrolladas en la Facultad de Agronomía de la Universidad de de San Carlos con el apoyo técnico de otras instituciones internacionales y con financiamiento nacional e internacional. Un mejor conocimiento de los genes asociados con la resistencia a los begomovirus sería de gran utilidad para el futuro desarrollo de mejores híbridos resistentes a begomovirus. I.2.2 Justificación del trabajo de investigación Hasta ahora no se cuenta con estudios que evalúen la efectividad de estos loci, genes Ty1, Ty2, Ty3, Ty3a y Ty4, en cuanto a conferir resistencia a los begomovirus. Se cuenta solo con un estudio que describe los resultados del cruce de líneas con genes de resistencia provenientes de diferentes especies silvestres, sin embargo este estudio no incluyó un análisis molecular de los loci de resistencia (Vidavski, et al., 2008). Por lo tanto, este estudio constituye el único que utiliza métodos moleculares para determinar la interacción de los genes de resistencia en los distintos genotipos. Por medio de la utilización de los marcadores fue posible construir líneas portadoras de diferentes combinaciones de genes de resistencia. Estos resultados serán utilizados para dirigir la investigación sobre cuáles genes utilizar en sobreposición para aumentar los niveles de resistencia y para producir híbridos con mayor durabilidad. Es posible que la sobreposición de los genes de resistencia resulte en un tipo de resistencia más fuerte y también más durable, haciendo más difícil al patógeno sobreponerla o quebrarla mediante mutación. La sobreposición de introgresiones para resistencia a begomovirus derivadas de diferentes accesiones de S. chilense y de diferentes especies silvestres como S. peruvianum y S. habrochaites tiene un buen potencial en el mejoramiento del tomate (Vidavsky et al., 2008). La utilización de varias fuentes de resistencia en un híbrido podría impedir la selección de formas virales con nuevas combinaciones genómicas que pudieran ser más agresivos en plantas con una sola introgresión de resistencia a begomovirus (García-Andrés et al., 2009). Este proyecto para evaluar a efectividad de introgresiones de resistencia a los begomovirus (Ty1, Ty2, Ty3, Ty3a, y Ty4) se inició 9
10 debido a la necesidad de comprender como los diferentes genes de resistencia responden a los begomovirus. Se espera que esta información contribuya a guiar la sobreposición de las introgresiones para resistencia a begomovirus en la formación de híbridos con una resistencia más durable. 10
11 I.3 OBJETIVOS E HIPOTESIS I.3.1 Objetivos I General I Específicos I.3.2 Hipótesis Evaluar la efectividad genética de cinco genes de resistencia a Begomovirus en tomate. Separación de los genes acoplados Ty3a de Ty4. Sobreposición de los genes Ty1, Ty2, Ty3 (o Ty3a) y Ty4, en una misma línea homocigótica. Evaluar el nivel de resistencia a begomovirus en líneas portadoras de los genes Ty1-Ty2-Ty3 (o Ty3a) y Ty4 con relación a líneas portadoras de solo uno de los cuatro genes y líneas sin ninguno de los genes. Si los genes a evaluar están relacionados a la resistencia, las líneas portadoras de los genes Ty1-Ty2-Ty3-Ty3a y Ty4, serán más resistentes que las que contengan solo uno o ninguno de estos genes. 11
12 I.4 METODOLOGÍA I.4.1 Localización El diseño de cebadores y la secuenciación de fragmentos de PCR se realizó en el Laboratorio del Prof. Douglas Maxwell en el Departamento de Patología Vegetal de la Universidad de Wisconsin-Madison. La determinación de los genotipos por PCR se realizó en el Laboratorio de Biotecnología Vegetal de la Facultad de Agronomía de la Universidad de San Carlos. Las evaluaciones de campo, determinación de fenotipos, se realizaron en una parcela experimental localizada en Sanarate, El Progreso, a 14º, 45 y 30 latitud Norte y 90º, 12 y 23 longitud Oeste, a una altura de 800 metros sobre el nivel del mar. Esta región corresponde al bosque seco subtropical, con días soleados durante la época seca y parcialmente nublados durante la estación lluviosa. La precipitación media anual es de 667 mm y la temperatura promedio es de 32 o C, con una temperatura máxima de 40º C y mínima de 18º C. I.4.2 Materiales I Germoplasma Las líneas que se utilizaron en el proyecto son: la línea Gh13, homocigota para Ty3; la línea Gc143-2, homocigota para Ty1 y Ty3; la línea Gc171, homocigota para Ty3a y Ty4; la línea CLN2116, homocigota para Ty2 y la línea 21-a (GTc645-1), homocigota para Ty3a. En todos los casos, excepto en el caso de Ty2, los genes de resistencia se originaron de la especie silvestre S. chilense, Ty2 tuvo su origen en la especie S. habrochaites. Posteriormente, la línea susceptible HUJ-VF, sin ninguno de estos genes de resistencia, será utilizada como progenitor en varios cruces. La población F2 utilizada para la separación de los genes Ty3a y Ty4 proviene de un híbrido producido mediante el cruce de la línea Gc171 con una línea susceptible. I.4.3 Métodos I Extracción del ADN La extracción de ADN se hizo utilizando el Kit de Extracción MasterPure TM Plant Leaf DNA Purification Kit, de EPICENTRE Biotechnologies. I Condiciones de PCR En un total de 25 µl de reacción: 2.5 µl 2.5 mm dntps, 2.5µl amortiguador 10X, 2.5µl 25mM MgCl 2, 0.1µl Taq polymerasa (Promega Corp., Madison, WI), 2.5µl de cada cebador a una concentración de 10µM, 5 µl de ADN a una concentración de 15 12
13 ng/µl y H 2 O. Los parámetros para el termociclador (MJ DNA Engine PT200 Thermocylcer, MJ Research Inc., Waltham, MA) fueron los siguientes: denaturación a 94 C durante 3 min, luego 35 ciclos a 94 C durante 30 segundos, hibridación a 53 C durante 1 min y extensión a 72 C durante 1 min, seguida de 72 C durante 10 minutos, luego la reacción se mantiene a 4 C. I Separación de los genes acoplados Ty3a y Ty4 Se sembró semilla de la generación F2 de un híbrido, producido por el cruce entre la línea Gc171, portadora de los genes Ty3a y Ty4 y una línea susceptible. Se tomaron muestras de tejido foliar de 77 plantas individuales para análisis molecular. Se determinó el genotipo de las plantas F2 mediante PCR, utilizando los cebadores específicos P6-25F2 y P6-25R5. Se determinó el genotipo de 77 las plantas F2, para detectar la presencia del gen Ty3a. Además, se determinó el genotipo en las 77 plantas con relación al gen Ty4, utilizando los cebadores P3-76F2 Y P3-76R2 (Ji et al., 2008). Se seleccionaron plantas F2 homocigotas para uno solo de los dos genes o para ambos genes (RR o Ty3a/Ty3a, Ty4/Ty4; RS o Ty3a/Ty3a, ty4/ty4; SR o ty3a/ty3a, Ty4/Ty4 y SS o ty3a/ty3a, ty4/ty4) y se permitió su autofecundación para la producción de familias F3. Las familias F3, con sus diferentes genotipos, se llevaron al campo para la determinación de su fenotipo o resistencia a begomovirus transmitidos por mosca blanca (Figura 2). I Segregación de los alelos Ty4 y ty4 Con el objeto de verificar la segregación de los alelos Ty4/ty4, se retrocruzó la planta heterocigota para el gen Ty4, L56 (Ty3a/ty3a, Ty4/ty4) con el hibrido la línea homocigota recesiva Silverado (ty3a/ty3a, ty4/ty4). Posteriormente se realizó el análisis genotípico de las plantas individuales, por medio de marcadores moleculares. Cualquier desviación significativa de la proporción esperada 1:1, a favor del genotipo ty3a/ty3a, ty4/ty4, confirmaría el efecto deletéreo o dañino de la presencia del alelo Ty4. Se consideró que, para tener una mayor confianza en el resultado, debería de repetirse la retrocruza utilizando líneas resistentes portadoras del gen Ty4 solo, y no en combinación con Ty3a. Para ello, se retrocruzó la planta heterocigota L17 (ty3a/ty3a, Ty4/ty4) con el hibrido Silverado (ty3a/ty3a, ty4/ty4) y también se realizó el cruce recíproco de estas plantas. I Combinación de los genes Ty2 y Ty3 Del cruce de la línea resistente Gh13, portadora del gene Ty3 (ty2/ty2, Ty3/Ty3), con la línea CLN2116 portadora del gen Ty2 (Ty2/Ty2, ty3/ty3) se obtuvo semilla F2 (Figura 3). Se determinó el genotipo de las plantas F2, utilizando los marcadores moleculares correspondientes y se identificaron los individuos recombinantes portadores uno o ambos genes de resistencia (genotipos RR, Ty2/Ty2, Ty3/Ty3; RS, Ty2/Ty2, ty3/ty3; SR, ty2/ty2, Ty3/Ty3 y SS, ty2/ty2, ty3/ty3), los cuales fueron transplantados en el invernadero para la obtención de semilla F3. Se verificó el genotipo de las familias F3 y se transplantaron a campo, en la parcela experimental localizada en Sanarate, para la determinación de su resistencia a begomovirus transmitidos por mosca blanca (Figuras 4 13
14 y 5). Se transplantaron 17 familias F3, las dos líneas parentales y un control susceptible en tres repeticiones de cinco plantas cada una. El nivel de infección de las plantas fue adecuado para establecer el nivel de resistencia. I Combinación de los genes Ty1, Ty3 y Ty2 Del cruce entre la línea resistente Gc143-2, portadora de los genes Ty1 y Ty3, y la línea B188-2, derivada del híbrido TyQueen, portadora del gen Ty2 (Ty2/Ty2) se obtuvo semilla F2. Utilizando los marcadores moleculares correspondientes, se determinó el genotipo de las plantas F2 y se identificaron los individuos recombinantes portadores de uno o ambos genes de resistencia (RR, Ty2/Ty2, Ty3/Ty3; RS, Ty2/Ty2, ty3/ty3; SR, ty2/ty2, Ty3/Ty3 y SS, ty2/ty2, ty3/ty3) los cuales fueron transplantados en el invernadero para la obtención de semilla F3. Se verificó el genotipo de las familias F3 y se transplantaron a campo para la determinación de su resistencia. Se transplantaron 17 familias F3, las dos líneas parentales y un control susceptible en tres repeticiones de cinco plantas cada una. El nivel de infección de las plantas fue adecuado para establecer el nivel de resistencia, ya que dos semanas después del transplante pudo determinarse que las plantas susceptibles empezaban a mostrar síntomas claros de virosis. I Combinación de los genes Ty3a y Ty2 La línea 21-a, portadora del gen Ty3a, fue cruzada con la línea B188-2, portadora del gen Ty2. Se evaluaron los individuos heterocigotos (Ty3a/ty3a, Ty2/ty2), comparando su resistencia con la de los genotipos parentales. Las líneas 21-a y B188-2 también fueron cruzada con la variedad susceptible HUJ-VF (ty2/ty2, ty3/ty3). Los individuos monohíbridos, heterocigotos Ty3a/ty3a, ty2/ty2 y ty3a/ty3a, Ty2/ty2 también fueron evaluados para resistencia con relación a los genotipos parentales. El genotipo de las plantas F1 fue verificado individualmente y se transplantaron a campo el 26 de agosto para la determinación del fenotipo de resistencia. Se transplantaron los tres híbridos (21- a X B188-2, 21-a X HUJ-VF y B188-2 X HUJ-VF), las tres líneas parentales y un control susceptible en tres repeticiones de cinco plantas cada una. I Combinación de los genes Ty3a y Ty3 La línea 21-a (Ty3a/Ty3a) fue cruzada con la línea portadora del gen Ty3 Gh13 (Ty3/Ty3). Se evalúo la resistencia del híbrido con relación a los genotipos parentales. El genotipo de las plantas F1 fue determinado individualmente y luego fue transplantado en el campo para la determinación de su fenotipo de resistencia. Se transplanto el híbrido (GTc645-1 X Gh13), las dos líneas parentales y un control susceptible en tres repeticiones de cinco plantas cada una. El genotipo de las plantas F1 fue determinado individualmente y luego, el fueron transplantadas en el campo para la determinación de su fenotipo de resistencia. Se transplanto el híbrido (21-a X Gh13), las dos líneas parentales y un control susceptible en tres repeticiones de cinco plantas cada una. 14
15 I Escala para evaluación de síntomas Para la determinación de la resistencia se utilizó una escala de 0 a 6, en la cual 0 significa ausencia de síntomas y 6 presencia severa de síntomas, una calificación menor de 3 se considera indicativa de resistencia. 15
16 PARTE II II. MARCO TEÓRICO II.1 Mejoramiento del tomate para resistencia a virosis transmitida por mosca blanca en Guatemala La virosis en el tomate producida por los geminivirus transmitidos por la mosca blanca (begomovirus), ha afectado significativamente la producción de tomate en Guatemala y otros países de Centro América durante más de una década (Polston y Anderson, 1997; Morales y Anderson, 2001; Mejía et al., 2002). Muchos campos de cultivo en las principales áreas de producción de Guatemala presentan un 100% de incidencia de infección por begomovirus y con frecuencia son abandonados. Cuando las plantas se infectan temprano en su desarrollo los síntomas son severos e incluyen un amarillamiento y deformación (acolochamiento) de las hojas y el achaparramiento de la planta. A pesar de que las prácticas culturales, tales como el uso de plántulas libres de virus o el uso de túneles cubiertos por hasta 30 días después del transplante, han reducido la severidad de esta enfermedad, los productores aun sostienen considerables pérdidas. El uso excesivo de insecticidas tiene un alto costo económico y un efecto negativo sobre el ambiente y la salud. La introducción de variedades resistentes sería una adición deseable a otras estrategias de manejo integrado para el control de esta enfermedad. Hasta hace poco no se contaba con variedades resistentes a los begomovirus en Guatemala y Centro América. En 1998 se inició un proyecto para seleccionar tomates con resistencia (Mejía, 1999 y 2001). Este proyecto se localizó en un área de intensa producción hortícola durante todo el año. El clima es cálido y seco y las poblaciones de mosca blanca son generalmente altas. Las plantaciones de tomate normalmente muestran un 100% de incidencia de infección por begomovirus. Esta área es considerada un lugar de alta variabilidad para begomovirus infecciosos del tomate, casi todos los begomovirus que presentes en Centro América se encuentran en esta área, con frecuencia se encuentran en infecciones mixtas (Nakhla et al., 2006). Se obtuvo germoplasma de tomate con resistencia al Tomato yellow leaf curl virus (TYLCV), un begomovirus monopartito del Hemisferio Oriental, y al Tomato mottle virus (ToMoV), un begomovirus bipartito presente en Florida. Este germoplasma fue obtenido utilizando diferentes fuentes de resistencia, de las especies silvestres L. hirsutum (Vidavsky y Czosnek, 1998), L. chilense (Scott et al., 1995), L. peruvianum (Friedmann et al., 1998) y L. pimpinellifolium (Laterrot, 1992). Las plántulas fueron producidas a campo abierto cerca del sitio experimental y estuvieron expuestas a las moscas blancas virulíferas provenientes de los campos infectados. No se utilizó ningún control específico contra el vector. Las plántulas se sembraron en un diseño de bloques al azar con cuatro replicaciones de diez plantas. Los síntomas se registraron utilizando una escala de severidad (DSI) de 0-4, en la cuál una calificación menor de 2 es considerada indicativa de resistencia. Se tomaron calificaciones de DSI a los 30 y 60 días después del 16
17 transplante. Durante la cosecha se registraron los pesos promedio por fruto y los rendimientos promedio por planta. Se seleccionaron individualmente las plantas resistentes durante varias generaciones hasta la obtención de líneas fenotípicamente homogéneas. Las líneas con resistencia de S. habrochaites (Vidavsky y Czosnek, 1998) fueron Gh1, Gh3 y Gh13 y fueron seleccionadas del híbrido FAVI 9; la línea Gh2 fue seleccionada del híbrido FAVI 12. La línea Gper11 seleccionada a partir de la línea Ty198 con resistencia derivada de L. peruvianum (Friedmann et al., 1998), Gper12 y Gper19 se obtuvieron de la línea TY197. Las líneas con resistencia proveniente de L. chilense (Scott, 1995) fueron Gc9 y Gc16 y fueron seleccionadas de las líneas Fla595-2 y Fla658-2BK respectivamente. La línea Gpimper10 se obtuvo de la población segregante Pimper J-13 con resistencia derivada de L. pimpinellifolium y L. peruvianum (Laterrot, 1992). La línea susceptible GC6 fue seleccionada de la variedad cubana HC-7880, con buena adaptación a condiciones tropicales. No todos los genotipos resistentes a TYLCV evaluados en Guatemala tuvieron niveles útiles de resistencia a los begomovirus locales; por ejemplo, las líneas TY 52, con resistencia derivada de L. chilense (Zamir et al., 1994) y H24, con resistencia de L. hirsutum (Kaloo and Banerjee, 1990) fueron susceptibles. A los 30 días después del transplante, la calificación de DSI de la líneas susceptible GC6 generalmente fue superior a 3, mientras que la de las líneas resistentes fue menor de 2. Los rendimientos por planta variaron de entre 148 gr. para GC6 a 2,312 gr. para Gc16. Se determinó el nivel de DSI para varios híbridos obtenidos de cruces entre las líneas más resistentes con genes de diferentes fuentes de resistencia. En todos los casos las calificaciones de DSI fueron menores y los rendimientos considerablemente más altos que el híbrido comercial susceptible Elios. La calificación de DSI de los híbridos fue similar a la de los progenitores. Se cruzaron líneas resistentes obtenidas de diferentes fuentes de resistencia con líneas susceptible para determinar las relaciones de dominancia. Los cruces de Gc9, Gc16, Gper11 o Gpimper10 con una línea o híbrido susceptible siempre produjeron poblaciones F1 altamente susceptibles. Solo los cruces de los progenitores resistentes portadores de genes de resistencia derivados de S. habrochaites con líneas o híbridos susceptible produjeron poblaciones F1 con DSIs menores de 2 a los 30 días después del transplante. Con el objeto de introducir en estos genotipos resistentes otros caracteres de interés, se realizaron cruces con líneas susceptible portadoras de algunas de estas características. Estas características incluyeron principalmente resistencia a otros patógenos y caracteres del fruto, tales como forma Saladette y buena firmeza, debido a que el 80% de los tomates cultivados en Guatemala producidos a campo abierto son del tipo Saladette y poseen buenas características de firmeza. Se seleccionaron líneas de estos híbridos en base a resistencia a begomovirus, forma del fruto, firmeza y rendimiento. Varias de estas líneas, las cuáles poseen excelentes niveles de resistencia, firmeza y rendimiento en combinación con otros caracteres deseables, han sido cruzadas para producir híbridos actualmente en evaluación para la producción comercial. Se cuenta con más de 30 líneas con altos niveles de resistencia con varios tamaños y forma 17
18 de frutos para el desarrollo de híbridos para los mercados de Guatemala y Centro América. Los resultados obtenidos indican que es posible el desarrollo de líneas con resistencia tanto a begomovirus monopartitos como bipartitos. Los primeros cruces con líneas resistentes indicaron que el nivel de resistencia de los híbridos podía ser aumentado cuando se introducían genes de resistencia de ambos progenitores. Por ejemplo, cuando la línea P (la línea que dió origen a la línea Gpimper10) fue cruzada con una línea susceptible, el híbrido mostró un bajo nivel de resistencia (DSI a los 60 días después del trasplante= 2.9, comparado con 3.1 en el control susceptible). Sin embargo, cuando se cruzó la misma línea con la línea resistente F (la línea de la que se derivó la línea Gh13), esta produjo un híbrido con un alto nivel de resistencia (DSI = 1.9). Este resultado no podría atribuirse solo a la dominancia, ya que la línea F cruzada con la línea susceptible tuvo un nivel intermedio de resistencia (DSI = 2.1). Las pruebas de campo mostraron que los cruces entre dos líneas resistentes produjeron los más altos niveles de resistencia. Estos cruces también indican que la resistencia derivada de S. habrochaites fue al menos parcialmente dominante, mientras que la derivada de S. pimpinellifolium y S. peruvianum fue de una naturaleza más recesiva. Los híbridos con una línea progenitora con resistencia derivada de S. habrochaites como uno de sus progeniores siempre fueron más resistentes. Estos resultados sugieren que la combinación de fuentes de resistencia provenientes de diferentes especies silvestres podría tener la ventaja de proporcionar mayor durabilidad (Mejía et al., 2005). Los híbridos entre líneas resistentes y susceptibles con alto valor hortícola han mostrado altos niveles de resistencia y buenos rendimientos. A partir de estos híbridos se han seleccionado líneas mejoradas con altos niveles de resistencia y buenas características hortícolas. Los cruces entre estas líneas resistentes mejoradas han resultado en híbridos con alta resistencia a begomovirus y buenas cualidades para el mercado guatemalteco. Hasta ahora se han evaluado más de mil híbridos experimentales y actualmente se cuenta con varios híbridos comerciales, los que ya están siendo utilizados por los productores. II.2 Los antepasados silvestres en el mejoramiento del tomate Las especies del genero Solanum (antes Lycopersicon) (Peralta et al., 2008) se han dividido en dos grupos principales de acuerdo al color del fruto, rojo o verde. También se han clasificado en dos complejos de acuerdo a su afinidad de hibridación con el tomate cultivado, lycopersicon y peruvianum. Las especies de fruto rojo son S. lycopersicon, S. lycopersicon var ceraciforme, S. pimpinellifolium y S. cheesmanii. Las especies de fruto verde son S. peruvianum, S. habrochaites (antes L. hirsutum), S. parviflorum, S. chmielewskii y S. penellii. El centro de distribución del género queda en los Andes, esta región abarca desde Ecuador al norte, hasta Chile al sur, las Islas Galapagos al oeste y al este la cordillera de los Andes. Con muy pocas excepciones, las especies silvestres son alógamas (de polinización cruzada), mientras que el tomate cultivado es autógamo (autofecundado). Existen muchas especies de insectos silvestres que son los responsables de la polinización de las especies silvestres en su habitat nativo. La distribución del tomate cultivado S. lycopersicon es la más amplia de todas las especies, los cultivares primitivos, incluyendo el tomatillo var. ceraciforme, se encuentran en una amplia 18
19 distribución desde Perú hasta México. En general, la variabilidad genética estimada por medio de marcadores moleculares y morfológicos es pobre en S. lycopersicon, especialmente en los cultivares modernos, lo cuál no es de extrañar debido a su naturaleza autógama. En la parte sur de la distribución, en donde se encuentran la mayoría de los cerasiformes primitivos, es en donde observa una mayor variabilidad. El máximo se encuentra sobre la costa peruana y ecuatoriana, principalmente debido a la introgresión de genes de la especie silvestre de fruto rojo S. pimpinellifolium. Esta alta variabilidad esta acompañada por una alta tasa de alogámia, la cual esta correlacionada con el nivel de exención del estigma. S. pimpinellifolium es una fuente importante de resistencia a enfermedades, este se cruza fácilmente con el tomate cultivado. Otra especie cercanamente emparentada es S. cheesmanii, esta es endémica de las islas Galápagos. En contraste con S. pimpinellifolium su fruto es de color anaranjado, únicamente una especie de abeja lo poliniza y ha sido distribuido por las tortugas. Esta especie ha servido como fuente de tolerancia a la salinidad y otros genes importancia, como el gen jointless (ausencia de cojinete). Las dos formas botánicas de L. cheesmanii pueden cruzarse fácilmente en ambas direcciones con el tomate cultivado. De las especies de fruto verde, S. peruvianum y S. chilense son las más distantes del tomate cultivado. Estas se cruzan sin mucha dificultad entre ellas, pero se necesita del rescate de embriones para la obtención de híbridos interespecíficos entre estas dos especies y el tomate cultivado. El resto de las especies de fruto verde pueden cruzarse sin mayor dificultad, pero solo en una dirección, utilizando S. lycopersicon como el progenitor femenino y las especies silvestres como donadoras de polen. Este fenómeno se conoce como incompatibilidad unilateral. La mayoría de las especies de fruto verde son autoincompatibles, lo cuál resulta en un alto nivel de polimorfismo debido a la alogámia. El nivel de polimorfismo de ADN dentro de accesiones y especies esta altamente correlacionado con el sistema de apareamiento. Las especies autoincompatibles son 10 veces más variables que las especies auto-compatibles. Las especies silvestres del tomate son muy útiles en el mejoramiento del tomate, han sido buenas fuentes de características deseables. II. 3 Introgresión de resistencia a Geminivirus transmitidos por mosca blanca A finales de la década de los 1960s dieron inicio varios programas de mejoramiento con el objetivo de producir variedades de tomate resistentes al geminivirus monopartito del Hemisferio Oriental Tomato Yellow Leaf Curl Virus (TYLCV). Estos programas se basan en la introgresión de la resistencia o tolerancia a partir de accesiones de especies silvestres a la especie cultivada Solanum lycopersicon (antes Lycopersicon esculentum). Dependiendo de la fuente, la resistencia se ha reportado como condicionada por de uno a cinco genes, recesivos o dominantes (Pico et al. 1996; Zakay et al. 1991). El primer cultivar comercial con tolerancia, Ty20, portador de resistencia derivada de S. peruvianum (Pilowsky y Cohen, 1990) mostró un tardío desarrollo de síntoma y de acumulación de ADN viral (Rom et al. 1993). Un gen de tolerancia derivado de S. chilense LA1969, Ty1, fue asignado al cromosoma 6 utilizando marcadores RFLP y ha sido introducido a S. lycopersicon (Michelson et al. 1994; Zakay et al., 1991). Otro gen de tolerancia derivado de S. pimpinellifolium fue asignado, utilizando marcadores 19
20 RAPD, también al cromosoma 6 pero a un locus diferente que Ty-1 (Chagué et al., 1997). Después de todos estos años, las mejores variedades disponibles muestran tolerancia al virus (Pico et al. 1996). Después de la infección, los rendimientos son superiores que los de las variedades susceptibles y los síntomas de la enfermedad son menores, pero todas las plantas contienen diferentes niveles de virus (Lapidot et al. 1997). Estas variedades tolerantes casi siempre requieren protección de los insectos virulíferos con insecticidas o telas durante el primer mes después del transplante. II.3.1 Solanum habrochaites Entre las ocho especies silvestres emparentadas con el tomate, Solanum habrochaites (antes Lycopersicon hirsutum) es una de las más diversas y es la fuente más rica de genes para resistencia a plagas y enfermedades del tomate cultivado (Miller y Tanksley, 1990; Rick, 1982). Por ejemplo, de las 16 plagas y 32 enfermedades del tomate para las cuáles se ha identificado resistencia en las especies silvestres, resistencia a 14 y 9, respectivamente, se encontraron en S. habrochaites (Rick, 1982; Rick, 1989). Sin embargo, la variación genética dentro de esta especie ha permanecido en gran medida inexplorada, principalmente por dificultades en la identificación y la transferencia de genes útiles por medio de los métodos convencionales de mejoramiento. El desarrollo de los marcadores y mapas moleculares facilitará sin duda la caracterización y transferencia de genes de S. habrochaites al tomate cultivado. Con el fin de obtener variedades resistentes a inoculaciones por mosca blanca y que no permitan la acumulación viral Vidavsky y Czosnek (1998) iniciaron un proyecto para obtener resistencia de las especies silvestres. En este proyecto se determinó que solo dos accesiones de S. habrochaites contenían la mayor proporción de individuos resistentes. Se cruzaron plantas de las accesiones LA1777 y LA386 y el híbrido resultante fue cruzado con plantas susceptibles de S. lycopersicon, las plantas silvestres fueron utilizadas como progenitores masculinos, seguida de una serie de autofecundaciones. Durante cada generación se seleccionaron individuos resistentes después de inoculación con moscas blancas virulíferas. Se obtuvo una línea estable en BC1F4, denominada 902, que no segregaba para resistencia y que presentaba buenas características hortícolas (frutos rojos de 80 a 120 gr., fertilidad, auto compatibilidad, vigor y buena morfología de planta). De acuerdo a los autores (Vidavsky y Czosnek, 1998) esta línea no permitió la replicación viral aun bajo fuerte presión de inoculación por mosca blanca de plántulas jóvenes y no necesita de protección por telas o insecticidas. La F1 con S. lycopersicon es tolerante, produce frutos de 120 a 150 gr. y no necesita protección de inoculación. Esta F1, híbridos FAVI 9 y FAVI 12, fue la que se evaluó en Guatemala, conjuntamente con otras fuentes de resistencia, en 1998 (Mejía, 1999). Del híbrido FAVI 9 se obtuvo la línea Gh13, portadora del gen Ty3 y de otros genes de resistencia aun desconocidos. II.3.2 Solanum chilense Se ha reportado la presencia resistencia al geminivirus transmitido por mosca blanca Tomato Yellow Leaf Curl Virus (TYLCV) en varias especies del género Solanum, 20
21 incluyendo S. peruvianum, S. pimpinellifolium, S. habrochaites y S. cheesmanii y se ha estudiado la herencia de algunas de estas resistencias (Hassan, 1984; Kasrawi et al., 1988; Kasrawi, 1989; Kalloo y Banerjee, 1990a; 1990b); Pilowsky y Cohen, 1974, 1990). Más recientemente, Zakay et al. (1991) reportó a la accesión LA1969 de L. chilense como más resistente a TYLCV que la proveniente de las especies silvestres anteriores en base a expresión de síntomas y criterios de detección de virus. Zamir et al. (1994) reportó resistencia a TYLCV, utilizando inoculación controlada y selección asistida por RFLP, derivada de LA1969. Esta resistencia fue determinada por un gen principal, parcialmente dominante, Ty1, localizado en el cromosoma 6. Simultáneamente, Laterrot (1992) llevaba a cabo un programa internacional para resistencia a TYLCV utilizando poblaciones derivadas de la selección recurrente de fuentes de resistencia de S. pimpinellifolium, S. cheesmanii, S. habrochaites y S. peruvianum. Más recientemente, Laterrot y Moretti (1994) reportaron la utilización de LA1969 como fuente de resistencia para TYLCV. En este proyecto se utilizó agroinoculación para seleccionar las plantas resistentes y se realizaron tres retrocruzas al tomate. El programa se desarrolló en dos direcciones: retrocruzamientos acelerados por agroinoculación y la retrocruza de los descendientes de plantas seleccionadas en varios países para resistencia a TYLCV. Selecciones de este programa también fueron evaluadas en Guatemala en 1998 (Mejía, 1999). Chagué et al. (1994) reportaron que LA1969 fue resistente al TYLCV de Taiwan (TTYLCV). Un geminivirus bipartito, Tomato Mottle Virus (ToMoV), fue detectado por primera vez en Florida a finales de los 1980 s (Abouzid et al., 1992; Polston et. al., 1993). Scott y Schuster (1991) tamizaron un número de accesiones de L. chilense así como accesiones de otras especies reportadas como tolerantes a geminivirus y otros virus en el tomate. Encontraron que 23 accesiones de L. chilense no presentaban síntomas de virosis mientras que las accesiones de las demás especies presentaban una mayor severidad de síntomas. A partir de entonces iniciaron un programa para introducir genes de resistencia de varias accesiones de L. chilense, incluyendo notablemente LA1932 y LA2779, al tomate. También de este programa se obtuvieron líneas resistentes las cuáles han sido utilizadas en Guatemala. La accesión LA1932 dio origen a la línea Gc171 portadora de los genes de resistencia Ty3a y Ty4 y de la accesión LA2779 se derivó la línea Gc143-2 portadora del gen de resistencia Ty3. II.4 El tomate como material experimental El tomate es un organismo ideal para investigación. El desarrollo del ADN recombinante y su aplicación al tomate promete resultar en descubrimientos importantes, los cuáles tendrán implicaciones en el futuro del mejoramiento de este cultivo. El mapa genético bien establecido con el que se cuenta actualmente no solo permitirá el marcaje de genes útiles para su eficiente transferencia en los programas de mejoramiento sino que también permitirá el mapeo de los genes introducidos por transformación genética. Los montajes de cromosomas en paquiteno en combinación con la hibridación in situ permitirá la localización física de muchos marcadores. La formación de híbridos intergenéricos entre Solanum lycopersicon y las especies parientes de género Solanum resultará en la transferencia de característica útiles. El tomate es considerado actualmente un organismo ideal para la investigación en genética molecular gracias a su 21
22 bien caracterizado genoma por métodos citogenéticos básicos. Actualmente se realiza un trabajo internacional para la secuenciación del genoma del tomate, este esfuerzo colaborativo es coordinado por la Solanacea Genomics Network ( II.5 Marcadores moleculares Los marcadores basados en las secuencias de nucleótidos del ADN han sido ampliamente desarrollados en el tomate y han venido a complementar a los marcadores morfológicos. Entre estos marcadores se cuenta con una gran variedad, incluyendo los RFLP s, los RAPD s, los microsatélites y los AFLP s. II.5.1Marcadores RFLP Se cuenta con esta técnica desde hace aproximadamente 20 años y proviene del campo de la genética molecular. En forma resumida, esta consiste en extraer el ADN total de la planta, digerirlo con un juego de enzimas de restricción y separar los fragmentos por electroforésis. Después de la digestión, los fragmentos son transferidos a una membrana (procedimiento conocido como Southern blotting) y seguidamente se hibridizan moléculas de ADN marcadas conocidas como sondas. Estas sondas pueden ser genes específicos o secuencias aleatorias, las cuáles reconocen segmentos específicos de ADN en la membrana. Aquellas que muestran complementariedad con la sonda aparecen en una película de rayos X como bandas. Cada enzima de restricción reconoce y corta una secuencia muy específica de nucleótidos, lo cuál resulta en diferentes patrones de bandeo dependiendo del juego de enzimas de restricción y sondas que se utilicen. La gran ventaja de la técnica de RFLP es su capacidad de detectar una cantidad de polimorfismo virtualmente ilimitada. Una gran ventaja de los RFLP s como marcadores genéticos es que son codominantes, el genotipo heterocigoto puede ser identificado. Se requiere de 5 a 10 ug de ADN y las membranas puede ser re-sondeadas. Varias membranas correspondientes a varios individuos pueden ser sondeadas simultáneamente para producir mapas de ligamiento. Las sondas se obtienen de bibliotecas (genotecas) de ADN, las cuales son bancos de fragmentos de 2 a 3 Kb los cuales han sido introducidos en plásmidos bacterianos. Existen dos clases principales de genotecas, 1) bibliotecas genómicas, las cuales consisten de fragmentos aleatorios de ADN, los cuales pueden incluir tanto secuencias codificantes como no-codificantes y 2) bibliotecas cadn, las cuales son obtenidas de ARN retro-transcrito a ADN y luego clonado en plásmidos. II.5.2 Marcadores basados en PCR Con el advenimiento de la técnica de reacción en cadena de la polimerasa (PCR), inventada por C. Mullis a mediados de los 1980 s, se revolucionó la genética molecular. Esta técnica permite la producción de grandes cantidades de ADN específico sin necesidad de clonar. El PCR explota ciertas características de la replicación del ADN. La ADN polimerasa utiliza ADN monocatenario como patrón o molde para la síntesis de un nuevo filamento complementario. Este ADN monocatenario se produce por calentamiento del ADN bicatenario a temperaturas cercanas a la ebullición, lo cual causa 22
23 la separación de los filamento o desnaturalización. La ADN polimerasa requiere de una molécula pequeña de ADN monocatenario para iniciar (cebar) la síntesis, esto se logra al proporcionar un cebador oligonucleotídico que se acopla al molde en el punto de iniciación. Es la elección de este cebador (primer) la que determina la región específica en el ADN a partir de la cuál se realizará la síntesis. Ambos filamentos de ADN pueden utilizarse como moldes para síntesis siempre y cuando se agreguen cebadores para cada filamento. Se seleccionan cebadores localizados a ambos lados de la región de ADN que se desea amplificar. El nuevo filamento sintetizado a partir del cebador se extiende más allá de la posición ocupada por el cebador en el filamento opuesto. Por lo tanto, en cada filamento sintetizado se generan nuevos sitios de unión para los cebadores. Seguidamente la mezcla de reacción es desnaturalizada por calor para separar los filamentos originales de los recién sintetizados, los cuales se vuelven disponibles para nuevos ciclos de hibridación y síntesis. El resultado neto es que al final de n ciclos la secuencia original se habrá amplificado 2 n veces. Los ingredientes para la reacción de amplificación son: 1) ADN blanco (target) a ser amplificado, típicamente ADN genómico, la cantidad necesaria es muy pequeña, aproximadamente 10 ng., 2) cebadores de 10 a 25 nucleótidos de largo, 3) los cuatro tipos de desoxiribonucleótidos (datp, dttp, dgtp y dctp), 4) ADN polimerasa resistente a la temperatura, como Taq polimerasa y 5) amortiguador, incluyendo MgCl. La reacción se realiza a través de los siguientes ciclos de temperatura: 1) 95 o C durante 2-3 minutos para la desnaturalización, 2) 60 a 65 o C durante 30 segundos, dependiendo de la afinidad de los cebadores al molde, para el acoplamiento de los cebadores, 3) 72 o C durante 1 minuto para la síntesis. Este paso final en el ciclo es seguido por la desnaturalización o separación de filamentos de ADN nuevos y viejos durante 30 segundos a 94 o C. Luego los ciclos se repiten de 30 a 60 veces. II Marcadores RAPDs El nombre de esta técnica deriva de random amplified polymorphic DNA. En ella se utilizan cebadores arbitrarios de solo 10 nucleótido de longitud y baja temperatura de hibridación. Los cebadores reconocen regiones homologas en el genoma que ocurren al azar y a partir de ellas amplifican productos de ADN. Estos productos son separados en geles de agarosa y visualizados por tinción con bromuro de etidio. Cada producto se deriva de una región del genoma que contiene dos segmentos cortos de ADN con homología con el cebador, estos segmentos deben estar presentes en filamentos opuestos y lo suficientemente cerca (a unos 3 Kb, máximo 5) el uno del otro para que la amplificación pueda ocurrir. Normalmente, el polimorfismo entre individuos ocurre por diferencias en secuencias en uno o ambos sitios de unión del cebador y puede manifestarse como presencia o ausencia de una banda RAPD específica, por lo tanto se comportan como marcadores dominantes. Otros tipos de polimorfismos que ocurren con menor frecuencia son deleciones y duplicaciones. Se requieren pequeñas cantidades de ADN para el desarrollo de estos marcadores, de 15 a 25 ng, el equipamiento es simple y no se utiliza radioactividad. Pueden existir problemas de reproducibilidad debido al uso de diferentes fuentes de enzimas, concentración de ADN y aparatos para amplificación (termocicladores). También, en la mayoría de los casos estos marcadores son específicos para una progenie, de manera que es difícil su utilización por varios laboratorios. Las 23
24 bandas RAPD contienen secuencias de ADN repetitivo dispersas, lo cuál las hace inútiles como sondas en experimentos de hibridación. Los cebadores de corta extensión para la identificación por RAPD amplifican simultáneamente otras bandas de regiones no ligadas, por lo tanto carecen de especificidad por sitios únicos en el genoma. También, muchas veces carecen de homología entre taxa relacionados. Cuando se encuentra un marcador RAPD asociado a una característica de interés este puede convertirse a un marcador más reproducible mediante varias estrategias. II Marcadores SCAR Derivan su nombre de sequence characterized amplified regions. Las bandas RAPD son clonadas y secuenciadas en sus extremos para diseñar cebadores más largos y completamente homólogos. Las primeras 10 bases (extremo 5 ) son idénticas al cebador arbitrario original. Estos generalmente amplificarán una sola banda con alta repetitividad. Algunas veces pueden detectarse polimorfismos de tamaño lo cuál resulta en marcadores codominantes. En otras ocasiones, la dominancia se retiene, o el polimorfismo se pierde y se producen bandas idénticas, estas pueden distinguirse en algunos casos por digestión con endonucleasas de corte frecuente (cortadoras de 4 bases). II Microsatélites Son secuencias de alta repetitividad que consisten patrones cortos, por ejemplo CT, CA, AT, CTT, etc. Fueron reportadas originalmente en secuencias humanas y su uso se ha ampliado a las plantas. El patrón más frecuente en plantas parece ser AT, GT, AG/TC, TAT (en arroz GAA, GCG). Para el desarrollo de estos marcadores, por lo general se requiere de un trabajo básico bastante costoso. Se requiere de la construcción de bibliotecas genómicas de clones pequeños, idealmente menores de 1 Kb, seguida del rastreo de las genotecas con oligos sintéticos que contengan el patrón de interés. Luego se aislan aquellos que hibridicen y se secuencian para determinar si verdaderamente contienen microsatélites. Una vez se encuentra. Se sintetizan oligos correspondientes a ambos extremos los cuáles son utilizados par amplificación. Las principales secuencias descubiertas en tomate son GACA/GATA las cuales son altamente polimórficas. Estas pueden distinguir variedades de tomate para uso industrial (Vosman et al., 1992), lo cual no se ha logrado con otros tipos de marcadores. Los loci de los microsatélites se mapean en las regiones centroméricas de la mayoría de los cromosomas del tomate (Granadillo y Tanksley, 1996). Más recientemente se han desarrollado nuevos microsatélites en el tomate con alelos múltiples, mediante la búsqueda en bases de datos de las Solanáceas de repeticiones de di o tetranucleótidos (Smulders et al., 1997). Las secuencias microsatélites son muy importantes en cultivos en los que no se cuenta con polimorfismo en otros marcadores. Generalmente tienen un alto número de alelos múltiples y alta heterocigosidad, entre 7 y 10 veces más que los RFLP s. II AFLP Derivan su nombre de amplified length polymorphism. Esta técnica fue desarrollada en Holanda por la compañía Key Gene. Consiste en la selección de RFLP s 24
25 específicos los cuáles luego son amplificados por PCR, esto elimina una gran cantidad de ADN no-polimórfico. El ADN es cortado con endonucleasas y luego se ligan adaptadores bicatenarios a los extremos de los fragmentos. De esta manera la secuencia de los adaptadores y los nucleótidos adyacentes sirven como sitios de unión para la amplificación. Se agregan nucleótidos selectivos a los extremos 3 de los cebadores, los cuáles reconocen solo a un subgrupo de sitios de restricción. Solo son amplificados los fragmentos de restricción en los cuáles los nucleótidos de ambos lados del sitio de restricción son complementarios a los nucleótidos selectivos. Para la digestión del ADN se utilizan dos enzimas, una de corte raro y otra de corte frecuente. Después de la amplificación los fragmentos son separados en geles de acrilamida para secuenciación. Los AFLP son marcadores dominantes. II.6 Aplicación de los marcadores moleculares en el tomate Una de las principales aplicaciones de los marcadores moleculares en el mejoramiento del tomate es el marcado de genes útiles, lo cuál se conoce como selección asistida por marcadores. II.6.1 Resistencia a nemátodos, Mi. Se han descubierto marcadores RFLP asociados al gen Mi que confiere resistencia a nemátodos y recientemente se han descrito marcadores basados en PCR (Williamson et al., 1994; Seah et al., 2007). II.6.2 Virus del mosaico del tabaco. Existen tres loci de resistencia a este virus, Tm-1, Tm-2 y Tm-2a. Se han desarrollado basados en PCR para los loci Tm-1 (Ohmori et al., 1995a), Tm-2 (Ohmori et al., 1995) y Tm-2a (Dax et al., 1994). II.6.3 Otros genes de resistencia a enfermedades. También existen marcadores ligados a los genes de resistencia a Cladosporium (Cf-2 a 9), Verticillium (Ve), Fusarium (I1, I2 y I3), tomato spotted wilt virus (Sw5), Pseudomonas (Pto), Xanthomonas y powdery mildew (Lv). II.7 Utilización de marcadores y variabilidad genética en el tomate Durante su evolución y proceso de evolución, la especie cultivada de tomate Solanum Lycopersicon Mill., sufrió varios cuellos de botella impuestos por la selección y la endogámia extrema de poblaciones limitadas, particularmente en Europa y los Estados Unidos de Norte América (Rick, 1976). Tales eventos causaron un agotamiento de la variabilidad dentro del tomate cultivado. Lo limitado de la variación genética hace al cultivo vulnerable a enfermedades y epidemias de plagas y amenaza el potencial para un mejoramiento sostenido (Tanksley y McCouch, 1997). Afortunadamente, existen ocho especies silvestres emparentadas del tomate que contienen una enorme riqueza de variabilidad genética para varios caracteres de importancia y todas ellas pueden ser hibridizadas con el tipo cultivado, aunque con mayor dificultad cuando se utilizan 25
26 especies lejanamente emparentadas tales como Solanum chilense o Solanum peruvianum (Rick, 1982). Solo una pequeña fracción de la variabilidad genética dentro de las especies silvestres emparentadas del tomate ha sido utilizada en el mejoramiento del tomate (Tanksley y McCouch, 1997) y esta se ha limitado principalmente a caracteres de resistencia a plagas y enfermedades de herencia simple. De particular importancia, sin embargo, son la variación genética relacionada con caracteres complejos de importancia agronómica, incluyendo tolerancia a factores ambientales adversos, resistencia a plagas y enfermedades, potencial de rendimiento y calidad nutricional. Los protocolos convencionales de genética de plantas y mejoramiento, a pesar de ser efectivos en varios aspectos, han sido poco eficientes para la identificación, selección y transferencia de genes con menos de un efecto mayor (Foolad, 1999). Además, la introgresión de genes de una especie silvestre a una variedad cultivada por medio de los procedimientos del mejoramiento convencional puede encontrarse con varias dificultades (Jones y Qualset, 1984). Las especies silvestres son deficientes con relación a muchas características necesarias en los sistemas modernos de cultivo. Durante la hibridación interespecífica, la introducción de genes exóticos en germoplasma élite puede conducir a la introducción de características agronómicas indeseables por arrastre de ligamiento. Por lo tanto, una tarea principal es la eliminación de una gran cantidad de genes indeseables al mismo tiempo que se mantienen unas pocas características deseables. Se requiere de una serie de retrocruzas al progenitor cultivado recurrente, de manera alternativa con generaciones de autofecundación y selección fenotípica, antes de obtener las combinaciones deseas de caracteres parentales. Este procedimiento, normalmente parte de una etapa previa al mejoramiento propiamente dicho, es laboriosa y costosa. Con el advenimiento de los marcadores moleculares y los mapas, tales limitaciones podrían ya no ser imposibles de superar. Los mapas moleculares de ligamiento pueden facilitar la determinación exacta del número, localización cromosómica y el efecto individual de genes o loci para caracteres cuantitativos (QTLs) que afectan a los caracteres complejos. Luego de su identificación, los QTLs deseables podrían se introducidos a la variedad cultivada y las características indeseables podrían ser eliminadas con relativa rapidez por medio de un procedimiento conocido como selección asistida por marcadores (MAS) (Tanksley et al., 1989). Recientemente, se han desarrollado mapas de marcadores moleculares para muchas especies vegetales incluyendo el tomate y han sido utilizados exitosamente para varias aplicaciones genéticas como marcaje de genes y mapeo de QTLs (Foolad et al., 1997, Peterson et al., 1988), MAS y desarrollo de germoplasma (Stuber, 1994) y clonación posicional de genes (Martin et al., 1993). La tecnología de marcadores puede utilizarse para facilitar la explotación de la variación genética cuantitativa dentro de germoplasmas exóticos del tomate y de esta manera ampliar la base genética del cultivo. II.8 Desarrollo de marcadores moleculares De enero a abril, 2007, Brenda E. García, recientemente graduada de la Facultad de Agronomía de la USAC, realizó una estadía en el laboratorio del Prof. Douglas P. Maxwell en el Departamento de Patología Vegetal de la Universidad de Wisconsin- 26
27 Madison. Por medio de la colaboración con colegas en la Universidad de Florida (UFL), la Universidad Hebrea de Jerusalén (HUJ) y el Centro Asiático para la Investigación y el Desarrollo de Hortalizas (AVRDC), se desarrollaron marcadores moleculares para la detección de cinco genes relacionados con la resistencia a begomovirus en el germoplasma desarrollado en Guatemala (Ji et al., 2007a). Estos marcadores moleculares utilizan métodos de PCR y son del tipo codominante SCAR (sequence characterized amplified region) y CAPS (cleavage amplified polymorphic sequences) (Garcia et al., 2007; Ji et al., 2007, Pérez de Castro et al., 2007). Así mismo, también se desarrollo, un nuevo marcador, tipo codominante SCAR, para el gen Mi, que confiere resistencia a nematodos. Esto último es importante, debido a que el gen de resistencia a begomovirus Ty-1, se encuentra localizado en la misma región que el gen Mi del cromosoma 6 (Sean et al., 2007). Garcia et al. (2007) desarrollaron un marcador codominante tipo SCAR a partir del marcador RFLP TO302 a una altura de 89 cm y este marcador efectivamente detectó la introgresión Ty2 en líneas del AVRDC. El gen Ty3 fue reportado recientemente por Ji y Scott (2006) en líneas derivadas de una introgresión de S. chilense y fue localizado cercano a los 25 cm en el cromosoma 6. Salus, Martin y Maxwell de la Universidad de Wisconsin-Madison en colaboración con Ji y Scott de la Universidad de Florida desarrollaron un marcador tipo SCAR utilizando los cebadores FLUW25 (Ji et al., 2007). Martin y Maxwell encontraron que una línea proveniente de la USAC, Gc171, contenía una secuencia diferente en la región Ty3 a los 25 cm y este gen es el denominado Ty3a (información no publicada). Jenson y Maxwell (información no publicada) han desarrollado un marcador tipo CAPS que diferencia a los genes Ty3 y Ty3a de la secuencia de Solanum lycopersicum en esa posición. Otro alelo, Ty4, ha sido localizado recientemente en la mitad superior del cromosoma 3 (Ji et al., 2008). II.8.1 Protocolos para la detección de los genes asociados con resistencia a begomovirus Estos métodos basados en PCR han sido desarrollados y evaluados en el laboratorio del Dr. Douglas P. Maxwell en la Universidad de Wisconsin-Madison, con la colaboración de la Ing. Brenda E. García (Maxwell y asociados, 2007, ver sitio de Internet ( El siguiente protocolo, relacionado con la detección del gen Ty3, se utiliza como ejemplo: Marcador tipo codominante SCAR para la detección del gen Ty3. II.8.2 Diseño de los primers FLUW-25 y P6-25F2/P6-25R5 Ji y Scott (2006a y 2006b) asignaron el gen Ty-3 para resistencia al Tomato yellow leaf curl virus (TYLCV), localizado en una introgresión procedente de LA2779 (S. chilense), a un lugar cerca del locus FER (a 25 cm) en el cromosoma 6. El gen FER esta localizado en el clon BAC 56B23 (AY678298). Ji y Scott diseñaron cebadores para PCR para amplificar secuencias cercanas al extremo 5 de este clon BAC. Estos cebadores fueron utilizados para amplificar fragmentos de PCR en la variedad susceptible a begomovirus Purple Russian, S. lycopersicum, y una línea resistente, Gh25, del programa de mejoramiento de tomate de la USAC, con una introgresión en esta región 27
28 (Mejía et al., 2005). Estas secuencias fueron comparadas y seguidamente se diseñaron cebadores en ambos sentidos (hacia adelante y hacia atrás), estos son el FLUW-25F (5 CAAGTGTGCATATACTTCATA(T/G)TCACC) y el FLUW-25R (5 CCATATATAACCTCTGTTTCTATTTCGAC), respectivamente. Se esperaba que estos cebadores produjeran fragmentos de PCR en S. lycopersicum y en la introgresión de un tamaño de 475 y 641pb, respectivamente. Se diseñó otro juego de primers, P6-25-F2, 5 GGTAGTGGAAATGATGCTGCTC y P6-25-R5,5 GCTCTGCCTATTGTCCCATATATAACC, para amplificar la indel ty-3/ty-3 de 143-nt y para producir un fragmento más pequeño que el juego de primers FLUW25 (Ji et al., 2007) (Fig. 1). Utilizando las líneas resistentes a begomovirus, con resistencia derivada de la accesión silvestre S. chilense LA2779, Gc9 y Ih902 (Vidavsky and Czosnek, 1998), se obtuvo el fragmento Ty-3 esperado de 450pb. En el caso de líneas sin la introgresión se obtuvo un fragmento ty-3 de 320pb. Con líneas con resistencia derivada de la accesión silvestre S. chilense LA1932, como la Gc171, se obtuvo un fragmento Ty-3a de 630pb. Los híbridos heterocigotos pudieron identificarse fácilmente por medio de estos primers los cuales amplifican dos fragmentos correspondientes a ty-3 de S. lycopersicum (320pb) y cualquiera de los dos fragmentos Ty-3 (450pb) o Ty-3a (630pb) bp --- Fig. 1. Fragmentos de PCR obtenidos con los P6-25-F2/P6-25-R5. Carril 1, Marcador en escalera de ADN Benchtop, Promega, de 100pb; Carril 2, M (ty-3/ty- 3); Carril 3, Gc9 (Ty-3/Ty-3); Carril 4, híbrido F1 Romelia (Ty-3/ty-3); Carril 5, Gc171 (Ty-3a/Ty-3a); Carril 6, híbrido F1 GTc191-3 (Ty-3a/ty-3); Carril 7, marcador de 100pb. Se determinó la secuencia de los fragmentos de los tres tamaños producidos por la amplificación con los primers P6-25-F2/P6-25-R5 (Maxwell et al., 2007). Los fragmentos de 320pb y 450pb correspondieron a secuencias de S. lycopersicum y de S. chilense LA2779 respectivamente. El fragmento de 650pb proveniente de Gc171 contenía dos inserciones grandes, en comparación con la secuencia de líneas derivadas de S. lycopersicum. 28
29 Fotografía 1. Determinación de síntomas de infección viral en familias F3 a los 30 días después del transplante por los investigadores Brenda García y Rudy Teni. Fotografía 2. Plantas de un híbrido resistente, en primer plano, y plantas susceptibles en la parcela experimental a los 45 después del transplante. 29
30 Fotografía 3. Plantas resistentes (DSI=1) y susceptibles (DSI=6) de familias F3 en el momento de la evaluación. Las plantas susceptibles muestran síntomas severos de infección por begomovirus, incluyendo achaparramiento y deformación de las hojas. Fotografía 4. Estudiantes de la Facultad de Agronomía en la parcela experimental durante la evaluación de síntomas en octubre, De izquierda a derecha primero Dr. Luis Mejía, cuarto Dr. Douglas Maxwell y quinto Ing. Amilcar Sánchez. 30
31 PARTE III III.1 RESULTADOS III.1.1 Segregación de los alelos Ty3a y Ty4 En la F2, la introgresión correspondiente al gen Ty3a en el cromosoma 6 presentó una segregación Mendeliana normal 1:2:1, mientras que la introgresión correspondiente al gen Ty4 en el cromosoma 3 presentó una deficiencia de los genotipos ty4/ty4 (Cuadro 1). Cuadro 1. Segregación de las introgresiones correspondientes a Ty-3a y Ty-4 en una población F 2. Introgresión Homocigota dominante Heterocigota Homocigota recesivo Total X 2 Valor de p Ty3a Ty <0.005 Esperado :2:1 Fuente: FODECYT Al determinar genotipo de las plantas F2 pudo observarse que los genotipos portadores de la introgresión correspondiente al gen ty4 se produjeron con menor frecuencia de la esperada. Por ejemplo, con el genotipo Aabb (Ty3a/ty3a, ty4/ty4) se esperaban 25/77 plantas pero solo se obtuvieron 10 (Cuadro 2). Cuadro 2. Número de plantas de tomate de cada genotipo en la población F 2. AA AA AA Aa Aa Aa aa aa aa To p BB Bb bb BB Bb bb BB Bb bb tal X 2 Observado < Esperado A = Ty-3a, a = ty-3, B = Ty-4, b = ty-4 Fuente: FODECYT Este resultado parece indicar que la presencia de la introgresión correspondiente al gen Ty4 contiene factores, posiblemente arrastrados de la especie silvestre, que interfieren con la viabilidad de los gametos que la poseen. Por lo tanto, es de interés determinar cual es la contribución real del gen Ty4 a la resistencia de campo frente a los 31
32 begomovirus transmitidos por mosca blanca. Estos resultados fueron enviados para a la publicación Report of thetomato Genetics Cooperative y apareció en el Volumen 58, en la página 18 (ver Anexo 1) Con el objeto de verificar la segregación de los alelos Ty4/ty4, se retrocruzó la línea L17, heterocigotas para el gen Ty4, ty3a/ty3a, Ty4/ty4, con el híbrido Silverado, homocigoto recesivo ty3a/ty3a, ty4/ty4. El análisis genotípico de las plantas individuales, por medio de marcadores moleculares, identificó la presencia de 5 plantas heterocigotas (Ty4/ty4) y 22 plantas homocigota recesivas (ty4/ty4). Este resultado constituye una desviación significativa de la proporción esperada 1:1 y parecería confirmar la hipótesis del efecto deletéreo asociado al alelo Ty4 (Cuadro 3). Cuadro 3. Análisis de Chi 2 para datos genotípicos obtenidos de la retrocruza L17 X Silverado. Genotipo Proporción Observado Esperado Desviación Varianza esperada (o) (e) (d) (d 2 ) d 2 /e X 2 p Ty4/ty4 1/ ty4/ty4 1/ Fuente: FODECYT En el cruce reciproco, sin embargo, el análisis genotípico de las plantas individuales identificó la presencia de 14 plantas heterocigotas (Ty4/ty4) y 12 plantas homocigota recesivas (ty4/ty4), lo cual constituye una desviación no significativa de la proporción esperada, indicando una segregación normal de los alelos Ty4 y ty4 al utilizar la línea resistente como progenitor masculino (Cuadro 4). Cuadro 4. Análisis de Chi 2 para datos genotípicos obtenidos de la retrocruza Silverado X L17. Genotipo Proporción Observado Esperado Desviación Varianza esperada (o) (e) (d) (d 2 ) d 2 /e X 2 p Ty4/ty4 1/ ty4/ty4 1/ Fuente: FODECYT Se realizó un análisis molecular de 14 plantas F2 homocigotas, utilizando los cebadores P3-76F2 Y P3-76R2 para la detección del gen Ty4 (ver secuencias abajo). Por medio de método de PCR y secuenciación del producto de PCR se determinó que hay correlación positiva entre el marcador y la presencia de la introgresión en el cromosoma 3, a 76 cm, por lo que se comprueba que el marcador esta asociado a la misma. El Cuadro 5 muestra un resumen de los resultados obtenidos en el laboratorio de Patología Vegetal de la Universidad de Wisconsin-Madison en junio del
33 Cuadro 5. Relación entre el genotipo mostrado por el marcador y la presencia de la introgresión correspondiente al gen Ty4 (según secuencia) para 5 plantas homocigota dominantes (Ty4/Ty4) y 9 plantas homocigota recesivas (ty4/ty4). NOMBRE INTROGRESIÓN PRESENTE AUSENTE GENOTIPO L1 X ty4/ty4 L3 X ty4/ty4 L6 X Ty4/Ty4 L7 X ty4/ty4 L9 X ty4/ty4 L11 X ty4/ty4 L14 X ty4/ty4 L15 X Ty4/Ty4 L17 X ty4/ty4 L19 X ty4/ty4 L33 X Ty4/Ty4 L36 X Ty4/Ty4 L39 X Ty4/Ty4 L57 X ty4/ty4 Fuente: FODECYT Secuencias de cebadores P3-76F2 y P3-76R2 P3-76 F2: CAA TGG TGT GCT CGT CGA ATC AGA ATC GGA G P3-76 R2: GTT CCT TTT CAG CAT AGG CAG TAT TCC ATT TCG III Introgresión relacionada con el gen Ty4 A continuación se presentan las secuencias parciales de Gc171-1 (portadora del gen Ty4) y HUJ-VF (susceptible a Begomoviruses) y la comparación de las mismas. Los polimorfismos de nucleótidos simples (SNP s), así como una inserción/deleción de regular tamaño. 33
34 Gc171-1 (secuencia parcial): CAATGGTGTGCTCGTCGAATCGAATCGGAGCATGGAGTCCCATTGTCNACTGNAAAGGGA 60 GGGCTTTGATGTGGCCTCCATATGGCACTGGGCAACATGTAAAGCTTCCTGGTCCTTCTA 120 TTAGGGAACCAAATGTTTACGACTTCGGGCACTCCTTATAAGCAGATGTTCGATGACCTC 180 CGCCGCAAAGACGTCGAACTGTAAGCCAAAATTGCATTTATGCCTTTTCTAATTATCGTC 240 TCCTTTTGCTAATTCCTTGAACTCTCTGTGTTTAAAATTTCGTGTGTCGCTCAATTCAGT 300 TACCATTAAATAACCCTAATATGTGTAGAGTCTGTTATTTGCGCTGCACGAAGTGAATTT 360 AAGGATTTTCCATCTGCTAGGATTTGTATGATCTTCTTGAAGCTTTTGGATGCCTAATAT 420 CCTGTTCTAGTTTCCTTACCTTAATGGACAATTAGCATAATAAAATCGGAGGAGTTAATG 480 TAAGGGTATTCATACAAGTATAAATTTTACAAGAACTTCTCGTTTCGTTAGACGCCAATG 540 GAGGTTTCTTTTTAGGGAAAAAGTGTCTATCATTATTTCAAATGAGACAGGGGTCTGTAA 600 CAGGATTTCTATGTGTATTTGGAGTTGTTTTATTCTTGCGGATAAATTTTAGAACTTTGT 660 TTAGTTTTTGACTTCTGAATACGGGTAAAGTTGCATATTTTGGAATACGAAATTCTCAAA 720 TGTTCCACGCCATGATAATGTGGTGTGTTCCTTTTCAGGACAAAAGTCACAACCTCCACC 780 GTGTTTCTATGCTTTGAGATGGCTATTATACCTAGAATTGTTGGCAAATATTGCTTTATT 840 TTCAGAATCCTCTTGTTTTAATTCAAATTAGGGTTGAGCCTCAGTTTCATTTCCAGGTAC 900 AAACGAAATGGAATACTGCCTATGCTGAAAAGGAAC 936 HUJ-VF (secuencia parcial): CAATGGTGTGCTCGTCGAATCAGAATCGGAGCATGGAGTCACATTGTCTACTGAAAAGGG 60 AGGGATTTGATGTGGCCTCATATGGCACTGGGCAACATGtaaagcttcCTGGTCCTTCTA 120 TTAGGGAACCAAATGTTTACGACTTCGGCACTCCTTATAAGCAGATGTTCGATGACCTCC 180 GCCGCAAAGACGTCGAACTGTAAGCCAAAATTGCATTTATGCCTTTTCTAATTATCGTCT 240 CCTTTTGCTAATTCCTTGAACTCTCTGTGTTTAAAATTTCGTGTGTTACTCAATTCAGTT 300 ACCATTAAATAACCCTAATATGTGTAGTGTCTGTTATTTGCGCTGCACGAAGTGAATTTA 360 AGGATTTTCCATCTGCTAGGATTTGAATGATCTTCTTGAAGCTTTTGGATGCCTAATATC 420 CTGTTCTAGTTTCCTTACCTTAATGGACAATTAGCATAATAAAATCGGAGGAGTTAATGT 480 AAGGGTATTCATATAAGTATAAATTTTACAAGAACTTCTCGTTTCGTTAGACGCCAATGG 540 AGGTTTCTTTTTAGGGAAAATGTGTCTATCATTATTTCACATGAGACAGGGGGTCTGTAA 600 CAGGATTTTCATGTGTATTTGGAGTTGTTTTATTCTTGCGGATAAATTTTAGCACTTGTT 660 TAGTTTTTGACTTCTGAATATGGGTAAAGTTGCATATTCTGGAATACGAAATTCTCAAAT 720 GTTCCACGCCATGATAATGTGGTGTGTTCCTTTTCAGGACAAAAcGTCACAACCTCCACC 780 GTGTTTCTATACTTTGAGATGGCTGATATTGCTTTATTTTCAGAATCCTCTTGTTTTAAT 840 TCAAATTAGGGTTGAGCCTCAATTTCAATTTCCAGGTACAAACGAAATGGAATACTGCCT 900 ATGCTGAAAAGGAAC
35 Comparación de Gc171 (secuencia de abajo) y HUJ-VF (secuencia de arriba) 1 CAATGGTGTGCTCGTCGAATCAGAATCGGAGCATGGAGTCACATTGTCTACTGAAAAGGG 1 CAATGGTGTGCTCGTCGAATC.GAATCGGAGCATGGAGTCCCATTGTCNACTGNAAAGGG 61 AGGGATTTGATGTGGCCT.CATATGGCACTGGGCAACATGTAAAGCTTCCTGGTCCTTCT 60 AGGGCTTTGATGTGGCCTCCATATGGCACTGGGCAACATGTAAAGCTTCCTGGTCCTTCT 120 ATTAGGGAACCAAATGTTTACGACTTC.GGCACTCCTTATAAGCAGATGTTCGATGACCT 120 ATTAGGGAACCAAATGTTTACGACTTCGGGCACTCCTTATAAGCAGATGTTCGATGACCT 179 CCGCCGCAAAGACGTCGAACTGTAAGCCAAAATTGCATTTATGCCTTTTCTAATTATCGT 180 CCGCCGCAAAGACGTCGAACTGTAAGCCAAAATTGCATTTATGCCTTTTCTAATTATCGT 239 CTCCTTTTGCTAATTCCTTGAACTCTCTGTGTTTAAAATTTCGTGTGTTACTCAATTCAG 240 CTCCTTTTGCTAATTCCTTGAACTCTCTGTGTTTAAAATTTCGTGTGTCGCTCAATTCAG 299 TTACCATTAAATAACCCTAATATGTGTAGTGTCTGTTATTTGCGCTGCACGAAGTGAATT 300 TTACCATTAAATAACCCTAATATGTGTAGAGTCTGTTATTTGCGCTGCACGAAGTGAATT 359 TAAGGATTTTCCATCTGCTAGGATTTGAATGATCTTCTTGAAGCTTTTGGATGCCTAATA 360 TAAGGATTTTCCATCTGCTAGGATTTGTATGATCTTCTTGAAGCTTTTGGATGCCTAATA 419 TCCTGTTCTAGTTTCCTTACCTTAATGGACAATTAGCATAATAAAATCGGAGGAGTTAAT 420 TCCTGTTCTAGTTTCCTTACCTTAATGGACAATTAGCATAATAAAATCGGAGGAGTTAAT 479 GTAAGGGTATTCATATAAGTATAAATTTTACAAGAACTTCTCGTTTCGTTAGACGCCAAT 480 GTAAGGGTATTCATACAAGTATAAATTTTACAAGAACTTCTCGTTTCGTTAGACGCCAAT 539 GGAGGTTTCTTTTTAGGGAAAATGTGTCTATCATTATTTCACATGAGACAGGGGGTCTGT 540 GGAGGTTTCTTTTTAGGGAAAAAGTGTCTATCATTATTTCAAATGAGACA.GGGGTCTGT 599 AACAGGATTTTCATGTGTATTTGGAGTTGTTTTATTCTTGCGGATAAATTTTAGCAC.TT 599 AACAGGATTTCTATGTGTATTTGGAGTTGTTTTATTCTTGCGGATAAATTTTAGAACTTT 658 GTTTAGTTTTTGACTTCTGAATATGGGTAAAGTTGCATATTCTGGAATACGAAATTCTCA 659 GTTTAGTTTTTGACTTCTGAATACGGGTAAAGTTGCATATTTTGGAATACGAAATTCTCA 718 AATGTTCCACGCCATGATAATGTGGTGTGTTCCTTTTCAGGACAAAACGTCACAACCTCC 719 AATGTTCCACGCCATGATAATGTGGTGTGTTCCTTTTCAGGACAAAA.GTCACAACCTCC 778 ACCGTGTTTCTATACTTTGAGATGGCTG...ATATTGCTTT 778 ACCGTGTTTCTATGCTTTGAGATGGCTATTATACCTAGAATTGTTGGCAAATATTGCTTT 816 ATTTTCAGAATCCTCTTGTTTTAATTCAAATTAGGGTTGAGCCTCAATTTCAATTTCCAG 838 ATTTTCAGAATCCTCTTGTTTTAATTCAAATTAGGGTTGAGCCTCAGTTTC.ATTTCCAG 876 GTACAAACGAAATGGAATACTGCCTATGCTGAAAAGGAAC 897 GTACAAACGAAATGGAATACTGCCTATGCTGAAAAGGAAC * Rasmussen, A., Maxwell, D Chromosome 3, 76 cm. Universidad de Wisconsin-Madison. Disponible en: 35
36 III. 1.2 Separación de los genes acoplados Ty3a y Ty4 Dentro de 77 plantas F2 analizadas, se encontraron 17 plantas homocigota dominantes (Ty3a/Ty3a), 12 homocigota recesivas (ty3a/ty3a) y 48 heterocigotas (Ty3a/ty3a). Con relación al gen Ty4, se encontraron 10 plantas homocigota dominantes (Ty4/Ty4), 42 homocigota recesivas (ty4/ty4) y 25 heterocigotas (Ty4/ty4). De estas se obtuvieron 17 familias F3 con los genotipos RR, RS, SR y SS los cuales fueron evaluados en el campo para la determinación de su fenotipo de resistencia (Cuadro 6). Cuadro 6. Índice de severidad (DSI) promedio para 17 familias F3, sus parentales y un control, a los 31 días después del trasplante y a los 44 días después del trasplante. Familia Genotipo DSI (0-6) DSI (0-6) 44 días 31 días Bloque I Bloque II Bloque III L6 ty3/ty3,ty4/ty L7 Ty3a/Ty3a,ty4/ty L8 Ty3a/Ty3a,Ty4/ty L9 Ty3a/Ty3a,ty4/ty L17 ty3/ty3,ty4/ty L30 ty3/ty3,ty4/ty L36 ty3/ty3,ty4/ty L47 Ty3a/Ty3a,Ty4/ty L51 Ty3a/Ty3a,ty4/ty L54 Ty3a/Ty3a,Ty4/ty L57 Ty3a/Ty3a,Ty4/ty L68 Ty3a/Ty3a,ty4/ty R Ty3a/Ty3a;Ty4/Ty S ty3/ty3,ty4/ty C ty3/ty3,ty4/ty Fuente: FODECYT Media Con los datos obtenidos a los 44 días después del trasplante se realizó un análisis de varianza. Este análisis indica que el modelo explica la variación existente entre las unidades experimentales, las cuales consistían en 5 plantas de cada familia, distribuidas en 3 bloques al azar, por lo que se realizó una prueba de t para las medias y se estimaron los componentes de varianza. Se determinó que existen diferencias significativas entre los distintos genotipos, con la excepción de ty3/ty3, Ty4/Ty4 y ty3/ty3, Ty4/ty4, para los cuales los valores promedio de DSI fueron similares. Esto indica que no existe diferencia entre las plantas con genotipo homocigoto resistente y heterocigoto para el gen Ty4. De igual manera ocurrió con los genotipos Ty3a/Ty3a; Ty4/Ty4, Ty3a/Ty3a; Ty4/ty4 y Ty3a/Ty3a; ty4/ty4, lo cual parece indicar que el gen Ty4 no aporta a la resistencia al encontrarse junto con gen Ty3a en la misma planta. 36
37 Cuadro 7. Influencia de los distintos factores del modelo sobre la varianza fenotípica observada. Componente de Varianza Media DSI Estimada Porcentaje de varianza explicada Genotipo % Bloque % Genotipo X bloque % Error % Fuente: FODECYT El cuadro anterior indica que el genotipo es el componente más significativo dentro del modelo, explicando el 75% de la variación total. Los datos sugirieron que el gen Ty3a aporta mayor resistencia a Begomovirus que el gen Ty4 en las plantas evaluadas. Figura 2. Índice de severidad de la enfermedad (DSI) de familias F3 de genotipos RR (Ty3a/Ty3a, Ty4/Ty4 and Ty3a/Ty3a, Ty4/ty4), RS (Ty3a/Ty3a, ty4/ty4), SR (ty3a/ty3a, Ty4/Ty4 y ty3a/ty3a, Ty4/ty4), RR-C, línea resistente Gc171 (Ty3a/Ty3a, Ty4/Ty4), SS-C, línea susceptible Gh44 (ty3a/ty3a, ty4/ty4) y SS-2, híbrido comercial control susceptible (ty3a/ty3a, ty4/ty4). Los números indican el DSI promedio de 15 plantas en bloques al azar de tres replicaciones de cinco plantas cada una. 37
38 III.1.3 Combinación de los genes Ty2 y Ty3 El hibrido Gh13 (Ty3/Ty3, ty2/ty2) X CLN2116 (ty3/ty3, Ty2/Ty2) tuvo un nivel de síntomas de 3.9 a los 50 días después del transplante. De este híbrido, se obtuvo 17 familias F3 provenientes de plantas F2 individuales. El genotipo de cada una de estas familias fue verificado antes del transplante para la determinación de su fenotipo de resistencia. Cuadro 8. Genotipos para los genes Ty2 y Ty3 de plantas F3 del cruce Gh13 X CLN2116. Familia Genotipo Familia Genotipo F3 F3 A1 ty3/ty3, Ty2/Ty2 SR A85 Ty3/Ty3, Ty2/Ty2 RR A5 Ty3/Ty3, Ty2/ty2 RR A90 ty3/ty3, ty2/ty2 SS A18 Ty3/Ty3, ty2/ty2 RS A91 ty3/ty3, Ty2/Ty2 SR A42 ty3/ty3, ty2/ty2 SS A92 ty3/ty3, Ty2/Ty2 SR A53 Ty3/Ty3, ty2/ty2 RS A97 ty3/ty3, ty2/ty2 SS A54 Ty3/Ty3, Ty2/Ty2 RR A112 Ty3/Ty3, Ty2/Ty2 RR A81 ty3/ty3, Ty2/Ty2 SR A117 Ty3/Ty3, Ty2/Ty2 RR A83 ty3/ty3, Ty2/Ty2 SR A144 Ty3/Ty3, ty2/ty2 RS A84 Ty3/Ty3, Ty2/Ty2 RR Fuente: FODECYT Estas familias F3 fueron evaluadas a los 36 días después del transplante para la determinación de su fenotipo de resistencia (Cuadro 9). 38
39 Cuadro 9. Combinación de los genes de resistencia Ty3 y Ty2. Replica 1, planta número Replica 2, planta número Replica 3, planta número No. Campo Familia F3 Genotipo #1 #2 #3 #4 #5 #1 #2 #3 #4 #5 #1 #2 #3 #4 #5 1 A1 SR A5 RR* A18 RS A42 SS A53 RS A54 RR* A81 SR A83 SR A84 RR* A85 RR* A90 SS A91 SR A92 SR A97 SS A112 RR* A117 RR* A144 RS Gh13 RS CLN2116 SR Silverado SS * Ty3/Ty3, Ty2/ty2 Fuente: FODECYT
40 Se realizó un análisis de varianza a los resultados anteriores (Cuadros 10 a 12). Cuadro 10. Efecto del genotipo sobre la variación observada. Fuente Grados de Suma de Cuadrado Valor F Probabilidad libertad cuadrados medio Genotipo <.0001 Bloque Genotipo X bloque Fuente: FODECYT Cuadro 11. Contribución de cada genotipo a la variación observada No. Genotipo DSI promedio Error Probabilidad 1 RR < RS < SR < SS <.0001 Fuente: FODECYT Cuadro 12. Contribución de cada familia a la resistencia observada. No. Familia DSI promedio Error Probabilidad A. Genotipo RR 1 A < A < A < A < A < A <.0001 B. Genotipo RS 7 A < A < A < Gh C. Genotipo SR 11 A < A < A < A < A < CLN <.0001 D. Genotipo SS 17 A < A < A < Silverado <.0001 Fuente: FODECYT
41 La comparación de las variancias de cada factor indica claramente que la varianza del genotipo resulta ser la más importante (0.78), siendo la varianza de las familias (0.60) segundo en importancia. Figura 3. Índice de severidad para familias F3 de genotipos RR (Ty2/Ty2, Ty3/Ty3), RS (Ty2/Ty2, ty3/ty3), SR (ty2/ty2, Ty3/Ty3), SS (ty2/ty2, ty3/ty3) and SS2 (ty2/ty2, ty3/ty3, híbrido commercial utilizado como control susceptible). Los números indican el DSI promedio de 15 plantas en bloques al azar de tres replicaciones de cinco plantas cada una. III.1.4 Combinación de los genes Ty1, Ty3 y Ty2 El híbrido Gc143-2 (Ty1Ty3/Ty1Ty3, ty2/ty2) X B188-2 (ty1ty3/ty1ty3, Ty2/Ty2) mostro un nivel de síntomas de 2.55 a los 50 días después del transplante. De este híbrido se obtuvo 17 familias F3 provenientes de plantas F2 individuales. El genotipo de cada una de estas familias fue verificado antes del transplante para la determinación de su fenotipo de resistencia (Cuadros 13 y 14). 41
42 Cuadro 13. Genotipos para los genes Ty2 y Ty3 de plantas F3 del cruce Gc143-2 X B Familia F3 Genotipo Familia F3 Genotipo F1 Ty3/Ty3, Ty2/Ty2 RR F89 ty3/ty3, ty2/ty2 SS F3 ty3/ty3, ty2/ty2 SS F91 ty3/ty3, Ty2/Ty2 SR F8 ty3/ty3, ty2/ty2 SS F92 Ty3/Ty3, ty2/ty2 RS F9 ty3/ty3, Ty2/Ty2 SR F97 Ty3/Ty3, Ty2/Ty2 RR F19 ty3/ty3, ty2/ty2 SS F99 ty3/ty3, ty2/ty2 SS F38 Ty3/Ty3, Ty2/Ty2 RR F108 Ty3/Ty3, Ty2/Ty2 RR F52 Ty3/Ty3, ty2/ty2 RS F120 Ty3/Ty3, Ty2/Ty2 RR F55 Ty3/Ty3, ty2/ty2 RS F122 ty3/ty3, Ty2/Ty2 SR F84 ty3/ty3, Ty2/Ty2 SR Fuente: FODECYT
43 Cuadro 14. Combinación de los genes de resistencia Ty1, Ty3 y Ty2. No. Cam po Replica 1, planta número Replica 2, planta número Replica 3, planta número Famili a F3 Genotipo #1 #2 #3 #4 #5 #1 #2 #3 #4 #5 #1 #2 #3 #4 #5 21 F1 RR F3 SS F8 SS F9 SR F19 SS F38 RR F52 RS F55 RS F F89 SS F91 SR F92 RS F97 RR F99 SS F108 RR F120 RR F122 SR Gc143-2 RS B188-2 SR Fuente: FODECYT
44 Se realizó un análisis de varianza a los resultados anteriores (Cuadros 15 a 17). Cuadro 15. Efecto del genotipo sobre la variación observada. Fuente Grados de Suma de Cuadrado Valor F Probabilidad libertad cuadrados medio Genotipo <.0001 Bloque Genotipo X bloque Fuente: FODECYT Cuadro 16. Contribución de cada genotipo a la variación observada Genotipo DSI promedio Error Probabilidad RR <.0001 RS <.0001 SR <.0001 SS <.0001 Fuente: FODECYT Cuadro 17. Contribución de cada familia a la resistencia observada. No. Familia DSI promedio Error Probabilidad A. Genotipo RR 1 F < F < F < F < F <.0001 B. Genotipo RS 7 F < F F < Gc C. Genotipo SR 11 B < F < F < F <.0001 D. Genotipo SS 15 F < F < F < F < F <.0001 Fuente: FODECYT La comparación de las variancias de cada factor indica claramente que la varianza del genotipo resulta ser la más importante (1.67), pero la varianza de las familias (1.37) dentro de los genotipos también es importante. Es decir que las distintas familias tienen 44
45 un componente propio importante, posiblemente debido a otros genes modificadores de la expresión de los genes de resistencia. Figura 4. Nivel de Resistencia de familias F3 de genotipes RR (Ty2/Ty2, Ty1-Ty3/Ty1- Ty3), RS (Ty2/Ty2, ty1-ty3/ty1-ty3), SR (ty2/ty2, Ty1-Ty3/Ty1-Ty3), SS (ty2/ty2, ty1- ty3/ty1-ty3) y SS2 (control comercial susceptible). Los números indican el DSI promedio de 15 plantas en bloques al azar de tres replicaciones de cinco plantas cada una. III.1.5 Combinación de los genes Ty3a y Ty2 La línea GTc645-1, portadora del gen Ty3a, fue cruzada con la línea B188-2, portadora del gen Ty2. Se evaluarán los individuos heterocigotos (Ty3a/ty3a, Ty2/ty2), comparando su resistencia con la de los genotipos parentales. Las líneas 21-a y B188-2 también fueron cruzadas con la variedad susceptible HUJ-VF (ty3/ty3, ty2/ty2). Los individuos monohíbridos, heterocigotos Ty3a/ty3a, ty2/ty2 y ty3a/ty3a, Ty2/ty2 también serán evaluados para resistencia con relación a los genotipos parentales. El genotipo de las plantas F1 fue verificado individualmente y se transplantaron a campo para la determinación del fenotipo de resistencia. Se transplantaron los tres híbridos (21-a X B188-2, 21-a X HUJ-VF y B188-2 X HUJ-VF), las tres líneas parentales y un control susceptible en tres repeticiones de cinco plantas cada una. A los 36 días después del transplante, se evaluaron las plantas individualmente (Cuadro 18). 45
46 Cuadro 18. Combinación de los genes de resistencia Ty3a y Ty2. Replica 1, planta número Replica 2, planta número Relica 3, planta número No. Campo Híbrido/ Línea Genotipo #1 #2 #3 #4 #5 #6 #7 #1 #2 #3 #4 #5 #6 #7 #1 #2 #3 #4 #5 # a X B188-2 RR a X HUJ-VF RS a X HUJ-VF RS HUJ-VF X B188-2 SR a RS B188-2 SR Silverado SS Fuente: FODECYT Cuadro 21. Combinación de los genes de Resistencia Ty3a y Ty3. Replica 1, planta número Replica 2, planta número Relica 3, planta número No. Campo Híbrido/ Línea Genotipo #1 #2 #3 #4 #5 #6 #7 #1 #2 #3 #4 #5 #6 #1 #2 #3 #4 #5 #6 21-a X Gh13 RR a RS Gh13 SR Silverado SS Fuente: FODECYT
47 Se realizó un análisis de varianza a los resultados anteriores (Cuadros 19 y 20). Cuadro 19. Contribución de cada factor a la variación observada. Fuente Grados de Suma de Cuadrado Valor F Probabilidad libertad cuadrados medio Genotipo <.0001 Familia X <.0001 genotipo Bloque Genotipo X bloque Fuente: FODECYT Cuadro 20. Contribución de cada familia a la variación observada. No. Familia DSI promedio Error Probabilidad 1 21-a X B a < a X HUJ-VF < B < HUJ-VF X B < Silverado <.0001 Fuente: FODECYT La comparación de las variancias de cada factor indica claramente que la varianza del genotipo es la más importante (2.15), siendo la varianza dentro de las familias (1.40) segunda en importancia. 47
48 Figura 5. Nivel de desarrollo de síntomas (DSI) de los genotipos heterocigotas RR* (Ty2/ty2, Ty3a/ty3), SR* (ty2/ty2, Ty3a/ty3a), RS* (Ty2/ty2, ty3a/ty3a) y de las líneas parentales homocigotas SR (ty2/ty2, Ty3a/Ty3a), RS (Ty2/Ty2, ty3/ty3) y el control susceptible SS (ty2/ty2, ty3/ty3). III.1.6 Combinación de los genes Ty3a y Ty3 La línea 21-a (Ty3a/Ty3a) fue cruzada con la línea portadora del gen Ty3 Gh13 (Ty3/Ty3). Se evalúo la resistencia del híbrido con relación a los genotipos parentales. El genotipo de las plantas F1 fue determinado individualmente y luego, fue transplantado en el campo para la determinación de su fenotipo de resistencia. Se transplantó el híbrido (21-a X Gh13), las dos líneas parentales y un control susceptible en tres repeticiones de cinco plantas cada una. A los 36 días después del transplante, se evaluaron las plantas individualmente (Cuadro 21). 48
49 Se realizó un análisis de varianza a los resultados anteriores (Cuadros 22 y 23). Cuadro 22. Contribución de cada factor a la variación observada. Fuente Grados de Suma de Cuadrado Valor F Probabilidad libertad cuadrados medio Genotipo <.0001 Bloque Genotipo X bloque Fuente: FODECYT Cuadro 23. Contribución de cada familia a la variación observada. No. Familia DSI promedio Error Probabilidad 1 21-a X Gh a Gh Silverado <.0001 Fuente: FODECYT Figura 6. Índice de severidad de la enfermedad (DSI) de los genotipos heterocigotas RR (21-a X Gh13, Ty3a/Ty3), SR (Gc21-a,Ty3a/Ty3a), RS (Gh13,Ty3/Ty3) y SS (híbrido comercial susceptible, ty3/ty3). 49
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80
 AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
o Y Cromosomas autosómicos: 22 pares Cromosoma sexual: 1 par (X o Y)
 Cromosomas autosómicos: 22 pares o Y Cromosoma sexual: 1 par (X o Y) Un alelo es dominante cuando su presencia se manifiesta siempre en el fenotipo, y el fenotipo es igual para el homocigótico que para
Cromosomas autosómicos: 22 pares o Y Cromosoma sexual: 1 par (X o Y) Un alelo es dominante cuando su presencia se manifiesta siempre en el fenotipo, y el fenotipo es igual para el homocigótico que para
CURSO DE POSTGRADO APLICACIÓN DE LOS MARCADORES MOLECULARES
 CURSO DE POSTGRADO APLICACIÓN DE LOS MARCADORES MOLECULARES Dr. Oscar Díaz Director del Programa FITOGEN Departamento de Acuicultura y Recursos Agroalimentarios Universidad de Los Lagos SELECCIÓN ASISTIDA
CURSO DE POSTGRADO APLICACIÓN DE LOS MARCADORES MOLECULARES Dr. Oscar Díaz Director del Programa FITOGEN Departamento de Acuicultura y Recursos Agroalimentarios Universidad de Los Lagos SELECCIÓN ASISTIDA
MARCADORES GENÉTICOS
 MARCADORES GENÉTICOS MARCADORES GENÉTICOS La diferencia entre individuos se denotaba señalando diferencias en su aspecto externo (fenotipo) Marcadores Morfológicos Carácter Informativo Variación POLIMORFISMO
MARCADORES GENÉTICOS MARCADORES GENÉTICOS La diferencia entre individuos se denotaba señalando diferencias en su aspecto externo (fenotipo) Marcadores Morfológicos Carácter Informativo Variación POLIMORFISMO
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ
 REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
Uso de nuevas tecnologías para el desarrollo de variedades de arroces INIA
 Viviana Becerra Mario Paredes Gabriel Donoso Carmen Rojo Eduardo Gutiérrez Uberlinda Luengo Fernando Saavedra Uso de nuevas tecnologías para el desarrollo de variedades de arroces INIA Chile es el lugar
Viviana Becerra Mario Paredes Gabriel Donoso Carmen Rojo Eduardo Gutiérrez Uberlinda Luengo Fernando Saavedra Uso de nuevas tecnologías para el desarrollo de variedades de arroces INIA Chile es el lugar
Métodos de Biología Molecular y de Ingeniería Genética
 Métodos de Biología Molecular y de Ingeniería Genética I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II.
Métodos de Biología Molecular y de Ingeniería Genética I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II.
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
Técnicas del ADN recombinante Introducción
 Técnicas del ADN recombinante Introducción Estructura primaria del ADN Enzimas de restricción (ER) Son enzimas (endonucleasas) de origen bacteriano que reconocen y cortan secuencias específicas (4-8 pb)
Técnicas del ADN recombinante Introducción Estructura primaria del ADN Enzimas de restricción (ER) Son enzimas (endonucleasas) de origen bacteriano que reconocen y cortan secuencias específicas (4-8 pb)
(FTG-7086/07) (FTG/RF-0721-RG)
 Identificación y selección de cultivares de tomate (Lycopersicon esculentum Mill.) tolerantes al complejo de virosis transmitido por Bemisia tabaci (Genn.) (Aleyrodidae) en América Central. (FTG-7086/07)
Identificación y selección de cultivares de tomate (Lycopersicon esculentum Mill.) tolerantes al complejo de virosis transmitido por Bemisia tabaci (Genn.) (Aleyrodidae) en América Central. (FTG-7086/07)
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola 2016 Dogma central de la biología
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola 2016 Dogma central de la biología
Marcadores Bioquímicos: Isoenzimas y Proteínas de Reserva
 Marcadores morfológicos Influencia del ambiente Bajo número Caracteres de madurez Entrenamiento y subjetividad Marcadores moleculares Sin influencia ambiental Cantidad ilimitada Análisis en fases tempranas
Marcadores morfológicos Influencia del ambiente Bajo número Caracteres de madurez Entrenamiento y subjetividad Marcadores moleculares Sin influencia ambiental Cantidad ilimitada Análisis en fases tempranas
Herencia y técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TPsNº 1 y 2 Herencia y técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Dogma central
Explicación de TPsNº 1 y 2 Herencia y técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Dogma central
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
Identificación y selección de cultivares de tomate ( Lycopersicon esculentum Mill.) tolerantes al complejo de virosis transmitido por Bemisia tabaci
 Identificación y selección de cultivares de tomate (Lycopersicon esculentum Mill.) tolerantes al complejo de virosis transmitido por Bemisia tabaci (Genn.) (Aleyrodidae) en América Central. (FTG-7086/07)
Identificación y selección de cultivares de tomate (Lycopersicon esculentum Mill.) tolerantes al complejo de virosis transmitido por Bemisia tabaci (Genn.) (Aleyrodidae) en América Central. (FTG-7086/07)
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
Genética cuantitativa
 Genética cuantitativa TLC Problema básico de estadística a) Calcule la media y la varianza muestrales de las medidas de altura del siguiente grupo de individuos: X 1 : 160 cm X 2 : 170 cm X 3 : 180 cm
Genética cuantitativa TLC Problema básico de estadística a) Calcule la media y la varianza muestrales de las medidas de altura del siguiente grupo de individuos: X 1 : 160 cm X 2 : 170 cm X 3 : 180 cm
El ADN de cada individuo afectado o no, se analiza utilizando una variedad
 LOS ESTUDIOS DE LIGAMIENTO PUEDEN MAPEAR GENES DE ENFERMEDADES CON UNA RESOLUCION DE ALREDEDOR DE 1 cm (centimorgan): 1cM =una región de 7.5 x 10 5 bases, pero en ese tamaño puede haber unos 50 genes distintos!
LOS ESTUDIOS DE LIGAMIENTO PUEDEN MAPEAR GENES DE ENFERMEDADES CON UNA RESOLUCION DE ALREDEDOR DE 1 cm (centimorgan): 1cM =una región de 7.5 x 10 5 bases, pero en ese tamaño puede haber unos 50 genes distintos!
Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento
 Dra. Gabriela Lacoste- QBP 2012 MLPA Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento Es una técnica que permite la detección de cambios en el
Dra. Gabriela Lacoste- QBP 2012 MLPA Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento Es una técnica que permite la detección de cambios en el
HERRAMIENTAS MOLECULARES APLICADAS A LA INVESTIGACIÓ PHYTOPHTHORA INFESTANS EN LA ESTACIÓ
 HERRAMIENTAS MOLECULARES APLICADAS A LA INVESTIGACIÓ INVESTIGACIÓN DE PHYTOPHTHORA INFESTANS EN LA ESTACIÓ ESTACIÓN CIPCIP-QUITO El tizón tardío de la papa causado por el hongo Phytophthora infestans continua
HERRAMIENTAS MOLECULARES APLICADAS A LA INVESTIGACIÓ INVESTIGACIÓN DE PHYTOPHTHORA INFESTANS EN LA ESTACIÓ ESTACIÓN CIPCIP-QUITO El tizón tardío de la papa causado por el hongo Phytophthora infestans continua
Identificación de cultivares de tomate con resistencia a begomovirus utilizando marcadores moleculares tipo SCAR y CAPS
 Artículo científico Biotecnología Vegetal Vol. 12, No. 2: 67-76, abril - junio, 2012 ISSN 2074-8647 (Versión electrónica) Identificación de cultivares de tomate con resistencia a begomovirus utilizando
Artículo científico Biotecnología Vegetal Vol. 12, No. 2: 67-76, abril - junio, 2012 ISSN 2074-8647 (Versión electrónica) Identificación de cultivares de tomate con resistencia a begomovirus utilizando
 http://history.nih.gov/exhibits/nirenberg/images/photos/01_mendel_pu.jpg Gregor Mendel (1822-1884) fue un monje católico y naturalista, nacido en Heinzendorf, Austria (actualmente pertenece a la República
http://history.nih.gov/exhibits/nirenberg/images/photos/01_mendel_pu.jpg Gregor Mendel (1822-1884) fue un monje católico y naturalista, nacido en Heinzendorf, Austria (actualmente pertenece a la República
TECNICAS DE BIOLOGIA MOLECULAR
 Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA
 BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA PROFESORADO CURSO 1994-1995: Dr. J. Ariño (Unidad de Bioquímica) Dra. F. Bosch (Unidad de Bioquímica) Dr. A. Sánchez (Unidad de Genética y Mejora)
BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA PROFESORADO CURSO 1994-1995: Dr. J. Ariño (Unidad de Bioquímica) Dra. F. Bosch (Unidad de Bioquímica) Dr. A. Sánchez (Unidad de Genética y Mejora)
La investigación participativa en Recursos Fitogenéticos como herramienta al servicio de agricultores y agricultoras
 La investigación participativa en Recursos Fitogenéticos como herramienta al servicio de agricultores y agricultoras María José Suso Llamas Instituto de Agricultura Sostenible (CSIC) Construyendo investigación
La investigación participativa en Recursos Fitogenéticos como herramienta al servicio de agricultores y agricultoras María José Suso Llamas Instituto de Agricultura Sostenible (CSIC) Construyendo investigación
Material complementario UD 5
 Material complementario UD 5 Componentes de la PCR ADN molde que es el que se quiere replicar. Oligonucleótidos iniciadores, primers o cebadores: - Son oligonucleótidos monocatenarios cuya secuencia es
Material complementario UD 5 Componentes de la PCR ADN molde que es el que se quiere replicar. Oligonucleótidos iniciadores, primers o cebadores: - Son oligonucleótidos monocatenarios cuya secuencia es
(FTG-7086/07) (FTG/RF-0721-RG)
 Identificación y selección de cultivares de tomate (Lycopersicon esculentum Mill.) tolerantes al complejo de virosis transmitido por Bemisia tabaci (Genn.) (Aleyrodidae) en América Central. (FTG-7086/07)
Identificación y selección de cultivares de tomate (Lycopersicon esculentum Mill.) tolerantes al complejo de virosis transmitido por Bemisia tabaci (Genn.) (Aleyrodidae) en América Central. (FTG-7086/07)
3. Materiales y Métodos
 3. Materiales y Métodos El análisis de haplotipos se realizó mediante la caracterización de variaciones alélicas reportadas con anterioridad. En total de analizaron cinco marcadores polimórficos: tres
3. Materiales y Métodos El análisis de haplotipos se realizó mediante la caracterización de variaciones alélicas reportadas con anterioridad. En total de analizaron cinco marcadores polimórficos: tres
Caracteristicas del ADN. Se rompen con las siguientes condiciones -Temperaturas >90 - ph >10.5
 PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
UNIVERSIDAD NACIONAL AGRARIA LA MOLINA
 UNIVERSIDAD NACIONAL AGRARIA LA MOLINA FACULTAD DE CIENCIAS Caracterización citogenética y molecular de las especies cultivadas del género Pachyrhizus Richard ex DC. Presentado por: Mónica Lucía Santayana
UNIVERSIDAD NACIONAL AGRARIA LA MOLINA FACULTAD DE CIENCIAS Caracterización citogenética y molecular de las especies cultivadas del género Pachyrhizus Richard ex DC. Presentado por: Mónica Lucía Santayana
Test Genéticos Moleculares
 CERPO Centro de Referencia Perinatal Oriente Facultad de Medicina, Universidad de Chile Test Genéticos Moleculares Dr. Guillermo Parrao Barrera Residente Medicina Materno Fetal Pontificia Universidad Católica
CERPO Centro de Referencia Perinatal Oriente Facultad de Medicina, Universidad de Chile Test Genéticos Moleculares Dr. Guillermo Parrao Barrera Residente Medicina Materno Fetal Pontificia Universidad Católica
Métodos de Mejora en Plantas Autógamas
 Métodos de Mejora en Plantas Autógamas Objetivo de la Mejora en Autógamas Concentrar diferentes genes útiles en un mismo genotipo. Aspectos a considerar En plantas completamente autógamas no hay recombinación
Métodos de Mejora en Plantas Autógamas Objetivo de la Mejora en Autógamas Concentrar diferentes genes útiles en un mismo genotipo. Aspectos a considerar En plantas completamente autógamas no hay recombinación
Tema 28. LIGAMIENTO II
 Tema 28. LIGAMIENTO II LIGAMIENTO EN LA ESPECIE HUMANA * Marcadores polimórficos para el estudio del ligamiento. * Haplotipo familiar y cambio de fase de ligamiento. QUÉ SON LOS LOCI MARCADORES? Los loci
Tema 28. LIGAMIENTO II LIGAMIENTO EN LA ESPECIE HUMANA * Marcadores polimórficos para el estudio del ligamiento. * Haplotipo familiar y cambio de fase de ligamiento. QUÉ SON LOS LOCI MARCADORES? Los loci
INSTITUTO NICARAGÜENSE DE TECNOLOGÍA AGROPECUARIA INTA VI Región (Matagalpa-Jinotega) PROGRAMA: Investigación Agrícola SUB PROGRAMA: Hortalizas
 INSTITUTO NICARAGÜENSE DE TECNOLOGÍA AGROPECUARIA INTA VI Región (Matagalpa-Jinotega) PROGRAMA: Investigación Agrícola SUB PROGRAMA: Hortalizas Incremento de Semilla de Líneas Promisorias de Tomate Tolerantes
INSTITUTO NICARAGÜENSE DE TECNOLOGÍA AGROPECUARIA INTA VI Región (Matagalpa-Jinotega) PROGRAMA: Investigación Agrícola SUB PROGRAMA: Hortalizas Incremento de Semilla de Líneas Promisorias de Tomate Tolerantes
UNIVERSIDAD DE SAN CARLOS DE GUATEMALA FACULTAD DE AGRONOMÍA ÁREA INTEGRADA TRABAJO DE GRADUACIÓN
 UNIVERSIDAD DE SAN CARLOS DE GUATEMALA FACULTAD DE AGRONOMÍA ÁREA INTEGRADA TRABAJO DE GRADUACIÓN DESARROLLO DE UN MARCADOR MOLECULAR, BASADO EN LA TÉCNICA DE REACCIÓN EN CADENA DE LA POLIMERASA, PARA
UNIVERSIDAD DE SAN CARLOS DE GUATEMALA FACULTAD DE AGRONOMÍA ÁREA INTEGRADA TRABAJO DE GRADUACIÓN DESARROLLO DE UN MARCADOR MOLECULAR, BASADO EN LA TÉCNICA DE REACCIÓN EN CADENA DE LA POLIMERASA, PARA
GENÉTICA Y BIOTECNOLOGÍA MARINA Profesores Beatriz Camara (UTFSM) José Gallardo (PUCV) Doctorado en Biotecnología I Semestre 2015
 GENÉTICA Y BIOTECNOLOGÍA MARINA Profesores Beatriz Camara (UTFSM) José Gallardo (PUCV) Doctorado en Biotecnología I Semestre 2015 1 MARCADORES GENETICOS Debe ser polimórfico (Genetic variations occurring
GENÉTICA Y BIOTECNOLOGÍA MARINA Profesores Beatriz Camara (UTFSM) José Gallardo (PUCV) Doctorado en Biotecnología I Semestre 2015 1 MARCADORES GENETICOS Debe ser polimórfico (Genetic variations occurring
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
TEMA 3 CAMBIOS EN LA LAS POBLACIONES AGRÍCOLAS
 TEMA 3 CAMBIOS EN LA ESTRUCTURA GENÉTICA DE LAS POBLACIONES AGRÍCOLAS TEMA 2.- DIVERSIDAD GENÉTICA EN AGRICULTURA 1.- SELECCIÓN 2.- TASA DE REPRODUCCIÓN RELATIVA EL PROCESO MICROEVOLUTIVO ACTUACIÓN DE
TEMA 3 CAMBIOS EN LA ESTRUCTURA GENÉTICA DE LAS POBLACIONES AGRÍCOLAS TEMA 2.- DIVERSIDAD GENÉTICA EN AGRICULTURA 1.- SELECCIÓN 2.- TASA DE REPRODUCCIÓN RELATIVA EL PROCESO MICROEVOLUTIVO ACTUACIÓN DE
VARIACIÓN INTER-INDIVIDUAL
 Facultad de Medicina Genética 1 er Curso TEMA 2 LA GEOGRAFÍA DEL GENOMA HUMANO 2.1 Estructura del genoma humano y variación inter-individual. 2.2 El ADN repetitivo. 2.1 ESTRUCTURA DEL GENOMA HUMANO Y VARIACIÓN
Facultad de Medicina Genética 1 er Curso TEMA 2 LA GEOGRAFÍA DEL GENOMA HUMANO 2.1 Estructura del genoma humano y variación inter-individual. 2.2 El ADN repetitivo. 2.1 ESTRUCTURA DEL GENOMA HUMANO Y VARIACIÓN
Métodos de Mejora en Plantas Autógamas
 Métodos de Mejora en Plantas Autógamas Objetivo Principal de la Mejora en Autógamas Concentrar diferentes genes útiles en un mismo genotipo. Aspectos a considerar En plantas completamente autógamas no
Métodos de Mejora en Plantas Autógamas Objetivo Principal de la Mejora en Autógamas Concentrar diferentes genes útiles en un mismo genotipo. Aspectos a considerar En plantas completamente autógamas no
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Reacción en cadena de la Polimerasa, sus aplicaciones y algunos elementos de ecología molecular. Polymerase Chain Reaction (PCR)
 Reacción en cadena de la Polimerasa, sus aplicaciones y algunos elementos de ecología molecular Polymerase Chain Reaction (PCR) PCR? PCR = síntesis en progresión exponencial de secuencias objetivos (locus)
Reacción en cadena de la Polimerasa, sus aplicaciones y algunos elementos de ecología molecular Polymerase Chain Reaction (PCR) PCR? PCR = síntesis en progresión exponencial de secuencias objetivos (locus)
Tipo Tm ( O C) Tamaño (pb) Segregación
 IV. RESULTADOS Se diseñaron iniciadores i-pcr para 12 marcadores AFLP, obteniendo la secuencia del producto de amplificación en 2 casos correspondientes a marcadores AFLP mayores a 190 pb con un contenido
IV. RESULTADOS Se diseñaron iniciadores i-pcr para 12 marcadores AFLP, obteniendo la secuencia del producto de amplificación en 2 casos correspondientes a marcadores AFLP mayores a 190 pb con un contenido
Qué método o métodos de laboratorio serían los más apropiados en las siguientes situaciones diagnósticas?:
 Qué método o métodos de laboratorio serían los más apropiados en las siguientes situaciones diagnósticas?: a) Diagnóstico prenatal de un feto masculino con riesgo de presentar distrofia muscular de Duchenne
Qué método o métodos de laboratorio serían los más apropiados en las siguientes situaciones diagnósticas?: a) Diagnóstico prenatal de un feto masculino con riesgo de presentar distrofia muscular de Duchenne
La metodología de extracción de ADN permitió obtener productos de PCR de buena
 V.- RESULTADOS. La metodología de extracción de ADN permitió obtener productos de PCR de buena calidad. En la figura 2 se puede observar un ejemplo de los bandeos presentados en algunos de los organismos
V.- RESULTADOS. La metodología de extracción de ADN permitió obtener productos de PCR de buena calidad. En la figura 2 se puede observar un ejemplo de los bandeos presentados en algunos de los organismos
Tema 1. El análisis genético
 Tema 1 El análisis genético Gen Genoma Genética Genómica Genómica estructural Genómica funcionall Mapas genéticos Mapas físicos EXPRESIÓN Transcriptoma Proteoma Interactoma FUNCIÓN Genética inversa ANÁLISIS
Tema 1 El análisis genético Gen Genoma Genética Genómica Genómica estructural Genómica funcionall Mapas genéticos Mapas físicos EXPRESIÓN Transcriptoma Proteoma Interactoma FUNCIÓN Genética inversa ANÁLISIS
ABREVIATURAS... XI I. INTRODUCCIÓN EL TOMATE... 3
 ÍNDICE ABREVIATURAS... XI I. INTRODUCCIÓN... 1 1 EL TOMATE... 3 1.1 Taxonomía... 3 1.2 Características generales... 3 1.3 La flor... 4 1.4 Características del fruto... 5 2 CUAJADO Y DESARROLLO DEL FRUTO...
ÍNDICE ABREVIATURAS... XI I. INTRODUCCIÓN... 1 1 EL TOMATE... 3 1.1 Taxonomía... 3 1.2 Características generales... 3 1.3 La flor... 4 1.4 Características del fruto... 5 2 CUAJADO Y DESARROLLO DEL FRUTO...
Por qué no nacemos con cuatro ojos, por ejemplo?
 GENÉTICA Por qué no nacemos con cuatro ojos, por ejemplo? EL CANGREJO DE HEIKE Eligiendo a los descendientes por los caracteres deseados, el hombre ha modificado muchas especies, e incluso ha creado razas
GENÉTICA Por qué no nacemos con cuatro ojos, por ejemplo? EL CANGREJO DE HEIKE Eligiendo a los descendientes por los caracteres deseados, el hombre ha modificado muchas especies, e incluso ha creado razas
INGENIERÍA GENÉTICA Apuntes de biología
 INGENIERÍA GENÉTICA Son técnicas para la manipulación del genoma; entre ellas, las principales: - Técnicas de ADN recombinante - Clonación de genes - Hibridación de ácidos nucleicos Técnica del ADN Recombinante:
INGENIERÍA GENÉTICA Son técnicas para la manipulación del genoma; entre ellas, las principales: - Técnicas de ADN recombinante - Clonación de genes - Hibridación de ácidos nucleicos Técnica del ADN Recombinante:
incognita, Fusarium oxysporum y Ralstonia solanacearum UTILIZANDO MARCADORES
 EVALUACIÓN DE GENOTIPOS DE TOMATE POR SU RESISTENCIA A Meloidogyne incognita, Fusarium oxysporum y Ralstonia solanacearum UTILIZANDO MARCADORES MOLECULARES TIPO SCAR CON FINES DE MEJORAMIENTO Rafael Morales
EVALUACIÓN DE GENOTIPOS DE TOMATE POR SU RESISTENCIA A Meloidogyne incognita, Fusarium oxysporum y Ralstonia solanacearum UTILIZANDO MARCADORES MOLECULARES TIPO SCAR CON FINES DE MEJORAMIENTO Rafael Morales
Poblaciones de premejora
 Poblaciones de premejora Apellidos, nombre Departamento Centro Pérez de Castro, Ana María (anpede1@btc.upv.es) Biotecnología Escuela Técnica Superior de Ingeniería Agronómica y del Medio Natural 1 Resumen
Poblaciones de premejora Apellidos, nombre Departamento Centro Pérez de Castro, Ana María (anpede1@btc.upv.es) Biotecnología Escuela Técnica Superior de Ingeniería Agronómica y del Medio Natural 1 Resumen
Gen. Locus. El lugar donde se localiza el gen en el cromosoma.
 GENÉTICA MENDELIANA Griffiths A., Wessler S., Lewontin R., Gelbart W., Suzuki D., Miller J. (2005) Introduction to Genetic Analysis (8 th ed). W.H. Freeman and Company, New York. (también ediciones recientes)
GENÉTICA MENDELIANA Griffiths A., Wessler S., Lewontin R., Gelbart W., Suzuki D., Miller J. (2005) Introduction to Genetic Analysis (8 th ed). W.H. Freeman and Company, New York. (también ediciones recientes)
GENÉTICA. La Genética (del griego genno γεννώ= dar a luz) es la ciencia de los genes, la herencia y la variación de los organismos.
 GENÉTICA La Genética (del griego genno γεννώ= dar a luz) es la ciencia de los genes, la herencia y la variación de los organismos. La genética se subdivide en tres ramos: Clásica o mendeliana: Se preocupa
GENÉTICA La Genética (del griego genno γεννώ= dar a luz) es la ciencia de los genes, la herencia y la variación de los organismos. La genética se subdivide en tres ramos: Clásica o mendeliana: Se preocupa
Técnicas moleculares en ecología y biología de la conservación
 Técnicas moleculares en ecología y biología de la conservación Tecnología que complementa el cuerpo teórico y empírico de la ecología y evolución. No es una disciplina en sí misma (Ecología molecular).
Técnicas moleculares en ecología y biología de la conservación Tecnología que complementa el cuerpo teórico y empírico de la ecología y evolución. No es una disciplina en sí misma (Ecología molecular).
Evaluación n de la Aptitud Combinatoria General y Específica en 21 progenies
 UNIVERSIDAD CENTRAL DEL ECUADOR FACULTAD DE CIENCIAS AGRÍCOLAS Evaluación n de la Aptitud Combinatoria General y Específica en 21 progenies de papa Solanum phureja para resistencia a Tizón n tardío (Phytophthora
UNIVERSIDAD CENTRAL DEL ECUADOR FACULTAD DE CIENCIAS AGRÍCOLAS Evaluación n de la Aptitud Combinatoria General y Específica en 21 progenies de papa Solanum phureja para resistencia a Tizón n tardío (Phytophthora
TEMA 5. MENDEL Y LOS GUISANTES
 TEMA 5. MENDEL Y LOS GUISANTES Gregor Mendel (1822-1884) fue un monje católico y naturalista, nacido en Heinzendorf, Austria (actualmente pertenece a la República Checa). Se le reconoce como el padre de
TEMA 5. MENDEL Y LOS GUISANTES Gregor Mendel (1822-1884) fue un monje católico y naturalista, nacido en Heinzendorf, Austria (actualmente pertenece a la República Checa). Se le reconoce como el padre de
Electroforesis de DNA
 Electroforesis de DNA DNA Topoisomerasas Izquierda Derecha Enzimas de restricción Clasificadas en 3 tipos, donde la mayoría pertenece al grupo II (proteínas monoméricas, poseen simetria rotacional y generalmente
Electroforesis de DNA DNA Topoisomerasas Izquierda Derecha Enzimas de restricción Clasificadas en 3 tipos, donde la mayoría pertenece al grupo II (proteínas monoméricas, poseen simetria rotacional y generalmente
PCR SIMULADA. Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO
 Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
TEMA 18: Selección Asistida por Marcadores y Selección Genómica
 TEMA 18: Selección Asistida por Marcadores y Selección Genómica 1 1.- Mejora genética convencional Mejora Genética Convencional Valores Fenotípicos Pedigrí Valores Fenotípicos Selección Mejora Genética
TEMA 18: Selección Asistida por Marcadores y Selección Genómica 1 1.- Mejora genética convencional Mejora Genética Convencional Valores Fenotípicos Pedigrí Valores Fenotípicos Selección Mejora Genética
Estimación de las Frecuencias Alélicas del D16S539 en una Muestra Poblacional de ascendencia andina ancashina. Tito Tadeo, Raúl Yhossef.
 ANTECEDENTES Los estudios del genoma humano han revelado la existencia de secuencias repetitivas en tándem en muchos sitios a lo largo de todos los cromosomas, que varían en el número de veces que la unidad
ANTECEDENTES Los estudios del genoma humano han revelado la existencia de secuencias repetitivas en tándem en muchos sitios a lo largo de todos los cromosomas, que varían en el número de veces que la unidad
TEMA 5 AVANCES DE LA GENÉTICA
 TEMA 5 AVANCES DE LA GENÉTICA Y MANTENIMIENTO DE LA DIVERSIDAD TEMA 5.- AVANCES DE LA GENÉTICA Y MANTENIMIENTO DE LA DIVERSIDAD 1.- AVANCES DE LA GENÉTICA 2.- MANTENIMIENTO DE LA DIVERSIDAD GENÉTICA: MEJORA
TEMA 5 AVANCES DE LA GENÉTICA Y MANTENIMIENTO DE LA DIVERSIDAD TEMA 5.- AVANCES DE LA GENÉTICA Y MANTENIMIENTO DE LA DIVERSIDAD 1.- AVANCES DE LA GENÉTICA 2.- MANTENIMIENTO DE LA DIVERSIDAD GENÉTICA: MEJORA
TAXONOMÍA MOLECULAR. Dra. Alicia Luque CEREMIC
 TAXONOMÍA MOLECULAR Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las condiciones ambientales. PLEOMORFISMO Debido al bajo poder
TAXONOMÍA MOLECULAR Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las condiciones ambientales. PLEOMORFISMO Debido al bajo poder
TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR
 TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR Microbiología a General Área Micología Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las
TAXONOMÍA MOLECULAR TAXONOMÍA MOLECULAR Microbiología a General Área Micología Dra. Alicia Luque CEREMIC Taxonomía clásica Carecen de reproducibilidad: los caracteres fenotípicos pueden variar con las
Genética. Examen Parcial. 17 de Enero de Apellidos Nombre Firma:
 Genética. Examen Parcial. 17 de Enero de 2011. Apellidos Nombre Firma: 1/6-La enfermedad de Tay-Sachs, en su forma infantil, está determinada por el alelo recesivo no funcional de un gen autosomico (a).
Genética. Examen Parcial. 17 de Enero de 2011. Apellidos Nombre Firma: 1/6-La enfermedad de Tay-Sachs, en su forma infantil, está determinada por el alelo recesivo no funcional de un gen autosomico (a).
RESULTADOS Y DISCUSIÓN
 RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
Principales virosis en los cultivos de tomate y pimiento. Leticia Rubio
 Principales virosis en los cultivos de tomate y pimiento Leticia Rubio Las virosis son un factor limitante para los cultivos de invernadero. El control solo se logra a través de variedades resistentes
Principales virosis en los cultivos de tomate y pimiento Leticia Rubio Las virosis son un factor limitante para los cultivos de invernadero. El control solo se logra a través de variedades resistentes
11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS. Actualmente existen diversos métodos en el mercado que pueden utilizarse
 11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS Actualmente existen diversos métodos en el mercado que pueden utilizarse para el diagnóstico de rotavirus, las cuales pueden realizarse directamente a partir
11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS Actualmente existen diversos métodos en el mercado que pueden utilizarse para el diagnóstico de rotavirus, las cuales pueden realizarse directamente a partir
PLANIFICACIÓN DE ASIGNATURA
 PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
Marcadores Moleculares en Pollos. Gabriela. M. Iglesias, M.V. MSc
 Marcadores Moleculares en Pollos Gabriela. M. Iglesias, M.V. MSc Marcadores moleculares en Pollos Camperos Utilización n en el mejoramiento animal: Evaluar los marcadores moleculares asociados a caracteres
Marcadores Moleculares en Pollos Gabriela. M. Iglesias, M.V. MSc Marcadores moleculares en Pollos Camperos Utilización n en el mejoramiento animal: Evaluar los marcadores moleculares asociados a caracteres
Marcadores basados en restricción e hibridación: RFLPs (Restriction Fragment Length Polymorphisms)
 Marcadores basados en restricción e hibridación: RFLPs (Restriction Fragment Length Polymorphisms) pellidos, nombre Departamento Centro Pérez de Castro, na María (anpede1@btc.upv.es) Picó Sirvent, María
Marcadores basados en restricción e hibridación: RFLPs (Restriction Fragment Length Polymorphisms) pellidos, nombre Departamento Centro Pérez de Castro, na María (anpede1@btc.upv.es) Picó Sirvent, María
Grupo de Mejora Genética de Tomate y Melón
 XXIII Foro INIA Horticultura al aire libre Jueves 14 de mayo de 2015 FAME INNOWA 2015.Torre-Pacheco (Murcia) Grupo de Mejora Genética de Tomate y Melón Rafael Fernández Muñoz y M. Luisa Gómez-Guillamón
XXIII Foro INIA Horticultura al aire libre Jueves 14 de mayo de 2015 FAME INNOWA 2015.Torre-Pacheco (Murcia) Grupo de Mejora Genética de Tomate y Melón Rafael Fernández Muñoz y M. Luisa Gómez-Guillamón
EL PARADIGMA POBLACIONAL: la variación en el seno de las poblaciones es la materia prima de la evolución
 EL PARADIGMA POBLACIONAL: la variación en el seno de las poblaciones es la materia prima de la evolución Lo único que se trasmite a la descendencia son genes Genotipo Fenotipo Expresión génica, desarrollo
EL PARADIGMA POBLACIONAL: la variación en el seno de las poblaciones es la materia prima de la evolución Lo único que se trasmite a la descendencia son genes Genotipo Fenotipo Expresión génica, desarrollo
Selección asistida por marcadores moleculares para tolerancia al frío en el Cono Sur Latinoamericano: una estrategia para enfrentar la inestabilidad
 Selección asistida por marcadores moleculares para tolerancia al frío en el Cono Sur Latinoamericano: una estrategia para enfrentar la inestabilidad climática FTG-8009/08 Argentina Uruguay Brasil Antecedentes
Selección asistida por marcadores moleculares para tolerancia al frío en el Cono Sur Latinoamericano: una estrategia para enfrentar la inestabilidad climática FTG-8009/08 Argentina Uruguay Brasil Antecedentes
DÍA NACIONAL DEL MAÍZ
 DÍA NACIONAL DEL MAÍZ Amalio Santacruz Varela 28 de septiembre de 2015 El contexto de los maíces nativos (2014) A nivel México Superficie sembrada 7.426 millones de hectáreas En riego 1.268 millones ha
DÍA NACIONAL DEL MAÍZ Amalio Santacruz Varela 28 de septiembre de 2015 El contexto de los maíces nativos (2014) A nivel México Superficie sembrada 7.426 millones de hectáreas En riego 1.268 millones ha
MEJORAMIENTO GENÉTICO DE ALÓGAMAS. Mercedes Rivas Fitotecnia 2009
 MEJORAMIENTO GENÉTICO DE ALÓGAMAS Mercedes Rivas Fitotecnia 2009 Especies alógamas: ALFALFA, TRÉBOL BLANCO, TRÉBOL ROJO, LOTUS DACTYLIS, FALARIS, FESTUCA, RAIGRÁS GIRASOL, MAIZ, SORGO*, CENTENO, REMOLACHA
MEJORAMIENTO GENÉTICO DE ALÓGAMAS Mercedes Rivas Fitotecnia 2009 Especies alógamas: ALFALFA, TRÉBOL BLANCO, TRÉBOL ROJO, LOTUS DACTYLIS, FALARIS, FESTUCA, RAIGRÁS GIRASOL, MAIZ, SORGO*, CENTENO, REMOLACHA
Tema 1: Mendelismo. La transmisión del material hereditario. Las leyes de la Herencia. Excepciones a las leyes de Mendel
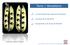 Tema 1: Mendelismo La transmisión del material hereditario Las leyes de la Herencia Excepciones a las leyes de Mendel Yolanda Loarce Tejada En 1866, Mendel, publicó los resultados de unos experimentos
Tema 1: Mendelismo La transmisión del material hereditario Las leyes de la Herencia Excepciones a las leyes de Mendel Yolanda Loarce Tejada En 1866, Mendel, publicó los resultados de unos experimentos
Alelo. Locus. La forma alternativa de un gen. El lugar donde se localiza el gen en el cromosoma.
 GENÉTICA MENDELIANA Griffiths A., Wessler S., Lewontin R., Gelbart W., Suzuki D., Miller J. (2005) Introduction to Genetic Analysis (8 th ed). W.H. Freeman and Company, New York. QH430/I59/2005 Strachan
GENÉTICA MENDELIANA Griffiths A., Wessler S., Lewontin R., Gelbart W., Suzuki D., Miller J. (2005) Introduction to Genetic Analysis (8 th ed). W.H. Freeman and Company, New York. QH430/I59/2005 Strachan
Ensayos de restricción
 Ensayos de restricción Vector con inserto= Vector recombinante 6500pb Objetivos de ensayos de restricción de plásmidos Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa.
Ensayos de restricción Vector con inserto= Vector recombinante 6500pb Objetivos de ensayos de restricción de plásmidos Conocer el principio de separación y detección de ácidos nucleicos en geles de agarosa.
BIOLOGIA MOLECULAR. Dra. Ma. Laura Tondo
 TÉCNICAS BASADAS EN HIBRIDACIÓN MOLECULAR BIOLOGIA MOLECULAR 2016 Dra. Ma. Laura Tondo DESNATURALIZACION Y RENATURALIZACION DEL DNA Cómo se mantienen unidas las dos hebras del DNA? - Puentes de hidrógeno
TÉCNICAS BASADAS EN HIBRIDACIÓN MOLECULAR BIOLOGIA MOLECULAR 2016 Dra. Ma. Laura Tondo DESNATURALIZACION Y RENATURALIZACION DEL DNA Cómo se mantienen unidas las dos hebras del DNA? - Puentes de hidrógeno
FONDO REGIONAL DE TECNOLOGÍA AGROPECUARIA Secretaría Técnica Administrativa INFORME DE SEGUIMIENTO TÉCNICO ANUAL DE LA EJECUCIÓN DEL PROYECTO
 INFORME DE SEGUIMIENTO TÉCNICO ANUAL DE LA EJECUCIÓN DEL PROYECTO Identificación y selección de cultivares de tomate (Lycopersicum esculentum Mill), tolerantes al complejo de virosis transmitido por Bemisia
INFORME DE SEGUIMIENTO TÉCNICO ANUAL DE LA EJECUCIÓN DEL PROYECTO Identificación y selección de cultivares de tomate (Lycopersicum esculentum Mill), tolerantes al complejo de virosis transmitido por Bemisia
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón
 RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos
 Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Qué estudia la genética? Qué importancia tiene la Genética Molecular para la arqueología?
 Qué estudia la genética? Qué importancia tiene la Genética Molecular para la arqueología? Todos los seres vivos somos herederos de un bien fundamental, el material genético, que se transmite de generación
Qué estudia la genética? Qué importancia tiene la Genética Molecular para la arqueología? Todos los seres vivos somos herederos de un bien fundamental, el material genético, que se transmite de generación
Cursos de SANIDAD. Principios de Biología Molecular A distancia 80 h
 Cursos de SANIDAD [ Principios de Biología Molecular ] A distancia 80 h PRINCIPIOS DE BIOLOGÍA MOLECULAR El curso de Principios de biología molecular da a conocer a los participantes la estructura y propiedades
Cursos de SANIDAD [ Principios de Biología Molecular ] A distancia 80 h PRINCIPIOS DE BIOLOGÍA MOLECULAR El curso de Principios de biología molecular da a conocer a los participantes la estructura y propiedades
Investigación en genes
 Investigación en genes Conocer el genóma de los organismos Modificar la dotación génica natural Tecnología a del ADN recombinante Conocimientos básicos que posibilitaron el desarrollo de la Tecnología
Investigación en genes Conocer el genóma de los organismos Modificar la dotación génica natural Tecnología a del ADN recombinante Conocimientos básicos que posibilitaron el desarrollo de la Tecnología
1. Lea el siguiente texto sobre un término básico de la genética.
 1. Lea el siguiente texto sobre un término básico de la genética. 2. El siguiente texto es una afirmación sobre un término relacionado con la genética. Uno de los genes para un rasgo determinado, que tiene
1. Lea el siguiente texto sobre un término básico de la genética. 2. El siguiente texto es una afirmación sobre un término relacionado con la genética. Uno de los genes para un rasgo determinado, que tiene
Evolución y Mejoramiento
 Domesticación y mejoramiento de los cultivos 1 Evolución y Mejoramiento Selección natural: Reproducción o supervivencia diferencial de individuos fenotípicamente diferentes 2008 2 Origen y evolución de
Domesticación y mejoramiento de los cultivos 1 Evolución y Mejoramiento Selección natural: Reproducción o supervivencia diferencial de individuos fenotípicamente diferentes 2008 2 Origen y evolución de
Región codificante. eucarionte
 Gen La unidad hereditaria básica. Por definición molecular, una secuencia de DNA necesaria para brindar un producto funcional a la célula: proteína o RNA. Región codificante eucarionte Alelo La forma alternativa
Gen La unidad hereditaria básica. Por definición molecular, una secuencia de DNA necesaria para brindar un producto funcional a la célula: proteína o RNA. Región codificante eucarionte Alelo La forma alternativa
BIOTECNOLOGÍA MOLECULAR UNIDAD N 2: Marcadores Moleculares de ADN
 BIOTECNOLOGÍA MOLECULAR UNIDAD N 2: Marcadores Moleculares de ADN Lic. Gustavo Bich Dra. María Mercedes Tiscornia. Cátedra de Biotecnología Molecular. 2017. INSTITUTO DE BIOTECNOLOGÍA MISIONES MARÍA EBE
BIOTECNOLOGÍA MOLECULAR UNIDAD N 2: Marcadores Moleculares de ADN Lic. Gustavo Bich Dra. María Mercedes Tiscornia. Cátedra de Biotecnología Molecular. 2017. INSTITUTO DE BIOTECNOLOGÍA MISIONES MARÍA EBE
Universidad de Los Andes Facultad de Medicina Instituto Inmunología Clínica Maestría en Inmunología
 TÉCNICAS EN BIOLOGÍA MOLECULAR RESPONSABLE: Dra. Lisbeth Berrueta. CREDITOS: 2 El curso de Técnicas de Biología Molecular está destinado a profesionales del área biomédica con conocimientos básicos relativos
TÉCNICAS EN BIOLOGÍA MOLECULAR RESPONSABLE: Dra. Lisbeth Berrueta. CREDITOS: 2 El curso de Técnicas de Biología Molecular está destinado a profesionales del área biomédica con conocimientos básicos relativos
Cursos de SANIDAD. Principios de Biología Molecular A distancia 80 h
 Cursos de SANIDAD [ Principios de Biología Molecular ] A distancia 80 h PRINCIPIOS DE BIOLOGÍA MOLECULAR El curso de Principios de biología molecular da a conocer a los participantes la estructura y propiedades
Cursos de SANIDAD [ Principios de Biología Molecular ] A distancia 80 h PRINCIPIOS DE BIOLOGÍA MOLECULAR El curso de Principios de biología molecular da a conocer a los participantes la estructura y propiedades
CURSO ACADÉMICO 2009 2010
 TITULACIÓN: BIOLOGÍA CURSO ACADÉMICO 2009 2010 GENÉTICA APLICADA CÓDIGO: 200810419 Departamento de adscripción: Parasitología, Ecología y Genética Área de conocimiento: Genética Ciclo: 2º Curso: 4º Tipo:
TITULACIÓN: BIOLOGÍA CURSO ACADÉMICO 2009 2010 GENÉTICA APLICADA CÓDIGO: 200810419 Departamento de adscripción: Parasitología, Ecología y Genética Área de conocimiento: Genética Ciclo: 2º Curso: 4º Tipo:
Pregunta PSU, Demre Modelo de admisión 2018
 Pregunta PSU, Demre Modelo de admisión 2018 La ley de la segregación de los caracteres de Mendel es una ley porque A) debe ser sometida a prueba cada vez que se hagan cruzamientos entre individuos que
Pregunta PSU, Demre Modelo de admisión 2018 La ley de la segregación de los caracteres de Mendel es una ley porque A) debe ser sometida a prueba cada vez que se hagan cruzamientos entre individuos que
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP
 SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
TALLER (Evaluación 20 de abril). C E R T A A P R I N C I P I O D E S E G R E G A C I O N I
 I. SOPA DE LETRAS TALLER (Evaluación 20 de abril). C E R T A A P R I N C I P I O D E S E G R E G A C I O N I A R D C Z F J Ñ V H I R A J V U B W L G B A C O R P I X P C D G N Z Q E H U L M F A F J A C
I. SOPA DE LETRAS TALLER (Evaluación 20 de abril). C E R T A A P R I N C I P I O D E S E G R E G A C I O N I A R D C Z F J Ñ V H I R A J V U B W L G B A C O R P I X P C D G N Z Q E H U L M F A F J A C
Genética (Caps )
 Genética (Caps. 12-14) Genética clásica o mendeliana (Capítulo 12 Laboratorio 9) Genética molecular (Cap. 13) Biotecnología vegetal (Cap. 14) Parcial 2, BIOL 3300, etc. 12: Genética Estudio de la herencia
Genética (Caps. 12-14) Genética clásica o mendeliana (Capítulo 12 Laboratorio 9) Genética molecular (Cap. 13) Biotecnología vegetal (Cap. 14) Parcial 2, BIOL 3300, etc. 12: Genética Estudio de la herencia
