La titina en el diagnóstico genético de cardiopatías familiares
|
|
|
- Pedro Blanco Juárez
- hace 7 años
- Vistas:
Transcripción
1 MÁSTER EN BIOTECNOLOGÍA AVANZADA ESPECIALIDAD SANITARIA La titina en el diagnóstico genético de cardiopatías familiares 21/01/2013 Iria Gómez Díaz Empresa: Health in code S.L. Tutor: Dr. Lorenzo Monserrat Iglesias
2 CONTENIDO 1. Introducción 1.1. La empresa y su campo de trabajo pág Titina. La proteína gigante pág Objetivos pág Material y métodos 3.1. Fase bioinfomática pág Fase de laboratorio pág Resultados y discusión pág Conclusiones pág Referencias bibliográficas pág. 28 La titina en el diagnóstico genético de cardiopatías familiares Página 2
3 1. INTRODUCCIÓN 1.1. La empresa y su campo de trabajo Health In Code (HIC) es una empresa de base biotecnológica, que surge como una spin off de la Universidad de A Coruña y que está situada en el edificio de O Fortín del Hospital Marítimo de Oza. Está especializada en el diagnóstico genético de enfermedades cardiovasculares y en la interpretación de los resultados, para así ofrecer a los clínicos la oportunidad de realizar una medicina individualizada para cada paciente. Su principal área de trabajo dentro de las enfermedades cardiovasculares son las cardiopatías familiares, que engloban la mayor parte de causas de muerte súbita en individuos jóvenes y deportistas, aunque también son causa importante en pacientes de mayor edad. Estas cardiopatías comparten una serie de características comunes: - Tienen una base genética y una presentación familiar, es decir, si un miembro de la familia padece la enfermedad existe la posibilidad de que el resto de familiares también estén afectados. - La muerte súbita puede ser la primera manifestación de estas enfermedades. Sin embargo, si existen síntomas previos suelen ser muy heterogéneos y su evolución difícil de predecir, ya que existen cientos de mutaciones descritas en genes que están asociadas con estas enfermedades. Por ello es fundamental realizar un diagnóstico genético que permita individualizar tanto el diagnóstico, como el pronóstico, el tratamiento de la enfermedad y la toma de medidas eficaces de prevención. Las cardiopatías familiares incluyen: - Miocardiopatías.- Enfermedades que afectan al miocardio, como por ejemplo: hipertrófica, dilatada, no compactada, restrictiva, a) La Miocardiopatía Hipertrófica (MHC) es una enfermedad hereditaria del músculo cardíaco que se caracteriza por la presencia de un engrosamiento anormal de las paredes del corazón sin causa aparente. Es la causa más frecuente de muerte súbita. La titina en el diagnóstico genético de cardiopatías familiares Página 3
4 Imagen 1. Corazón sano frente a corazón enfermo MHC. b) La Miocardiopatía Dilatada (MCD) se define por la presencia de dilatación y disfunción sistólica ventricular izquierda en ausencia de condiciones anormales de sobrecarga. Afecta a 1 de cada 3000 individuos y representa la tercera causa más común de fallo cardíaco, siendo la primera causa de trasplante. Imagen 2. Corazón sano frente a corazón enfermo MCD. - Canalopatías.- Enfermedades que afectan a los canales del corazón, responsables de la transmisión de los impulsos eléctricos, como por ejemplo: Síndrome de QT largo Síndrome de QT corto Síndrome de Brugada - Otros síndromes de afección vascular que también se caracterizan por tener una base genética y una presentación familiar, como por ejemplo el síndrome de Marfan. La titina en el diagnóstico genético de cardiopatías familiares Página 4
5 Por lo tanto, Health In Code estudia los genes que codifican las proteínas implicadas en la función del músculo cardíaco en busca de las mutaciones responsables de enfermedades cardiacas hereditarias Titina. La proteína gigante. La titina es la proteína más grande encontrada en mamíferos (varía entre 2970 y 4200 kd según la isoforma), codificada por un único gen que está ubicado en el brazo largo del cromosoma 2 (región 2q31), constituida por 368 exones y 7 isoformas (según el NCBI, 11 isoformas según Uniprot). Se localiza tanto en el músculo cardíaco como en el esquelético y, estructuralmente, se sitúa en el interior de los sarcómeros donde se une e interactúa con otras proteínas importantes, principalmente con la actina y miosina. Imagen 3. Localización de la titina. - Estructura Se trata de una proteína estructuralmente compleja, con cerca de 1 μm de largo, que se extiende desde la línea Z hasta la línea M del sarcómero. De este modo, la titina abarca 4 La titina en el diagnóstico genético de cardiopatías familiares Página 5
6 zonas distintas del sarcómero, lo que conlleva a dividirla en 4 regiones que se corresponden con la línea M, la banda A, la banda I y la línea Z. La región de la línea Z contiene el dominio amino-terminal y dominios kinasa, mientras que en la región de la línea M está ubicado el dominio carboxilo-terminal y regiones de secuencia única. La región de la banda A contiene la mayor parte de la molécula de titina que, en esta zona, se encuentra formada por dominios semejantes a la fibronectina-3 (llamados Fn3) y a la inmunoglobulina (llamados dominios Ig), además representa un componente integrante del filamento grueso, por lo tanto, esta región no es extensible y su constitución es semejante en las diferentes isoformas de la molécula. Por el contrario, la región de la banda I es muy extensible, presentando capacidad de alargarse o enrollarse, funcionando así como una unión elástica entre el filamento grueso y la línea Z. Estructuralmente, la región de la banda I es la más compleja ya que está constituida por dominios Ig, dominios N2 y segmentos PEVK (ricos en prolina (P), glutamato (E), valina (V) y lisina (K). Imagen 4. Estructura de la titina. Catro-Ferreira et al. The role of titin in the modulation of cardiac function and its pathophysiological implications Arq Bras Cardiol 2011;96: Isoformas En el corazón adulto, de las 7 isoformas que puede presentar esta macromolécula, sólo se expresan dos de ellas, N2B y N2BA, de las cuales la más común es N2B. La diferencia entre ambas isoformas reside en la banda I, concretamente en el dominio N2. La titina en el diagnóstico genético de cardiopatías familiares Página 6
7 La isoforma N2B contiene en el dominio N2 el elemento N2B, este elemento está constituido por una región con 3 dominios Ig y una región única de 572 residuos. Sin embargo, la isoforma N2BA está formada por un elemento N2B junto con uno N2A, este último está compuesto por una región con 4 dominios Ig y una región única de 106 residuos. Imagen 6. Diferencias entre la isoforma N2B y N2BA. Martín LeWinter et al. Cardiac titin: A multifunctional giant. Circulation 2010, 121: Cómo funciona? Inicialmente, la región extensible de la titina, la banda I, se encuentra en su estado acortado mientras el sarcómero está en fase de reposo. Sin embargo, a medida que se va estirando el sarcómero también lo hace junto a él la titina, generando así una fuerza en la proteína que se conoce como fuerza pasiva, y que tenderá al reposo. A continuación, el sarcómero se acorta por debajo del tamaño que presenta en estado de reposo, lo que provoca que el filamento grueso entre en contacto con la región de la línea Z. Esta situación crea en la titina otra fuerza, conocida como fuerza de restauración, que provoca que la región de la banda I se extienda ahora en dirección opuesta. La titina en el diagnóstico genético de cardiopatías familiares Página 7
8 Imagen 5. Mecanismo de generación de fuerza pasiva y de restauración. Henk L. Granzier et al. The giant protein titin: A major player in myocardial mechanics, signaling, and disease Circulation Research 2004;94: La isoforma N2BA al poseer un elemento más en la región de la banda I es más extensible y menos rígida que N2B. De este modo, los cardiomiocitos que expresan mayores cantidades de la isoforma N2B son más rígidos que aquellos que expresan una mayor proporción relativa de la isoforma N2BA. Esto conlleva que los cardiomiocitos con un elevado grado de expresión de N2B no sólo ejercen una mayor tensión pasiva durante el estiramiento, sino que también generan una mayor fuerza para recuperar el largo de reposo después de la contracción y que el sarcómero vuelve al reposo. De ese modo, en los ventrículos con elevadas proporciones de N2B se acorta la duración de la fase inicial de diástole permitiendo la formación de un fenómeno de succión ventricular que provoca el aumento del llenado ventricular, lo que puede ser importante en las situaciones de ejercicio físico u otras situaciones de taquicardia. La propiedad elástica además de permitir la regulación del largo del sarcómero que influye en la función cardíaca diastólica, también interviene en la función sistólica por el efecto que ejerce en la modulación de la relación de Frank-Starling. Según la ley de Frank- Starling el corazón posee una capacidad intrínseca de adaptarse a volúmenes crecientes de sangre, es decir, cuanto más se llena de sangre un ventrículo durante la diástole, mayor será el volumen de sangre expulsado en la siguiente contracción sistólica ya que la fuerza de La titina en el diagnóstico genético de cardiopatías familiares Página 8
9 contracción aumentará. La titina, como determinante de la rigidez ventricular y de las propiedades pasivas del ventrículo, influye en la relación presión-volumen telediastólica. Entre sus diversas acciones también destaca su papel como sensor biomecánico, dada su capacidad de distenderse y acortarse, es sensible al grado de estiramiento del cardiomiocito, siendo capaz después de transmitir esa señal biomecánica por la unión e interacción que establece con varias proteínas estructurales y señalizadoras, por lo tanto, está implicada en mecanismos de señalización intracelular. Además la titina mantiene también la estructura del sarcómero permitiendo el correcto alineamiento de los miofilamentos de actina y miosina, así la pérdida de la titina origina una organización anormal del sarcómero. A la par, comienza a investigarse la implicación de la titina en el proceso de doblaje de proteínas, regulación de la expresión de genes y regulación de varios canales iónicos. - Regulación Por otro lado, las funciones de la titina pueden ser moduladas por alteraciones a largo plazo, mediante la modificación de la proporción relativa entre las dos isoforma, o a corto plazo, por alteraciones post translacionales, como la alteración del estado de fosforilación de la titina o de su unión al ión calcio. Tales alteraciones le permiten al miocardio modificar su elasticidad en razón de las diversas alteraciones hemodinámicas a que está sujeto. - Implicación en enfermedades cardíacas Igualmente, la titina por medio de la modificación de la proporción de sus isoformas y/o de su estado de fosforilación, parece ser un elemento clave en la fisiopatología de varias enfermedades cardíacas como la cardiomiopatía dilatada (CMD), cardiomiopatía hipertrófica (CHM) y la insuficiencia cardíaca diastólica (ICD). Se ha demostrado que enfermos con ICD presentan una alteración en la proporción relativa de las isoformas de titina, con aumento de la isoforma N2B (más rígida) lo que parece explicar el aumento de la rigidez ventricular observado en esos enfermos. De tal manera que, mientras que en un corazón humano sano la proporción N2BA:N2B normal es aproximadamente de 30:70, en los enfermos con ICD esa relación era de 17:83. También La titina en el diagnóstico genético de cardiopatías familiares Página 9
10 se demostró que la hipofosforilación del segmento N2B parece contribuir significativamente al aumento de la tensión pasiva verificada en los casos de ICD. Además en enfermos con cardiomiopatía dilatada se observó una alteración de la relación entre las isoformas de la titina, con un aumento relativo de la isoforma más distensible, la N2BA. Es también en la cardiomiopatía dilatada o hipertrófica donde parece ocurrir alteraciones en la fosforilación de la titina. Con todo ello, la titina parece ser un potencial blanco de manipulación farmacológica constituyendo así un nuevo blanco terapéutico. 2. OBJETIVOS La finalidad de un buen diagnóstico genético de cardiopatías familiares consiste en identificar la enfermedad, ayudar al pronóstico clínico del paciente e identificar, en el caso de que existan, a otros familiares afectados y, de este modo, tomar medidas eficaces de prevención. El método actual validado para el diagnóstico genético de pacientes es la secuenciación automática de Sanger. Sin embargo, las pruebas genéticas que se obtienen por Sanger presentan problemas de rendimiento, automatización y rentabilidad, especialmente al tratarse de enfermedades multigénicas como las cardiopatías. Esto provoca que en un diagnóstico clínico rutinario sólo se evalúen los genes que están habitualmente afectados por la enfermedad, lo que limita el significado de los resultados. Por el contrario, las nuevas técnicas de ultrasecuenciación o NGS se presentan como una estrategia que ha ido abaratando costes y aumentando la capacidad y velocidad de secuenciación, asimismo permite realizar pruebas con todos los genes conocidos implicados en la enfermedad para establecer una mejor predicción de los resultados. La titina en el diagnóstico genético de cardiopatías familiares Página 10
11 Existen numerosas publicaciones que apoyan y describen la importancia de NGS e incluso su aplicación en miocardiopatías, sin embargo las técnicas de NGS todavía no están validadas para emitir diagnósticos clínicos concluyentes. Por este motivo se siguen empleando las técnicas de secuenciación por Sanger para confirmar los hallazgos obtenidos con NGS. En este proyecto, se pretende confirmar por Sanger mutaciones que se han detectado por NGS en la proteína de la titina. En Health In Code hasta este momento no se habían diagnosticado mutaciones en dicha proteína y, por lo tanto, requiere de su puesta a punto en el laboratorio. 3. MATERIAL Y MÉTODOS 3.1. Fase bioinformática Se introducen en la base de datos HIC mutaciones de Health In Code las 27 variantes encontradas por NGS que se quieren confirmar por Sanger (Tabla 1). De estas variantes sólo se tiene la posición a nivel cromosómico, con el fin de poder agilizar el trabajo, se convierten estas posiciones para obtener la posición a nivel proteico y genómico de laboratorio (Tabla 2). Muestra P. cr. Inicial P. cr. Final Referencia Observada C G G A G C TTC ATCTGC TGT T A ATCTGAAGG CTGAAGGCT G A La titina en el diagnóstico genético de cardiopatías familiares Página 11
12 C A A G C T C G TTC T C C T C G A G T C G A C T G C C T T C C T C T Tabla 1. Variantes que se han encontrado en las diferentes muestras por NGS. P.cr. inicial hace referencia a la posición cromosómica donde empieza la variación observada y P.cr. final a la posición cromosómica donde finaliza. Referencia refleja el nucleótido/s que hay en la posición indicada en la secuencia de referencia. Observada muestra el nucleótido/s que hay en la muestra. Muestra P. cr. Inicial P. cr. Final P. genómica de laboratorio Cambio aa g g>c p.a25686p g.61345c>t p.r5441x g c>g p.p24914r g _129754delgaa p.10170_10170del g _176215delgcagat p.5547_5548del g _235890delaca p.15801_15801del g.27390a>t p.d1186v g _272136insccttcagat p.p24711delinspsdp g _272125insagccttcag p.k24707delinskpse g c>t p.r8985c g.87601g>t p.r6646i g t>c p.s13302p g.48248g>a Intrónica g g>c p.a7807p g _129754delgaa p.10170_10170del g a>g p.e14985g g g>a p.e11694k La titina en el diagnóstico genético de cardiopatías familiares Página 12
13 g g>c p.e10289q g t>c Intrónica g a>g p.n21581s g.28390c>t p.r1304c g g>a p.r26175q g c>g p.t8910s g g>a p.r20744q g a>g p.e26299g g g>a p.r24899h g g>a p.e11694k Tabla 2. Posición de las variantes que se han encontrado en las diferentes muestras por NGS. P.cr. inicial hace referencia a la posición cromosómica donde empieza la variación observada y P.cr. final a la posición cromosómica donde finaliza. P. genómica de laboratorio la posición genómica de la variante junto al cambio nucleotídico. Cambio aa. muestra el cambio aminoacídico, en los casos en los que se hace la anotación intrónica indica que el cambio no ha tenido lugar dentro del exón y, por lo tanto, no hay cambio aminoacídico, pero se estudiará igualmente porque puede afectar al splicing de la proteína. Se descarga del NCBI ( la secuencia completa de titina y se localiza a qué exón pertenece cada variante para, posteriormente, diseñar los primers correspondientes. Existen algunos exones que debido a su gran tamaño no se van a poder amplificar, por lo tanto, en estos casos se dividen en amplicones de 500 pb aproximadamente y se le asigna a cada fragmento del exón, en función de su posición, una letra. Se consideran amplicones de 500pb porque lo máximo que es capaz de discriminar correctamente el secuenciador es 700pb aproximadamente. Asimismo, se observa que hay dos variantes que están repetidas en dos muestras diferentes, por lo tanto, se diseñarán finalmente 25 primers. Se diseñan los primers para cada variante, mediante el programa Primer Express v2.0, del siguiente modo: los primers deben amplificar el exón ± 20 nucleótidos flanqueantes del intrón que se considera que afectan al splicing de la proteína. la longitud de cada primer debe de encontrarse entre 19-24pb. la Tm no debe de ser muy baja y debe de ser similar entre ambos primers, con una diferencia máxima de 5ºC. el contenido en C/G debe de situarse en torno al 45-60%. se debe tratar de eludir las zonas repetitivas. La titina en el diagnóstico genético de cardiopatías familiares Página 13
14 se debe de evitar la formación de una horquilla en el extremo 3 para que el primer no autoanille. también se debe de evitar que se formen más de 3 enlaces C/G entre cada pareja de primers para que no autoanillen entre sí. Imagen 7. Diseño de los primers del exón 97 de N2B. Una vez finalizado el diseño, se confirma que los primers son específicos y que no van amplificar con otras zonas del genoma, para ello se emplea un programa de búsqueda de similitud de secuencias como BLAT de Genome Browser ( Al mismo tiempo se emplea la herramienta PCR in silico para comprobar el tamaño del amplicón, este dato será necesario a la hora de realizar electroforesis. Imagen 8. BLAT de los primers Fw y Rv del exón 97 de N2B. La titina en el diagnóstico genético de cardiopatías familiares Página 14
15 Imagen 9. PCR in silico del exón 97 de N2B. Se introduce en una tabla de la base de datos el nombre de cada uno de los exones o fragmentos para los que se han diseñado los primers, el tamaño del amplicón, la posición genómica dónde empieza y termina cada exón/fragmento, así como la posición dónde empieza el exón menos 20 pb y la posición dónde termina cada exón más 20 pb (esto último sólo se realizará en caso de que no se dividiera el exón en fragmentos). A continuación se crean los test de PCR y secuencia para cada uno de los exones/fragmentos, se activan y se hacen simulaciones, de esta manera cuando se optimicen los primers se podrán analizar las muestras con total normalidad Fase de laboratorio Una vez se reciben en el laboratorio los primers que se han diseñado, se resuspenden a una concentración de 100µM y se preparan diluciones a 5µM para PCR y reacciones de secuencia. En el caso de las diluciones de PCR se incluyen en una mix los primers forward y reverse, de tal forma que se añade en un eppendorf 180µl de H 2 O miliq, 10µl de forward y 10µl de reverse. Sin embargo, para las reacciones de secuencia se preparan 2 eppendorfs distintos, uno para el primer forward y otro para el reverse, de tal modo que, se añade en un eppendorf 95µl de H 2 O miliq y 5µl de forward o de reverse. La titina en el diagnóstico genético de cardiopatías familiares Página 15
16 Para poner a punto los primers de PCR, se utilizan 3 muestras de ADN control del laboratorio y una vez que se conozcan las condiciones óptimas se repiten dichas PCR con los ADN problema. Con dicho fin, se prepara una mix por cada primer, que incluye los siguientes volúmenes por pocillo: 6,25µl de buffer + enzima ( Kapa2G Ready mix de Kapa Biosystems), 1,5µl del mix de primers, 1µl de solución Q y 2,25µl de H 2 Od autoclavada. Se dispensan las mixes en una placa de 96 pocillos de Bio-Rad y después se le añade a cada pocillo 1,5µl del ADN correspondiente, manteniendo siempre para cada primer un control negativo, que incluirá 1,5µl de H 2 Od autoclavada adicional. Se pone en un termociclador Veriti 96-well de Applied Biosystems que nos permite seleccionar una Ta diferente cada dos columnas, de este modo, en una sola placa se hacen todas las PCRs. Las Ta ( C) de cada primer, así como el tamaño del amplicón (pb), son las que se muestran en la tabla: Nombre del exón/fragmento Ta ( C) Tamaño (pb) TTN-Ex108-N2B TTN-Ex132E-N2B TTN-Ex143-N2A TTN-Ex146-N2B TTN-Ex147-N2A TTN-Ex154E-N2B TTN-Ex154H-N2B TTN-Ex154J-N2B TTN-Ex154L-N2B TTN-Ex162-N2B TTN-Ex166-N2B TTN-Ex177-N2B TTN-Ex186B-N2B TTN-Ex186C-N2B TTN-Ex186H-N2B TTN-Ex186K-N2B TTN-Ex21-N2B TTN-Ex23-N2B TTN-Ex43-N2B TTN-Ex46M-Novex TTN-Ex46P-Novex La titina en el diagnóstico genético de cardiopatías familiares Página 16
17 TTN-Ex65-N2B TTN-Ex79-N2A TTN-Ex86-N2B TTN-Ex97-N2B Tabla 3. Listado de primers a optimizar. El programa que se utiliza en el termociclador Veriti 96-well de Applied Biosystems para esta enzima es el que recomienda la casa comercial Kapa Biosystems: Programa PCR Desnaturalización inicial Desnaturalización Anillamiento Extensión Extensión final Temperatura ( C) 95 C 1 minuto 95 C 10 segundos 35 ciclos Ta 10 segundos 72 C 1 minutos 72 C 30 segundos Tabla 4. Programa del termociclador para PCR. Una vez que el termociclador finaliza el programa, se realiza una electroforesis con los productos de PCR. Para ello, se prepara un gel de agarosa al 2% en el que se carga en un pocillo 3µl de marcador de peso molecular y en los demás pocillos la mezcla de 7,5µl de tampón de carga + 2µl de producto de PCR. Se corre durante 30 minutos a 300V y 150mA. Posteriormente se revelan las fotos del gel y se comprueba que los amplicones tengan el tamaño indicado en la Tabla 3. Se observa que existen 2 fragmentos (TTN-Ex65-N2B y TTN-Ex186C-N2B) en los que se observa una estela detrás de la banda y 3 fragmentos (TTN-Ex186H-N2B, TTN- Ex143-N2A y TTN-Ex154J-N2B) que presentan unas bandas muy tenues. En el caso de los 2 primeros fragmentos se repite la electroforesis y se deja correr 40 minutos a 300V y 150mA. Por otro lado, para los 3 últimos fragmentos indicados, se comprueba que las bandas se observan nítidas cambiando las condiciones de la mix de PCR: 6,25µl de buffer + enzima ( Kapa2G Ready mix de Kapa Biosystems), 2µl del mix de primers, 1µl de solución Q y 1,75µl de H 2 Od autoclavada. La titina en el diagnóstico genético de cardiopatías familiares Página 17
18 Una vez que se ha visto que todas las bandas de los productos de PCR son correctas, se introducen en la base de datos las condiciones de las mixes utilizadas así como las Ta a las que se ha programado el termociclador. Con el fin de eliminar los primers y dnts sobrantes, y así purificar los productos de PCR, se prepara en un eppendorf la siguiente mix (los volúmenes que se señalan son los correspondientes a un pocillo): 0,28µl de Exonucleasa I de Thermo scientific, 0,55µl de Fastap de Thermo scientific y 2,2µl H 2 Od autoclavada. Se dispensan 3µl de la mezcla en cada pocillo de la placa de Bio-Rad, y 1,4µl de cada producto de PCR en un pocillo diferente. Se pone en un termociclador Veriti 96-well de Applied Biosystems con el siguiente programa: Programa purificación Desfosforilación rápida Inactivación completa Temperatura ( C) 37 C 15 minutos 85 C 15 minutos Tabla 5. Programa del termociclador para la purificación de PCR. A continuación, para preparar la reacción de secuencia se mezcla en un eppendorf (los volúmenes que se señalan son los correspondientes a un pocillo): 0,25µl de BDTs (BigDye Terminator Cycle Sequencing Kit de Applied Biosystems), 1,75µl de buffer de secuencia (BigDye Terminator Cycle Sequencing Kit de Applied Biosystems) y 6,25µl H 2 Od autoclavada. En una placa de 96 pocillos de Bio-Rad se dispensan 8,2 µl por pocillo de la mix, seguidamente se añade 1µl de primer forward o 1µl de primer reverse según corresponda en cada pocillo y finalmente 1µl del producto de PCR purificado. Se pone en un termociclador Veriti 96-well de Applied Biosystems con el siguiente programa La titina en el diagnóstico genético de cardiopatías familiares Página 18
19 Programa secuencia Desnaturalización inicial Desnaturalización Anillamiento Extensión Temperatura ( C) 96 C 1 minuto 96 C 10 segundos 25 ciclos 58 C 5 segundos 60 C 4 minutos Tabla 6. Programa del termociclador para una reacción de secuencia. Posteriormente, se purifica la reacción de secuencia y para ello se prepara la mezcla a continuación descrita (los volúmenes que se señalan son los correspondientes a un pocillo): 5µl de BigDye XTerminator (BigDye XTerminator Purification Kit de Applied Biosystems), 22,5µl de SAM solution (BigDye XTerminator Cycle Purification Kit de Applied Biosystems) y 27,5µl H 2 Od autoclavada. Se le añade a cada pocillo de la anterior placa 55µl, se cubre con un film y se envuelve en papel de aluminio para que no incida sobre ella la luz. Se agita durante 30 minutos e inmediatamente se centrifuga a 1000g durante 2 minutos, acto seguido se le retira el film a la placa y se le coloca un septa, se dispone en un soporte y se introduce en el secuenciador 3730 de Applied Biosystems. Finalmente, se crea un archivo para el secuenciador en el cual se le indica qué muestra y qué exón/fragmento corresponde a cada pocillo, y se comienza la secuenciación. 4. RESULTADOS Y DISCUSIÓN Tras llevar a cabo todas las pruebas pertinentes para confirmar por Sanger las variantes en los distintos exones o fragmentos de titina, se puede afirmar que tras los ensayos realizados en el laboratorio se ha comprobado que las condiciones óptimas de amplificación de los primers son: La titina en el diagnóstico genético de cardiopatías familiares Página 19
20 Para TTN-Ex186H-N2B, TTN-Ex143-N2A y TTN-154J-N2B. Volumen (µl) Buffer + enzima 6,25 Primer 2 Solución Q 1 H 2 O 1,75 ADN 1,5 Tabla 7. Condiciones de PCR para los fragmentos arriba indicados. Para todos los demás exones y fragmentos se ha visto que las condiciones de PCR son: Volumen (µl) Buffer + enzima 6,25 Primer 1,5 Solución Q 1 H 2 O 2,25 ADN 1,5 Tabla 8. Condiciones de PCR generales. Comparando las diferencias entre ambas condiciones de PCR se puede apreciar que éstas residen únicamente en el volumen del primer (Tabla 9). Por lo tanto, se puede pensar que las bandas tenues que se observaban eran debidas, simplemente, a que el volumen dispensado de estos primers no era suficiente para amplificar las muestras. Tabla 7. Condiciones específicas Tabla 8. Condiciones generales Buffer + enzima 6,25 µl 6,25 µl Primer 2 µl 1,5 µl Solución Q 1 µl 1 µl H 2 O 1,75 µl 2,25 µl ADN 1,5 µl 1,5 µl Tabla 9. Comparación de las distintas condiciones de PCR. La titina en el diagnóstico genético de cardiopatías familiares Página 20
21 A continuación se muestran las fotos de las electroforesis realizadas, donde se puede comprobar lo anteriormente citado en el apartado 3.2. TTN-EX186C-N2B TTN-EX143-N2A TTN-EX186H-N2B TTN-EX154J-N2B Imagen 10. Electroforesis (parte 1). TTN-EX65-N2B Imagen 11. Electroforesis (parte 2). Tal y como se señala en las fotografías (Imagen 10 y 11), las bandas de los fragmentos TTN-Ex186H-N2B, TTN-Ex143-N2A y TTN-Ex154J-N2B presentan unas bandas demasiado tenues como para esperar que la lectura del secuenciador sea correcta. Asimismo los fragmentos TTN-Ex186C-N2B y TTN-Ex65-N2B muestran una estela tras la banda. Se repite la electroforesis de estos dos con un tiempo de carrera de 40 minutos, y se observa lo siguiente: La titina en el diagnóstico genético de cardiopatías familiares Página 21
22 Imagen 12. Electroforesis de TTN-EX65-N2B (izquierda) y TTN-Ex186C-N2B (derecha). Como se muestra en la imagen de electroforesis para los fragmentos TTN-Ex65-N2B y TTN-Ex186C-N2B (Imagen 12), con una carrera mayor han desaparecido las estelas que se observaban tras las bandas. Probablemente haya ocurrido porque al aumentar el tiempo, en esta ocasión, ha migrado mejor. Seguidamente se muestra la electroforesis de los 3 fragmentos, TTN-Ex186H- N2B,TTN-Ex143-N2A y TTN-Ex154J-N2B, a los que se les asignó unas condiciones de PCR específicas (tabla 7): TTN-EX186H-N2B TTN-EX143-N2A TTN-EX154J-N2B Imagen 13. Electroforesis de PCR con condiciones específicas. En esta ocasión, las bandas de los fragmentos señalados (Imagen 13) se observan con la calidad suficiente como para continuar el proceso de optimización. Para finalizar, el siguiente y último punto de observación de resultados es el análisis de los datos obtenidos por el secuenciador 3730 de Applied Biosystems mediante la aplicación informática de Variant Reporter, también de Applied Biosystems. Variant Reporter recoge los datos que exporta el secuenciador al finalizar el análisis de las muestras y facilita su La titina en el diagnóstico genético de cardiopatías familiares Página 22
23 análisis, comparando los resultados de cada secuencia con una secuencia de referencia y señalando las variaciones que presenta respecto a ésta, además también permite hacer comparaciones entre diferentes secuencias o indicar en qué región del exón/intrón se está analizando. Señala el exón que se analiza Señala en qué región del exón se analiza Señala las variantes que se encontraron Señala las muestras que se analizan Imagen 14. Software de Variant Reporter. Con ayuda del Variant Reporter se analizan todas las secuencias, es decir, se va comprobando que tanto la secuencia forward de la muestra como la reverse coincida con la secuencia de referencia, si no es así se anota cuál es la variante o variantes que presenta. Además, al ser la primera vez que se secuencian estos fragmentos de titina, se comprueba que la región de lectura sea del tamaño esperado. Las variaciones que se pueden encontrar habitualmente en una secuencia son: Cambios nucleotídicos sinónimos o no sinónimos Inserciones que no modifican la pauta de lectura Deleciones que no modifican la pauta de lectura Frameshifts, cambios en la pauta de lectura provocados por inserciones o deleciones La titina en el diagnóstico genético de cardiopatías familiares Página 23
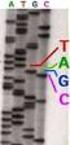
24 Igualmente, se pueden encontrar en los 20pb de las regiones intrónicas flanqueantes variaciones que pueden afectar al splicing de la proteína. Para predecirlo se emplea un programa informático Alamut, de Interactive Biosoftware, que compara en diferentes bases de datos. A continuación se muestra el ejemplo de una secuencia que no presenta ninguna variante en el exón TTN-Ex23-N2B. En la línea indicada como Ref muestra la secuencia de referencia con la que se compara, la línea Sum señala el sumatorio de la secuencia forward y reverse de la muestra problema. Las bandas moradas que aparecen debajo de Ref y Sum hacen referencia a la calidad con la que se da el resultado de cada nucleótido. Por último se muestra el electroferograma de la secuencia forward y reverse Imagen 15. Software de Variant Reporter. Análisis del exón TTN-Ex23-N2B de una muestra. En el siguiente caso se muestra como se visualizan 2 muestras a la vez para el exón TTN-Ex21-N2B. En la primera muestra (situada en la parte superior) se observa que no presenta variantes para dicho exón. Sin embargo, la segunda muestra (situada en la parte inferior de la imagen) presenta, en la posición señalada, 2 picos en el electroferograma: uno de color negro correspondiente a la G y otro verde correspondiente a la A, lo que está indicando que este paciente presenta una variante en heterocigosis. La titina en el diagnóstico genético de cardiopatías familiares Página 24
25 Imagen 16. Software de Variant Reporter. Análisis del exón TTN-Ex21-N2B de dos muestras. Tras analizar con el Variant Reporter las secuencias de todas las muestras requeridas se puede afirmar que se han detectado las siguientes variantes: Muestra P. genómica de laboratorio Cambio aa. Significado del cambio 1445 g g>c p.a25686p No sinónimo 1445 g.61345c>t p.r5441x Stop 1492 g c>g p.p24914r No sinónimo 1391 g _129754delgaa p.10170_10170del Deleción 1494 g _176215delgcagat p.5547_5548del Deleción 1300 g _235890delaca p.15801_15801del Deleción 1300 g.27390a>t p.d1186v No sinónimo 1701 g _272136insccttcagat p.p24711delinspsdp Inserción 1701 g _272125insagccttcag p.k24707delinskpse Inserción 1701 g c>t p.r8985c No sinónimo 1377 g.87601g>t p.r6646i No sinónimo 1498 g t>c p.s13302p No sinónimo 1498 g.48248g>a Intrónica Splicing 1625 g g>c p.a7807p No sinónimo 1711 g _129754delgaa p.10170_10170del Deleción 1711 g a>g p.e14985g No sinónimo 1711 g g>a p.e11694k No sinónimo 1711 g g>c p.e10289q No sinónimo La titina en el diagnóstico genético de cardiopatías familiares Página 25
26 1711 g t>c Intrónica Splicing 1381 g a>g p.n21581s No sinónimo 1381 g.28390c>t p.r1304c No sinónimo 1446 g g>a p.r26175q No sinónimo 1446 g c>g p.t8910s No sinónimo 1629 g g>a p.r20744q No sinónimo 1668 g a>g p.e26299g No sinónimo 1390 g g>a p.r24899h No sinónimo 1287 g g>a p.e11694k No sinónimo Tabla 10. Posición de las variantes que se han encontrado en el laboratorio. P. genómica de laboratorio es la posición genómica de la variante junto al cambio nucleotídico. Cambio aa. muestra el cambio aminoacídico, en los casos en los que se hace la anotación intrónica indica que el cambio no ha tenido lugar dentro del exón y, por lo tanto, no hay cambio aminoacídico, pero se estudiará igualmente porque puede afectar al splicing de la proteína. Significado del cambio hace referencia a cómo afecta esa variante en la secuencia. Comparando los resultados de la Tabla 10, que se han obtenido por el método de secuenciación de Sanger, con los de las Tablas 1 y 2, que se han obtenido por NGS, se puede confirmar que los resultados alcanzados son los esperados y, por lo tanto, que los resultados obtenidos por ambos métodos de secuenciación son los mismos. Basándose en estos datos, y apoyado en numerosos artículos y revisiones científicas, se demuestra que aunque todavía no se realicen diagnósticos genéticos por NGS, parece que la técnica ya está ampliamente desarrollada para su implantación. La titina en el diagnóstico genético de cardiopatías familiares Página 26
27 5. CONCLUSIÓN Health In Code cuenta con un laboratorio de análisis genético que emplea tecnología Sanger para el diagnóstico de cardiopatías familiares. Sin embargo, se está iniciando en un nuevo servicio basado en el diagnóstico genético empleando nuevas tecnologías de secuenciación masiva (NGS). El estudio de un paciente con NGS permite analizar gran cantidad de genes a un costo mucho más bajo y detectar miles de variantes genéticas que pueden estar asociadas a otras patologías. Estos son algunas de las ventajas que presentan las técnicas de NGS frente a la ya tradicional técnica de Sanger, y son motivos suficientes para que Health In Code esté muy interesado en adentrarse en el mundo de NGS. Así, artículos y revisiones, en los cuales se confirman que los resultados obtenidos por Sanger coinciden con los de NGS, son esenciales para poner definitivamente a punto esta técnica y dar el salto al diagnóstico genético por secuenciación masiva. La titina en el diagnóstico genético de cardiopatías familiares Página 27
28 6. REFERENCIAS BIBLIOGRÁFICAS 5.1. Lecturas principales 1. Tobias Voelkel, Wolfgang A Linke. Conformation-regulated mechanosensory control via titin domains in cardiac muscle. Pflugers Arch - Eur J Physiol 2011;462: Henk Granzier, Siegfried Labeit. Structure function relations of the giant elastic protein titin in striated and smooth muscle cells. Muscle Nerve 2007;36: Martin M LeWinter, Henk Granzier. Cardiac titin: A multifunctional giant. Circulation 2010, 121: Wolfgang A Linke. Sense and stretchability: The role of titin and titin-associated proteins in myocardial stress-sensingand mechanical dysfunction. Cardiovascular research 2008;77: Henk L. Granzier, Siegfried Labeit. The giant protein titin: A major player in myocardial mechanics, signaling, and disease. Circulation Research 2004;94: Ricardo Castro-Ferreira, Ricardo Fontes-Carvalho, Inês Falcão-Pires, Adelino F Leite- Moreira. The role of titin in the modulation of cardiac function and its pathophysiological implications. Arq Bras Cardiol 2011;96: Lectura complementaria 7. Henk Granzier, Yiming Wu, Labeit Siegfried, Martin LeWinter. Titin: Physiological function and role in cardiomyopathy and failure. Heart Failure Reviews 2005;10: Larissa Tskhovrebova, John Trinick. Roles of titin in the structure and elasticity of the sarcomere. Journal of Biomedicine and Biotechnology 2010;612482:1-7 La titina en el diagnóstico genético de cardiopatías familiares Página 28
29 9. Aikaterini Kontrogianni-Konstantopoulos, Maegen A Ackermann, Amber L Bowman, Solomon V Yap, Robert J. Bloch. muscle giants: Molecular scaffolds in sarcomerogenesis. Physiol Rev 2009;89: Tskhovrebova L, Trinick J. Role of titin in vertebrate striated muscle. Phil. Trans. R. Soc. Lond. B 2002; 357: Makarenko I, Opitz CA, Leake MC, Neagoe C, Kulke M, Gwathmey JK, Del Monte F, Hajjar RJ, Linke WA. Passive stiffness changes caused by upregulation of compliant titin isoforms in human dilated cardiomyopathy hearts. Circulation Researh 2004;95: La titina en el diagnóstico genético de cardiopatías familiares Página 29
Protocolos de preparación de muestras
 Protocolos de preparación de muestras Contenido 1. TIPOS DE MUESTRA 2. PROTOCOLO DE PREPARACIÓN DE MUESTRA 2.1 Producto de PCR crudo (sin purificar). 2.2 Producto de PCR crudo en gel (sin purificar). 2.3
Protocolos de preparación de muestras Contenido 1. TIPOS DE MUESTRA 2. PROTOCOLO DE PREPARACIÓN DE MUESTRA 2.1 Producto de PCR crudo (sin purificar). 2.2 Producto de PCR crudo en gel (sin purificar). 2.3
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
Test Genéticos Moleculares
 CERPO Centro de Referencia Perinatal Oriente Facultad de Medicina, Universidad de Chile Test Genéticos Moleculares Dr. Guillermo Parrao Barrera Residente Medicina Materno Fetal Pontificia Universidad Católica
CERPO Centro de Referencia Perinatal Oriente Facultad de Medicina, Universidad de Chile Test Genéticos Moleculares Dr. Guillermo Parrao Barrera Residente Medicina Materno Fetal Pontificia Universidad Católica
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
Temas actuales: Next Generation Sequencing (NGS) Bioinformática Elvira Mayordomo
 Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introducción a la Secuenciación Masiva y a la Bioinformática
 Introducción a la Secuenciación Masiva y a la Bioinformática Dietmar Fernández Orth, PhD 24 de Abril de 2014 1 El DNA (Ácido desoxirribonucleico) contiene la información genética usada en el desarrollo
Introducción a la Secuenciación Masiva y a la Bioinformática Dietmar Fernández Orth, PhD 24 de Abril de 2014 1 El DNA (Ácido desoxirribonucleico) contiene la información genética usada en el desarrollo
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80
 AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
TECNICAS DE BIOLOGIA MOLECULAR
 Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Código: IDK-010 Ver: 1. Proteína G C825T. Sistema para la detección de la mutación C825T en el gene de la proteína G.
 C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
figura1 : esquema PCR introducción a la PCR termocicladoresnahita
 Gracias a sus altas prestaciones e increíble precio, los termocicladores Nahita son los equipos de elección de gran cantidad de laboratorios y centros de investigación para llevar a cabo la reacción en
Gracias a sus altas prestaciones e increíble precio, los termocicladores Nahita son los equipos de elección de gran cantidad de laboratorios y centros de investigación para llevar a cabo la reacción en
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR
 PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
Diagnóstico Genético Preimplantacional Indicaciones Actuales. Dr. Joaquín Moreno Valencia Febrero 2009
 Diagnóstico Genético Preimplantacional Indicaciones Actuales Dr. Joaquín Moreno Valencia Febrero 2009 DGP El diagnóstico Genético Preimplantacional(DGP) se presenta como una forma muy precoz de diagnóstico
Diagnóstico Genético Preimplantacional Indicaciones Actuales Dr. Joaquín Moreno Valencia Febrero 2009 DGP El diagnóstico Genético Preimplantacional(DGP) se presenta como una forma muy precoz de diagnóstico
La primera parte del proyecto consistió en la obtención de un control positivo mediante
 7. DISCUSIÓN La primera parte del proyecto consistió en la obtención de un control positivo mediante técnicas de clonación molecular. E. coli, a diferencia de otras bacterias que presentan competencia
7. DISCUSIÓN La primera parte del proyecto consistió en la obtención de un control positivo mediante técnicas de clonación molecular. E. coli, a diferencia de otras bacterias que presentan competencia
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006
 IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
3. COMPONENTES. Tampón de electroforesis concentrado 2 x 50 ml 10 X (2 envases 500ml)
 PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
Material complementario UD 5
 Material complementario UD 5 Componentes de la PCR ADN molde que es el que se quiere replicar. Oligonucleótidos iniciadores, primers o cebadores: - Son oligonucleótidos monocatenarios cuya secuencia es
Material complementario UD 5 Componentes de la PCR ADN molde que es el que se quiere replicar. Oligonucleótidos iniciadores, primers o cebadores: - Son oligonucleótidos monocatenarios cuya secuencia es
3. Materiales y Métodos
 3. Materiales y Métodos El análisis de haplotipos se realizó mediante la caracterización de variaciones alélicas reportadas con anterioridad. En total de analizaron cinco marcadores polimórficos: tres
3. Materiales y Métodos El análisis de haplotipos se realizó mediante la caracterización de variaciones alélicas reportadas con anterioridad. En total de analizaron cinco marcadores polimórficos: tres
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA
 UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
TaqMan Roundup Ready Quantification Kit. Part No:
 TaqMan Roundup Ready Quantification Kit Part No: 4466335 1. Introducción Los organismos modificados genéticamente (OMG) se encuentran ampliamente distribuidos, siendo la soja y el maíz los vegetales que
TaqMan Roundup Ready Quantification Kit Part No: 4466335 1. Introducción Los organismos modificados genéticamente (OMG) se encuentran ampliamente distribuidos, siendo la soja y el maíz los vegetales que
INFORMACIÓN AL USUARIO
 INFORMACIÓN AL USUARIO 1. SERVICIOS QUE SE OFRECEN 2. CONDICIONES QUE DEBEN CUMPLIR LAS MUESTRAS 3. INFORMACIÓN ADICIONAL 4. CÓMO HACER USO DEL SERVICIO 5. RESULTADOS 1.- SERVICIOS QUE SE OFRECEN Nuestro
INFORMACIÓN AL USUARIO 1. SERVICIOS QUE SE OFRECEN 2. CONDICIONES QUE DEBEN CUMPLIR LAS MUESTRAS 3. INFORMACIÓN ADICIONAL 4. CÓMO HACER USO DEL SERVICIO 5. RESULTADOS 1.- SERVICIOS QUE SE OFRECEN Nuestro
DETERMINACIÓN DEL FACTOR Rh por PCR
 Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Instrucciones de uso. BAG Cycler Check. Kit de para la validación de la uniformidad de la temperatura en los termocicladores
 Instrucciones de uso BAG Cycler Check Kit de para la validación de la uniformidad de la temperatura en los termocicladores listo para usar, prealicuotado REF 7104 (10 tests) REF 71044 (4 tests) Contenidos
Instrucciones de uso BAG Cycler Check Kit de para la validación de la uniformidad de la temperatura en los termocicladores listo para usar, prealicuotado REF 7104 (10 tests) REF 71044 (4 tests) Contenidos
3. MATERIALES Y MÉTODOS
 3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
RESULTADOS Y DISCUSIÓN
 RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
RESULTADOS Y DISCUSIÓN Extracción de ADN en sangre periférica La técnica de extracción por GeneClean empleada en este trabajo dio un buen rendimiento, ya que la cantidad de ADN y el nivel de purificación
Actualización en nuevas técnicas de diagnóstico genético molecular disponibles en Chile
 Actualización en nuevas técnicas de diagnóstico genético molecular disponibles en Chile Dra. Teresa Aravena Clínica INDISA Hospital Clínico de la Universidad de Chile Hospital Dr. Sótero del Río Exámenes
Actualización en nuevas técnicas de diagnóstico genético molecular disponibles en Chile Dra. Teresa Aravena Clínica INDISA Hospital Clínico de la Universidad de Chile Hospital Dr. Sótero del Río Exámenes
PCR Múltiplex. Listeria monocytogenes
 PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
Técnicas moleculares.
 Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Papel de la Titina en la Modulación de la Función Cardíaca y sus Implicaciones Fisiopatológicas
 Papel de la Titina en la Modulación de la Función Cardíaca y sus Implicaciones Fisiopatológicas Ricardo Castro-Ferreira, Ricardo Fontes-Carvalho, Inês Falcão-Pires, Adelino F. Leite-Moreira Serviço de
Papel de la Titina en la Modulación de la Función Cardíaca y sus Implicaciones Fisiopatológicas Ricardo Castro-Ferreira, Ricardo Fontes-Carvalho, Inês Falcão-Pires, Adelino F. Leite-Moreira Serviço de
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP
 SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES
 PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS
 Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90. País: ESPAÑA
 XII OLIMPIADA IBEROAMERICANA DE BIOLOGÍA SETIEMBRE 2018 LOJA-ECUADOR EXAMEN PRÁCTICO BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90 TIEMPO PERMITIDO: 120 minutos País: ESPAÑA ESCRIBA EL CÓDIGO EN EL SIGUIENTE
XII OLIMPIADA IBEROAMERICANA DE BIOLOGÍA SETIEMBRE 2018 LOJA-ECUADOR EXAMEN PRÁCTICO BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90 TIEMPO PERMITIDO: 120 minutos País: ESPAÑA ESCRIBA EL CÓDIGO EN EL SIGUIENTE
Proyecto de Fin de Cursos
 Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Métodos de secuenciación masiva. Introducción. Métodos de secuenciación masiva
 Introducción Secuenciación de Sanger -Mezcla de dntps y ddntps -Amplificación -Electroforesis Los fragmentos migran de acuerdo a su tamaño Secuenciación de Sanger Mejoras al método de Sanger Uso de terminadores
Introducción Secuenciación de Sanger -Mezcla de dntps y ddntps -Amplificación -Electroforesis Los fragmentos migran de acuerdo a su tamaño Secuenciación de Sanger Mejoras al método de Sanger Uso de terminadores
TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones
 TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones Dra. Mariana L. Ferramola Curso de Técnicas Moleculares Aplicadas a Bioquímica Clínica. Área de Qca. Biológica
TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones Dra. Mariana L. Ferramola Curso de Técnicas Moleculares Aplicadas a Bioquímica Clínica. Área de Qca. Biológica
Descifrando las bases genéticas de la ceguera
 Descifrando las bases genéticas de la ceguera Servicio de interpretación clínica del genoma germinal que analiza mediante secuenciación de nueva generación un panel de más de 300 genes con utilidad clínica,
Descifrando las bases genéticas de la ceguera Servicio de interpretación clínica del genoma germinal que analiza mediante secuenciación de nueva generación un panel de más de 300 genes con utilidad clínica,
Medir con cámaras infrarrojas
 El pie diabético es una degeneración de la estructura vascular de los pies. Surge a partir de que se produce un engrosamiento vascular y el flujo sanguíneo se atrofia. Con el paso del tiempo se forman
El pie diabético es una degeneración de la estructura vascular de los pies. Surge a partir de que se produce un engrosamiento vascular y el flujo sanguíneo se atrofia. Con el paso del tiempo se forman
PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN DE LAS COMPETENCIAS PROFESIONALES CUESTIONARIO DE AUTOEVALUACIÓN PARA LAS TRABAJADORAS Y TRABAJADORES
 MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
Caracteristicas del ADN. Se rompen con las siguientes condiciones -Temperaturas >90 - ph >10.5
 PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
Informe técnico del proyecto. de diagnóstico genético de pacientes con. síndrome de Dravet y espectros asociados
 Informe técnico del proyecto de diagnóstico genético de pacientes con síndrome de Dravet y espectros asociados Introducción El objetivo principal de este proyecto es realizar un diagnóstico genético a
Informe técnico del proyecto de diagnóstico genético de pacientes con síndrome de Dravet y espectros asociados Introducción El objetivo principal de este proyecto es realizar un diagnóstico genético a
Grecia Manuela Marrón Liñares Tutora: Dra. Lucía Núñez Fernández INIBIC: Grupo de Cardiología
 DESARROLLO METODOLÓGICO PARA UN ESTUDIO FARMACOGENÉTICO DE LOS POLIMORFISMOS DE LOS GENES HCN4 Y CYP3A4 Y SU EFECTO EN LA RESPUESTA A LA IVABRADINA EN PACIENTES CON INSUFICIENCIA CARDÍACA. METHODOLOGICAL
DESARROLLO METODOLÓGICO PARA UN ESTUDIO FARMACOGENÉTICO DE LOS POLIMORFISMOS DE LOS GENES HCN4 Y CYP3A4 Y SU EFECTO EN LA RESPUESTA A LA IVABRADINA EN PACIENTES CON INSUFICIENCIA CARDÍACA. METHODOLOGICAL
Epidemiología molecular Clostridium difficile
 Epidemiología molecular Clostridium difficile Dra Paola Pidal Laboratorio biomédico Nacional y de Referencia Clostridium difficile: Epidemiología molecular Disciplina que resulta de la aplicación e integración
Epidemiología molecular Clostridium difficile Dra Paola Pidal Laboratorio biomédico Nacional y de Referencia Clostridium difficile: Epidemiología molecular Disciplina que resulta de la aplicación e integración
PRÁCTICA DE GENÉTICA
 PRÁCTICA DE GENÉTICA Diagnóstico molecular de una enfermedad genética, seguimiento en una genealogía y consejo genético. Profesores responsables: Paloma Martínez Rodríguez y José L. Bella Sombría NORMAS
PRÁCTICA DE GENÉTICA Diagnóstico molecular de una enfermedad genética, seguimiento en una genealogía y consejo genético. Profesores responsables: Paloma Martínez Rodríguez y José L. Bella Sombría NORMAS
Código: KIT-001 Ver: 1. Pre-FRAXA. Sistema para la detección del número de repetidos CGG en el gene FMR1 PM F M M F M M M C. Reg.
 Pre-FRAXA Sistema para la detección del número de repetidos CGG en el gene FMR1 PM F M M F M M M C Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. www.atgen.com.uy
Pre-FRAXA Sistema para la detección del número de repetidos CGG en el gene FMR1 PM F M M F M M M C Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. www.atgen.com.uy
EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL EXAMEN DHE MOLECULARES EN EL EXAMEN DHE
 X Curso de Formación sobre la Protección de las Obtenciones Vegetales para Países Iberoamericanos Montevideo (URUGUAY) 12 al 16 de Diciembre de 2011 EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL
X Curso de Formación sobre la Protección de las Obtenciones Vegetales para Países Iberoamericanos Montevideo (URUGUAY) 12 al 16 de Diciembre de 2011 EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de
 Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de ASEBIR). 1 2 3 4 Anexo2: Ejemplo de protocolo para translocaciones
Anexo 1: Enfermedades monogénicas diagnosticadas hasta la fecha, en España, mediante DGP (Fuente: datos propios del grupo de interés en DGP de ASEBIR). 1 2 3 4 Anexo2: Ejemplo de protocolo para translocaciones
UNIDAD DE SECUENCIACIÓN HOSPITAL UNIVERSITARIO SANT JOAN DE DEU
 UNIDAD DE SECUENCIACIÓN HOSPITAL UNIVERSITARIO SANT JOAN DE DEU La Unidad de secuenciación del Hospital Universitario Sant Joan de Déu, es una unidad de reciente creación que nace con el objetivo de prestar
UNIDAD DE SECUENCIACIÓN HOSPITAL UNIVERSITARIO SANT JOAN DE DEU La Unidad de secuenciación del Hospital Universitario Sant Joan de Déu, es una unidad de reciente creación que nace con el objetivo de prestar
ANÁLISIS DE LA EXPRESIÓN DE LOS GENES XPR2 Y DAP1 (CODIFICANTES DE LA PROTEASA ALCALINA Y LA DIPEPTIDIL AMINOPEPTIDASA) DE Yarrowia lipolytica
 ANÁLISIS DE LA EXPRESIÓN DE LOS GENES XPR2 Y DAP1 (CODIFICANTES DE LA PROTEASA ALCALINA Y LA DIPEPTIDIL AMINOPEPTIDASA) DE Yarrowia lipolytica El sistema proteolítico del hongo dimórfico Yarrowia lipolytica
ANÁLISIS DE LA EXPRESIÓN DE LOS GENES XPR2 Y DAP1 (CODIFICANTES DE LA PROTEASA ALCALINA Y LA DIPEPTIDIL AMINOPEPTIDASA) DE Yarrowia lipolytica El sistema proteolítico del hongo dimórfico Yarrowia lipolytica
GENOTIPADO 1. MICROSATÉLITES
 GENOTIPADO 1. MICROSATÉLITES El uso de marcadores moleculares para la identificación de genotipos de interés mediante microsatélites es susceptible de ser analizadas a través de la instrumentación existente
GENOTIPADO 1. MICROSATÉLITES El uso de marcadores moleculares para la identificación de genotipos de interés mediante microsatélites es susceptible de ser analizadas a través de la instrumentación existente
Reacción en cadena de la polimerasa (PCR)
 Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
MAESTRÍA EN INGENIERÍA BIOMÉDICA
 MAESTRÍA EN INGENIERÍA BIOMÉDICA Bases Físico-Matemáticas de los Sistemas Biológicos Docentes: Msc. Ing. Sandra Wray Dr. Ing. Ricardo L. Armentano Dr. Ing. Leandro J. Cymberknop 2015 Las células cardíacas
MAESTRÍA EN INGENIERÍA BIOMÉDICA Bases Físico-Matemáticas de los Sistemas Biológicos Docentes: Msc. Ing. Sandra Wray Dr. Ing. Ricardo L. Armentano Dr. Ing. Leandro J. Cymberknop 2015 Las células cardíacas
Introducción a la Bioinformática
 Introducción a la Bioinformática Genómica y bioinformática: Nuevas áreas de biotecnología Genómica-Ciencia que se encarga de las estrategias de clonación, secuenciación y análisis de genes. Cómo los científicos
Introducción a la Bioinformática Genómica y bioinformática: Nuevas áreas de biotecnología Genómica-Ciencia que se encarga de las estrategias de clonación, secuenciación y análisis de genes. Cómo los científicos
DETECCIÓN DE C. jejuni EN HAMBURGUESA DE POLLO POR PCR TRADICIONAL PRT-712.04-082
 Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Ficha descriptiva SERVICIOS CENTRALES DEL CENTRO DE APOYO TECNOLÓGICO DE LA URJC. Nombre de la UNIDAD/Técnica: Genómica y Citometría de Flujo.
 Página 1 de 5 Nombre de la UNIDAD/Técnica: Genómica y Citometría de Flujo. Responsable: José Antonio Mas Gutiérrez Teléfono: 914 888 645 / 914 887 184 Email joseantonio.mas@urjc.es Principios de la Técnica
Página 1 de 5 Nombre de la UNIDAD/Técnica: Genómica y Citometría de Flujo. Responsable: José Antonio Mas Gutiérrez Teléfono: 914 888 645 / 914 887 184 Email joseantonio.mas@urjc.es Principios de la Técnica
Métodos de secuenciación de ADN
 Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos
 Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Pregunta y Conoce tu Respuesta
 En LMC Pregunta y Conoce tu Hematológica Citogenética Molecular Conoce la leucemia mieloide crónica Qué es la leucemia mieloide crónica? La leucemia mieloide crónica (LMC) es una enfermedad en la que las
En LMC Pregunta y Conoce tu Hematológica Citogenética Molecular Conoce la leucemia mieloide crónica Qué es la leucemia mieloide crónica? La leucemia mieloide crónica (LMC) es una enfermedad en la que las
Código: IDK-003 Ver: 1. Factor V Leiden
 Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
Sistema para detección de la alteración G1691A en el gene que codifica para el Factor V de la coagulación humana Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN
 COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN OBJETIVO: el presente trabajo práctico tiene como objetivo principal que el alumno adquiera destreza en el análisis de electroferogramas generados por equipos
COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN OBJETIVO: el presente trabajo práctico tiene como objetivo principal que el alumno adquiera destreza en el análisis de electroferogramas generados por equipos
TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones
 TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones Dra. Mariana L. Ferramola Curso de Técnicas Moleculares Aplicadas a Bioquímica Clínica. Área de Química Biológica
TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones Dra. Mariana L. Ferramola Curso de Técnicas Moleculares Aplicadas a Bioquímica Clínica. Área de Química Biológica
Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento
 Dra. Gabriela Lacoste- QBP 2012 MLPA Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento Es una técnica que permite la detección de cambios en el
Dra. Gabriela Lacoste- QBP 2012 MLPA Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento Es una técnica que permite la detección de cambios en el
MARCADORES GENÉTICOS
 MARCADORES GENÉTICOS MARCADORES GENÉTICOS La diferencia entre individuos se denotaba señalando diferencias en su aspecto externo (fenotipo) Marcadores Morfológicos Carácter Informativo Variación POLIMORFISMO
MARCADORES GENÉTICOS MARCADORES GENÉTICOS La diferencia entre individuos se denotaba señalando diferencias en su aspecto externo (fenotipo) Marcadores Morfológicos Carácter Informativo Variación POLIMORFISMO
MANUAL DE PROCEDIMIENTOS DEL SECUENCIADOR GENETICO: 3500 xl GENETIC ANALIZER de Applied Biosystems
 MANUAL DE PROCEDIMIENTOS DEL SECUENCIADOR GENETICO: 3500 xl GENETIC ANALIZER de Applied Biosystems INDICE I. Introducción II. Objetivo General del Manual A. Protocolo de Secuenciación A.1 Propósito A.2
MANUAL DE PROCEDIMIENTOS DEL SECUENCIADOR GENETICO: 3500 xl GENETIC ANALIZER de Applied Biosystems INDICE I. Introducción II. Objetivo General del Manual A. Protocolo de Secuenciación A.1 Propósito A.2
Centro Medico Nacional Siglo XXI Hospital de Especialidades. Alergia e Inmunología Clínica
 Centro Medico Nacional Siglo XXI Hospital de Especialidades Complejo Mayor de Histocompatibilidad Dra. Patricia María O Farrill Romanillos R3AIC México, D.F. a 21 de Junio del 2012 Definición Moléculas
Centro Medico Nacional Siglo XXI Hospital de Especialidades Complejo Mayor de Histocompatibilidad Dra. Patricia María O Farrill Romanillos R3AIC México, D.F. a 21 de Junio del 2012 Definición Moléculas
EXAMEN DE LA PRIMERA PARTE (30%) NOMBRE CÓDIGO FECHA. El examen consta de un total de 20 puntos y el tiempo máximo para contestar es de 1 hora.
 1 PONTIFICIA UNIVERSIDAD JAVERIANA MAESTRÍA EN INFORMÁTICA CURSO DE BIOINFORMÁTICA EXAMEN DE LA PRIMERA PARTE (30%) NOMBRE CÓDIGO FECHA El examen consta de un total de 20 puntos y el tiempo máximo para
1 PONTIFICIA UNIVERSIDAD JAVERIANA MAESTRÍA EN INFORMÁTICA CURSO DE BIOINFORMÁTICA EXAMEN DE LA PRIMERA PARTE (30%) NOMBRE CÓDIGO FECHA El examen consta de un total de 20 puntos y el tiempo máximo para
PROCEDIMIENTO GENERAL. Gestión de Incidencias y Acciones Correctivas RAZÓN SOCIAL DE LA EMPRESA. Código PG-12 Edición 0. Índice:
 Índice: 1. TABLA RESUMEN... 2 2. OBJETO... 2 3. ALCANCE... 2 4. RESPONSABILIDADES... 3 5. ENTRADAS... 4 6. SALIDAS... 4 7. PROCESOS RELACIONADOS... 4 8. DIAGRAMA DE FLUJO... 5 9. DESARROLLO... 6 9.1. DETECCIÓN
Índice: 1. TABLA RESUMEN... 2 2. OBJETO... 2 3. ALCANCE... 2 4. RESPONSABILIDADES... 3 5. ENTRADAS... 4 6. SALIDAS... 4 7. PROCESOS RELACIONADOS... 4 8. DIAGRAMA DE FLUJO... 5 9. DESARROLLO... 6 9.1. DETECCIÓN
1 MATERIALES Y MÉTODOS
 1 MATERIALES Y MÉTODOS El presente protocolo experimental contempla la amplificación del DNA de las bacterias y virus causantes de las ETS Neisseria gonorrhoeae, Chlamydia trachomatis y VPH mediante PCR
1 MATERIALES Y MÉTODOS El presente protocolo experimental contempla la amplificación del DNA de las bacterias y virus causantes de las ETS Neisseria gonorrhoeae, Chlamydia trachomatis y VPH mediante PCR
Código: IDK-006 Ver: 1 MTHFR A1298C. Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa
 Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
Sistema para detección de la alteración A1298C en el gene de la Mutilen Tetrahidrofolato Reductasa Reg. MSP 21199 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336
Genes. Estructura. Promotores. Intrones y exones. Pseudogenes. Genética 1 er Curso. Facultad de Medicina TEMA 0-2
 Facultad de Medicina Genética 1 er Curso TEMA 0-2 EXPRESIÓN GÉNICA:FLUJO DE INFORMACIÓN GENÉTICA Genes. El código genético. Dogma central de la Biología Molecular.. Genes Apartados Estructura Promotores
Facultad de Medicina Genética 1 er Curso TEMA 0-2 EXPRESIÓN GÉNICA:FLUJO DE INFORMACIÓN GENÉTICA Genes. El código genético. Dogma central de la Biología Molecular.. Genes Apartados Estructura Promotores
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
PCR SIMULADA. Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO
 Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Técnicas de Biología Molecular. Biol 3051L
 Técnicas de Biología Molecular Biol 3051L Objetivos El cortar el DNA con enzimas de restricción y hacer corridas de geles usando electroforesis es muchas veces el primer paso para estudiar un gen. En este
Técnicas de Biología Molecular Biol 3051L Objetivos El cortar el DNA con enzimas de restricción y hacer corridas de geles usando electroforesis es muchas veces el primer paso para estudiar un gen. En este
NUEVAS CAPACIDADES PARA LA MEDICINA GENÓMICA EN CANARIAS. División de Genómica
 NUEVAS CAPACIDADES PARA LA MEDICINA GENÓMICA EN CANARIAS División de Genómica El material genético en el núcleo celular Conformación 3D del ADN Descubrimiento del ADN El ADN es una doble hélice (1953)
NUEVAS CAPACIDADES PARA LA MEDICINA GENÓMICA EN CANARIAS División de Genómica El material genético en el núcleo celular Conformación 3D del ADN Descubrimiento del ADN El ADN es una doble hélice (1953)
MANUAL DE USUARIOS DEL SITIO WEB REMAS
 MANUAL DE USUARIOS DEL SITIO WEB REMAS RED DE ESTACIONES METEOROLÓGICAS AUTOMATIZADAS DEL ESTADO DE SONORA Versión 1. Mayo de 2015 CESAVESON SIAFESON Contenido I. Introducción... 2 II. Sitio web... 2 III.
MANUAL DE USUARIOS DEL SITIO WEB REMAS RED DE ESTACIONES METEOROLÓGICAS AUTOMATIZADAS DEL ESTADO DE SONORA Versión 1. Mayo de 2015 CESAVESON SIAFESON Contenido I. Introducción... 2 II. Sitio web... 2 III.
Detección de ADN de Trypanosoma cruzi por Reacción en Cadena de la Polimerasa en Tiempo Real
 Detección de ADN de Trypanosoma cruzi por Reacción en Cadena de la Polimerasa en Tiempo Real Prác%ca Profesional Obligatoria Área Parasitología Docentes: Dra. Victoria Alonso (valonso@
Detección de ADN de Trypanosoma cruzi por Reacción en Cadena de la Polimerasa en Tiempo Real Prác%ca Profesional Obligatoria Área Parasitología Docentes: Dra. Victoria Alonso (valonso@
CURSO ACADÉMICO 2009 2010
 TITULACIÓN: BIOLOGÍA CURSO ACADÉMICO 2009 2010 GENÉTICA APLICADA CÓDIGO: 200810419 Departamento de adscripción: Parasitología, Ecología y Genética Área de conocimiento: Genética Ciclo: 2º Curso: 4º Tipo:
TITULACIÓN: BIOLOGÍA CURSO ACADÉMICO 2009 2010 GENÉTICA APLICADA CÓDIGO: 200810419 Departamento de adscripción: Parasitología, Ecología y Genética Área de conocimiento: Genética Ciclo: 2º Curso: 4º Tipo:
Investigación en evolución
 Investigación en evolución Análisis de secuencias de: nucleótidos en DNA (genómica) aminoácidos en Proteínas (proteómica) Comparación de secuencias de: nucleótidos aminoácidos Bioinformática Análisis de
Investigación en evolución Análisis de secuencias de: nucleótidos en DNA (genómica) aminoácidos en Proteínas (proteómica) Comparación de secuencias de: nucleótidos aminoácidos Bioinformática Análisis de
- Las mezclas de amplificación se presentan en formato monotest en tubos de PCR de 0.2 ó 0.5 ml, identificadas con diferentes colores.
 ! El diagnóstico de los linfomas malignos es una de las áreas que más dificultad reviste dentro de la histopatología. Si bien muchos casos se diagnostican a través de los datos histomorfológicos e inmunohistoquímicos,
! El diagnóstico de los linfomas malignos es una de las áreas que más dificultad reviste dentro de la histopatología. Si bien muchos casos se diagnostican a través de los datos histomorfológicos e inmunohistoquímicos,
Caracterización de la expresión del gen reca
 Figura 3.8. (A) Diagrama que representa los fragmentos amplificados a partir del promotor lexa de F. nucleatum usados en experimentos EMSA. Las posiciones numeradas se refieren al punto de inicio de traducción
Figura 3.8. (A) Diagrama que representa los fragmentos amplificados a partir del promotor lexa de F. nucleatum usados en experimentos EMSA. Las posiciones numeradas se refieren al punto de inicio de traducción
GAC-Opendata Manual de Usuario
 GAC-Opendata Manual de Usuario Introducción GAC es la abreviatura de Global Aquifer Control. GAC-OpenData es: Una aplicación multinavegador que permite acceder a los datos de los acuíferos. Un conjunto
GAC-Opendata Manual de Usuario Introducción GAC es la abreviatura de Global Aquifer Control. GAC-OpenData es: Una aplicación multinavegador que permite acceder a los datos de los acuíferos. Un conjunto
6.1 Células competentes y transformación. posterior transformación de éstas para la elaboración de un control positivo. La
 6. RESULTADOS 6.1 Células competentes y transformación La primera parte experimental del trabajo consistió en la obtención de células competentes y posterior transformación de éstas para la elaboración
6. RESULTADOS 6.1 Células competentes y transformación La primera parte experimental del trabajo consistió en la obtención de células competentes y posterior transformación de éstas para la elaboración
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR
 KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
KITS EDUCATIVOS DE BIOLOGÍA MOLECULAR Índice General Aula GENYCA. Flujo de trabajo. 3 Kits Educativos de Técnicas Básicas 5 P1-A. Extracción de ADN de Sangre 6 P1-B. Extracción de ADN en Columna 7 P2.
QUÉ ES? La sucesión de estas cuatro letras en el ADN constituye el peculiar lenguaje de esta molécula, que forma frases del tipo:
 QUÉ ES? La sucesión de estas cuatro letras en el ADN constituye el peculiar lenguaje de esta molécula, que forma frases del tipo: GGCTATTAAACGATCGGGTC... El texto íntegro del ADN de una célula humana está
QUÉ ES? La sucesión de estas cuatro letras en el ADN constituye el peculiar lenguaje de esta molécula, que forma frases del tipo: GGCTATTAAACGATCGGGTC... El texto íntegro del ADN de una célula humana está
ES 2 525 416 A1 ESPAÑA 11. Número de publicación: 2 525 416. Número de solicitud: 201330715 C04B 18/16 (2006.01) 20.05.2013
 19 OFICINA ESPAÑOLA DE PATENTES Y MARCAS ESPAÑA 11 21 Número de publicación: 2 2 416 Número de solicitud: 1371 1 Int. CI.: C04B 18/16 (06.01) 12 SOLICITUD DE PATENTE A1 22 Fecha de presentación:.0.13 43
19 OFICINA ESPAÑOLA DE PATENTES Y MARCAS ESPAÑA 11 21 Número de publicación: 2 2 416 Número de solicitud: 1371 1 Int. CI.: C04B 18/16 (06.01) 12 SOLICITUD DE PATENTE A1 22 Fecha de presentación:.0.13 43
Límites de la anonimización y la confidencialidad
 H o s p i t a l U n i v e r s i t a r i o L a P a z M a d r i d Límites de la anonimización y la confidencialidad Julián Nevado/Pablo Lapunzina- INGEMM- Instituto de Genética Médica y Molecular Hospital
H o s p i t a l U n i v e r s i t a r i o L a P a z M a d r i d Límites de la anonimización y la confidencialidad Julián Nevado/Pablo Lapunzina- INGEMM- Instituto de Genética Médica y Molecular Hospital
Protocolos de secuenciación de ADN
 1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
Created by Simpo PDF Creator Pro (unregistered version) http://www.simpopdf.com MUSCULO ESQUELÉTICO
 MUSCULO ESQUELÉTICO INTRODUCCIÓN Clasificación funcional del musculo: 40% Musculo cardiaco Musculo liso MUSCULO ESQUELÉTICO MUSCULO ESQUELÉTICO Forman la mayor parte del musculo. Constituyen hasta el 40%
MUSCULO ESQUELÉTICO INTRODUCCIÓN Clasificación funcional del musculo: 40% Musculo cardiaco Musculo liso MUSCULO ESQUELÉTICO MUSCULO ESQUELÉTICO Forman la mayor parte del musculo. Constituyen hasta el 40%
Recursos didácticos digitales en Educación Permanente Mª Luisa Miras Cidad
 El entorno LIM Después de ver y analizar brevemente algunas herramientas de autor que se utilizan frecuentemente para crear materiales educativos, nos decidimos por LIM para empezar a elaborar los nuestros.
El entorno LIM Después de ver y analizar brevemente algunas herramientas de autor que se utilizan frecuentemente para crear materiales educativos, nos decidimos por LIM para empezar a elaborar los nuestros.
UNIVERSIDAD AUTONOMA DE TAMAULIPAS
 R-RS-01-25-03 UNIVERSIDAD AUTONOMA DE TAMAULIPAS UNIDAD ACADEMICA MULTIDISCIPLINARIA REYNOSA-AZTLAN NOMBRE DE LA CARRERA QUÍMICO FARMACÉUTICO BIÓLOGO NOMBRE DE LA ASIGNATURA LABORATORIO DE BIOLOGÍA MOLECULAR
R-RS-01-25-03 UNIVERSIDAD AUTONOMA DE TAMAULIPAS UNIDAD ACADEMICA MULTIDISCIPLINARIA REYNOSA-AZTLAN NOMBRE DE LA CARRERA QUÍMICO FARMACÉUTICO BIÓLOGO NOMBRE DE LA ASIGNATURA LABORATORIO DE BIOLOGÍA MOLECULAR
Facultad de Ciencias Universidad de Zaragoza
 INSTRUCCIONES BÁSICAS PARA UTILIZAR LA APLICACIÓN DE SOLICITUD DE RESERVA DE AULAS Y OTROS ESPACIOS COMUNES Facultad de Ciencias Universidad de Zaragoza ÍNDICE 1.- BREVE DESCRIPCIÓN...2 2.- CONSULTAR INFORMACIÓN
INSTRUCCIONES BÁSICAS PARA UTILIZAR LA APLICACIÓN DE SOLICITUD DE RESERVA DE AULAS Y OTROS ESPACIOS COMUNES Facultad de Ciencias Universidad de Zaragoza ÍNDICE 1.- BREVE DESCRIPCIÓN...2 2.- CONSULTAR INFORMACIÓN
1. Cuál es el complemento cromosómico diploide de la especie?: a. 2n=3 b. n=3 c. n=6 d. 2n=6
 Prototipo 1er parcial BCM-2016 PROBLEMA 1 El dibujo muestra un esquema del complemento cromosómico de tres individuos de una especie de monos. La célula dibujada de cada individuo, es una célula somática
Prototipo 1er parcial BCM-2016 PROBLEMA 1 El dibujo muestra un esquema del complemento cromosómico de tres individuos de una especie de monos. La célula dibujada de cada individuo, es una célula somática
El genoma humano. Conceptos
 CardioChip El genoma humano. Conceptos Herencia: conjunto de caracteres que los seres vivos reciben de sus progenitores. Gen: unidad mínima de herencia. Compuesto por ADN, una macromolécula formada por
CardioChip El genoma humano. Conceptos Herencia: conjunto de caracteres que los seres vivos reciben de sus progenitores. Gen: unidad mínima de herencia. Compuesto por ADN, una macromolécula formada por
PROGRAMACION EN VISUAL BASIC
 PROGRAMACION EN VISUAL BASIC EJEMPLO 1 En el presenta artículo se explicará la forma de hacer un sencillo programa en el lenguaje de programación VB 6.0 el cual arroja como resultado el valor en ohms de
PROGRAMACION EN VISUAL BASIC EJEMPLO 1 En el presenta artículo se explicará la forma de hacer un sencillo programa en el lenguaje de programación VB 6.0 el cual arroja como resultado el valor en ohms de
