Es necesario entonces, poder caracterizarlas y clasificarlas: Por lo expuesto, los objetivos del presente trabajo son:
|
|
|
- María Josefa Álvarez Serrano
- hace 6 años
- Vistas:
Transcripción
1 OBJETIVOS
2 Objetivos La necesidad de demostrar la producción de β-lactamasas por un microorganismo aislado en el laboratorio clínico, ha llegado a ser de crucial importancia en los últimos años, debido a la emergencia de cepas productoras de nuevos tipos de β-lactamasas y su diversificación. En muchos casos son difíciles de diferenciar entre ellas. Es necesario entonces, poder caracterizarlas y clasificarlas: - según sus características fenotípicas: perfil de actividad, punto isoeléctrico y actividad hidrolítica, - según características genotípicas: PCR con iniciadores específicos para el gen que codifica la β-lactamasa, clonación y secuenciación. Por lo expuesto, los objetivos del presente trabajo son: - Evaluar la incidencia de cepas de enterobacterias, productoras de β-lactamasas plasmídicas, con sensibilidad disminuida a cefalosporinas de tercera generación. - Definir cualitativa y cuantitativamente el fenotipo de resistencia de estas cepas. - Caracterizar e identificar las β-lactamasas implicadas en la resistencia. 43
3 MATERIALES Y METODOS
4 1) Cepas estudiadas Se han estudiado la sensibilidad de un total de 13,800 cepas de la familia Enterobacteriaceae aisladas de muestras clínicas entre los años 1997 y 1999 en el Hospital de la Santa Creu i Sant Pau. De estas 10,057 pertenecieron a las especies de Escherichia coli (7705), Klebsiella pneumoniae (491), Salmonella enterica (913) y Proteus mirabilis (948). A través de la técnica de difusión en agar (siguiendo las recomendaciones de la NCCLS (National Committee for Clinical Laboratory Standards, 1995, 1998 y 1999) se seleccionan 53 cepas por presentar sensibilidad disminuida a cefalosporinas de tercera generación y/o aztreonam: 41 cepas de E. coli, 9 cepas de K. pneumoniae, 2 cepa de S. enterica y 1 cepa de P. mirabilis. El origen clínico de la mayoría de muestras era de orina 35 (67,3%), seguido de sangre 5 (9,6%) (Tabla 3, 6 y 9). Fueron obtenidas por los procedimientos microbiológicos habituales y se identificaron mediante las pruebas bioquímicas convencionales para enterobacterias. 2) Determinación de la sensibilidad a los antibióticos 2.1 Técnica de difusión Se trata de ensayar la sensibilidad de las cepas bacterianas haciéndolas crecer en placas de agar donde se depositan discos de papel inpregnados con los antibióticos a estudiar. Si sembramos en una placa con un medio de cultivo de forma homogénea una bacteria y colocamos unos discos de papel cargados con concentraciones determinadas de cada antibiótico, éste al ser depositado sobre el agar, difunde casi instantáneamente a través del agar, formándose un gradiente radial de concentración del antibiótico alrededor del disco. 47
5 Se incuba la placa a 35ºC-37ºC durante 18 horas y cuando el microorganismo empieza a crecer, este gradiente ya está formado: el microorganismo crecerá dando lugar a un halo de inhibición o zona sin crecimiento, alrededor del disco. En la zona radial del agar en la que empiezan a desarrollarse colonias, estará la concentración inhibitoria mínima (CIM) del antibiótico con el que el disco estaba cargado. Mediremos el diámetro del halo y lo expresaremos en milimetros. Mediante líneas de regresión CIM/halos de inhibición, podremos correlacionar ámbos parámetros. Se utiliza el medio Mueller Hinton (Oxoid, Unipath Limited, Basingstoke, United Kingdon) y los discos (Oxoid) estudiados son: ampicilina (30 µg), piperacilina (100 µg), amoxicilina-ácido clavulánico (30/10 µg), cefazolina (30 µg), cefuroxima (30 µg), cefoxitin (30 µg), cefotaxima (30 µg), ceftazidima (30 µg), cefepima (30 µg), imipenem (10 µg), aztreonam (30 µg), acido nalidíxico (30 µg), ciprofloxacino (5 µg), cloranfenicol (30 µg), tetraciclina (30 µg), estreptomicina (10 µg), kanamicina (30 µg), neomicina (30 µg), gentamicina (30 µg), tobramicina (30 µg), amikacina (30 µg), sulfamidas (300 µg), trimetoprim (30 µg), cotrimoxazol (110 µg), colistina (30 µg), y fosfomicina (50 µg). El tamaño del inóculo, las condiciones de incubación así como los criterios de lectura e interpretación de los resultados fueron los establecidos por el National Committee for Clinical Laboratory Standars (NCCLS) Determinación de la concentración inhibitoria mínima (CIM) El estudio de la sensibilidad bacteriana in vitro se efectúa determinando la concentración inhibitoria mínima (CIM), que es la mínima concentración de antibiótico expresado en µg/ml que inhibe el crecimiento del microorganismo. El estudio de éste parámetro, que cuantifica la sensibilidad, se lleva a cabo mediante técnicas de dilución en medio líquido (o caldo) o en medio sólido. 48
6 2.2.1 Técnica de dilución en medio sólido Se prepara una serie de tubos con cantidades decrecientes de un antibiótico, para conseguir una serie doble progresiva de concentraciones. Se incorporan las diluciones al agar fundido, a temperatura alrededor de 50ºC y se vierten posteriormente en placas de Petri que se dejaron solidificar. Esta técnica permite estudiar simultáneamente la CIM de un gran número de cepas a un antimicrobiano. Se inoculan las placas con una cantidad conocida de los microorganismos a estudiar, con la ayuda de un replicador de Steers (Multipoint inoculator), para obtener una concentración de microorganismo en el agar de 10 4 UFC. Tras la incubación a 35-37ºC durante 18 horas se determina el valor de la CIM del antibiótico para cada microorganismo, que corresponde a la placa con la menor concentración de antibiótico en la que el crecimiento bacteriano está inhibido. Se incluyen en todas las series dos cepas de control E.coli ATCC y E.coli ATCC Los antimicrobianos estudiados son: ampicilina, amoxicilina, ácido clavulánico, ticarcilina (Smith Kline Beecham); piperacilina, cefoperazona (Sigma); tazobactam (Lederle Cyanamid); cefuroxima, ceftazidima (Glaxo S.A.); cefoxitina (Merck Sharp & Dohme); cefotaxima (Hoechst Marion Roussel); cefepime, aztreonam (Bristol Myers Squibb) y cefpodoxima (Sankyo Co). Los disolventes y diluyentes utilizados para cada antibiótico son los recomendados por el fabricante Técnica de microdilución La técnica de microdilución es una técnica de dilución en caldo que utiliza placas de poliestireno con micropocillos en lugar de tubos de ensayo, utilizándo por tanto, volumenes finales menores. 49
7 Se utilizan placas comercializadas (Sensititre R EMIZA 9EF) con 26 antibióticos desecados por placa. La metodología seguida fue la propuesta por el fabricante, siguiendo las normativas del NCCLS. Los antibióticos estudiados y sus concentraciones fueron: ampicilina (1-16 µg/ml), amoxicilina-ácido clavulánico (4/2-16/8 µg/ml), cefuroxima (4-16 µg/ml), cefoxitina (4-16 µg/ml), cefazolina (8 y 16 µg/ml), piperacilina (16-32 µg/ml), ticarcilina (16-64 µg/ml), cefixima (8 y 16 µg/ml), piperacilina/tazobactam (8/4-64/4 µg/ml), gentamicina (1-8 µg/ml), amikacina (4-32 µg/ml), tobramicina (2-8 µg/ml), fosfomicina+g6p (128 µg/ml), imipenem (0,25-8 µg/ml), ampicilina/sulbactam (8/4 y 16/8 µg/ml), meropenem (0,5-8 µg/ml), aztreonam (4-16 µg/ml), ciprofloxacino (0,25-2 µg/ml), norfloxacino (4 y 8 µg/ml), tetraciclina (4 y 8 µg/ml), levofloxacino (0,25-2 µg/ml), trimetropin/sulfametoxazol (0,5/9,5-2/38 µg/ml), cefepime (1-16 µg/ml), cloranfenicol (8 y 16 µg/ml), cefotaxima (0,25-32 µg/ml) y ceftazidima (0,25-16 µg/ml) Método de E-test E test (AB Biodisk, Solna, Sweden) es un método innovador para cuantificar la actividad antimicrobiana, al determinar la CIM por técnica de difusión. Consiste en una tira de plástico no poroso de 5 cm de largo por 5 mm de ancho que incorpora un gradiente predefinido de un antimicrobiano equivalente a 15 concentraciones dobles progresivas. Siguiendo la metodología de difusión, una vez se ha inoculado la placa de agar con el microorganismo, se coloca la tira de E-test sobre la superficie, produciéndose de forma inmediata una difusión del antibiótico desde el soporte de plástico hasta el agar, creándose de este modo a lo largo de la tira un gradiente exponencial de las concentraciones del antimicrobiano. Tras la incubación de las placas, a 35ºC durante 18 horas, se puede observar una zona de inhibición elipsoidal y simétrica. El punto en que el extremo de la zona de inhibición intersecciona con la tira, es el valor de la CIM. 50
8 E-test es un método alternativo para el estudio cuantitativo de la sensibilidad antimicrobiana del que cabe destacar su sencillez y buena correlación con la técnica estándar de dilución. En este estudio se utilizaron tiras de E-test de cefotaxima/cefotaxima-ácido clavulánico y ceftazidima/ceftazidima-ácido clavulánico, valorándose la reducción de la CIM de cefotaxima y ceftazidima en presencia de inhibidores de β-lactamasas como el ácido clavulánico. Dicha reducción de CIM sugeriría la presencia de BLEAs en las cepas estudiadas. 2.3 Prueba de doble difusión con disco (placa de sinergia) Para la detección de β-lactamasas de espectro extendido se emplea la prueba de doble difusión con disco. La placa de agar Mueller Hinton se baña con la cepa a estudiar; en el centro de la placa se coloca un disco de amoxicilina-ácido clavulánico 2:1 (30 µg), y radialmente se colocaron los siguientes discos: cefotaxima (30 µg), ceftazidima (30 µg), aztreonam (30 µg) y cefepime (30 µg). Estos discos son puestos a 30mm del disco de amoxicilina-ácido clavulánico y cuando no se observa sinergia de esta forma se cambió a 25mm. Las placas se incuban a 37ºC por 18 horas. Se considera la prueba positiva cuando se observa un aumento del halo de inhibición en cualquiera de los discos de cefalosporinas de tercera generación o el de aztreonam, en la zona próxima al disco que contiene el ácido clavulánico 75. 3) Procedimientos para el análisis epidemiológico de las cepas Se denomina así, al proceso de analizar aislados múltiples de un especie dada, para determinar si ellos vienen de una sola cepa o de diferentes y múltiples cepas, que están causando alguna infección clínica. De esta forma se puede decir si la serie de aislados obtenidos durante un brote epidémico o durante el curso de una infección en un mismo paciente son clonales, es decir descienden de un precursor común. 51
9 3.1 Técnicas fenotípicas Aquellas técnicas que detectan las características expresadas por los microorganismos. Están limitadas inherentemente por la capacidad del microorganismo para modificar la expresión de los genes subyacentes Biotipado El biotipado se refiere al patrón metabólico expresado por un aislado. Para realizar el biotipado de las cepas de E. coli se utiliza una serie bioquímica 141 con doce test dividido en grupos de tres: un grupo de fermentación del dulcitol, sacarosa y rafinosa; otro grupo producción de lisina descarboxilasa y ornitina descarboxilasa y detección de la β- glucoronidasa; otro de fermentación de la ramnosa y el sorbitol y producción de gas en el tubo de glucosa; y fermentación de la lactosa, adonitol y sorbosa. Luego se va dando puntos por cada reacción positiva: 1 punto para el primer test de cada grupo, 2 puntos para el segundo test y 4 puntos para el tercer test. Los biotipos se expresan en un código de 4 dígitos. La lectura se realiza primero a las 24 horas luego a las 48 horas Antibiotipado El antibiotipado esta dado por el patrón de resistencia de los microorganismos a los diferentes antimicrobianos (ver apartado 2) Serotipado El serotipado se basa en la observación de que microorganismos de la misma especie pueden diferir en los determinantes antigénicos expresados en la superficie celular. El serotipado, de las cepas de Salmonella enterica se realizó en el Servicio de Enterobacterias del Centro Nacional de Microbiología, Instituto Carlos III Majadahonda, Madrid. 52
10 La realización del serogrupo de las cepas de E. coli, se realizó con anticuerpos específicos de los siguientes antígenos O: O:1, O:2, O:4, O:6, O:8, O:9, O:15, O:18 y O:83 La prueba se considera positiva cuando se observa una aglutinación franca al enfrentar el anticuerpo con la cepa problema, negativa cuando no se observa aglutinación y rugoso cuando la cepa autoaglutina. 4) Selección de las cepas estudiadas con cefotaxima Esta selección se realiza por el método de Szybalski 35 que consiste en la utilización de placas de Mueller Hinton con un gradiente determinado de un antibiótico de elección. Primero se debe conocer la CIM de la cepa a seleccionar ya que las placas de Mueller Hinton, en este caso con cefotaxima, se preparan a una concentracción dos veces superior a la CIM de la cepa a estudiar. Las placas se preparan con 18 ml de agar Mueller Hinton a 50ºC más 2 ml de cefotaxima a una concentración diez veces superior a la que se quiere obtener en el volumen final. Agregar la mezcla del agar en la placa petri inclinada (para esto se coloca un lapiz sobre la mesa de trabajo y sobre él se apoya la placa), hasta que el agar quede a 1 cm del borde de la placa que se apoya en el lapiz. Dejar secar, cuando se enfríe, colocar la placa en posición horizontal y agregar agar Mueller Hinton a 50ºC hasta el borde más alto de agar solidificado previamente. Es importante señalar el lado donde hay más agar con antibiótico. Una vez solidificadas las placas, dejar secar en la estufa a 37ºC durante más o menos 1 hora. Luego sembrar en el medio de la placa 100 µl de una suspensión de la cepa en suero fisiológico a 0,8 McFarland y extender uniformemente por toda la placa con asa de vidrio. Incubamos a 37ºC durante 18 horas. El crecimiento que se observa es un gradiente desde el sitio de menor concentración del antibiótico (cefotaxima) al sitio de mayor concentración de antibiótico. A continuación, se seleccionan las colonias más próximas al sitio de mayor concentración del antibiótico. De estas colonias se debe realizar la CIM por el método de dilución en caldo Mueller Hinton ajustado con cationes. Se usa como control la cepa E. coli ATCC Aquellas cuya CIM haya incrementado, se volverán a seleccionar (se utiliza el tubo de mayor concentración de antibiótico donde se observa crecimiento), hasta obtener colonias cuya CIM no pueda subir más, o alcanzar la concentración de 1024 µg/ml. 53
11 De esta forma se seleccionaron 16 cepas de E. coli productoras de β-lactamasa compatible con CTX-M-9 y TEM-1 y 5 cepas de E. coli con β-lactamasa compatible con SHV-2. 5) Determinación del punto isoeléctrico 5.1 Extracción de -lactamasas La extracción de las β-lactamasas se realiza por medio de la lisis celular llevada a cabo mediante sonicación. El contenido resultante corresponde a todo el contenido proteíco celular (incluida la β-lactamasa) y se denomina extracto crudo. Procedimiento: Se siembra una o dos colonias de la cepa, se pone en crecimiento en 5 ml de caldo tripticasa soya (CT) durante 2 horas en agitación a 37ºC; una vez se alcanza el cultivo exponencial (aproximadamente a las 3 horas) se coloca 2 ml de este cultivo en 200 ml de caldo Luria-Bertani, LB (método de Mathew y cols., 1975) 94. Se agrega ampicilina (antibiotico necesario para la inducción de la β-lactamasa) en una concentración de 100 µg/ml y se incuba en agitación durante 18 horas a 37ºC. Se hace un antibiograma del cultivo para comprobar la pureza del mismo así como, la expresión de la β-lactamasa. Se centrifuga el cultivo a 4,000 rpm a 4ºC durante 30 minutos, el sedimento se resuspende en 10 ml de agua bidestilada y la suspensión se centrifuga nuevamente durante 30 minutos a 10,000 rpm a 4ºC. El sedimento se resuspende en 5 ml de agua bidestilada ultrapura (si es <5 mg en 5 ml y si >5 mg en una proporción 1:1) y la suspensión celular se somete a sonicación (Ultrasonicador: Labsonic 200, B. Braun Biotech International), 1 minuto en 15 µm a 4ºC por tres veces, con períodos de descansos intercalados de 30 segundos. Observar una disminución en la turbidez. Luego las células lisadas se centrifugan a 14,000 rpm a 4ºC durante 30 minutos y el sobrenadante, que es el extracto crudo proteíco se conserva a 20ºC. 54
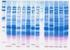
12 5.2 Análisis por la técnica de isoelectroenfoque en gel de poliacrilamida El isoelectroenfoque (IEF) es un método de determinación del pi de una proteína mediante electroforesis, en un gel que contiene un gradiente de ph. Este gradiente de ph se establece con anfolitos. Estas son sustancias anfotéricas de bajo peso molecular que migran en un campo eléctrico hasta llegar a su pi donde su carga eléctrica es zero o neutra. Cuando se utiliza una mezcla de diferentes anfolitos es cuando se crea el gradiente de ph. Cuando se aplica una proteína al gel, esta puede estar cargada positiva o negativamente, dependiendo del ph local de aquel punto del gel. Después de la aplicación de la corriente, la proteína se moverá hacia el ánodo o al cátodo (dependiendo de su carga) hasta que encuentra la región en el gel donde la carga neta será 0 y por lo tanto parará de migrar, el ph de este punto se define como pi. Junto con la proteína problema corren proteínas de pi conocido, lo cual permite la estimación, mediante la correlación de éstas, del pi de nuestra proteína problema. Procedimiento: El análisis por isoelectroenfoque 8 del extracto crudo, se realiza en gel de poliacrilamida con anfólitos de un rango de ph 3,5 a 11 (Pharmacia Biotech 3,5 9,5 y Serva 9-11). El volumen de extracto analizado varía, entre 20µl y 40µl. Las condiciones de la corrida del gel son de 1000V, 50 ma y 2W durante 18 horas. Cuando es necesario, porque no sé define bien alguna β-lactamasa con pi bajo (pi esperados fueron ácidos), sé realizan geles con anfólitos de un rango de ph 3,5 a 9,5 (Pharmacia Biotech 3,5 9,5 y Pharmacia Biotech 4-6), en este caso las condiciones de corrida son las siguientes: voltaje máximo 1500 V, amperaje máximo de 50 ma y una potencia fija de 50W durante 4 horas. La focalización en ambos casos se realiza a 4ºC. La actividad enzimática se revela mediante el método iodométrico con geles de agar con una concentración final de 125 µg/ml de ceftriaxona, para poder observar si la β- lactamasas tiene actividad frente a las cefalosporinas de tercera generación, y luego una vez visualizada la actividad y quitando este gel, se revela con un segundo gel con una concentración final de 125 µg/ml de penicilina. El gel de revelado es superpuesto al gel 55
13 de IEF y en los puntos donde se hayan focalizado las β-lactamasas estas degradarán el anillo β-lactámico del antibiótico con la consecuente liberación de protones que reducirán el ioduro a iodo, este proceso conlleva un cambio de color del almidón que pasará de azul a blanco. Como controles se colocan extractos crudos de las β-lactamasas plasmídicas SHV-1 (p453 pi 7,6), SHV-3 (pud18 pi 7,1), SHV-5 (TR349 pi 8,2), TEM-1 (RL111 pi 5,4), TEM-2 (RL114 pi 5,6), TEM-3 (CF1304 pi 6,3), CTX-M-4 (R893 pi 8,4), CTX-M-9 (MSP492 pi 8,0) y CTX-M-9 y TEM-1 (785-D pi 8,0 y pi 5,4), los cuales se corren paralelamente a los extractos de las cepas problemas. 6) Conjugación Es un método de apareamiento sexual donde el plásmido o el material genético se transmite desde una célula bacteriana donadora hasta otra célula bacteriana receptora, a través de un apéndice especial denominado pili sexual. La célula donadora posee un plásmido (factor F; fertilidad) que permite la síntesis del pilus sexual o pilus conjugativo. La célula receptora está relacionada estrechamente con la donadora y contiene en su superficie lugares de reconocimiento. El material genético pasa a través del punto formado por el pilus dejando en la bacteria donadora generalmente una copia del material genético transferido. Procedimiento: Para la obtención de los transconjugantes se utiliza E. coli BM694 Cla, resistente al ácido nalidíxico, como cepa receptora, cuyo fenotipo se describe en la tabla 20, 21 y 22. Se siembran las cepas donadoras y la receptora en placas de agar sangre, incubar h a 37ºC. Inocular una colonia de cada una de estas placas, en 3 ml de CT durante 2-3 h a 37ºC en agitación, es decir, hasta que esten en la fase de crecimiento exponencial. La conjugación se realiza en medio sólido, poniendo en contacto 50 µl de un cultivo en fase exponencial de crecimiento, tanto de la cepa donadora como de la cepa receptora, en el centro de una placa de agar sangre muy seca. Para evitar que la suspensión se 56
14 expanda en toda la placa se utiliza un disco de papel de filtro estéril (Millipore ), el que se coloca en el centro y sobre el cual se realiza la mezcla. Una vez seca la gota se incuba 18h a 37ºC, evitando mover bruscamente la placa. Pasado ese tiempo de incubación, recoger el crecimiento mediante una asa de siembra y colocar el contenido en un tubo que contiene 2 ml de suero fisiológico. Hacer diluciones de éste, obteniendo unas concentraciones finales en placa de 10-1, 10-2 y Sembrar en placas de Mueller Hinton suplementado con ampicilina 100 µg/ml, cefotaxima 2 µg/ml y ácido nalidíxico 50 µg/ml, lo que permite la selección de las cepas transconjugantes, lo que será diferente para cada conjugación. Incubar h a 37ºC. Hacer un antibiograma y biotipado, para excluir las mutantes naturales, de la cepa transconjugante y además comprobar el punto isoeléctrico (pi). 7) Detección y caracterización de las -lactamasas por amplificación del ADN mediante la reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR) se utiliza para amplificar de forma exponencial un segmento de ADN existente entre dos regiones de secuencia conocida. Se utilizan dos oligonucleótidos como iniciadores para una serie de reacciones en cadena que constan de ciclos de desnaturalización, renaturalización y extensión. Debido a que los productos de un ciclo completo sirven como moldes para el segundo, cada ciclo sucesivo duplica la cantidad del producto del ADN, obteniendo al final del proceso un número de moléculas de ADN de 2 X, siendo X el número de ciclos realizados. En nuestro caso se va a realizar la técnica de la PCR para la amplificación de los genes codificantes de las β-lactamasas y posteriormente, en algunos casos, para la secuenciación. El procedimiento seguido se realiza a partir de muestras que contienen el ADN molde procedente de una extracción de ADN a partir de una colonia. Procedimiento: Los genes bla TEM, bla SHV, bla CTX-M-9, bla AMPC, bla CMY-2, ampr y la región variable de los integrones son amplificados utilizando los iniciadores que se detallan en la tabla 2. 57
15 Las condiciones de amplificación para bla TEM son: desnaturalización durante 4 min a 94ºC, 35 ciclos (desnaturalización 1 min a 94ºC, renaturalización 2 min a 48ºC, extensión 2 min a 72ºC), extensión 10 min a 72ºC y parada de la reacción a 4ºC. El fragmento amplificado a obtener es de 938 pb. Las condiciones de amplificación para bla SHV son: desnaturalización durante 5 min a 95ºC, 30 ciclos (desnaturalización 30 segundos a 94ºC, renaturalización 30 segundos a 68ºC, extensión 50 segundos a 72ºC), extensión 3 min a 72ºC y parada de la reacción a 4ºC. Los iniciadores son los descritos por Nüesch-Inderbinen y cols. en El fragmento amplificado a obtener es de 1017 pb. Se utiliza como cepa control E. coli SHV-1 (J53 R1010) y E. coli SHV-2 (DH5x, MPB-2). Para la amplifiación del gen bla CTX-M-9 se utilizan los iniciadores CTX-M-9 IATG y CTX-M-9 ISTOP descritos por Sabaté y cols. (2000) 145, y las condiciones son: desnaturalización durante 5 min a 94ºC, 30 ciclos (desnaturalización 1 min a 95ºC, renaturalización 1 min a 55ºC, extensión 1 min a 72ºC), extensión 7 min a 72ºC y parada de la reacción a 4ºC. El tamaño del fragmento amplificado a obtener es de 850 pb. Cuando se utilizaron los iniciadores CTX-M-9 EATG y CTX-M-9 ESTOP, descritos por Sabaté 145, las condiciones de amplificación son: desnaturalización durante 5 min a 94ºC, 30 ciclos (desnaturalización 1 min a 95ºC, renaturalización 1 min a 57ºC, extensión 2 min a 72ºC), extensión 7 min a 72ºC y parada de la reacción a 4ºC. El tamaño del fragmento amplificado a obtener es de 1.5 Kb. Se utiliza como cepa control la cepa de E. coli MSP492. Las condiciones de amplificación para bla AMPC, que utiliza los iniciadores degenerados ampc A1 y ampc A2, son los descritos por Marchese y cols en que permiten la amplificación del gen ampc de E. coli, Enterobacter cloacae y Citrobacter freundii así como de otros genes que codifican para β-lactamasas plasmídicas CMY-2, LAT-21, LAT-2 y BIL-1 entre otras. Las condiciones de amplificación son: desnaturalización durante 5 min a 94ºC, 35 ciclos (desnaturalización 30 segundos a 94ºC, renaturalización 30 segundos a 48ºC, extensión 1 min a 72ºC), extensión 10 min a 72ºC y parada de la reacción a 4ºC. El tamaño del fragmento amplificado a obtener es de 561 pb. Como cepa control se utiliza E. coli MI 1443 (cepa sin el gen ampc cromosómico y con el gen ampc de Enterobacter clonado en el plásmido p18ec). Para la amplificación del gen bla CMY-2 se diseñan los iniciadores CMY-2 ATG y CMY-2 STOP y las condiciones de amplificación son: desnaturalización durante 5 min a 94ºC, 30 ciclos (desnaturalización 30 segundos a 94ºC, renaturalización 58
16 30 segundos a 50ºC, extensión 1 min a 72ºC), extensión 10 min a 72ºC y parada de la reacción a 4ºC. El tamaño del fragmento amplificado a obtener es de 1017 pb. Como cepa control se utiliza Citrobacter freundii 70HB (aislada del LCR de un paciente de HSP). Para la amplificación del gen ampr se realiza dos PCRs en una se utiliza los iniciadores amprd y amprmr y en la otra amprd y amprfr; las condiciones en ambos casos son: desnaturalización durante 5 min a 94ºC, 35 ciclos (desnaturalización 30 segundos a 94ºC, renaturalización 30 segundos a 52ºC, extensión 1 min a 72ºC), extensión 10 min a 72ºC y parada de la reacción a 4ºC. El tamaño del fragmento amplificado a obtener es de 550 y 1100 pb, respectivamente. Como cepa control se coloca la misma cepa anterior de C. freundii. Y finalmente para la amplificación de fragmentos internos de las zonas 5'-CS y 3'-CS de los integrones de la clase I se utilizan los iniciadores descritos por Lévesque y cols. en y las condiciones de amplificación son: desnaturalización durante 5 min a 94ºC, 35 ciclos (desnaturalización 1 min a 94ºC, renaturalización 1 min a 55ºC, extensión 5 min a 72ºC con un augmento de 1 seg en cada ciclo), extensión 10 min a 72ºC y parada de la reacción a 4ºC. El tamaño del fragmento amplificado depende en cada caso del número de genes cassettes insertados en el integrón. Como cepa control se utiliza Salmonella enterica serovar Typhimurium 44S (que contenia dos integrones de 1000 pb y 1200 pb). Las concentraciones finales de la mezcla para la reacción de amplificación son: tampón de reacción 1X (2 mm Tris-HCl ph 7.5, 10 mm KCl, 1.5 mm MgCl 2 ), Taq ADN polimerasa 0.03 U (Expand TM High Fidelity, Roche) y 400 mm dinucleótidos trifosfatos (dntps). El ADN que se utiliza como cebador para la amplificación es obtenido a partir de una colonia resuspendida en 100 µl de agua esteril miliq y dicha suspensión es hervida durante 10 min a 95ºC. La cantidad de ADN de la que se parte para cada reacción de amplificación depende del gen en cuestión, siendo de 2 µl para bla TEM y bla SHV, 50 µl para bla CTX-M-9 (tanto cuando se utilizan los iniciadores internos CTX-M- 9 IATG y CTX-M-9 ISTOP como los externos CTX-M-9 EATG y CTX-M-9 ESTOP ), 15 µl para la amplificación de bla AMPC, bla CMY-2 y ampr y finalmente para la ampificación de la región interna de los integrones se utiliza 10 µl de ADN. Como cepas controles se utilizan las mismas que las utilizadas en la técnica de isoelectroenfoque, excepto en aquellos casos donde se detalla la cepa control utilizada. 59
17 Los fragmentos amplificados por PCR son separados mediante la técnica de electroforesis en geles de agarosa al 1.5%, teñidos en bromuro de etidio (0.5 µg/ml) y visualizados con luz ultravioleta (ver apartado 8.2). Todas las reacciones de amplificación se realizan en el termociclador GeneAmp PCR System 2400 (Perkin Elmer, Roche Diagnostic Systems). Tabla 2: Secuencias nucleotidicas de los oligonucleótidos usados para la amplificación por PCR y la secuencia del ADN Iniciadores Secuencia Nucleotidica 5 a 3 Iniciadores usados para la amplificación por PCR TEM 3 1 TEM 4 1 AGT GTC GAC TTA CCA ATG CTT AAT CAG T AAA GAA TTC TAA ATA CAT TCA AAT ATG SHV A SHV B CTX-M-9 IATG CTX-M-9 ISTOP CTX-M-9 EATG CTX-M-9 ESTOP ampc A1 ampc A2 CMY-2 ATG 1 CMY-2 STOP 1 5'-CS 3'-CS CGC CGG GTT ATT CTT ATT TGT CGC TCT TTC CGA TGC CGC CGC CAG TCA GTG ACA AAG AGA GTG CAA CGG ATG ATT CTC GCC GCT GAA GCC CGT CTC TTT GAC TGA CGA CCC TGG AAG AAT AAC TTC CGG CGG GGA ATT CCT (AT)TG CTG CGC (GCT)CT GCT CGG GAT CCC TGC CAG TTT TGA TAA AA ATG ATG AAA AAA TCG TTA TGC TTG CAG CTT TTC AAG AAT GCG C GGC ATC CAA GCA GCA AGC AAG CAG ACT TGA CCT GAT amprd amprmr amprfr GA(CT) AAA C(GT)G TTA AAT TTA TAA TCC AGC CCT TCG GCG GC TTT ATT TGT GCA GCA CCC CG Iniciadores usados en la secuenciación 1 CMY-2 MIG GAC GTT AGG GAT AAA GCC GC P5M P6M P7M P8M TAG TTT GCG CAA CGT TGT TG CTA TTC TCA GAA TGA CTT GG GCC ACA TAG CAG AAC TTT AA TAA AGT TGC AGG ACC ACT TC 1 Estos iniciadores también son utilizados en la secuenciación 60
18 8) Técnicas de análisis del ADN La mayoria de las técnicas aplicadas en este trabajo estan basadas en los protocolos descritos en los libros Molecular cloning: a laboratory manual 147, y Current protocols in molecular biology 5, introduciendo pequeñas modificaciones. 8.1 Endonucleasas de restricción Las endonucleasas de restricción son enzimas, purificadas a partir de hongos o bacterias que presentan una marcada especificidad respecto a secuencias cortas de ADN y que actúan sobre ellas rompiendo los enlaces fosfodiester y por tanto cortando la cadena del ADN. Cada enzima de restricción presenta requerimientos de fuerza iónica y condiciones específicas de reacción, así que en cada caso se tiene que utilizar el tampón de restricción pertinente. Como norma general se digiere el ADN con aproximadamente 5 unidades de enzima por µg de ADN, a una concentración de glicerol del 5% y a la temperatura recomendada, durante un mínimo de 4 horas. Los productos de restricción son analizados en geles de agarosa. Para confirmar la existencia de una β-lactamasa de espectro ampliado derivada de SHV se va a realizar la digestión parcial de los productos de amplificación con los iniciadores específicos de SHV, con la enzima de restricción NheI 116. Esta enzima de restricción reconoce 4 pb y corta en la diana GCT:AGC. Procedimiento: Para realizar la digestión se coloca en un ependorf 25,7 µl de agua miliq estéril, 4 µl de tampón de restricción, 10 µl del ADN amplificado con iniciadores SHV y 0,3 µl de la enzima NheI. Se mezcla e incuba a baño maria a 37ºC sin agitación durante 18 horas. Preparar un gel de agarosa de una concentración final al 1,5% para correr los productos de la digestión. Se utiliza como marcador de peso molecular el Marker VIII (Roche). Los fragmentos que se esperan encontrar cuando hay digestión es de 769 pb y 248 pb, lo 61
19 cual nos indica que no es una β-lactamasa SHV-1, y por lo tanto se puede tratar de una BLEA de la familia SHV, como por ejemplo SHV Separación de fragmentos de ADN en geles de agarosa La electroforesis en geles de agarosa es una técnica útil para la separación analítica o preparativa de fragmentos de ADN de tamaño entre 0,1 y 25 Kb. Esta se basa en la migración unidireccional del ADN a través de una matriz porosa cuando se le aplica un campo eléctrico. La visualización de los fragmentos de ADN se consigue mediante la incorporación al gel de un colorante fluorescente que se intercala entre las dos cadenas de ADN. Este colorante (en nuestro caso el bromuro de etidio) revela la presencia de una banda de ADN o ARN al ser iluminada con luz ultravioleta, de longitud de onda corta (310nm). La técnica es muy sensible, permitiendo detectar en un gel bandas de hasta 5ng de ADN. Esta técnica también permite la cuantificación de bandas de ADN mediante comparación con patrones de concentración conocida. La resolución de los geles de agarosa se modula por la misma concentración de agarosa, permitiendo discriminar entre bandas de peso molecular próximos mediante la variación de concentraciones de agarosa. El peso molecular de las bandas resulta de la interpolación sobre una linea de regresión realizada mediante los datos obtenidos de la migración electroforética de bandas de peso molecular conocidos, donde la migración es proporcional al logaritmo del peso molecular. Todos los productos de la amplificación se van a separar por electroforesis convencional en geles de agarosa. Para preparar un gel de agarosa se mezcla 150 ml de tampón TBE 0,5X (TBE 10X: 89mM Tris-HCl - 2,5 mm EDTA - 89 mm ácido bórico ph 8,3) y 1,2 gr de agarosa (Ultrapure ADN grade agarosa, Bio Rad), para conseguir una concentración final de 0,8%. Una vez calentada esta mezcla y completada su homogenización debe ser teñida con bromuro de etidio (100 mg/ml, esto es 8 µl de bromuro de etidio en 150 ml del gel de agarosa). Esta mezcla se deja enfriar sobre la cubeta de electroforesis, que previamente se ha sellado, y a la que se ha colocado el peine respectivo. La muestra se cargará tras mezclar 18 µl del ADN amplificado con 2 µl de tampón de muestra 10X. Como marcador de peso molecular se utiliza el Marker 62
20 III (21226, 5148, 4973, 4268, 3530, 2027, 1904, 1584, 1375, 947, 831 y 564 pares de bases) y VIII (1114, 900, 692, 501, 489, 404, 320, 242, 190, 147, 124, 110, 67 y 37 pares de bases), ambos de Roche. Una vez cargados los pocillos en el gel de agarosa, se debe conectar la fuente de poder (Bio Rad) y aplicar un voltaje de 70V durante aproximadamente 2 horas. El bromuro de etidio intercalado entre las bases de ADN nos permitirá visualizar y fotografiar el gel mediante un transiluminador de luz ultravioleta. 9) Secuenciación directa de los productos de la PCR Para llevar a cabo las reacciones de secuenciación del ADN se va a utilizar el método basado en el marcaje con fluorocromos, en nuestro caso el marcaje está en los iniciadores. Este método fue descrito por Sanger y cols. en , y utiliza una mezcla de desoxinucleótidos y dideoxinucleótidos. De esta forma primero se van incorporando desoxinucleótidos, a la copia del ADN molde, pero cuando se incorpora un dideoxinucleótido, el fragmento de ADN que se estaba fabricando queda interrumpido. De esta manera, separando los fragmentos generados en la reacción de secuenciación a través de una electroforesis en gel de poliacrilamida y determinando la fluorescencia, a través de un rayo láser, se podrá determinar la secuencia de nucleótidos. La secuenciación de los productos resultantes de las PCR de CMY-2 y TEM, se realiza mediante el sistema automático ALFexpress (Amersham Pharmacia Biotech, Uppsala, Sweden). Los iniciciadores que se utilizan están detallados en la tabla 2. Procedimiento: Se purifica el fragmento de ADN amplificado para así eliminar los iniciadores y que estos no puedan interferir en la reacción de secuenciación. Para esto se utilizan dos enzimas hidrolíticas: la fosfatasa alcalina y la exonucleasa I (PCR Product Pre- Sequencing Kit, USB Corporation). La fosfatasa alcalina remueve los remanentes de los dntps y la exonucleasa I remueve los iniciadores residuales de cadena simple producida por la PCR. Por cada 5 µl de amplificado de la PCR colocar 1 µl de fosfatasa alcalina y 1 µl de exonucleasa I, mezclar e incubar a 37ºC durante 15 minutos. Luego 63
21 inactivar las enzimas por calor a 80ºC durante 15 minutos y guardar a 4ºC. El ADN está ahora listo para ser secuenciado. Las reacciones de secuencia se realizan con el kit fmol ADN Sequencing System (Promega, USA). 1). Rotular cuatro eppendorfs con las letras A, C, G y T, por muestra a secuenciar, y depositar en cada uno de ellos 2 µl de la mezcla de d/ddntp respectivo (d/ddatp, d/ddctp, d/ddgtp y d/ddttp). 2) Por cada muestra a secuenciar se prepara en dos tubos eppendorf la siguiente mezcla: entre 4 y 40 fmols del ADN molde de la presecuencia (esta cantidad se determina con el GeneQuant de Amersham Pharmacia Biotech o utilizando geles de agarosa); 1,5 µl de iniciador marcado de una concentración de 1,5 pmols/µl (en uno el iniciador de la secuencia directa y en el otro de la secuencia inversa); 5 µl del tampón de la reacción; 1 µl de la Taq polimerasa; y agua miliq hasta completar 16 µl. 3) En uno de los juegos de eppendorfs con los 2 µl de d/ddntps colocar 4 µl de la mezcla con el iniciador marcado para la secuencia directa y en el otro juego de cuatro eppendorfs colocar 4 µl de la mezcla con el iniciador marcado para la secuencia inversa. 4) Se coloca en el termociclador a 30 ciclos de: 95ºC durante 30 segundos, luego, dependiendo de la temperatura de hibridación del iniciador que se utiliza, se coloca a dicha temperatura durante 30 segundos y finalmente a 70ºC durante un minuto. Al final del programa colocar a 4ºC. 5) Parar la reacción de secuenciación añadiendo 3 µl de la solución stop (formamida 100% desionizada y dextran blue 2000). Se guarda a 20ºC hasta su utilización. Para realizar el gel de secuenciación primero debe prepararse los vidrios lavándolos con un detergente catiónico (para evitar la mínima interacción con el rayo láser). Enjuagar abundantemente con agua destilada, secar y repasar con papel de celulosa impregnado con etanol al 70%. Se debe hacer lo mismo con los separadores y el peine de los pocillos. Preparar la solución fijadora con 1 ml de etanol al 100%, 250 µl ácido acético al 10%, 3 µl de bind-silane (Amersham Pharmacia Biotech). Esto se realiza para fijar la acrilamida a los vidrios y permitir así que los pocitos mantengan su forma. Con la solución fijadora, se unta 5 cm del vidrio exterior y 3 cm del vidrio interior mediante un papel de celulosa. Montar los vidrios y limpiar las superficies exteriores con alcohol. Utilizar el ReproGel High Resolution (Amersham Pharmacia Biotech), cuando la 64
22 secuencia que se quiere obtener es de 450 pb aproximadamente o el ReproGel Long Read (Amersham Pharmacia Biotech) cuando lo que se va a obtener es una secuencia de aproximadamente 750 pb. En el primer caso, se utiliza espaciadores de 0,5 mm y en el segundo caso se utiliza los espaciadores de 0,3 mm. Volcar el contenido del gel entre los vidrios teniendo en cuenta que no se formen burbujas. Colocar el peine y aplicar luz ultravioleta durante 10 minutos. Para realizar la secuenciación, iniciar el programa ALFwin. Se inicia el secuenciador ALFexpress (Pharmacia Biotech, Uppsala, Sweden). Programar el secuenciador con las condiciones necesarias para la corrida de un gel High Resolution o Long Read, en función de la secuencia que se espera obtener. Las condiciones de corrida van a ser para High Resolution: 1500 V, 60 ma, 25 W, 55ºC durant 450 min y para Long Read: 1500 V, 60 ma, 25 W, 55ºC durant 700 min. Situar el gel de poliacrilamida en el ALFexpress y preparar 1 L de tampon de electroforesis TBE 0,5X por cubeta. Alinear la luz láser y una vez que el gel ha llegado a los 55ºC, retirar el peine y limpiar los pocillos de los restos de acrilamida mediante una jeringa que inyecta tampón de cubeta a presión encima de cada pocillo. Inmediatamente desnaturalizar las muestras 3 minutos a 70ºC y colocar a 4ºC. Cargar el gel con todo el volumen de las muestras, colocar los electrodos y comenzar la corrida electroforética. La lectura de la secuencia se realiza a través del software ALFwin Analysis Sequence (Amersham Pharmacia Biotech, Uppsala, Sweden). 10) Análisis computacional del ADN y de las secuencias aminoácidicas deducidas Para analizar las diferentes secuencias de ADN, se va ha utilizar el programa DNAstar (1999) que va a permitir el alineamiento múltiple de todas las secuencias nucleotídicas obtenidas y las aminoacídicas deducidas. Se va a utilizar el programa BLAST utilizando el software disponible vía Internet en la red del "National Center for Biothechnology Information" ( y en la red "The Pedro's Biomolecular Research Tools" ( para la investigación de secuencias de ADN y proteínas homólogas. 65
TEMA 12. Pruebas de sensibilidad a los antimicrobianos
 TEMA 12 Pruebas de sensibilidad a los antimicrobianos Tema 12. Pruebas de sensibilidad a los antimicrobianos 1. Nacimiento de la era de los antibióticos 2. Pruebas de sensibilidad a los antimicrobianos
TEMA 12 Pruebas de sensibilidad a los antimicrobianos Tema 12. Pruebas de sensibilidad a los antimicrobianos 1. Nacimiento de la era de los antibióticos 2. Pruebas de sensibilidad a los antimicrobianos
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
Detección de mutaciones en el gen rpob de M. tuberculosis 8 MATERIALES Y MÉTODOS
 8 MATERIALES Y MÉTODOS 8.2 CEPAS DE M. TUBERCULOSIS. Se utilizó 1 cepa control y se analizaron un total de 13 extracciones diferentes de DNA de cepas de M. tuberculosis previamente aisladas de muestras
8 MATERIALES Y MÉTODOS 8.2 CEPAS DE M. TUBERCULOSIS. Se utilizó 1 cepa control y se analizaron un total de 13 extracciones diferentes de DNA de cepas de M. tuberculosis previamente aisladas de muestras
BETALACTAMASAS COMO MECANISMO DE RESISTENCIA EN BACTERIAS GRAM NEGATIVAS Y SU DETECCIÓN POR PCR CONVENCIONAL
 BETALACTAMASAS COMO MECANISMO DE RESISTENCIA EN BACTERIAS GRAM NEGATIVAS Y SU DETECCIÓN POR PCR CONVENCIONAL SANDRA YAMILE SAAVEDRA ROJAS Bacterióloga UCMC MSc Microbiología UN Laboratorio de Microbiología
BETALACTAMASAS COMO MECANISMO DE RESISTENCIA EN BACTERIAS GRAM NEGATIVAS Y SU DETECCIÓN POR PCR CONVENCIONAL SANDRA YAMILE SAAVEDRA ROJAS Bacterióloga UCMC MSc Microbiología UN Laboratorio de Microbiología
PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS. Es un ácido capaz de formar sales con iones cargados
 Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006
 IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
GUÍA DE TRATAMIENTO ANTIMICROBIANO EMPÍRICO DE MICROORGANISMOS MULTIRRESISTENTES
 GUÍA DE TRATAMIENTO ANTIMICROBIANO EMPÍRICO DE MICROORGANISMOS MULTIRRESISTENTES Comisión de Infecciones y Terapéutica Antimicrobiana Hospital Universitario Basurto Marzo 2013 1 TRATAMIENTO EMPÍRICO DE
GUÍA DE TRATAMIENTO ANTIMICROBIANO EMPÍRICO DE MICROORGANISMOS MULTIRRESISTENTES Comisión de Infecciones y Terapéutica Antimicrobiana Hospital Universitario Basurto Marzo 2013 1 TRATAMIENTO EMPÍRICO DE
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
5. MATERIALES Y MÉTODOS
 5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS
 Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS DICIEMBRE 2006 Tratamiento de Imágenes por Computadora Proyecto
Universidad de la República Oriental del Uruguay Facultad de Ingeniería Instituto de Ingeniería Eléctrica MANUAL DE USUARIO ELECTROFORESIS DICIEMBRE 2006 Tratamiento de Imágenes por Computadora Proyecto
Objetivos. S Determinar la presencia de bacterias coliformes en una muestra de agua
 Análisis del agua S Objetivos S Determinar la presencia de bacterias coliformes en una muestra de agua Introducción S Las tres pruebas básicas para determinar la presencia de bacterias coliformes (asociada
Análisis del agua S Objetivos S Determinar la presencia de bacterias coliformes en una muestra de agua Introducción S Las tres pruebas básicas para determinar la presencia de bacterias coliformes (asociada
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR).
 Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
- Clonar un gen (molde ADN o ARN)
 APLICACIONES PCR Aplicaciones de la PCR - Clonar un gen (molde ADN o ARN) - Secuencia conocida - Genes homólogos en especies próximas (primers degenerados) - Información de secuencia proteica (primers
APLICACIONES PCR Aplicaciones de la PCR - Clonar un gen (molde ADN o ARN) - Secuencia conocida - Genes homólogos en especies próximas (primers degenerados) - Información de secuencia proteica (primers
Proyecto de Fin de Cursos
 Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Universidad de la República Oriental del Uruguay Proyecto de Fin de Cursos ELECTROFORESIS EN GEL DE MOLÉCULAS DE ADN Noviembre de 2006 Carolina Etchart C.I. 3.757.708-8 Marcelo Lavagna C.I. 3.508.715-8
Técnicas de Estudio de las células
 Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
Técnicas de Estudio de las células Microscopia Preparaciones permanentes: Fijación Deshidratación Inclusión Corte Fijación: Acidos, solventes orgánicos como alcohol, aldehídos (Formaldehído, glutaraldehídos)
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN
 INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
INTERPRETACIÓN DE GELES DE DNA DIGERIDOS CON ENZIMAS DE RESTRICCIÓN El empleo de las enzimas de restricción y los plásmidos es habitual en el laboratorio de Biología Molecular. Con este ejercicio se pretende
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015
 CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
PRÁCTICAS DE MICROBIOLOGÍA CLÍNICA (CURSO )
 PRÁCTICAS DE MICROBIOLOGÍA CLÍNICA (CURSO 2012 2013) PRÁCTICAS A REALIZAR ANÁLISIS CUALITATIVO DE ORINA PRUEBA DE BAUER KIRBY (ANTIBIOGRAMA) PRUEBA DE EPSILON (DETERMINACIÓN DE LA CMI) ANÁLISIS CUALITATIVO
PRÁCTICAS DE MICROBIOLOGÍA CLÍNICA (CURSO 2012 2013) PRÁCTICAS A REALIZAR ANÁLISIS CUALITATIVO DE ORINA PRUEBA DE BAUER KIRBY (ANTIBIOGRAMA) PRUEBA DE EPSILON (DETERMINACIÓN DE LA CMI) ANÁLISIS CUALITATIVO
RESULTADOS Y DISCUSIÓN
 RESULTADOS Y DISCUSIÓN Estimación de la Concentración Proteica del Filtrado de Cultivo de Mycobacterium tuberculosis H37Rv La curva estándar con la que se estimó la concentración proteica del filtrado
RESULTADOS Y DISCUSIÓN Estimación de la Concentración Proteica del Filtrado de Cultivo de Mycobacterium tuberculosis H37Rv La curva estándar con la que se estimó la concentración proteica del filtrado
Caracteristicas del ADN. Se rompen con las siguientes condiciones -Temperaturas >90 - ph >10.5
 PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
LA NUEVA BIOTECNOLOGÍA
 LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
LA NUEVA BIOTECNOLOGÍA Ingeniería genética: técnicas que permiten manipular la información genética de un ser vivo. TECNOLOGÍA TRADICIONAL DEL ADN RECOMBINANTE CLONACIÓN DE GENES: Obtención de muchas copias
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO
 SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
PRUEBAS DE SENSIBILIDAD A AGENTES ANTIMICROBIANOS
 PRACTICA Nº2 DE MALALTIES INFECCIOSES I PRUEBAS DE SENSIBILIDAD A AGENTES ANTIMICROBIANOS Introducción La determinación de la sensibilidad a los agentes antimicrobianos es una de las principales funciones
PRACTICA Nº2 DE MALALTIES INFECCIOSES I PRUEBAS DE SENSIBILIDAD A AGENTES ANTIMICROBIANOS Introducción La determinación de la sensibilidad a los agentes antimicrobianos es una de las principales funciones
DETECCIÓN DE C. jejuni EN HAMBURGUESA DE POLLO POR PCR TRADICIONAL PRT-712.04-082
 Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Práctico: Genética bacteriana 2007.
 Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
 EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS Los hongos poseen un genoma complejo consistente en: ADN nuclear (n ADN) ADN mitocondrial (mt ADN) en algunos casos ADN plasmídico EXTRACCIÓN DE ADN DE HONGOS FILAMENTOSOS
ISP Instituto de Salud Pública de Chile Bacteriología
 Instituto de Salud Pública de Chile Bacteriología Métodos de Susceptibilidad Verificaciones - Errores - Actualizaciones NCCLS. TM M. Soledad Prat Agosto 2004 TEMAS A DESARROLLAR Control de Calidad: Acciones
Instituto de Salud Pública de Chile Bacteriología Métodos de Susceptibilidad Verificaciones - Errores - Actualizaciones NCCLS. TM M. Soledad Prat Agosto 2004 TEMAS A DESARROLLAR Control de Calidad: Acciones
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
8. PRUEBAS DE SUSCEPTIBILIDAD PARA Mycobacterium tuberculosis. Estudios científicos han determinado que al menos el 1 % de los bacilos tuberculosos
 8. PRUEBAS DE SUSCEPTIBILIDAD PARA Mycobacterium tuberculosis. Estudios científicos han determinado que al menos el 1 % de los bacilos tuberculosos de un paciente determinado son resistentes in Vitro a
8. PRUEBAS DE SUSCEPTIBILIDAD PARA Mycobacterium tuberculosis. Estudios científicos han determinado que al menos el 1 % de los bacilos tuberculosos de un paciente determinado son resistentes in Vitro a
TEMA 14. Métodos inmunológicos para la identificación microbiana
 TEMA 14 Métodos inmunológicos para la identificación microbiana Tema 14. Métodos inmunológicos para la identificación microbiana 1. Introducción 2. Detección de antígenos 2.1. Obtención de anticuerpos
TEMA 14 Métodos inmunológicos para la identificación microbiana Tema 14. Métodos inmunológicos para la identificación microbiana 1. Introducción 2. Detección de antígenos 2.1. Obtención de anticuerpos
INFORME DE RESULTADOS
 INFORME DE RESULTADOS Los análisis realizados al producto con Aloe vera, se desarrollaron de acuerdo a lo establecido en CCAYAC-P-062, para determinar la dilución correspondiente con las 3 cepas analizadas
INFORME DE RESULTADOS Los análisis realizados al producto con Aloe vera, se desarrollaron de acuerdo a lo establecido en CCAYAC-P-062, para determinar la dilución correspondiente con las 3 cepas analizadas
3. MATERIALES Y MÉTODOS
 3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
3. MATERIALES Y MÉTODOS El proceso general llevado a cabo en la presente tesis se ilustra en el siguiente esquema: Exudado uretral/ cervical y/o biopsias Extracción del ADN PCR Digestión Algoritmo computacional:
41. Comprobación de colonias transformantes mediante PCR y electroforesis
 41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
1. Contaminación de agua y alimentos por microorganismos patógenos 1
 INTRODUCCIÓN GENERAL 1. Contaminación de agua y alimentos por microorganismos patógenos 1 2. Papel del agua en la transmisión de enfermedades infecciosas 2 2.1. Las aguas residuales 2 2.2. El agua potable
INTRODUCCIÓN GENERAL 1. Contaminación de agua y alimentos por microorganismos patógenos 1 2. Papel del agua en la transmisión de enfermedades infecciosas 2 2.1. Las aguas residuales 2 2.2. El agua potable
DETECCIÓN DE RESISTENCIA BACTERIANA POR MÉTODOS MOLECULARES EN BACTERIAS GRAM. Dra. Norma Velázquez Guadarrama
 DETECCIÓN DE RESISTENCIA BACTERIANA POR MÉTODOS MOLECULARES EN BACTERIAS GRAM POSITIVAS Y NEGATIVAS. Dra. Norma Velázquez Guadarrama Laboratorio de Investigación en Bacteriología Antibiótico Toda sustancia
DETECCIÓN DE RESISTENCIA BACTERIANA POR MÉTODOS MOLECULARES EN BACTERIAS GRAM POSITIVAS Y NEGATIVAS. Dra. Norma Velázquez Guadarrama Laboratorio de Investigación en Bacteriología Antibiótico Toda sustancia
PROCEDIMIENTO DETECCIÓN Y ENUMERACIÓN POR NUMERO MAS PROBABLE (NMP) CON ETAPA DE PRE- ENRIQUECIMIENTO DE ENTEROBACTERIACEAE EN ALIMENTOS
 Página 1 de 5 1. OBJETIVO Estimar el número de Enterobacteriaceae presentes en el alimento. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a todas las muestras de alimentos de consumo humano
Página 1 de 5 1. OBJETIVO Estimar el número de Enterobacteriaceae presentes en el alimento. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a todas las muestras de alimentos de consumo humano
ANEXOS. Anexo 1 Transformación mediante electroporación
 Anexo 1 Transformación mediante electroporación ANEXOS En este método de transformación de E. coli se requieren células electrocompetentes. La preparación de estas células consta de los siguientes pasos:
Anexo 1 Transformación mediante electroporación ANEXOS En este método de transformación de E. coli se requieren células electrocompetentes. La preparación de estas células consta de los siguientes pasos:
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
BIORREACTRES. Estudios básicos y aplicados sobre procesos de fermentación
 BIORREACTRES Ing. en Alimentos Estudios básicos y aplicados sobre procesos de fermentación Mg. Anahí V. Cuellas Docente investigadora Universidad Nacional de Quilmes TRABAJO PRACTICO Obtención de enzimas
BIORREACTRES Ing. en Alimentos Estudios básicos y aplicados sobre procesos de fermentación Mg. Anahí V. Cuellas Docente investigadora Universidad Nacional de Quilmes TRABAJO PRACTICO Obtención de enzimas
GUIA DE PROBLEMAS DE BIOLOGÍA MOLECULAR BIOQUÍMICA- AÑO 2016 PROBLEMAS DE CLONADO, TRANSFORMACIÓN, PCR E HIBRIDACIÓN MOLECULAR
 GUIA DE PROBLEMAS DE BIOLOGÍA MOLECULAR BIOQUÍMICA- AÑO 2016 PROBLEMAS DE CLONADO, TRANSFORMACIÓN, PCR E HIBRIDACIÓN MOLECULAR 1) Se desea clonar en el sitio EcoRI del puc19 un inserto de 800 pb cortado
GUIA DE PROBLEMAS DE BIOLOGÍA MOLECULAR BIOQUÍMICA- AÑO 2016 PROBLEMAS DE CLONADO, TRANSFORMACIÓN, PCR E HIBRIDACIÓN MOLECULAR 1) Se desea clonar en el sitio EcoRI del puc19 un inserto de 800 pb cortado
PLANIFICACIÓN DE ASIGNATURA
 PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
PLANIFICACIÓN DE ASIGNATURA I IDENTIFICACION GENERAL DE LA ASIGNATURA CARRERA BIOQUIMICA DEPARTAMENTO BIOLOGIA ASIGNATURA BIOLOGIA MOLECULAR II CÓDIGO 1681 PRERREQUISITOS Biología Molecular I CREDITOS
PRÁCTICA 1: MÉTODOS DE RUPTURA CELULAR
 PRÁCTICA 1: MÉTODOS DE RUPTURA CELULAR FUNDAMENTO TEÓRICO Para estudiar la estructura y/o las funciones metabólicas de los distintos compartimentos celulares hay que aislarlos del resto de los orgánulos.
PRÁCTICA 1: MÉTODOS DE RUPTURA CELULAR FUNDAMENTO TEÓRICO Para estudiar la estructura y/o las funciones metabólicas de los distintos compartimentos celulares hay que aislarlos del resto de los orgánulos.
DESARROLLO COGNOSCITIVO Y APRENDIZAJE
 Prof. Jhonis Valbuena Cátedra: Análisis Instrumental La Electrofóresis es un método de la Química Analítica que utiliza corriente eléctrica controlada con la finalidad de separar biomolèculas según su
Prof. Jhonis Valbuena Cátedra: Análisis Instrumental La Electrofóresis es un método de la Química Analítica que utiliza corriente eléctrica controlada con la finalidad de separar biomolèculas según su
Por qué en un resultado positivo siempre cambia a amarillo intenso pero solo algunos emiten fluorescencia?
 Biología 3725 L COLILERT Por qué en un resultado positivo siempre cambia a amarillo intenso pero solo algunos emiten fluorescencia? ENTEROLERT Cuál es el propósito de utilizar enterolert, que nos dice
Biología 3725 L COLILERT Por qué en un resultado positivo siempre cambia a amarillo intenso pero solo algunos emiten fluorescencia? ENTEROLERT Cuál es el propósito de utilizar enterolert, que nos dice
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
CONTROL EN CONTINUO DE SISTEMAS DE DESINFECCIÓN
 CONTROL EN CONTINUO DE SISTEMAS DE DESINFECCIÓN OBJETIVOS I. Caracterizar la carga microbiana presente en distintos puntos de la EDAR y comprobar su variabilidad. II. Estudiar la viabilidad del uso de
CONTROL EN CONTINUO DE SISTEMAS DE DESINFECCIÓN OBJETIVOS I. Caracterizar la carga microbiana presente en distintos puntos de la EDAR y comprobar su variabilidad. II. Estudiar la viabilidad del uso de
 Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Anexo Conservar a 4 o C.
 Anexo 1 Purificación de ADN viral 1. Partir de un sobrenadante de un cultivo celular infectado y clarificado. 2. Concentrar las formas brotantes de los baculovirus por centrifugación (centrifugrar 1,5
Anexo 1 Purificación de ADN viral 1. Partir de un sobrenadante de un cultivo celular infectado y clarificado. 2. Concentrar las formas brotantes de los baculovirus por centrifugación (centrifugrar 1,5
INCIDENCIA Y SENSIBILIDAD DE LA POBLACIÓN MICROBIANA AEROBIA EN EL RÍO TAMBRE. Encontro Galego-Portugues Química. A Coruña 2002
 Título INCIDENCIA Y SENSIBILIDAD DE LA POBLACIÓN MICROBIANA AEROBIA EN EL RÍO TAMBRE Rodríguez 1, A.J., Díaz 1, M.T., Gómez 1, M.J., Araujo 2, M., Garrido 2, M.J. Encontro Galego-Portugues Química. A Coruña
Título INCIDENCIA Y SENSIBILIDAD DE LA POBLACIÓN MICROBIANA AEROBIA EN EL RÍO TAMBRE Rodríguez 1, A.J., Díaz 1, M.T., Gómez 1, M.J., Araujo 2, M., Garrido 2, M.J. Encontro Galego-Portugues Química. A Coruña
CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL
 LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
3. MATERIALES Y MÉTODOS. 3.1 Muestreo de agua en Hermosillo, Sonora.
 3. MATERIALES Y MÉTODOS 3.1 Muestreo de agua en Hermosillo, Sonora. Se hizo un muestreo previo de agua en Hermosillo, Sonora para determinar los niveles de arsénico en agua potable y seleccionar aquellas
3. MATERIALES Y MÉTODOS 3.1 Muestreo de agua en Hermosillo, Sonora. Se hizo un muestreo previo de agua en Hermosillo, Sonora para determinar los niveles de arsénico en agua potable y seleccionar aquellas
ELECTROFORESIS AVANZADA
 Ref. ELECAVANZADA (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref. ELECAVANZADA (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa
 38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
Documentos de Referencia para Diagnóstico de Semilla de Papa de Canadá
 SECRETARIA DE AGRICULTURA, GANADERÍA. DESARROLLO RURAL, PESCA Y ALIMENTACIÓN Dirección General de Sanidad Vegetal Centro Nacional de Referencia Fitosanitaria Subdirección de Diagnóstico Fitosanitario Documentos
SECRETARIA DE AGRICULTURA, GANADERÍA. DESARROLLO RURAL, PESCA Y ALIMENTACIÓN Dirección General de Sanidad Vegetal Centro Nacional de Referencia Fitosanitaria Subdirección de Diagnóstico Fitosanitario Documentos
Procedimientos en Microbiología Clínica
 Procedimientos en Microbiología Clínica Recomendaciones de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica Editor: Juan J. Picazo Coordinador: José A. García Rodríguez Rafael Cantón
Procedimientos en Microbiología Clínica Recomendaciones de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica Editor: Juan J. Picazo Coordinador: José A. García Rodríguez Rafael Cantón
QUE ES? Es la búsqueda de anticuerpos preformados contra los linfocitos de un posible donante en el suero de un paciente.
 CROSS MATCH QUE ES? Es la búsqueda de anticuerpos preformados contra los linfocitos de un posible donante en el suero de un paciente. Pueden estar específicamente dirigidos contra los antígenos HLA o contra
CROSS MATCH QUE ES? Es la búsqueda de anticuerpos preformados contra los linfocitos de un posible donante en el suero de un paciente. Pueden estar específicamente dirigidos contra los antígenos HLA o contra
2. INFORMACIÓN DEL PERMISO
 1. INFORMACIÓN DEL CERTIFICADO Número de certificado: 14CDA38EEC5 Fecha de la última actualización del conjunto de datos: 2015-04-21 URL del conjunto de datos: http://ipt.sibcolombia.net/crsib/resource.do?r=255_ptar_20150420
1. INFORMACIÓN DEL CERTIFICADO Número de certificado: 14CDA38EEC5 Fecha de la última actualización del conjunto de datos: 2015-04-21 URL del conjunto de datos: http://ipt.sibcolombia.net/crsib/resource.do?r=255_ptar_20150420
Instituto Nacional. de Salud PROTOCOLO PARA DETECCION DE KPC EN ENTEROBACTERIAS
 PROTOCOLO PARA DETECCION DE KPC EN ENTEROBACTERIAS Fuente: Servicio Antimicrobianos, de Enfermedades Infecciosas, INEI ANLIS Dr. Carlos G. Malbrán 1. ANTECEDENTES Las infecciones por enterobacterias productoras
PROTOCOLO PARA DETECCION DE KPC EN ENTEROBACTERIAS Fuente: Servicio Antimicrobianos, de Enfermedades Infecciosas, INEI ANLIS Dr. Carlos G. Malbrán 1. ANTECEDENTES Las infecciones por enterobacterias productoras
En qué contextos está indicado el tratamiento de la infección por enterica no tifoidea y cuáles son los antimicrobianos de elección?
 ADQUISICIÓN IN VIVO, TRAS TRATAMIENTO CON AMOXICILINA/CLAVULÁNICO, DE UNA AMPC PLASMÍDICA POR UN AISLADO DE SALMONELLA TYPHIMURIUM MULTIRRESISTENTE Y PORTADOR DE INTEGRONES, RECUPERADO DE UNA NIÑA CON
ADQUISICIÓN IN VIVO, TRAS TRATAMIENTO CON AMOXICILINA/CLAVULÁNICO, DE UNA AMPC PLASMÍDICA POR UN AISLADO DE SALMONELLA TYPHIMURIUM MULTIRRESISTENTE Y PORTADOR DE INTEGRONES, RECUPERADO DE UNA NIÑA CON
TEMA 3 EL DIAGNÓSTICO MICROBIOLÓGICO
 TEMA 3 EL DIAGNÓSTICO MICROBIOLÓGICO FUNDAMENTOS DEL DIAGNÓSTICO DE LAS INFECCIONES DE VIAS URINARIAS METODOLOGÍA UTILIZADA PARA EL DIAGNÓSTICO DE INFECCION URINARIA DIAGNÓSTICO MICROBIOLÓGICO DIAGNOSTICO
TEMA 3 EL DIAGNÓSTICO MICROBIOLÓGICO FUNDAMENTOS DEL DIAGNÓSTICO DE LAS INFECCIONES DE VIAS URINARIAS METODOLOGÍA UTILIZADA PARA EL DIAGNÓSTICO DE INFECCION URINARIA DIAGNÓSTICO MICROBIOLÓGICO DIAGNOSTICO
SECUENCIACIÓN. 1.1.1. Purificación mediante columnas. 1.1.2. Protocolos manuales.
 SECUENCIACIÓN En la Unidad se realiza la secuenciación automática del DNA mediante electroforesis capilar y mediante el uso de la química BigDye Terminator v3.1 de Applied Biosystems. Todas las muestras
SECUENCIACIÓN En la Unidad se realiza la secuenciación automática del DNA mediante electroforesis capilar y mediante el uso de la química BigDye Terminator v3.1 de Applied Biosystems. Todas las muestras
PRÁCTICO N 3. Técnicas para la observación, aislamiento y conservación de microorganismos
 PRÁCTICO N 3 Técnicas para la observación, aislamiento y conservación de microorganismos Objetivos Conocer el manejo del microscopio óptico, sus partes y utilidad en el laboratorio de microbiología. Conocer
PRÁCTICO N 3 Técnicas para la observación, aislamiento y conservación de microorganismos Objetivos Conocer el manejo del microscopio óptico, sus partes y utilidad en el laboratorio de microbiología. Conocer
CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA. Grado en Medicina
 DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Electroforesis de proteínas en geles de poliacrilamida
 Electroforesis de proteínas en geles de poliacrilamida Electroforesis de proteínas en geles de poliacrilamida: Introducción y técnica básica La electroforesis de proteínas en geles con una matriz de poliacrilamida,
Electroforesis de proteínas en geles de poliacrilamida Electroforesis de proteínas en geles de poliacrilamida: Introducción y técnica básica La electroforesis de proteínas en geles con una matriz de poliacrilamida,
Pruebas de susceptibilidad antimicrobiana
 Pruebas de susceptibilidad antimicrobiana CONSIDERACIONES GENERALES Todas las pruebas que se discutirán, dependen del cultivo bacteriano in vitro Requieren de un tiempo relativamente largo para la obtención
Pruebas de susceptibilidad antimicrobiana CONSIDERACIONES GENERALES Todas las pruebas que se discutirán, dependen del cultivo bacteriano in vitro Requieren de un tiempo relativamente largo para la obtención
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 1 Virología y Micología Veterinaria Trabajo Práctico No. 4 ELECTROFORESIS La electroforesis es una técnica utilizada para separar grandes moléculas por tamaño a través de la aplicación de un campo eléctrico
1 Virología y Micología Veterinaria Trabajo Práctico No. 4 ELECTROFORESIS La electroforesis es una técnica utilizada para separar grandes moléculas por tamaño a través de la aplicación de un campo eléctrico
FRAGMENTO OLIGO 5-3 Hebra SECUENCIA 5' - - > 3' TAMAÑO (pb) F1. hmtl 569 L AACCAAACCCCAAAGACACC hmth2982 H CTGATCCAACATCGAGGTCG
 ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
TEMA 3: Expresión Génica
 TEMA 3: Expresión Génica Genómica Estructural: composición de los Genomas ADN Génico y Relacionado: 37% (1.5% CODIFICANTE, EXONES!!) ADN No Codificante: 63% (44 % ELEMENTOS TRANSPONIBLES) 1.5% 44% CONCEPTO
TEMA 3: Expresión Génica Genómica Estructural: composición de los Genomas ADN Génico y Relacionado: 37% (1.5% CODIFICANTE, EXONES!!) ADN No Codificante: 63% (44 % ELEMENTOS TRANSPONIBLES) 1.5% 44% CONCEPTO
Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497.
 Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497. Extracción de DNA genómico El DNA de A. brasilense CBG 497 se extrajo siguiendo el método descrito por la casa comercial distribuidora
Construcción de la biblioteca en cósmidos de Azospirillum brasilense CBG 497. Extracción de DNA genómico El DNA de A. brasilense CBG 497 se extrajo siguiendo el método descrito por la casa comercial distribuidora
PROCEDIMIENTO DETECCION NOROVIRUS RT-qPCR EN EXTRACTO VIRAL PROVENIENTE DE MOLUSCOS BIVALVOS. PRT-712.07.01-097 Página 1 de 7
 PRT-712.07.01-097 Página 1 de 7 1. OBJETIVO Realizar la detección molecular de genes de Norovirus en muestras de moluscos bivalvos. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a concentrados
PRT-712.07.01-097 Página 1 de 7 1. OBJETIVO Realizar la detección molecular de genes de Norovirus en muestras de moluscos bivalvos. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a concentrados
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN DE LAS COMPETENCIAS PROFESIONALES CUESTIONARIO DE AUTOEVALUACIÓN PARA LAS TRABAJADORAS Y TRABAJADORES
 MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato
 Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
Introducción Taller Ciencia para Jóvenes CIMAT 2012 Bachillerato julio 8 14 Cinvestav Campus Guanajuato Visita al CINVESTAV - Irapuato. 19 julio, 2012. Las bacterias son los organismos más abundantes que
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL EXAMEN DHE MOLECULARES EN EL EXAMEN DHE
 X Curso de Formación sobre la Protección de las Obtenciones Vegetales para Países Iberoamericanos Montevideo (URUGUAY) 12 al 16 de Diciembre de 2011 EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL
X Curso de Formación sobre la Protección de las Obtenciones Vegetales para Países Iberoamericanos Montevideo (URUGUAY) 12 al 16 de Diciembre de 2011 EL EMPLEO DE ANÁLISIS BIOQUÍMICOS Y MOLECULARES EN EL
Grado en Química. 4º Curso Bioquímica. Guión de Prácticas
 Grado en Química 4º Curso Bioquímica Guión de Prácticas UTILES A TRAER POR EL ALUMNO Bata Gafas de Seguridad Cuaderno de Laboratorio NORMAS DE TRABAJO Antes de empezar Antes de empezar cada práctica, el
Grado en Química 4º Curso Bioquímica Guión de Prácticas UTILES A TRAER POR EL ALUMNO Bata Gafas de Seguridad Cuaderno de Laboratorio NORMAS DE TRABAJO Antes de empezar Antes de empezar cada práctica, el
Diagnostico del Virus de Papiloma Humano por papanicolau y PCR en un grupo de adolescentes y mujeres jóvenes.
 Diagnostico del Virus de Papiloma Humano por papanicolau y PCR en un grupo de adolescentes y mujeres jóvenes. Instituto Conmemorativo Gorgas de Estudios de la Salud, Centro de Investigación en Reproducción
Diagnostico del Virus de Papiloma Humano por papanicolau y PCR en un grupo de adolescentes y mujeres jóvenes. Instituto Conmemorativo Gorgas de Estudios de la Salud, Centro de Investigación en Reproducción
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Prácticas de Ingeniería genética
 Prácticas de Ingeniería genética 5 3 ATCA TAG ATCG T G G C T AA T AA T AG T G G AA CC T C A A 3 T T C T T 5 Guión de alumnos (2012-2013) laczα http://www.bbm1.ucm.es/public_html/public_html//teach/biol/web05/index.html
Prácticas de Ingeniería genética 5 3 ATCA TAG ATCG T G G C T AA T AA T AG T G G AA CC T C A A 3 T T C T T 5 Guión de alumnos (2012-2013) laczα http://www.bbm1.ucm.es/public_html/public_html//teach/biol/web05/index.html
Universidad de Los Andes Facultad de Medicina Instituto Inmunología Clínica Maestría en Inmunología
 TÉCNICAS EN BIOLOGÍA MOLECULAR RESPONSABLE: Dra. Lisbeth Berrueta. CREDITOS: 2 El curso de Técnicas de Biología Molecular está destinado a profesionales del área biomédica con conocimientos básicos relativos
TÉCNICAS EN BIOLOGÍA MOLECULAR RESPONSABLE: Dra. Lisbeth Berrueta. CREDITOS: 2 El curso de Técnicas de Biología Molecular está destinado a profesionales del área biomédica con conocimientos básicos relativos
Logros. Crecimiento bacteriano. Crecimiento. Finalidad de las bacterias
 Fisiología bacteriana Dr. Juan C. Salazar jcsalazar@med.uchile.cl Programa de Microbiología y Micología ICBM, Facultad de Medicina Finalidad de las bacterias Logros Crecimiento Cómo lo hacen? Herramientas
Fisiología bacteriana Dr. Juan C. Salazar jcsalazar@med.uchile.cl Programa de Microbiología y Micología ICBM, Facultad de Medicina Finalidad de las bacterias Logros Crecimiento Cómo lo hacen? Herramientas
2. ESTRATEGIA PARA EFECTUAR LA CLONACION Y EXPRESION DEL GEN
 1. CONTROL FINAL Nombre Internacional y nombre de la vacuna Nombre del propietario Nombre y dirección del fabricante Número de Lote Fecha de fabricación Fecha de caducidad Temperatura de almacenamiento
1. CONTROL FINAL Nombre Internacional y nombre de la vacuna Nombre del propietario Nombre y dirección del fabricante Número de Lote Fecha de fabricación Fecha de caducidad Temperatura de almacenamiento
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico
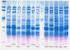 17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
NOVEDADES 2014 CLINICAL AND LABORATORY STANDARDS INSTITUTE (CLSI)
 NOVEDADES 2014 CLINICAL AND LABORATORY STANDARDS INSTITUTE (CLSI) Melina Rapoport Servicio Antimicrobianos Instituto Nacional de Enfermedades Infecciosas. ANLIS Dr. C. G. Malbrán Se describe aquí un breve
NOVEDADES 2014 CLINICAL AND LABORATORY STANDARDS INSTITUTE (CLSI) Melina Rapoport Servicio Antimicrobianos Instituto Nacional de Enfermedades Infecciosas. ANLIS Dr. C. G. Malbrán Se describe aquí un breve
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Purificación de proteínas
 Purificación de proteínas Purificación Proceso que cuenta con varias etapas cuyo objetivo es lograr la concentración diferencial de la proteína o molécula de interés Condiciones generales a evaluar Calidad
Purificación de proteínas Purificación Proceso que cuenta con varias etapas cuyo objetivo es lograr la concentración diferencial de la proteína o molécula de interés Condiciones generales a evaluar Calidad
Página: 1. Cantidad. Unidad Medida. Precio Unitario. Característica/ RENG Codigo Producto Concentracion Presentacion Lote05 Reactivos de Laboratorio
 Lote05 Reactivos de Laboratorio 1 9292 A.S.O. Kit/50-150 900 2 10109 AC. Nalidixico 3 3383 Acido Clorhidrico 1 N Litro 4 2444 Acido Úrico Manual Kit/100-400 P 804 5 10089 Agar Chocolate 500 g 132 6 10088
Lote05 Reactivos de Laboratorio 1 9292 A.S.O. Kit/50-150 900 2 10109 AC. Nalidixico 3 3383 Acido Clorhidrico 1 N Litro 4 2444 Acido Úrico Manual Kit/100-400 P 804 5 10089 Agar Chocolate 500 g 132 6 10088
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA
 UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE ADN
 Servicio de Secuenciación de ADN UNIVERSIDAD DE LA LAGUNA Edf. Servicios Generales - IUBO Avda. Francisco Sánchez s/n Tfno.: 922 318 644 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE ADN SERVICIO DE SECUENCIACIÓN
Servicio de Secuenciación de ADN UNIVERSIDAD DE LA LAGUNA Edf. Servicios Generales - IUBO Avda. Francisco Sánchez s/n Tfno.: 922 318 644 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE ADN SERVICIO DE SECUENCIACIÓN
PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN DE LAS COMPETENCIAS PROFESIONALES CUESTIONARIO DE AUTOEVALUACIÓN PARA LAS TRABAJADORAS Y TRABAJADORES
 MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
MINISTERIO DE EDUCACIÓN SECRETARÍA DE ESTADO DE EDUCACIÓN Y FORMACIÓN PROFESIONAL DIRECCIÓN GENERAL DE FORMACIÓN PROFESIONAL INSTITUTO NACIONAL DE LAS CUALIFICACIONES PROCEDIMIENTO DE EVALUACIÓN Y ACREDITACIÓN
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Separación e identificación de lípidos de membrana
 Separación e identificación de lípidos de membrana Objetivos Separación e identificación de especies de fosfolípidos por cromatografía de capa fina (TLC, Thin Layer Chromatography) TLC monodimensional
Separación e identificación de lípidos de membrana Objetivos Separación e identificación de especies de fosfolípidos por cromatografía de capa fina (TLC, Thin Layer Chromatography) TLC monodimensional
Resultados Práctica Transformación
 Resultados Práctica Transformación Eficiencia de Transformación Para cálculo de eficiencia Número de colonias transformadas Cantidad de plásmido agregado a bacterias que están en placa a. Cantidad
Resultados Práctica Transformación Eficiencia de Transformación Para cálculo de eficiencia Número de colonias transformadas Cantidad de plásmido agregado a bacterias que están en placa a. Cantidad
BIOLOGIA CELULAR Y MOLECULAR. LABORATORIO No 5: ENZIMAS DE RESTRICCION
 BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 5: ENZIMAS DE RESTRICCION 1. INTRODUCION Las enzimas de restricción, también conocidas como endonucleasas, son enzimas que cortan los enlaces fosfodiéster del
BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 5: ENZIMAS DE RESTRICCION 1. INTRODUCION Las enzimas de restricción, también conocidas como endonucleasas, son enzimas que cortan los enlaces fosfodiéster del
BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA
 BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA PROFESORADO CURSO 1994-1995: Dr. J. Ariño (Unidad de Bioquímica) Dra. F. Bosch (Unidad de Bioquímica) Dr. A. Sánchez (Unidad de Genética y Mejora)
BIOQUIMICA Y GENETICA MOLECULAR APLICADA A LA VETERINARIA PROFESORADO CURSO 1994-1995: Dr. J. Ariño (Unidad de Bioquímica) Dra. F. Bosch (Unidad de Bioquímica) Dr. A. Sánchez (Unidad de Genética y Mejora)
