5. MATERIALES Y MÉTODOS
|
|
|
- Celia Montoya Nieto
- hace 8 años
- Vistas:
Transcripción
1 5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System Perkin Elmer, GeneAmp PCR System Applied Biosystems, GeneAmp PCR Systems Transformación bacteriana Plásmidos Para la obtención del control positivo se optó por la transformación bacteriana con un vector plásmido. Las cepas de Escherichia coli que se emplearon fueron DH5α y XL1-blue proporcionadas amablemente por el Dr. Ignacio Martínez Laguna del Instituto de Ciencias de la Universidad Autónoma de Puebla. El plásmido empleado para la transformación fue paulo-p185-t315i-gwb (Beissert T, 2006) que contiene el gen híbrido humano bcr-abl1 con la mutación T315I. El plásmido contiene los genes de resistencia a puromicina y el gen beta-lactamasa como marcadores de transformación. La figura 5.1 muestra el mapa del plásmido para control positivo. Se recibieron 5 μg de plásmido y se diluyeron en 100µL de buffer TE estéril. La muestra se almacenó a -20 C. 34
2 El plásmido fue construido por el Dr. Tim Beissert y proporcionado cordialmente por el Dr. Martin Ruthardt de la Universidad J. W. Goethe, Frankfurt, Alemania. Fig Mapa del plásmido paulo-p185-t315i-gwb (Beissert T, 2006). En el texto se empleará la forma abreviada paulo-t315i para referirse al mismo. En todos los experimentos de transformación se empleó como plásmido control puc18 cuyo mapa genético se muestra en la figura
3 Fig Mapa genético de puc18 y puc19 (Fermentas Life Sciences, 2006) Células competentes Para la obtención de células competentes se empleó el protocolo reportado por Zhiming Tu et al. (2005), al cual se le hicieron algunas modificaciones para adecuarlo a las condiciones imperantes. La preparación de las soluciones empleadas se describe en la sección de Anexos 9.1, 9.2 y 9.3. El método consistió en inocular 2 ml de medio líquido S.O.C. fresco e incubar a 37 C durante toda la noche en agitación constante. Después tomar 1 ml del medio con crecimiento 36
4 y colocarlo en 20 ml de S.O.C. e incubar nuevamente a 37 C con agitación vigorosa hasta alcanzar una densidad óptica OD 600 de (2-3 horas aproximadamente). Una vez que se obtiene la OD 600 adecuada, los siguientes pasos son siempre a una temperatura de 4 C. Pasar los 20 ml de medio crecido a dos tubos falcon de 15 ml (10 ml en cada tubo) y centrifugar durante 10 min a 4000 rpm. Eliminar el sobrenadante, resuspender el pellet en 5 ml de solución TB e incubar la solución a 4 C durante 25 minutos. Posteriormente centrifugar, eliminar el sobrenadante y disolver el pellet en 1 ml de la solución TB. Mantener siempre a 4 C Transformación bacteriana Para la transformación de las células competentes previamente obtenidas se siguió el protocolo complementario (Tu Z et al., 2005). Una vez que se obtienen las células competentes, tomar 100 µl de dicha solución y colocarlas en un tubo Eppendorf y añadir 5 μl de la solución de plásmido paulo-t315i junto con 1 μl de DMSO. Incubar en hielo durante 30 min y posteriormente calentar durante 90 segundos exactamente a 42 C. Incubar nuevamente en hielo durante 5 min y agregar 400 µl de medio S.O.C. líquido e incubar a 37 C durante 45 min. Tomar 50 μl y colocarlos sobre una placa de S.O.C. con ampicilina e incubar toda la noche. Lo anterior también se realizó con el plásmido control puc18. 37
5 La eficiencia de la transformación en ufc/µg se calculó mediante las siguientes fórmulas: ufc transformadas = No. de colonias Coeficiente dilución (Vol. original de transformación) Volumen empleado en la placa Eficiencia de transformación = ufc transformadas DNA de plásmido (en µg) Lisis bacteriana y extracción del plásmido Previo a la lisis y extracción es necesario crecer colonias bacterianas aisladas en 2 ml de medio LB-Ampicilina durante toda la noche con agitación. Posteriormente se adiciona 1 ml de medio crecido previamente y se colocan en 100 ml de LB-Ampicilina con agitación vigorosa a 37 C durante toda la noche. El protocolo empleado es una modificación de los métodos de Birnboim y Doly (1979) y Ish- Horowicz y Burke (1981) de acuerdo a Sambrook (1990). El método consistió en colocar los 100 ml de medio con crecimiento en 2 tubos Falcon de 50 ml y centrifugar a 4000 rpm durante 10 min a 4 C. Eliminar cuidadosamente el sobrenadante y añadir 3 ml de Solución I fría y agitar con vórtex. Añadir 1 ml de solución STE, agitar y centrifugar. Después de retirar el sobrenadante, resuspender el pellet en solución I como se ha señalado previamente y añadir 1 ml de una solución fresca de lisozima ph 8.0 y 2 ml de solución II fría recién preparada. Mezclar con movimientos suaves, almacenar a temperatura 38
6 ambiente durante 10 min. Agregar 3 ml de solución III helada, agitar con vórtex y almacenar sobre hielo durante 5 min. A continuación centrifugar a 4000 rpm durante 5 min a 4 C y transferir el sobrenadante en un tubo fresco. Subsiguientemente añadir un volumen igual de una solución de fenol:cloroformo (1:1), mezclar con vórtex y centrifugar como se ha descrito previamente, transferir el sobrenadante a un tubo fresco. Para precipitar el DNA de doble cadena añadir 2 ml de isopropanol, mezclar y almacenar 10 minutos a temperatura ambiente; centrifugar como se ha descrito previamente. Retirar el sobrenadante con mucho cuidado y resuspender el pellet de DNA con 2 ml de etanol frío al 70% por duplicado. Centrifugar y retirar el sobrenadante, permitir secar el pellet de DNA durante minutos a temperatura ambiente. Finalmente disolver el pellet de DNA en 1 ml de TE con RNAasa. Almacenar a - 20 C. El análisis del plásmido se hizo en geles de agarosa al 0.8 %. La presencia de trna en grandes cantidades requirió de otro tratamiento de RNAasa durante 2 horas o por toda la noche para evitar digestiones parciales. Posteriormente se volvió a extraer con volúmenes iguales de fenol/cloroformo. Se agregó 1/10 parte del volumen de 3 M de acetato de sodio ph 5.2 y se precipitó con 2 volúmenes de etanol absoluto Cuantificación de DNA La cuantificación de DNA se realizó midiendo la densidad óptica a 260 nm. Un valor de OD 260 igual a 1 corresponde a 50 μg/ml de DNA de doble cadena. 39
7 Para calcular la pureza se midió también el valor de OD a 280 nm. El cociente obtenido a partir de OD 260 / OD 280 nos proporciona el valor de pureza. Un valor de es aceptable. Para calcular el número de copias presente en una determinada cantidad de dsdna se empleó la siguiente ecuación: # de copias de dsdna = (cantidad dsdna) (6.022x1023 ) #pb 1x10 3 (660) En donde la cantidad de DNA debe estar expresada en mg. El número de Avogadro corresponde a 6.022x10 23 ; # pb es la longitud de la cadena de DNA expresado en pares de bases y finalmente 660 (g/mol) corresponde al peso molecular de un par de bases presente en cualquier cadena de dsdna. 5.3 Amplificación por ARMS-PCR La amplificación se realizó en una mezcla para PCR constituida por 2.5 µl de buffer 10xPCR con una concentración de MgCl 2 de 15mM, 2.5 μl de 2mM dntps, 2.5 μl de solución de primers 5µM, 1 U de Taq DNA polimerasa y 1 µl de muestra de DNA para un volumen final de 25 μl. Se emplearon tres primers diferentes distribuidos en dos mezclas. Dos primers son aleloespecíficos y están diseñados para ARMS-PCR a los cuales se les hizo un cambio en el penúltimo nucleótido del extremo 3 para originar un desajuste de bases ( mismatch ) y aumentar la especificidad por parte de Taq DNA-polimerasa. Se designó con la letra C al 40
8 oligonucleótido inespecífico que es común para los dos alelos. Para la mezcla de PCR se emplearon dos mezclas de primers denominadas mezcla T (primer T + primer C) y mezcla I (primer I + primer C). La secuencia de los primers se muestra a continuación: Oligonucleótido Secuencia Primer T 5 AGC CCC CGT TCT ATA TCA TCT C 3 Primer I 5 AGC CCC CGT TCT ATA TCA TCT T 3 Primer C 5 AGC AGC ACC ACG GCG TTC ACC T 3 Tabla 5.1. Secuencia de los tres primers empleados para la amplificación por ARMS-PCR. La base subrayada fue introducida para generar un desajuste de bases. El perfil de temperaturas en cada ciclo para la amplificación por PCR se muestra en la siguiente tabla: Paso Temperatura Tiempo Ciclos Primera desnaturalización 94 C 2 minutos 1 Desnaturalización 94 C 20 segundos Hibridación 60 C 30 segundos 35 Extensión 72 C 30 segundos Post-ciclo (extensión) 72 C 2 minutos 1 Tabla 5.2. Perfil de temperaturas empleadas para amplificación por ARMS-PCR. Una muestra de paciente positivo T315I fue proporcionada por el Dr. Guillermo Ruiz Argüelles. Dicha muestra funcionó como control positivo temporal. 41
9 5.4 Caracterización de los productos de amplificación Los productos de amplificación se analizaron en geles de poliacrilamida al 4.5 % empleando el marcador de puc18 digerido con Hpa II. En todos los casos los productos de amplificación tienen una longitud de 100 pares de bases. Para poder comprobar que los productos amplificados son los buscados se realizaron digestiones de dichos productos con las enzimas Hpa II y Dde I. Las mezclas de digestión consistieron en 7 µl de agua, 2 µl del buffer, 1 µl de la enzima de restricción correspondiente y 10 μl del producto de amplificación (volumen final 20 μl). Las digestiones están listas en 2 h aproximadamente a 37 C para ambas enzimas, sin embargo se dejaron toda la noche para evitar posibles digestiones parciales. El análisis de los fragmentos de restricción se hizo en geles de poliacrilamida al 6.0 % y posterior tinción con bromuro de etidio. 5.5 Especificidad y sensibilidad analíticas Para medir la especificidad analítica del método se realizaron amplificaciones con DNA humano normal con la mezcla I de primers. Se utilizaron 3 diferentes concentraciones de primers para forzar una amplificación sobre DNA humano (control negativo). Las concentraciones de primers fueron 1x, 2x y 4x considerando x la concentración indicada en el protocolo para la mezcla de PCR descrito en la sección 5.3. La concentración inicial de 42
10 control negativo fue de μg/ml. Las diluciones del control negativo en TE para amplificación con la mezcla I fueron 1:3, seis diluciones en total. Después se realizaron diluciones de control positivo/control negativo. La concentración de control negativo empleada en las diluciones con control positivo corresponde a aquella concentración mínima con la que sí se obtuvo producto de amplificación con la mezcla I. Las diluciones corresponden a 1:3 (control positivo: control negativo) partiendo de una concentración inicial de copias de control positivo y control negativo. De dichas soluciones se hicieron amplificaciones con las mezclas de primer I y T a concentraciones 1x, 2x y 4x. De esta forma se obtiene una relación entre la concentración mínima de control positivo que puede ser amplificada y observada en un gel respecto a una concentración dada de control negativo determinándose así la sensibilidad analítica. 43
CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL
 LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
DETECCIÓN DE C. jejuni EN HAMBURGUESA DE POLLO POR PCR TRADICIONAL PRT-712.04-082
 Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA. Grado en Medicina
 DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS
 Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Protocolos de secuenciación de ADN
 1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
DANAGENE SALIVA KIT. Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones
 DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O. Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua
 C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR
 TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Andrés Ciocchini, Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN) por partición diferencial
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Andrés Ciocchini, Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN) por partición diferencial
39. Aislamiento y purificación del DNA de un plásmido recombinante
 39. Aislamiento y purificación del DNA de un plásmido recombinante Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento de Bioquímica
39. Aislamiento y purificación del DNA de un plásmido recombinante Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento de Bioquímica
V MATERIALES Y MÉTODOS
 V MATERIALES Y MÉTODOS 5.1 Aislamiento de bacterias e identificación 5.1.1 Cepas tomadas del cepario de la Universidad de las Américas-Puebla En el cepario del Departamento de Química y Biología hay algunas
V MATERIALES Y MÉTODOS 5.1 Aislamiento de bacterias e identificación 5.1.1 Cepas tomadas del cepario de la Universidad de las Américas-Puebla En el cepario del Departamento de Química y Biología hay algunas
Práctico: Genética bacteriana 2007.
 Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
Reacción en cadena de la polimerasa (PCR)
 Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO
 Actividad Experimental SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO Investigación previa 1.- Investigar las medidas de seguridad que hay que mantener al manipular KOH y H SO, incluyendo que acciones
Actividad Experimental SEPARACIÓN DE ALUMINIO A PARTIR DE MATERIAL DE DESECHO Investigación previa 1.- Investigar las medidas de seguridad que hay que mantener al manipular KOH y H SO, incluyendo que acciones
Código: IDK-010 Ver: 1. Proteína G C825T. Sistema para la detección de la mutación C825T en el gene de la proteína G.
 C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
3. COMPONENTES. Tampón de electroforesis concentrado 2 x 50 ml 10 X (2 envases 500ml)
 PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
1 MATERIALES Y MÉTODOS
 1 MATERIALES Y MÉTODOS El presente protocolo experimental contempla la amplificación del DNA de las bacterias y virus causantes de las ETS Neisseria gonorrhoeae, Chlamydia trachomatis y VPH mediante PCR
1 MATERIALES Y MÉTODOS El presente protocolo experimental contempla la amplificación del DNA de las bacterias y virus causantes de las ETS Neisseria gonorrhoeae, Chlamydia trachomatis y VPH mediante PCR
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES
 PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
COMO ABORDAR Y RESOLVER ASPECTOS PRÁCTICOS DE MICROBIOLOGÍA
 COMO ABORDAR Y RESOLVER ASPECTOS PRÁCTICOS DE MICROBIOLOGÍA 3. CÁLCULOS DE BIOMASA Inés Arana, Maite Orruño e Isabel Barcina Departamento Inmunología, Microbiología y Parasitología Universidad del País
COMO ABORDAR Y RESOLVER ASPECTOS PRÁCTICOS DE MICROBIOLOGÍA 3. CÁLCULOS DE BIOMASA Inés Arana, Maite Orruño e Isabel Barcina Departamento Inmunología, Microbiología y Parasitología Universidad del País
III. METODOLOGÍA EXPERIMENTAL. La metodología empleada se muestra en el diagrama de flujo de la Figura 4 y se presenta en las siguientes secciones:
 III. METODOLOGÍA EXPERIMENTAL La metodología empleada se muestra en el diagrama de flujo de la Figura 4 y se presenta en las siguientes secciones: 1. Área de Estudio y Recolecta de las Muestras (sección
III. METODOLOGÍA EXPERIMENTAL La metodología empleada se muestra en el diagrama de flujo de la Figura 4 y se presenta en las siguientes secciones: 1. Área de Estudio y Recolecta de las Muestras (sección
CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA)
 CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA) PROTOCOLO ADA FUNDAMENTO DEL METODO: La adenosindesaminasa (ADA) es una enzima del catabolismo de las purinas que cataliza la conversión de la adenosina en inosina
CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA) PROTOCOLO ADA FUNDAMENTO DEL METODO: La adenosindesaminasa (ADA) es una enzima del catabolismo de las purinas que cataliza la conversión de la adenosina en inosina
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
ELECTROFORESIS AVANZADA
 Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR
 PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS.
 NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz porosa
 INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala
 Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala IICA/Red SICTA-CIAT INFORME DE AVANCE Gerardo Gallego - Harold
Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala IICA/Red SICTA-CIAT INFORME DE AVANCE Gerardo Gallego - Harold
DETERMINACIÓN DEL FACTOR Rh por PCR
 Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
III. METODOLOGÍA EXPERIMENTAL
 III. METODOLOGÍA EXPERIMENTAL 3.1 Análisis Previos Se utilizaron los filtros con muestra de partículas suspendidas en el aire PM 10 que el Instituto Municipal de Ecología (IME) proporcionó para llevar
III. METODOLOGÍA EXPERIMENTAL 3.1 Análisis Previos Se utilizaron los filtros con muestra de partículas suspendidas en el aire PM 10 que el Instituto Municipal de Ecología (IME) proporcionó para llevar
NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN
 NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN 1. PROCEDIMIENTOS BÁSICOS: En este apartado pretendemos detallar algunos factores muy importantes a la hora de realizar
NORMAS DE TRABAJO EN BIOLOGÍA MOLECULAR Y GUÍA DE PROBLEMAS HABITUALES Y SU SOLUCIÓN 1. PROCEDIMIENTOS BÁSICOS: En este apartado pretendemos detallar algunos factores muy importantes a la hora de realizar
LABORATORIO DE QUÍMICA ANALÍTICA INFORME PRESENTADO A LA PROF. ANGELA SIFONTE
 LABORATORIO DE QUÍMICA ANALÍTICA INFORME PRESENTADO A LA PROF. ANGELA SIFONTE TURBIDIMETRÍA INFORME N 9. PRESENTADO POR LOS BRS.: WILLIAM CHEN CHEN C.I.: 16.113.714 YUSMARY DE ABREU C.I.: 15.914.973 CARACAS,
LABORATORIO DE QUÍMICA ANALÍTICA INFORME PRESENTADO A LA PROF. ANGELA SIFONTE TURBIDIMETRÍA INFORME N 9. PRESENTADO POR LOS BRS.: WILLIAM CHEN CHEN C.I.: 16.113.714 YUSMARY DE ABREU C.I.: 15.914.973 CARACAS,
Curso de biología molecular CIMAT 2010
 Curso de biología molecular CIMAT 2010 INTRODUCCIÓN Este curso-taller tiene como propósito mostrar de manera teórica y práctica algunos principios básicos de biología molecular e ingeniería genética. Conocerás
Curso de biología molecular CIMAT 2010 INTRODUCCIÓN Este curso-taller tiene como propósito mostrar de manera teórica y práctica algunos principios básicos de biología molecular e ingeniería genética. Conocerás
GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA
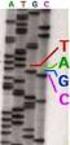 Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
3/22/2010. Iván Ferrer Rodríguez, PhD Catedrático. En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR.
 Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO
 PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO I. OBJETIVO Determinar la presencia de bacterias Mesófilas Aerobias en una muestra de agua potable por la técnica de
PRACTICA Núm. 16 RECUENTO DE BACTERIAS MESOFILAS AEROBIAS EN AGUA PARA CONSUMO HUMANO I. OBJETIVO Determinar la presencia de bacterias Mesófilas Aerobias en una muestra de agua potable por la técnica de
DANAGENE RNA PURIFICATION KIT
 DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
FRAGMENTO OLIGO 5-3 Hebra SECUENCIA 5' - - > 3' TAMAÑO (pb) F1. hmtl 569 L AACCAAACCCCAAAGACACC hmth2982 H CTGATCCAACATCGAGGTCG
 ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
ELECTROFORESIS BASICA
 Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz
 IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
Guía de Problemas. Espectrofotometría: Técnicas de Muestreo, Análisis e Interpretación de Datos Ingeniería Ambiental
 Guía de Problemas Espectrofotometría: 1- La absortividad molar de una solución del complejo formado por Bi(III) y tiourea es 9,32x10 3 L/mol x cm a 470 nm. Cuál es la absorbancia de una solución 3,79x10-5
Guía de Problemas Espectrofotometría: 1- La absortividad molar de una solución del complejo formado por Bi(III) y tiourea es 9,32x10 3 L/mol x cm a 470 nm. Cuál es la absorbancia de una solución 3,79x10-5
La primera parte del proyecto consistió en la obtención de un control positivo mediante
 7. DISCUSIÓN La primera parte del proyecto consistió en la obtención de un control positivo mediante técnicas de clonación molecular. E. coli, a diferencia de otras bacterias que presentan competencia
7. DISCUSIÓN La primera parte del proyecto consistió en la obtención de un control positivo mediante técnicas de clonación molecular. E. coli, a diferencia de otras bacterias que presentan competencia
I. 15microlitros de agua esteril, perforar el pozo y diluir. II. Se tomaron 2 microlitros para la primera transformación.
 23 de agosto 2007 Se comenzó la elaboración de la bitácora Medio LB Broth, Billar (Luria-Bertani) 25gr/litro Se preparó 500 mililitros agregando 12.5 gramos Se diluyó y esterilizó por autoclave Medio LB
23 de agosto 2007 Se comenzó la elaboración de la bitácora Medio LB Broth, Billar (Luria-Bertani) 25gr/litro Se preparó 500 mililitros agregando 12.5 gramos Se diluyó y esterilizó por autoclave Medio LB
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO
 AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO E l estudio del genoma de los seres vivos a sido uno d los principales objetivos de la Biología. Desde los trabajos de Mendel (1866),
AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO E l estudio del genoma de los seres vivos a sido uno d los principales objetivos de la Biología. Desde los trabajos de Mendel (1866),
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR)
 METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
RESOLUCIÓN OENO 5/2007
 RESOLUCIÓN OENO 5/2007 CODEX - GLICOSIDASA LA ASAMBLEA GENERAL Visto el Artículo 2, párrafo 2 iv del acuerdo del 3 de abril de 2001 por el cual se creó la Organización Internacional de la Viña y el Vino,
RESOLUCIÓN OENO 5/2007 CODEX - GLICOSIDASA LA ASAMBLEA GENERAL Visto el Artículo 2, párrafo 2 iv del acuerdo del 3 de abril de 2001 por el cual se creó la Organización Internacional de la Viña y el Vino,
ENFERMEDADES INFECCIOSAS GRADO EN VETERINARIA
 FACULTAD DE VETERINARIA DEPARTAMENTO DE SANIDAD ANIMAL ENFERMEDADES INFECCIOSAS GRADO EN VETERINARIA GUIÓN DE PRÁCTICAS DE LABORATORIO CURSO 2014 2015 MÓDULO: PORCINO Síndrome respiratorio y reproductor
FACULTAD DE VETERINARIA DEPARTAMENTO DE SANIDAD ANIMAL ENFERMEDADES INFECCIOSAS GRADO EN VETERINARIA GUIÓN DE PRÁCTICAS DE LABORATORIO CURSO 2014 2015 MÓDULO: PORCINO Síndrome respiratorio y reproductor
Comprobando la calidad del DNA genómico mediante electroforesis en gel
 Comprobando la calidad del DNA genómico mediante electroforesis en gel GELES 1 Y 2 Agarosa 0.8 %, electroforesis a 1 V/cm Muestras: Se ha cargado muestras de DNA genómico no digerido, antes (líneas impares)
Comprobando la calidad del DNA genómico mediante electroforesis en gel GELES 1 Y 2 Agarosa 0.8 %, electroforesis a 1 V/cm Muestras: Se ha cargado muestras de DNA genómico no digerido, antes (líneas impares)
MATERIALES Y MÉTODOS. Cepas Utilizadas
 MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
5 Materiales y Métodos
 5 Materiales y Métodos Los tejidos de Cenchurs ciliaris utilizados en esta investigación fueron obtenidos por pretratamiento con H 2 SO 4 al 0.15 M, a 135ºC, para eliminar la hemicelulosa. Con los tejidos
5 Materiales y Métodos Los tejidos de Cenchurs ciliaris utilizados en esta investigación fueron obtenidos por pretratamiento con H 2 SO 4 al 0.15 M, a 135ºC, para eliminar la hemicelulosa. Con los tejidos
CAPÍTULO VI. Expresión de genes relacionados con apoptosis
 CAPÍTULO VI Expresión de genes relacionados con apoptosis 1.- Introducción 2.- Protocolos para análisis genéticos 3.- Resultados en MCF-7 4.- Discusión 1.- Introducción Como se ha descrito anteriormente,
CAPÍTULO VI Expresión de genes relacionados con apoptosis 1.- Introducción 2.- Protocolos para análisis genéticos 3.- Resultados en MCF-7 4.- Discusión 1.- Introducción Como se ha descrito anteriormente,
Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez
 Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez 1 Objetivos Entender la técnica: Reacción de Polimerasa en Cadena (PCR por sus siglas en inglés)
Detección de la secuencia Alu mediante la técnica Reacción en Cadena de la Polimerasa (PCR ) N. Rodríguez 1 Objetivos Entender la técnica: Reacción de Polimerasa en Cadena (PCR por sus siglas en inglés)
Técnicas de ADN recombinante: la manipulación genética
 Técnicas de ADN recombinante: la manipulación genética A partir de los años 70 se desarrollaron las herramientas de la biología molecular o la ingeniería genética o lo que se ha llamado técnicas del ADN
Técnicas de ADN recombinante: la manipulación genética A partir de los años 70 se desarrollaron las herramientas de la biología molecular o la ingeniería genética o lo que se ha llamado técnicas del ADN
6.1 Células competentes y transformación. posterior transformación de éstas para la elaboración de un control positivo. La
 6. RESULTADOS 6.1 Células competentes y transformación La primera parte experimental del trabajo consistió en la obtención de células competentes y posterior transformación de éstas para la elaboración
6. RESULTADOS 6.1 Células competentes y transformación La primera parte experimental del trabajo consistió en la obtención de células competentes y posterior transformación de éstas para la elaboración
CUESTIONES. 1. Por qué la PCR convencional no es cuantitativa?
 2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
11.1 Expresión de los transcritos del subcomplejo SG-SSPN en el tejido adiposo parametrial.
 11. RESULTADOS Mediante este trabajo se identificó la expresión de los transcritos de α, β, γ, δ, ε y ζ SG y sspn en tejido adiposo parametrial de la rata y se localizó la proteína sspn en la membrana
11. RESULTADOS Mediante este trabajo se identificó la expresión de los transcritos de α, β, γ, δ, ε y ζ SG y sspn en tejido adiposo parametrial de la rata y se localizó la proteína sspn en la membrana
Obtención y prueba de corazones y vigas extraídos de concreto endurecido
 el concreto en la obra editado por el instituto mexicano del cemento y del concreto, A.C. Abril 2009 Obtención y prueba de corazones y vigas extraídos de concreto endurecido Primera parte 20 Problemas,
el concreto en la obra editado por el instituto mexicano del cemento y del concreto, A.C. Abril 2009 Obtención y prueba de corazones y vigas extraídos de concreto endurecido Primera parte 20 Problemas,
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA"
 P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
Laboratorio de Termodinámica TEMPERATURA. Objetivos
 Objetivos TEMPERATURA Comprender el fundamento termodinámico de la medición de la temperatura Construir la curva de calentamiento del agua Obtener mediciones de temperatura con tres termómetros distintos
Objetivos TEMPERATURA Comprender el fundamento termodinámico de la medición de la temperatura Construir la curva de calentamiento del agua Obtener mediciones de temperatura con tres termómetros distintos
CAPÍTULO III RESULTADOS Y ANÁLISIS DE RESULTADOS
 CAPÍTULO III RESULTADOS Y ANÁLISIS DE RESULTADOS Para el proceso de purificación del aceite, se pudo observar, en el momento del filtrado algunas partículas sólidas retenidas en los diferentes filtros
CAPÍTULO III RESULTADOS Y ANÁLISIS DE RESULTADOS Para el proceso de purificación del aceite, se pudo observar, en el momento del filtrado algunas partículas sólidas retenidas en los diferentes filtros
PRÁCTICA 5. CALORIMETRÍA
 PRÁCTICA 5. CALORIMETRÍA INTRODUCCIÓN Al mezclar dos cantidades de líquidos a distinta temperatura se genera una transferencia de energía en forma de calor desde el más caliente al más frío. Dicho tránsito
PRÁCTICA 5. CALORIMETRÍA INTRODUCCIÓN Al mezclar dos cantidades de líquidos a distinta temperatura se genera una transferencia de energía en forma de calor desde el más caliente al más frío. Dicho tránsito
UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I
 UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I DRAS. MARÍA LUISA ORTIZ Y ANA PEDREGOSA 1 PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA
UNIVERSIDAD DE MAYORES PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA Y BIOTECNOLOGÍA MICROBIOLOGÍA I DRAS. MARÍA LUISA ORTIZ Y ANA PEDREGOSA 1 PRÁCTICAS DE MICROBIOLOGÍA DEPARTAMENTO DE BIOMEDICINA
PCR. Técnica inventada por Kary Mullis :1987. Premio Nobel 1993. Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN
 PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
DETERMINACIÓN DE GLUTEN EN ALIMENTOS PARA CELÍACOS INDICE
 DETERMINACIÓN DE GLUTEN EN ALIMENTOS PARA CELÍACOS INDICE 1 Objetivo 2 2 Alcance 2 3 Desarrollo 2 4 Anexo 8 1.0. Objetivo Determinación de gluten en alimentos para celíacos. 2.0. Alcance Este método analítico
DETERMINACIÓN DE GLUTEN EN ALIMENTOS PARA CELÍACOS INDICE 1 Objetivo 2 2 Alcance 2 3 Desarrollo 2 4 Anexo 8 1.0. Objetivo Determinación de gluten en alimentos para celíacos. 2.0. Alcance Este método analítico
1.- OBJETIVO 2.- ALCANCE
 Código: M-FMED-LFIS-02 Revisión: 02 Página: 1 de 5 1.- OBJETIVO Establecer las instrucciones para la preparación de los reactivos que se utilizan en las prácticas tradicionales del Laboratorio de Ciencias
Código: M-FMED-LFIS-02 Revisión: 02 Página: 1 de 5 1.- OBJETIVO Establecer las instrucciones para la preparación de los reactivos que se utilizan en las prácticas tradicionales del Laboratorio de Ciencias
Práctica 5 CINÉTICA ENZIMÁTICA: DETERMINACIÓN ESPECTOFOTOMÉTRICA DE LA CONSTANTE DE MICHAELIS-MENTEN DE LA PAPAÍNA
 Práctica 5 CINÉTICA ENZIMÁTICA: DETERMINACIÓN ESPECTOFOTOMÉTRICA DE LA CONSTANTE DE MICHAELIS-MENTEN DE LA PAPAÍNA 1. Objetivo Se pretende calcular la constante de Michaelis-Menten (K M ), la constante
Práctica 5 CINÉTICA ENZIMÁTICA: DETERMINACIÓN ESPECTOFOTOMÉTRICA DE LA CONSTANTE DE MICHAELIS-MENTEN DE LA PAPAÍNA 1. Objetivo Se pretende calcular la constante de Michaelis-Menten (K M ), la constante
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico
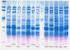 17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
Laboratorio. Objetivos I N T R O D U C C I Ó N. Al finalizar este laboratorio el estudiante podrá:
 Laboratorio 12 Biología molecular Objetivos Al finalizar este laboratorio el estudiante podrá: 1. Conocer los principios básicos de la técnica de electroforesis y su aplicación al análisis del ADN. 2.
Laboratorio 12 Biología molecular Objetivos Al finalizar este laboratorio el estudiante podrá: 1. Conocer los principios básicos de la técnica de electroforesis y su aplicación al análisis del ADN. 2.
Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml
 Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml Para la purificación de ADN genómico de todos los kits de colección Oragene y ORAcollect. Visite nuestro sitio
Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml Para la purificación de ADN genómico de todos los kits de colección Oragene y ORAcollect. Visite nuestro sitio
23. Hidrólisis ácida y enzimática del glucógeno
 23. Hidrólisis ácida y enzimática del glucógeno Emilia Martínez Galisteo, Carmen Alicia Padilla Peña, Concepción García Alfonso, José Antonio Bárcena Ruiz, Jesús Diez Dapena. Departamento de Bioquímica
23. Hidrólisis ácida y enzimática del glucógeno Emilia Martínez Galisteo, Carmen Alicia Padilla Peña, Concepción García Alfonso, José Antonio Bárcena Ruiz, Jesús Diez Dapena. Departamento de Bioquímica
MMP. MÉTODOS DE MUESTREO Y PRUEBA DE MATERIALES
 LIBRO: PARTE: TÍTULO: CAPÍTULO: MMP. MÉTODOS DE MUESTREO Y PRUEBA DE MATERIALES 5. MATERIALES PARA SEÑALAMIENTO Y DISPOSITIVOS DE SEGURIDAD 01. Pinturas para Señalamiento 003. Contenido de Pigmento en
LIBRO: PARTE: TÍTULO: CAPÍTULO: MMP. MÉTODOS DE MUESTREO Y PRUEBA DE MATERIALES 5. MATERIALES PARA SEÑALAMIENTO Y DISPOSITIVOS DE SEGURIDAD 01. Pinturas para Señalamiento 003. Contenido de Pigmento en
METODO DE FILTRACIÓN POR MEMBRANA PARA DETERMINACION DE COLIFORMES Y E. coli EN AGUA
 PRT-712.03-009 Página 1 de 7 1. OBJETIVO Este método se utiliza para medir la calidad sanitaria del agua potable. 2. CAMPO DE APLICACIÓN Y ALCANCE Se aplica a agua clorada o agua naturales de muy baja
PRT-712.03-009 Página 1 de 7 1. OBJETIVO Este método se utiliza para medir la calidad sanitaria del agua potable. 2. CAMPO DE APLICACIÓN Y ALCANCE Se aplica a agua clorada o agua naturales de muy baja
PROTOCOLO REGISTRO DE SEMILLA PRE-INOCULADA
 Dirección General de Servicios Agrícolas Ministerio de Ganadería, Agricultura y Pesca República Oriental del Uruguay Av. Millán 4703, Montevideo. CP 12.900. Teléfono: (598) -2304 3992 Web: http://www.mgap.gub.uy
Dirección General de Servicios Agrícolas Ministerio de Ganadería, Agricultura y Pesca República Oriental del Uruguay Av. Millán 4703, Montevideo. CP 12.900. Teléfono: (598) -2304 3992 Web: http://www.mgap.gub.uy
UNIDAD 3: SOLUCIONES
 UNIDAD 3: SOLUCIONES 1 Las soluciones son mezclas homogéneas. Estas constan de dos o más componentes en una única fase, por ejemplo agua con sal de cocina, o azúcar en agua Para estudiar o trabajar con
UNIDAD 3: SOLUCIONES 1 Las soluciones son mezclas homogéneas. Estas constan de dos o más componentes en una única fase, por ejemplo agua con sal de cocina, o azúcar en agua Para estudiar o trabajar con
TÉCNICAS GENÓMICAS. 2) Cuantificación del número de virus presentes en muestras de sangre o suero: carga vírica
 TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
TÉCNICAS GENÓMICAS Utilidad/Objetivos: 1) Detección de microorganismos directamente en muestras clínicas identificando un fragmento específico del genoma del microorganismo concreto 2) Cuantificación del
Normalización de soluciones de NaOH 0,1N y HCl 0,1N.
 Laboratorio N 1: Normalización de soluciones de NaOH 0,1N y HCl 0,1N. Objetivos: - Determinar la normalidad exacta de una solución de hidróxido de sodio aproximadamente 0,1 N, utilizando biftalato de potasio
Laboratorio N 1: Normalización de soluciones de NaOH 0,1N y HCl 0,1N. Objetivos: - Determinar la normalidad exacta de una solución de hidróxido de sodio aproximadamente 0,1 N, utilizando biftalato de potasio
EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS
 EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS Objetivos: Mª Jesús González García Mª Amparo Mora Alcácer COLEGIO AVE Mª DE PENYA-ROJA Aprender a trabajar en el laboratorio y apreciar el orden,
EFECTO DE LA DIGESTIÓN SOBRE PROTEINAS, GRASAS Y GLÚCIDOS Objetivos: Mª Jesús González García Mª Amparo Mora Alcácer COLEGIO AVE Mª DE PENYA-ROJA Aprender a trabajar en el laboratorio y apreciar el orden,
Y 7 elementos se requieren en cantidades menores a 1o ppm, conocidos como micronutrientes (Cloro, Zinc, Boro, Hierro, Cobre, Manganeso, Molibdeno)
 5 SOLUCIÓN NUTRITIVA INTRODUCCIÓN Para un desarrollo adecuado las plantas necesitan de 16 elementos esenciales, de los cuales 9 se requieren en cantidades mayores a 40 ppm conocidos como macronutrientes
5 SOLUCIÓN NUTRITIVA INTRODUCCIÓN Para un desarrollo adecuado las plantas necesitan de 16 elementos esenciales, de los cuales 9 se requieren en cantidades mayores a 40 ppm conocidos como macronutrientes
Trabajo Práctico N o 1
 1 Trabajo Práctico N o 1 Soluciones y diluciones OBJETIVOS - Conocer todas las formas de indicar la concentración química de una solución. - Ser capaz de calcular la concentración de una solución a partir
1 Trabajo Práctico N o 1 Soluciones y diluciones OBJETIVOS - Conocer todas las formas de indicar la concentración química de una solución. - Ser capaz de calcular la concentración de una solución a partir
FACULTAD REGIONAL ROSARIO
 FACULTAD REGIONAL ROSARIO CATEDRA BIOTECNOLOGIA ROFESOR: Ing. Eduardo Santambrosio JTP: Ing. Marta Ortega AUX 1ª : Ing. Pablo Garibaldi PCR (Polymerase chain reaction) Reacción en cadena de la polimerasa
FACULTAD REGIONAL ROSARIO CATEDRA BIOTECNOLOGIA ROFESOR: Ing. Eduardo Santambrosio JTP: Ing. Marta Ortega AUX 1ª : Ing. Pablo Garibaldi PCR (Polymerase chain reaction) Reacción en cadena de la polimerasa
1. Título. Cuantificacion de ADN por espectrofluorometría mediante el uso de Quant- it PicoGreen dsaadnssay Kit (Invitrogen ).
 1. Título. Cuantificacion de ADN por espectrofluorometría mediante el uso de QuantiT PicoGreen dsaadnssay Kit (Invitrogen ). 2. Finalidad. El presente documento describe el Procedimiento Normalizado de
1. Título. Cuantificacion de ADN por espectrofluorometría mediante el uso de QuantiT PicoGreen dsaadnssay Kit (Invitrogen ). 2. Finalidad. El presente documento describe el Procedimiento Normalizado de
ELECTROFORESIS EN GELES DE AGAROSA. Agustín Garrido. agugarrido@hotmail.com
 1 ELECTROFORESIS EN GELES DE AGAROSA Agustín Garrido agugarrido@hotmail.com Introducción En este trabajo práctico se utilizó la técnica de electroforesis. Este proceso se basa en la migración de las moléculas
1 ELECTROFORESIS EN GELES DE AGAROSA Agustín Garrido agugarrido@hotmail.com Introducción En este trabajo práctico se utilizó la técnica de electroforesis. Este proceso se basa en la migración de las moléculas
TEMA 11 LA MATERIA EN EL UNIVERSO
 TEMA 11 LA MATERIA EN EL UNIVERSO TEMA 11 LA MATERIA EN EL UNIVERSO QUÉ ES LA MATERIA? Materia es todo aquello que tiene volumen (ocupa un espacio) y que tiene una determinada masa (por tanto, pesa). QUÉ
TEMA 11 LA MATERIA EN EL UNIVERSO TEMA 11 LA MATERIA EN EL UNIVERSO QUÉ ES LA MATERIA? Materia es todo aquello que tiene volumen (ocupa un espacio) y que tiene una determinada masa (por tanto, pesa). QUÉ
GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS
 GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS Leidy Diana Ardila Leal Docente. INTRODUCCIÓN En esta práctica se va a realizar la extracción
GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS Leidy Diana Ardila Leal Docente. INTRODUCCIÓN En esta práctica se va a realizar la extracción
OBTENCIÓN DE MUTANTES
 OBTENCIÓN DE MUTANTES parp::hyg DE Fusarium oxysporum f. sp. lycopersici MEDIANTE LA TÉCNICA PCR DE DOBLE FUSIÓN. Gallegos Almanza I. A. (1) ; Martínez Cadena M. G. (2) ; Ferrel Cano L. E. (2). (1) Facultad
OBTENCIÓN DE MUTANTES parp::hyg DE Fusarium oxysporum f. sp. lycopersici MEDIANTE LA TÉCNICA PCR DE DOBLE FUSIÓN. Gallegos Almanza I. A. (1) ; Martínez Cadena M. G. (2) ; Ferrel Cano L. E. (2). (1) Facultad
APÉNDICE. Apéndice 1. Espectrofotómetro. (Users Manual 2100 Series Spectrophotometer) 68
 APÉNDICE Apéndice 1. Espectrofotómetro (Users Manual 2100 Series Spectrophotometer) 68 El espectrofotómetro de la marca UNICO serie 2100 UV posee un rango de longitud de onda de 200-1000 nm. La técnica
APÉNDICE Apéndice 1. Espectrofotómetro (Users Manual 2100 Series Spectrophotometer) 68 El espectrofotómetro de la marca UNICO serie 2100 UV posee un rango de longitud de onda de 200-1000 nm. La técnica
AQUAGESTION. ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico.
 AQUAGESTION ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico. Noviembre 2011 ACTUALIDAD Este año Sernapesca confirma
AQUAGESTION ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico. Noviembre 2011 ACTUALIDAD Este año Sernapesca confirma
versus PCR en Tiempo Real PCR convencional no es cuantitativo
 PCR convencional o PCR de punto final versus PCR en Tiempo Real PCR convencional no es cuantitativo S Lobos C - U de Chile 1 PCR clásico La cantidad de producto no está relacionada con la cantidad de DNA
PCR convencional o PCR de punto final versus PCR en Tiempo Real PCR convencional no es cuantitativo S Lobos C - U de Chile 1 PCR clásico La cantidad de producto no está relacionada con la cantidad de DNA
ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN
 Universidad Centroccidental Lisandro Alvarado Decanato de Ciencias de la Salud ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN Octubre 2009 INTRODUCCION La molécula de ADN (que es la que se
Universidad Centroccidental Lisandro Alvarado Decanato de Ciencias de la Salud ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN Octubre 2009 INTRODUCCION La molécula de ADN (que es la que se
BIOTECNOLOGÍA. 1.- Ingeniería Genética, ADN recombinante
 BIOTECNOLOGÍA 1.- Ingeniería Genética, ADN recombinante Técnica del ADN recombinante: es la más usada en ingeniería genética, esta basada en el uso de las enzimas de restricción o endonucleasas de restricción
BIOTECNOLOGÍA 1.- Ingeniería Genética, ADN recombinante Técnica del ADN recombinante: es la más usada en ingeniería genética, esta basada en el uso de las enzimas de restricción o endonucleasas de restricción
Tecnologías basadas en la PCR
 Tecnologías de marcadores moleculares para estudios de diversidad genética de plantas: Módulo de aprendizaje Tecnologías basadas en el ADN Tecnologías basadas en la PCR Fundamentos de la PCR Derechos de
Tecnologías de marcadores moleculares para estudios de diversidad genética de plantas: Módulo de aprendizaje Tecnologías basadas en el ADN Tecnologías basadas en la PCR Fundamentos de la PCR Derechos de
4. Materiales y Métodos. Los equipos que a continuación se mencionan se encuentran en el laboratorio de
 39 4. Materiales y Métodos 4.1 Equipos Los equipos que a continuación se mencionan se encuentran en el laboratorio de Ingeniería Ambiental de la Universidad de las Américas Puebla y en el Laboratorio de
39 4. Materiales y Métodos 4.1 Equipos Los equipos que a continuación se mencionan se encuentran en el laboratorio de Ingeniería Ambiental de la Universidad de las Américas Puebla y en el Laboratorio de
II. METODOLOGÍA. El proceso de elaboración del biodiesel se constituye de siete pasos fundamentales: 6.1. DETERMINACIÓN DE LOS GRAMOS DE CATALIZADOR
 II. METODOLOGÍA 6. PROCESO DE ELABORACIÓN El proceso de elaboración del biodiesel se constituye de siete pasos fundamentales: 1. Determinación de los gramos de catalizador 2. Preparación del Metóxido de
II. METODOLOGÍA 6. PROCESO DE ELABORACIÓN El proceso de elaboración del biodiesel se constituye de siete pasos fundamentales: 1. Determinación de los gramos de catalizador 2. Preparación del Metóxido de
