Cursos de Formación de la UCTS (2011) Tecnologías de alto rendimiento en genómica
|
|
|
- Julián Suárez Pérez
- hace 7 años
- Vistas:
Transcripción
1 Cursos de Formación de la UCTS (2011) Plataforma de Genómica / Plataforma de Diagnóstico Molecular Tecnologías de alto rendimiento en genómica 2ª Parte: Tecnologías de ultrasecuenciación y de enriquecimiento de secuencia.
2 Programa del curso De Sanger hacia NGS 454 de Roche Desarrollo de la tecnología Cómo funciona Aplicaciones Comparación con otros Sistemas NGS Sistema Nimblegen Cómo funciona Formatos Aplicaciones Análisis de datos de alta densidad (UEB)
3 Cualquier DNA puede ser secuenciado
4 Nature Reviews Genetics 9, , 2008 Genomas Secuenciados
5 Cronología de la Secuenciación 1953 Francis Crick and James Watson describen el modelo de la doble hélice del DNA Método secuenciación Wandering spot, Maxam y Gilbert 1975 Método secuenciación plus and minus, Sanger y Coulson 1ª GENERACIÓN 1996 Pal Nyrén & Mostafa Ronagh i publican método de la pirosecuenciación en el Royal Institute of Technology (Stockholm) Se publica la primera versión del genoma humano. Science 291 (5507): ; Nature 409 (6822): Proyecto Genoma Humano (13 años). U.S. Department of Energy and the NIH 1ª GENERACIÓN DNA sequencing by chemical degradation by Maxam y Gilbert. 2. Chain-terminator method by Sanger et al. Método usado durante los proximos 30 años phi X 174 Primer genoma de DNA completo secuenciado 11 genes en 5386 bases (cadena sencilla) Applied Biosystems comercializa el primer secuenciador automático, El modelo ABI 370. NGS:2ª GENERACIÓN Life Science comercializa el 1er ultrasecuenciador GS20 (20Mpb) 2006 Lanzamiento de SOLEXA (Illumina) Genoma de Venter mediante sec. Sanger automática (4 años) Lanzamiento de GS FLX de Roche (100Mbp) SOLID de Applied Biosystem Serie de reactivos Titanium de Roche (500Mbp). Genoma Watson mediante 454/ROCHE Nature 452, (17 April 2008) El Instituto Naiconal de Salud (NIH) empieza secuenciación a gran escala de diversos microorganismos, ej. E.coli NGS:3ª GENERACIÓN Genomes Project Método de secuenciaciación SingleMolecularRealTime
6 1ª Generación Secuenciación Método Sanger Fragmentación de DNA Clonaje en Vectores; Transformación Bacterias; crecimiento y aislamiento vector DNA Ciclo Secuenciación Sanger sequencing: - Long reads ( bp) - Low throughput (192 reactions/run) Secuencia: 3 GACTAGATACGACGAGCGTGA 5 Primer: 5 CTGAT Electroforesis ( 1 Secuencia/Capilar) CTATGCTCG Polimerasa dntps ddntps marcados
7 2ª Generación Secuenciación Los Instrumentos de secuenciación de 2ª generación pueden generar tantos datos en un día como los generados por varios cientos de secuenciadores con capilares tipo Sanger, obtenidos por una sola persona.
8 2ª Generación Secuenciación ROCHE GS FLX 454 GS FLX+ 454 GS Junior 454 illumina Solexa Life Technology SOLiD 3System SOLiD 4 System 5500 System 5500xl System Ion Torrent System
9 Servicio Ultrasecuenciación UCTS GS 454 de ROCHE GS FLX GS Junior
10 Cúantas muestras se pueden secuenciar por run? 1ª Generación 3100 ABI Metal coated PTP reduces crosstalk 29 µm well diameter (20/bead) 3,400,000 wells per PTP 2ª Generación GS ROCHE 96p-Plates 384p-Plates PicoTiterPlate_FLX 70x70mm PicoTiterPlate_Junior
11 GS FLX/Junior 454 Troughput PTP Gaskets 35 -Tamaño de lo que quiero secuenciar -Coverage -Multiplexar (MIDS) N= (GxC)/Mbp por región PTP Donde: N= num de muestras que puedo secuenciar en un run G= tamaño de lo que quiero secuenciar C=Coverage (C= N * L / G)
12 GS FLX/Junior 454 Workflow gdna, Amplicones, cdna 1.Calidad & Cantidad Material de partida 2. Construcción Librería 3. Amplificación mediante empcr 4. Secuenciación Datos Obtenidos
13 1. Calidad & Cantidad Material de partida 1.1 Calidad mediante Chips Bioanalyzer; gel agarosa gdna, RNA 1.2 Cuantificación mediante Picogreen (gdna) o Ribogreen (RNA) y = 34,577x - 61,596 R 2 = 0,9994 Fluorescence Lam bda DNA (ng/m L) Fluorímetro FLx800
14 GS FLX/Junior 454 Workflow gdna, Amplicones, RNA 1.Calidad & Cantidad Material de partida 2. Construcción Librería 3. Amplificación mediante empcr 4. Secuenciación Datos Obtenidos
15 2. Construcción Librería Fragmentación Selección Tamaño Ligación Adaptadores Librería Shotgun Librería Pair-End Librería cdna gdna, RNA PCR con Fusion Primers Librería Amplicones Adaptador A (44 bases): Adaptador B (44 bases) Fusion Primers Primer Amplificación Primer 4 nucleótidos Secuenciación Key Biotina Primer Amplificación Primer 4 nucleótidos Secuenciación Key Adaptador A Target Adaptador B Target
16 2. Construcción Librería: Fragmentación gdna Librerías Shotgun NEBULIZACIÓN Rotura utilizando nitrógeno a alta presión DNA genómico Fragmentos de DNA de doble cadena 2.1 bar (30psi) Librerías Pair-End HYDROSHEAR gdna Fuerzas de rotura hidrodinámicas Orificio gdna fragmentado
17 2. Construcción Librería: Fragmentación RNA Librerías cdna RNA Random Primers First Strand Synthesis Solución de Fragmentación de RNA Second Strand Synthesis Fragmentos de cdna de doble cadena
18 2. Construcción Librería: Selección fragmentos gdna Nebulizado: DNA 7500 Lab Chip AMPure beads SPRI (Solid Phase Reversible Immobilization) DNA 7500 LabChip 50pb-1000pb 300pb-1000pb gdna fragmentado con Hydroshear: RNA Pico 6000 LabChip Electroelución 500pb-600 nt Tamaño medio de nt (dep. del contenido en GC) Menos del 10% 300 nt, no adaptor dimers Conc >0.2 ng/µl (Ribogreen )
19 2. Construcción Librería Inmobilización Fragmentos y aislamiento de la Librería: AB AB Melt Solution BB AA 4 tipos de productos resultan de la ligación Los productos con Biotina (AB, BA, BB) se unen a bolas magnéticas que llevan estreptavidina. Los products AA son lavados y eliminados. Mediante Melt Solution (NaOH0.1N) las cadenas no biotiniladas de cada fragmento de dsdna son aisladas. Ambas cadenas de los fragmentos BB quedarán unidas a las bolas. Sólo se aislan cadenas de DNA sencilla AB constituyendo la librería.
20 2. Construcción Librería: Q&Q Librería Molecules/µl = - Num de Avogadro es 6.022x1023 (moléculas/mole) x10 9 (gramos/mole) es peso molecular medio de nts. -Perfil típico de una librería ssdna (Agilent 2100 RNA Pico 6000 LabChip): Tamaño medio de bp -Cuantificación mediante Ribogreen -Dilución de trabajo para empcr
21 GS FLX/Junior 454 Workflow gdna, Amplicones, cdna 1.Calidad & Cantidad Material de partida 2. Construcción Librería 3. Amplificación mediante empcr 4. Secuenciación Datos Obtenidos
22 3. Amplificación mediante empcr Antes de la empcr: high-speed shaker -1 starting effective fragment per microreactor - ~10 6 microreactors per ml - All processed in parallel (Amplificación clonal)
23 3. Amplificación mediante empcr Después de la PCR: Rotura y Recuperación Contaje % Recuperación= DNA-beads/ml Input beads x100 Enrequecimiento de beads con DNA: Melt dsdna Unión de Primer marcado con Biotina a bolas de captura con ssdna Adición de bolas magnéticas con estreptavidina Melt % Enrequecimiento= DNA-beads/ml Input beads x100
24 empcr Titulación sólo para GS FLX Antes de la empcr: Cuántas copias de librería por Beads de captura son óptimas? 1. Procesar 4 tubos emulsiones Tubo Moléculas de Librería por Bead de Captura (cpb) Vol Librería Diluida µl µl µl µl 2. Recuperación y enrequecimiento de cada tubo 3. Contaje de las beads enriquecidas 4. Escoger el ratio copia/bead con aproximadamente un 8% de enrequecimiento
25 GS FLX/Junior 454 Workflow gdna, Amplicones, cdna 1.Calidad & Cantidad Material de partida 2. Construcción Librería 3. Amplificación mediante empcr 4. Secuenciación Datos Obtenidos
26 4. Secuenciación Metal coated PTP reduces crosstalk 29 µm well diameter (20/bead) 3,400,000 wells per PTP Gaskets
27 4. Secuenciación Secuenciación mediante síntesis Química basada en la pirosecuenciación Sulfurylase Luciferase Luciferina Polimerasa añade nucleótidos (datp) Se libera pirofosfato (PPi) Sulfurilasa crea ATP a partir del PPi Luciferasa hidroliza ATP y usa luciferina para producir luz. Light + oxyluciferin
28 4. Secuenciación Flujo de Reactivos Nucleotides are flowed sequentially across the PTPone at a time (200 cycles à4 bases) Pyrophosphate signal generation upon complimentary nucleotide incorporation dark otherwise The CCDcamera is generating a image after every flow The signal strength is proportional to the number of nucleotides incorporated
29 Flowgama y Base calling: 4. Secuenciación
30 4. Secuenciación:Ejemplo
31 MULTIPLEXACIÓN DE MUESTRAS MIDS: -Los MIDs son secuencias cortas que se añaden a los fragmentos a secuenciar durante la generación de librería y permiten identificar cada muestra de manera individual. Primer Amplificación Primer 4 nucleótidos Secuenciación Key Biotina Primer Amplificación Primer 4 nucleótidos Secuenciación Key Adaptador A Target MIDS Adaptador B Target MIDS MIDS MIDS -Permite aumentar el número de muestras por PTP: -separación física: gaskets pérdida física de espacio en la placa -separación por código de barras -Utilizando las dos posibilidades anteriores, aumenta el número de muestras a secuenciar por placa: -Kit comercial de 12 MIDs (diseñados por Roche) 12 muestras/reg. -División de la PTP en 16 reg. con gaskets TOTAL: 12 MIDs/reg. * 16 reg. = 192 muestras por PTP (máx) (INCLUSO MÁS)
32 Multiplexado de Muestras Multiplexado de amplicones MID2-Amplicón 2 MID4-Amplicón 4 MID1-Amplicón 1 MID3-Amplicón 3 MID5-Amplicón 5 MID6-Amplicón 6 Amplicón 11 Amplicón 7 Amplicón 8 Amplicón 9 Amplicón 10 Amplicón 12
33 SISTEMA GS FLX 454-APLICACIONES -Secuenciación de DNA a partir de muestras de especies extinguidas (shot-gun, paired-end) -Estudios de epigenética: amplicones -ChIP y secuenciación de los fragmentos de DNA presentes en los IPs -Metilación: conversión con bisulfito, amplificación de las regiones conteniendo islas CpG y secuenciación. -Ensamblaje de genomas eucariotas y procariotas completos, tanto de novo como resecuenciación (shot-gun +paired-end) -SAGE (Serial Analysis of Gene Expression Ditags): análisis cuantitativo y cualitativo del transcriptoma (shot-gun) -Caracterización y cuantificación de poblaciones virales a través de la secuenciación de genes diana (ej: transcriptasa reversa en VIH). Detección de quasiespecies (amplicones). -Metagenómica: estudio del contenido genómico en una mezcla compleja de microorganismos (microbiota, muestras medioambientales). Determinación tanto cuantitativa como cualitativa (shot-gun, retrotranscripción de RNA total o de mrna, amplicones de 16S rrna)
34 SISTEMA GS FLX 454-APLICACIONES -Secuenciación de genomas de pequeño tamaño (virales, mitocondriales) o de plásmidos (shot-gun) -Secuenciación de RNAs de pequeño tamaño (micrornas, sirnas): generación del cdna de doble cadena como material de partida (shot-gun) -Detección de SNPs, InDels, CNV (shot-gun) -Análisis del transcriptoma (partiendo de RNA total o mrna), cuantitativo o cualitativo (comparación de niveles de expresión) (retrotranscripción y shot-gun) -Enriquecimiento de regiones del genoma/captura del exoma utilizando arrays de captura de Nimblegen. Secuenciación de las regiones capturadas (shot-gun). En función de la aplicación, puede ser necesario completar los datos de 454 utilizando otras tecnologías, p.ej. Resolución de homopolímeros utilizando Sanger o lecturas cortas de Illumina. En general, se recomienda validar siempre los resultados utilizando otro tipo de aproximaciones: arrays, secuenciación Sanger, PCR a tiempo real, otras tecnologías de ultrasecuenciación...
35 Especificaciones Sistemas GS FLX & GS Junior
36 El futuro de la secuenciación 454
37 Programa del curso De Sanger hacia NGS 454 de Roche Desarrollo de la tecnología Cómo funciona Aplicaciones Comparación con otros Sistemas NGS Sistema Nimblegen Cómo funciona Formatos Aplicaciones Análisis de datos de alta densidad (UEB)ç
38 Comparación Plataformas secuenciación GS FLX 454 Chemistry based on pirosequencing Sample amplified by emulsion PCR Read length bp >1 million reads per run Mb of sequence ~10 hours run HiSeq 2000-Illumina Chemistry based on reversible terminators Sample amplified by solidphase amplification Read length 2x100 bp 3 billions reads per run 600 Gb of sequence 2-11 days run ABI SOLID 5500xl Chemistry based on sequencing by ligation Sample amplified by emulsion PCR Read length bp million reads per run Gb of sequence 4-8 days run
39 Comparación Plataformas secuenciación
40 Comparación Plataformas secuenciación
41 Comparación Plataformas secuenciación
42 Ejemplos de Genomas humanos secuenciados Nature Reviews Genetics 11, (January 2010)
43 Comparación Plataformas secuenciación 1ª Generación 2ª Generación
44 3ª Generación Secuenciación SCIENCE Vol JANUARY 2009 Real-Time DNA Sequencing from Single Polymerase Molecules John Eid, * Adrian Fehr, * Jeremy Gray, * Khai Luong, * John Lyle, * Geoff Otto, * Paul Peluso, * David Rank, * Primo Baybayan, Brad Bettman, Arkadiusz Bibillo, Keith Bjornson, Bidhan Chaudhuri, Frederick Christians, Ronald Cicero, Sonya Clark, Ravindra Dalal, Alex dewinter, John Dixon, Mathieu Foquet, Alfred d Gaertner, Paul Hardenbol, Cheryl Heiner, Kevin Hester, David Holden, Gregory Kearns, Xiangxu Kong, Ronald Kuse, Yves Lacroix, Steven Lin, Paul Lundquist, Congcong Ma, Patrick Marks, Mark Maxham, Devon Murphy, Insil Park, Thang Pham,, Michael Phillips, Joy Roy, Robert Sebra, Gene Shen, Jon Sorenson, Austin Tomaney, Kevin Travers, Mark Trulson, John Vieceli, Jeffrey Wegener, Dawn Wu, Alicia A Yang, Denis Zaccarin, Peter Zhao, Frank Zhong, Jonas Korlach, Stephen n Turner. Press Release Pacific Biosciences Announces Early Access Customers for Its Single Molecule Real Time System Eleven Leading Companies Support Launch of Third-generation DNA Sequencing MENLO PARK, Calif., Feb 23, 2010 Pacific Biosciences, a private company developing a disruptive technology platform for real-time detection of biological events at single molecule resolution, today announced the 10 institutions that have purchased its Single Molecule Real Time (SMRT(TM)) DNA sequencing system as part of the company's early access program in North America.
45 Programa del curso De Sanger hacia NGS 454 de Roche Desarrollo de la tecnología Cómo funciona Aplicaciones Comparación con otros Sistemas NGS Sistema Nimblegen Cómo funciona Formatos Aplicaciones Análisis de datos de alta densidad (UEB)
46 NIMBLEGEN: Arrays de Captura Los arrays de captura de secuencia de Nimblegen permiten capturar y enriquecer regiones génicas de interés, contiguas o no, con una elevada sensibilidad y especificidad, que luego pueden amplificarse y secuenciarse mediante tecnologías de alto rendimiento (454/Illumina). -Este sistema permite secuenciar regiones de interés en vez de genomas completos, con lo cual el coste de la secuenciación se reduce considerablemente. Técnicamente, el proceso también es menos costoso. -Sistema flexible: las regiones de interés pueden ser contiguas o no en el genoma. -Nimblegen diseña los arrays a la carta, solamente es necesario facilitarles las coordenadas de los genes diana. 1) Formato sólido -Arrays a la carta, con dos posibles tamaños de captura: 5 Mb ó 30 Mb por array. -Arrays de captura del exoma: prediseñados, contienen exones humanos codificantes y 551 exones para mirna (34 Mb), utilizando 2,1 millones de sondas. El listado de genes que contienen estos arrays puede consultarse en la web de Nimblegen ( -2) Formato en solución -Arrays de captura del exoma: prediseñados, contienen exones humanos codificantes y 551 exones para mirna (34 Mb). Existe una versión LR (long-read) optimizada para secuenciación con 454. Disponible en dos formatos, para 4 reacciones y para 48 reacciones. Próximamente existirá este formato para arrays de 5 Mb.
47 NIMBLEGEN: Arrays de Captura
48 PROTOCOLO DE ARRAYS DE CAPTURA EN SÓLIDO 3. Pre-capture amplification 4. Hybridization a) Ensamblaje del array b) Carga del array c) Hibridación: 42º C, h
49 PROTOCOLO DE ARRAYS DE CAPTURA EN SOLUCIÓN Streptavidin beads Pre-capture amplification Primers biotinilados 3. Hybridization 47 ºC, horas
50 CONTROL DE CALIDAD DE LA CAPTURA MEDIANTE qpcr La eficiencia teórica de una qpcr es del 100% y significa que las secuencias diana se doblan en cada ciclo, es decir, que E=2. Sin embargo, la eficiencia real nunca es del 100% y por eso el valor de E debe calcularse empíricamente para cada sonda. Los locus control NSC permiten determinar el enriquecimiento de un pequeño set de locus control estandarizados que se encuentran dentro de un rango de eficiencias de captura conocidas. Estos ensayos permiten hacer una estimación aproximada del enriquecimiento de poblaciones mayores de genes diana sin necesidad de secuenciarlos. Si la qpcr de estos locus control indica una captura correcta, es muy problable que los locus experimentales de interés también hayan sido capturados satisfactoriamente.
51 TECNOLOGÍA DE NIMBLEGEN -Arrays de enriquecimiento de secuencia -CGH arrays CGX / CNV / Whole genome / Whole genome-exon focused / Custom -ChIP-chip arrays Whole genome / Promoter / Custom -Arrays de metilación Whole genome / Promoter / Custom -Arrays de expresión génica Whole genome / Promoter / Custom
52 OTRAS TECNOLOGÍAS DE ENRIQUECIMIENTO DE SECUENCIA -Sistema SureSelect (Agilent): arrays de captura en solución. Optimizada para la secuenciación con Illumina, SOLiD y 454. Existen versiones prediseñadas para capturar el exoma y el noma humanos, o bien pueden diseñarse ensayos a la carta (captura de 3.3 ó 6.6 Mb). Las muestras pueden indexarse después de la captura para optimizar el rendimiento de la ultrasecuenciación (=MIDs de Roche). Existe también un formato sólido que permite capturar hasta 1 Mb. -Sistema Febit (ABI). Para ver una descripción de cómo funciona el sistema:
53 PAUTAS PARA EL DISEÑO EXPERIMENTAL DE UN ESTUDIO DE ULTRASECUENCIACIÓN
54 UCTS WORKFLOW Researcher Statistics and Bioinformatics (UEB) UCTS EXPERIMENTAL DESIGN QUALITY SAMPLES COLLECTION RESULTS CHECKING EXPERIMENTS SAMPLE PROCESSING DATA ANALYSIS SEQUENCING UEB UCTS Others
55 El futuro de la secuenciación 454
56 Fin
57 I. ANÁLISIS DE VARIANTES VIRALES UTILIZANDO UN ENSAYO DE AMPLICONES PAUTAS PARA LA GENERACIÓN DE UNA LIBRERÍA DE AMPLICONES -La secuenciación puede hacerse de manera unidireccional o bidireccional (en ambos sentidos, forward y reverse, separadamente). -Si la secuenciación es unidireccional, la generación de los amplicones es también unidireccional. Este método es más cómodo, especialmente cuando se quiere trabajar con diferentes loci diana en muchas muestras, utilizando para la amplificación primers genespecíficos (1 set por loci ). -En general se obtienen mejores resultados utilizando secuenciación bidireccional. Esto es especialmente importante cuando se desean buscar variantes con baja frecuencia en una población. Si la secuenciación es bidireccional deben amplificarse los fragmentos de PCR con primers para ambos sentidos, forward y reverse. -Para la generación de la librería de amplicones debe usarse una polimerasa de alta fidelidad. -La cuantificación de la librería debe hacerse siempre por un método fluorimétrico (Picogreen ) porque los métodos espectrofotométricos (absorbancia) tienden a sobreestimar la cantidad de ADN.
58 Programa del curso De Sanger hacia NGS 454 de Roche Desarrollo de la tecnología Cómo funciona Aplicaciones Comparación con otros Sistemas NGS Sistema Nimblegen Cómo funciona Formatos Aplicaciones Análisis de datos de alta densidad (UEB)
59 I. ANÁLISIS DE VARIANTES VIRALES UTILIZANDO UN ENSAYO DE AMPLICONES Diseño de los primers para generar una librería de amplicones Usuario a) Librería de amplicones unidireccional Primers genéricos SIN adaptadores de secuenciación para GS FLX Titanium y SIN MIDs. Generación de una librería de tipo shot-gun donde se añaden los adaptadores de secuenciación y los MIDs. b) Librería de amplicones bidireccional -Se une a las Beads de captura (EmPCR) -Complementaria a los primers de amplificación & secuenciación Sequencing key 400 bp bp (máx. 800 bp)* Fusion primer MIDs: Multiplex Identifiers (diseñados por Roche) *Amplicones <200 pb ó >1000 bp también pueden secuenciarse con modificaciones en el protocolo.
60 II. SECUENCIACIÓN DE NOVO DE GENOMAS BACTERIANOS UTILIZANDO LAS APLICACIONES PAIRED-END Y SHOT-GUN
61 II. SECUENCIACIÓN DE NOVO DE GENOMAS BACTERIANOS UTILIZANDO LAS APLICACIONES PAIRED-END Y SHOT-GUN
62 II. SECUENCIACIÓN DE NOVO DE GENOMAS BACTERIANOS UTILIZANDO LAS APLICACIONES PAIRED-END Y SHOT-GUN
63 II. SECUENCIACIÓN DE NOVO DE GENOMAS BACTERIANOS UTILIZANDO LAS APLICACIONES PAIRED-END Y SHOT-GUN
64 EL MÉTODO DE SECUENCIACIÓN DEPENDE DEL TAMAÑO DEL GENOMA DNA mitocondrial humano: 16.5 Kb Saccharomyces cerevisiae: 12.5 Mb Human exome: 34 Mb Human genome: 3 Gb Drosophila melanogaster: Mb E. coli O157:H7: 5.44 Mb Caenorhabditis elegans: 100 Mb Sanger Nº de bases
65 SECUENCIACIÓN DE GENOMAS HUMANOS COMPLETOS!!!! Nature 463, (18 Feb.2010) Suppl. Info
66 SECUENCIACIÓN DE GENOMAS HUMANOS COMPLETOS AÑO 2010 Nature 463, (18 Feb.2010) Genomas completos 1 bosquimano (total 4 participantes) + 1 bantú Bosq. 1: 454 Titanium shotgun (10.2x) + 20 Kb paired-end (12.3x) Bosq. 2: 454 Titanium (2x) Bantú: SOLiD 3.0 (> 30x) Todos: captura del exoma con arrays de Nimblegen + secuenciación 454 Titanium > 16x Validación: comparación de la secuencia completa con la secuencia del exoma, resecuenciación del genoma completo de dos individuos con Illumina (23.2x, 7.2x), validación de SNPs (Illumina arrays, Taqman, Sanger).
67 SECUENCIACIÓN DE GENOMAS HUMANOS COMPLETOS AÑO 2010 Nature 463, (11 Feb.2010) Nature 463, (21 Jan. 2010)
68 Solexa Genome Analyzer de Illumina
69 SOLEXA Workflow 1. Fragmentación y Ligación de adaptadores 2. Amplificación en fase sólida y generación de cluster mediante Bridge amplificación 3. Flujo de Terminadores reversibles e incorporación de una base en cada ciclo Incorporación 4 dntps Cada uno marcado con un fluorocromo Lavado, imagen de los 4 colores Corte de fluorocromos y grupos terminadores, lavado Incorporación 4 dntps Cada uno marcado con un fluorocromo Lavado, imagen de los 4 colores Se repiten los ciclos 4. Flujo de Terminadores reversibles e incorporación de una base en cada ciclo
70 SOLiD 3 de Applied Biosystem
71 1. Generación Librería 2. empcr y enriquecimiento SOLiD Workflow (Sequencing by Oligonucleotide Ligation and Detection ) 4.Secuenciación mediante Ligación 3. Deposición beads
72 Secuenciación mediante ligación
73 Comparación Plataformas secuenciación: Método amplificación 454 SOLID Polonotor SOLEXA
74 CÓMO PROCEDER PARA HACER UN ARRAY DE CAPTURA DE SECUENCIA 1. El investigador recibe el array diseñado de Nimblegen. La UCTS hace el proceso completo: generación de la librería a partir del DNA genómico con los adaptadores de secuenciación para GS FLX 454, captura de secuencia mediante el array y ultrasecuenciación. 2. El investigador envía el DNA genómico a Nimblegen para que ellos generen la librería y hagan la captura de secuencia. El investigador recibirá la muestra capturada con unos adaptadores genéricos. La UCTS genera otra librería con los adaptadores para 454 y procede con la ultrasecuenciación. REQUISITOS DEL DNA DE PARTIDA DNA genómico humano no amplificado y no fragmentado (no debe mostrar un smear en gel de agarosa/bionanalyzer) UCTS Mín. 10 µg de DNA, C 300 ng/µl en TE, cuantificado por un método fluorimétrico como el Picogreen (UCTS), con A 260 /A y A 260 /A DNA humano o de otros organismos (QC por el propio investigador) DNA genómico humano no amplificado y no fragmentado (no debe mostrar un smear en gel de agarosa/bionanalyzer) NIMBLEGEN Mín. 21 µg de DNA, C= ng/µl, con A 260 /A y A 260 /A Sólo aceptan DNA genómico humano.
75
Temas actuales: Next Generation Sequencing (NGS) Bioinformática Elvira Mayordomo
 Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Temas actuales: Next Generation Sequencing (NGS) Bioinformática 16-3-16 Elvira Mayordomo Veremos Historia Plataformas NGS Aplicaciones Retos bioinformáticos Dogma central Secuenciación de Sanger El DNA
Introducción a la Secuenciación Masiva y a la Bioinformática
 Introducción a la Secuenciación Masiva y a la Bioinformática Dietmar Fernández Orth, PhD 24 de Abril de 2014 1 El DNA (Ácido desoxirribonucleico) contiene la información genética usada en el desarrollo
Introducción a la Secuenciación Masiva y a la Bioinformática Dietmar Fernández Orth, PhD 24 de Abril de 2014 1 El DNA (Ácido desoxirribonucleico) contiene la información genética usada en el desarrollo
NEXT GENERATION SEQUENCING TECHNOLOGIES AND APPLICATIONS
 CURS OF BIOINFORMATICS FOR BIOMEDICAL RESEARCH Vall d Hebron Institut de Recerca (VHIR) Institut d Investigació Sanitària acreditat per l Instituto de Salud Carlos III (ISCIII) NEXT GENERATION SEQUENCING
CURS OF BIOINFORMATICS FOR BIOMEDICAL RESEARCH Vall d Hebron Institut de Recerca (VHIR) Institut d Investigació Sanitària acreditat per l Instituto de Salud Carlos III (ISCIII) NEXT GENERATION SEQUENCING
Aplicaciones y Tendencias en secuenciación de ADN
 Aplicaciones y Tendencias en secuenciación de ADN Agus%n Arasanz Duque Unidad de Genómica Centres Cien%fics i Tecnològics Universidad de Barcelona Aplicaciones y Tendencias en secuenciación de ADN Introducción
Aplicaciones y Tendencias en secuenciación de ADN Agus%n Arasanz Duque Unidad de Genómica Centres Cien%fics i Tecnològics Universidad de Barcelona Aplicaciones y Tendencias en secuenciación de ADN Introducción
INGENIERÍA GENÉTICA. Sinónimos: Manipulación genética Clonaje génico Tecnología del DNA recombinante
 INGENIERÍA GENÉTICA Es una revolución metodológica que ha puesto a nuestra disposición gran número de técnicas procedentes de diversos campos de la ciencia con un objetivo común muy importante: el acceso
INGENIERÍA GENÉTICA Es una revolución metodológica que ha puesto a nuestra disposición gran número de técnicas procedentes de diversos campos de la ciencia con un objetivo común muy importante: el acceso
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP
 SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
I SIMPOSIO Biopsia líquida
 I SIMPOSIO Biopsia líquida Detección de mutaciones en biopsia líquida con tecnología CLART SANTIAGO DE COMPOSTELA, 30/01/16 Biopsia líquida: ADNtc Biopsia líquida comprende tres biomarcadores diferentes:
I SIMPOSIO Biopsia líquida Detección de mutaciones en biopsia líquida con tecnología CLART SANTIAGO DE COMPOSTELA, 30/01/16 Biopsia líquida: ADNtc Biopsia líquida comprende tres biomarcadores diferentes:
Métodos de secuenciación de ADN
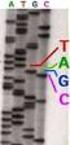 Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
Secuenciar una molécula de ADN consiste en determinar en qué orden se disponen los cuatro nucleótidos (A, T, C y G) que componen la molécula.
 SECUENCIACIÓN DEL ADN Secuenciar una molécula de ADN consiste en determinar en qué orden se disponen los cuatro nucleótidos (A, T, C y G) que componen la molécula. El primer método diseñado para secuenciar
SECUENCIACIÓN DEL ADN Secuenciar una molécula de ADN consiste en determinar en qué orden se disponen los cuatro nucleótidos (A, T, C y G) que componen la molécula. El primer método diseñado para secuenciar
2001 < + 2006 89:8"I%;(
 ! "#$ $%&"'( ))! - ' (()*+ 1953 1977./0 1 / + 1973./, C).0-1975 1987./,C)- 5+,.0- + *+, -+./0123 "#$% 4 55167 %( '-9 $) "';1A2+ ' (()*+, '(()*+ 1996 2005 200 7 2008 23 D4 5)" / %E (+ 2001 < + %2A(512F5G
! "#$ $%&"'( ))! - ' (()*+ 1953 1977./0 1 / + 1973./, C).0-1975 1987./,C)- 5+,.0- + *+, -+./0123 "#$% 4 55167 %( '-9 $) "';1A2+ ' (()*+, '(()*+ 1996 2005 200 7 2008 23 D4 5)" / %E (+ 2001 < + %2A(512F5G
NUEVAS TECNOLOGÍAS EN LOS LABORATORIOS DE GENÉTICA CLÍNICA
 NUEVAS TECNOLOGÍAS EN LOS LABORATORIOS DE GENÉTICA CLÍNICA Sebastián Blesa Luján Laboratorio de Estudios Genéticos, Fundación para la Investigación del Hospital Clínico Universitario de Valencia. TÉCNICAS
NUEVAS TECNOLOGÍAS EN LOS LABORATORIOS DE GENÉTICA CLÍNICA Sebastián Blesa Luján Laboratorio de Estudios Genéticos, Fundación para la Investigación del Hospital Clínico Universitario de Valencia. TÉCNICAS
Plataforma de Genómica
 Plataforma de Genómica Dr. Víctor J. Asensio Técnico de Plataforma 27 de febrero de 2015 La Plataforma de Genómica del IdisPa está ubicada en el Hospital Universitario Son Espases; consta de dos salas
Plataforma de Genómica Dr. Víctor J. Asensio Técnico de Plataforma 27 de febrero de 2015 La Plataforma de Genómica del IdisPa está ubicada en el Hospital Universitario Son Espases; consta de dos salas
Dra. Ing. Agr. Sabrina Costa Tártara Departamento de Tecnología
 Que podemos encontrar en los ácidos nucleicos de los organismos? Megabases de datos inexploradas que nos pueden dar información sobre la diversidad de los organismos. Dra. Ing. Agr. Sabrina Costa Tártara
Que podemos encontrar en los ácidos nucleicos de los organismos? Megabases de datos inexploradas que nos pueden dar información sobre la diversidad de los organismos. Dra. Ing. Agr. Sabrina Costa Tártara
Cursos de Formación de la UCTS (2011) Tecnologías de alto rendimiento en genómica
 Cursos de Formación de la UCTS (2011) Plataforma de Genómica / Plataforma de Diagnóstico Molecular Tecnologías de alto rendimiento en genómica 2ª Parte: Tecnologías de ultrasecuenciación y de enriquecimiento
Cursos de Formación de la UCTS (2011) Plataforma de Genómica / Plataforma de Diagnóstico Molecular Tecnologías de alto rendimiento en genómica 2ª Parte: Tecnologías de ultrasecuenciación y de enriquecimiento
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
NTC = NO TEMPLATE CONTROL
 NTC = NO TEMPLATE CONTROL Químicos usados en la qpcr para detectar los productos: específicos o inespecíficos Inespecíficos: agentes intercalantes que se unen al DNA doble hebra y detectan cualquier producto.
NTC = NO TEMPLATE CONTROL Químicos usados en la qpcr para detectar los productos: específicos o inespecíficos Inespecíficos: agentes intercalantes que se unen al DNA doble hebra y detectan cualquier producto.
Cursos de SANIDAD. Principios de Biología Molecular A distancia 80 h
 Cursos de SANIDAD [ Principios de Biología Molecular ] A distancia 80 h PRINCIPIOS DE BIOLOGÍA MOLECULAR El curso de Principios de biología molecular da a conocer a los participantes la estructura y propiedades
Cursos de SANIDAD [ Principios de Biología Molecular ] A distancia 80 h PRINCIPIOS DE BIOLOGÍA MOLECULAR El curso de Principios de biología molecular da a conocer a los participantes la estructura y propiedades
Genética Medicina Nicolás Jouve Curso
 7. Los antecedentes metodológicos de los Proyectos Genoma. Historia del Proyecto Genoma Humano. Fundamentos y Objetivos. Metodología básica: corte de ADN, separación de fragmentos, clonación, librerías
7. Los antecedentes metodológicos de los Proyectos Genoma. Historia del Proyecto Genoma Humano. Fundamentos y Objetivos. Metodología básica: corte de ADN, separación de fragmentos, clonación, librerías
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
Electroforesis de DNA
 Electroforesis de DNA DNA Topoisomerasas Izquierda Derecha Enzimas de restricción Clasificadas en 3 tipos, donde la mayoría pertenece al grupo II (proteínas monoméricas, poseen simetria rotacional y generalmente
Electroforesis de DNA DNA Topoisomerasas Izquierda Derecha Enzimas de restricción Clasificadas en 3 tipos, donde la mayoría pertenece al grupo II (proteínas monoméricas, poseen simetria rotacional y generalmente
Cursos de SANIDAD. Principios de Biología Molecular A distancia 80 h
 Cursos de SANIDAD [ Principios de Biología Molecular ] A distancia 80 h PRINCIPIOS DE BIOLOGÍA MOLECULAR El curso de Principios de biología molecular da a conocer a los participantes la estructura y propiedades
Cursos de SANIDAD [ Principios de Biología Molecular ] A distancia 80 h PRINCIPIOS DE BIOLOGÍA MOLECULAR El curso de Principios de biología molecular da a conocer a los participantes la estructura y propiedades
Soluciones validadas que aumentan la eficiencia en la rutina de trabajo de tu laboratorio
 Cristina Gómez, X workshop MRAMA iq-check TM Real Time PCR y medios cromogénicos RAPID Soluciones validadas que aumentan la eficiencia en la rutina de trabajo de tu laboratorio BIO-RAD la compañía Fundada
Cristina Gómez, X workshop MRAMA iq-check TM Real Time PCR y medios cromogénicos RAPID Soluciones validadas que aumentan la eficiencia en la rutina de trabajo de tu laboratorio BIO-RAD la compañía Fundada
Protocolo de desarrollo y producción de paneles génicos de apoyo al diagnóstico
 Protocolo de desarrollo y producción de paneles génicos de apoyo al diagnóstico 1 ÍNDICE CAPÍTULO PÁGINA 1. Objeto... 3 2. Definiciones... 3 3. Proceso de producción.... 4 4. Tiempo de respuesta en la
Protocolo de desarrollo y producción de paneles génicos de apoyo al diagnóstico 1 ÍNDICE CAPÍTULO PÁGINA 1. Objeto... 3 2. Definiciones... 3 3. Proceso de producción.... 4 4. Tiempo de respuesta en la
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón
 RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
LA EVOLUCIÓN DE LA GENÓMICA
 UAM 10 julio 2014 1 LA EVOLUCIÓN DE LA GENÓMICA 2 LOS DOS OBJETIVOS DE LA GENÓMICA IDENTIFICAR GENES CUANTIFICAR GENES DNA & RNA 3 CONOCER LOS GENES Y SUS VARIANTES IMPACTA EN EL ESTADO DE SALUD Y ENFERMEDAD
UAM 10 julio 2014 1 LA EVOLUCIÓN DE LA GENÓMICA 2 LOS DOS OBJETIVOS DE LA GENÓMICA IDENTIFICAR GENES CUANTIFICAR GENES DNA & RNA 3 CONOCER LOS GENES Y SUS VARIANTES IMPACTA EN EL ESTADO DE SALUD Y ENFERMEDAD
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80
 AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
TECNICAS DE BIOLOGIA MOLECULAR
 Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
APLICACIONES DE LAS TECNOLOGÍAS DE NGS
 APLICACIONES DE LAS TECNOLOGÍAS DE NGS Rosa M. Prieto APLICACIONES DE LAS TECNOLOGÍAS DE NGS 1. ESTUDIO DE QUASIESPECIES VIRALES (HEPATITIS B Y C) UTILIZANDO AMPLICONES 2. ESTUDIOS DE METAGENÓMICA 3. SECUENCIACIÓN
APLICACIONES DE LAS TECNOLOGÍAS DE NGS Rosa M. Prieto APLICACIONES DE LAS TECNOLOGÍAS DE NGS 1. ESTUDIO DE QUASIESPECIES VIRALES (HEPATITIS B Y C) UTILIZANDO AMPLICONES 2. ESTUDIOS DE METAGENÓMICA 3. SECUENCIACIÓN
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO
 SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
1. Contaminación de agua y alimentos por microorganismos patógenos 1
 INTRODUCCIÓN GENERAL 1. Contaminación de agua y alimentos por microorganismos patógenos 1 2. Papel del agua en la transmisión de enfermedades infecciosas 2 2.1. Las aguas residuales 2 2.2. El agua potable
INTRODUCCIÓN GENERAL 1. Contaminación de agua y alimentos por microorganismos patógenos 1 2. Papel del agua en la transmisión de enfermedades infecciosas 2 2.1. Las aguas residuales 2 2.2. El agua potable
Utilidad de las técnicas de biología molecular en el manejo de bacterias fastidiosas.
 Utilidad de las técnicas de biología molecular en el manejo de bacterias fastidiosas. Estudio rutinario de las bacterias en el laboratorio Examen microscópico (en fresco, Gram, Ziehl- Neelsen, otros) Cultivo
Utilidad de las técnicas de biología molecular en el manejo de bacterias fastidiosas. Estudio rutinario de las bacterias en el laboratorio Examen microscópico (en fresco, Gram, Ziehl- Neelsen, otros) Cultivo
Single Cell Genomics. María Méndez Lago. Seminario tecnológico Facultad de Farmacia, UB 5.05.2015
 Single Cell Genomics María Méndez Lago 5.05.2015 Seminario tecnológico Facultad de Farmacia, UB Contenido 1. Introducción a la secuenciación de nueva generación (NGS) y a la genómica de células individuales
Single Cell Genomics María Méndez Lago 5.05.2015 Seminario tecnológico Facultad de Farmacia, UB Contenido 1. Introducción a la secuenciación de nueva generación (NGS) y a la genómica de células individuales
PCR a tiempo real. Sección de Biología Molecular, Servicio de Apoyo a la Investigación, Universidad de Murcia
 PCR a tiempo real Sección de Biología Molecular, Servicio de Apoyo a la Investigación, Universidad de Murcia PCR a tiempo real (PCRrt) Aplicaciones: - Cuantificación de ácidos nucleicos (AQ). - Estudio
PCR a tiempo real Sección de Biología Molecular, Servicio de Apoyo a la Investigación, Universidad de Murcia PCR a tiempo real (PCRrt) Aplicaciones: - Cuantificación de ácidos nucleicos (AQ). - Estudio
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN
 COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN OBJETIVO: el presente trabajo práctico tiene como objetivo principal que el alumno adquiera destreza en el análisis de electroferogramas generados por equipos
COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN OBJETIVO: el presente trabajo práctico tiene como objetivo principal que el alumno adquiera destreza en el análisis de electroferogramas generados por equipos
TECNICAS BASADAS EN ADN FINGERPRINTING SECUENCIACIÓN
 TECNICAS BASADAS EN ADN FINGERPRINTING SECUENCIACIÓN Nombre: Dra. Claudia Etchebehere Institución: Grupo de Ecología Microbiana. Instituto de Investigaciones Biológicas Clemente Estable, Montevideo, Uruguay
TECNICAS BASADAS EN ADN FINGERPRINTING SECUENCIACIÓN Nombre: Dra. Claudia Etchebehere Institución: Grupo de Ecología Microbiana. Instituto de Investigaciones Biológicas Clemente Estable, Montevideo, Uruguay
Repaso de PCR y qpcr
 Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Por qué es importante estudiar la función del genoma?
 GENÓMICA FUNCIONAL Por qué es importante estudiar la función del genoma? La función de los genes sólo puede ser enteramente entendida en el contexto de todo el organismo y su contenido genético El entender
GENÓMICA FUNCIONAL Por qué es importante estudiar la función del genoma? La función de los genes sólo puede ser enteramente entendida en el contexto de todo el organismo y su contenido genético El entender
Nuevas perspectivas en el diagnóstico de Clostridium difficile. Evelyn Rodríguez Cavallini
 Nuevas perspectivas en el diagnóstico de Clostridium difficile Evelyn Rodríguez Cavallini Pruebas diagnósticas Detectan productos de C. difficile Métodos de cultivo Detección de genes o deleciones GDH
Nuevas perspectivas en el diagnóstico de Clostridium difficile Evelyn Rodríguez Cavallini Pruebas diagnósticas Detectan productos de C. difficile Métodos de cultivo Detección de genes o deleciones GDH
 Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Simposio SEAP Marcadores Moleculares Emergentes y Su Potencial Utilidad Clínica
 Simposio SEAP Marcadores Moleculares Emergentes y Su Potencial Utilidad Clínica Fausto J. Rodríguez M.D. Profesor Asistente Departamento de Patología Clínica Mayo Rochester, MN Resumen Nuevas Tecnologías
Simposio SEAP Marcadores Moleculares Emergentes y Su Potencial Utilidad Clínica Fausto J. Rodríguez M.D. Profesor Asistente Departamento de Patología Clínica Mayo Rochester, MN Resumen Nuevas Tecnologías
UNIVERSIDAD JUAREZ AUTOMA DE TABASCO
 UNIVERSIDAD JUAREZ AUTOMA TABASCO DIVISIÒN ACAMICA CIENCIAS BIOLOGICAS LICENCITURA EN BIOLOGIA ASIGNATURA: INGENIERIA GENETICA AREA FORMACION: TRANSVERSAL HORAS TEORICAS: 3 HORAS PRACTICAS: 3 CREDITOS:
UNIVERSIDAD JUAREZ AUTOMA TABASCO DIVISIÒN ACAMICA CIENCIAS BIOLOGICAS LICENCITURA EN BIOLOGIA ASIGNATURA: INGENIERIA GENETICA AREA FORMACION: TRANSVERSAL HORAS TEORICAS: 3 HORAS PRACTICAS: 3 CREDITOS:
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones
 TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones Dra. Mariana L. Ferramola Curso de Técnicas Moleculares Aplicadas a Bioquímica Clínica. Área de Qca. Biológica
TÉCNICAS DE DIAGNÓSTICO MOLECULAR Variantes de PCR; detección de delecciones e inserciones Dra. Mariana L. Ferramola Curso de Técnicas Moleculares Aplicadas a Bioquímica Clínica. Área de Qca. Biológica
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica
 Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema1 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 1 Genoma Humano Proyecto Genoma Humano Controversias
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema1 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 1 Genoma Humano Proyecto Genoma Humano Controversias
Transcriptómica bacteriana
 Transcriptómica bacteriana Fernando Rojo CNB-CSIC CH3-(CH2)n-CH3 CH3-(CH2)n-CH2OH CH3-(CH2)n-CHO CH3-(CH2)n-COOH CH3-(CH2)n-COSCoA β-oxidación Qué é pretende la biotecnología bacteriana? Utilizar bacterias
Transcriptómica bacteriana Fernando Rojo CNB-CSIC CH3-(CH2)n-CH3 CH3-(CH2)n-CH2OH CH3-(CH2)n-CHO CH3-(CH2)n-COOH CH3-(CH2)n-COSCoA β-oxidación Qué é pretende la biotecnología bacteriana? Utilizar bacterias
1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr. Criterios generales para el diseño de cebadores para RT qpcr
 Diseño de cebadores para RT qpcr ACTIVIDADES 1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr con SYBR Green 2 Mostración del diseño de un par de cebadores para cuantificar
Diseño de cebadores para RT qpcr ACTIVIDADES 1 Guías Generales para el Diseño de cebadores para cuantificación de ARNm por RT qpcr con SYBR Green 2 Mostración del diseño de un par de cebadores para cuantificar
Universidad de Los Andes Facultad de Medicina Instituto Inmunología Clínica Maestría en Inmunología
 TÉCNICAS EN BIOLOGÍA MOLECULAR RESPONSABLE: Dra. Lisbeth Berrueta. CREDITOS: 2 El curso de Técnicas de Biología Molecular está destinado a profesionales del área biomédica con conocimientos básicos relativos
TÉCNICAS EN BIOLOGÍA MOLECULAR RESPONSABLE: Dra. Lisbeth Berrueta. CREDITOS: 2 El curso de Técnicas de Biología Molecular está destinado a profesionales del área biomédica con conocimientos básicos relativos
Secuenciación del ARN
 Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN
Biotina: se usa uno de los dntps biotinilado. Detección: Streptavidina conjugada con enzima (ALP) o anti-biotina. Usos: NB, SB, hibridación in situ
 Northern blot Marcación no radioactiva: Digoxigenina: se usa uno de los dntps marcado con digoxigenina Detección: Anticuerpo conjugado con enzima (ALP) o fluorocromo Biotina: se usa uno de los dntps
Northern blot Marcación no radioactiva: Digoxigenina: se usa uno de los dntps marcado con digoxigenina Detección: Anticuerpo conjugado con enzima (ALP) o fluorocromo Biotina: se usa uno de los dntps
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Estrategias para la secuenciación de Genomas y NGS
 Taller ensamblaje y anotación de Genomas y Metagenomas usando Galaxy Estrategias para la secuenciación de Genomas y NGS Alejandro Reyes PhD Sep=embre 12 de 2015 Universidad de los Andes El camino al genoma
Taller ensamblaje y anotación de Genomas y Metagenomas usando Galaxy Estrategias para la secuenciación de Genomas y NGS Alejandro Reyes PhD Sep=embre 12 de 2015 Universidad de los Andes El camino al genoma
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos
 Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
Técnicas moleculares para el diagnóstico de microorganismos fitopatógenos Dra. Ing. Agr. Sandra Alaniz Unidad de Fitopatología setiembre de 2015 Unidades taxonómicas Dominio Orden Familia Género Especie
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006
 IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
IV Curso Avanzado WHO GSS 2006 Buenos Aires 15 al 24 de mayo de 2006 REACCION EN CADENA DE LA POLIMERASA (PCR) EN EL DIAGNOSTICO DE Campylobacter jejuni/coli Viernes 19 de mayo María Rosa Viñas Servicio
DISTRIBUCIÓN HORARIA DE LA ASIGNATURA SEGÚN NORMATIVA
 GUÍA DOCENTE CURSO: 2016-17 DATOS BÁSICOS DE LA ASIGNATURA Asignatura: Genética Molecular Código de asignatura: 49152204 Plan: Grado en Biotecnología (Plan 2015) Año académico: 2016-17 Ciclo formativo:
GUÍA DOCENTE CURSO: 2016-17 DATOS BÁSICOS DE LA ASIGNATURA Asignatura: Genética Molecular Código de asignatura: 49152204 Plan: Grado en Biotecnología (Plan 2015) Año académico: 2016-17 Ciclo formativo:
Alberts Molecular Biology of the Cell 4ª, 5ª o 6ª Ed. (2002, 2008, 2014) Lehninger Principles of Biochemistry 4ª, 5ª o 6ª Ed. (2004, 2008, 2012)
 Bibliografía: Alberts Molecular Biology of the Cell 4ª, 5ª o 6ª Ed. (2002, 2008, 2014) Lehninger Principles of Biochemistry 4ª, 5ª o 6ª Ed. (2004, 2008, 2012) Lodish Molecular Cell Biology 5ª, 6ª o 7ª
Bibliografía: Alberts Molecular Biology of the Cell 4ª, 5ª o 6ª Ed. (2002, 2008, 2014) Lehninger Principles of Biochemistry 4ª, 5ª o 6ª Ed. (2004, 2008, 2012) Lodish Molecular Cell Biology 5ª, 6ª o 7ª
DISTRIBUCIÓN HORARIA DE LA ASIGNATURA SEGÚN NORMATIVA
 GUÍA DOCENTE CURSO: 2017-18 DATOS BÁSICOS DE LA ASIGNATURA Asignatura: Ingeniería Genética Código de asignatura: 49152202 Plan: Grado en Biotecnología (Plan 2015) Año académico: 2017-18 Ciclo formativo:
GUÍA DOCENTE CURSO: 2017-18 DATOS BÁSICOS DE LA ASIGNATURA Asignatura: Ingeniería Genética Código de asignatura: 49152202 Plan: Grado en Biotecnología (Plan 2015) Año académico: 2017-18 Ciclo formativo:
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR).
 Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
Conceptos generales de Biología Molecular. Extracción de DNA y Reacción en Cadena de la Polimerasa (PCR). Olga Redondo González MIR 3 Análisis Clínicos Hospital Universitario de Guadalajara, 25 abril 2007
TIPIFICACION HLA. Bioq. Eliana Palomino Lab. De Histocompatibilidad Hospital Privado
 TIPIFICACION HLA Bioq. Eliana Palomino Lab. De Histocompatibilidad Hospital Privado Tipificación HLA: Estudio mediante el cual se determina cuales de todas las variantes conocidas del locus HLA están presentes
TIPIFICACION HLA Bioq. Eliana Palomino Lab. De Histocompatibilidad Hospital Privado Tipificación HLA: Estudio mediante el cual se determina cuales de todas las variantes conocidas del locus HLA están presentes
Investigación en genes
 Investigación en genes Conocer el genóma de los organismos Modificar la dotación génica natural Tecnología a del ADN recombinante Conocimientos básicos que posibilitaron el desarrollo de la Tecnología
Investigación en genes Conocer el genóma de los organismos Modificar la dotación génica natural Tecnología a del ADN recombinante Conocimientos básicos que posibilitaron el desarrollo de la Tecnología
La Patología a Molecular y el Control de Calidad
 UNIDAD DIDÁCTICA 7: LOS PROCESOS EN ANATOMÍA PATOLÓGICA La Patología a Molecular y el Control de Calidad Asunción n Olmo Sevilla Master Diagnóstica S. L. Patología Molecular: Patología del futuro X Personal:
UNIDAD DIDÁCTICA 7: LOS PROCESOS EN ANATOMÍA PATOLÓGICA La Patología a Molecular y el Control de Calidad Asunción n Olmo Sevilla Master Diagnóstica S. L. Patología Molecular: Patología del futuro X Personal:
Seminario Genética. Técnicas de Biología Molecular
 Seminario Genética Técnicas de Biología Molecular Contenidos Extracción de ADN Enzimas de restricción PCR Southern Blot Secuenciación método de Sanger y automática 1º Extracción de ADN Se pueden tomar
Seminario Genética Técnicas de Biología Molecular Contenidos Extracción de ADN Enzimas de restricción PCR Southern Blot Secuenciación método de Sanger y automática 1º Extracción de ADN Se pueden tomar
HOSPITAL UNIVERSITARI VALL D'HEBRON INSTITUT DE RECERCA RED NACIONAL DE CENTROS DE CÁNCER PROGRAMA DE GENÓMICA Y PROTEÓMICA
 HOSPITAL UNIVERSITARI VALL D'HEBRON INSTITUT DE RECERCA RED NACIONAL DE CENTROS DE CÁNCER PROGRAMA DE GENÓMICA Y PROTEÓMICA CATÁLOGO DE OFERTA DE SERVICIOS GENÓMICOS I. INFORMACIÓN GENERAL: NOMBRE DE UNIDAD:
HOSPITAL UNIVERSITARI VALL D'HEBRON INSTITUT DE RECERCA RED NACIONAL DE CENTROS DE CÁNCER PROGRAMA DE GENÓMICA Y PROTEÓMICA CATÁLOGO DE OFERTA DE SERVICIOS GENÓMICOS I. INFORMACIÓN GENERAL: NOMBRE DE UNIDAD:
La caja de herramientas para el reumatólogo del siglo XXI: el lenguaje de la Genómica
 La caja de herramientas para el reumatólogo del siglo XXI: el lenguaje de la Genómica Ana María Blasini Santelli Centro Nacional de Enfermedades Reumáticas, Hospital Universitario de Caracas, Universidad
La caja de herramientas para el reumatólogo del siglo XXI: el lenguaje de la Genómica Ana María Blasini Santelli Centro Nacional de Enfermedades Reumáticas, Hospital Universitario de Caracas, Universidad
PCR Punto de No Retorno de la Biología Molecular
 TÉCNICAS DE ANÁLISIS EN BIOLOGÍA MOLECULAR CURSO DE BIOTECNOLOGÍA ELEMENTAL Biotechnology Explorer Julio 2012 PCR Punto de No Retorno de la Biología Molecular Qué es un gen Técnicas de aislamiento y estudio
TÉCNICAS DE ANÁLISIS EN BIOLOGÍA MOLECULAR CURSO DE BIOTECNOLOGÍA ELEMENTAL Biotechnology Explorer Julio 2012 PCR Punto de No Retorno de la Biología Molecular Qué es un gen Técnicas de aislamiento y estudio
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA SECUENCIACIÓN Y ENSAMBLADO
 TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA SECUENCIACIÓN Y ENSAMBLADO Luis Valencia Cabrera Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia
TÉCNICAS INTELIGENTES EN BIOINFORMÁTICA SECUENCIACIÓN Y ENSAMBLADO Luis Valencia Cabrera Mario de J. Pérez Jiménez Grupo de investigación en Computación Natural Dpto. Ciencias de la Computación e Inteligencia
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Métodos de determinación del virus del papiloma humano (HPV) en cribado de cáncer de cérvix
 Métodos de determinación del virus del papiloma humano (HPV) en cribado de cáncer de cérvix Beatriz Bellosillo Servicio de Anatomía Patológica Hospital del Mar, Barcelona Virus del Papiloma Humano- HPV
Métodos de determinación del virus del papiloma humano (HPV) en cribado de cáncer de cérvix Beatriz Bellosillo Servicio de Anatomía Patológica Hospital del Mar, Barcelona Virus del Papiloma Humano- HPV
Biocomputación. http://bioinfo2.ugr.es/ Curso 2011/2012 José L. Oliver (oliver@ugr.es) Michael Hackenberg (hackenberg@ugr.es) Bioinformatics, UGR
 Biocomputación Curso 2011/2012 José L. Oliver (oliver@ugr.es) Michael Hackenberg (hackenberg@ugr.es) biocomp/ De ~nada a ~todo Biología Molecular: un gen una proteína un gen un laboratorio un gen una tesis
Biocomputación Curso 2011/2012 José L. Oliver (oliver@ugr.es) Michael Hackenberg (hackenberg@ugr.es) biocomp/ De ~nada a ~todo Biología Molecular: un gen una proteína un gen un laboratorio un gen una tesis
El laboratorio de Microbiología y Parasitología en el diagnóstico de las enfermedades tropicales importadas
 El laboratorio de Microbiología y Parasitología en el diagnóstico de las enfermedades tropicales importadas Juan Carlos Rodríguez S. Microbiología Hospital General Universitario de Alicante Universidad
El laboratorio de Microbiología y Parasitología en el diagnóstico de las enfermedades tropicales importadas Juan Carlos Rodríguez S. Microbiología Hospital General Universitario de Alicante Universidad
Técnicas moleculares.
 Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Organización y estructura de genomas
 Organización y estructura de genomas ORGANIZACIÓN DE LOS GENOMAS 1. Un gene es un segmento de DNA que al expresarse da un producto funcional que puede ser una proteína o un RNA. 2. Un genoma es el conjunto
Organización y estructura de genomas ORGANIZACIÓN DE LOS GENOMAS 1. Un gene es un segmento de DNA que al expresarse da un producto funcional que puede ser una proteína o un RNA. 2. Un genoma es el conjunto
APLICADAS A LA ECOLOGÍA MICROBIANA
 TÉCNICAS MOLECULARES APLICADAS A LA ECOLOGÍA MICROBIANA Áreas de estudio tradicionales en la Ecología Microbiana: - Estudio de la diversidad microbiana, incluye aislamiento, identificación y cuantificación
TÉCNICAS MOLECULARES APLICADAS A LA ECOLOGÍA MICROBIANA Áreas de estudio tradicionales en la Ecología Microbiana: - Estudio de la diversidad microbiana, incluye aislamiento, identificación y cuantificación
Viejas y nuevas tecnologías. enfermedades genéticas
 Viejas y nuevas tecnologías en el estudio de enfermedades genéticas Miguel A. Garcia-Gonzalez, PhD. Hospital Clínico San Carlos 5 de Abril, 2014 Contenido 1 Por qué? 2 Qué es la secuenciación? 3 Tipos
Viejas y nuevas tecnologías en el estudio de enfermedades genéticas Miguel A. Garcia-Gonzalez, PhD. Hospital Clínico San Carlos 5 de Abril, 2014 Contenido 1 Por qué? 2 Qué es la secuenciación? 3 Tipos
Técnicas de marcado no radioactivo
 Sondas moleculares Técnicas de marcado no radioactivo Mariano N. Belaich, Vanina A. Rodríguez, Facundo Temprana Universidad Nacional de Quilmes Sondas moleculares Fragmento de DNA o RNA (marcado) de tamaño
Sondas moleculares Técnicas de marcado no radioactivo Mariano N. Belaich, Vanina A. Rodríguez, Facundo Temprana Universidad Nacional de Quilmes Sondas moleculares Fragmento de DNA o RNA (marcado) de tamaño
Reacción en cadena de la Polimerasa, sus aplicaciones y algunos elementos de ecología molecular. Polymerase Chain Reaction (PCR)
 Reacción en cadena de la Polimerasa, sus aplicaciones y algunos elementos de ecología molecular Polymerase Chain Reaction (PCR) PCR? PCR = síntesis en progresión exponencial de secuencias objetivos (locus)
Reacción en cadena de la Polimerasa, sus aplicaciones y algunos elementos de ecología molecular Polymerase Chain Reaction (PCR) PCR? PCR = síntesis en progresión exponencial de secuencias objetivos (locus)
Evolución de la genética molecular hasta el chip 50K
 Jornada Sobre Selección Genómica en los Programas de Mejora Genética Animal Madrid, 5 de Mayo de 2010 Evolución de la genética molecular hasta el chip 50K Armand Sánchez Bonastre Departamento de Ciencia
Jornada Sobre Selección Genómica en los Programas de Mejora Genética Animal Madrid, 5 de Mayo de 2010 Evolución de la genética molecular hasta el chip 50K Armand Sánchez Bonastre Departamento de Ciencia
ABREVIATURAS... XI I. INTRODUCCIÓN EL TOMATE... 3
 ÍNDICE ABREVIATURAS... XI I. INTRODUCCIÓN... 1 1 EL TOMATE... 3 1.1 Taxonomía... 3 1.2 Características generales... 3 1.3 La flor... 4 1.4 Características del fruto... 5 2 CUAJADO Y DESARROLLO DEL FRUTO...
ÍNDICE ABREVIATURAS... XI I. INTRODUCCIÓN... 1 1 EL TOMATE... 3 1.1 Taxonomía... 3 1.2 Características generales... 3 1.3 La flor... 4 1.4 Características del fruto... 5 2 CUAJADO Y DESARROLLO DEL FRUTO...
Catálogo de Servicios GenXPro
 Catálogo de Servicios GenXPro Aplicando las mejores y más sensibles técnicas de preparación de muestras, en combinación con las técnicas más modernas de secuenciación de última generación, GenXPro ofrece
Catálogo de Servicios GenXPro Aplicando las mejores y más sensibles técnicas de preparación de muestras, en combinación con las técnicas más modernas de secuenciación de última generación, GenXPro ofrece
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ
 REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
Ingeniería Genética en Eucariontes
 Universidad de Chile Facultad de Ciencias Químicas y Farmacéuticas Departamento de Bioquímica y Biología Molecular l Ingeniería Genética en Eucariontes Bibliografía : Recombinant DNA, Third Edition: Genes
Universidad de Chile Facultad de Ciencias Químicas y Farmacéuticas Departamento de Bioquímica y Biología Molecular l Ingeniería Genética en Eucariontes Bibliografía : Recombinant DNA, Third Edition: Genes
Secuenciación de Nueva Generación (NGS) para la Caracterización y Análisis de Organismos Genéticamente Modificados (OGM)
 Secuenciación de Nueva Generación (NGS) para la Caracterización y Análisis de Organismos Genéticamente Modificados (OGM) Marco legal De acuerdo con el Protocolo de Cartagena sobre seguridad de la biotecnología
Secuenciación de Nueva Generación (NGS) para la Caracterización y Análisis de Organismos Genéticamente Modificados (OGM) Marco legal De acuerdo con el Protocolo de Cartagena sobre seguridad de la biotecnología
Marcadores moleculares (MM)
 Marcadores moleculares (MM) Un marcador genético o marcador molecular es un segmento de ADN situado en un lugar específico del genoma (locus) y cuya herencia genética se puede rastrear. Puede ser un gen
Marcadores moleculares (MM) Un marcador genético o marcador molecular es un segmento de ADN situado en un lugar específico del genoma (locus) y cuya herencia genética se puede rastrear. Puede ser un gen
Tema 8. Mapas físicos Genómica estructural
 Tema 8 Mapas físicos Genómica estructural Genoma Genómica Genómica estructural Genómica funcional Mapas genéticos Mapas físicos EXPRESIÓN Transcriptoma Proteoma Interactoma FUNCIÓN Genética inversa Secuenciacion
Tema 8 Mapas físicos Genómica estructural Genoma Genómica Genómica estructural Genómica funcional Mapas genéticos Mapas físicos EXPRESIÓN Transcriptoma Proteoma Interactoma FUNCIÓN Genética inversa Secuenciacion
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
Medicina y Biología Molecular y Celular
 Medicina y Biología Molecular y Celular Molecular Cell Biology is at the center of Modern Medicine Diseases such as cancer, heart disease, diabetes and arthritis all involve molecules and/or cells that
Medicina y Biología Molecular y Celular Molecular Cell Biology is at the center of Modern Medicine Diseases such as cancer, heart disease, diabetes and arthritis all involve molecules and/or cells that
Instituto de Biomedicina y Biotecnología de Cantabria (IBBTEC) SERVICIO DE SECUENCIACIÓN MASIVA
 1. DESCRIPCIÓN DEL SERVICIO El servicio de Secuenciación Masiva tiene como finalidad el proporcionar asesoramiento y soporte técnico a los grupos de investigación interesados en realizar proyectos de ultrasecuenciación.
1. DESCRIPCIÓN DEL SERVICIO El servicio de Secuenciación Masiva tiene como finalidad el proporcionar asesoramiento y soporte técnico a los grupos de investigación interesados en realizar proyectos de ultrasecuenciación.
Métodos de secuenciación de ácidos nucleicos. Gonzalo Greif. Unidad de Biología Molecular. Institut Pasteur Montevideo. 1. Un poco de historia.
 Gonzalo Greif. Unidad de Biología Molecular. Institut Pasteur Montevideo. 1. Un poco de historia 2. Secuenciación por método de Maxxam y Gilbert 3. Secuenciación por método de Sanger a. Avances en el método
Gonzalo Greif. Unidad de Biología Molecular. Institut Pasteur Montevideo. 1. Un poco de historia 2. Secuenciación por método de Maxxam y Gilbert 3. Secuenciación por método de Sanger a. Avances en el método
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
Informe técnico del proyecto. de diagnóstico genético de pacientes con. síndrome de Dravet y espectros asociados
 Informe técnico del proyecto de diagnóstico genético de pacientes con síndrome de Dravet y espectros asociados Introducción El objetivo principal de este proyecto es realizar un diagnóstico genético a
Informe técnico del proyecto de diagnóstico genético de pacientes con síndrome de Dravet y espectros asociados Introducción El objetivo principal de este proyecto es realizar un diagnóstico genético a
Roche y Tecnologías de Secuenciación de Alto Rendimiento Miguel Álvarez Tejado. 16 Octubre 2014
 Roche y Tecnologías de Secuenciación de Alto Rendimiento Miguel Álvarez Tejado. 16 Octubre 2014 IMPORTANT NOTICE Intended Use Unless explicitly stated otherwise, all Roche products and services referenced
Roche y Tecnologías de Secuenciación de Alto Rendimiento Miguel Álvarez Tejado. 16 Octubre 2014 IMPORTANT NOTICE Intended Use Unless explicitly stated otherwise, all Roche products and services referenced
Yolanda Loarce Tejada. Universidad de Alcalá Patrimonio de la Humanidad
 Yolanda Loarce Tejada GENOMA: conjunto de genes que llevan los organismos (1920) GENÓMICA: el estudio de los genomas Estructural Funcional Comparativa El significado de una PALABRA depende de la secuencia
Yolanda Loarce Tejada GENOMA: conjunto de genes que llevan los organismos (1920) GENÓMICA: el estudio de los genomas Estructural Funcional Comparativa El significado de una PALABRA depende de la secuencia
CARACTERISTICAS GENERALES DEL GENOMA. D ra. M a r í a I s a b e l F o n s e c a
 CARACTERISTICAS GENERALES DEL GENOMA D ra. M a r í a I s a b e l F o n s e c a Genoma (gen, origen) Es el conjunto total del material genético hereditario presente en la célula, tanto nuclear como extranuclear,
CARACTERISTICAS GENERALES DEL GENOMA D ra. M a r í a I s a b e l F o n s e c a Genoma (gen, origen) Es el conjunto total del material genético hereditario presente en la célula, tanto nuclear como extranuclear,
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA
 UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO FACULTAD DE MEDICINA VETERINARIA Y ZOOTECNIA. Estudio comparativo del diagnóstico del virus del PRRS por RT-PCR, PCR anidada y RT-PCR en tiempo real. Zoalli Angélica
