Clase PCR II. Reacción en cadena de la polimerasa. Biología Molecular, Bioquímica 2016 Ivana Kraiselburd
|
|
|
- Juan Antonio Cabrera Núñez
- hace 5 años
- Vistas:
Transcripción
1 Clase PCR II Reacción en cadena de la polimerasa Biología Molecular, Bioquímica 2016 Ivana Kraiselburd
2 PCR: es una técnica para amplificar ácidos nucleicos in vitro, que permite la obtención de grandes cantidades de un fragmento específico de DNA a partir de un molde complejo El ADN de interés es amplificado exponencialmente mediante la síntesis enzimática dirigida por una DNA polimerasa termoestable cebada por 2 oligonucleótidos cebadores (o primers) y por el agregado de desoxirribonucleótidos trifosfatos (dntps)
3 Etapas de cada ciclo de la PCR Desnaturalización: el ADN doble hebra se separa en dos hebras a altas temperaturas (94-95ºC, durante 45 s-1 min). Depende del contenido de GC del molde y su longitud Hibridación de los cebadores a la zona específica de cada una de las hebras por complementariedad de bases. Temperaturas más bajas (55-65º C, durante 30 seg- 1min). T y t dependen de las características particulares de los cebadores utilizados Elongación o extensión del cebador por la DNA polimerasa, la cual cataliza la incorporación de desoxirribonucleótidos siguiendo la cadena molde en la dirección 5-3. Esta etapa será realizada a la temperatura óptima de reacción de la ADN polimerasa (72 C para la Taq polimerasa). Los tiempos dependerán de la prosesividad de la enzima y de la longitud del fragmento a amplificar
4 Conceptos de la PCR Sensibilidad: cantidad mínima de DNA necesaria para que se produzca la amplificación, es decir, para obtener un producto (banda en gel). Se relaciona con los falsos negativos, ya que puede ocurrir que una muestra sea positiva pero sea dada como negativa porque no se ha amplificado por no tener suficiente cantidad de DNA Especificidad: se refiere a la obtención de un solo producto amplificado. Viene determinada por los oligos y la especificidad con la que se unen al DNA molde. De esta forma, si los oligos tienen más de un sitio al que se pueden unir aparecerá más de un producto amplificado Eficiencia- rendimiento: se refiere a la amplificación máxima que se puede obtener en un número determinado de ciclos Fidelidad: se refiere a los errores que comete la DNA polimerasa durante la amplificación. Este concepto es de especial importancia en la secuenciación, pero en otros casos no es tan importante. Una buena fidelidad permite evitar falsos positivos y/o negativos
5 Sensibilidad N de moléculas blanco presentes en la muestra Complejidad de las secuencias blanco Sensibilidad Diseño del Primer Enzima Optimización de la Reacción Concentración de Magnesio Formulación del Buffer Temperatura de Anillado Especificidad-Fidelidad T de anillado (compromiso) Número de ciclos Concentración de Magnesio Concentración y balance de Nucleótidos dntps (10-50 M) Elección de la Enzima Concentración de Primers-cebadores suficientemente alta para la unión rápida al DNA blanco
6 Componentes esenciales de la PCR estándar DNA polimerasa termoestable Oligonucleótidos iniciadores (primers o cebadores) Cationes divalentes: Mg 2+ (MgCl 2 ) Buffer (para mantener el ph de la enzima) DNA molde Desoxirribonucleótidos trifosfatos dntps: se deben utilizar soluciones equimoleculares de los desoxirribonucleótidos datp, dgtp, dctp y dttp H 2 O de alta calidad (doble destilada estéril o MiliQ). Cationes monovalentes Co-Solventes: estabilizan la enzima, influencia la procesividad y/o la T m.
7 DNA polimerasas termoestables Llevan a cabo la síntesis de DNA dependiente del molde Diferentes orígenes:taq Thermus aquaticus, Pwo Pyrococcus woesei, Pfu Pyrococcus furiosus, Tli Thermococcus littoralis Estabilidad: Taq 9 min a 97 C (40 min a 95 C), Pwo >2 hr a 100 C Procesividad: número promedio de nucleótidos añadidos a la cadena de ADN que se sintetiza, antes de separarse de la cadena molde Fidelidad: frecuencia de incorporación de un nucleótido equivocado (depende MgCl 2, dntps, ph, daño térmico, etc). Fidelidad: Taq baja; Pfu alta; Pfx alta Actividad correctora (proofreading, exonucleasa 3-5 ) y/o reparadora (exonucleasa 5-3 )
8 Características de algunas DNA polimerasas termoestables Taq Stoffel Vent Pfu Fuente Thermus aquaticus Taq truncada sin exo 5-3 Thermococus litoralis Pirococus furiosus Peso molecular Velocidad Procesividad 94 kda 61 kda? 92kDa 75nt/s + 50nt/s + 80nt/s 60 nt/s Vida ½ (95 C) 40 min 90 min 360 min + 2h Exo 5-3 SI NO SI Exo 3-5 NO NO SI SI Mg 2+ óptima 1,5 mm 3 mm 2 mm 1,5-2 mm KCl óptima 50 mm 10 mm 10 mm 10 mm
9 Taq polimerasa Aislada de Thermus aquaticus Fidelidad baja Tasa de error total para la Taq polimerasa: introducción de errores de aproximadamente 1 cada nucleótidos No posee actividad 3-5 exonucleasa Alta procesividad Es muy importante determinar la concentración adecuada de la taq polimerasa en la PCR (0.5 y 2.5 U por reacción) Presenta actividad transferasa terminal en el extremo 3 (agrega una A al extremo 3, especialmente si en el extremo hay una C). Pfu, Pwo y Tli generan extremos romos
10 DNA molde Puede ser ssdna o dsdna (simple o doble cadena) En general se utilizan cantidades que van desde 10 a 500 ng dependiendo de la naturaleza de la secuencia a amplificar, ADN genómico (1-100 ng) o plasmídico (2-10 ng), representación del gen de interés en el genoma, etc Usualmente se utilizan varios miles de copias, ej: 1µg de humano, 10 ng de levadura, 1 ng de bacteriano DNA circular cerrado es levemente menos efectivo que el DNA lineal Se puede amplificar a partir de una sola molécula de DNA molde, pero las condiciones deben estar muy optimizadas Debe estar completamente desnaturalizado (etapa previa) Calidad del molde (integridad: no fragmentado en trozos más pequeños de lo que queremos amplificar; proceso de extracción: que la muestra no presente agentes que inhiban a la actividad de la polimerasa como trazas de fenol o alcoholes o quelantes como EDTA que reducen la concentración de iones de Mg en la solución)
11 Oligonucleótidos iniciadores (primers, cebadores) Dos oligonucleótidos cebadores simple hebra complementarios a los extremos de la secuencia que se desea amplificar Presentan entre nt de longitud Se conoce su secuencia Es el factor más importante para la eficiencia y la especificidad del proceso Deben estar presentes en exceso (10 13 = 30 ciclos, 1kb) comparado con el DNA molde Requieren de un cuidadoso diseño Solución stock 10 µm, se usan 0,1-1 µm final en cantidad equimolar de cada oligonucleótido
12 Diseño de cebadores Manual o por programas de bioinformática Complementarios a la secuencia deseada Secuencia del oligo: contenido de GC entre 45-55% Tamaño estándar: 18 a 25 nt Extremos 3 de los oligos quedan enfrentados y deben estar bien apareados Evitar formación de estructuras secundarias: dímeros, autocomplementariedad (palíndromos o largos segmentos de polipurinas y polimirimidinas) e intercomplementariedad Agregado de linkers o sitios de restricción
13 Diseño de cebadores Consideraciones sobre la T melting y T annealing Temperatura de melting o fusión es la temperatura a la que la mitad de los oligos se encuentran hibridados y la mitad se encuentran como simple hebra. A mayor contenido GC más alta T m Si los primers se encuentran mal apareados, la amplificación será menos eficiente o puede no funcionar. Los cebadores con T m más altas pueden aparearse inespecíficamente a temperaturas más bajas y el cebador con la T m más baja puede no aparearse a temperaturas más altas Solamente se puede programar una temperatura en el termociclador o equipo de PCR. Si los cebadores poseen diferentes T m se elige como T annealing la más baja
14 Diseño de cebadores Las temperaturas de melting de los oligonucleótidos pueden ser estimadas usando la fórmula de Wallace: T m = 2(A+T) + 4(G+C) T a = T m - 4 (válida para oligos entre bases de rango, sino se usan fórmulas más complejas) Reglas de diseño: Longitud= 18-25bp Contenido de GC entre 45-55% Ambos cebadores deben tener T m similares (no mas de 5 ºC de diferencia entre ambos) Evitar las secuencias repetidas. No deberían presentar estructuras secundarias
15 Evitar secuencias repetidas 1. Caso de extremos 3 repetidos 5 -NNNNNNNNNNNNNNNTATA-3 5 -NNNNNNNNNNNNNNNTATA NNNNNNNNNNNNNNNTATA-3 3 -ATATNNNNNNNNNNNNNNN PCR Dímero del primer
16 Evitar secuencias repetidas 2. Caso de extremos 5 repetidos 5 -TATANNNNNNNNNNNNNNN-3 5 -TATANNNNNNNNNNNNNNN-3 3 -NNNNNNNNNNNNNNNATAT TATANNNNNNNNNNNNNNN PCR No hay extensión
17 Evitar secuencias repetidas 3. Caso de formación de horquillas 5 -NNNNNNNNNNNNNNNGCATGC-3 5 -NNNNNNNNNNNNNNNNNNNNN-3 5 -NNNNNNNNNNNNNNNGCA 3 -CGT PCR Productos de PCR no deseados
18 Temperatura y tiempo de anillado Rango estándar: entre 50 C - 65 C, s Mejoras en la especificidad: Incrementar la temperatura de anillado (incrementos de 2-5 C son recomendados) puesto que reduce las posibilidades de priming no específico y por lo tanto la formación de producto no específico Reducir los tiempos de anillado. Durante el anillado, los primers se hibridan rápidamente. Tiempos muy largos no mejoran normalmente la producción y pueden producir un aumento en el apareamiento inespecífico y así mayores cantidades de productos no específicos
19 Estrategias para lograr una selectiva amplificación en los primeros ciclos evitando el apareamiento inespecífico Hot start disminuye la inespecificidad causada por el anillado y la elongación a bajas T (agregado de la enzima taq polimerasa o el Mg 2+ a altas T ). Exclusión de un reactivo (dntps, taq, Mg 2+, primers) de la mezcla de reacción hasta que la reacción llega a la temperatura de desnaturalización del ciclo 94 C También puede ser con anti-taq (inhibe la actividad de la Taq polimerasa hasta la etapa de desnaturalización) Touchdown PCR (TD-PCR). Es una modificación de la PCR en la cual la temperatura de anillado inicial es mayor que la T m óptima de los primers y la T a es gradualmente reducida en los subsiguientes ciclos hasta que la T a óptima es alcanzada. Una reducción gradual de la temperatura hasta una temperatura más permisiva durante el curso de los ciclos favorece la amplificación del fragmento deseado
20 Iones Mg 2+ Cofactor requerido para las DNA polimerasas Estabiliza el DNA de doble cadena y eleva la T m, es importante para controlar la especificidad de la reacción Mg 2+ bajo, apareamiento más estricto. Pero muy pocos iones Mg 2+ producen un bajo rendimiento de producto de PCR Mg 2+ alto, aumenta el rendimiento de los productos pero favorece la inespecificidad promoviendo la incorporación errónea Exceso de Mg 2+ reduce la fidelidad de las DNA polimerasas y puede causar la generación de productos no deseados (en un gel aparecen como una escalera) Los dntps secuestran iones Mg 2+ (un cambio importante en la concentración de dntps en el buffer puede requerir el ajuste de la concentración de Mg 2+ en la mezcla de reacción) EDTA en el molde puede afectar la concentración de Mg 2+ libre. La Taq polimerasa requiere Mg 2+ libre para su actividad
21 Concentración de Magnesio Se utiliza en una concentración entre 1 y 4 mm La concentración de magnesio afecta los siguientes procesos: hibridación de los cebadores, temperatura de desnaturalización, especificidad del producto, formación de dímeros entre cebadores y la actividad y fidelidad enzimática Alto Mg 2+ : alto rendimiento; productos inespecíficos Bajo Mg 2+ : bajo rendimiento; alta especificidad Mejorar la especificidad: disminuir la concentración. Cuidado: concentraciones inadecuadas de Mg 2+ pueden dar lugar a un más bajo rendimiento Mejorar la eficiencia: aumentar la concentración. Cuidado: exceso de Mg 2+ tiende a causar reacciones no específicas
22 dntps Las concentraciones de los 4 desoxinucleótidos trifosfato (datp, dctp, dttp, dgtp) deben ser iguales de modo que ocurra la incorporación exacta (si un dntp está en una concentración más alta que otro será incorporado preferencialmente) Rango estándar de concentración: µm Las concentraciones de dntps utilizados en una reacción son determinados por la afinidad de la enzima por el sustrato (la Km de la enzima). Usar concentraciones mayores que la Km (Km para los dntps µm) Se preparan soluciones stock (5-10mM) que contienen cantidades equimoleculares de los dntps La concentración de dntps óptima depende de: Longitud del producto (200 µm puede alcanzar a sintetizar 12,5 µg DNA de 400 bp) MgCl 2 Astringencia (exigencias impuestas por el sistema, rigurosidad)
23 dntps Mejorar la eficiencia: para secuencias molde largas, incrementar la cantidad de dntps Mejorar la fidelidad: bajar la concentración dntps pero tener en cuenta la concentración de Mg 2+ que debe ser disminuida en una proporción equimolar (cambio en su concentración afecta la concentración de Mg 2+ disponible ya que el Mg 2+ forma un complejo soluble con los dntps) Mejorar la especificidad: reducir la concentración de dntp pero observar que la concentración de Mg 2+ proporción equimolar se debe bajar en una Alto dntps baja fidelidad alto rendimiento Bajo dntps alta fidelidad bajo rendimiento
24 Buffer de Reacción Proporciona las condiciones de reacción, que son importantes para la actividad de la enzima Por lo general está formado por: 10 mm tris-hcl (ph=8.4) 50 mm KCl 1.5 mm MgCl 2 Adyuvantes o aditivos: pueden ayudar en la práctica a aumentar la especificidad y fidelidad de la reacción en cadena de la polimerasa. El dimetilsulfóxido (DMSO) añadido al buffer de la reacción en un 10% contribuye a la disminución de la estructura secundaria del ADN. También se pueden usar detergentes como el tween 20, laureth 12 (0.1%) o Tritón x10, que ayudan a estabilizar la enzima. Existen también protocolos que incorporan polietilenglicol (PEG), glicerol, formamida, albúmina sérica bovina (BSA), etc, aunque no son imprescindibles Agua de buena calidad (destilada, desionizada y estéril o agua milliq) para completar el volumen final de la reacción
25 Número de ciclos Rango Estándar: ciclos Se necesitan >25 ciclos para amplificar un gen de copia única a partir de DNA genómico de mamífero Pocos ciclos: bajan el rendimiento Muchos ciclos: aumentan la inespecificidad Algunos componentes se tornan limitantes después de 30 ciclos El efecto plateau favorece las amplificaciones inespecíficas por lo que incrementar el n de ciclos no incrementa la especificidad o la eficiencia Mayor a 40 ciclos: se aumenta cantidad y complejidad de los productos no específicos. Mejorar la especificidad: Reducir el número de ciclos
26 Ejemplo de un protocolo de PCR COMPONENTE VOLUMEN (µl) CONCENTRACIÓN FINAL Agua autoclavada y ultra filtrada (ph 7) 20,7 10X Buffer de PCR 2,5 1X Mezcla dntps (25mM de cada nucleótido) 0,2 200 µm (cada nucleótido) Mezcla de primers (25 pmoles/µl cada uno) 0,4 0,4 µm (cada primer) Taq DNA polimerasa (10U/ µl) 0,2 2 U totales en 25 µl Molde DNA genómico (100 ng/ µl) ng totales en 25 µl c.s.p o C 30x o C o C 2 72 o C o C Molde Cebadores Enzima, longitud producto
27 Detección y Análisis de los resultados Línea 1: fragmento de PCR de aproximadamente 1850 pares de bases de longitud. Línea 2 y 4: los fragmentos son de aproximadamente 800 bases de longitud. Línea 3: no hay producto formado, la PCR falló. Línea 5: múltiples bandas son formadas a causa de que uno de los primers se unió a diferentes regiones del molde de DNA.
28 Consideraciones prácticas importantes PCR es una técnica muy sensible: evitar contaminaciones, ya que es posible que cantidades traza de ADN contaminante sirvan de ADN molde (producto no deseado, falsos positivos) 1. Contaminación por arrastre de ADN diana amplificado en PCR anteriores 2. Contaminación cruzada entre muestras, lo que supone la transferencia de ADN diana de una muestra a otra 3. ADN genómico de preparados de muestras anteriores Normas que ayudan a evitar las contaminaciones: Zonas de trabajo exclusivas para PCR, dotadas de su propio material, separadas de los pasos anteriores (extracción de ADN genómico, etc) y posteriores (corrida electroforética de los productos) Uso de guantes por el manipulador Utilización de reactivos y tubos estériles Cuidado en pipeteo del ADN (evitarse la formación de aerosoles) Las mesas y estantes se lavados periódicamente con lavandina al 10 %, seguida de etanol al 70 % y de ser posible tratadas con luz UV Preparación de la mezcla para la PCR (mezcla maestra) Controles: negativo (todos los componentes de la reacción excepto el ADN molde); positivo (material que se haya utilizado con éxito en PCR anteriores)
29 Introducir sitios de restricción mediante PCR Uno distinto en cada extremo: clonado direccional. Ventaja: no se puede ligar sobre sí mismo y entra en la orientación correcta. Hay dos formas de introducir sitios: 1) agregar el sitio en el 5 del primer o 2) agregarlo internamente introduciendo mismatch. 1) Introducir un sitio BamHI (GGATCC) en el 5 del primer Secuencia del gen: 5 CTTCTGCACACAACTGATGTTCACTAGCAACCTC 3 Primer directo o forward: 5 GCGGATCCCTTCTGCACACAACTG 3 24 nt T m 1= 48= Ta1= 44 T m 2= 76 Ta2=72 T m 1= sólo con la parte que hibrida inicialmente; T m 2= con el oligo completo Anillado de la PCR: primeros 5-10 ciclos a 44º, siguientes a 72º 2) Interno: secuencia del gen: 5 CTTCTGCACACAACTGATGTTCACTAGCAACCTC 3 GgA t cc Primer directo: 5 CTTCTGGATCCAACTGATG TTCAC 3 Restar 4º por cada mismatch T m = 70 Ta= 70 12= 58º Hacer la PCR completa con esta temperatura 29
30
31 Bibliografía Sambrook & Russel - Molecular Cloning - Vol. 1, 2, 3-3rd edition Chapter 8, , c2000 CSHL Press PCR: Clinical Diagnostics and Research, Rolfs, Schuller, Finckh and Weber-Rolfs, Chapter 1: PCR Principles and Reaction Components, Pag. 1-21, Springer-Verlag.
Material complementario UD 5
 Material complementario UD 5 Componentes de la PCR ADN molde que es el que se quiere replicar. Oligonucleótidos iniciadores, primers o cebadores: - Son oligonucleótidos monocatenarios cuya secuencia es
Material complementario UD 5 Componentes de la PCR ADN molde que es el que se quiere replicar. Oligonucleótidos iniciadores, primers o cebadores: - Son oligonucleótidos monocatenarios cuya secuencia es
 Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Química Biológica Patológica Técnicas de Biología Molecular aplicadas a Bioquímica Clínica Tema 2 Dra. Silvia Varas qbpatologica.unsl@gmail.com Tema 2 PCR Historia Bases Optimización de una reacción de
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 3 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2013 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Bioq. Mariana L. Ferramola 2014 Definición de PCR Es la amplificación enzimática de un fragmento de interés
Reacción en cadena de la
 Reacción en cadena de la polimerasa (PCR) Biología Molecular Año 2018 Elena G. Orellano orellano@gmail.com ; eorellan@fbioyf.unr.edu.ar Consulta: jueves 16 hs Contenidos a desarrollar PRINCIPIOS Y EQUIPAMIENTO:
Reacción en cadena de la polimerasa (PCR) Biología Molecular Año 2018 Elena G. Orellano orellano@gmail.com ; eorellan@fbioyf.unr.edu.ar Consulta: jueves 16 hs Contenidos a desarrollar PRINCIPIOS Y EQUIPAMIENTO:
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Bases Moleculares. Efrén Santos
 Bases Moleculares Efrén Santos ADN, ARN, proteínas ADN, contiene la información genética ARN, muy similar al ADN. (1) Copia temporal del ADN (2) Parte funcional y estructural del aparato traductor Proteinas,
Bases Moleculares Efrén Santos ADN, ARN, proteínas ADN, contiene la información genética ARN, muy similar al ADN. (1) Copia temporal del ADN (2) Parte funcional y estructural del aparato traductor Proteinas,
Caracteristicas del ADN. Se rompen con las siguientes condiciones -Temperaturas >90 - ph >10.5
 PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
PCR Caracteristicas del ADN Se rompen con las siguientes condiciones -Temperaturas >90 C - ph >10.5 Baja astringencia tiene uniones parciales o imperfectas. Renaturalización Dependiendo de: -Contenido
3/22/2010. Iván Ferrer Rodríguez, PhD Catedrático. En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR.
 Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS
 TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
TEMA 14 NUEVAS TECNOLOGÍAS PARA EL ESTUDIO DE LAS ENFERMEDADES INFECCIOSAS: MÉTODOS GENÉTICOS Tema 14. Nuevas tecnologías para el estudio de enfermedades infecciosas: métodos genéticos Métodos genéticos
Fundamento de la Reacción en Cadena de la Polimerasa (PCR)
 Fundamento de la Reacción en Cadena de la Polimerasa (PCR) Eva Mas, Julio Poza, Jesús Ciriza, Pilar Zaragoza, Rosario Osta y Clementina Rodellar. Laboratorio de Genética, Bioquímica y Grupos Sanguíneos.
Fundamento de la Reacción en Cadena de la Polimerasa (PCR) Eva Mas, Julio Poza, Jesús Ciriza, Pilar Zaragoza, Rosario Osta y Clementina Rodellar. Laboratorio de Genética, Bioquímica y Grupos Sanguíneos.
PCR. Dr. Francisco Pérez Bravo Laboratorio de Genómica Nutricional Departamento de Nutrición Facultad de Medicina Universidad de Chile
 FUNDAMENTOS BASICOS EN PCR Dr. Francisco Pérez Bravo Laboratorio de Genómica Nutricional Departamento de Nutrición Facultad de Medicina Universidad de Chile Manipulación del DNA en el uso Principio diagnóstico
FUNDAMENTOS BASICOS EN PCR Dr. Francisco Pérez Bravo Laboratorio de Genómica Nutricional Departamento de Nutrición Facultad de Medicina Universidad de Chile Manipulación del DNA en el uso Principio diagnóstico
PCR gen 16S ARNr bacteriano
 PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
PCR gen 16S ARNr bacteriano Ref. PCR16S 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y la práctica de la Reacción en Cadena de la Polimerasa
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA"
 P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
PCR. Técnica inventada por Kary Mullis :1987. Premio Nobel 1993. Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN
 PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología de los Alimentos Ingeniería en alimentos 2015 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente una región
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Microbiología General Microbiología M de los Alimentos Licenciatura en Bioquímica i Ingeniería en alimentos c Microbiología r 2015 Licenciatura en Biotecnología
Seminario Genética. Técnicas de Biología Molecular
 Seminario Genética Técnicas de Biología Molecular Contenidos Extracción de ADN Enzimas de restricción PCR Southern Blot Secuenciación método de Sanger y automática 1º Extracción de ADN Se pueden tomar
Seminario Genética Técnicas de Biología Molecular Contenidos Extracción de ADN Enzimas de restricción PCR Southern Blot Secuenciación método de Sanger y automática 1º Extracción de ADN Se pueden tomar
Repaso de PCR y qpcr
 Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
Repaso de PCR y qpcr 1. Los siguientes oligonucleótidos se utilizaron para amplificar una región específica de DNA o RNA, el diseño es variable de acuerdo a el propósito de los oligonucleótidos, a continuación
REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
REACCIÓN EN CADENA DE LA POLIMERASA (PCR) Lic. en Bioquímica Lic. en Biotecnología Microbiología de los Alimentos 2016 Desarrollada por Kary Mullis en 1986 Técnica in vitro que permite amplificar enzimáticamente
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Conceptos
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987)
 EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
EXTRACCIÓN DE ADN (Doyle & Doyle, 1987) MATERIALES Y REACTIVOS (Grado biología molecular a analítico) Cloroformo Alcohol Isoamilico Isopropanol. Etanol al 70%. Bloques de Hielo Microcentrifuga Bloque de
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80
 AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
AMPLIFICACIÓN Y DETECCIÓN POR PCR DEL LOCUS HUMANO D1S80 Ref. PCR1 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción
HIBRIDACIÓN DE ÁCIDOS NUCLEICOS: SOUTHERN BLOT
 HIBRIDACIÓN DE ÁCIDOS NUCLEICOS: SOUTHERN BLOT HIBRIDACION DE ACIDOS NUCLEICOS APAREAMIENTO COMPLEMENTARIO DE BASES ENTRE DOS ACIDOS NUCLEICOS DE HEBRA SIMPLE PRODUCTO DE HEBRA DOBLE. DNA/DNA RNA/RNA DNA/RNA
HIBRIDACIÓN DE ÁCIDOS NUCLEICOS: SOUTHERN BLOT HIBRIDACION DE ACIDOS NUCLEICOS APAREAMIENTO COMPLEMENTARIO DE BASES ENTRE DOS ACIDOS NUCLEICOS DE HEBRA SIMPLE PRODUCTO DE HEBRA DOBLE. DNA/DNA RNA/RNA DNA/RNA
Polymerase Chain Reaction (PCR)
 Polymerase Chain Reaction (PCR) Un poco de historia La técnica de PCR fue inventada por Kary B. Mullis en 1983. La primer publicación sobre PCR apareció en 1985, aunque el principio básico de replicar
Polymerase Chain Reaction (PCR) Un poco de historia La técnica de PCR fue inventada por Kary B. Mullis en 1983. La primer publicación sobre PCR apareció en 1985, aunque el principio básico de replicar
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón
 RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
Métodos de secuenciación de ADN
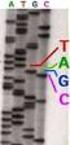 Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
Métodos de secuenciación de ADN Valeria Tekiel Breve historia. 1871 Descubrimiento de los ácidos nucleicos 1903 Los cromosomas son unidades hereditarias 1910 Los genes están en los cromosomas 1944 El ADN
BIOLOGIA MOLECULAR. Dra. Ma. Laura Tondo
 TÉCNICAS BASADAS EN HIBRIDACIÓN MOLECULAR BIOLOGIA MOLECULAR 2016 Dra. Ma. Laura Tondo DESNATURALIZACION Y RENATURALIZACION DEL DNA Cómo se mantienen unidas las dos hebras del DNA? - Puentes de hidrógeno
TÉCNICAS BASADAS EN HIBRIDACIÓN MOLECULAR BIOLOGIA MOLECULAR 2016 Dra. Ma. Laura Tondo DESNATURALIZACION Y RENATURALIZACION DEL DNA Cómo se mantienen unidas las dos hebras del DNA? - Puentes de hidrógeno
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR
 ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
ESTUDIO DE POLIMORFISMOS ALU HUMANOS POR PCR Ref. PCRALU 1. OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena
Otras enzimas usadas en la manipulación de ácidos nucleicos DNA polimerasas
 Otras enzimas usadas en la manipulación de ácidos nucleicos DNA polimerasas -dependientes de DNA -dependientes de RNA -independientes del molde RNA polimerasas -dependientes de DNA -independientes de DNA
Otras enzimas usadas en la manipulación de ácidos nucleicos DNA polimerasas -dependientes de DNA -dependientes de RNA -independientes del molde RNA polimerasas -dependientes de DNA -independientes de DNA
Secuenciar una molécula de ADN consiste en determinar en qué orden se disponen los cuatro nucleótidos (A, T, C y G) que componen la molécula.
 SECUENCIACIÓN DEL ADN Secuenciar una molécula de ADN consiste en determinar en qué orden se disponen los cuatro nucleótidos (A, T, C y G) que componen la molécula. El primer método diseñado para secuenciar
SECUENCIACIÓN DEL ADN Secuenciar una molécula de ADN consiste en determinar en qué orden se disponen los cuatro nucleótidos (A, T, C y G) que componen la molécula. El primer método diseñado para secuenciar
BIOLOGIA CELULAR Y MOLECULAR. LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR)
 BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR) 1. INTRODUCCIÓN La Reacción en Cadena de la Polimerasa (PCR) es una técnica in Vitro que imita la habilidad natural
BIOLOGIA CELULAR Y MOLECULAR LABORATORIO No 7: REACCION EN CADENA DE LA POLIMERASA (PCR) 1. INTRODUCCIÓN La Reacción en Cadena de la Polimerasa (PCR) es una técnica in Vitro que imita la habilidad natural
Purinas (R); Pirimidinas (Y)
 Replicación del ADN La doble hélice Purinas (R); Pirimidinas (Y) Tautómeros Puentes de H normales a) Una vez que la replicación comienza, todo el ADN es replicado. b) Como regla general, cuando la replicación
Replicación del ADN La doble hélice Purinas (R); Pirimidinas (Y) Tautómeros Puentes de H normales a) Una vez que la replicación comienza, todo el ADN es replicado. b) Como regla general, cuando la replicación
Protocolos de preparación de muestras
 Protocolos de preparación de muestras Contenido 1. TIPOS DE MUESTRA 2. PROTOCOLO DE PREPARACIÓN DE MUESTRA 2.1 Producto de PCR crudo (sin purificar). 2.2 Producto de PCR crudo en gel (sin purificar). 2.3
Protocolos de preparación de muestras Contenido 1. TIPOS DE MUESTRA 2. PROTOCOLO DE PREPARACIÓN DE MUESTRA 2.1 Producto de PCR crudo (sin purificar). 2.2 Producto de PCR crudo en gel (sin purificar). 2.3
Técnicas moleculares aplicadas en Bioquímica Clínica-2017 Dra. María Gabriela Lacoste
 Técnicas moleculares aplicadas en Bioquímica Clínica-2017 Dra. María Gabriela Lacoste TIPOS DE PCR SEGÚN LA MUTACION A DETECTAR GRANDES DELECIONES MUTACIONES PUNTUALES Y DELECIONES PEQUEÑAS PCR Multiplex
Técnicas moleculares aplicadas en Bioquímica Clínica-2017 Dra. María Gabriela Lacoste TIPOS DE PCR SEGÚN LA MUTACION A DETECTAR GRANDES DELECIONES MUTACIONES PUNTUALES Y DELECIONES PEQUEÑAS PCR Multiplex
11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS. Actualmente existen diversos métodos en el mercado que pueden utilizarse
 11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS Actualmente existen diversos métodos en el mercado que pueden utilizarse para el diagnóstico de rotavirus, las cuales pueden realizarse directamente a partir
11. MÉTODOS PARA LA DETECCIÓN DEL ROTAVIRUS Actualmente existen diversos métodos en el mercado que pueden utilizarse para el diagnóstico de rotavirus, las cuales pueden realizarse directamente a partir
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ
 REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
REGULACIÓN DE LA EXPRESIÓN GÉNICA EN PROCARIOTAS ANÁLISIS DE LA EXPRESIÓN DE LACZ Para conocer la organización del ADN se desarrollaron métodos que permitieron conocer la secuencia exacta de nucleótidos,
PCR SIMULADA. Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO
 Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCR Simulada (4 prácticas) 1. OBJETIVO DEL EXPERIMENTO PCR SIMULADA El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
TRABAJO PRÁCTICO Nº 4 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) INTRODUCCIÓN La reacción en cadena de la polimerasa o PCR (del inglés Polymerase Chain Reaction) es una técnica relativamente simple y poderosa
Cuál de estas 3 posibilidades de replicación es la que ocurre?
 Cuál de estas 3 posibilidades de replicación es la que ocurre? Marcaje del DNA bacteriano con N 15 Experimento de Meselson-Stahl (1958) 1. Bacteria crecida en N 14 2. Bacteria crecida en N 15 3. Incubación
Cuál de estas 3 posibilidades de replicación es la que ocurre? Marcaje del DNA bacteriano con N 15 Experimento de Meselson-Stahl (1958) 1. Bacteria crecida en N 14 2. Bacteria crecida en N 15 3. Incubación
Técnicas del ADN recombinante Introducción
 Técnicas del ADN recombinante Introducción Estructura primaria del ADN Enzimas de restricción (ER) Son enzimas (endonucleasas) de origen bacteriano que reconocen y cortan secuencias específicas (4-8 pb)
Técnicas del ADN recombinante Introducción Estructura primaria del ADN Enzimas de restricción (ER) Son enzimas (endonucleasas) de origen bacteriano que reconocen y cortan secuencias específicas (4-8 pb)
Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias
 Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola 2016 Dogma central de la biología
Explicación de TP Nº 1 Técnicas de biología molecular utilizadas en el diagnóstico de enfermedades hereditarias Química Biológica Patológica Dra. Mariana L. Ferramola 2016 Dogma central de la biología
Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática.
 Departamento de Bioquímica Médica y Biología Molecular Práctica 3 Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática. Curso 1 Medicina (Abril) 2012 BIOQUÍMICA
Departamento de Bioquímica Médica y Biología Molecular Práctica 3 Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática. Curso 1 Medicina (Abril) 2012 BIOQUÍMICA
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular
 Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Unidad9. Principios de Ingeniería Genética Aplicaciones de Biología Molecular Aislamiento, análisis y manipulación de ácidos nucleicos Generación de moléculas de DNA recombinante Ingeniería Genética AISLAMIENTO
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Cuál de estas 3 posibilidades de replicación es la que ocurre?
 Cuál de estas 3 posibilidades de replicación es la que ocurre? Marcaje del DNA bacteriano con N 15 Experimento de Meselson-Stahl (1958) 1. Bacteria crecida en N 14 2. Bacteria crecida en N 15 3. Incubación
Cuál de estas 3 posibilidades de replicación es la que ocurre? Marcaje del DNA bacteriano con N 15 Experimento de Meselson-Stahl (1958) 1. Bacteria crecida en N 14 2. Bacteria crecida en N 15 3. Incubación
TECNICAS DE BIOLOGIA MOLECULAR
 Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR IV- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com TIPO DE MUTACION DEFINE
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento
 Dra. Gabriela Lacoste- QBP 2012 MLPA Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento Es una técnica que permite la detección de cambios en el
Dra. Gabriela Lacoste- QBP 2012 MLPA Multiplex Ligation-dependent Probe Amplification Amplificación Múltiple de Sondas dependientes de Ligamiento Es una técnica que permite la detección de cambios en el
Bases moleculares de la herencia
 Bases moleculares de la herencia i. Qué moléculas son las portadoras de la información hereditaria? ii. Cómo se preserva y trasmite la información hereditaria? iii. Cómo se expresa esta información? Dogma
Bases moleculares de la herencia i. Qué moléculas son las portadoras de la información hereditaria? ii. Cómo se preserva y trasmite la información hereditaria? iii. Cómo se expresa esta información? Dogma
Laboratorio de Ecología de Enfermedades Facultad de Ciencias Veterinarias Universidad Nacional del Litoral Santa Fe, Argentina. PCR en Tiempo Real
 Laboratorio de Ecología de Enfermedades Facultad de Ciencias Veterinarias Universidad Nacional del Litoral Santa Fe, Argentina PCR en Tiempo Real Dr. Lucas Monje Consejo Nacional de Investigaciones Científicas
Laboratorio de Ecología de Enfermedades Facultad de Ciencias Veterinarias Universidad Nacional del Litoral Santa Fe, Argentina PCR en Tiempo Real Dr. Lucas Monje Consejo Nacional de Investigaciones Científicas
PCR Múltiplex. Listeria monocytogenes
 PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
PCR Múltiplex Listeria monocytogenes PCR-múltiplex En una sola reacción de PCR se amplifican al mismo tiempo 2 o más genes Condiciones similares de PCR para los genes amplificar: Desnaturalización Alineamiento
Código: KIT-001 Ver: 1. Pre-FRAXA. Sistema para la detección del número de repetidos CGG en el gene FMR1 PM F M M F M M M C. Reg.
 Pre-FRAXA Sistema para la detección del número de repetidos CGG en el gene FMR1 PM F M M F M M M C Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. www.atgen.com.uy
Pre-FRAXA Sistema para la detección del número de repetidos CGG en el gene FMR1 PM F M M F M M M C Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. www.atgen.com.uy
Polymerase Chain Reaction (PCR)
 Polymerase Chain Reaction (PCR) Un poco de historia La técnica de PCR fue inventada por Kary B. Mullis en 1983. La primer publicación sobre PCR apareció en 1985, aunque el principio básico de replicar
Polymerase Chain Reaction (PCR) Un poco de historia La técnica de PCR fue inventada por Kary B. Mullis en 1983. La primer publicación sobre PCR apareció en 1985, aunque el principio básico de replicar
UNIVERSIDAD NACIONAL DE MISIONES Facultad de Ciencias Exactas Químicas y Naturales COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN
 COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN OBJETIVO: el presente trabajo práctico tiene como objetivo principal que el alumno adquiera destreza en el análisis de electroferogramas generados por equipos
COLOQUIO N 6/15 FUNDAMENTOS DE SECUENCIACIÓN OBJETIVO: el presente trabajo práctico tiene como objetivo principal que el alumno adquiera destreza en el análisis de electroferogramas generados por equipos
SEGUNDA SESIÓN: AMPLIFICACIÓN DE LOS GENES inla E inlc DE Listeria monocytogenes POR PCR-MÚLTIPLE.
 SEGUNDA SESIÓN: AMPLIFICACIÓN DE LOS GENES inla E inlc DE Listeria monocytogenes POR PCR-MÚLTIPLE. La reacción en cadena de la polimerasa (PCR por sus siglas en inglés), es un procedimiento rápido para
SEGUNDA SESIÓN: AMPLIFICACIÓN DE LOS GENES inla E inlc DE Listeria monocytogenes POR PCR-MÚLTIPLE. La reacción en cadena de la polimerasa (PCR por sus siglas en inglés), es un procedimiento rápido para
OBJETIVOS. Comprender la forma a través de la cual nuestro material genético se mantiene en el tiempo. Analizar el trayecto de la información genética
 REPLICACIÓN NM4 OBJETIVOS Comprender la forma a través de la cual nuestro material genético se mantiene en el tiempo. Analizar el trayecto de la información genética CONOCIMIENTOS PREVIOS NECESARIOS COMPOSICIÓN
REPLICACIÓN NM4 OBJETIVOS Comprender la forma a través de la cual nuestro material genético se mantiene en el tiempo. Analizar el trayecto de la información genética CONOCIMIENTOS PREVIOS NECESARIOS COMPOSICIÓN
Acción de la enzima de restricción EcoRI
 Acción de la enzima de restricción EcoRI S Lobos C - U de Chile 1 S Lobos C - U de Chile 2 S Lobos C - U de Chile 3 Nucleic Acids Res. 2001 September 15; 29(18): 3705 3727. Structure and function of type
Acción de la enzima de restricción EcoRI S Lobos C - U de Chile 1 S Lobos C - U de Chile 2 S Lobos C - U de Chile 3 Nucleic Acids Res. 2001 September 15; 29(18): 3705 3727. Structure and function of type
Métodos de Biología Molecular y de Ingeniería Genética
 Métodos de Biología Molecular y de Ingeniería Genética I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II.
Métodos de Biología Molecular y de Ingeniería Genética I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II.
APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS
 Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Reacción en cadena de la polimerasa (Polymerase Chain Reaction, PCR)
 Reacción en cadena de la polimerasa (Polymerase hain Reaction, PR) Apellidos, nombre Pérez de astro, Ana María Departamento entro Biotecnología ETSIAMN 1 Resumen La reacción en cadena de la polimerasa
Reacción en cadena de la polimerasa (Polymerase hain Reaction, PR) Apellidos, nombre Pérez de astro, Ana María Departamento entro Biotecnología ETSIAMN 1 Resumen La reacción en cadena de la polimerasa
Enzimas utilizadas en Biología molecular
 s utilizadas en Biología molecular Nucleasas DNAsas Endonucleasas inespecíficas. DNAsa I (páncreas bovino) Molde Degrada DNA dc, sc. A bajas concentraciones genera nicks Sondas, Nick translation Dnasa
s utilizadas en Biología molecular Nucleasas DNAsas Endonucleasas inespecíficas. DNAsa I (páncreas bovino) Molde Degrada DNA dc, sc. A bajas concentraciones genera nicks Sondas, Nick translation Dnasa
BIOTECNOLOGÍA MOLECULAR UNIDAD N 2: Marcadores Moleculares de ADN
 BIOTECNOLOGÍA MOLECULAR UNIDAD N 2: Marcadores Moleculares de ADN Lic. Gustavo Bich Dra. María Mercedes Tiscornia. Cátedra de Biotecnología Molecular. 2017. INSTITUTO DE BIOTECNOLOGÍA MISIONES MARÍA EBE
BIOTECNOLOGÍA MOLECULAR UNIDAD N 2: Marcadores Moleculares de ADN Lic. Gustavo Bich Dra. María Mercedes Tiscornia. Cátedra de Biotecnología Molecular. 2017. INSTITUTO DE BIOTECNOLOGÍA MISIONES MARÍA EBE
3. Materiales y Métodos
 3. Materiales y Métodos El análisis de haplotipos se realizó mediante la caracterización de variaciones alélicas reportadas con anterioridad. En total de analizaron cinco marcadores polimórficos: tres
3. Materiales y Métodos El análisis de haplotipos se realizó mediante la caracterización de variaciones alélicas reportadas con anterioridad. En total de analizaron cinco marcadores polimórficos: tres
Electroforesis de DNA
 Electroforesis de DNA DNA Topoisomerasas Izquierda Derecha Enzimas de restricción Clasificadas en 3 tipos, donde la mayoría pertenece al grupo II (proteínas monoméricas, poseen simetria rotacional y generalmente
Electroforesis de DNA DNA Topoisomerasas Izquierda Derecha Enzimas de restricción Clasificadas en 3 tipos, donde la mayoría pertenece al grupo II (proteínas monoméricas, poseen simetria rotacional y generalmente
DUPLICACION DEL ADN. Dra Carmen Aída Martínez
 DUPLICACION DEL ADN Dra Carmen Aída Martínez Definción El proceso de replicación de ADN es el mecanismo que permite al ADN duplicarse (es decir, sintetizar una copia idéntica) Replicación La reproducción
DUPLICACION DEL ADN Dra Carmen Aída Martínez Definción El proceso de replicación de ADN es el mecanismo que permite al ADN duplicarse (es decir, sintetizar una copia idéntica) Replicación La reproducción
PCR. Qué es la PCR? T a de fusión de oligonucleótidos (t m )
 % a de fusión de oligonucleótidos (t m ) Qué es la PCR? La reacción en cadena de la polimerasa (PCR: polymerase chain reaction) es una técnica de amplificación de secuencias de DNA in vitro t m = 2 (A
% a de fusión de oligonucleótidos (t m ) Qué es la PCR? La reacción en cadena de la polimerasa (PCR: polymerase chain reaction) es una técnica de amplificación de secuencias de DNA in vitro t m = 2 (A
A. Anexo: Extracción de dsrna por columnas de exclusión de Sephadex G-50
 A. Anexo: Extracción de dsrna por columnas de exclusión de Sephadex G-50 (Tomado de Guzmán, 1994; Rodríguez, 2006) 1. En un tubo eppendorf tapa rosca estéril colocar 100 mg de material vegetal pulverizado
A. Anexo: Extracción de dsrna por columnas de exclusión de Sephadex G-50 (Tomado de Guzmán, 1994; Rodríguez, 2006) 1. En un tubo eppendorf tapa rosca estéril colocar 100 mg de material vegetal pulverizado
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP
 SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
SOLICITUD DEL SERVICIO DE SECUENCIACION CENTRO DE DETECCION BIOMOLECULAR EN ENFERMEDADES EMERGENTES/BUAP Usuario: Institución: Fecha: Departamento: e-mail: Teléfono: Ext.: Investigador Principal: e-mail:
41. Comprobación de colonias transformantes mediante PCR y electroforesis
 41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
41. Comprobación de colonias transformantes mediante PCR y electroforesis Aurora Galván Cejudo, Manuel Tejada, Antonio Camargo, José Javier Higuera, Vicente Mariscal, Emilio Fernández Reyes Departamento
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
CUESTIONES. 1. Por qué la PCR convencional no es cuantitativa?
 2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
REPLICACIÓN. La importancia de la replicación del ADN en la medicina. El experimento de Meselson y Stahl. Tres posibles modelos de replicación
 REPLICACIÓN La importancia de la del ADN en la medicina Propiedades generales del proceso de Biosíntesis del ADN El mecanismo de la La maquinaria de la La Replicación en Eucariotas Telómeros y Telomerasas
REPLICACIÓN La importancia de la del ADN en la medicina Propiedades generales del proceso de Biosíntesis del ADN El mecanismo de la La maquinaria de la La Replicación en Eucariotas Telómeros y Telomerasas
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Definición de PCR Es la amplificación enzimática de un
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Definición de PCR Es la amplificación enzimática de un
BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90. País: ESPAÑA
 XII OLIMPIADA IBEROAMERICANA DE BIOLOGÍA SETIEMBRE 2018 LOJA-ECUADOR EXAMEN PRÁCTICO BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90 TIEMPO PERMITIDO: 120 minutos País: ESPAÑA ESCRIBA EL CÓDIGO EN EL SIGUIENTE
XII OLIMPIADA IBEROAMERICANA DE BIOLOGÍA SETIEMBRE 2018 LOJA-ECUADOR EXAMEN PRÁCTICO BIOLOGÍA MOLECULAR TOTAL DE PUNTOS: 90 TIEMPO PERMITIDO: 120 minutos País: ESPAÑA ESCRIBA EL CÓDIGO EN EL SIGUIENTE
Módulo III: Vías de la información genética
 Módulo III: Vías de la información genética Bibliografía: Mathews, Van Holde, Ahern, Bioquimica, Lenhinger Voet &Voet Mónica Marín Prof. de Bioquímica, FC Este material no tiene fines comerciales Tiene
Módulo III: Vías de la información genética Bibliografía: Mathews, Van Holde, Ahern, Bioquimica, Lenhinger Voet &Voet Mónica Marín Prof. de Bioquímica, FC Este material no tiene fines comerciales Tiene
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
CLONAR-CLONADO-CLONACIÓN
 CLONAR-CLONADO-CLONACIÓN Se refiere a los procesos (conjunto de métodos) empleados para crear copias idénticas de fragmentos de ADN, copias idénticas de células o copias idénticas de organismos Se pueden
CLONAR-CLONADO-CLONACIÓN Se refiere a los procesos (conjunto de métodos) empleados para crear copias idénticas de fragmentos de ADN, copias idénticas de células o copias idénticas de organismos Se pueden
Test Genéticos Moleculares
 CERPO Centro de Referencia Perinatal Oriente Facultad de Medicina, Universidad de Chile Test Genéticos Moleculares Dr. Guillermo Parrao Barrera Residente Medicina Materno Fetal Pontificia Universidad Católica
CERPO Centro de Referencia Perinatal Oriente Facultad de Medicina, Universidad de Chile Test Genéticos Moleculares Dr. Guillermo Parrao Barrera Residente Medicina Materno Fetal Pontificia Universidad Católica
PROPIEDADES DE LOS ACIDOS NUCLEICOS Y TECNICAS DE DETECCION
 PROPIEDADES DE LOS ACIDOS NUCLEICOS Y TECNICAS DE DETECCION 1. RNA NORTHERN BLOT CONVENCIONAL NORTHERN BLOT REVERSO (MICROARRAY) HIBRIDACION IN SITU 2. DNA SOUTHERN BLOT ANALISIS DE GENOTECAS Estructura
PROPIEDADES DE LOS ACIDOS NUCLEICOS Y TECNICAS DE DETECCION 1. RNA NORTHERN BLOT CONVENCIONAL NORTHERN BLOT REVERSO (MICROARRAY) HIBRIDACION IN SITU 2. DNA SOUTHERN BLOT ANALISIS DE GENOTECAS Estructura
Técnicas moleculares.
 Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
UNIVERSIDAD NACIONAL DE GRAL. SAN MARTÍN ECyT BIOLOGÍA CPU. Biología. Información genética, ADN, Cromosomas
 UNIVERSIDAD NACIONAL DE GRAL. SAN MARTÍN ECyT BIOLOGÍA CPU Biología Información genética, ADN, Cromosomas Qué es la información genética? Es la información biológica heredable: Toda la información que
UNIVERSIDAD NACIONAL DE GRAL. SAN MARTÍN ECyT BIOLOGÍA CPU Biología Información genética, ADN, Cromosomas Qué es la información genética? Es la información biológica heredable: Toda la información que
Técnicas de Ingeniería Genética
 Técnicas de Ingeniería Genética REACCIÓN EN CADENA DE LA POLIMERASA: PCR Kary Mullis de Cetus Corporation.1985 Video PCR Tapa que calienta Termociclador Tubos de pared delgada Taq polimerasa Taq polimerasa:
Técnicas de Ingeniería Genética REACCIÓN EN CADENA DE LA POLIMERASA: PCR Kary Mullis de Cetus Corporation.1985 Video PCR Tapa que calienta Termociclador Tubos de pared delgada Taq polimerasa Taq polimerasa:
TECNICAS DE BIOLOGIA MOLECULAR
 Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR III- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com GENERALIDADES Electroforesis
Química Biológica Patológica TECNICAS DE BIOLOGIA MOLECULAR III- Parte Aplicadas al diagnóstico de Enfermedades Genéticas Tema:1 (1) Dra. Silvia Varas qbpatologica.unsl@gmail.com GENERALIDADES Electroforesis
Seminario de Problemas
 Seminario de Problemas 1. Qué uniones relacionan: a) dos deoxirribosas entre si; b) 2 bases enfrentadas? 2. Un segmento de ADN de cadena simple tiene la siguiente composición de bases: A 31,5 % C 23,7
Seminario de Problemas 1. Qué uniones relacionan: a) dos deoxirribosas entre si; b) 2 bases enfrentadas? 2. Un segmento de ADN de cadena simple tiene la siguiente composición de bases: A 31,5 % C 23,7
Criterios para diseñar primers. Iván Ferrer Rodríguez, Ph.D. Catedrático
 Criterios para diseñar primers Iván Ferrer Rodríguez, Ph.D. Catedrático 1 Qué es un primer? Es una cadena corta de nucleótidos, un oligonucleótido. Sirve como punto de partida para la replicación del DNA.
Criterios para diseñar primers Iván Ferrer Rodríguez, Ph.D. Catedrático 1 Qué es un primer? Es una cadena corta de nucleótidos, un oligonucleótido. Sirve como punto de partida para la replicación del DNA.
ADN y RNA estructura - estabilidad
 ADN y RNA estructura - estabilidad Objetivos propiedades físicas, químicas y biológicas(función) del ADN plasmídico y genómico Interacción con el entorno y otras moléculas Aplicación de los conceptos integrados:
ADN y RNA estructura - estabilidad Objetivos propiedades físicas, químicas y biológicas(función) del ADN plasmídico y genómico Interacción con el entorno y otras moléculas Aplicación de los conceptos integrados:
ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS. Aislamiento de plásmido. M. en C. J. Abraham León Domínguez
 ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS Aislamiento de plásmido M. en C. J. Abraham León Domínguez La transformación bacteriana es un proceso muy útil en biología molecular. Conjugación Transformación
ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS Aislamiento de plásmido M. en C. J. Abraham León Domínguez La transformación bacteriana es un proceso muy útil en biología molecular. Conjugación Transformación
ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS. Aislamiento de plásmido
 ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS Aislamiento de plásmido Transferencia de material genético intercelular Transformación Conjugación Transformación Transducción Existe algún mecanismo
ESTRUCTURA, PROPIEDADES Y FUNCIÓN DE ÁCIDOS NUCLÉICOS Aislamiento de plásmido Transferencia de material genético intercelular Transformación Conjugación Transformación Transducción Existe algún mecanismo
PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS. Es un ácido capaz de formar sales con iones cargados
 Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
Extracción de ADN Genética molecular PROPIEDADES FISICOQUÍMICAS DE LOS ÁCIDOS NUCLEICOS Es un ácido capaz de formar sales con iones cargados positivamente (cationes). Es soluble en soluciones concentradas
NTC = NO TEMPLATE CONTROL
 NTC = NO TEMPLATE CONTROL Químicos usados en la qpcr para detectar los productos: específicos o inespecíficos Inespecíficos: agentes intercalantes que se unen al DNA doble hebra y detectan cualquier producto.
NTC = NO TEMPLATE CONTROL Químicos usados en la qpcr para detectar los productos: específicos o inespecíficos Inespecíficos: agentes intercalantes que se unen al DNA doble hebra y detectan cualquier producto.
Materia de Articulación CEBI_A5
 CARRERA DE ESPECIALIZACION EN BIOTECNOLOGIA INDUSTRIAL FCEyN-INTI Materia de Articulación CEBI_A5 Biología Molecular l Replicación ió del ADN La división celular requiere la duplicación del material genético
CARRERA DE ESPECIALIZACION EN BIOTECNOLOGIA INDUSTRIAL FCEyN-INTI Materia de Articulación CEBI_A5 Biología Molecular l Replicación ió del ADN La división celular requiere la duplicación del material genético
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO
 SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
SSI DERMATOPHYTE PCR KIT PRUEBA PCR DERMATOFITO 2 Prueba de PCR para la detección de dermatofitos y Trichophyton rubrum Para uso diagnóstico in vitro Aplicación La prueba de PCR para dermatofitos en uñas
INGENIERÍA GENÉTICA. Sinónimos: Manipulación genética Clonaje génico Tecnología del DNA recombinante
 INGENIERÍA GENÉTICA Es una revolución metodológica que ha puesto a nuestra disposición gran número de técnicas procedentes de diversos campos de la ciencia con un objetivo común muy importante: el acceso
INGENIERÍA GENÉTICA Es una revolución metodológica que ha puesto a nuestra disposición gran número de técnicas procedentes de diversos campos de la ciencia con un objetivo común muy importante: el acceso
