10. MATERIALES Y MÉTODOS
|
|
|
- María Dolores Parra Ponce
- hace 8 años
- Vistas:
Transcripción
1 10. MATERIALES Y MÉTODOS A continuación se presenta un esquema general de la estrategia experimental del presente trabajo. Esquema de trabajo Modelo animal (Ratas hembras) Disección de los tejidos (TAP y ME) Extracción del ARN total (Trizol ) Frotis (TAP) Cuanficación e integridad del ARN total indirecta (A / gel agarosa 1.0 %) (sspn) Inmunofluorescencia Observación al microscopio Síntesis de ADNc (RT-PCR) de fluorescencia (20X / 40X) Diseño de oligonucleótidos Amplificación del ADNc (PCR) (α, β, γ, δ, ε, ζ SGs y sspn) Secuenciación de los productos amplificados (sgcz y sspn) TAP, Tejido adiposo parametrial; ME, Músculo esquelético.
2 10.1 Modelo animal. Se utilizaron 6 ratas hembras de 14 semanas de edad (adultas) de la cepa Sprague- Dawley. A partir del destéte y hasta el día del experimento se alimentaron a libre demanda con una dieta comercial (Tabla 2). Los animales se mantuvieron bajo ciclos de luz-oscuridad de 12 hrs. Tabla 2. Composición de la dieta comercial. Componente Porcentaje total Hidratos de carbono 67.5% Proteínas 22.5% Lípidos 5.0% Minerales 4.0% Vitaminas 1.0% Formulab Diet Disección de los tejidos. A las 14 semanas de edad, las ratas fueron anestesiadas con pentobarbital sódico (35 mg/kg de peso corporal) vía peritoneal, esto se realizó con la finalidad de evitar el sufrimiento innecesario del animal. Posteriormente, se disecó el tejido adiposo parametrial (TAP) y el músculo esquelético (ME; quadriceps femoris) de cada rata. El tejido adiposo parametrial se separó de los oviductos y de los ovarios. Una vez obtenidos, los tejidos se lavaron con solución salina fisiológica (NaCl 0.9%) fría. Con material de disección estéril se cortó cada uno de los tejidos en pequeños pedazos los cuales se colocaron en tubos de 1.5 ml estériles y se congelaron en nitrógeno líquido. Las muestras se almacenaron a 70 C hasta realizar los ensayos posteriores. Este procedimiento requiere de destreza manual ya que es necesario que se realice en el menor tiempo posible para evitar la degradación del ARN Extracción del ARN total.
3 El ARN total fue aislado a partir de los adipocitos y de los miocitos por medio del método de extracción fenólica con TRIZOL (Reagent Invitrogen TM Life Technologies) de Chomczynsky (Chomczynsky y Sacchi, 1987). Este método se basa en el uso de una solución monofásica de fenol e isotiocianato de guanidina para la lisis de las células y la separación de la muestra en dos fases (acuosa y orgánica). Seguida de la extracción y precipitación del ARN total con cloroformo e isopropanol respectivamente a partir de la fase acuosa. Cada tejido se molió en un mortero agregando nitrógeno líquido hasta obtener un polvo fino. De este polvo se pesaron de 0.10 a 0.15 g en un tubo de 1.5 ml, y se adicionó 1.0 ml de TRIZOL (fenol-sales de guanidina), el tejido se homogenizó con un homogenizador (Polytron PT 1200, Kinematica AG). Las aspas del polytron fueron previamente lavadas con NaOH 1.0 N en agua DEPC (dietilpirocarbonato) 1.0%, y por último con agua DEPC al 1.0%. El tejido homogenizado se centrifugó a 10,000 rpm por 10 min a 4 C; en este paso se eliminan los restos celulares, si la muestra es tejido adiposo se observa un anillo de grasa en la parte superior. El sobrenadante se transfirió a un tubo eppendorf y se adicionaron 200 µl de cloroformo frío, se agitó vigorosamente durante 15 seg. y se incubó en hielo por 3 minutos. Posteriormente la muestra se centrifugó a 10,000 rpm por 10 min a 4 C; en este paso se forman dos fases por diferencia de densidad; la fase fenólica orgánica, contiene al ADN y restos de proteínas desnaturalizadas y la fase superior acuosa contiene el ARN en cloroformo. Esta fase se transfirió a un tubo de 1.5 mly se adicionaron 500 µl de isopropanol el cual precipita al ARN. La pastilla se lavó con etanol al 75%, y se centrífugo por 5 minutos a 7,500 rpm a 4 C (2 veces). En el último lavado, el etanol se eliminó y la pastilla se secó por aproximadamente 5 minutos. Al final, la pastilla de ARN se disolvió en µl de agua DEPC estéril dependiendo de su tamaño, se agitó en vortex y se tomó una alícuota la cual se almacenó a 20ºC para más adelante cuantificar y determinar la integridad del RNA total. Al resto se le agregó 1/10 del volumen total de acetato de potasio 2.0 M y 2 volúmenes de etanol absoluto y se almacenó a -70 C para los subsecuentes ensayos (RT-PCR s) Determinación de la concentración, pureza e integridad del ARN total.
4 Para conocer la concentración del ARN total se preparó una dilución 1:50 por duplicado con agua DEPC y se midió la absorbancia a 260 nm y a 280 nm en un espectrofotómetro (Ultrospec 2000 Pharmacia Biotech TM ) con lámpara UV. Una vez que se obtuvieron los valores de absorbancia de los ARN s, a partir de la siguiente ecuación se puede conocer su concentración: Concentración de ARN (µg / µl) = (Absorbancia a 260 nm)(dilución)(40)/1000 Sabiendo que 1 densidad óptica a 260 nm (1 OD 260 ) = 40 µg de ARN. La relación de absorbancias a 260/280 indica la pureza del ARN, se considera que las relaciones cercanas a 2.0 unidades de OD 260 son las óptimas. Para determinar la integridad del ARN total extraído, se sometió una muestra del mismo a electroforésis horizontal en gel de agarosa al 1.0% teñido con bromuro de etidio (0.5 µg/ml). La técnica de electroforésis se basa en el desplazamiento en este caso del ARN a través de una matriz porosa formada por la agarosa polimerizada, siendo el tamaño del poro determinado por la concentración de ésta en una solución de sales. Al aplicarse una corriente eléctrica a través del gel, los ácidos nucleicos que poseen una carga negativa conferida por los grupos fosfato, migran hacia el ánodo en condiciones de ph neutro (Maniatis et al. 1989). Se mezclaron 5.0 µl de la muestra de ARN con 2.0 µl de buffer TBE 1X y 2.0 µl de colorante azul de bromofenol (0.25%), el cual contiene glicerol al 30%. La electroforesis se llevo a cabo a 90 voltios durante 50 minutos. El ARN se observó en un transiluminador con luz ultravioleta a 260 nm. La radiación UV es absorbida por el ARN y emitida al bromuro de etidio que a su vez reemite la radiación a 590 nm en la región rojo-naranja del espectro visible. Se tomó una fotografía de las muestras de ARN. La electroforésis se realizó en una cámara de electroforesis con peine previamente lavados con NaOH 1.0 N en agua DEPC al 1.0%. El tamaño del gel empleado fue de 10.0 x 6.0 cm con un espesor de 7.0 mm. Para preparar este gel se requirieron los siguientes reactivos: - Solución amortiguadora de corrida TBE 1X (Tris-borato 90 mm, ph 8.3; y EDTA 2.0 mm).
5 - Agarosa (Pronadisa) 10.5 Síntesis de ADNc (RT). El ADNc se sintetizó a partir del ARN total obtenido de ME y TAP por medio de la transcripción reversa, utilizando hexámeros. Las moléculas de ARNm funcionan como molde y los hexámeros al azar actúan como primers. Además, se requiere la presencia de dntp s que funcionan como sustrato para la transcriptasa reversa, de lo que resulta una hebra de ADN complementario. El RT-PCR se realizó en dos fases: Fase 1. 1) 2.0 µg de ARN 2) 0.5 µl de hexámeros (100 mm) 3) agua inyectable (completar el volumen de la reacción a 20.0 µl) En la primera fase, la reacción se sometió a un ciclo de desnaturalización a 72 C por 3 minutos en un termociclador (T1 Termocycler, Biometra TM ), transcurrido este tiempo el tubo que contiene la reacción se puso en hielo para evitar su renaturalización y se agregó lo siguiente:
6 Fase 2. 1) 4.0 µl de solución amortiguadora 5X (Tris-HCl 250 mm, ph 8.3, KCl 375 mm y MgCl 2 15 mm). 2) 1.0 µl de mezcla de dinucleótidos trifosfato (dntp s) [10 mm de adenina trifosfato (datp), citosina trifosfato (dctp), guanina trifosfato (dgtp), y timina trifosfato (dttp) con NaOH 10 mm, ph 7.0]. 3) 1.0 µl (200U/µl) de la enzima transcriptasa reversa M-MLV (virus de la leucemia murina de Moloney Invitrogen TM Life Technologies). 4) 2.0 µl del detergente ditiotreitol (DTT) 0.1 M como inhibidor de RNAsas. Esta mezcla se sometió a un ciclo de síntesis del ADNc a 37 C por 60 minutos y a un ciclo final de desnaturalización de la enzima a 94 C por 6 minutos en el termociclador. Una vez que terminaron todos los ciclos, el ADNc se almacenó a -20 C Diseño de los oligonucleótidos. Para llevar a cabo los ensayos de PCR se diseñaron oligonucleótidos específicos para cada uno de los transcritos de los miembros del subcomplejo SG-SSPN, así como de la enzima gliceraldehído 3 fosfato deshidrogenasa (gapdh), cuyo transcrito fue considerado como control de expresión constitutiva. a) Sarcoglicanos El diseño de los oligonucleótidos para cada uno de los SGs se realizó a partir de una región homóloga (> 90%) de la rata (Rattus norvegicus) con el ratón (Mus musculus), obtenida mediante el alineamiento de las secuencias reportadas para cada transcrito (subunidades α, β, γ, δ, ε ó ζ) en ambas especies. En la tabla 3 se enlista el número de acceso de la página web ( para cada secuencia consultada, al igual que las posiciones inicial y final de la secuencia homóloga determinada.
7 Tabla 3. Secuencias homólogas de la rata y del ratón para los genes de sarcoglicanos y el gen de sarcospan. Transcrito No. Acceso a Identidad b Homología Ubicación sitio blanco c RN MM (%) RN MM sgca XM_ NM_ / sgcb XM_ NM_ / sgcg XM_ NM_ / sgcd XM_ NM_ / sgce AY NM_ / sgcz XM_ NM_ / sspn NW_ NM_ / \Rn4_ / / a Se refiere al número de acceso de la secuencia reportada para cada transcrito en GeneBank NCBI (URL: a excepción de sarcospan de rata cuyo número de acceso corresponde a las secuencias contiguas del cromosoma 4. b Se refiere al número de pares de bases idénticas con respecto al número total de pares de bases que comprende la región homóloga. c Los números corresponden a las posiciones inicial y final de la secuencia homóloga determinada conforme a la secuencia del gen dela rata y el ratón reportada en la base de datos GeneBank de NCBI. MM, Mus musculus (ratón), RN, Rattus norvegicus (rata); SG, sarcoglicano; SSPN, sarcospan
8 El diseño de los oligonucleótidos ( primers ) dentro de la secuencia homóloga para cada transcrito se realizó con el programa OLIGO 4.1 (Primer analysis software, National Biosciences Inc ). Los parámetros para este diseño fueron los siguientes: Longitud equivalente a 20 nt Cantidad de GC 50% Temperatura de alineamiento 60 C Alta compatibilidad (Probabilidad reducida de formación estable de asas y dímeros). NOTA: El diseño de los oligos se realizó únicamente con regiones exónicas eludiendo aquellas secuencias entre las uniones exón-intrón con el fin de evitar una probable amplificación inespecífica del ADN genómico. b) Sarcospan En vista de que a la fecha no se ha reportado la secuencia del gen de sarcospan en la rata, el diseño de los oligonucleótidos fue a partir de una región homóloga ( 90%) de la rata (Rattus norvegicus) con el ratón (Mus musculus) que se obtuvo mediante el alineamiento de la secuencia reportada para el ARNm de sspn del ratón (NM_010656) contra el genoma de la rata (NW_ Rn4_2219). Posteriormente, se secuenció el producto amplificado del tamaño esperado con la finalidad de corroborar que el transcrito obtenido corresponde al gen sspn. c) Gliceraldehído 3-fosfato deshidrogenasa No fue necesario diseñar oligonucleótidos para gapdh en rata, ya que los diseñados para ratón se probaron y funcionaron. La secuencia de los oligonucleótidos, la temperatura de fusión y el tamaño esperado de los productos amplificados para cada transcrito se enlistan en la tabla 4.
9 Tabla 4. Secuencia de los oligonucleótidos utilizados para la reacción de PCR. Nombre a Gen blanco Tamaño producto T m ( C) Secuencia (5 > 3 ) Longitud (nt) de PCR (pb) α SG-F sgca AGA GGC TGC TGC TGC TGA TT 20 α SG-R GAG GTG CCA AGG TGT CAT AG 20 β SG-F sgcb CTG GCT GTC ATC AAC CTG CT 20 β SG-R AAC ACG CCT TCA TTG CCT CG 20 γ SG-F sgcg CCA GAG CCA GCA CTT ACA GA 20 γ SG-R GAA GAG GGA GTG GTC AGA GA 20 δ SG-F sgcd TAC GGC TGG AGG AAA AGA TG 20 δ SG-R TGG CGT AGA GAG GTT GTA AG 20 ε SG-F sgce GGA ACG CCT CAA TGC TAT CA 20 ε SG-R TGA GTC TGG TGT GGC AAG TT 20 ζ SG-F sgcz TGA AGT GAG AGC CAG TGA AG 20 ζ SG-R GCA GAC GCA CAG TTC ATA CA 20 ζ SG-F1 sgcz TGC AGG TGA CTT CAA GGC TA 20 ζ SG-R1 GTG AAG AAG AGA AGG AAC CG 20 ζ SG-F1 sgcz TGC AGG TGA CTT CAA GGC TA 20 ζ SG-R GCA GAC GCA CAG TTC ATA CA 20 ζ SG-F sgcz TGA AGT GAG AGC CAG TGA AG 20 ζ SG-R1 GTG AAG AAG AGA AGG AAC CG 20 Sspn-F sspn GCT AGT CAG AGA CAC TCC GT 20 Sspn-R GCA GCC CAA CAC CCA CAT AA 20 Gapdh-F gapdh ATC CCA TCA CCA TCT TCC AG 20 Gapdh-R TGT GGT CAT GAG TCC TTC CA 20 a Dentro del nombre las letras F ó R denotan que se trata de un oligonucleótido forward ó reversa respectivamente. gapdh, gliceraldehído-3-fosfato dehidrogenasa; sgca, α sarcoglicano; sgcb, β sarcoglicano; sgcd, δ sarcoglicano; sgcg, γ sarcoglicano; sgce, ε sarcoglicano; sgcz, ζ sarcoglicano; sspn, sarcospan; T m, temperatura de fusión Amplificación de ADNc (RT-PCR).
10 La PCR consiste en la amplificación logarítmica de un segmento definido de ADN. Para amplificar las regiones transcritas de los miembros del complejo SG-SSPN, los ensayos de PCR fueron realizados con los siguientes reactivos: 1) 2.5 µl de ADNc 2) 0.5 µl de mezcla de dinucleótidos trifosfato (dntp s) [10 mm de adenina trifosfato (datp), citosina trifosfato (dctp), guanina trifosfato (dgtp), y timina trifosfato (dttp) con NaOH 10 mm, ph 7.0]. 3) 2.5 µl de solución amortiguadora para PCR 10X (Tris-HCl 200 mm, ph 8.4; KCl 500 mm) 4) 1.5 µl de MgCl 2 (50 mm) 5) 1.0 µl (20 pmoles) de cada uno de los oligonucleótidos (forward y reverse) 6) 0.2 µl de Taq-polimerasa (5U/µl) (Invitrogen TM Life Technologies) 7) 15.8 µl de agua para completar la reacción a un volumen total de 25.0 µl La amplificación se realizó bajo las siguientes condiciones: i. 5 min, 94ºC (desnaturalización inicial) ii. 30 ciclos de: 30 seg., 94ºC (desnaturalización) 30 seg., T m (alineamiento) 30 seg., 72ºC (extensión) iii. 7 min. 72ºC (extensión final) Después de la amplificación, las muestras se almacenaron a 20 C. Los productos obtenidos del PCR se sometieron a electroforesis en un gel de agarosa horizontal al 2.0% teñido con bromuro de etidio (0.5 µg/ml). De los 25.0 µl del volumen total de reacción del PCR se tomaron 5.0 µl y se mezclaron con 2.0 µl de colorante de corrida, azul cianolxileno al 0.25% en 30% de glicerol y 2.0 µl de buffer TBE 1X.
11 Para la preparación y electroforesis del gel se requirieron los siguientes reactivos y equipo: - Solución amortiguadora de corrida TBE 1X (Tris-borato 90 mm, ph 8.3; y EDTA 2.0 mm). - Agarosa (Pronadisa) - Cámara de electroforesis con peine previamente lavados con agua destilada. El tamaño del gel empleado fue de 13.0 x 13.0 cm con un espesor de 7.0 mm. Al gel se le aplicaron 100 voltios durante 50 minutos. Todos los ensayos fueron realizados por triplicado. Los productos amplificados por PCR se visualizaron en el gel por medio de luz UV y se les tomó una fotografía. NOTA: Debido a la baja expresión del transcrito de ζ sarcoglicano, se realizaron ensayos de PCR anidado y semianidado mediante combinaciones de dos pares de oligonucleótidos específicos para verificar la presencia del transcrito correspondiente. Las condiciones de amplificación para este transcrito requirieron los siguientes cambios: 30 ciclos con el primer par de oligonucleótidos (ζ SG-F y ζ SG-R) bajo las condiciones antes descritas. Posteriormente, 1.0 µl del producto de la primera amplificación fue sometido a 30 ciclos con el segundo par de oligonucleótidos respectivo (ζ SG-F1 y ζ SG-R1; ζ SG-F1 y ζ SG-R; ζ SG-F y ζ SG-R1) (ver tabla 4) bajo condiciones idénticas a la primera amplificación Secuenciación de los productos amplificados de los transcritos de sspn y scgz. Para la purificación de la banda correspondiente al tamaño esperado del producto amplificado, se siguió el protocolo de extracción rápida en gel por centrifugación en columnas de resina-silica (Marligen Bioscience TM Inc.).
12 Para la reacción de secuenciación se utilizaron los siguientes reactivos: 1) 4.0 µl de mix Big Dye Terminator v3.1 (Applied Biosystems TM ) 2) 2.0 µl de solución amortiguadora 5X (Applied Biosystems TM ) 3) 0.32 µl de oligonucleótidos (oligo forward ó reverse) 4) 50 ng de producto de PCR 5) agua inyectable (completar el volumen final de la reacción a 20.0 µl) La reacción se realizó bajo las siguientes condiciones: i. 3 min, 96ºC (desnaturalización inicial) ii. 25 ciclos de: 10 seg., 96ºC (desnaturalización) 10 seg., T m (alineamiento) 4 min., 60ºC (extensión) La rampa de temperatura se ajustó a 1.0 ºC / seg. Al terminar los 25 ciclos de la reacción, a cada tubo se le adicionaron 80.0 µl de isopropanol al 65% y se dejó incubando durante 30 minutos a temperatura ambiente. Posteriormente se centrifugó a 14,000 rpm por 25 minutos a temperatura ambiente. Mediante un extractor con vacío se retiró completamente el isopropanol y se adicionaron 300 µl de etanol al 60%. Se dejó incubando durante 10 minutos a temperatura ambiente y después de ese tiempo se centrifugó a 14,000 rpm durante 15 minutos. Nuevamente con vacío se eliminó el etanol por completo. Finalmente, las muestras se secaron a 55ºC durante ~5 min. en una centrífuga al vacío (Concentrator 5301 Eppendorf TM ). Las muestras procesadas se secuenciaron en un equipo Abi Prism TM 377 DNA Sequencer (Applied Biosystems TM ) en el Centro de Instrumentos por la Dra. Norma Pernas Buitrón Alineamiento de la secuencia del transcrito de sspn y su correspondiente secuencia de aminoácidos.
13 La secuencia obtenida para el transcrito amplificado de sarcospan, así como su secuencia traducida (obtenida con el programa DNAMAN versión 3.0 Lynnon BioSoft 94-97) proveniente de tejido adiposo parametrial se sometió a un alineamiento múltiple de agrupamiento jerárquico con el programa Multalin versión (I.N.R.A. Francia 1989, 1991, 1994, 1996). El alineamiento múltiple se realizó contra las secuencias reportadas para el gen y la proteína sarcospan en ratón (NM_010656, NP_ respectivamente) y humano (NM_005086, NP_ respectivamente) en análisis separados Ensayos de inmunofluorescencia indirecta. Con la finalidad de determinar la localización de sarcospan en los adipocitos se disecó el tejido adiposo parametrial de la rata (TAP). Con el tejido fresco se realizaron frotis sobre portaobjetos previamente tratados con poli-l-lisina. Los portaobjetos fueron sumergidos toda la noche a temperatura ambiente en una solución de poli-l-lisina (Sigma-Aldrich Co.) disuelta 1:10 con agua desionizada. Antes del bloqueo, el tejido adiposo de los frotis se permeabilizó con Triton X-100 al 0.3% p/v, PBS 1X a temperatura ambiente durante 3 minutos. Para bloquear la señal de fondo (señal inespecífica) los frotis se incubaron con solución bloqueadora, la cual se compone de amortiguador de sales de fosfato de sodio (PBS) 0.5 M, albúmina sérica de bovino al 5.0% p/v, suero fetal bovino al 5.0% p/v y 0.5% p/v de gelatina de laboratorio durante 60 minutos a temperatura ambiente. Transcurrido este tiempo las muestras se incubaron con el anticuerpo primario correspondiente (el cual fue donado por el Dr. Dominique Mornet de la Facultad de Farmacia del INSERM Montpellier, Francia) (tabla 5) a 4 C toda la noche. Al término de este tiempo, las preparaciones se lavaron cuatro veces con PBS 1X frío por 5 minutos cada vez. Enseguida se incubaron por una hora a temperatura ambiente con el anticuerpo secundario respectivo enumerado en la tabla 5. Transcurrido el tiempo de incubación con el anticuerpo secundario, las laminillas se lavaron cuatro veces con PBS 1X frío, 5 minutos cada vez. Posteriormente se eliminó el exceso de solución amortiguadora y adicionaron 10 µl de medio de montaje Vecta Shield con 6-diamidino-2-fenilindol, DAPI (Laboratorios Vector), el cual tiñe los núcleos de color azul. Finalmente, se colocó un cubreobjetos sobre la muestra sellando
14 las orillas del mismo con un poco de barniz de uñas para evitar que la muestra se secara, y que el cubreobjetos se deslizara sobre el portaobjetos. Las señales de fluorescencia de las muestras se observaron bajo un microscopio de fluorescencia láser con un aumento de 20X y 40X y se les tomó una fotografía. Tabla 5. Lista de anticuerpos para inmunofluorescencia. No. Nombre Dilución a Región/dominio blanco PRIMARIO 1 Sarcospan : (policlonal, conejo) 1:50 región COO - (últimos 13 aa) SECUNDARIO 2 Cabra anti-conejo IgG-FITC 1:100 - Jackson Immunoresearch Laboratories, Inc. (URL: a Se refiere a la dilución recomendada para ensayos de inmunofluorescencia. FITC, Conjugado isotiociantato de fluoresceína (Fluorocormo).
11.1 Expresión de los transcritos del subcomplejo SG-SSPN en el tejido adiposo parametrial.
 11. RESULTADOS Mediante este trabajo se identificó la expresión de los transcritos de α, β, γ, δ, ε y ζ SG y sspn en tejido adiposo parametrial de la rata y se localizó la proteína sspn en la membrana
11. RESULTADOS Mediante este trabajo se identificó la expresión de los transcritos de α, β, γ, δ, ε y ζ SG y sspn en tejido adiposo parametrial de la rata y se localizó la proteína sspn en la membrana
ELABORACIÓN N DE MEZCLA PARA PCR. Raquel Asunción Lima Cordón
 ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
ELABORACIÓN N DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede
Módulo 1 Biología Molecular Genética Molecular II 2009. Módulo 1. Amplificación de un fragmento de ADN genómico por PCR
 Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
Módulo 1 Amplificación de un fragmento de ADN genómico por PCR En este primer módulo se amplificará por PCR, mediante el uso de oligonucleótidos específicos, un fragmento de ADN genómico de Aspergillus
- Clonar un gen (molde ADN o ARN)
 APLICACIONES PCR Aplicaciones de la PCR - Clonar un gen (molde ADN o ARN) - Secuencia conocida - Genes homólogos en especies próximas (primers degenerados) - Información de secuencia proteica (primers
APLICACIONES PCR Aplicaciones de la PCR - Clonar un gen (molde ADN o ARN) - Secuencia conocida - Genes homólogos en especies próximas (primers degenerados) - Información de secuencia proteica (primers
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN
 5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. DETECCION DE GENES DE VIRULENCIA MEDIANTE HIBRIDACIÓN CON SONDAS DE ADN Materiales - Eppendorf de 1,5 ml estériles - Eppendorf de pared fina para PCR - Puntas de pipeta estériles desechables - Guantes
5. MATERIALES Y MÉTODOS
 5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
5. MATERIALES Y MÉTODOS 5.1 Equipos Los siguientes termocicladores se emplearon para la amplificación por PCR: - Perkin Elmer, GeneAmp PCR System 2400. - Perkin Elmer, GeneAmp PCR System 9600. - Applied
Easy PDF Creator is professional software to create PDF. If you wish to remove this line, buy it now.
 Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
Unidad Curricular: Virología y Micología Veterinaria 1 TRABAJO PRÁCTICO No. 3 AMPLIFICACIÓN DE GENES VIRALES Reacción en Cadena de la Polimerasa, conocida como PCR (por sus siglas en inglés: Polimerase
DETECCIÓN DE C. jejuni EN HAMBURGUESA DE POLLO POR PCR TRADICIONAL PRT-712.04-082
 Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Página 1 de 5 1. OBJETIVO Detectar la presencia de Campylobacter jejuni en hamburguesa de pollo mediante técnica de PCR. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a muestras de hamburguesa
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa.
 Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
Detección de patógenos en alimentos, utilizando la técnica de PCR, reacción en cadena de la polimerasa. Lic. Esp. Mariano Diego Massari 1er Congreso Bioquímico Córdoba 2011 8ª Jornada de Actualización
DANAGENE RNA PURIFICATION KIT
 DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
DANAGENE RNA PURIFICATION KIT REF.0801.1 100 EXTRACCIONES REF.0801.2 500 EXTRACCIONES 1.INTRODUCCION Este kit permite la permite la obtención de ARN total a partir de cultivos celulares, tejidos animales,
CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL
 LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
LIBRO DE MEMORIAS 2 CURSO DE EXTENSIÓN TEÓRICO PRÁCTICO HERRAMIENTAS EFICIENTES PARA LA IDENTIFICACIÓN DE LEVADURAS DE INTERÉS AGROINDUSTRIAL Santiago de Cali, 23 al 28 de agosto de 2010 3 Libro de memorias
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015
 CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
CLASE DE RESOLUCIÓN DE PROBLEMAS DE PCR LIC. BIOTECNOLOGÍA 2015 DEFINICIÓN PCR: Constituye la técnica más importante para amplificar ácidos nucleicos in vitro. Ambas hebras del DNA de interés son replicadas
Materiales para la secuenciación de ADN
 Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
Introduccion La Secuenciación Sanger es un método de secuenciación de ADN en el que el ADN diana se desnaturaliza y se hibrida con un cebador de oligonucleótidos, que se extiende entonces gracias a la
PCR gen 18S ARNr humano
 PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
PCR gen 18S ARNr humano Ref.PCR18S 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA. Grado en Medicina
 DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
DEPARTAMENTO DE BIOQUÍMICA Y BIOLOGÍA MOLECULAR III FACULTAD DE MEDICINA UNIVERSIDAD COMPLUTENSE DE MADRID CUADERNO DE PRÁCTICAS GENETICA MOLECULAR HUMANA Grado en Medicina CURSO ACADÉMICO 2014/2015 INDICE
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR)
 METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
METODOLOGÍAS UTILIZADAS EN INVESTIGACIÓN BÁSICA Y APLICADA REACCION EN CADENA DE LA POLIMERASA (PCR) Centro de de Microscopía Electrónica Facultad de de Ciencias Médicas Universidad Nacional de de Córdoba
PCR. Componentes del PCR. PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa)
 PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
PCR PCR Polymerase Chain Reaction (Reacción en Cadena de la Polimerasa) Es una técnica de biología molecular descrita en 1986 por Kary Mullis. (Nobel de química en 1993) Necesidad de pruebas de diagnóstico
CUESTIONES. 1. Por qué la PCR convencional no es cuantitativa?
 2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
2º BIOQUÍMICA CUESTIONES 1. Por qué la PCR convencional no es cuantitativa? En la PCR convencional, la cuantificación del ADN de la muestra es complicada debido a que la eficacia de amplificación va disminuyendo
Protocolos de secuenciación de ADN
 1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
1 Protocolos de secuenciación de ADN Introducción El método de secuenciación utilizado aquí es el desarrollado por Sanger en 1975. Este consiste en la incorporación de didesixinucleótidos marcados fluorescentemente
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA"
 P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
P.C.R. "Técnica de amplificación enzimática de secuencias específicas de DNA" Paso 1: Desnaturalización del DNA Paso 2: Hibridación de los "Primers" Paso 3: Extensión Paso 1: Desnaturalización del DNA
Código: IDX-016 Ver: 1 TOXO. Sistema para la detección de la presencia de ADN de Toxoplasma gondii. Reg. MSP 21205
 Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
Sistema para la detección de la presencia de ADN de Toxoplasma gondii Reg. MSP 21205 Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. info@atgen.com.uy www.atgen.com.uy
PROCEDIMIENTO DETECCION NOROVIRUS RT-qPCR EN EXTRACTO VIRAL PROVENIENTE DE MOLUSCOS BIVALVOS. PRT-712.07.01-097 Página 1 de 7
 PRT-712.07.01-097 Página 1 de 7 1. OBJETIVO Realizar la detección molecular de genes de Norovirus en muestras de moluscos bivalvos. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a concentrados
PRT-712.07.01-097 Página 1 de 7 1. OBJETIVO Realizar la detección molecular de genes de Norovirus en muestras de moluscos bivalvos. 2. CAMPO DE APLICACIÓN Y ALCANCE Aplicar este procedimiento a concentrados
El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN.
 PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
PROGRAMA CONTROL DE CALIDAD DE MUESTRAS El Banco Nacional de ADN oferta un control de calidad de muestras de ADN y ARN. El programa completo de control de calidad de muestras de ADN y ARN engloba diferentes
Biotecnología. Anotación de genes. Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla
 Biotecnología Anotación de genes Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla Localizar los genes Segundo paso en el análisis del genoma ab initio: propiedades estadísticas
Biotecnología Anotación de genes Dpto. Ciencias de la Computación e Inteligencia Artificial Universidad de Sevilla Localizar los genes Segundo paso en el análisis del genoma ab initio: propiedades estadísticas
APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS
 Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
Prácticas docentes en la COD: 10-71 APLICACIÓN DE LA PCR: DIAGNÓSTICO DE PARÁSITOS FUNDAMENTO TEÓRICO La PCR es una técnica que permite llevar a cabo la síntesis in vitro de fragmentos de ADN. Está basada
MANUAL DE PRÁCTICAS DE LABORATORIO
 MANUAL DE PRÁCTICAS DE LABORATORIO INGENIERIA ENBIOTECNOLOGÍA INGENIERÍA GENÉTICA FICHA TECNICA FICHA TÉCNICA SEMINARIO DE BIOTECNOLOGÍA MÉDICO-FARMACÉUTICA Fecha: Nombre del catedrático: 10-Julio-2012
MANUAL DE PRÁCTICAS DE LABORATORIO INGENIERIA ENBIOTECNOLOGÍA INGENIERÍA GENÉTICA FICHA TECNICA FICHA TÉCNICA SEMINARIO DE BIOTECNOLOGÍA MÉDICO-FARMACÉUTICA Fecha: Nombre del catedrático: 10-Julio-2012
velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz porosa
 INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
INTRODUCCIÓN En general, la electroforesis es una técnica que separa las moléculas en base a sus diferentes velocidades de movimiento mediante la aplicación de un campo eléctrico a través de una matriz
Practica la genética Ficha didáctica del profesorado Bachillerato
 Ficha didáctica del profesorado Bachillerato www.eurekamuseoa.es Extracción de ADN Cuál es la función de cada uno de los ingredientes utilizados para realizar la disolución tampón para la visualización
Ficha didáctica del profesorado Bachillerato www.eurekamuseoa.es Extracción de ADN Cuál es la función de cada uno de los ingredientes utilizados para realizar la disolución tampón para la visualización
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O. Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua
 C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
C A P Í T U L O 3 M A T E R I A L E S Y M É T O D O Se ejecutaron varias pruebas para la inactivación de Escherichia Coli ATCC 25922 en agua destilada utilizando Dióxido de Titanio dopado con Nitrógeno,
Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml
 Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml Para la purificación de ADN genómico de todos los kits de colección Oragene y ORAcollect. Visite nuestro sitio
Protocolo de laboratorio para purificación manual de ADN a partir de una muestra de 0,5 ml Para la purificación de ADN genómico de todos los kits de colección Oragene y ORAcollect. Visite nuestro sitio
FRAGMENTO OLIGO 5-3 Hebra SECUENCIA 5' - - > 3' TAMAÑO (pb) F1. hmtl 569 L AACCAAACCCCAAAGACACC hmth2982 H CTGATCCAACATCGAGGTCG
 ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
ANEXO 1 Protocolo para la PCR para la amplificación del mtdna en 10 fragmentos solapantes: Se prepara la siguiente mezcla: Tampón ( TRIS 100 mm, MgCl 2 12 mm, KCl 500 mm, ph 8,3): 10X dntps : 10 mm (cada
Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri
 Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri OBJETIVO Desarrollar un banco de datos a partir de plantas de origen indudable y del uso
Identificación varietal en vid por técnicas de Biología Molecular. Lic. Luciana Garcia Lic. Carolina Chiconofri OBJETIVO Desarrollar un banco de datos a partir de plantas de origen indudable y del uso
3. COMPONENTES. Tampón de electroforesis concentrado 2 x 50 ml 10 X (2 envases 500ml)
 PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
PCR SIMULADA Ref.PCR Simulada (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
3/22/2010. Iván Ferrer Rodríguez, PhD Catedrático. En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR.
 Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
Iván Ferrer Rodríguez, PhD Catedrático En el 1983, Kary Mullis dio a conocer la Técnica de reacción en cadena de la Polimerasa o PCR. Síntesis "in vitro" de secuencias específicas de ADN son amplificadas.
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón
 RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
RT- PCR diagnóstica del Cotton leafroll dwarf virus (CLRDV) en plantas de algodón La identificación rápida y precisa de los patógenos que afectan a los cultivos de interés agronómico es crítica para predecir
DETERMINACIÓN DEL FACTOR Rh por PCR
 Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
Ref.PCRRh DETERMINACIÓN DEL FACTOR Rh por PCR 1.OBJETIVO DEL EXPERIMENTO El objetivo de este experimento es introducir a los estudiantes en los principios y práctica de la Reacción en Cadena de la Polimerasa
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR)
 MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
MICROBIOLOGÍA DE LOS ALIMENTOS 2015 INGENIERÍA EN ALIMENTOS LICENCIATURA EN BIOQUÍMICA TRABAJO PRÁCTICO N 1 REACCIÓN EN CADENA DE LA POLIMERASA (PCR) OBJETIVOS - Definir PCR, conocer los reactivos que
Bioquímica III- 2009
 Facultad de Ciencias Exactas, Universidad Nacional de La Plata Bioquímica III- 2009 Trabajo Práctico Nro 4 Extracción de RNA y DNA bacteriano INTRODUCCIÓN El RNA es el ácido nucleico más abundante en la
Facultad de Ciencias Exactas, Universidad Nacional de La Plata Bioquímica III- 2009 Trabajo Práctico Nro 4 Extracción de RNA y DNA bacteriano INTRODUCCIÓN El RNA es el ácido nucleico más abundante en la
AQUAGESTION. ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico.
 AQUAGESTION ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico. Noviembre 2011 ACTUALIDAD Este año Sernapesca confirma
AQUAGESTION ISAv: Un acercamiento actualizado en la detección de sus variantes en la región. Departamento de Desarrollo y Laboratorio Diagnóstico. Noviembre 2011 ACTUALIDAD Este año Sernapesca confirma
Reacción en cadena de la polymerasa (PCR)
 Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Reacción en cadena de la polymerasa (PCR) 1 PCR Que es? Es una técnica in vitro diseñada para amplificar una región específica de DNA. Es extremadamente sensible, con la capacidad de amplificar millones
Código: IDK-010 Ver: 1. Proteína G C825T. Sistema para la detección de la mutación C825T en el gene de la proteína G.
 C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
C825T Sistema para la detección de la mutación C825T en el gene de la proteína G. Valdense 3616. 11700. Montevideo. Uruguay. Teléfono (598) 2 336 83 01. Fax (598) 2 336 71 60. Info@atgen.com.uy www.atgen.com.uy
FILTROS ELECTROPOSITIVOS Y REACCIÓN DE PCR PARA LA CONCENTRACIÓN Y DETECCIÓN SIMULTÁNEA DE VIRUS Y BACTERIAS EN AGUAS. Víctor Ramírez Angulo
 FILTROS ELECTROPOSITIVOS Y REACCIÓN DE PCR PARA LA CONCENTRACIÓN Y DETECCIÓN SIMULTÁNEA DE VIRUS Y BACTERIAS EN AGUAS Víctor Ramírez Angulo Coordinación de Tratamiento y Calidad del Agua, Instituto Mexicano
FILTROS ELECTROPOSITIVOS Y REACCIÓN DE PCR PARA LA CONCENTRACIÓN Y DETECCIÓN SIMULTÁNEA DE VIRUS Y BACTERIAS EN AGUAS Víctor Ramírez Angulo Coordinación de Tratamiento y Calidad del Agua, Instituto Mexicano
Técnicas de Biología Molecular. Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Extracción de ácidos nucleicos o o DNA RNA Tipos de Muestras Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Extracción de ácidos nucleicos o o DNA RNA Tipos de Muestras Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR
 TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Andrés Ciocchini, Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN) por partición diferencial
TRABAJO PRÁCTICO Nº 5: ÁCIDOS NUCLEICOS DOCENTES: Andrés Ciocchini, Gabriela Niemirowicz. DESCRIPCIÓN DE LOS MÉTODOS A UTILIZAR Purificación de ácido desoxiribonucleico (ADN) por partición diferencial
GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS
 GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS Leidy Diana Ardila Leal Docente. INTRODUCCIÓN En esta práctica se va a realizar la extracción
GUIA DE LABORATORIO PRACTICA 8 EXTRACCIÓN ADN PROGRAMA DE ENFERMERIA CURSO INTEGRADO DE PROCESOS BIOLOGICOS Leidy Diana Ardila Leal Docente. INTRODUCCIÓN En esta práctica se va a realizar la extracción
Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala
 Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala IICA/Red SICTA-CIAT INFORME DE AVANCE Gerardo Gallego - Harold
Caracterización morfo-agronómica y molecular de frijoles criollos de color rojo claro en Nicaragua y de frijoles negros en Ipala, Guatemala IICA/Red SICTA-CIAT INFORME DE AVANCE Gerardo Gallego - Harold
CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA)
 CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA) PROTOCOLO ADA FUNDAMENTO DEL METODO: La adenosindesaminasa (ADA) es una enzima del catabolismo de las purinas que cataliza la conversión de la adenosina en inosina
CUANTIFICACIÓN DE ADENOSIN DESAMINASA (ADA) PROTOCOLO ADA FUNDAMENTO DEL METODO: La adenosindesaminasa (ADA) es una enzima del catabolismo de las purinas que cataliza la conversión de la adenosina en inosina
Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a
 Introducción Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a los investigadores verificar la expresión de una proteína, determinar la cantidad relativa
Introducción Western Blotting (o inmunotransferencia) es un procedimiento estándar de laboratorio que permite a los investigadores verificar la expresión de una proteína, determinar la cantidad relativa
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa
 38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
38. Purificación de ADN plasmídico y electroforesis del mismo en gel de agarosa José Luis Caballero Repullo, Enriqueta Moyano, Juan Muñoz Blanco Departamento de Bioquímica y Biología Molecular, Campus
1) a) En la figura señale, englobando con un trazo continuo, lo siguiente:
 CÁTEDRA: BIOQUÍMICA Carreras: Farmacia Profesorado en Química Licenciatura en Química Licenciatura en Alimentos ÁCIDOS NUCLEICOS 1) a) En la figura señale, englobando con un trazo continuo, lo siguiente:
CÁTEDRA: BIOQUÍMICA Carreras: Farmacia Profesorado en Química Licenciatura en Química Licenciatura en Alimentos ÁCIDOS NUCLEICOS 1) a) En la figura señale, englobando con un trazo continuo, lo siguiente:
ELECTROFORESIS AVANZADA
 Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECAVANZADA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS AVANZADA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
DANAGENE SALIVA KIT. Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones
 DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
DANAGENE SALIVA KIT Ref.0603.4 50 Extracciones / Ref.0603.41 160 Extracciones 1.INTRODUCCION DANAGENE SALIVA Kit provee un método para la extracción de ADN genómico de alta calidad a partir de muestras
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES
 PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
PROBLEMAS MÁS FRECUENTES Y POSIBLES SOLUCIONES NO SE PRODUCE REACCIÓN O ÉSTA DECAE POCO DESPUÉS DE COMENZAR No hay ADN o hay menos del necesario No hay cebador o la concentración es inferior a la necesaria.
1 MATERIALES Y MÉTODOS
 1 MATERIALES Y MÉTODOS El presente protocolo experimental contempla la amplificación del DNA de las bacterias y virus causantes de las ETS Neisseria gonorrhoeae, Chlamydia trachomatis y VPH mediante PCR
1 MATERIALES Y MÉTODOS El presente protocolo experimental contempla la amplificación del DNA de las bacterias y virus causantes de las ETS Neisseria gonorrhoeae, Chlamydia trachomatis y VPH mediante PCR
ELECTROFORESIS EN GELES DE AGAROSA. Agustín Garrido. agugarrido@hotmail.com
 1 ELECTROFORESIS EN GELES DE AGAROSA Agustín Garrido agugarrido@hotmail.com Introducción En este trabajo práctico se utilizó la técnica de electroforesis. Este proceso se basa en la migración de las moléculas
1 ELECTROFORESIS EN GELES DE AGAROSA Agustín Garrido agugarrido@hotmail.com Introducción En este trabajo práctico se utilizó la técnica de electroforesis. Este proceso se basa en la migración de las moléculas
Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática.
 Departamento de Bioquímica Médica y Biología Molecular Práctica 3 Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática. Curso 1 Medicina (Abril) 2012 BIOQUÍMICA
Departamento de Bioquímica Médica y Biología Molecular Práctica 3 Metodología y aplicaciones de la amplificación de material genético: Introducción a la bioinformática. Curso 1 Medicina (Abril) 2012 BIOQUÍMICA
4. Materiales y Métodos. Los equipos que a continuación se mencionan se encuentran en el laboratorio de
 39 4. Materiales y Métodos 4.1 Equipos Los equipos que a continuación se mencionan se encuentran en el laboratorio de Ingeniería Ambiental de la Universidad de las Américas Puebla y en el Laboratorio de
39 4. Materiales y Métodos 4.1 Equipos Los equipos que a continuación se mencionan se encuentran en el laboratorio de Ingeniería Ambiental de la Universidad de las Américas Puebla y en el Laboratorio de
ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN
 Universidad Centroccidental Lisandro Alvarado Decanato de Ciencias de la Salud ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN Octubre 2009 INTRODUCCION La molécula de ADN (que es la que se
Universidad Centroccidental Lisandro Alvarado Decanato de Ciencias de la Salud ACTIVIDAD PRACTICA No. 8 BIOLOGIA CELULAR EXTRACCIÓN DE ADN Octubre 2009 INTRODUCCION La molécula de ADN (que es la que se
Criterios para diseñar primers. Iván Ferrer Rodríguez, Ph.D. Catedrático
 Criterios para diseñar primers Iván Ferrer Rodríguez, Ph.D. Catedrático 1 Qué es un primer? Es una cadena corta de nucleótidos, un oligonucleótido. Sirve como punto de partida para la replicación del DNA.
Criterios para diseñar primers Iván Ferrer Rodríguez, Ph.D. Catedrático 1 Qué es un primer? Es una cadena corta de nucleótidos, un oligonucleótido. Sirve como punto de partida para la replicación del DNA.
Guía Práctica 9. Producción de micelio en medio líquido para extracción de ADN. Micelio de hongos. Contenido
 Guía Práctica 9 Micelio de hongos Producción de micelio en medio líquido para extracción de ADN Guillermo Castellanos, Experto en Investigación 2 Carlos Jara, Ing. Agr. M.Sc., Asociado en Investigación
Guía Práctica 9 Micelio de hongos Producción de micelio en medio líquido para extracción de ADN Guillermo Castellanos, Experto en Investigación 2 Carlos Jara, Ing. Agr. M.Sc., Asociado en Investigación
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS.
 NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
NMX-F-070-1964. MÉTODO DE PRUEBA PARA LA DETERMINACIÓN DE TIAMINA. THIAMINE DETERMINATION. TEST METHOD. NORMAS MEXICANAS. DIRECCIÓN GENERAL DE NORMAS. ASUNTO Con fundamento en lo dispuesto en los Artículos
TaqMan GMO Maize Quantification Kit. Part No: 4481972
 TaqMan GMO Maize Quantification Kit Part No: 4481972 1. Introducción Los organismos modificados genéticamente (OMG) se encuentran ampliamente distribuidos, siendo la soja y el maíz los vegetales que ocupan
TaqMan GMO Maize Quantification Kit Part No: 4481972 1. Introducción Los organismos modificados genéticamente (OMG) se encuentran ampliamente distribuidos, siendo la soja y el maíz los vegetales que ocupan
Reacción en cadena de la polimerasa (PCR)
 Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Dr. Alejandro Leal Reacción en cadena de la polimerasa (PCR) La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés de "polymerase chain reaction") es un método de amplificación in vitro
Tecnología HaloPlex by Genycell Biotech España
 Paneles de genes custom-made Tecnología HaloPlex by Genycell Biotech España SERVICIO NGS CLÍNICA HALO: OVERVIEW 1. DISEÑO DEL PANEL DE GENES Y DEL KIT DE CAPTURA 2. PREPARACIÓN DE LA MUESTRA EN EL LABORATORIO
Paneles de genes custom-made Tecnología HaloPlex by Genycell Biotech España SERVICIO NGS CLÍNICA HALO: OVERVIEW 1. DISEÑO DEL PANEL DE GENES Y DEL KIT DE CAPTURA 2. PREPARACIÓN DE LA MUESTRA EN EL LABORATORIO
Utilidad de la Biología Molecular en el Diagnóstico y Vigilancia del Dengue
 Utilidad de la Biología Molecular en el Diagnóstico y Vigilancia del Dengue José Usme Ciro Unidad de Secuenciación y Análisis Genómico Grupo de Virología Instituto Nacional de Salud Virus dengue (DENV)
Utilidad de la Biología Molecular en el Diagnóstico y Vigilancia del Dengue José Usme Ciro Unidad de Secuenciación y Análisis Genómico Grupo de Virología Instituto Nacional de Salud Virus dengue (DENV)
Reacción en cadena de la Polimerasa (PCR)
 Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Definición de PCR Es la amplificación enzimática de un
Explicación de TP Nº 2 Reacción en cadena de la Polimerasa (PCR) Química Biológica Patológica Dra. Mariana L. Ferramola Dra. Gabriela Lacoste 2015 Definición de PCR Es la amplificación enzimática de un
Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz
 IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
IDEXX VetLab Suite IDEXX SNAP Tests Laboratorio de Referencia IDEXX Nuevos paneles de PCR Real Time: El diagnóstico molecular más rápido, fiable y eficaz Obtenga respuestas definitivas con la IDEXX RealPCR
CICLO 2 : Fraccionamiento subcelular
 Curso de Fisicoquímica Biológica 2006 Licenciatura de Bioquímica. Facultad de Ciencias. CICLO 2 : Fraccionamiento subcelular OBJETIVOS GENERALES: 1) Obtención de preparados enriquecidos en fracciones subcelulares
Curso de Fisicoquímica Biológica 2006 Licenciatura de Bioquímica. Facultad de Ciencias. CICLO 2 : Fraccionamiento subcelular OBJETIVOS GENERALES: 1) Obtención de preparados enriquecidos en fracciones subcelulares
FACULTAD REGIONAL ROSARIO
 FACULTAD REGIONAL ROSARIO CATEDRA BIOTECNOLOGIA ROFESOR: Ing. Eduardo Santambrosio JTP: Ing. Marta Ortega AUX 1ª : Ing. Pablo Garibaldi PCR (Polymerase chain reaction) Reacción en cadena de la polimerasa
FACULTAD REGIONAL ROSARIO CATEDRA BIOTECNOLOGIA ROFESOR: Ing. Eduardo Santambrosio JTP: Ing. Marta Ortega AUX 1ª : Ing. Pablo Garibaldi PCR (Polymerase chain reaction) Reacción en cadena de la polimerasa
Práctico: Genética bacteriana 2007.
 Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
Práctico: Genética bacteriana 2007. Actividad integrada Departamento de Genética Departamento de Bacteriología y Virología. OBJETIVOS Objetivos generales El estudiante al terminar la actividad práctica
o c Insertos Tinciones Hematológicas www.comacmexico.com 01 (55) 2163 4127 Lerdo # 30 Cuajimalpa México, D.F 05000 TINCIÓN DE WRIGHT WRIGHT BUFFER
 c ma o c TINCIÓN DE WRIGHT WRIGHT BUFFER ANTES DE USAR: a) Dejar madurar el colorante de Wright 5 días después de su elaboración. Insertos Tinciones Hematológicas. Colocar el frotis perfectamente seco
c ma o c TINCIÓN DE WRIGHT WRIGHT BUFFER ANTES DE USAR: a) Dejar madurar el colorante de Wright 5 días después de su elaboración. Insertos Tinciones Hematológicas. Colocar el frotis perfectamente seco
PCR. Técnica inventada por Kary Mullis :1987. Premio Nobel 1993. Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN
 PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
PCR PCR Técnica inventada por Kary Mullis :1987 Premio Nobel 1993 Amplifica una ÚNICA secuencia a partir de una mezcla compleja de ADN Aprovecha los requerimientos de la replicación Templete ADN Nucleótidos
III. METODOLOGÍA EXPERIMENTAL
 III. METODOLOGÍA EXPERIMENTAL 3.1 Análisis Previos Se utilizaron los filtros con muestra de partículas suspendidas en el aire PM 10 que el Instituto Municipal de Ecología (IME) proporcionó para llevar
III. METODOLOGÍA EXPERIMENTAL 3.1 Análisis Previos Se utilizaron los filtros con muestra de partículas suspendidas en el aire PM 10 que el Instituto Municipal de Ecología (IME) proporcionó para llevar
23. Hidrólisis ácida y enzimática del glucógeno
 23. Hidrólisis ácida y enzimática del glucógeno Emilia Martínez Galisteo, Carmen Alicia Padilla Peña, Concepción García Alfonso, José Antonio Bárcena Ruiz, Jesús Diez Dapena. Departamento de Bioquímica
23. Hidrólisis ácida y enzimática del glucógeno Emilia Martínez Galisteo, Carmen Alicia Padilla Peña, Concepción García Alfonso, José Antonio Bárcena Ruiz, Jesús Diez Dapena. Departamento de Bioquímica
AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO
 AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO E l estudio del genoma de los seres vivos a sido uno d los principales objetivos de la Biología. Desde los trabajos de Mendel (1866),
AISLAMIENTO DE ÁCIDO DESOXIRRIBONUCLEICO (DNA) GENÓMICO Y PLASMÍDICO E l estudio del genoma de los seres vivos a sido uno d los principales objetivos de la Biología. Desde los trabajos de Mendel (1866),
CAPÍTULO 4 DESARROLLO EXPERIMENTAL
 CAÍTUL 4 DEALL EXEIMETAL 4.1 Generalidades Los reactivos utilizados en las reacciones fueron de marca Aldrich. El desarrollo de las reacciones y los productos de reacción se siguió por medio de cromatografía
CAÍTUL 4 DEALL EXEIMETAL 4.1 Generalidades Los reactivos utilizados en las reacciones fueron de marca Aldrich. El desarrollo de las reacciones y los productos de reacción se siguió por medio de cromatografía
Prácticas de Análisis Instrumental
 Prácticas de Análisis Instrumental Asignatura: Análisis Instrumental Alumno: Daniel González Mancebo Practica 1. DETERMINACIÓN DE CONSTANTES DE EQUILIBRIO MEDIANTE ESPECTROFOTOMETRÍA UV- VISIBLE. Lo primero
Prácticas de Análisis Instrumental Asignatura: Análisis Instrumental Alumno: Daniel González Mancebo Practica 1. DETERMINACIÓN DE CONSTANTES DE EQUILIBRIO MEDIANTE ESPECTROFOTOMETRÍA UV- VISIBLE. Lo primero
Documentos de Referencia para Diagnóstico de Semilla de Papa de Canadá
 SECRETARIA DE AGRICULTURA, GANADERÍA. DESARROLLO RURAL, PESCA Y ALIMENTACIÓN Dirección General de Sanidad Vegetal Centro Nacional de Referencia Fitosanitaria Subdirección de Diagnóstico Fitosanitario Documentos
SECRETARIA DE AGRICULTURA, GANADERÍA. DESARROLLO RURAL, PESCA Y ALIMENTACIÓN Dirección General de Sanidad Vegetal Centro Nacional de Referencia Fitosanitaria Subdirección de Diagnóstico Fitosanitario Documentos
AUTOR: JORGE CONTRERAS PINEDA 1. Titulo:
 Página 1 de 1 1. Titulo: Electroforesis de DNA 2. Objetivo Conocer los principios básicos de la electroforesis horizontal en geles de agarosa y aplicarlo para la separación de DNA humano, plasmídico, recombinante
Página 1 de 1 1. Titulo: Electroforesis de DNA 2. Objetivo Conocer los principios básicos de la electroforesis horizontal en geles de agarosa y aplicarlo para la separación de DNA humano, plasmídico, recombinante
imegen Alfa-1-AT Manual de Usuario Referencia: IMG-211
 Manual de Usuario imegen Alfa-1-AT Genotipado de las mutaciones Glu342Lys (PI-Z) y Glu264Val (PI-S) del gen SERPINA1 mediante PCR a tiempo real Referencia: Fabricado en España Garantías y responsabilidades
Manual de Usuario imegen Alfa-1-AT Genotipado de las mutaciones Glu342Lys (PI-Z) y Glu264Val (PI-S) del gen SERPINA1 mediante PCR a tiempo real Referencia: Fabricado en España Garantías y responsabilidades
Usando Modelos de Markov para buscar genes
 Usando Modelos de Markov para buscar genes Anotando un genoma Una vez que tenemos la secuencia de un genoma, lo siguiente es ver qué es lo que está escrito ahí. A eso se le llama anotar el genoma. Qué
Usando Modelos de Markov para buscar genes Anotando un genoma Una vez que tenemos la secuencia de un genoma, lo siguiente es ver qué es lo que está escrito ahí. A eso se le llama anotar el genoma. Qué
Técnicas moleculares.
 Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Técnicas moleculares. DMTV Carolina Acevedo Curso de Microbiología 2015 SÍNTESIS IN VITRO DE UNA GRAN CANTIDAD DE COPIAS DE UN SEGMENTO DE ADN EXISTENTE EN UNA MUESTRA. O sea. Amplificamos en forma exponencial
Normalización de soluciones de NaOH 0,1N y HCl 0,1N.
 Laboratorio N 1: Normalización de soluciones de NaOH 0,1N y HCl 0,1N. Objetivos: - Determinar la normalidad exacta de una solución de hidróxido de sodio aproximadamente 0,1 N, utilizando biftalato de potasio
Laboratorio N 1: Normalización de soluciones de NaOH 0,1N y HCl 0,1N. Objetivos: - Determinar la normalidad exacta de una solución de hidróxido de sodio aproximadamente 0,1 N, utilizando biftalato de potasio
BIOLOGIA MOLECULAR. 04 de septiembre de 2017 Dra. Silvana Petrocelli
 CLASE DE PROBLEMAS: CLONADO BIOLOGIA MOLECULAR Lic. en Biotecnología 04 de septiembre de 2017 Dra. Silvana Petrocelli Problema 1-Clonado en fase-guía de problemas 2017 Se desea expresar un gen de superóxido
CLASE DE PROBLEMAS: CLONADO BIOLOGIA MOLECULAR Lic. en Biotecnología 04 de septiembre de 2017 Dra. Silvana Petrocelli Problema 1-Clonado en fase-guía de problemas 2017 Se desea expresar un gen de superóxido
PCR. Qué es la PCR? T a de fusión de oligonucleótidos (t m )
 % a de fusión de oligonucleótidos (t m ) Qué es la PCR? La reacción en cadena de la polimerasa (PCR: polymerase chain reaction) es una técnica de amplificación de secuencias de DNA in vitro t m = 2 (A
% a de fusión de oligonucleótidos (t m ) Qué es la PCR? La reacción en cadena de la polimerasa (PCR: polymerase chain reaction) es una técnica de amplificación de secuencias de DNA in vitro t m = 2 (A
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico
 17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
17.- Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de DNA plasmídico Carmen Alicia Padilla Peña, Jesús Diez Dapena, Emilia Martínez Galisteo, José
7.012 Serie de ejercicios 5
 Nombre Grupo 7.012 Serie de ejercicios 5 Pregunta 1 Al estudiar los problemas de esterilidad, usted intenta aislar un hipotético gen de conejo que explique la prolífica reproducción de estos animales.
Nombre Grupo 7.012 Serie de ejercicios 5 Pregunta 1 Al estudiar los problemas de esterilidad, usted intenta aislar un hipotético gen de conejo que explique la prolífica reproducción de estos animales.
ELECTROFORESIS BASICA
 Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
Ref.ELECBASICA (4 prácticas) 1.OBJETIVO DEL EXPERIMENTO ELECTROFORESIS BASICA El objetivo de este experimento es introducir a los alumnos en el conocimiento de la teoría electroforética y familiarizarse
MATERIALES Y MÉTODOS. Cepas Utilizadas
 MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
MATERIALES Y MÉTODOS Cepas Utilizadas Para el presente trabajo se utilizaron tres cepas Tipo: Mycobacterium aviumintracellulare (proporcionada al Laboratorio Estatal de Salud Pública por el Instituto Nacional
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR
 PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
PROBLEMAS MAS FRECUENTES DURANTE LA SECUENCIACIÓN NO SE PRODUCE REACCION O ESTA DECAE POCO DESPUÉS DE COMENZAR Las causas pueden ser las siguientes: No hay ADN o se encuentra en muy baja concentración.
Técnicas de Biología Molecular
 Técnicas de Biología Molecular DNA I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II. Separación electroforética
Técnicas de Biología Molecular DNA I. Enzimas de restricción Análisis Las enzimas de restricción fueron aisladas de bacterias; su función natural es proteger contra DNA extraño II. Separación electroforética
PCR en Tiempo Real. Introducción
 Introducción Página 1 de 6 Introducción general La reacción en cadena de la polimerasa, cuyas iniciales en inglés son PCR ("polymerase chain reaction"), es una técnica que fue desarrollada por Kary Mullis
Introducción Página 1 de 6 Introducción general La reacción en cadena de la polimerasa, cuyas iniciales en inglés son PCR ("polymerase chain reaction"), es una técnica que fue desarrollada por Kary Mullis
INTRODUCCIÓN A LA METODOLOGÍA MOLECULAR. 2002 `Derechos Reservados Pontificia Universidad Javeriana Instituto de Genética Humana Bogotá COLOMBIA
 INTRODUCCIÓN A LA METODOLOGÍA MOLECULAR 2002 `Derechos Reservados Pontificia Universidad Javeriana Instituto de Genética Humana Bogotá COLOMBIA Equipos de Laboratorio Un equipo de laboratorio es un conjunto
INTRODUCCIÓN A LA METODOLOGÍA MOLECULAR 2002 `Derechos Reservados Pontificia Universidad Javeriana Instituto de Genética Humana Bogotá COLOMBIA Equipos de Laboratorio Un equipo de laboratorio es un conjunto
OBTENCIÓN DE MUTANTES
 OBTENCIÓN DE MUTANTES parp::hyg DE Fusarium oxysporum f. sp. lycopersici MEDIANTE LA TÉCNICA PCR DE DOBLE FUSIÓN. Gallegos Almanza I. A. (1) ; Martínez Cadena M. G. (2) ; Ferrel Cano L. E. (2). (1) Facultad
OBTENCIÓN DE MUTANTES parp::hyg DE Fusarium oxysporum f. sp. lycopersici MEDIANTE LA TÉCNICA PCR DE DOBLE FUSIÓN. Gallegos Almanza I. A. (1) ; Martínez Cadena M. G. (2) ; Ferrel Cano L. E. (2). (1) Facultad
GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA
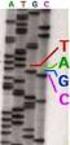 Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
Página 1 de 16 GUÍA PARA LA SECUENCIACIÓN AUTOMÁTICA DE DNA SERVICIO DE SECUENCIACIÓN-S.C.S.I.E. UNIVERSITAT DE VALÈNCIA Página 2 de 16 CONTENIDOS: 1.- Secuenciación automática de DNA 2.- Tipos de molde
Tipos de Muestras. Técnicas de Biología Molecular. Extracción de ácidos nucleicos. Extracción de DNA genómico. Muestra de sangre en papel filtro
 Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Técnicas de Biología Molecular Dr. Luis Salazar N Depto. de Ciencias Básicas / UFRO 2004 Tipos de Muestras Extracción de ácidos nucleicos o DNA o RNA Sangre total (EDTA) Saliva Líquido Amniótico Bulbo
Preguntas de selectividad en Andalucía. Ácidos nucleicos. Análisis e interpretación de imágenes, esquemas, figuras...
 Año 2001 Describa las funciones más relevantes de los nucleótidos. Cite un ejemplo de nucleótido que participe en cada una de ellas [1,5]. Explique las funciones de los distintos tipos de RNA que participan
Año 2001 Describa las funciones más relevantes de los nucleótidos. Cite un ejemplo de nucleótido que participe en cada una de ellas [1,5]. Explique las funciones de los distintos tipos de RNA que participan
5. MATERIAL Y MÉTODOS
 5. MATERIAL Y MÉTODOS 5.1 ANIMALES Se utilizaron setenta ratas Wistar adultas, hembras (Bioterio Claude Bernard, BUAP) con un peso entre 170 y 240 g. Se colocaron en cajas de policarbonato con aserrín
5. MATERIAL Y MÉTODOS 5.1 ANIMALES Se utilizaron setenta ratas Wistar adultas, hembras (Bioterio Claude Bernard, BUAP) con un peso entre 170 y 240 g. Se colocaron en cajas de policarbonato con aserrín
BIOLOGÍA GENERAL Y METODOLOGÍA DE LAS CIENCIAS TRABAJO PRÁCTICO Nº 2 GENÉTICA
 BIOLOGÍA GENERAL Y METODOLOGÍA DE LAS CIENCIAS TRABAJO PRÁCTICO Nº 2 GENÉTICA Objetivos: Diferenciar los niveles de organización y compactación del material genético. Comprender los principios básicos
BIOLOGÍA GENERAL Y METODOLOGÍA DE LAS CIENCIAS TRABAJO PRÁCTICO Nº 2 GENÉTICA Objetivos: Diferenciar los niveles de organización y compactación del material genético. Comprender los principios básicos
DETERMINACIÓN DE GLUTEN EN ALIMENTOS PARA CELÍACOS INDICE
 DETERMINACIÓN DE GLUTEN EN ALIMENTOS PARA CELÍACOS INDICE 1 Objetivo 2 2 Alcance 2 3 Desarrollo 2 4 Anexo 8 1.0. Objetivo Determinación de gluten en alimentos para celíacos. 2.0. Alcance Este método analítico
DETERMINACIÓN DE GLUTEN EN ALIMENTOS PARA CELÍACOS INDICE 1 Objetivo 2 2 Alcance 2 3 Desarrollo 2 4 Anexo 8 1.0. Objetivo Determinación de gluten en alimentos para celíacos. 2.0. Alcance Este método analítico
